微生物降解利用木质纤维素的协同作用
2010-02-09梁朝宁薛燕芬马延和
梁朝宁,薛燕芬,马延和
1 中国科学院微生物研究所 微生物资源国家重点实验室,北京 100101
2 中国科学院研究生院,北京 100049
1 概述
生物质 (Biomass) 主要由一系列作为结构支撑、储能组分的多糖构成。而它的重要组成之一——木质纤维素分布广泛、成本低廉并且在利用过程中对环境的副作用小,因而是生物能源研究领域中颇具潜力的原料。木质纤维素的重要来源是可再生的农业残料 (如秸秆) 和工业废料 (如纸浆废料等)。它的主要组成是纤维素、半纤维素和木质素[1]。纤维素是由葡萄糖单元经 β-1,4-糖苷键连接起来的同源多糖长链。最小的重复单位是纤维二糖。纤维素高级结构中,规则排列的糖链间易形成氢键,从而形成结构严谨、致密的微晶结构。在对里氏木霉Trichoderma reesei纤维素降解酶系研究后,发现天然纤维素降解是由多种酶协同作用完成的。首先,内切葡聚糖酶 (EC 3.2.1.4) 从内部随机切割多糖糖链,从而生成长短不一的短链。随后,外切纤维素酶 (EC 3.2.1.74) 从糖链末端进行水解,生成 D-葡萄糖;或者外切葡聚糖纤维二糖水解酶 (EC 3.2.1.91) 水解葡聚糖生成D-纤维二糖。最后,β-葡萄糖苷酶 (EC 3.2.1.21) 将这些短链纤维素 (尤其是纤维二糖) 水解生成葡萄糖[2]。半纤维素是由几种不同类型的单糖 (包括五碳糖和六碳糖) 构成的异质多聚体。它的含量和组成因来源于不同植物或同一植物的不同部位而有所差别。由于复杂的结构特征,水解利用半纤维素往往需要多种酶共同作用,如木聚糖酶、甘露聚糖酶等。木质素是由聚合的芳香醇构成的一类物质,主要存在于植物的次生壁中,通过与多糖组分 (尤其是半纤维素) 共价结合形成交织网来硬化细胞壁,起抗压作用。木质纤维素结构组成的复杂性,是植物界对抗微生物侵扰的天然屏障,同时也制约了降解、利用这一生物资源的相关技术的开发[3-4]。
为了提高木质纤维素的水解利用、降低生产成本,一种策略是发掘新酶资源或者提高天然酶的性能;但单一酶往往难以实现这一复杂混合物的有效水解,于是另一研究策略是开发利用能通过协同作用、高效降解木质纤维素的体系,包括多酶体系和菌群[4]。这就好比构建一个降解木质纤维素的工作车间,而每一种酶 (菌) 都是车间内的机器。本文概述了现有微生物降解利用木质纤维素体系的研究进展,如纤维小体和在此基础上理性设计的人工纤维小体、菌群共培养、工程菌改造等,展望了这些技术的应用前景,以期实现天然生物质木质纤维素到目的产品乙醇的高效转化。
2 游离酶降解木质纤维素
可降解纤维素的好氧微生物,如好氧真菌 (如里氏木霉) 或绝大多数的好氧细菌 (如 Thermobifida fusca),它们能合成多种纤维素水解酶。在降解木质纤维素底物时,这些酶蛋白在表达顺序、组成比例、水解方式上相互协调,可逐步实现底物的有效降解[5-6]。酶系之间的协同作用包括:内切酶与外切酶的协同作用,外切酶之间的协同作用,以及外切酶与 β-葡糖苷酶的协同作用等。此外,酶系中还存在多功能酶,有些酶由多个不同家族的催化区域构成,如Anaerocellum thermophilum、Caldicellulosiruptor sp.或Paenibacillus polymyxa GS01产生的多功能纤维素酶,同时具备纤维素酶、半纤维素酶活性[2-3];有些酶则具有多个底物结合区 CBM[7],可以与多种底物相结合。多功能酶蛋白分子内的催化单元促进了协同作用,使得水解过程能有序高效进行。关于这一部分内容可参考Lynd等的综述[8],本文不作详述。
3 纤维小体降解木质纤维素
发现于厌氧微生物中的多酶催化体系——纤维小体 (Cellulosome) 具有完备的协同方式。纤维小体是多种纤维素酶、半纤维素酶依靠锚定-粘附机制形成的、能通过细胞粘附蛋白附着在细菌细胞壁上,高效彻底地降解天然纤维素材料的多酶复合体结构。它是由 Bayer等从纤维素降解细菌 Clostridium thermocellum中首次分离得到的[9]。通常,纤维小体由两部分组成:其一是各种具有催化能力且能协同作用的酶类;其二是非催化功能的支架 (Scaffoldin),由于它由多种黏连蛋白 (Cohesin) 模块组成,因此是多种水解酶组合形成复合体的支撑结构[10]。现已在多种微生物中发现纤维小体,它们主要分布于厌氧细菌Clostridium属Cluster Ⅲ中[2],但有研究表明在好氧细菌 (如 Thermobifida fusca) 或一些厌氧真菌 (如 Neocallimastix、Piromyces和 Orpinomyces)中可能也存在类纤维小体结构[11-12]。纤维小体高效降解天然纤维素的 2个关键因素是:第一,结构中包含多个对不同底物具有结合功能的CBM;第二,多个不同家族的酶蛋白在空间位置上邻近,在催化方式上具有协同作用。纤维小体对纤维素类生物质的降解有广阔的工业应用前景,在此基础上,模拟纤维小体的协同作用方式、人工设计构建纤维小体也是世界范围内的研究课题。
3.1 纤维小体的结构研究
为了合理设计人工纤维小体,有必要深入理解纤维小体的构型和组成。Hammel等用X射线散射的方法深入研究了纤维小体的结构。研究表明,纤维小体组装过程与被称为连接区的支架蛋白上的特定区域有关。这一区域的结构柔性有效地克服了相邻黏附蛋白之间的空间位阻效应[13]。Madkour和Mayer用电子显微镜 (Electron microscopy,EM) 观察了C. thermocellum的纤维小体的超微结构,发现在球状颗粒的孔洞中存在的可能是支架蛋白的微丝结构,而催化蛋白则分布于外壳层上[14]。但是Mingardon指出,EM观察中因为使用了固定技术,可能不能真实反映纤维小体的生理结构;在结合并降解包含结晶纤维素和无定形纤维素的异质底物时,纤维小体的拓扑结构可能会发生变化[15]。
3.2 人工设计纤维小体
人工纤维小体的概念1994年由Bayer等提出。它的基本理念是设计构建一个分泌型的纤维素酶类或半纤维素酶类的复合体,通过各酶之间的协同作用,高效降解木质纤维素[16]。为了实现这一设想,通过基因重组技术,不同来源的纤维素酶与来自于纤维小体的锚定蛋白 (Dockerin) 相融合,并组装在能与锚定蛋白互补结合的黏连蛋白构成的人工支架蛋白上形成有机复合体。Murashima等首次实现了体外重组人工纤维小体的合成与组装。他们构建的纤维小体结构包括:催化区域、支架蛋白、CBM区、细胞壁结合区和黏连蛋白[16],见图 1。这一基本结构框架为日后人们设计人工纤维小体提供了依据。
3.2.1 人工纤维小体的设计实例
人工纤维小体技术的优势在于:首先,为了更好地发挥协同作用,来自不同天然纤维小体的酶蛋白可以重新组合;其次,将不同来源、可作用于不同底物的水解酶组合构建的人工纤维小体,有助于扩大底物作用范围。比之天然游离酶,通过合理设计纤维小体、发挥各催化单元之间的协同作用,人工纤维小体在催化能力和底物作用范围等方面都有所改进。

图1 纤维小体结构框架示意图[17]Fig. 1 A simplified schematic of general cellulosome composition[17].
Fierobe等设计合成了一种由 2种纤维素酶(Cel9G和Cel48F) 构成双功能的纤维小体[18]。在此基础上,这一小组又将不同来源的黏连蛋白将 2种纤维素酶和 1种半纤维素酶组合起来。这一三功能人工纤维小体的催化能力较之游离酶提高了7倍[19]。Cha等设计构建的微型人工支架用来自 Clostridium cellulovorans的黏连蛋白将内切葡聚糖酶或木聚糖酶组合起来,使这一复合体对多种纤维素类底物的催化能力较游离酶都有显著提高[20]。同样,Mingardon等将一种真菌来源的纤维素酶插入细菌纤维小体,使得水解活力较之游离酶提高了26倍[11]。
虽然有很多成功的人工纤维小体报道,但并不能把纤维小体简单地理解为纤维素酶类的聚合体。天然纤维小体微妙的结构、组成说明它的催化能力和底物作用范围的设计遵循着一定的内在规律。如Thermobifida fusca的糖苷水解酶家族6的两种纤维素酶的CBM区被替换成锚定蛋白,从而将在天然纤维小体中从未发现过的家族6纤维素酶引入纤维小体,但这一人工纤维小体尽管对不定型纤维素底物的催化能力是游离型酶的14倍,却对可溶底物 (如羧甲基纤维素钠) 的催化能力却有所下降[21]。而用同样方法将糖苷水解酶家族 48的外切纤维素酶插入纤维小体,与游离酶相比,纤维小体对可溶纤维素和结晶纤维素的催化能力都有所提高[22]。这说明,纤维小体各催化单元的选择上具有一定的偏好性;比之糖苷水解酶家族6的纤维素酶,家族48的纤维素酶可能更适于纤维小体的构建。
纤维小体的组成、各组成单元的空间位置等都对这一复合体的协同方式、催化能力有影响。Mingardon等试图通过对纤维小体的两种主要构成酶 (Cel48和Cel9的纤维素酶) 融合CBM区或锚定蛋白来提高纤维小体的催化能力,但是结果表明,改造后的纤维小体虽然对结晶型纤维素的催化能力提高,但是对不定型纤维素的水解能力下降。同时,比之仅包含野生型蛋白的人工纤维小体,改造后、包含不同CBM区或者锚定蛋白的纤维素酶构建的各种纤维小体对结晶纤维素的催化能力均有所下降[15]。对人工纤维小体来说,组成单元的拓扑结构和各组成纤维素酶的可活动范围都对纤维小体的催化能力有重要影响。在另一篇文章中,为了研究催化区域的柔性与催化能力的关系,纤维素酶 Cel5A的催化区域与锚定蛋白由不同长度的连接区相连接后插入纤维小体中后,测试各组合的性质发现:催化能力与连接蛋白的长度无关,而与锚定蛋白与催化中心结合的位置相关[23]。为了研究纤维小体各组成单元之间以及纤维小体酶蛋白与游离酶的相互作用关系,Bayer等在纤维小体组成酶 Cel48和 Cel9构建了一系列包含不同 CBM或锚定蛋白组合的嵌合重组蛋白,但有趣的是,纤维小体构成酶转化为游离酶时仍具有一定协同能力,而反之,大多数游离酶只有在不作改造 (如不增加 CMB区或锚定蛋白),以野生型的形态组装成为纤维小体时才具有协同能力[23-24]。
这些实验说明为了开发高效降解人工纤维小体,研究者仍需要深入研究纤维小体的协同机制和各个组成部分在整个催化体系中发挥的功用。
3.2.2 人造纤维小体的异源合成和组装
人工纤维小体可在多种原核表达系统中合成并组装,生成有催化活性的复合体。
Murashima曾利用胞外蛋白酶缺陷型 Bacillus subtilis WB800合成来自于C. cellulovorans的微型纤维小体[25]。能够产生不具有纤维素酶活性的纤维小体的C.acetobutylicum ATCC 824也被用于来自C. thermocellum和C. cellulolyticum的纤维小体的合成并分泌至胞外[26]。而C. thermocellum的一种甘露聚糖酶和部分支架蛋白也可以C. acetobutylicum作为宿主得到活性表达[27]。
4 纤维素降解细菌共培养
细菌共培养体系也是提高木质纤维素水解、增加产物利用率,从而提高最终目的产物得率的技术。设计一个稳定的共培养体系,不仅要考虑各组成菌的代谢过程中的协同作用,而且需优化培养条件 (如培养基成分、培养温度等) 以维持组成菌群的平衡。
现有的共培养体系多是以仅能发酵六碳糖的C. thermocellum为核心,组合其他嗜热厌氧微生物 (如 Thermoanaerobactium saccharolyticum、Thermoanaerobacter ethanolicus、C. thermohydrosulfuricum、Thermoanaerobium brockii等) 构建[28-30],见图2。菌群在代谢作用上具有协同作用,如纤维素可被C. thermocellum高效水解成纤维二糖,进而发酵成乙醇等目的产物;而半纤维素被C. thermocellum水解生成木二糖等五碳糖产物,随之可被其他组成菌发酵成乙醇等终产物。
共培养体系的优势在于有机结合了多种代谢途径,降低了单一菌发酵产生的有害产物对整个发酵系统的负面影响,同时简化了发酵工艺的操作流程。但是,在木质纤维素的降解中,共培养体系技术仍处于研究初期,菌群之间的协同方式有待进一步研究。
5 其他策略
以上分别介绍了利用酶系或菌系降解木质纤维素的体系,而进一步的研究目标是将这两者有机结合起来,将多酶体系协同作用的策略应用于工业菌株的改造中。利用基因工程技术,将异源的纤维素水解酶类转入工程菌中,一方面可提高天然纤维素降解菌株发酵生成产物的得率,降低产物抑制作用;另一方面,通过引入新酶、利用其在功能上与菌株原有代谢途径的协调,可改造非纤维素水解类菌株,使其直接发酵生产乙醇等目的产物。Kondo的研究小组将里氏木酶的内切葡聚糖酶和纤维二糖酶,以及Aspergillu aculeatus的β-葡萄糖苷酶用表面展示技术在酿酒酵母Saccharomyces cerevisiae的细胞表面表达,改造后菌株可利用不定型纤维素直接发酵生产乙醇[31]。同样,将木聚糖酶[32]或淀粉酶[33]在酿酒酵母的细胞表面表达后,改造菌株可发酵利用木聚糖或淀粉类底物合成乙醇。这一技术将纤维素降解酶类生产与工程菌相结合,实现了一步转化木质纤维素底物为目的产品乙醇,有利于提高生产效率,降低生产成本,是颇具应用潜力的新型技术。
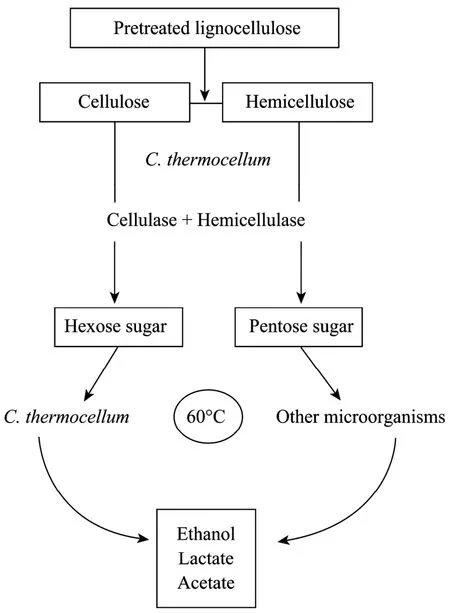
图2 以C. thermocellum为中心、降解木质纤维素共培养体系示意图[17]Fig. 2 Simplified process using C. thermocellum and other microorganisms in co-culture for degradation of lignocellulose[17].
6 小结
木质纤维素作为植物界抵抗微生物侵扰的天然屏障,结构复杂,组成多样。单一功能的酶往往无法胜任木质纤维素的降解,难以实现对它的充分利用。为了实现这一天然生物资源的高效降解,研究者提出联合生物加工技术 (Consolidated bioprocessing,CBP) 的概念,将纤维素酶生产、纤维素水解和乙醇发酵结合起来,旨在实现一步发酵完成木质纤维素到生物乙醇的转化[34]。而多酶 (菌) 体系可通过纤维素降解酶或代谢途径之间的协同作用,能以“加工工厂”的形式、以群体协同作用的方式实现这一天然多聚物的有效降解。为了实现这一目的,有必要对多酶体系各组成单元的协同作用机理进行深入的研究,比如酶单元的催化中心与底物结合区之间的关系,酶与底物之间的关系,纤维小体中各成员的空间位置、结构组成对有效降解底物 (尤其是结晶纤维素) 的影响,共培养体系中各菌群代谢之间的协同方式等。基于这些研究,人工设计构建的多酶体系 (如人工纤维小体)、多菌体系 (如共培养)以及将酶和菌株有机结合的体系 (工程菌代谢网络改造) 将有助于研究者认识纤维素降解的有机体系,在生物炼制细胞工厂中发挥作用。
REFERENCES
[1] Himmel ME, Ding SY, Johnson DK, et al. Biomass recalcitrance: engineering plants and enzymes for biofuels production. Science, 2007, 315(5813): 804−807.
[2] Schwarz WH. The cellulosome and cellulose degradation by anaerobic bacteria. Appl Microbiol Biotechnol, 2001, 56(5): 634−649.
[3] Wingren A, Galbe M, Zacchi G. Techno-economic evaluation of producing ethanol from softwood: comparison of SSF and SHF and identification of bottlenecks. Biotechnol Prog, 2003, 19: 1109−1117.
[4] Ding SY, Xu Q, Crowley M, et al. A biophysical perspective on the cellulosome: new opportunities for biomass conversion. Curr Opin Biotechnol, 2008, 19(3): 218−227.
[5] József M, Johan K, Dora L, et al. Hydrolysis of microcrystalline cellulose by cellobiohydrolase I and endoglucanase II from Trichoderma reesei: adsorption, sugar production pattern, and synergism of the enzymes. Biotechnol Bioeng, 1998, 59(5): 621−634.
[6] Watson D, Wilson D, Walker L. Synergism in binary mixtures of Thermobifida fusca cellulases Cel6B, Cel9A, and Cel5A on BMCC and Avicel. Appl Biochem Biotechnol, 2002, 101(2): 97−111.
[7] Cai S, Li J, Hu FZ, et al. Cellulosilyticum ruminicola, a newly described rumen bacterium that possesses redundant fibrolytic genes in the genome and degrades lignocellulose by multiple CBM-borne fibrolytic enzymes. Appl Environ Microbiol, 2010: 3124−3109.
[8] Lynd LR, Weimer PJ, van Zyl WH, et al. Microbial cellulose utilization: fundamentals and biotechnology. Microbiol Mol Biol Rev, 2002, 66(3): 506−577.
[9] Bayer EA, Kenig R, Lamed R. Adherence of Clostridium thermocellum to cellulose. J Bacteriol, 1983, 156(2): 818−827.
[10] Bayer EA, Shimon LJW, Shoham Y, et al. Cellulosomesstructure and ultrastructure. J Struct Biol, 1998, 124(2/3): 221−234.
[11] Mingardon F, Chanal A, Lopez-Contreras AM, et al. Incorporation of fungal cellulases in bacterial minicellulosomes yields viable, synergistically acting cellulolytic complexes. Appl Environ Microbiol, 2007, 73(12): 3822−3832.
[12] Li XL, Chen H, Ljungdahl LG. Two cellulases, CelA and CelC, from the polycentric anaerobic fungus Orpinomyces strain PC-2 contain N-terminal docking domains for a cellulase-hemicellulase complex. Appl Environ Microbiol, 1997, 63(12): 4721−4728.
[13] Hammel M, Fierobe H-P, Czjzek M, et al. Structural basis of cellulosome efficiency explored by small angle X-ray scattering. J Biol Chem, 2005, 280(46): 38562−38568.
[14] Madkour M, Mayer F. Structural organization of the intact bacterial cellulosome as revealed by electron microscopy. Cell Biol Int, 2003, 27(10): 831−836.
[15] Mingardon F, Chanal A, Tardif C, et al. Exploration of new geometries in cellulosome-like chimeras. Appl Environ Microbiol, 2007, 73(22): 7138−7149.
[16] Bayer EA, Morag E, Lamed R. The cellulosome-a treasure-trove for biotechnology. Trends Biotechnol, 1994, 12(9): 379−386.
[17] Maki M, Leung KT, Qin W. The prospects of cellulase-producing bacteria for the bioconversion of lignocellulosic biomass. Int J Biol Sci, 2009, 5: 500−516.
[18] Fierobe HP, Bayer EA, Tardif C, et al. Degradation of cellulose substrates by cellulosome chimeras. J Biol Chem, 2002, 277(51): 49621−49630.
[19] Fierobe HP, Mingardon F, Mechaly A, et al. Action of designer cellulosomes on homogeneous versus complex substrates. J Biol Chem, 2005, 280(16): 16325−16334.
[20] Cha J, Matsuoka S, Chan H, et al. Effect of multiple copies of cohesins on cellulase and hemicellulase activities of Clostridium cellulovorans mini-cellulosomes. J Microbiol Biotechnol, 2007, 17(11): 1782−1788.
[21] Caspi J, Irwin D, Lamed R, et al. Thermobifida fusca family-6 cellulases as potential designer cellulosome components. Biocatal Biotrans, 2006, 24(1): 3−12.
[22] Caspi J, Irwin D, Lamed R, et al. Conversion of Thermobifida fusca free exoglucanases into cellulosomal components: comparative impact on cellulose-degrading activity. J Biotechnol, 2008, 135(4): 351−357.
[23] Caspi J, Barak Y, Haimovitz R, et al. Effect of linker length and dockerin position on conversion of a Thermobifida fusca endoglucanase to the cellulosomal mode. Appl Environ Microbiol, 2009, 75(23): 7335−7342.
[24] Vazana Y, Morais S, Barak Y, et al. Interplay between Clostridium thermocellum family-48 and family-9 cellulases in the cellulosomal versus non-cellulosomal states. Appl Environ Microbiol, 2010, 76(10): 3236−3243.
[25] Murashima K, Chen CL, Kosugi A, et al. Heterologous production of Clostridium cellulovorans engB, using protease-deficient Bacillus subtilis, and preparation of active recombinant cellulosomes. J Bacteriol, 2002, 184(1): 76−81.
[26] Perret S, Casalot L, Fierobe HP, et al. Production of heterologous and chimeric scaffoldins by Clostridium acetobutylicum ATCC 824. J Bacteriol, 2004, 186(1): 253−257.
[27] Perret S, Belaich A, Fierobe HP, et al. Towards designer cellulosomes in Clostridia: mannanase enrichment of the cellulosomes produced by Clostridium cellulolyticum. J Bacteriol, 2004, 186(19): 6544−6552.
[28] Wiegel J. Formation of ethanol by bacteria. A pledge for the use of extreme thermophilic anaerobic bacteria in industrial ethanol fermentation processes. Cell Mol Life Sci, 1980, 36(12): 1434−1446.
[29] Lamed R, Zeikus JG. Ethanol production by thermophilic bacteria: relationship between fermentation product yields of and catabolic enzyme activities in Clostridium thermocellum and Thermoanaerobium brockii. J Bacteriol, 1980, 144(2): 569−578.
[30] Ng TK, Ben-Bassat A, Zeikus JG. Ethanol production by thermophilic bacteria: fermentation of cellulosic substrates by cocultures of Clostridium thermocellum and Clostridium thermohydrosulfuricum. Appl Environ Microbiol, 1981, 41(6): 1337−1343.
[31] Fujita Y, Ito J, Ueda M, et al. Synergistic saccharification, and direct fermentation to ethanol, of amorphous cellulose by use of an engineered yeast strain codisplaying three types of cellulolytic enzyme. Appl Environ Microbiol, 2004, 70(2): 1207−1212.
[32] Katahira S, Fujita Y, Mizuike A, et al. Construction of a xylan-fermenting yeast strain through codisplay of xylanolytic enzymes on the surface of xylose-utilizing Saccharomyces cerevisiae cells. Appl Environ Microbiol, 2004, 70(9): 5407−5414.
[33] Shigechi H, Koh J, Fujita Y, et al. Direct production of ethanol from raw corn starch via fermentation by use of a novel surface-engineered yeast strain codisplaying glucoamylase and α-amylase. Appl Environ Microbiol, 2004, 70(8): 5037−5040.
[34] Lynd LR, Zyl WHv, McBride JE, et al. Consolidated bioprocessing of cellulosic biomass: an update. Curr Opin Biotechnol, 2005, 16(5): 577−583.
