蓖麻bZIP基因家族的全基因组鉴定、生物信息学及表达分析
2025-01-28王冬艳张春玲冯研王丽新昝睿狄建军




摘要" 为了解蓖麻bZIP基因家族成员的组成和家族进化关系及特征,本研究利用生物信息学鉴定家族成员,进行基因结构、蛋白质保守基序分析及系统发育分析,同时分析其蛋白的理化性质、磷酸化位点和互作关系,以及该基因在组织中的表达模式和表达量。结果表明,共鉴定出46个蓖麻bZIP基因家族成员,位于10个染色体上;基因结构与蛋白质保守基序较为复杂,可分为11个亚族,包含25个基序;与拟南芥进化相似,较为保守;bZIP蛋白为亲水性蛋白,位于细胞核、细胞膜和叶绿体,主要通过丝氨酸的磷酸化来发挥生物学作用,蛋白RcbZIP18、RcbZIP27、RcbZIP2、RcbZIP17和RcbZIP46之间的联系较多,通过蛋白间的相互作用参与蓖麻各个生长时期;该基因家族均参与叶、茎、根和胚等生长时期的发育调控,其中RcbZIP10、RcbZIP25和RcbZIP44在叶片和种子中表现出明显的组织差异性。上述结果为蓖麻bZIP基因家族成员的功能研究提供参考。
关键词" 蓖麻;bZIP家族;生物信息学分析;系统进化;表达分析
中图分类号" S565.6" " " "文献标识码" A" " " "文章编号" 1007-7731(2025)02-0027-09
DOI号" 10.16377/j.cnki.issn1007-7731.2025.02.006
Genome-wide identification, bioinformatics and expression analysis of bZIP gene family of Ricinus communis
WANG Dongyan1" " ZHANG Chunling2" " FENG Yan1" " WANG Lixin1" " ZAN Rui1" " DI Jianjun1,3,4,5,6
(1College of Life Science and Food Engineering, Inner Mongolia Minzu University, Tongliao 028000, China;
2Tongliao Agricultural and Animal Husbandry Academy, Tongliao 028015, China;
3Inner Mongolia Key Laboratory of Castor Breeding and Comprehensive Utilization, Tongliao 028000, China;
4Key Laboratory of Castor Breeding of the State Et hnic Affairs Commission, Tongliao 028000, China;
5Inner Mongolia Industrial Engineering Research Center of Universities for Castor, Tongliao 028000, China;
6Inner Mongolia Engineering Research Center of Industrial Technology Innovation of Castor, Tongliao 028000, China)
Abstract" To understand the composition, evolutionary relationship and characteristics of Ricinus communis bZIP gene family members, bioinformatics was used to identify family members, analyze gene structure, protein conserved motifs, phylogenetic analysis, and analysis of protein physicochemical properties, phosphorylation sites, and interactions, as well as gene expression patterns and expression levels in tissues. The results showed that 46 members of the Ricinus communis bZIP gene family were identified, which were located on 10 chromosomes.The gene structure and protein conserved motifs were complex, which can be divided into 11 subfamilies, including 25 motifs. Similar to Arabidopsis, the evolution was more conservative; bZIP protein was a hydrophilic protein located in the nucleus, cell membrane and chloroplast. It played a biological role mainly through the phosphorylation of serine.The proteins RcbZIP18, RcbZIP27, RcbZIP2, RcbZIP17 and RcbZIP46 were associated with each other, participating in each growth cycle of Ricinus communis through the interaction between proteins. They are involved in the developmental regulation of leaf, stem, root and embryo during growth,RcbZIP10, RcbZIP25 and RcbZIP44 showed significant organizational differences in leaves and seeds These results provide references for the functional study of Ricinus communis bZIP gene family members .
Keywords" Ricinus communis L.; bZIP family; bioinformatics analysis; phylogeny; expression analysis
转录因子(Transcription factor,TF)是一类在特定的时间与空间下,目的基因与特定的序列进行专一性结合的蛋白质分子[1]。其作为细胞过程(如分化和发育)的关键调节因子,通过信号通路对外部干扰做出反应,在植物生长发育、代谢反应和抗逆反应等多种过程中发挥重要作用[2-3]。碱性亮氨酸拉链(Basic leucine zipper,bZIP)是真核生物中较为熟知的转录因子家族之一[4]。bZIP基因家族广泛存在于植物中,在多种生物过程中起着重要作用,如孙耀国等[5]研究表明,西洋梨的bZIP基因家族参与该植物的生长发育、种子成熟、光信号响应和环境胁迫等过程;曹丽茹等[6]研究发现,玉米bZIP转录因子ZmbZIP26在多个部位表达,且在氮胁迫和高盐条件下表达量上调20倍左右,在高温和干旱胁迫下表达量也呈上调趋势;潘婷婷[7]研究表明,在水稻中共鉴定出89个bZIP基因,其中调控水稻开花的新基因OsbZIP65在多种部位表达,在根、茎和穗中表达量较高,在叶片等部位表达量较低。游冉[8]研究发现,过表达OsbZIP34基因能够延迟植物开花,并参与调控水稻抽穗。目前,在野菊、水稻、玉米、大豆和睡莲等多种植物中,已有bZIP转录因子的基因组鉴定及生物学分析[9],但关于bZIP转录因子在蓖麻(Ricinus communis)中的分子进化研究相对较少。
蓖麻是大戟科油料植物[10],其种子含油量较高,蓖麻油中富含的蓖麻油酸在生活与工业中有着广泛的用途[11-12],为再生能源提供优质原料[13]。蓖麻油及其衍生物可代替部分石油产品,被称为能够再生的“绿色石油”[14]。然而,受到环境和种植效益的影响,蓖麻产量逐年下降,使产品供不应求。因此,利用基因工程和生物技术改良蓖麻的产量与性状对生产具有重要意义[15]。
本研究主要通过生物信息学技术对蓖麻bZIP转录因子家族的蛋白和基因的保守结构、系统进化及其基因表达模式等进行分析,以了解bZIP转录因子家族在蓖麻中的进化过程[16],为蓖麻bZIP转录因子家族在油脂代谢调控机制中的功能研究提供参考,有利于提高蓖麻育种效率和培育高产优质抗逆品种,对蓖麻稳产增产有重要作用。
1 材料与方法
1.1 数据来源
使用的蓖麻全基因组序列、编码序列、蛋白质序列和注释文件均下载自网站Oil plant database(https://oilplants.iflora.cn),蓖麻基因组文件下载自蓖麻基因组数据库 (https://castorbean.jcvi.org./index.php),拟南芥bZIP基因家族的基因与蛋白质序列下载自TAIR数据库(Araport11,https://www.arabidopsis.org/)[17],蓖麻的各组织相关表达量数据下载自GEO数据库(https://www.ncbi.nlm.nih.gov)。
1.2 家族成员鉴定
下载蓖麻的蛋白质序列和70个拟南芥bZIP基因家族成员序列,然后进行本地BLASTP比对(E-value≤1e-10)[18]。再从Pfam数据库(http://pfam.xfam.org/)下载bZIP基因家族典型结构域的蛋白质序列,进行本地HMMER比对[19]。采用这两种方法进行家族成员的鉴定,将比对结果进行整合,删除多余的重复序列。分别使用NCBI CDD工具(https://www.ncbi.nlm.nih.gov/cdd/)与EBI InterPro工具(https://www.ebi.ac.uk/interpro/)对候选序列进行结构域检测[20-21],使用Clustal Omega软件进行多重比对[22-23],从而确定蓖麻bZIP基因家族的成员。
1.3 基因结构与蛋白质保守基序分析
根据下载的蓖麻基因组注释文件编写脚本,获得bZIP基因家族成员的基因、内含子与外显子位点等信息。使用MEME在线工具(https://meme-suite.org/tools/meme)对蓖麻bZIP蛋白进行保守基序分析[24],基序数目设置为10个。使用Evoview在线工具对基因结构、蛋白质保守基序进行可视化分析[25]。
1.4 系统发育进化分析
在植物转录因子数据库(http://planttfdb.gao-lab.org/)下载模式植物拟南芥bZIP转录因子的家族成员基因序列。运用MEGA7软件对拟南芥和蓖麻bZIP基因家族成员采用Clustal W方法进行多序列比对[26]。选用相邻连接法(Neighbor-joining,NJ),设定Bootstrap值1 000进行校正,构建进化树。导出Newick Export文件。用EvolView(https://evolgenius.info//evolview-v2/)进行进化树美化[27]。
1.5 蛋白的理化性质分析
利用在线工具ExPASy的ProtParam工具(https://web.expasy.org/protparam/)对基因家族成员蛋白的氨基酸数目、分子量、理论等电点和亲水性等理化性质进行分析[28]。在Plant-mPLoc[25]中进行蛋白质的亚细胞定位分析。
1.6 磷酸位点预测
利用在线网站NetPhos3.1(NetPhos 3.1-DTU Health Tech -Bioinformatic Services /netphos-3.1/)对潜在的蛋白磷酸化位点数量进行预测[29],主要为苏氨酸(Threonine,Thr)、酪氨酸(Tyrosine,Tyr)和丝氨酸(Serine,Ser)。
1.7 蛋白互作网络分析
参考蓖麻蛋白质数据库,利用在线网站String (https://cn.string-db.org/)对蓖麻bZIP家族成员蛋白进行蛋白质网络互作建模,从而预测该基因家族蛋白质间的相互作用[30]。
1.8 表达模式分析
在NCBI GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)获取蓖麻在Ⅱ/Ⅲ期胚乳、Ⅴ/Ⅵ期胚乳、萌芽种子、叶片和雄花5个组织中基因的表达量[28]。得到的reads count 值进行FPKM标准化处理。对蓖麻的bZIP基因家族进行层次聚类,使用R4.1.0软件(https://www.r-project.org/)的Pheatmap包绘制基因表达量热图。
1.9 荧光定量分析
选择具有代表性的差异基因,测定其在蓖麻种子和叶片中的表达量。提取蓖麻种子和叶片RNA,使用反转录试剂盒PrimeScript TMRT Master Mix(TaKaRa)反转录合成cDNA。用TB Green Premix Ex TaqTM Ⅱ试剂(TaKaRa)进行荧光定量PCR试验,待测样本设3个重复。参照2-ΔΔCt方法计算bZIP在蓖麻不同组织基因的相对表达量。
2 结果与分析
2.1 家族成员鉴定
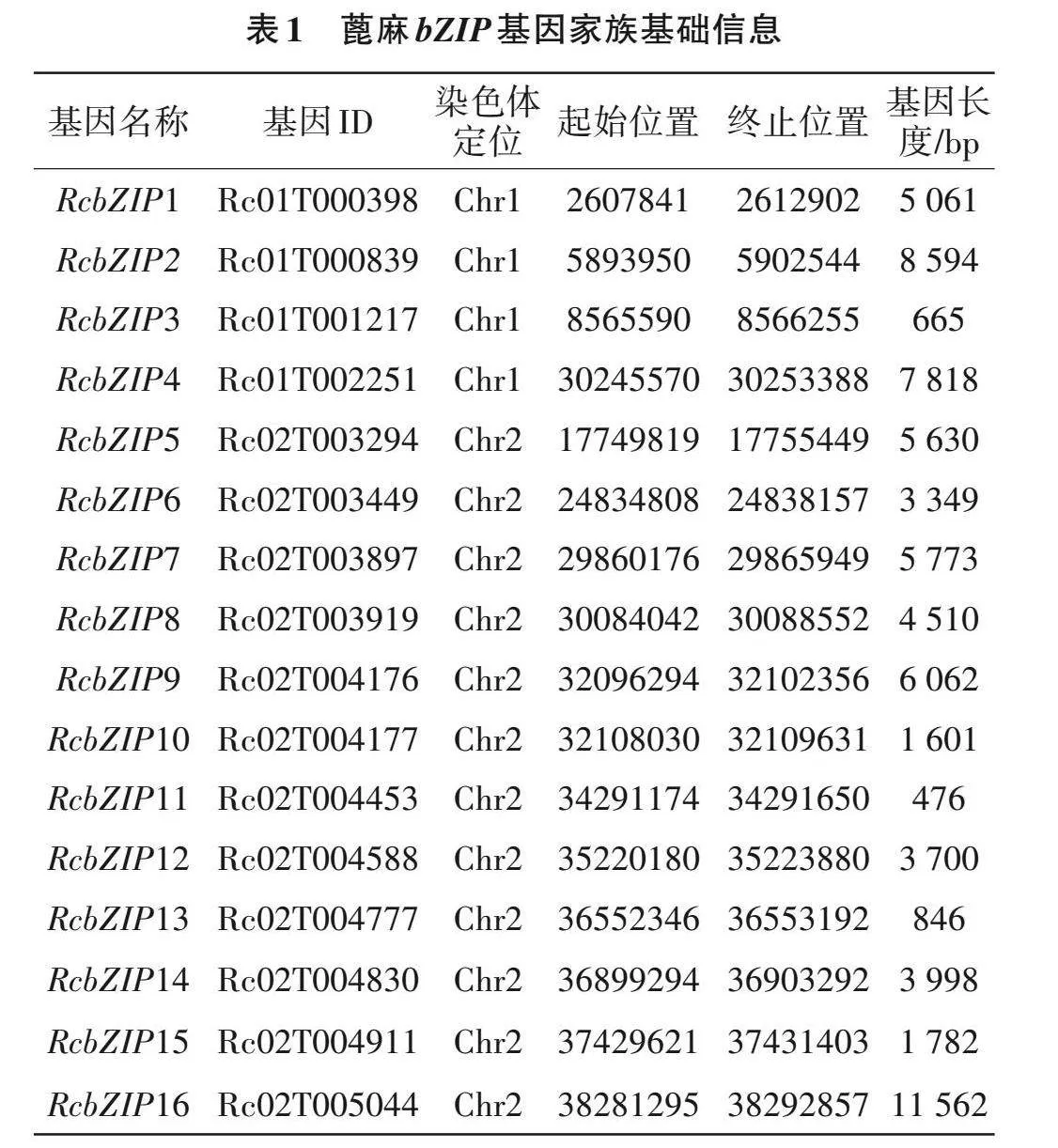
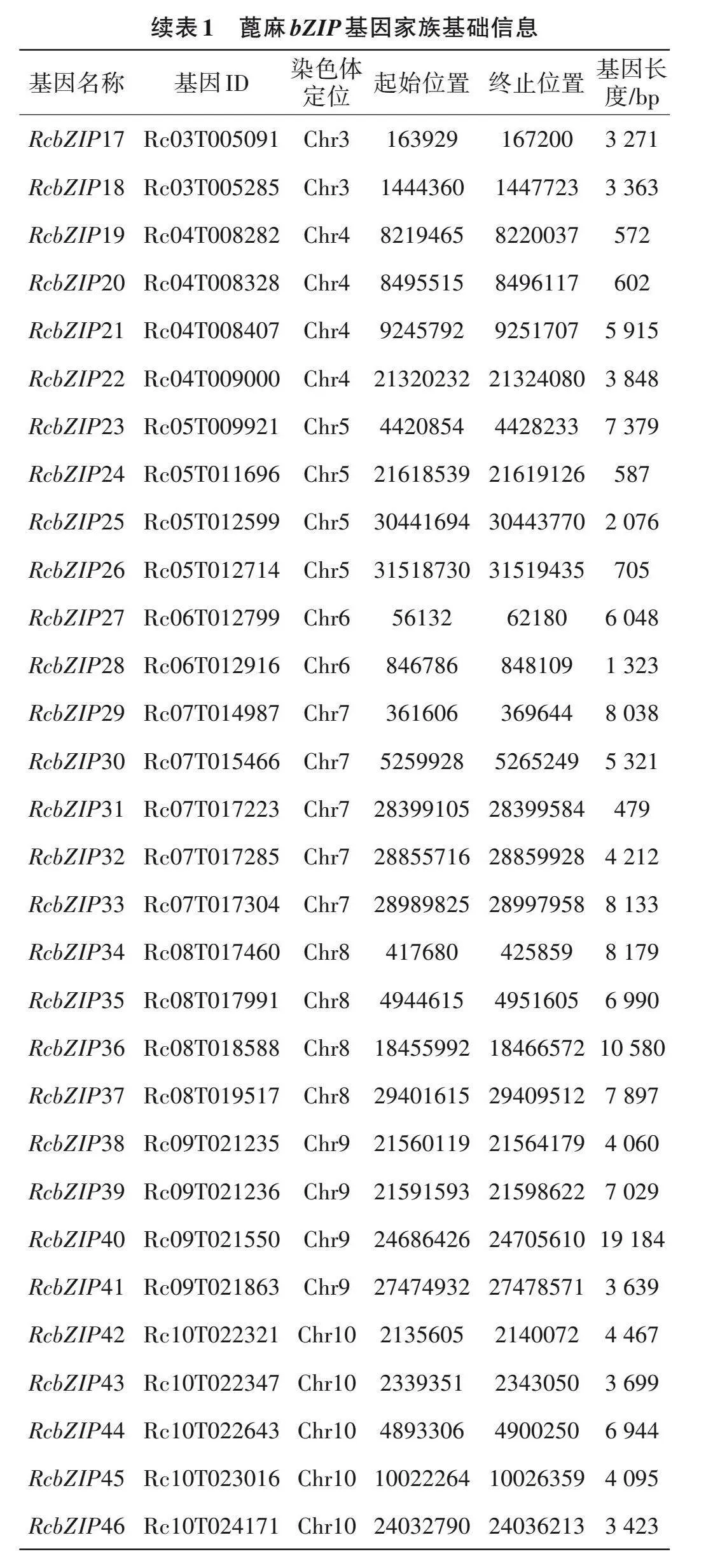
由表1可知,通过比对鉴定共得到46个蓖麻bZIP基因家族成员,按照基因在染色体上的定位,命名为RcbZIP1~RcbZIP46,基因长度在476~19 184 bp。对蓖麻基因组注释文件进行分析,发现46个蓖麻bZIP基因分布在10条染色体上。其中,2号染色体上分布的bZIP基因最多,有12个;3和6号染色体分布较少,各2个基因;1、4、5、8和9号染色体上均分布4个基因;7和10号染色体上各分布5个基因。
2.2 蓖麻bZIP基因结构与蛋白质保守基序
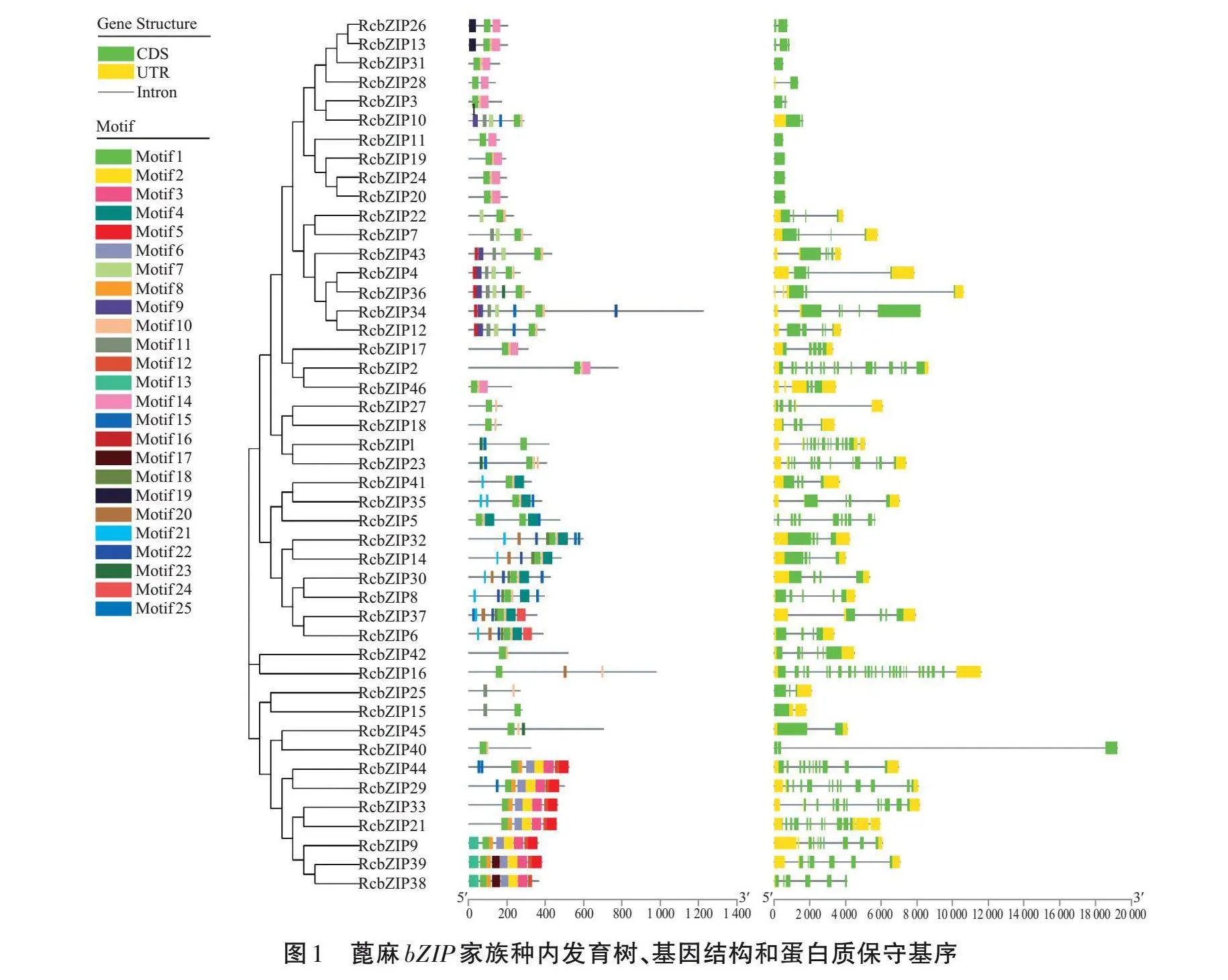
由表2可知,46个蓖麻bZIP基因家族成员分为11个亚族,第Ⅶ亚组包含了9个基因,是包含基因最多的亚族,Ⅲ组、Ⅷ组、Ⅸ组和Ⅹ组bZIP基因最少,各组仅有2个基因。除了Ⅰ组和Ⅱ组的部分基因无内含子外,其余bZIP基因家族成员均含有内含子和外显子,内含子数0~25个,外显子数1~26个;其中,Ⅱ组和Ⅲ组组内基因内含子数和外显子数完全一致。
基序是介于生物大分子二级和三级结构之间的一种结构层次,也被称为模体(Motif)[31]。11个亚族共25个Motif,单个成员含有的Motif范围在2~10个,其中Motif1为所有基因共有,经比较发现,Motif1是典型的类锌指结构CX2CX6CX3C,是bZIP基因家族的标志;Ⅰ~Ⅴ组内家族成员同时具有Motif1和Motif3,Ⅵ~Ⅺ组内家族成员同时具有Motif1和Motif2;其中,Ⅴ组组内家族成员Motif种类及排布完全一致,Ⅱ组除了bZIP11外,其他家族成员Motif种类及排布完全一致,各组内家族成员Motif种类差别较小。表明蓖麻bZIP家族的蛋白质保守基序结构较复杂,但与对应的基因结构基本一致(图1)。
2.3 系统发育进化树
由图2可知,蓖麻转录因子保守结构域和系统进化树将进化树分为5个亚族,蓖麻转录因子家族成员在各个亚族中呈不均匀分布。Ⅴ组中含有蓖麻bZIP基因最多为14个,Ⅰ、Ⅱ、Ⅲ和IV组中分别含有9、8、6和9个蓖麻bZIP基因。蓖麻和拟南芥的bZIP基因在多个分支中均有分布,表明二者可能具有相似的进化方式。
2.4 蛋白质理化性质
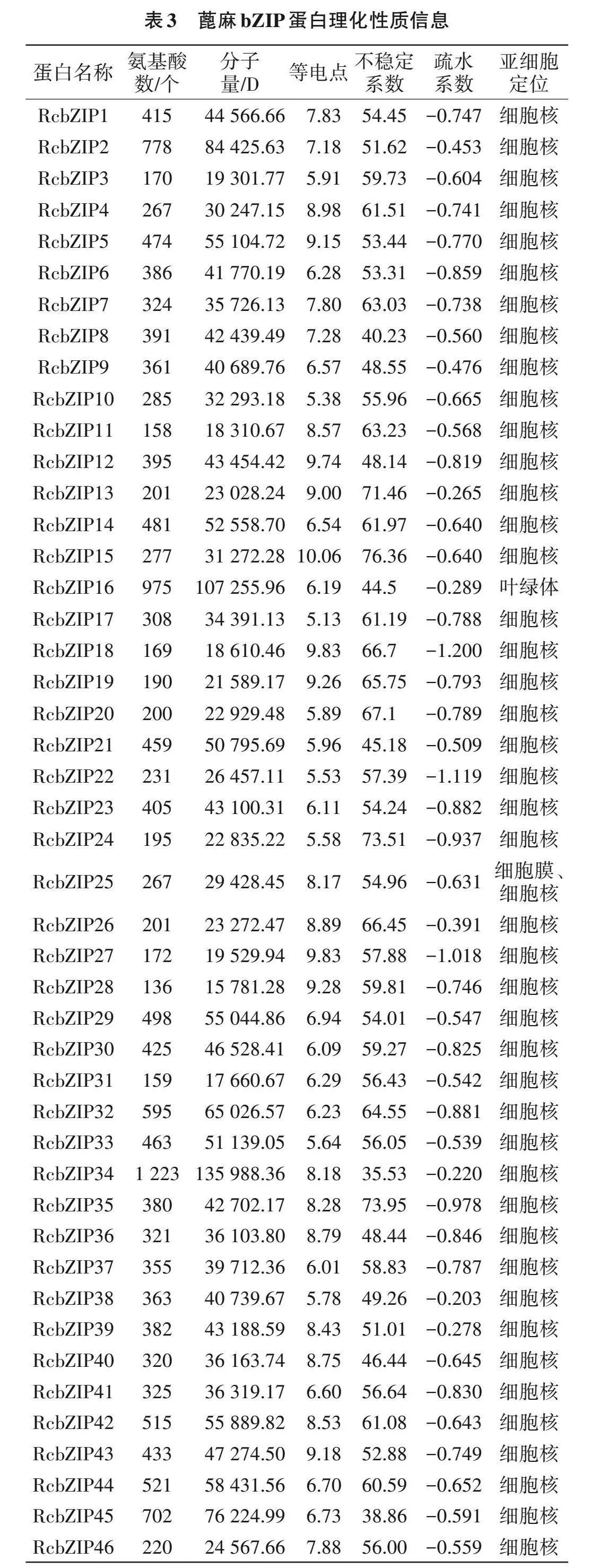
蛋白质理化性质方面,46个蓖麻bZIP蛋白的氨基酸数目和分子量有较大差异,其中有9个蛋白的氨基酸数小于200,分子量较小;RcbZIP28只有136个氨基酸,分子量最小,为15 781.28 D;RcbZIP34则有1 223个氨基酸,分子量最高,为135 988.36 D。RcbZIP17等电点最低,为5.13;RcbZIP15等电点最高,为10.06。另外,46个蓖麻bZIP蛋白的疏水系数均为负值,即蓖麻bZIP蛋白均为亲水性蛋白。在亚细胞定位中,除了RcbZIP16位于叶绿体,其余45个蓖麻bZIP蛋白全部分布于细胞核内,而RcbZIP25在细胞核内和细胞膜中均含有(表3)。
2.5 蛋白磷酸化位点预测
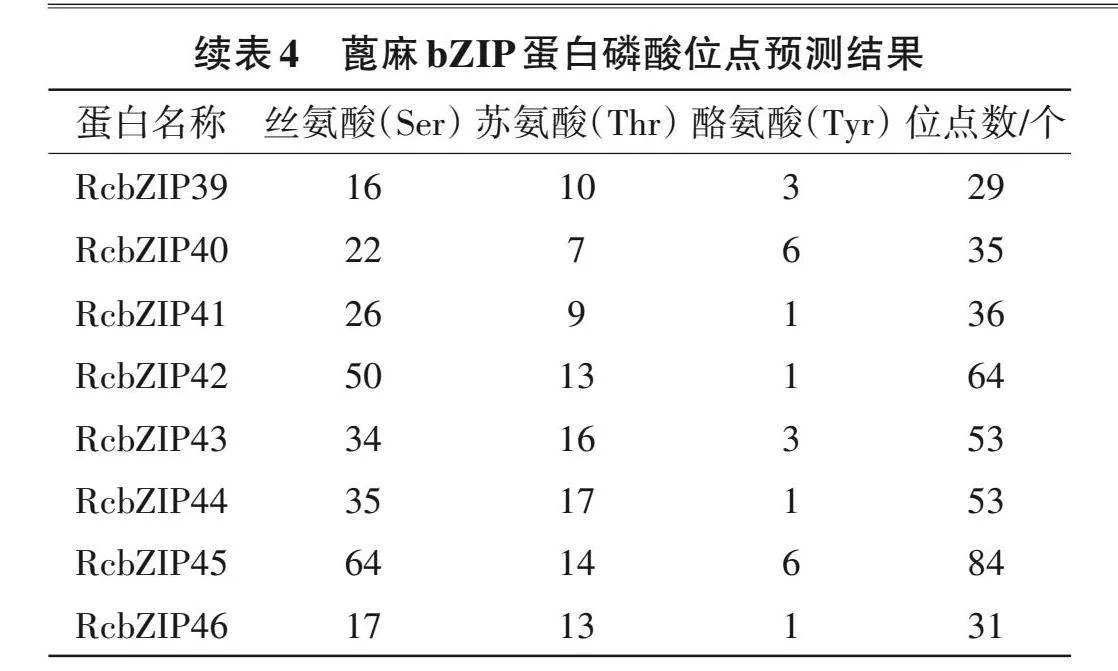
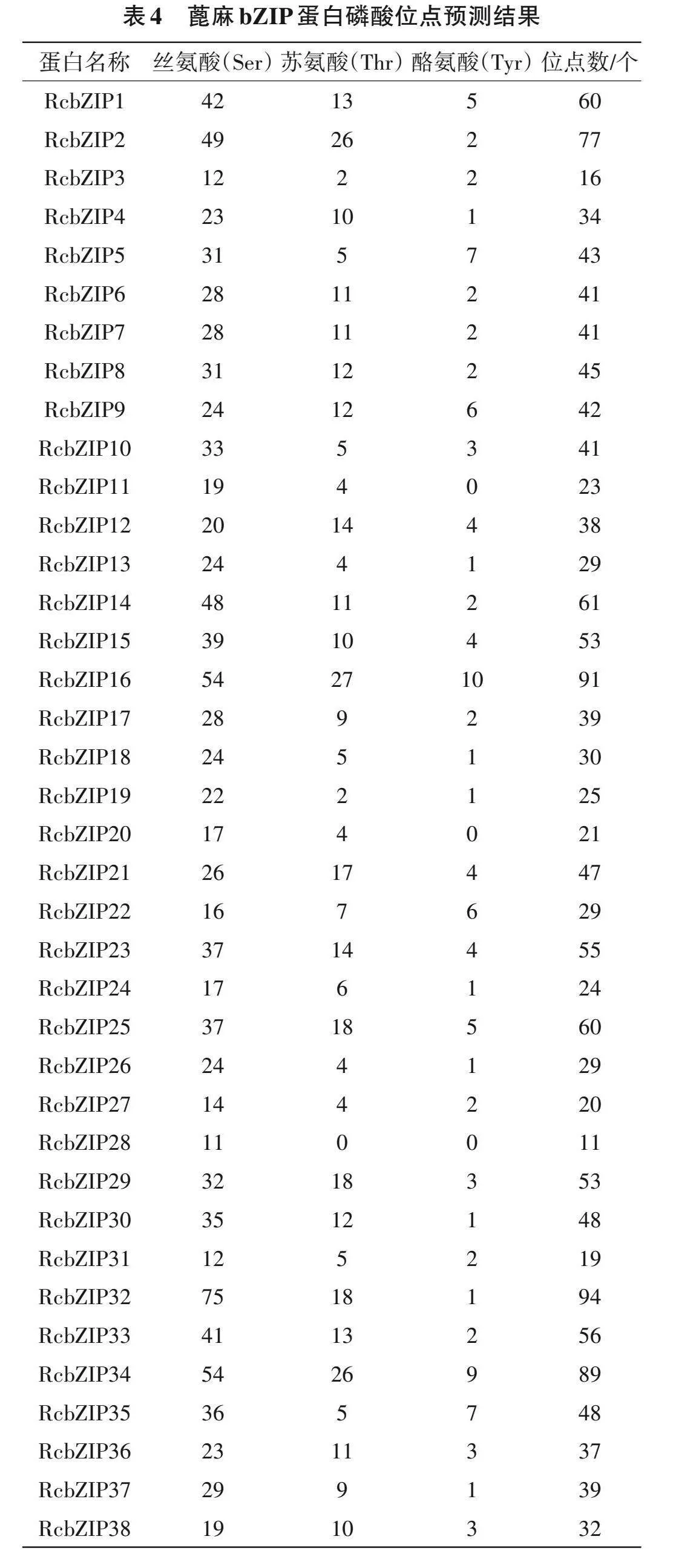
由表4可知,各基因家族成员的磷酸化位点均是丝氨酸(Ser)占比最大,且其磷酸位点数量明显高于苏氨酸(Thr)和酪氨酸(Tyr)。其中bZIP32具有94个磷酸化位点,而RcbZIP28磷酸化位点最少,仅11个。RcbZIP32有最多的丝氨酸(Ser)位点,为75个,RcbZIP16有最多的苏氨酸(Thr)位点,为27个。说明RcbZIP家族可能主要通过丝氨酸的磷酸化来发挥生物学作用。
2.6 蛋白互作网络分析
由图3可知,在bZIP蛋白基因家族中,各家族成员相互作用,其中以RcbZIP18、RcbZIP27、RcbZIP2、RcbZIP17和RcbZIP46之间的联系较多,其他蛋白之间也相互联系。
2.7 蓖麻bZIP基因家族表达模式
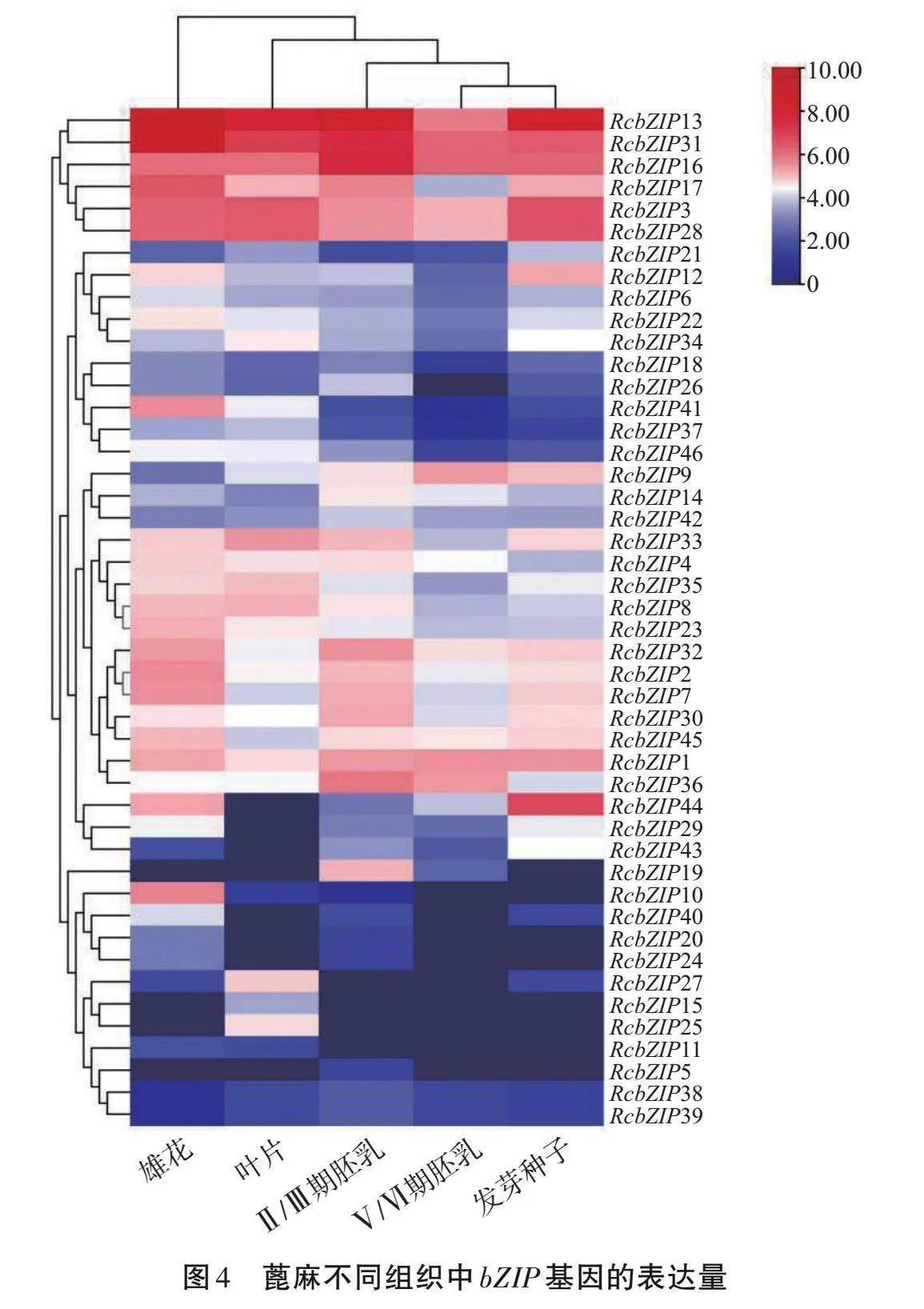
在GEO数据库中下载蓖麻各个组织的bZIP基因表达量数据,其结果见图4。蓖麻bZIP基因在不同组织中的表达量差异较大,具有组织特异性。其中多数的蓖麻bZIP基因仅在特定的组织部位中高表达,例如RcbZIP13、RcbZIP31、RcbZIP16、RcbZIP3和RcbZIP28在蓖麻各个生长时期的表达量较高,而RcbZIP20、RcbZIP24、RcbZIP15、RcbZIP11、RcbZIP5、RcbZIP38和RcbZIP39则在蓖麻各个生长时期的表达量均相对较低;其中,RcbZIP3和RcbZIP28,以及RcbZIP38和RcbZIP39在蓖麻各个生长时期表达量基本一致。
2.8 bZIP基因的荧光定量表达
选择具有代表性的差异基因,在蓖麻种子和叶片中测定表达量,结果如图5所示。4个基因的荧光定量表达量与GEO数据库表达量大致相同。RcbZIP13在叶片和种子中均为高表达,而RcbZIP10、RcbZIP25和RcbZIP44在叶片和种子中具有组织差异性表达。RcbZIP44基因在种子中的表达量显著高于在叶片中的表达量。
3" 结论与讨论
植物中bZIP转录因子家族成员较多,是分布较广且高度保守的转录因子,参与植物的生长调控过程。安爽等[32]研究发现,不同基因bZIP在蓝莓不同组织或是不同pH条件下表达模式有差异,具有抗逆调控作用;靳正伟等[33]通过对蓖麻bZIP基因家族成员在不同组织和种子发育阶段的表达规律的研究,鉴别出RcbZIP8、RcbZIP31和RcbZIP40基因的表达可能与种子油脂累积具有较强的相关性。本研究在蓖麻全基因组水平上共鉴定了46个bZIP转录因子家族成员。种内系统发育树显示,根据基因结构和蛋白质保守基序,46个RcbZIP基因被分为11个亚族,这11个亚族的蛋白质保守基序结构较复杂,但与对应的基因结构基本一致,其中外显子与内含子结构与分布,可能携带着基因家族的进化痕迹。蓖麻和拟南芥种间系统发育树表明,RcbZIP基因家族成员与拟南芥家族成员在进化上相似,均较为保守。通过基因的组织表达分析可以直观了解其功能,本研究发现,基因在组织中的表达量具有明显的组织特异性。多数蓖麻bZIP基因家族成员在蓖麻发育过程均存在较高的表达,推测该基因家族在蓖麻发育过程中发挥了重要的作用。蓖麻是重要的油料作物,胚乳是其油脂积累的重要部位,部分bZIP基因在胚组织中的表达量低,推测这些基因与油脂积累无关,或是油脂积累的负调控因子。部分基因在胚组织中的表达量较高,推测这些基因可能与蓖麻油脂积累与代谢通路密切相关,可能是甘油三酯合成代谢通路中的关键转录调控因子或关键酶,在油脂形成与累积过程中发挥关键作用。蛋白磷酸化分析结果显示,丝氨酸是该基因家族的主要磷酸化位点,预测丝氨酸的磷酸化修饰对蓖麻bZIP基因家族的生物功能起着至关重要的作用。同时,该基因家族成员之间相互作用,进而参与蓖麻的各个生长时期。
综上,本研究在全基因组水平上共筛选与鉴定出了46个蓖麻bZIP基因家族成员,采用生物信息学手段对其进行分析,发现46个蓖麻bZIP基因家族成员,可分为11个亚族,进化保守,位于细胞核、细胞膜和叶绿体中,主要通过丝氨酸的磷酸化来发挥生物学作用;参与叶、茎、根和胚等生长时期的发育调控,其中bZIP44基因在种子中的表达量明显高于在叶片中的表达量,推测在种子油脂形成中发挥作用。研究结果为bZIP基因家族功能研究提供理论基础,并为改良种质及优良品种选育提供参考。
参考文献
[1] 宿明星,孙颖颢,施鹤,等. 植物非生物胁迫相关转录因子研究方法[J]. 生物技术通报,2015,31(1):51-60.
[2] 岳晓琦,杨成坤,张月,等. 荔枝RAV家族转录因子的全基因组鉴定及表达分析[J]. 植物生理学报,2022,58(6):1092-1104.
[3] 代梦媛,高梅,李文昌. 蓖麻GRF转录因子家族生物信息学鉴定及表达分析[J]. 分子植物育种,2021,19(22):7383-7390.
[4] 代春艳,张笑晗,王晓丽,等. 芥菜bZIP基因家族的鉴定和表达分析[J]. 分子植物育种,2024,22(7):2104-2118.
[5] 孙耀国,蔡天润,姬行舟,等. 西洋梨全基因组bZIP基因家族生物信息学分析[J]. 林业与生态科学,2021,36(1):24-34.
[6] 曹丽茹,马晨晨,庞芸芸,等. 玉米bZIP转录因子ZmbZIP26的分子特征、亚细胞定位及响应非生物胁迫的表达分析[J]. 华北农学报,2023,38(4):1-10.
[7] 潘婷婷. OsbZIP65的克隆与调控水稻开花的机制解析[D]. 大庆:黑龙江八一农垦大学,2023.
[8] 游冉. 水稻转录因子bZIP34调控胚乳淀粉合成和抽穗期的机制研究[D]. 扬州:扬州大学,2023.
[9] LI H Y,LI L X,SHANGGUAN G D,et al. Genome-wide identification and expression analysis of bZIP gene family in Carthamus tinctorius L.[J]. Scientific reports,2020,10:15521.
[10] 丛娇娇,王晓宇,李平,等. 蓖麻耐盐基因HKT的克隆及表达载体构建[J]. 分子植物育种,2018,16(14):4648-4657.
[11] 李威,李国瑞,黄凤兰,等. 蓖麻RcDELLA基因的克隆及序列分析[J]. 华北农学报,2018,33(1):14-19.
[12] 杨丽凤,常如慧,李国瑞,等. 喷施草铵膦与草甘膦对蓖麻叶片生理生化的影响研究[J]. 内蒙古民族大学学报(自然科学版),2018,33(5):411-416.
[13] 郑鹭. 蓖麻种质资源鉴定评价及遗传多样性ISSR与SRAP分析[D]. 福州:福建农林大学,2007.
[14] 张伟,龚文照,赵广,等. 蓖麻油及其下游产业市场分析与发展探讨[J]. 山西化工,2016,36(3):24-27.
[15] OGUNNIYI D S. Castor oil:a vital industrial raw material[J]. Bioresource technology,2006,97(9):1086-1091.
[16] 杨晗. 基于隐马尔科夫模型的噬菌体尾部模块识别[D]. 哈尔滨:哈尔滨工业大学,2022.
[17] 陈渝,邓洁,陈君愉,等. 番茄FAR1/FHY3转录因子家族的全基因组鉴定及表达分析[J]. 植物生理学报,2021,57(10):1983-1995.
[18] MOUNT D W. Using the basic local alignment search tool (BLAST)[J]. Cold spring harbor protocols,2007,2007(7):pdb.top17.
[19] FINN R D,CLEMENTS J,EDDY S R. HMMER web server:interactive sequence similarity searching[J]. Nucleic acids research,2011,39(2):29-37.
[20] LU S N,WANG J Y,CHITSAZ F,et al. CDD/SPARCLE:the conserved domain database in 2020[J]. Nucleic acids research,2020,48(1):265-268.
[21] ZDOBNOV E M,APWEILER R. InterProScan-an integration platform for the signature-recognition methods in InterPro[J]. Bioinformatics,2001,17(9):847-848.
[22] SIEVERS F,HIGGINS D G. Clustal omega,accurate alignment of very large numbers of sequences[M]//Methods in molecular biology. Totowa,NJ:Humana Press,2013.
[23] WATERHOUSE A M,PROCTER J B,MARTIN D M A,et al. Jalview Version 2-a multiple sequence alignment editor and analysis workbench[J]. Bioinformatics,2009,25(9):1189-1191.
[24] BAILEY T L,ELKAN C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers[J]. Proceedings. international conference on intelligent systems for molecular biology,1994,2:28-36.
[25] SUBRAMANIAN B,GAO S H,LERCHER M J,et al. Evolview v3:a webserver for visualization,annotation,and management of phylogenetic trees[J]. Nucleic acids research,2019,47(1):270-275.
[26] CHEN J W. Dynamic research of growth,N,P,K and active ingredients in Angelica dahurica var. Formosana from Sichuan province[J]. China journal of Chinese materia medica,2010,35(21):2812-2817.
[27] GUO C L,GUO R R,XU X Z,et al. Evolution and expression analysis of the grape (Vitis vinifera L.) WRKY gene family[J]. Journal of experimental botany,2014,65(6):1513-1528.
[28] GASTEIGER E,HOOGLAND C,GATTIKER A,et al. Protein identification and analysis tools on the ExPASy server[M]//The proteomics protocols handbook. Totowa,NJ:Humana Press,2005.
[29] 练从龙,赖钟雄,卢秉国,等. 荔枝古树胚性愈伤组织Fe-SOD成员基因克隆及生物信息学分析[J]. 热带作物学报,2014,35(1):74-81.
[30] SZKLARCZYK D,FRANCESCHINI A,KUHN M,et al. The STRING database in 2011:functional interaction networks of proteins,globally integrated and scored[J]. Nucleic acids research,2011,39(1):561-568.
[31] 段强,何智彪,李国瑞,等. 蓖麻LBD基因家族全基因组鉴定、进化和表达分析[J]. 北方农业学报,2021,49(6):1-14.
[32] 安爽,刘金莲,占晓慧,等. 蓝莓bZIP转录因子基因预测及其在根中表达分析[J]. 浙江农业科学,2021,62(10):1977-1983.
[33] 靳正伟. 蓖麻bZIP家族鉴定及与油脂基因互作分析[D]. 合肥:中国科学技术大学,2014.
(责任编辑:胡立萍)
