深度卷积网络模型可自动识别与分割胰腺及其肿瘤:基于3DV-Net
2024-12-31陈菲李茂林蒋玉婷李康安
摘要:目的 探讨基于V-Net的深度卷积神经网络模型在胰腺及其肿瘤自动识别和分割任务中的有效性和可行性。方法 回顾性分析2012年5月~2019 年11月于上海交通大学医学院附属第一人民医院就诊且经病理证实为胰腺癌的186例患者的增强CT影像资料,经过筛选,共纳入108例胰腺癌病例,随机搜集同期37例正常胰腺病例用于对照,最终共纳入145例数据,构成本研究的数据集。采用五折交叉验证方法,在动脉期CT图像上进行人工标注感兴趣区域(ROI),包括胰腺头颈部、体尾部和肿瘤,通过计算敏感度、特异度、F1分数等指标评估模型对胰腺肿瘤的识别能力,并进行Kappa一致性验证。采用Dice系数定量评估模型的分割能力,并获取可视化结果进一步评估。结果 基于V-Net的模型识别胰腺肿瘤的敏感度为0.852、特异度为1.000、阳性预测值为1.000、阴性预测值为0.698,F1分数高达0.920。一致性验证显示,Kappa系数为0.746(Plt;0.05)。在分割任务中,胰腺肿瘤、胰腺体尾部和胰腺头颈部的Dice系数分别为0.722±0.290、0.602±0.175、0.567±0.200。结论 本研究构建基于V-Net的深度卷积网络模型,有效完成了胰腺及其肿瘤自动识别与分割,验证了该方法的有效性和可行性,为探索胰腺肿瘤领域人工智能应用提供有力支持。
关键词:胰腺肿瘤;V-Net;深度学习;卷积神经网络;人工智能;自动分割
A deep convolutional network model based on3D V-Net can automatically recognize and
segment pancreas and its tumors
CHEN Fei 1 , LI Maolin 1 , JIANG Yuting 2 , LI Kang'an 1
1 Department of Radiology, First People's Hospital Affiliated to Shanghai Jiaotong University Medical College, Shanghai 201620, China;
2 Department of Radiology, Jintan First People's Hospital, Changzhou 213200, China
Abstract: Objective To explore the effectiveness and feasibility of the deep convolutional neural network model, based on V-Net, for automatic recognition and segmentation of the pancreas and its tumors. Methods A retrospective analysis wasconducted on the enhanced CT imaging data of 186 patients with pathologically confirmed pancreatic cancer who visited FirstPeople's Hospital Affiliated to Shanghai Jiaotong University Medical College from May 2012 to November 2019. Afterscreening, a total of 108 cases of pancreatic cancer were included, and 37 cases of normal pancreas during the same periodwere randomly collected for comparison, resulting in a final dataset of 145 cases for this study. This paper employed a five-foldcross-validation method and manually annotated regions of interest on arterial phase CT images, including the pancreatichead and neck, body and tail, and tumors. The model's ability to identify pancreatic tumors was evaluated by calculatingmetrics such as sensitivity, specificity, F1 score, and Kappa consistency verification was performed. Dice coefficient was used toquantitatively assess the model's segmentation capability, and visual results were obtained for further evaluation. Results TheV-Net based model for identifying pancreatic tumors has a sensitivity of 0.852, a specificity of 1.000, a positive predictive valueof 1.000, a negative predictive value of 0.698, and an F1 score as high as 0.920. The consistency verification shows that theKappa coefficient is 0.746 (Plt;0.05). In the segmentation task, the mean Dice for pancreatic tumors, pancreatic body and tail,pancreatic head and neck were 0.722±0.290, 0.602±0.175, 0.567±0.200, respectively. Conclusion We constructed a deepconvolutional network model based on V-Net, which successfully achieved automatic identification and segmentation of thepancreas and tumors. Our findings demonstrated the effectiveness and feasibility of this approach, offering robust support forthe exploration of artificial intelligence applications in the field of pancreatic tumor research.
Keywords: pancreatic tumor; V-Net; deep learning; convolutional neural network; artificial intelligence; automaticsegmentation
胰腺癌预后较差,总体5年相对生存率约10%[1] 。研究数据显示,TNM I期(T 1-2 N 0 M 0 )或直径lt;1 cm的胰腺癌患者5年生存率可达75%[2] ,目前临床普遍认为,早期诊断与及时手术干预是改善胰腺癌患者预后的唯一有效途径[3] 。胰腺癌疑似病例首选的影像学检查方法是多排螺旋CT薄层扫描,最好为亚毫米级[4, 5] 。影像诊断依赖于医师的视觉评估,存在差错与观察者间差异,亟需建立精准稳定的诊断与预测手段。近年来,人工智能凭借其强大的计算能力和卓越的学习能力在临床实践中展现出显著优势[6] 。器官和病变的精准分割,对于人工智能进一步辅助疾病诊断、明确病情分期以及精准制定治疗计划来说,具有至关重要的意义[7]。但在胰腺CT分割研究中,准确性相较于其他腹部器官仍显不足[8] 。胰腺分割研究主要分为基于传统方法和基于深度学习卷积神经网络两类。既往研究提出了基于残差的多尺度感受野网络,并采用了多尺度跨域的信息融合策略,有效解决了过分割、欠分割及形状不一致等问题;但其属于传统方法,需依赖人工设定参数和手动提取图像特征,自动化程度低[9] 。鉴于医学图像数据庞大且分割受操作者影响大,基于深度学习卷积神经网络的自动分割算法可以提高效率和准确性,如有研究提出了创新的深度学习网络,该网络基于可形变U-Net架构,结合了Q学习算法,通过充分考虑上下文信息,实现精确的胰腺分割[10]。目前,无论是基于传统方法还是深度学习卷积神经网络的半自动及自动分割算法,其结果仍然未能完全满足临床的实际需求。V-Net可以看作是3D U-Net的变体,是三维医学图像分割卷积神经网络[11] ,它基于全卷积网络,接受端-到-端训练,是一种全自动识别系统。已有研究将其应用于3D前列腺图像的分割任务中,成功实现了对整个前列腺MRI图像的一次性精准分割。本文拟构建基于V-Net先进深度学习技术的分割模型,应用于胰腺及其病灶的精确分割。既往研究普遍采用监督学习方法,即需要对每一份输入数据进行标注。深度学习在医学图像处理中的应用高度依赖于高质量、充足且多样化的标注数据[12] 。然而,医学图像的人工标注成本高昂,需医生及专家具备丰富知识与经验,尤其三维医学图像的体素标注尤为耗时;加之患者群体与采集参数的差异性,医学影像标注数据的稀缺性问题日益显著,成为制约深度学习分割方法发展的一大瓶颈[13]。本研究尝试采用人工稀疏三维标注方法,缩减标注成本同时提升数据利用效率。本研究拟构建并优化一种基于3D V-Net架构的深度卷积神经网络模型,旨在验证其在胰腺及其肿瘤自动识别与分割任务中的有效性与可行性。模型在V-Net架构中创新性地融入了CoordConv层[14],实现图像信息与对应坐标信息的深度融合。此外,本研究采用人工稀疏三维标注方法并引入Mean Teachers[15] 半监督学习 [16] 策略,使模型能够充分利用未标记数据进行学习,提升其泛化性能。
1 资料与方法
1.1 研究对象
本研究采用连续性入组方式,回顾性分析2012年5月~2019年11月于上海交通大学医学院附属第一人民医院接受诊治且经病理证实为胰腺癌的186例患者的增强CT影像资料。纳入标准:年龄≥18岁;病史记录详实,经病理证实确诊为胰腺癌,检查前未行胰腺切除手术,胰腺大体解剖位置正常;重建图像层厚需≤3 mm,确保图像清晰、完整,质量满足胰腺及肿瘤轮廓勾画需求。排除标准:曾接受胰腺相关手术,致胰腺结构或位置改变;大量腹水,影响脏器对比度;图像伪影多,影响诊断;图像层厚gt;3 mm。最终纳入108例胰腺癌病例。另随机选取同期37例正常胰腺病例作为对照,半年后电话随访,均未发生胰腺相关疾病,且血生化指标(CA199)均在正常值范围内。本研究最终纳入145例数据用于胰腺及肿瘤识别和分割,包括108例病变胰腺和37例正常胰腺。本研究已获得伦理委员会批准(审批号:2020-044),患者信息均经匿名化处理。
1.2 CT影像采集方法
本研究采用16排及以上多排螺旋CT进行上腹部动态增强扫描,数据采集源自 Siemens(SomatomForce、Somatom Definition Flash),GE(Discovery、Revolution)及Philips的CT扫描仪。实施了统一扫描方案:动态、无间隔螺旋扫描,管电压120 kV,管电流采用自动毫安秒技术。患者仰卧,先行平扫,后静脉注射90~100 mL含碘造影剂(浓度gt;300 mgI/L,注射速率3~5 mL/s)进行增强扫描。大部分病例采用动静脉期延迟扫描(动脉期25~30 s,门静脉期60~70 s),少部分采用胰腺期延迟扫描(35~50 s)。图像重建层厚≤3 mm。扫描范围主要为上腹部膈顶至双肾下极,可扩展至胸部和骨盆,少部分病例以包含胰腺为主,从肝门至肾门水平。
1.3 数据标注
本研究选择在动脉期CT图像上进行人工稀疏三维标注,辅以平扫、静脉期及三维重建图像作为参考。首先由具有3年以上诊断经验的放射科医师沿着胰腺及肿瘤边缘手动勾画感兴趣区(ROI),ROI标注分为胰腺头颈部、体尾部及胰腺肿瘤。稀疏三维标注策略即灵活选取代表性层面进行标注,避免连续且相似层面重复标注,来平衡标注工作量与数据代表性。标注结果经过高年资主治及以上级别医师的严格审阅与修正,以确保标注质量。整体标注策略灵活自由,符合用户习惯,确保胰腺所在总层面中至少有10%的数据被标注,同时保留90%的未标注数据,可提升用户标注效率。
1.4 数据预处理
对于胰腺肿瘤的多模态医学图像进行图像配准和标准化处理。采用随机裁剪数据增强技术增加数据多样性,提升模型泛化能力。使用Normalize函数进行归一化处理,统一样本统计分布。图像数据采用双线性插值,标签数据采用最近邻插值,以保留图像细节并保持标签数据类型一致。通过上述预处理步骤,得到了处理后的图像数据及其对应的掩膜图像。在导入网络训练前,需对数据集进行随机化分配。
1.5 基于V-Net的改进模型
本研究基于V-Net模型进行改进,通过在卷积层前加入 CoordConv 层来增强坐标信息的学习,并引入Mean Teachers算法以提升在少量标注数据下的分割效果。实验采用kernel_size为(1,1,4)、stride为(1,1,4)的卷积层,缩减输入尺寸至(128,128,80)后输入V-Net。研究中运用了Dropout层、批量归一化和L2正则化等多种正则化技术,旨在应对数据不平衡与未标记数据所带来的挑战。同时,采用基于Dice系数的损失函数和SGD优化函数优化模型参数。
采用五折交叉验证评估模型,数据集内病变与正常胰腺随机分布,以提高训练准确性及泛化能力。使用TensorBoard工具,可视化跟踪准确率和损失函数,提升评估效率。实验环境基于Anaconda创建的Python 3.8环境,并安装了 PyTorch、Pillow 和 OpenCV 库,使用PyTorch DataLoader进行数据加载训练参数设置为:初始学习率为0.01,每1500次迭代学习率衰减10倍,训练总迭代次数为6000,Dropout率为0.5,动量值为0.9。
1.6 统计学分析
采用SPSS27.0统计软件进行数据分析和处理,计算敏感度、特异度、阳性预测值、阴性预测值和F1分数,评估基于V-Net深度卷积神经网络模型对胰腺肿瘤的识别性能。采用Kappa一致性验证和Dice系数的计算与定量评估,分析模型的准确性和稳定性。
2 结果
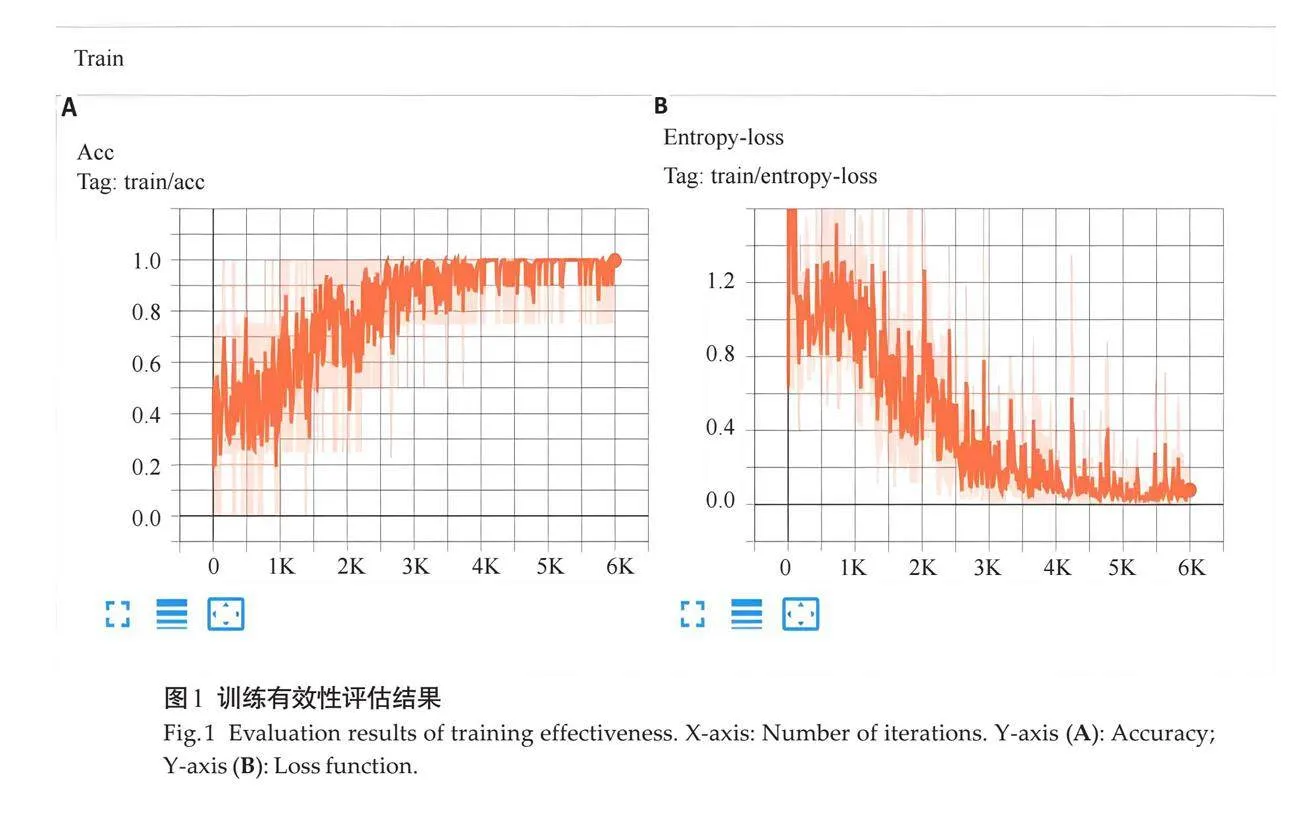
2.1 训练有效性评估结果
在训练过程中,本模型共进行了6000次迭代,其性能通过训练准确率和训练熵损失2个关键指标得到了全面评估。随着迭代次数的增加,训练准确率稳步上升,并在大约2000次迭代后趋于稳定(图1A)。训练熵损失的下降趋势显示,初期迅速降低,大约在2000次迭代后整体仍呈下降趋势(图1B)。模型在训练过程中的动态性能提升,训练准确率有所下降,而熵损失上升,模型仍正常收敛,当熵损失不再持续下降时,准确率开始稳定,模型权值逐渐稳定。
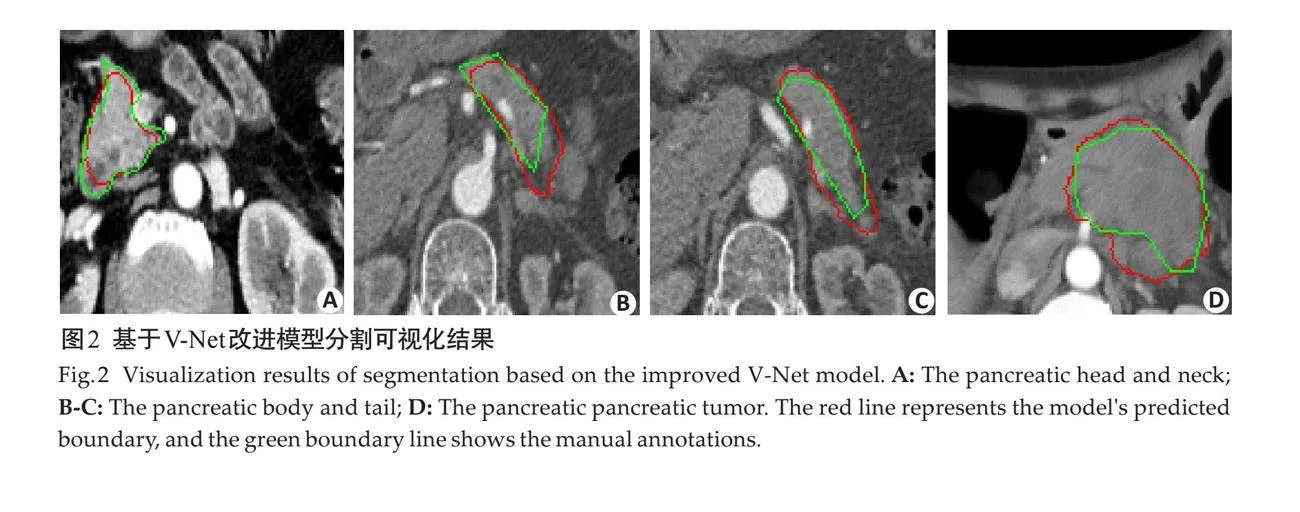
2.2 分割可视化结果
分割结果与原图中的手工标注的对比,依次为胰腺头部的分割结果(图2A)、胰腺体尾部的分割结果(图2B~C)以及胰腺肿瘤的分割结果(图2D)。
2.3 肿瘤识别评估
模型识别结果显示,本模型肿瘤识别敏感度为0.852,特异度为1.000,阳性预测值与F1分数分别为1.000、0.920,阴性预测值为0.698。Kappa一致性验证显示,Kappa系数为0.746(Plt;0.05)。
2.4 自动分割定量评估
采用Dice系数对肿瘤及胰腺分割进行定量评估,结果显示,胰腺肿瘤的Dice系数为0.722±0.290,胰腺体尾部为0.602±0.175,胰腺头颈部为0.567±0.200。
3 讨论
近年来,胰腺癌的发病率和死亡率逐年递增[17, 18] 。目前,医学影像领域还存在医疗资源不足、专业医生缺口巨大、误诊率偏高、诊断效率低等痛点[19] 。传统影像学诊断胰腺癌受医师水平影响,而人工智能有望打破此限,并提升效率,减轻医师压力[20] 。目前,胰腺人工智能应用的研究仍然相对较少,大多数早期研究主要集中于图像采集与优化[21] 。胰腺独特的解剖结构使其成为腹部分割的难点,加之医学图像中胰腺及病灶标注数据稀缺,导致胰腺自动分割面临诸多挑战。既往人工智能在胰腺研究中主要聚焦正常胰腺[21],建模数据主要来自欧美人群的公开数据集[22, 23] 。本研究聚焦深度卷积神经网络在胰腺及病灶分割中的应用,训练数据集全部来源于国内本土人群,充分代表我国人群特征。
本研究旨在开发一种针对胰腺及其病灶的自动分割算法,并深入探索及实现一种灵活、自由且高效的标注方法策略。模型构建设计遵循影像科医生诊断的核心原则“先定位后定性”。本研究旨在完成两项任务:其一,识别肿瘤的存在与否。鉴于正常胰腺图像中不存在肿瘤,模型仅能观察到背景信息,因此本研究纳入了正常病例,可通过对背景数据进行表征学习,使模型明确在正常影像中不应检测出肿瘤,从而对其学习进行约束;正常胰腺影像亦可用于检验模型的准确度,若模型在正常影像中错误地检测到肿瘤,则表明其泛化能力尚待提升。其二,模型需按照解剖位置将胰腺分割为头颈部和体尾部,以便于后续更深入的研究,并实现对病变位置的精准定位。本研究有望提高模型的诊断效率及准确性,为医师检测与识别病灶提供有力辅助,有效减轻医生的工作负担,并减少漏诊情况。
本文在 V-Net 中创新性地引入 CoordConv 和Mean Teachers,这些技术增强了V-Net模型的特性和性能,使其能更准确地捕捉到图像中的细节和结构信息。V-Net采用体积卷积(3D卷积)作为输入,使模型能够同时处理3个空间维度的数据,从而更有效地学习切片间的上下文信息,这也使得V-Net在处理三维医学图像数据时表现出显著的优势。坐标卷积是一种新型的卷积方式,通过在卷积操作中引入额外的坐标通道,将图像与坐标信息融合,增强了模型的空间感知力,从而更精准地分割和识别图像目标。Mean Teachers是一种半监督学习算法,通过设立教师和学生2个模型,教师模型权重为学生模型的时间加权平均,提升了半监督学习性能,使模型在部分标记数据下也能取得良好效果。胰腺癌病灶在动脉期多呈乏血供状态,有助于区分肿瘤/非肿瘤。有团队在构建深度学习胰腺自动分割模型时发现,静脉期的Dice相似系数略高于动脉期,但差异无统计学意义[24]。有研究提出了一种新的深度学习框架,该框架结合了体素分类和边界重建两种方法,以迭代的方式共同提高器官分割的性能;还引入了一种标签完善策略,以补全大量未标记体素的标签,进而将这些数据嵌入到训练集中,以增强模型的性能[25]。基于上述理论和研究,鉴于大部分胰腺癌在动脉期强化程度通常弱于正常胰腺实质,呈相对低密度改变这一特点,本研究选择尝试仅在动脉期进行稀疏人工标注,同时结合平扫和静脉期图像作为辅助判断手段。研究表明,在人工智能应用于胰腺癌及其癌前病变的诊断与预测方面,其AUC为 0.790~0.999,准确度为 77.66%~99.20%,敏感度为76.64%~100%,特异度为85.59%~98.50%[26] 。有学者提出了多尺度分割分类网络,该网络采用由粗到细的分割策略,在303例PDAC患者和136例健康对照的CT扫描中,识别肿瘤敏感度为 94.1% 的,特异度为98.5%[27] 。有研究通过筛选222例胰腺癌患者和190例胰腺正常者的CT图像,训练了一个卷积神经网络模型,其二分类模型在诊断平扫图像时,准确率达95.47%,敏感度为91.58%,特异度为98.27%,与放射科医师的诊断准确率相当[28] 。也有研究利用319例患者的腹部增强CT图像,经过手动筛查确保图像质量后,训练出一种深度学习模型应用于肿瘤诊断,表现优异,AUC达0.871,F1分数为88.5%[29] 。这些研究共同展示了人工智能在胰腺癌诊断中的潜力和价值。但上述研究均基于卷积神经网络,并逐步由二维图像处理向三维卷积方法演进,或尝试将两者融合,且训练时均要求对每份输入数据进行详尽标注。与之不同的是,本研究完全基于三维卷积神经网络构建模型,并采用了一种高效的稀疏三维标注策略,仅在动脉期进行标注,显著减少了标注工作量。通过这一方法,本研究在肿瘤识别上取得了较好成果:敏感度为 0.852,特异度为 1.000,阳性预测值为1.000,阴性预测值为 0.698,且 F1分数达到了 0.920。与既往上述研究相比,本研究的结果不仅处于较高水平,而且充分验证了所提模型算法框架的高效性和实用性。另外,本文还进行了一致性检验,Kappa系数为0.746(Plt;0.05),说明模型分类结果与金标准之间具有良好的一致性。
本研究使用Dice系数定量评估肿瘤及胰腺分割的准确性,将胰腺分割为肿瘤、体尾部和头颈部3部分分析。剔除Dice系数lt;0.1的样本后,胰腺肿瘤、胰腺体尾部、胰腺头颈部的Dice系数分别0.722±0.290、0.602±0.175、0.567±0.200。Dice系数作为衡量分割准确性的重要指标,其值越高表示分割结果与真实情况越接近。所有类别的Dice系数均数gt;0.5,表明分割方法准确,尤其在复杂肿瘤结构分割上性能优越。较低Dice系数的胰腺部分可能与病变胰腺高比例相关,因其结构和形态变化增加分割难度。有研究提出了腹盆19个正常组织的自动勾画(其中胰腺DSC=0.62)[16] ,以及有研究提出了腹部8个正常组织自动勾画的V-Network(其中胰腺DSC=0.75)[30] ,本研究聚焦病变胰腺,验证了本研究模型在胰腺肿瘤分割上的优越性,凸显了算法的鲁棒性和泛化能力。
本研究存在一些不足之处,可能导致偏倚:样本量差异(正常与病变胰腺),尽管通过统计模拟验证未发现显著差异;样本不平衡,早期胰腺癌病例较少;样本量较少,可能引发过拟合或欠拟合问题,影响模型识别准确性。另外,本研究使用Labelme软件进行标注,该软件仅支持jpg格式,因此采用无损jpg格式数据,虽满足本研究需求,但相比DICOM格式,jpg在医学图像处理中存在一定压缩和失真,数据精确性和完整性相对较低,给本研究带来一定的局限性。采用高精度的深度学习模型有望在一定程度上弥补这一不足。综上,本研究通过多维度全面评估了所构建的基于改进V-Net模型,包括训练有效性、分割结果的可视化展示,以及定性与定量的综合分析。研究结果表明,该模型在胰腺及其肿瘤的识别与分割方面展现出优异性能,有效性和可行性得到充分验证。这一成果为胰腺肿瘤的计算机辅助诊断开辟了新途径,有望提升早期胰腺癌的诊断效率,推动精准医疗的进步,具有广阔的应用前景。未来研究将从标注比例探索、算法优化、数据增强与后处理技术改进,以及扩充高质量、标准化的标注数据集等多方面努力,旨在持续提升模型性能,实现更精准、更高效的医疗影像分析,为胰腺癌等疾病的计算机辅助诊断提供更强支持。
参考文献:
[1] National Cancer Institute. Pancreas cancer[EB/OL]. [2021-02-18]https://seer.cancer.gov/statfacts/html/pancreas.html.
[2] 孔祥毓. 胰腺癌早期筛查中的问题和进展[J]. 中华胰腺病杂志,2021, 21(1): 16-9.
[3] Cai J, Chen HD, Lu M, et al. Advances in the epidemiology ofpancreatic cancer: trends, risk factors, screening, and prognosis[J].Cancer Lett, 2021, 520: 1-11.
[4] Tempero MA, Malafa MP, Al-Hawary M, et al. Pancreaticadenocarcinoma, version 2.2021, NCCN clinical practice guidelinesin oncology[J]. J Natl Compr Canc Netw, 2021, 19(4): 439-57.
[5] 中华医学会外科学分会胰腺外科学组. 中国胰腺癌诊治指南(2021)[J]. 中国实用外科杂志, 2021, 41(7): 725-38.
[6] Holmes JH, Sacchi L, Bellazzi R, et al. Artificial intelligence inmedicine AIME 2015[J]. Artif Intell Med, 2017, 81: 1-2.
[7] Barat M, Chassagnon G, Dohan A, et al. Artificial intelligence: acritical review of current applications in pancreatic imaging[J]. JpnJ Radiol, 2021, 39(6): 514-23.
[8] 李庆勃, 苏 丹. 基于V-Net的腹部多器官图像分割[J]. 数字技术与应用, 2019, 37(1): 89, 91.
[9] Li FY, Li WS, Shu YC, et al. Multiscale receptive field based onresidual network for pancreas segmentation in CT images[J].Biomed Signal Process Contr, 2020, 57: 101828.
[10]Man YZ, Huang Y, Feng JY, et al. Deep Q learning driven CTpancreas segmentation with geometry-aware U-net[J]. IEEE TransMed Imaging, 2019, 38(8): 1971-80.
[11]Milletari F, Navab N, Ahmadi SA. V-net: fully convolutional neuralnetworks for volumetric medical image segmentation
[C]//2016Fourth International Conference on 3D Vision (3DV). Stanford, CA,USA. IEEE, 2016: 565-71.
[12]Kleppe A, Skrede OJ, De Raedt S, et al. Designing deep learningstudies in cancer diagnostics[J]. Nat Rev Cancer, 2021, 21(3):199-211.
[13]Peng J, Wang Y. Medical image segmentation with limitedsupervision: a review of deep network models
[J/OL]. IEEE Access,2021. doi:10.1109/ACCESS.2021.3062380.
[14]Liu R, Lehman J, Molino P, et al. An intriguing failing ofconvolutional neural networks and the CoordConv solution
[EB/OL].
[2018-12-03]. http://arxiv.org/abs/1807.03247
[15]Tarvainen A, Valpola H. Mean teachers are better role models:Weight-averaged consistency targets improve semi-supervised deeplearning results
[EB/OL].
[2018-04-16]. https://doi. org/10.48550/arXiv.1703.01780
[16]Zhou XR, Takayama R, Wang S, et al. Deep learning of the sectionalappearances of 3D CT images for anatomical structure segmentationbased on an FCN voting method[J]. Med Phys, 2017, 44(10):5221-33.
[17]McGuigan A, Kelly P, Turkington RC, et al. Pancreatic cancer: a review of clinical diagnosis, epidemiology, treatment and outcomes[J]. World J Gastroenterol, 2018, 24(43): 4846-61.
[18]Chen X, Yi B, Liu Z, et al. Global, regional and national burden ofpancreatic cancer, 1990 to 2017: Results from the Global Burden ofDisease Study 2017[J]. Pancreatology, 2020, 20(3): 462-9.
[19]陈校云, 张旭东, 舒 婷. 中国医疗人工智能发展报告(2020)
[M]. 北京: 社会科学文献出版社, 2020.
[20]田永刚, 曹贞子, 白飞虎, 等. 人工智能在消化系肿瘤的研究进展[J]. 中华胃肠内镜电子杂志, 2019, 6(2): 82-5.
[21]Zheng H, Chen Y, Lu J, et al. Deep pancreas segmentation withuncertain regions of shadowed sets[J]. Magn Reson Imaging, 2020,68:45-52.
[22]Fu M, Wu WM, Hong XF, et al. Hierarchical combinatorial deeplearning architecture for pancreas segmentation of medicalcomputed tomography cancer images[J]. BMC Syst Biol, 2018, 12(Suppl 4): 56.
[23]Oliveira B, Queirós S, Morais P, et al. A novel multi-atlas strategywith dense deformation field reconstruction for abdominal andthoracic multi-organ segmentation from computed tomography[J].Med Image Anal, 2018, 45: 108-20.
[24]王希恒, 薛华丹, 曲太平, 等. 基于深度学习胰腺分割算法在双期CT上提升胰腺临界区域分割能力分析[J]. 中华医学杂志, 2021, 101(7): 470-5.
[25]Sheng T, Mathai TS, Shieh A, et al. Weakly-supervised detection ofbone lesions in CT[J]. Proc SPIE Int Soc Opt Eng, 2024. doi:10.1117/12.3008823.
[26]Huang BW, Huang HR, Zhang ST, et al. Artificial intelligence inpancreatic cancer[J]. Theranostics, 2022, 12(16): 6931-54.
[27]Zhu Z, Xia YD, Xie LX, et al. Multi-scale coarse-to-finesegmentation for screening pancreatic ductal adenocarcinoma
[M]//Lecture Notes in Computer Science. Cham: Springer InternationalPublishing, 2019: 3-12.
[28]Ma H, Liu Z, Zhang J, et al. Construction of a convolutional neuralnetwork classifier developed by computed tomography images forpancreatic cancer diagnosis[J]. World J Gastroenterol, 2020, 26(34): 5156-68.
[29]Si K, Xue Y, Yu X, et al. Fully end-to-end deep-learning-baseddiagnosis of pancreatic tumors[J]. Theranostics, 2021, 11(4):1982-90.
[30]Gibson E, Giganti F, Hu YP, et al. Automatic multi-organsegmentation on abdominal CT with dense V-networks[J]. IEEETrans Med Imaging, 2018, 37(8): 1822-34.(编辑:张庭玉)
