基于生物信息学分析溃疡性结肠炎DNA甲基化标志物
2024-12-12海双双刘怡红赵海波于瑞淼


[摘要]"目的"基于生物信息学方法分析溃疡性结肠炎(ulcerative"colitis,UC)的DNA甲基化生物标志物。方法"采用基因表达综合数据库(Gene"Expression"Omnibus,GEO)的芯片数据GSE81211、GSE75214、GSE87466分析UC的差异甲基化位点和差异表达基因,得到异常甲基化修饰的差异表达基因,并进行富集分析和蛋白质互作网络(protein-protein"interaction,PPI)分析。使用谱芯片GSE36807验证核心基因的表达水平并绘制受试者操作特征曲线验证核心基因诊断效能。结果"共筛选出42个高甲基化修饰低表达基因,53个低甲基化修饰高表达基因。功能富集分析显示主要与先天免疫反应、T细胞受体信号通路、上调细胞外信号调节激酶(extracellular"regulated"kinase,ERK)1和ERK2级联反应相关。基因富集的信号通路为细胞黏附分子和代谢途径。结论"7个异常甲基化修饰差异表达基因SELL、SLAMF1、FCGR2A、CD86、CTLA4、CCR7、PTPRC可能是UC的潜在生物标志物及治疗靶点。
[关键词]"溃疡性结肠炎;生物信息学;DNA甲基化;表观遗传学
[中图分类号]"R574.6""""""[文献标识码]"A""""""[DOI]"10.3969/j.issn.1673-9701.2024.35.009
Bioinformatics-based"analysis"of"DNA"methylation"markers"in"ulcerative"colitis
HAI"Shuangshuang1,"LIU"Yihong1,"ZHAO"Haibo1,"YU"Ruimiao2
1.Department"of"Gastroenterology,"the"First"Hospital"of"China"Medical"University,"Shenyang"110000,"Liaoning,"China;"2.Department"of"Gastroenterology,"Chifeng"Hospital,"Chifeng"024000,"Inner"Mongolia,"China
[Abstract]"Objective"To"analyze"DNA"methylation"biomarkers"of"ulcerative"colitis"(UC)"based"on"bioinformatics"methods."Methods"Chip"data"from"Gene"Expression"Omnibus"(GEO),"specifically"GSE81211,"GSE75214,"and"GSE87466,"were"used"to"analyze"differentially"methylated"sites"and"differentially"expressed"genes"in"UC."This"led"to"the"identification"of"differentially"expressed"genes"with"abnormal"methylation"modifications."These"genes"were"then"subjected"to"enrichment"analysis"and"protein-protein"interaction"(PPI)"network"analysis."The"expression"levels"of"core"genes"were"validated"using"the"spectral"chip"GSE36807,"and"diagnostic"efficacy"of"these"core"genes"was"verified"by"plotting"receiver"operating"characteristic"curves."Results"A"total"of"42"genes"with"high"methylation"modifications"and"low"expression,"and"53"genes"with"low"methylation"modifications"and"high"expression"were"identified."Functional"enrichment"analysis"showed"that"these"genes"were"mainly"associated"with"innate"immune"responses,"T"cell"receptor"signaling"pathways,"and"upregulation"of"extracelluar"regulated"kinase"(ERK1)"and"ERK2"cascade"reactions."The"enriched"signaling"pathways"of"these"genes"involved"cell"adhesion"molecules"and"metabolic"pathways."Conclusion"Seven"differentially"expressed"genes"with"abnormal"methylation"modifications"SELL,"SLAMF1,"FCGR2A,"CD86,"CTLA4,"CCR7,"and"PTPRC"may"serve"as"potential"biomarkers"and"therapeutic"targets"for"UC.
[Key"words]"Ulcerative"colitis;"Bioinformatics;"DNA"methylation;"Epigenetics
溃疡性结肠炎(ulcerative"colitis,UC)是一种慢性复发性炎症性肠道疾病,与克罗恩病(Crohn’s"disease,CD)统称为炎症性肠病(inflammatory"bowel"disease,IBD),遗传因素、环境因素、肠道微生物群和免疫系统均参与UC的病理生理过程[1]。UC的诊断基于非特异性症状、内窥镜检查和组织学特征相结合,有时很难与其他疾病区分。尽管有几种药物可用于治疗UC,但高达15%的患者对药物治疗无反应,且容易产生并发症。因此,迫切需要更好地了解UC的发病机制,寻找更有效的治疗方法[2]。越来越多的研究表明,DNA甲基化、非编码RNA表达和组蛋白修饰的异常表观遗传学改变对IBD的发生、发展发挥重要作用[2–4]。
表观遗传学指在不改变遗传信息的情况下调控基因表达的机制,包括多种调控方式,是基因型、环境和疾病之间的重要联系。由于表观遗传学具有遗传性、可逆性和动态性,更有利于调节寄主的发育、分化和功能。表观遗传学参与许多生理功能,如维持肠道上皮的稳态,调节免疫细胞和免疫系统等[3]。在IBD的表观遗传学研究中,DNA甲基化是研究最多的方向之一[4]。本研究采用DNA甲基化芯片和表达谱芯片数据集联合分析DNA甲基化在UC发病机制中的可能作用,探索异常甲基化基因与疾病病理之间的可能联系,寻找临床诊断和治疗的潜在靶点。
1""资料与方法
1.1""数据来源
从基因表达综合数据库(Gene"expression"omnibus,GEO)中下载UC的表达谱芯片GSE75214、GSE87466数据集,验证集GSE36807数据集,DNA甲基化芯片GSE81211数据集。所有数据均来自肠道组织。
1.2""差异表达基因筛选
使用R语言中的limma包筛选GSE75214"和GSE87466数据集中的差异表达基因(differentially"expressed"gene,DEGs),筛选条件为|log2FC(差异倍数)|gt;1,调整P(P"adj)lt;0.05,绘制火山图。使用CHAMP包对GSE81211数据集进行差异甲基化位点(DNA-methylation"profiles,DMP)分析,筛选条件|delta"beta值|gt;0.2和Padjlt;0.05,得到与健康对照相比下UC肠组织的DMP和异常甲基化基因,绘制火山图和热图。将表达上调基因与低甲基化基因通过Venny软件取交集得到低甲基化修饰下高表达基因;将表达下调基因与高甲基化基因使用Venny软件取交集得到高甲基化修饰下低表达基因,绘制韦恩图。
1.3""DEGs功能富集分析
使用R语言ClusterProfiler包对异常甲基化修饰DEGs进行京都基因和基因组百科全书(Kyoto"Encyclopedia"of"Genes"and"Genomes,KEGG)和基因本体(gene"ontology,GO)分析,使用org.Hs.eg.db、ggplot2包对富集分析结果进行可视化。
1.4""蛋白质互作网络和子模块构建
将获取的异常甲基化修饰DEGs输入STRING数据库进行蛋白质互作网络(protein-protein"interaction,PPI)分析,以最小互作评分gt;0.4分为筛选条件,下载相关数据,利用Cytoscape"3.9.1软件进行分析并可视化,使用MCODE进行模块划分,筛选条件设置为MCODEgt;18、Node截断值=0.2、Degree"截断值=2、K值=2、最大深度=100,得到高度连通区域的子网。将MCODE子网络基因与CytoHubba中的MCC算法的前10个基因取交集得到核心基因。
1.5""验证核心基因的表达
验证核心基因在GSE36807数据集中的表达水平,使用GEO在线工具GEO2R查找DEGs在肠组织中的表达量。绘制受试者操作特征(receiver"operating"characteristic,ROC)曲线验证核心基因诊断效能。
1.6""统计学方法
采用SPSS"22.0统计学软件对数据进行处理分析,计量资料以均数±标准差(")表示,组间比较采用t检验,计数资料以例数(百分率)[n(%)]表示,组间比较采用χ2检验。Plt;0.05为差异有统计学意义。将异常甲基化修饰DEGs输入STRING在线网站,下载相关数据,导入Cytoscape软件,绘制PPI网络图。
2""结果
2.1""异常甲基化DEGs筛选结果
差异基因分析流程见图1。UC表达谱芯片GSE75214、GSE87466差异基因火山图及DNA甲基化芯片GSE81211数据集差异甲基化位点见图2。低甲基化修饰表达上调基因,低甲基化高表达基因53个,高甲基化低表达基因42个。
2.2""异常甲基化修饰DEGs富集分析结果
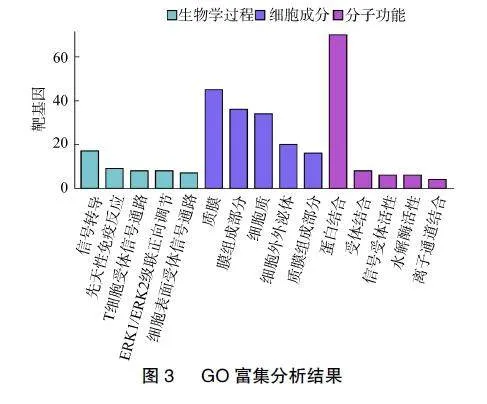
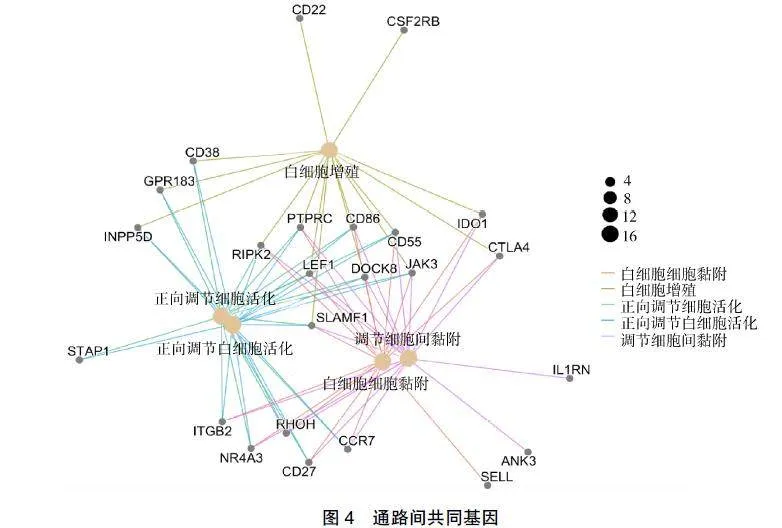
对95个异常甲基化修饰DEGs富集分析,GO分析结果显示,异常甲基化修饰DEGs多富集在信号转导、先天免疫反应、T细胞受体信号通路、上调细胞外信号调节激酶(extracellular"regulated"kinase,ERK)1和ERK2级联、细胞表面受体信号通路等生物学过程中,见图3。KEGG分析显示基因富集的信号通路为细胞黏附分子、代谢途径和酒精性肝脏疾病,通路间的共同基因见图4。
2.3""异常甲基化修饰DEGs的PPI及核心基因分析
MCODE子网络包含13个节点、138条边,MCODE值为11.5。将10个分数最高的基因与MCODE子网络基因取交集得到8个核心基因,分别为蛋白酪氨酸
磷酸酶受体C型(protein"tyrosine"phosphatase"receptor"type"C,PTPRC)、CD69、CD86、L-选择素(selectin"L,SELL)、Fc伽马受体Ⅱa"(Fc"gamma"receptor"Ⅱa,FCGR2A)、细胞毒性T淋巴细胞相关蛋白4(cytotoxic"T-lymphocyte"associated"protein"4,CTLA4)、C-C趋化因子受体7(C-C"motif"chemokine"receptor"7,CCR7)、信号淋巴细胞激活分子家族成员1"(signaling"lymphocytic"activation"molecule"family"member"1,SLAMF1)。8个核心基因均为低甲基化修饰基因。
2.4""验证核心基因的表达水平及诊断效能
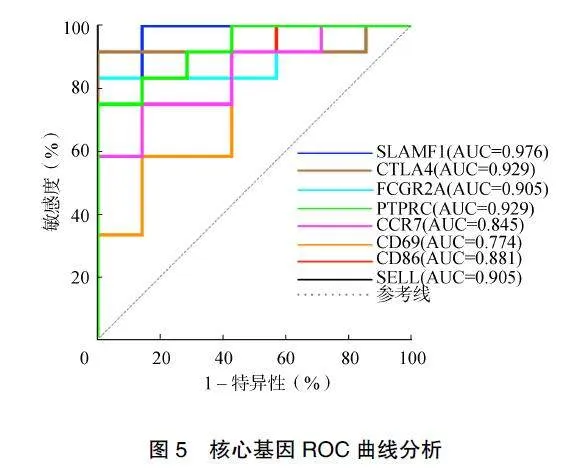
使用GSE36807数据集验证8个核心基因的表达水平,7个低甲基化修饰基因在UC患者肠组织中的表达水平均高于健康对照组,差异有统计学意义(Plt;0.05),而CD69在UC患者和健康对照者的肠组织中差异无统计学意义(Pgt;0.05)。绘制ROC曲线验证核心基因的诊断效能,曲线下面积(area"under"the"curve,AUC)为0.845~0.976,7个核心基因诊断效能较好,见图5。
3""讨论
研究表明DNA甲基化在诊断IBD中发挥重要作用,具有较高的敏感度及特异性[5]。来自肠组织的DNA甲基化标志物能较准确地区分CD和UC[6]。在儿童IBD患者的肠组织中发现特异性的DNA甲基化修饰,疾病诊断的特异性和敏感度均非常高[7]。这表明发掘IBD的特异性DNA甲基化模式具有重要的临床意义。
本研究联合多个芯片数据进行分析,筛选出95个具有异常甲基化修饰位点的差异表达基因,并进行PPI、GO和KEGG富集分析。异常甲基化修饰的差异表达基因多富集在信号转导、先天免疫反应、T细胞受体信号通路、上调ERK1和ERK2级联、细胞表面受体信号通路等生物学过程和细胞黏附分子、代谢途径等信号通路,与之前其他UC相关的研究结果一致[8]。IBD患者的肠道先天性和适应性免疫增强,先天免疫细胞响应微生物和受损组织的信号,产生炎症细胞因子和刺激适应性免疫系统T细胞和B细胞因子,炎症T细胞及其促炎相关细胞因子在肠组织中的积累[9]。细胞黏附分子是细胞表面的蛋白质,负责细胞与细胞、细胞与细胞外基质之间的相互作用,参与细胞生长分化、伸展和移动、激活与信号转导、炎症等重要病理生理过程,如T细胞通过表达黏附分子黏附在内皮细胞上,破坏T细胞和血管内皮之间的相互作用从而阻断T细胞浸润治疗UC[10]。因此,通过内皮细胞中黏附分子的表达来破坏免疫细胞浸润可能是治疗UC的有效策略[11]。ERK1和ERK2是丝氨酸/苏氨酸蛋白激酶,属于丝裂原活化蛋白激酶家族成员,参与胞内外信号转导,调节细胞增殖分化、蛋白质磷酸化等。UC患者炎性结肠黏膜组织可见ERK1、ERK2活化增强。在动物实验中抑制中性粒细胞ERK1、ERK2磷酸化可改善肠道炎症,表明ERK1、ERK2可能与UC的发病机制及疾病进展相关[12]。
本研究最终筛选出SELL、SLAMF1、FCGR2A、CD86、CTLA4、CCR7、PTPRC"7个有异常甲基化修饰位点的差异表达基因可能与UC密切相关。SELL是一种I型跨膜糖蛋白和细胞黏附分子,在大多数循环白细胞中表达,参与白细胞运输的调节,介导许多白细胞-内皮相互作用。SELL在UC中的研究较少,在正常和病理条件下,淋巴细胞迁移到胃肠黏膜形成肠道相关淋巴组织是由MAdCAM-1介导的,这种淋巴细胞归巢系统参与慢性炎症性胃肠道疾病的发病机制[13]。MAdCAM-1的表达与内镜下UC严重程度相关,与黏膜炎症和随后的复发有关,可作为UC复发和治疗效果的标志物[14]。SLAMF1调节多种免疫细胞的激活和分化,因此参与先天性和适应性免疫反应的调节和互联。有报道称SLAMF1在诊断为UC的前几年上调,高度预测UC的诊断,能够表征UC的临床前全身炎症[15]。FCGR2A是UC的易感基因,全基因组关联研究分析结果显示FCGR2A变体可改变其编码的抗体受体FCGR2A对IgG的结合亲和力[16]。在UC患者的结肠黏膜中,FCGR2A调控IgG免疫复合物,诱导NLRP3和反应性氧依赖性的白细胞介素-1β和中性粒细胞募集趋化因子的产生[17]。CD86是一种在抗原呈递细胞上表达的分子,提供T细胞活化和生存所需的共刺激信号,阻断CD86可有效抑制T细胞应答,在启动免疫应答中发挥重要作用。抑制树突状细胞(dendritic"cell,DC)表面抗原呈递细胞-Ⅱ和CD86表达,能够恢复结肠炎小鼠模型中TH17/Treg比值,改善小鼠UC[18]。CTLA4是T细胞上的一种跨膜受体,目前在肿瘤治疗中作为免疫检查点的研究非常丰富。CTLA4被认为与许多自身免疫性疾病相关,表达水平与免疫细胞浸润呈正相关,具有基因多态性,可增加UC风险[19]。CCR7被报道为新的UC易感识别位点,可能参与白细胞的快速募集和不恰当的保留导致肠道慢性炎症。使用布拉酵母菌可显著降低表达CCR7的IBD"骨髓DC,抑制T细胞共刺激和炎症相关迁移和DC动员,促进上皮恢复[20]。PTPRC可调节调节T细胞和巨噬细胞之间的相互作用,增加促炎细胞因子产生,在先天免疫系统中起着至关重要的作用。此外,PTPRC被鉴定为肝肾纤维化疾病中表达改变的潜在重要基因,并可能参与IBD纤维化途径的早期激活或纤维化和炎症的重叠[21]。这7个异常甲基化修饰的差异表达基因参与调节免疫反应,可能在UC的发病机制中起作用,AUC为0.845~0.976,具有良好诊断效能,或能作为诊断UC的生物标志物。
本研究存在一定的不足。本研究使用的样本量较小,未进行相关实验验证。综上,本研究基于生物信息学进行联合分析,筛选出7个异常DNA甲基化修饰的核心基因,分析了可能影响UC发生、发展的信号通路,为研究UC的DNA甲基化生物标志物及发病机制提供选择。
利益冲突:所有作者均声明不存在利益冲突。
[参考文献]
[1] SU"H,"CHIU"Y,"CHIU"C,"et"al."Inflammatory"bowel"disease"and"its"treatment"in"2018:"Global"and"Taiwanese"status"updates[J]."Formos"Med"Assoc,"2019,"118(7):"1083–1092.
[2] KAPLAN"G."The"global"burden"of"IBD:"From"2015"to"2025[J]."Nat"Rev"Gastroenterol"Hepatol,"2015,"12(12):"720–727.
[3] VENTHAM"N,"KENNEDY"N,"NIMMO"E,"et"al."Beyond"gene"discovery"in"inflammatory"bowel"disease:"The"emerging"role"of"epigenetics[J]."Gastroenterology,"2013,"145(2):"293–308.
[4] NOBLE"A,"NOWAK"J,"ADAMS"A,"et"al."Defining"interactions"between"the"genome,"epigenome,"and"the"environment"in"inflammatory"bowel"disease:"Progress"and"prospects[J]."Gastroenterology,"2023,"165(1):"44–60.
[5] MCDERMOTT"E,"RYAN"E,"TOSETTO"M,"et"al."Dna"methylation"profilingnbsp;in"inflammatory"bowel"disease"provides"new"insights"into"disease"pathogenesis[J]."Crohns"Colitis,"2016,"10(1):"77–86.
[6] TAHARA"T,"HIRATA"I,"NAKANO"N,"et"al."Comprehensive"DNA"methylation"profiling"of"inflammatory"mucosa"in"ulcerative"colitis[J]."Inflamm"Bowel"Dis,"2017,"23(1):"165–173.
[7] HOWELL"K,"KRAICZY"J,"NAYAK"K,"et"al."DNA"methylation"and"transcription"patterns"in"intestinal"epithelial"cells"from"pediatric"patients"with"inflammatory"bowel"diseases"differentiate"disease"subtypes"and"associate"with"outcome[J]."Gastroenterology,"2018,"154(3):"585–598.
[8] XAVIER"R,"PODOLSKY"D."Unravelling"the"pathogenesis"of"inflammatory"bowel"disease[J]."Nature,"2007,"448(7152):"427–434.
[9] GEREMIA"A,"BIANCHERI"P,"ALLAN"P,"et"al."Innate"and"adaptive"immunity"in"inflammatory"bowel"disease[J]."Autoimmun"Rev,"2014,"13(1):"3–10.
[10] WEST"N,"HEGAZY"A,"OWENS"B,"et"al."Oncostatin"M"drives"intestinal"inflammation"and"predicts"response"to"tumor"necrosis"factor-neutralizing"therapy"in"patients"with"inflammatory"bowel"disease[J]."Nat"Med,"2017,"23(5):"579–589.
[11] VERSTOCKT"B,"VETRANO"S,"SALAS"A,"et"al."Sphingosine"1-phosphate"modulation"and"immune"cell"trafficking"in"inflammatory"bowel"disease[J]."Nat"Rev"Gastroenterol"Hepatol,"2022,"19(6):"351–366.
[12] SAHA"K,"SUBRAMENIUM"GANAPATHY"A."Autophagy"reduces"the"degradation"and"promotes"membrane"localization"of"occludin"to"enhance"the"intestinal"epithelial"tight"junction"barrier"against"paracellular"macromolecule"flux[J]."Crohns"Colitis,"2023,"17(3):"433–449.
[13] DEKRIJGER"M,"WILDENBERG"M,"MOOKHOEK"A,"et"al."Expression"of"MAdCAM-1"and"gut-homing"T"cells"in"inflamed"pouch"mucosa[J]."Crohns"Colitis,"2021,"15(9):"1491–1499.
[14] UCHIYAMA"K,"TAKAGI"T,"MIZUSHIMA"K,"et"al."Mucosal"addressin"cell"adhesion"molecule"1"expression"reflects"mucosal"inflammation"and"subsequent"relapse"in"patients"with"ulcerative"colitis[J]."Crohns"Colitis,"2023,"17(5):"786–794.
[15] BERGEMALM"D,"ANDERSSON"E,"HULTDIN"J,"et"al."Systemic"inflammation"in"preclinical"ulcerative"colitis[J]."Gastroenterology,"2021,"161(5):"1526–1539.
[16] MCGOVERN"D,"GARDET"A,"TÖRKVIST"L,"et"al."Genome-wide"association"identifies"multiple"ulcerative"colitis"susceptibility"loci[J]."Nat"Genet,"2010,"42(4):"332–337.
[17] CASTRO"T,"DENNISON"T,"FERDINAND"J,"et"al."Anti-commensal"IgG"drives"intestinal"inflammation"and"type"17"immunity"in"ulcerative"colitis[J]."Immunity,"2019,"50(4):"1099–1114.
[18] ZHENG"K,"JIA"J,"YAN"S,"et"al."Paeoniflorin"ameliorates"ulcerative"colitis"by"modulating"the"dendritic"cell-"mediated"TH17/Treg"balance[J]."Inflammopharmacology,"2020,"28(6):"1705–1716.
[19] SASSON"S,"SLEVIN"S,"CHEUNG"V,"et"al."Interferon-"gamma-producing"CD8+"tissue"resident"memory"T"cells"are"a"targetable"hallmark"of"immune"checkpoint"inhibitor-nbsp;colitis[J]."Gastroenterology,"2021,"161(4):"1229–1244.
[20] THOMAS"S,"METZKE"D,"SCHMITZ"J,"et"al."Anti-"inflammatory"effects"of"saccharomyces"boulardii"mediated"by"myeloid"dendritic"cells"from"patients"with"Crohn’s"disease"and"ulcerative"colitis[J]."Am"J"Physiol"Gastrointest"Liver"Physiol,"2011,"301(6):"1083–1092.
[21] JERALA"M,"HAUPTMAN"N,"KOJC"N,"et"al."Expression"of"fibrosis-related"genes"in"liver"and"kidney"fibrosis"in"comparison"to"inflammatory"bowel"diseases[J]."Cells,"2022,"11(3):"314.
(收稿日期:2024–07–04)
(修回日期:2024–10–28)
