基于全基因组重测序技术对平菇白色变异菌株的鉴定及遗传变异研究
2024-11-07夏会楠郝刘斌田雨李海康王春霞郑素月



摘要:以生产上收集的一个平菇白色变异菌株(Po50)、黑色出发菌株(Po51)和一个白色生产菌株(Po9)为材料,采用拮抗、酯酶同工酶和全基因组重测序方法对3个菌株进行鉴定。拮抗和酯酶同工酶结果表明,白色变异菌株和黑色出发菌株之间无明显拮抗和酶谱差异,与生产平菇白色菌株差异较大;进一步通过全基因组重测序技术鉴定结果表明,3个菌株鉴定出大量的SNP(单核苷酸多态性位点)、InDel(插入缺失位点)、SV(结构变异位点)。平菇白色变异菌株Po50检测到SNP、InDel突变总数为1 504 391个,生产菌株Po9检测到SNP、InDel突变总数为1 371 818个,黑色出发菌株Po51检测到SNP、InDel突变总数为1 501 877个,通过生物信息手段分析白色变异菌株黑色出发菌株以及对照菌株Po9基因组间的结构差异,获得遗传变异图谱,可以对变异菌株进行快速精准鉴定。
关键词:平菇;变异菌株;全基因组重测序技术;酯酶同工酶
中图分类号:S646.1+40.1 文献标志码:A
文章编号:1002-1302(2024)18-0041-09
收稿日期:2023-09-20
基金项目:河北省现代农业产业技术体系食用菌创新团队建设专项资金(编号:HBCT2023090207);河北省农业科技成果转化项目(编号:202360101010014)。
作者简介:夏会楠(1998—),女,河北承德人,硕士,从事食用和药用真菌研究。E-mail:2199797807@qq.com。
通信作者:郑素月,博士,教授,硕士生导师,主要从事食用菌遗传育种研究。E-mail:zhengsuyue@sina.com。
平菇(Pleurotus ostreatus)栽培广泛,含有人体所需的大量生物活性物质、矿物质和维生素,具有很高的营养价值。由于其栽培技术简单、生长周期短、收益快、适应性强,为食用菌生产主栽品种之一。平菇产业中存在的主要问题是育种工作落后,品种单一,生产缺乏优良品种,因此平菇新品种种质资源选育十分重要,经常规鉴定后在分子层面进行精准分析,有利于更方便快捷地选育新品种。聂兴华等以野生板栗为试验材料,通过重测序技术确定46份野生板栗的分类,发现野生板栗和栽培板栗属于同种,为我国野生板栗提供了更准确的边界范围[1]。沈秀芬等通过对5个香菇进行重测序,分析这些菌株中的插入/缺失位点开发InDel标记,可将44个香菇菌株分为4个亚群[2]。侯炳豪等以铁观音茶树为试验材料,通过重测序技术对铁观音无性繁殖后代进行遗传差异研究,根据重测序数据得出在全基因组和编码区范围内,各个样本之间总变异位点数均差别不大,各样本中涉及突变的基因数基本一致,推测铁观音品种的种性在无性繁殖过程中总体保持稳定[3]。
全基因组重测序技术对已知基因序列的物种进行不同个体间的基因组重测序,并在此基础上对个体进行差异性分析[4],通过序列对比,在平菇基因组水平上开发大量分子标记如鉴定食用菌中大量的SNP单核苷酸多态性位点、InDel插入缺失位点、SV结构变异位点[5-6],通过生物信息手段,分析平菇白色变异菌株、栽培菌株Po9以及黑色出发菌株基因组间的结构差异,获得遗传变异图谱。全基因组重测序技术有助于快速发现重要性状的遗传变异以及对变异菌株精准鉴定[7-8]。
1 材料与方法
1.1 试验材料
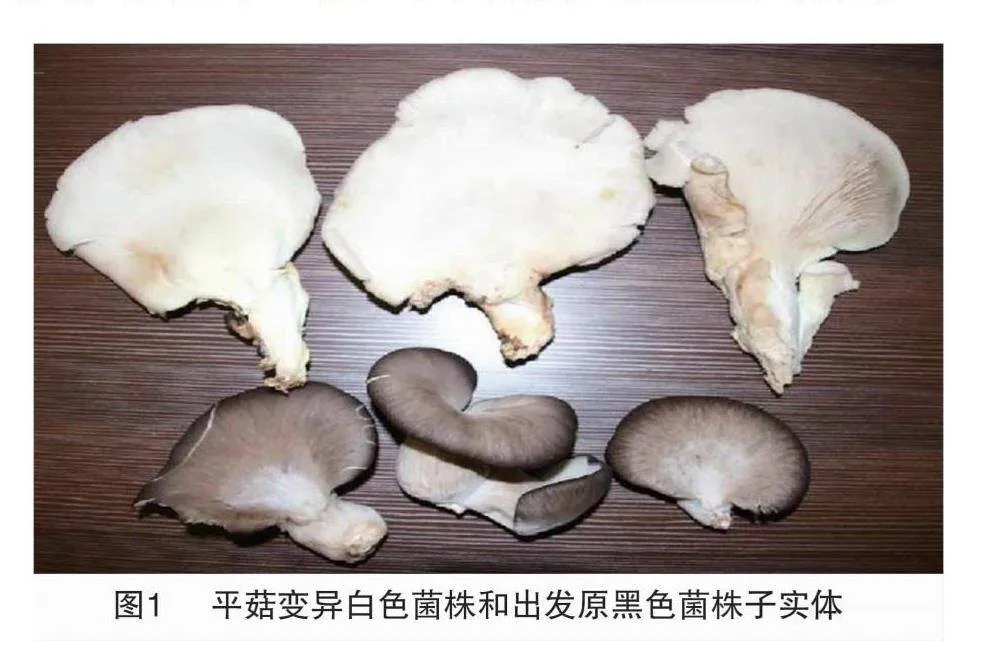
试验于2023年2—5月在河北工程大学食用菌研究室内进行,在平菇系统选育过程中,发现变异白色平菇菌株1个,变异的白色子实体和对应的出发菌株形态见图1。对采集的白色变异菌株子实体和黑色出发菌株子实体进行组织分离,获得纯菌种,白色变异菌株编号为Po50,出发黑色菌株编号为Po51。生产上栽培的白色平菇编号为Po9,以上材料均由河北工程大学食用菌实验室分离保藏。
1.2 试验方法
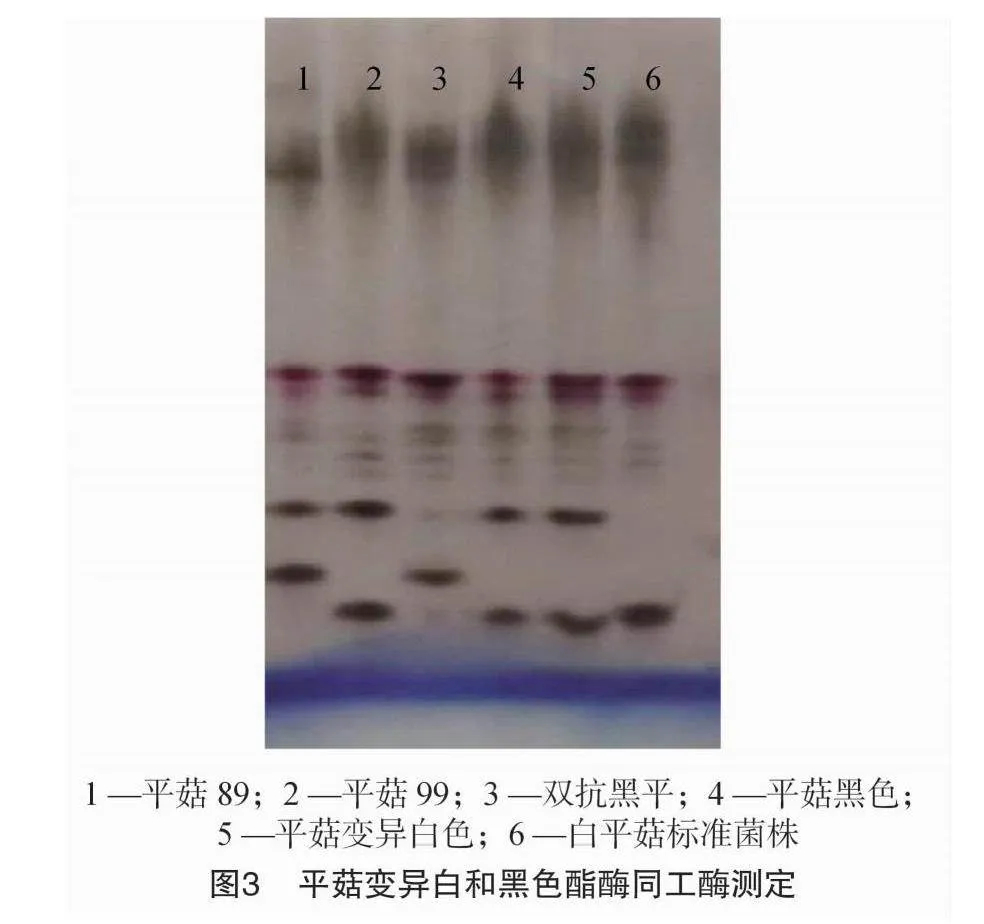
1.2.1 拮抗和酯酶同工酶测定 以平菇89、平菇99、双抗黑平以及生长上栽培的白平菇Po9作为对照菌株,对白色变异菌株和黑色出发菌株进行拮抗和酯酶同工酶测定[9]。
1.2.2 全基因组重测序技术
对平菇白色变异菌株Po50、黑色出发菌株Po51和生产对照白色菌株Po9进一步进行基因组重测序技术,文库构建和重测序数据均由北京诺禾致源科技股份有限公司提供。
1.2.3 SNP/InDel检测及注释统计
采用GATK 4.0.9.0软件进行MarkDuplicates、BaseRecalibrator处理后,再用GATK软件中的HaplotypeCaller进行SNP、InDel检测,并对变异位点进行质控标记。
2 结果与分析
2.1 白色变异菌株亲缘关系鉴定
拮抗测定结果表明,平菇白色菌株Po50与出发黑色菌株Po51无拮抗作用,二者均与平菇99无拮抗作用(图2)。酯酶同工酶图谱显示平菇白色变异菌株与原黑色菌株及标准99菌株酯酶同工酶图谱相同,与2个标准菌株(平菇89、双抗黑平、平菇黑色)和生产栽培白平菇Po9有较大差异(图3),可以初步确定出发黑色菌株为平菇99的同物异名,而白色变异菌株在遗传上与黑色出发菌株无显著差异。
2.2 全基因组重测序技术
2.2.1 测序数据质量评估及对比
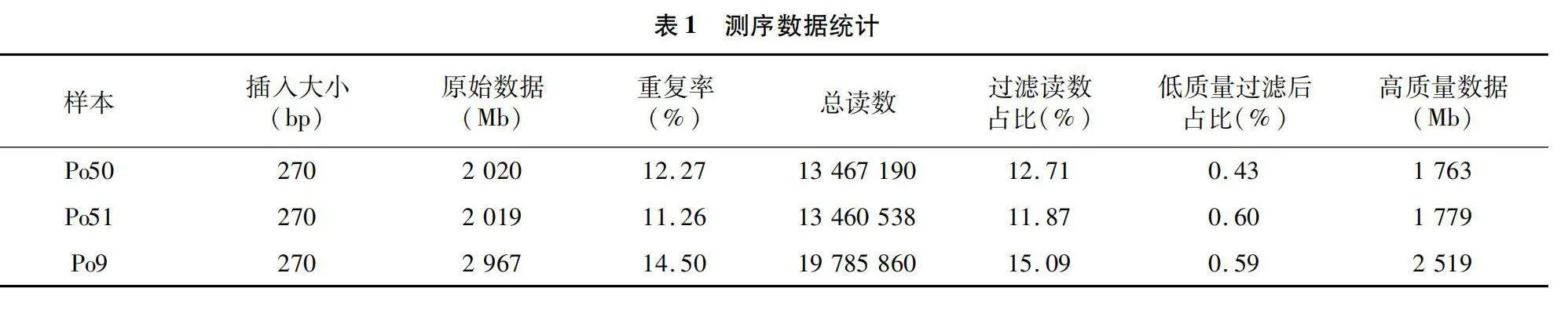
利用Illumina PE150测序,由于原始测序数据可能包含低质量序列、接头序列等,为了保证信息分析结果的可靠性,需要过滤这些杂质,从而得到高质量clean data,共获得2 020 Mb的平菇白色变异菌株、2 019 Mb的平菇黑色出发菌株、2 967 Mb的Po9基因组原始数据(raw data)。经质量评估和过滤杂质,最终平菇白色变异菌株获得1 763 Mb高质量数据 clean data、平菇黑色出发菌株获得1 779 Mb高质量数据clean data、Po9获得2 519 Mb高质量数据clean data,采用测序平台对样品进行测序,数据统计见表1。
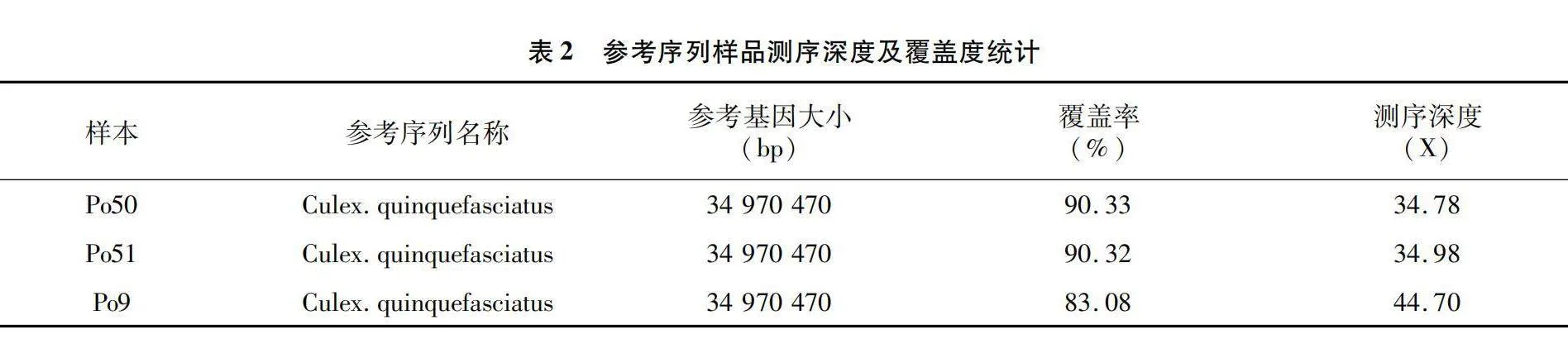
参考基因组对比结果将有效的测序数据通过BWA对比参考基因组得到最初对比结果,利用最初对比结果进行mapping率、duplication等的统计。重测序数据与参考数据样品对比平菇白色变异菌株Po50检测到的reads数目为11 754 734个,黑色平菇Po51共检测到reads数目为11 862 522个,Po9菌株共检测到16 799 756个 reads。整体对比率分别为80.48、81.11、75.83,覆盖率分别为90.33%、90.32%、83.08%(表2)。
2.2.2 SNP及InDel突变检测
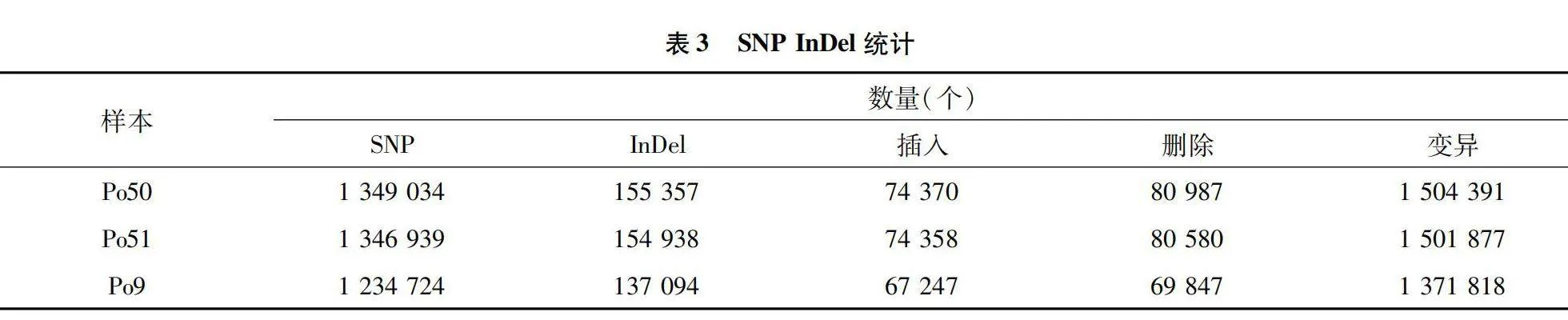
随着食用菌基因组的公布,通过全基因组重测序以及生物技术挖掘平菇基因组SNP标记简便快捷,SNP主要是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性。SNP所表现的多态性只涉及到单个碱基的变异,这种变异可由单个碱基的转换(transition)或颠换(transversion)所引起。将所测得的数据采用GATK软件进行MarkDuplicates、BaseRecalibrator处理后,在利用GATK软件中的HaplotypeCaller进行SNP检测,最后在对检测的结果进行质量过滤。最终得到SNP、InDel统计结果(表3),分别检测到突变总数为1 504 391、1 501 877、1 371 818个。
2.2.3 SNP注释
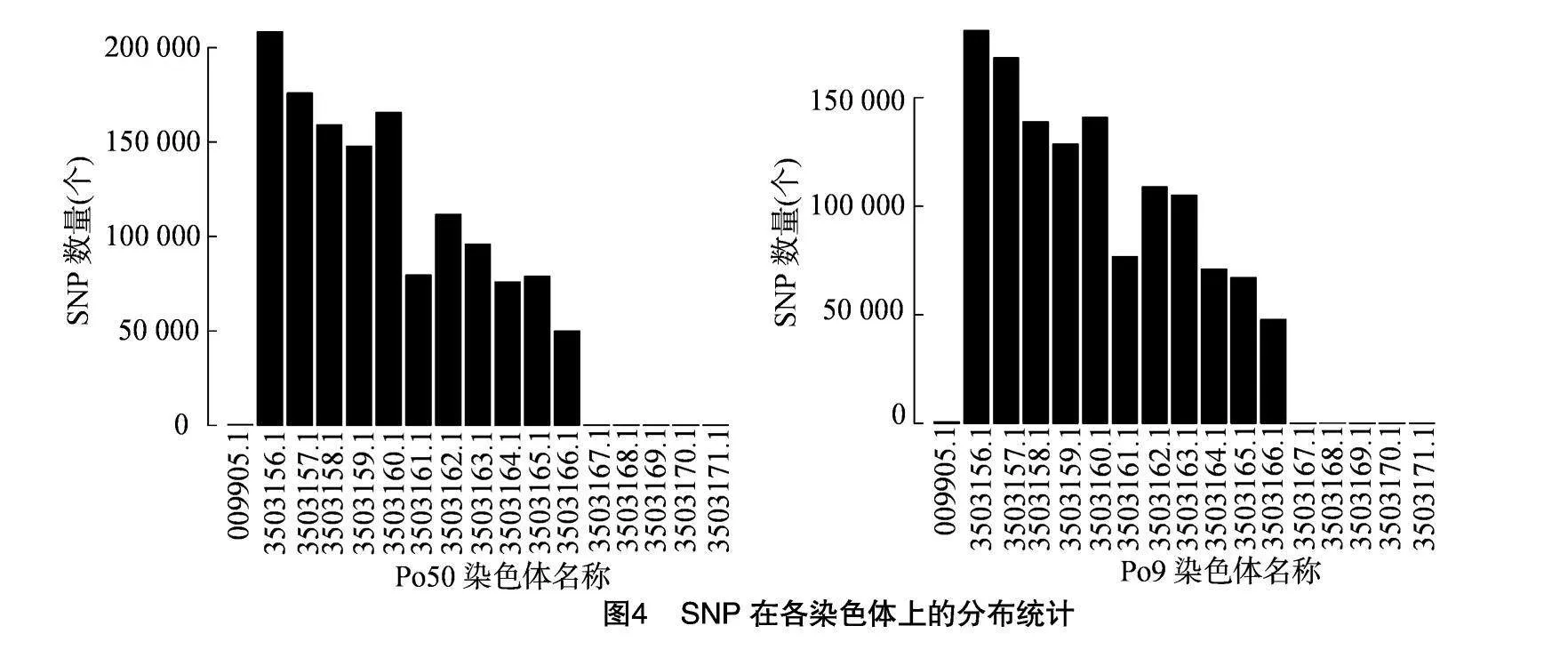
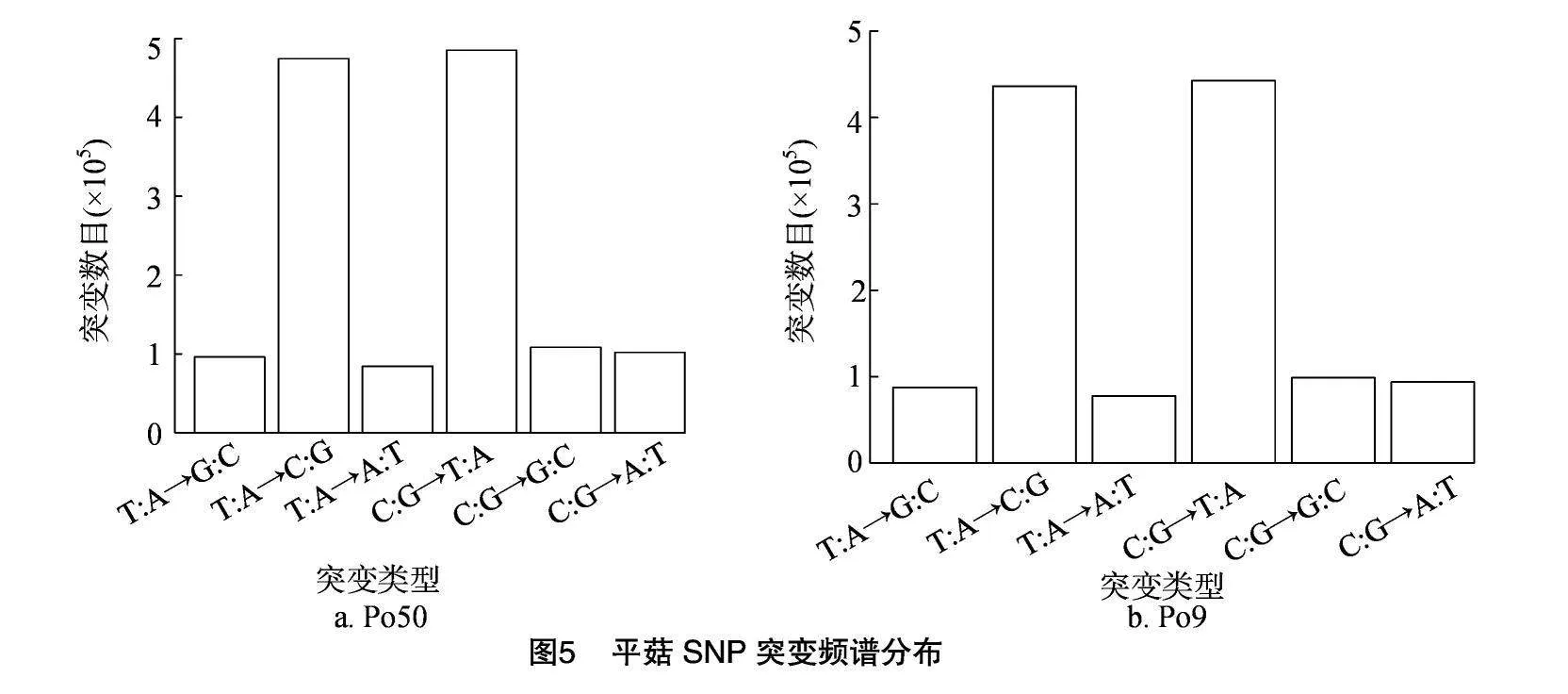
采用使用ANNOVAR对检测到的SNP进行注释,可对变异组合进行多层次的组合筛选。SNP在各染色体上分布统计见图4,Po50在染色体3503156.1上SNP数目最多,其次是染色体3503157.1、3503160.1、3503158.1,在染色体3503167.1、3503168.1、3503169.1、3503170.1、3503171.1、009905.1上SNP数目最少。Po9在染色体3503156.1、3503157.1上 SNP数目最多,在染色体3503167.1、3503168.1、3503169.1、3503170.1、3503171.1、009905.1上SNP数目最少。通过突变频谱分析可以直观看出点突变包含6种类型:T∶A→G∶C,T∶A→C∶G,T∶A→A∶T,C∶G→T∶A,C∶G→G∶C 和 C∶G→A∶T,在Po51、Po50、Po9中各种突变类型的比例存在某种突变类型的偏好性,其中T∶A→C∶G、C∶G→A∶T类型的突变较多,突变频谱分布见图5。
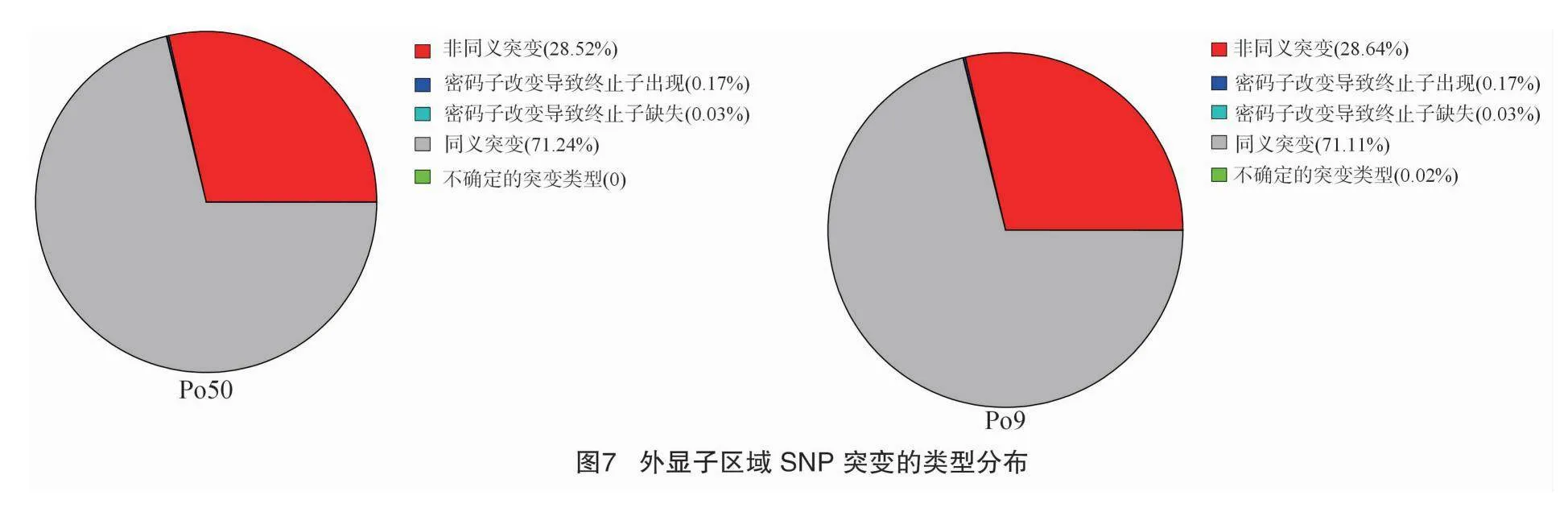
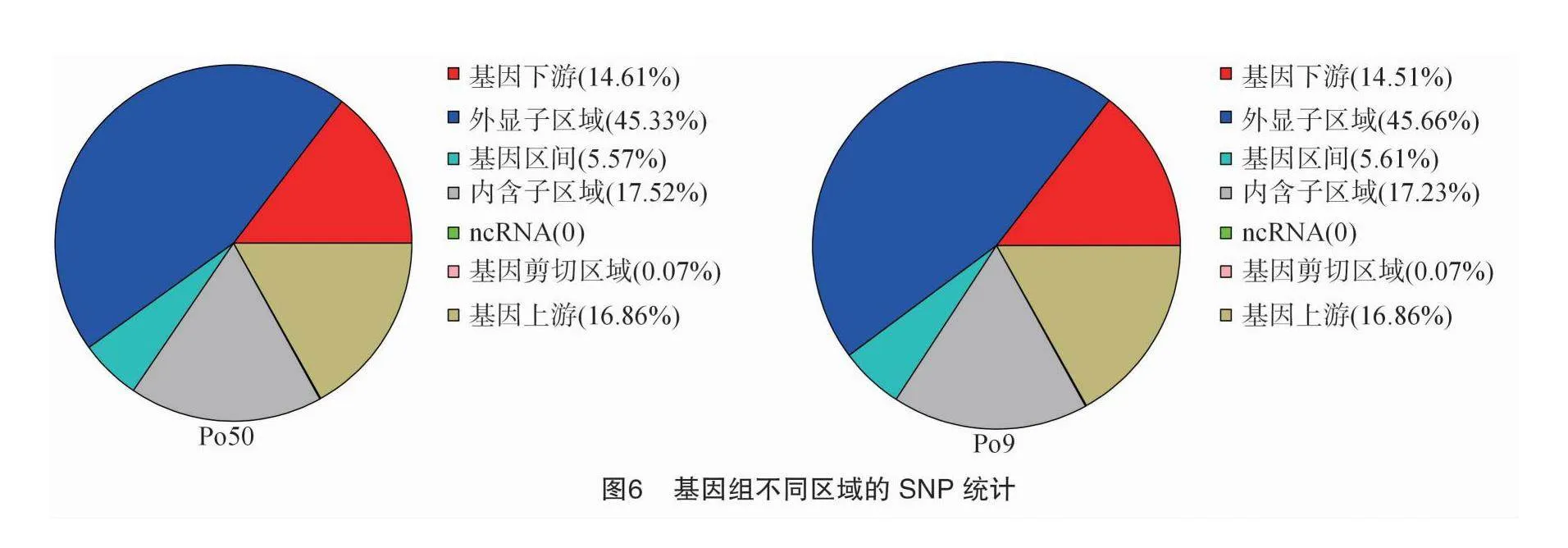
根据ANNOVAR的注释结果,SNP在基因组各区域分布统计见图6,SNP变异主要集中于发生在外显子区域,比e71d63d306b1d50358201d0558687f6d4af4b42aab7079ad363ff0e68f703025例在45.33%~45.66%,发生在基因下游的SNP变异比例在14.51%~14.61%,基因上游、基因区间、内含子区域、ncRNA区域、基因剪切区域比例分别占16.86%、5.57%~5.61%、17.23%~17.52%、0、0.07%。其中,在外显子区域发生同义突变的SNP比例为71.11%~71.24%,非同义突变为28.52%~28.64%,使基因转录提前终止突变为0.17%,失去终止密码子突变为0.03%(图7)。
2.2.4 InDel注释及统计
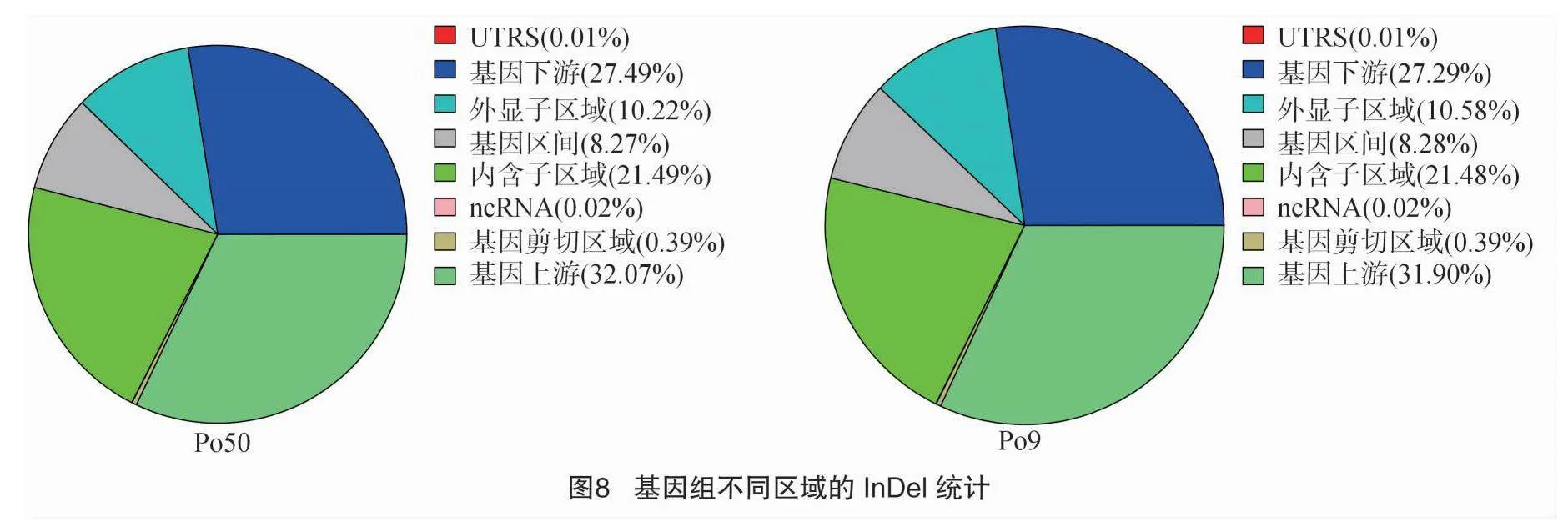
利用 ANNOVAR 对检测出的 InDel 进行注释,InDel突变检测与注释方法与SNP检测注释方法一致。 InDel在基因组各区域分布统计见图8,InDel变异主要集中于发生在基因上游外显子区域,比例在31.90%~32.07%;发生在基因下游的 InDel变异比例在27.29%~27.49%,基因区间、内含子区域、ncRNA区域、基因剪切区域比例分别占8.27%~8.28%、21.48%~21.49%、0.02%、0.39%。总体来说,InDel突变在
全基因组范围内的表现与SNP突变基本一致。在外显子区域内,移码缺失突变所占比例最高,占33.98%~34.20%,移码插入突变占外显子区域突变总数的27.48%~28.24%,非移码插入突变占17.56%~18.63%(图9)。
在基因外显子区域的SNP(InDel)位点中,部分位点导致基因编码中氨基酸改变,而引起基因产物的突变,可能会影响平菇基因的生物学功能。
3 平菇差异基因的GO功能富集分析
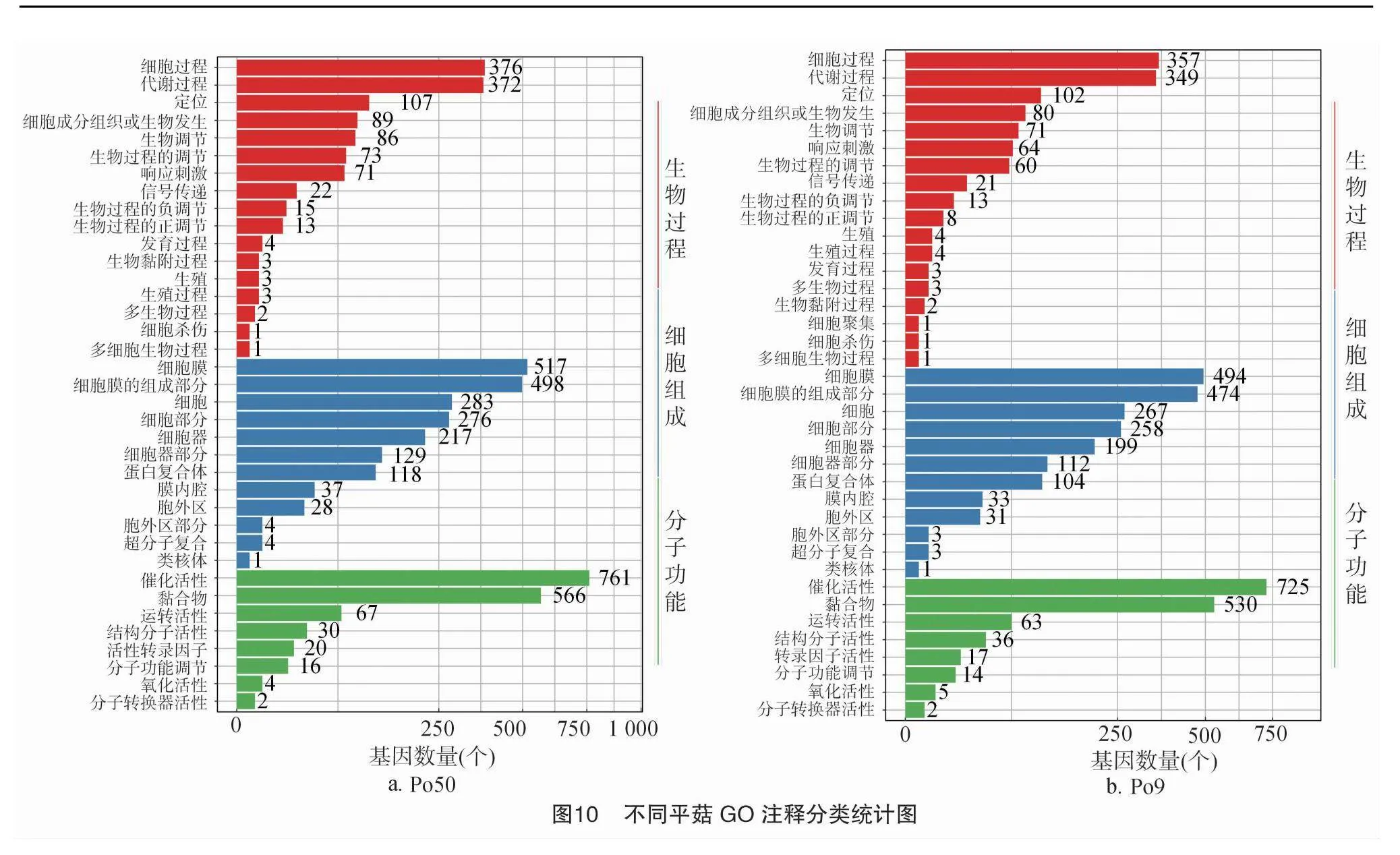
使用BLAST2GO v2.5对所测Po50、Po9基因进行GO功能注释(图10),Po50共注释到5 760个基因,注释到37个类别,主要包括生物过程(biological process,BP)、细胞组成(cellular component,CC)、分子功能(molecular function,MF)3个类别,其中CC数量最多,注释到12个条目,总数为2 112个,最多的集中在细胞膜,占总数的24.5%,数量达到517个,其次是细胞和细胞部分(GO:0009696),分别占总数的13.4%和13.1%,数量为283个及276个;MF注释8条,共有1 466个基因,最多的是催化活性部分(GO:0000155),占总数的51.9%,数量为761个,黏合物(GO:0046872)较少于活性催化部分,占总数的38.6%,数量为566个。其次是运转活性、结构分子活性、活性转录因子,分别占总数的4.6%、2.0%、1.4%;氧化活性、分子转换器活性最少,分别占总数的0.3%、0.1%。
Po9共注释到5 386个基因,注释到38个类别,主要包括生物过程、细胞组成、分子功能3个类别,其中CC数量最多,注释到12个条目,总数为1 979个,占3个类别总数的43.8%。最多的集中在细胞膜(GO:0016021),占总数的25.0%,数量达到494个:细胞膜组成部分(GO:000021)少于细胞膜,占总数的24.0%,数量为474个,其次是细胞、细胞部分。MF注释8条,共有1 392个基因,最多的是催化活性部分(GO:0000155),占总数的52.1%,数量为725个,黏合物(GO:0046872)较少于活性催化部分,占总数的38.1%,数量为530个。其次是转录活性,占总数的4.5%,数量为63个。在生物过程中Po9较Po50有1个基因注释到细胞聚集类别中,生物过程及分子功能Po9涉及的基因数量,与Po50基本一致。
分别筛选出Po50、Po9的差异基因,将差异基因向GO数据库映射,进行分析,主要富集在生物过程、分子功能和细胞组成相关的3个 GO 类别中,Po50、Po9在生物过程中参与细胞过程上调表达基因分别有372个和349个,代谢过程上调表达基因分别有357个和376个;在细胞组分过程中细胞膜上调表达基因分别有517个和494个,细胞膜组成过程中上调表达基因分别有498个和474个;在分子功能中催化活性上调表达基因分别有761个和725个,黏合物上调表达基因分别有566个和494个(图11)。
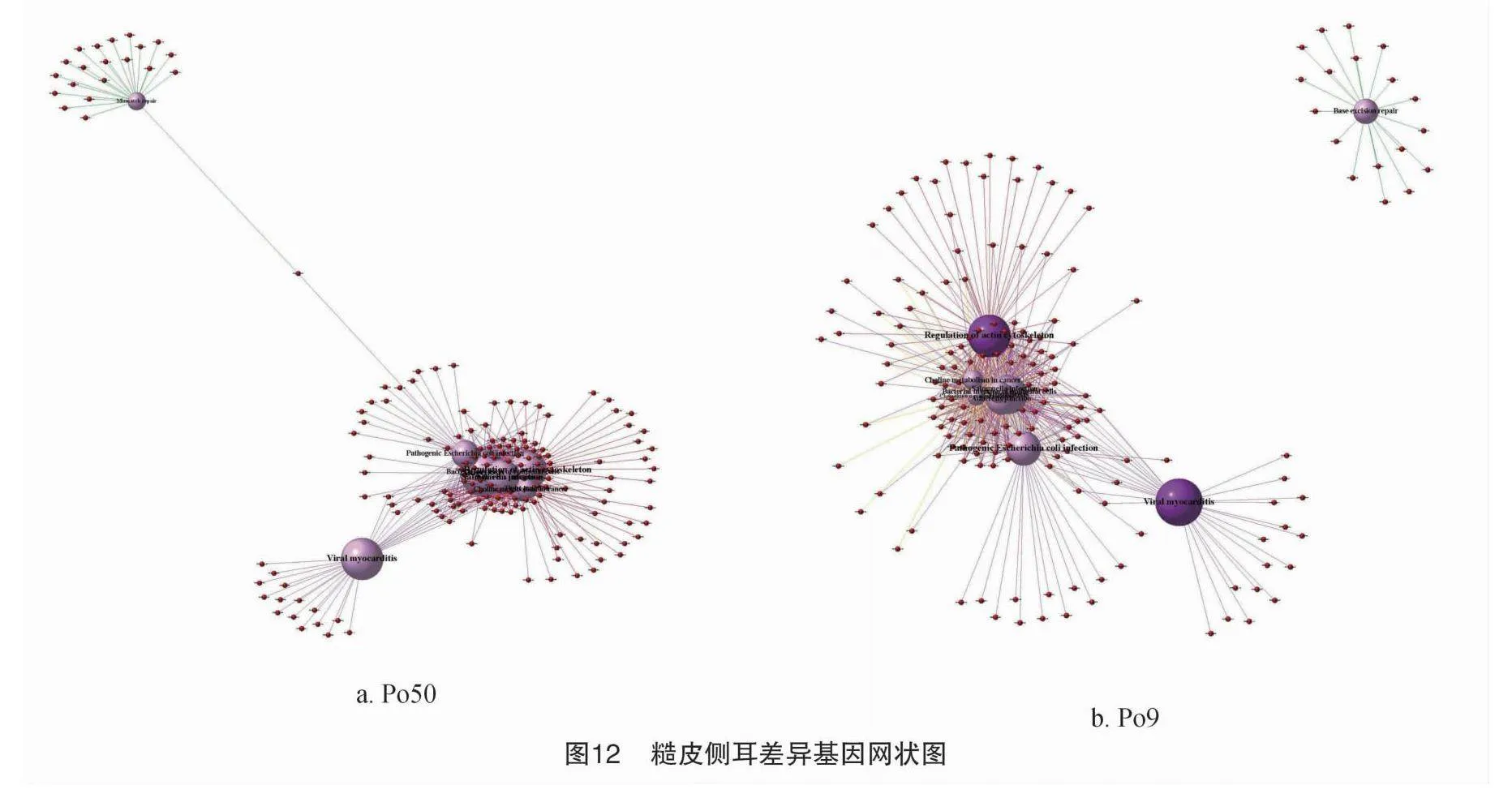
3.1 不同差异基因indel网状图
Po50选择10个显著的代谢通路如图12所示:肌动蛋白细胞骨架调控(regulation of actin cytoskeleton)由PC9H_000055等102个基因控制;志贺菌病的致病过程(shigellosis)由PC9H_000055等90个基因控制;致病性大肠杆菌感染(pathogenic escherichia colii nfection)由PC9H_000055等92个差异基因控制;错配修复(mismatch repair)由PC9H_bMkmRpjPmsYcWVy01BjqIt0/ln7g3cTLk74CKl8Qbm8=000717等21个差异基因控制。附着连接(adherens junction)由PC9H_000055等67个差异基因控制;表皮细胞细菌入侵(bacterial invasion of epithelial cells)由PC9H_000055等71个差异基因控制;癌症中的胆碱代谢(choline metabolism in cancer)由PC9H_000055等76个差异基因控制,其中还有紧密连接(tight junction)、沙门氏菌感染(salmonella infection)病毒性心肌炎(viral myocarditis)是个显著通路关系网状图。
Po9选择10个显著的代谢通路:沙门氏菌感染由PC9H_000055等68个差异基因控制;肌动蛋白细胞骨架调控由PC9H_000055等101个差异基因控制;志贺菌病的致病过程由PC9H_000055等84个基因控制;附着连接由PC9H_000055等65个差异基因控制;病毒性心肌炎由PC9H_000271等30个差异基因控制;癌症中的胆碱代谢由PC9H_000055等71个差异基因控制;致病性大肠杆菌感染,由PC9H_000249等89个差异基因控制;表皮细胞细菌入侵由PC9H_000249等67个差异基因控制;趋化因子信号通路由PC9H_000249等67个差异基因控制。
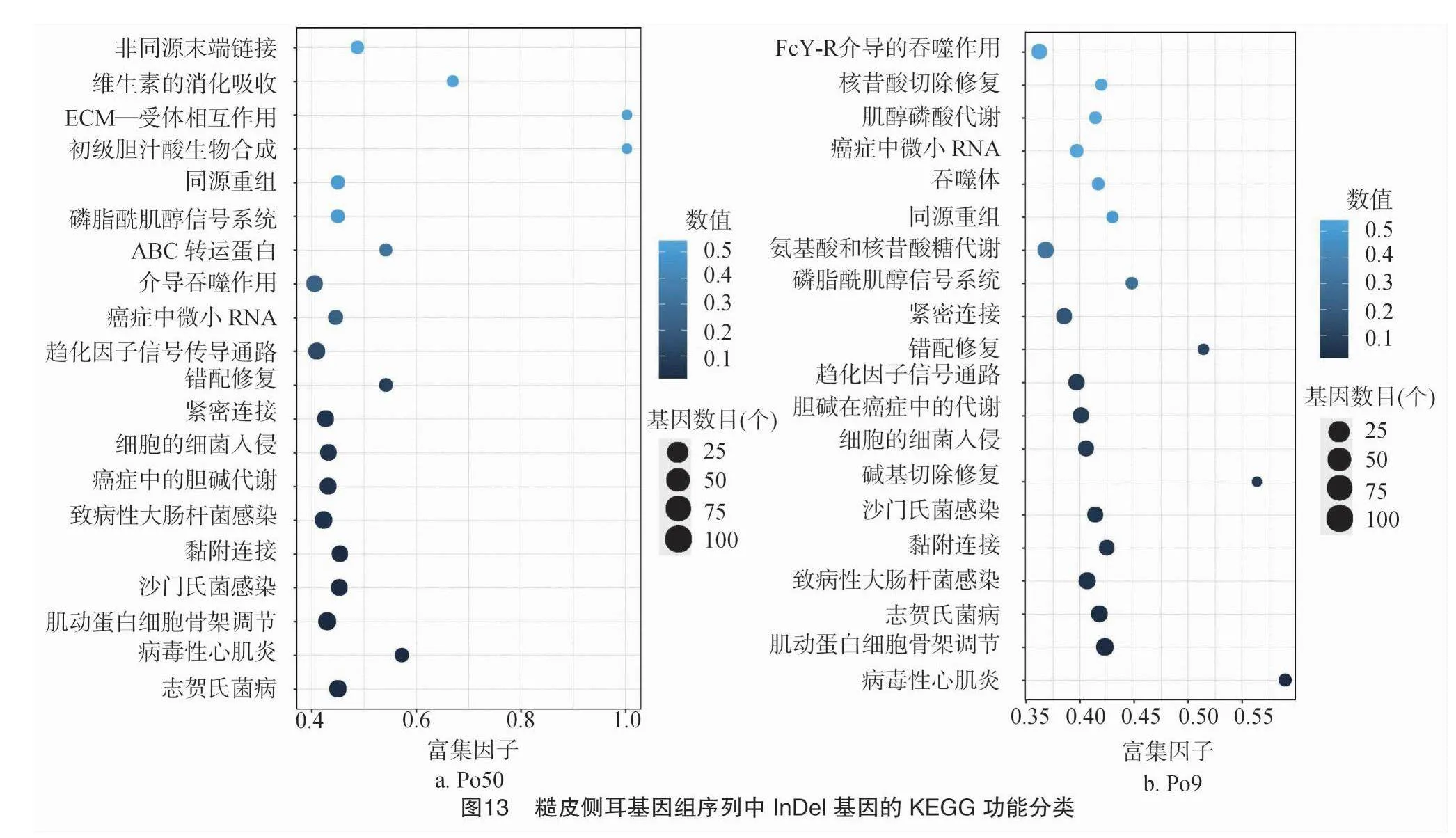
3.2 不同糙皮侧耳差异基因的KEGG代谢通路富集分析
将糙皮侧耳差异基因通过KEGG数据库进行Pathway分析,选取KEGG数据库里的真菌类,选取显著的通路制作散点图。对所得到的转录本进行验证和注释并筛选出的差异基因进行富集通路注释。将生物代谢通路分为6个类别:细胞过程(cellular processes)、生物体系统(organismal systems)、环境信息处理(environmental information processing)、遗传信息处理(genetic information processing)、人类疾病(human diseases)、新陈代谢(metabolism)。这些差异基因被分配到7个第1层级通路途径第2层44个KEGG通路途径,其中差异基因大部分富集在新陈代谢、疾病、生物体系统以及细胞过程上,其次是生物系统以及环境信息处理上,其中氨基酸和核苷酸糖代谢、紧密连接、白细胞骨架调节和附着连接在新陈代谢、细胞过程占主导地位(图13)。
4 讨论与结论
全基因组重测序已经广泛应用到人类、动物、植物的基因组和转录组分析中[9-12],然而全基因组重测序技术在食用菌中应用较少,通常是应用重测序技术进行ISSR分子标记的开发,在SNP以及InDel标记研究较少[13-15]。
本试验通过对白色变异菌株进行全基因组重测序从基因组水平精准快速地挖掘了大量的单核苷酸多态性位点SNP,插入缺失位点InDel分析不同个体间的结构差异。Illumina PE150测序平菇白色变异菌株获得 1 763 Mb高质量数据clean data、平菇黑色出发菌株获得 1 779 Mb高质量数据 clean data、Po9获得2 519 Mb高质量数据。在进行全基因组测序时还会检测到低质量的数据,这些低质量数据会影响基因组的分析,因此基因型对于基因组数据开发SNP以及InDel十分重要,所以重测序数据质量评估至关重要。重测序数据与参考数据样品对比平菇白色变异菌株Po50检测到的reads数目为11 754 734个,黑色平菇Po51共检测到reads数目为11 862 522个,Po9菌株共检测到16 799 756个 reads。分别检测到Po51 SNP突变总数1 504 391、1 501 877、1 371 818个。Po50在染色体503 156.1上SNP数目最多。通过突变频谱分析发现其中T∶A→C∶G、C∶G→A∶T类型的突变较多,SNP变异主要集中于发生在外显子区域。InDel变异主要集中于发生在基因上游外显子区域。Po50、Po9测基因进行GO功能注释,Po50共注释到5 760个基因,注释到38个类别,其中CC数量最多,注释到12个条目,总数为2 433个。将糙皮侧耳差异基因通过KEGG数据库进行Pathway分析,可将生物代谢通路分为6个类别:细胞过程、生物体系统、环境信息处理、遗传信息处理、人类疾病、新陈代谢。这些差异基因被分配到7个第1层级通路途径第2层44个KEGG通路途径[16-18]。本试验通过重测序技术对平菇白色变异菌株进行鉴定,然而定位到导致颜色变异的基因还有待进一步研究。
参考文献:
[1]聂兴华,张 煜,刘 松,等. 基于基因组重测序的野生板栗遗传特征和分类地位研究[J]. 园艺学报,2023,50(8):1622-1636.
[2]沈秀芬,章炉军,张美彦,等. 利用InDel标记分析中国香菇菌株的遗传多样性与群体结构[J]. 菌物学报,2021,40(9):2266-2281.
[3]侯炳豪,高 婷,魏月德,等. 基于高深度基因组重测序的‘铁观音’茶树无性繁殖后代遗传变异研究[J]. 园艺学报,2023,50(7):1505-1517.
[4]Zhao W,Fan L,Wu W J,et al. Re-sequencing and transcriptomic analysis reveal differential expression patterns and sequence variation in glucosyltransferase gene related to anthocyanin biosynthesis in walnut (Juglans regia L.)[J]. Scientia Horticulturae,2023,317:112077.
[5]Zhang Q P,Zhang Y P,Liu W S,et al. Re-sequencing and morphological data revealed the genetics of stone shell and kernel traits in apricot[J]. Frontiers in Plant Science,2023,14:1196754.
[6]Kim J Y,Hwang J E,Eo S H,et al. Development of InDel markers for interspecific hybridization between hill pigeons and feral pigeons based on whole-genome re-sequencing[J]. Scientific Reports,2022,12(1):22618.
[7]Shan T F,Li Y Q,Pang S J. Identification of a genomic region linked with sex determination of Undaria pinnatifida (Alariaceae) through genomic resequencing and genetic linkage analyses of a segregating gametophyte family[J]. Journal of Phycology,2023,59(1):193-203.
[8]Cheng Q,Sun L,Qiao H,et al. Loci underlying leaf agronomic traits identified by re-sequencing celery accessions based on an assembled genome[J]. iScience,2022,25(7):104565.
[9]刘晓雪,王 强,张彬彬,等. 基于酯酶和ISSR技术的平菇单核菌株遗传多样性分析[J]. 江苏农业科学,2022,50(9):27-32.
[10]ShresthaS,Fu Y Q,Michael V N,et al.ftXyIQGdctfetEbRlIEYW7VWtLLRoIhM1OdUDfBZkTI= Whole genome re-sequencing and bulk segregant analysis reveals chromosomal location for Papaya ringspot virus W resistance in squash[J]. Frontiers in Plant Science,2022,13:848631.
[11]Zhao W,Zhang Y P,Zhang J P,et al. QTL mapping by whole genome re-sequencing and analysis of candidate genes for salt tolerance in linseed (Linum usitatissmum L.)[J]. Oil Crop Science,2022,7(2):80-85.
[12]刁兴旺,吴莉君,何 红,等. 芒果炭疽病抗感品种全基因组重测序分析[J]. 江苏农业科学,2022,50(23):55-61.
[13]Qing J,Meng Y D,He F,et al. Whole genome re-sequencing reveals the genetic diversity and evolutionary patterns of Eucommia ulmoides[J]. Molecular Genetics and Genomics:MGG,2022,297(2):485-494.
[14]Song Z,Zhang Z,Dong J C,et al. Mapping immature fruit colour‐related genes via bulked segregant analysis combined with whole‐genome re‐sequencing in pepper (Capsicum annuum)[J]. Plant Breeding,2022,141(2):277-285
[15]黄平仙,高永明,刘乃新,等. 基于全基因组重测序技术分析甜菜InDel标记[J]. 中国糖料,2020,42(3):1-6.
[16]Wassana K,Pumipat T,Orarat M,等. 基于SNPs全基因组测序技术对泰国辣椒地方品种遗传多样性分析和辣椒素含量关联分析[J]. 辣椒杂志,2019,17(2):37-46.
[17]宋海岩,孙淑霞,李 靖,等. 基于SSR标记检测与重测序技术的3个李品种鉴定与遗传背景简析[J]. 中国南方果树,2023,52(3):94-101.
[18]李 淦. 基于全基因组重测序解析中国甜柿遗传多样性[D]. 武汉:华中农业大学,2022.
