珊瑚益生菌筛选及其益生特性分析
2024-10-15苏宏飞刘缘曾璐佳王姝颖黄沁愉何旭聪











苏宏飞(1985-),博士,副教授,广西大学海洋学院海洋生物与生态教研室主任,主要从事海洋生物资源多样性与功能化利用研究工作。主持国家自然科学基金项目“基于‘益生菌’假说的鹿角杯形珊瑚抗病机制研究”、广西自然科学基金项目“基于转座子突变文库的鹿角珊瑚抗病分子机制研究”“种间珊瑚微生物组移植的抗病作用及机制研究”等6项;作为主要成员参与国家自然科学基金重点项目、广西科技重大专项等国家级或省部级科研项目10余项;在《Journal of the Science of Food and Agricul-ture》《International Journal of Molecular Sciences》《南方农业学报》《热带海洋学报》等国内外权威期刊上发表学术论文30余篇,其中SCI收录论文23篇;获授权国家发明专利1项;获广西教学成果奖一等奖1项。
摘要:【目的】挖掘珊瑚潜在益生菌并明确其益生特性,为珊瑚益生菌资源库提供新材料,也为珊瑚疾病的微生物防治研究提供理论依据。【方法】分别以蛋白酶和β-葡萄糖苷酶活性筛选胞外酶产生菌株,以溶珊瑚弧菌为指示菌筛选具拮抗活性的菌株,以紫色杆菌为指示菌筛选有群体淬灭活性的菌株,通过16S rRNA测序对所获菌株进行种属鉴定,然后利用缸内添加试验验证益生菌的抗病功能,并通过全基因组分析预测益生菌的益生特性。【结果】通过原位模拟培养法,从7种珊瑚样品中共分离纯化获得271株细菌,其中,136株具有蛋白酶活性,87株具有β-葡萄糖苷酶活性,20株具有群体淬灭活性,28株对溶珊瑚弧菌有拮抗活性。既能产生胞外酶又具有拮抗活性或群体淬灭活性的菌属有芽孢杆菌属、假芽孢杆菌属、微小杆菌属、鲁杰氏菌属、鞘脂菌属、假交替单胞菌属、弧菌属、微球菌属、诺卡菌属和鞘氨醇单胞菌属。益生菌抗病作用缸内验证试验发现,芽孢杆菌GXU-Z9株能有效维持鹿角杯形珊瑚的光合效率和虫黄藻密度,保护珊瑚免受溶珊瑚弧菌引起的白化现象。基于全基因组数据分析发现,芽孢杆菌GXU-Z9株具有非常活跃的氨基酸代谢和运输能力,具备为珊瑚宿主提供必需和非必需氨基酸的潜力;通过产生对珊瑚有益的化合物(抗生素等)而抵御弧菌感染,并通过降解纤维类或蛋白类颗粒物质来补充珊瑚能量需求;同时,其全基因组中包含多种氨基酸生物合成相关基因及B族维生素合成相关基因,具有为珊瑚宿主提供氨基酸和维生素以获得能量的潜力。【结论】芽孢杆菌GXU-Z9株能有效维持鹿角杯形珊瑚的光合效率和虫黄藻密度,展现出良好的抗病功能;芽孢杆菌GXU-Z9株全基因组中含有编码几丁质酶和β-葡萄糖苷酶的相关基因,能大量合成珊瑚的必需氨基酸和维生素等营养物质,具有辅助珊瑚消化碳水化合物的酶类物质、提供氨基酸和维生素等益生特性。
关键词:珊瑚;益生菌;芽孢杆菌属;拮抗活性;群体淬灭活性;益生特性
中图分类号:S917.1文献标志码:A文章编号:2095-1191(2024)07-1873-14
Screening of coral probiotics and their probiotic characteristics
SU Hong-fei LIU Yuan ZENG Lu-jia WANG Shu-ying HUANG Qin-yu HE Xu-cong
(1Coral Reef Research Center,Guangxi University/Guangxi Laboratory on the Study of Coral Reefs in the South ChinaSea,Nanning,Guangxi 530004,China;2School of Marine Sciences,Guangxi University,Nanning,Guangxi 530004,China;3School of Resources,Environment and Materials,Guangxi University,Nanning,Guangxi 530004,China)
Abstract:【Objective】This study investigated potential probiotics in coral and elucidated their probiotic characteris-tics,thereby contributing novel materials to the coral probiotic resource library and offering theoretical foundation formi-crobial control research in coral disease management.【Method】Extracellular enzyme producing strains were screened using protease andβ-glucosidase activities,antagonistic active strains was screened with Vibrio coralliilyticus as indicatorstrain,quorum quenching active strains was screened with Chromobacteriumviolaceum as indicator strain,and strainidentification on species and genus was performed via 16S rRNA sequencing.Then,the anti-disease function of probiotics was verified by in-tank addition test,and the probiotic properties of probiotics were predicted by whole genome analysis.【Result】A total of 271 bacterial strains were isolated and purified from 7 coral samples using an in situ simulated cultiva-tionmethod.Activity screening indicated that 136 strains exhibited protease activity,87 strains exhibitedβ-glucosidase ac-tivity,20 strains exhibited quorum quenching activity,and 28 strains exhibited antagonistic activity against V.coralliilyti-cus.The bacterial genera capable of producing extracellular enzymes and displaying antagonistic or quorum quenching ac-tivities included Bacillus sp.,Fictibacillus sp.,Exiguobacterium sp.,Ruegeria sp.,Sphingobium sp.,Pseudoalteromo-nas sp.,Vibrio sp.,Micrococcus sp.,Nocardia sp.,and Sphingomonassp.The in-tank validation test of probiotic diseaseresistance demonstrated that the Bacillus sp.GXU-Z9 strain could effectively sustain the photosynthetic efficiency and zooxanthellae density of Pocilloporadamicornis,thereby protecting the coral from bleaching induced by V.coralliilyti-cus.Whole genome data analysis revealed that Bacillus sp.GXU-Z9 strain possessed highly active amino acid metabolism and transport capabilities,indicating its potential to supply both essential and non-essential amino acids to coral hosts.Ad-ditionally,it could produce compounds beneficial to corals,such as antibiotics,which helped in resisting Vibrio infec-tions,and could supplement the coral’s energy requirements by degrading fibrous or protein particulate matter.Concur-rently,its genome encompassed multiple genes associated with amino acid biosynthesis and B Vitamin synthesis,which had the potential to supply essential amino acids and vitamins to the coral hosts,thereby facilitating their energy acquisi-tion.【Conclusion】Bacillus sp.GXU-Z9 strain can effectively sustain the photosynthetic efficiency and zooxanthellae den-sity of P.damicornis,exhibiting remarkable disease resistance.The whole genome of Bacillus sp.GXU-Z9 strain includes genes encoding chitinase andβ-glucosidase,enabling the synthesis of essential amino acids and vitamins in substantial amounts.These capabilities assist the coral in digesting carbohydrate enzymes and the provision of amino acids and vita-mins.
Key words:corals;probiotics;Bacillus sp.;antagonistic activity;quorum quenching;probiotic properties
Foundation items:Youth Science Fund Project of National Natural Science Foundation of China(41706158);Guangxi Natural Science Foundation(2023GXNSFAA026388)
0引言
【研究意义】近年来,因病害引起的珊瑚白化现象逐年攀升,且由于气候变化和人类活动的干扰,如沿海开发、海水富营养化及过度捕捞等,珊瑚疾病的暴发或将愈发严重(Ward and Lafferty,2004)。珊瑚疾病已成为全球珊瑚礁严重退化的主要原因之一(Bourne et al.,2009)。至今,已报道了165种珊瑚的22种疾病,最常见的疾病有黑带病、白色瘟疫、白化综合征、骨骼侵蚀、黑斑病和黄带病(Morais et al.,2022)。全球主要的珊瑚礁区,包括远岸礁区和保护区都暴发过珊瑚疾病,导致珊瑚覆盖率大幅下降(Aeby et al.,2003;Williamset al.,2011)。以美国佛罗里达州2014年暴发的石珊瑚组织脱落病(Stonycoral tissue loss disease,SCTLD)最严重(Spadafore et al.,2021),从暴发开始至2016年珊瑚密度约下降30%,活组织损失超过60%(Waltonetal.,2018)。溶珊瑚弧菌(Vibrio coralliilyticus)是引起合并感染及加剧SCTLD的病原菌(Ushijimaetal.,2020)。值得注意的是,溶珊瑚弧菌是一种温度依赖性病原菌,27℃以上溶珊瑚弧菌通过特有的毒力机制包括鞭毛运动、群体感应和毒力因子分泌,导致鹿角杯形珊瑚(Pocilloporadamicornis)发生细菌性白化(Kimes et al.,2012)。这种高传染性和致死性疾病对珊瑚群落极具破坏性及威胁性,对珊瑚礁生态系统的结构和功能产生巨大影响,因此迫切需要了解珊瑚疾病暴发的原因及传播方式,并采取合理的保护措施和有效的治疗方案。【前人研究进展】自Reshef等(2006)提出珊瑚益生菌假说并主张珊瑚通过调控自身的共附生细菌群落应对环境压力以来,越来越多学者将研究重点聚焦在益生菌对珊瑚及其共生体的影响。Peixoto等(2017)在珊瑚益生菌假说的基础上提出珊瑚有益微生物(BMCs)这一概念,同时强调了BMCs潜在的有益机制:第一,BMCs通过介导养分循环(碳循环、氮循环等)为珊瑚宿主提供能量。大多数珊瑚都依靠共生藻类光合作用产生的有机化合物提供能量,用于生长、繁殖及钙化(Davy et al.,2012)。此外,珊瑚所需的氮源主要由固氮细菌提供,通过硝化、反硝化和氨化等途径参与氮循环(Wegleyetal.,2007),尤其是珊瑚白化后更依赖于宿主主动或被动捕食海洋环境中的有机化合物碎屑(纤维素、琼脂、几丁质等),且这些化合物由珊瑚肠道微生物分解代谢后为珊瑚补充营养(Reshefetal.,2006)。第二,BMCs能通过产生抗生素或与病原体进行生态位竞争的方式抑制病原体定殖与生长。Krediet等(2013)研究发现,4种发光杆菌(Photobac-terium sp.)、1种微小杆菌(Exiguobacterium sp.)和1种弧菌(Vibrio sp.)能通过生态竞争而抑制白痘病原体——粘质沙雷氏菌(Serratia mar-cescens)的定殖与生长;Rosado等(2019)通过拮抗作用成功从鹿角杯形珊瑚中分离出能抑制溶珊瑚弧菌生长的7株菌株,其中5株隶属于假交替单胞菌属(Pseudoal-teromonas sp.),其余2株分别为盐单胞菌属(Halomo-nas sp.)和科贝特氏菌属(Cobetia sp.),且发现这些菌株组成的菌团能显著增强珊瑚对溶珊瑚弧菌和热应激的抵抗力;Kitamura等(2021)研究证实,从丛生盔形珊瑚(Galaxeafascicularis)中分离获得的鲁杰氏菌属(Ruegeria sp.)细菌能抑制溶珊瑚弧菌的体外生长。因此,对病原菌具有生长抑制活性或与其进行生态位竞争的细菌是珊瑚益生菌的重要来源。第三,细菌可通过群体感应(Quorum sen-sing,QS)监测种群密度并相应调节其基因表达,而群体淬灭(Quo-rum quenching,QQ)能干扰细菌的群体感应(Ng and Bassler,2009)。细菌群体会通过信号分子如AHL和AI等调节基因表达的模式,从而影响细菌的生物发光、定殖、毒力因子和胞外酶的产生等(Tait et al.,2010)。弧菌属(Vibrio sp.)细菌是导致多种珊瑚疾病的主要病原体,多种弧菌都能产生AHL或AI,从而通过群体感应机制来控制相关毒力基因表达并诱导珊瑚白化,且温度对其群体感应有直接影响,如溶珊瑚弧菌在高于27℃的条件下会引起鹿角杯形珊瑚白化和组织裂解(Kimes etal.,2012);施罗氏弧菌(V.shiloi)在夏季高温时会黏附在大西洋枇杷珊瑚(Oculina patagonica)的黏液上,产生肽毒素和超氧化物歧化酶,并引起珊瑚白化(Rubio-Portillo et al.,2014)。目前已发现部分细菌能通过降解群体感应信号分子而破坏细菌的群体感应及减弱毒力。AHL内酯酶和AHL酰化酶均能有效减少AHL的积累,并下调其毒力基因表达。其中,能编码AHL内酯酶的AiiA基因在芽孢杆菌属(Bacillus sp.)中广泛存在(Donget al.,2000);AHL酰化酶如PvdQ、AiiD、QuiP等已在多种细菌中被发现,包括假交替单胞菌属、节杆菌属(Arthrobacter sp.)及链霉菌属(Streptomycessp.)等(Rudrappa et al.,2008;Tang and Zhang,2014)。【本研究切入点】珊瑚共附生微生物数量庞大、多样性高,通常具有产蛋白酶、纤维素酶等胞外酶活性,以及病原菌拮抗与群体淬灭等活性,可能扮演着益生菌角色而发挥抗病功能。【拟解决的关键问题】采集海南三亚鹿回头海域(18°13′N,109°28′E)的7种珊瑚,以蛋白酶和β-葡萄糖苷酶活性筛选胞外酶产生菌株,以溶珊瑚弧菌为指示菌筛选具拮抗活性的菌株,以紫色杆菌(Chromobacteriumviolaceum)为指示菌筛选有群体淬灭活性的菌株,通过16S rRNA测序对所获菌株进行种属鉴定,利用缸内添加试验验证益生菌的抗病功能,并通过全基因组分析预测益生菌的益生特性,旨在为珊瑚益生菌资源库提供新材料,也为珊瑚疾病的微生物防治研究提供理论依据。
1材料与方法
1.1试验材料
1.1.1珊瑚样品和试验菌株珊瑚样品采集自海南三亚鹿回头海域,主要包括鹿角杯形珊瑚、美丽鹿角珊瑚(Acropora austera)、盾形陀螺珊瑚(Turbi-nariapeltata)、滨珊瑚(Porites sp.)、扁脑珊瑚(Platy-gyra sp.)、蔷薇珊瑚(Montipora sp.)和角蜂巢珊瑚(Favites sp.)。溶珊瑚弧菌购自海洋微生物菌种保藏管理中心,紫色杆菌CV12614购自美国菌种保藏中心。
1.1.2主要试剂氯化钠、丙三醇、无水乙醇和二甲苯购自国药集团化学试剂有限公司;葡萄糖、七叶苷和柠檬酸铁购自天津市风船化学试剂科技有限公司;酵母提取物和琼脂粉购自天津市北方天医化学试剂厂;蛋白胨购自天津博迪化工股份有限公司;2216E琼脂购自青岛海博生物技术有限公司;2×Easy Taq®PCRSuperMix购自北京全式金生物技术股份有限公司;蛋白酶K购自上海碧云天生物技术股份有限公司;DNA提取试剂盒购自美国MP Bio-medicals公司。
1.1.3培养基配制Marine Agar(MA)培养基:胰蛋白胨5.0 g,酵母粉1.0 g,葡萄糖0.5 g,柠檬酸铁0.1 g,一级水500.0 mL,无菌海水500.0 mL,琼脂粉10.0 g(配制固体培养基时添加),pH调至7.2~7.5。原位模拟培养基(Certner and Vollmer,2015):用0.22μm有机过滤膜将珊瑚组织过滤得到无菌珊瑚组织悬液,取总体积1/10的组织悬液加入灭菌后降温至60℃的MA固体培养基中。2216E固体培养基:2216E琼脂粉52.4 g,一级水1 L。Luria Bertani(LB)培养基:胰蛋白胨15.0 g,氯化钠15.0 g,酵母提取粉10.0 g,琼脂粉10.0 g(配制固体培养基时添加),pH调至7.4。七叶苷固体培养基:酵母提取粉15.0 g,氯化钠15.0 g,琼脂粉10.0 g,蛋白胨5.0 g,柠檬酸铁铵0.3 g,七叶苷0.1 g,一级水500.0 mL,无菌海水500.0 mL。酪蛋白固体培养基:2216E琼脂粉52.4 g,酪蛋白10.0 g,一级水1 L。2%琼脂固体培养基:琼脂粉20.0 g,一级水1 L。
1.2珊瑚共附生细菌分离培养
按照Yamamura等(2003)的蔗糖密度梯度离心法收集珊瑚微生物,具体步骤如下:(1)称取1.0 g珊瑚置于10.0 mL无菌海水中,涡旋振荡10min。(2)取10 mL的离心管,从下到上分别加入50%、40%、30%、20%和10%蔗糖溶液各1.0 mL,最上层加入1.0 mL无菌珊瑚组织悬液。(3)200 r/min离心5min后,每层吸取1.0 mL,分别稀释到10-1~10-7等7个浓度梯度,取200.0μL不同浓度的稀释液分别涂布在原位模拟培养基上,25℃培养(Certner and Vollmer,2015)。(4)挑取单菌落在原位模拟培养基上进行分区划线培养,直至获得色泽、形态单一的菌落,再将其接种于MA液体培养基中培养至对数期,转移至20%甘油管中,-80℃保存备用。
1.3珊瑚潜在益生菌筛选
1.3.1胞外酶产生菌株筛选胞外蛋白酶活性菌筛选参考Li等(2017)的方法,采用打孔器挑取1.2分离获得的单菌落,转移至酪蛋白固体培养基上,25℃培养至出现水解圈,挑取阳性菌落在MA培养基上进行分区划线培养。同样,胞外β-葡萄糖苷酶活性菌采用打孔器挑取1.2分离获得的单菌落,接种至七叶苷固体培养基上,25℃培养观察,出现黑色水解圈的菌株即为β-葡萄糖苷酶活性菌。
1.3.2拮抗活性菌株筛选参考蒋庆茹等(2012)的方法,通过平板扩散法筛选拮抗活性菌株。取200.0μL培养至OD600 nm达0.6~1.0的溶珊瑚弧菌悬液,涂布于MA固体培养基上。以无菌圆形滤纸片(直径6mm)浸泡于MA液体培养基后,置于涂布有溶珊瑚弧菌的MA固体培养基上作为空白对照;将1.2分离获得的单菌落接种至MA液体培养基中,160 r/min摇床培养至OD600 nm达0.6~1.0,用滤纸片浸泡发酵液后放置在涂布有溶珊瑚弧菌的MA固体培养基上,周围有透明抑菌圈的菌落即为拮抗活性菌株。
1.3.3群体淬灭活性菌株筛选紫色杆菌CV12614具有群体感应机制,当其浓度达到一定阈值后,会释放信号分子C6-HSL与特定转录调节蛋白结合,而调控紫色素相关基因表达(Martinelli et al.,2004)。将牛津杯均匀放置在2%琼脂固体培养基上作为第一层;将含有琼脂的LB培养基高温灭菌后降温至40℃,加入其体积10%的紫色杆菌CV12614悬液,混匀,倒在琼脂固体培养基上作为群体淬灭活性筛选培养基的第二层。冷却凝固后,取出牛津杯,将1.2分离获得的单菌落发酵液加入孔中,每孔200.0μL。29℃培养观察,周围出现透明圈即为群体淬灭活性菌株(王为善等,2009)。
1.4菌种鉴定及风险预测
采用DNA提取试剂盒提取菌株DNA,以27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R(5'-T ACGACTTAACCCCAATCGC-3')为引物,进行16SrRNA扩增。PCR反应体系30.0μL:DNA模版0.5μL,引物27F和1492R各1.0μL,2×Easy Taq®PCRSuperMix 15.0μL,ddH2O 12.5μL。扩增程序:95℃预变性5 min;94℃30 s,55℃40 s,72℃90 s,进行30个循环;72℃延伸10min。经琼脂糖凝胶电泳检测合格的PCR扩增产物,送至生工生物工程(上海)股份有限公司进行16S rRNA测序,测序结果进行BLAST比对分析,选择同源相似性最高的菌株作为候选益生菌,并在Web of Science上查阅文献评估所有菌株的致病风险。
1.5缸内验证试验
1.5.1菌株培养及计数将溶珊瑚弧菌和候选益生菌接种至MA液体培养基中,在29℃下160r/min摇床培养,使用光密度分光光度计(VarioskanTM LUX)在600 nm处测定菌液OD600 nm,带入公式计算菌液浓度(Okwadha and Li,2010)。菌液浓度(CFU/mL)=8.59×107×Z1.3627(Z为OD600 nm)。
1.5.2益生菌作用验证以鹿角杯形珊瑚为受体,试验开始前2周从健康珊瑚中截取3~5 cm长的断枝,将其固定在碳酸钙基座上,放回原缸[温度为(29±0.5)℃]进行恢复培养。设空白对照组(C)、弧菌对照组(V)和益生菌处理组(VT),每组设3个生物学重复。溶珊瑚弧菌、候选益生菌在环境中的终浓度分别为5.00×104和5.00×105 CFU/mL,根据OD600 nm和终浓度分别计算致病菌与候选益生菌的添加体积。每天观察珊瑚的表型变化。
1.5.3最大光化学效率(Fv/Fm)测定虫黄藻光合系统PSII的Fv/Fm是由最大光化学量子产额反映。黑暗中适应30 min后,采用脉冲幅度调制荧光仪(PAM)进行测定,每个珊瑚每天重复测定6次,取平均值。
1.5.4虫黄藻密度测定在15 mL离心管中加入经0.45μm滤膜过滤后的海水10.0 mL,试验结束后将珊瑚剪成碎块,放入离心管中振荡,直到珊瑚的组织全脱落。取出碎块后,对获得的组织进行匀浆并平均分成3等份,6000 r/min离心1 min,弃上清液,加入1.0 mL的5%甲醛固定后利用血球计数板进行计数。虫黄藻密度计算公式:D=C×V/(M/G)。式中,D为虫黄藻密度(105 Cells/cm2),C为虫黄藻浓度(Cells/mL),V为冲洗液体积(mL),M为包裹珊瑚样本的铝箔纸重量(g),G为铝箔纸单位面积重量(g/cm2)(刘旭,2020)。
1.6全基因组测序分析
以芽孢杆菌GXU-Z9株为受试候选益生菌,采用接种环在2216E固体培养基上划线培养,挑取单菌落接种至MA液体培养基中,在30℃下160r/min摇床培养24 h。取10.0 mL菌液,10000 r/min离心1 min,收集菌体,液氮速冻后送至上海凌恩生物科技有限公司进行全基因组测序。使用Unicycler 0.4.8对原始数据进行拼接,利用R语言circlize包绘制基因组Circos图;预测基因序列分别在Nr、KEGG、COG、GO、CARD和CAZy等数据库中进行比对,以获得预测基因的注释信息。
2结果与分析
2.1潜在益生菌筛选结果
通过原位模拟培养法,从7种珊瑚样品中共分离纯化获得271株菌株。在七叶苷固体培养基上,有87株菌株周围出现黑色圈(图1-A),说明这些菌株具有产β-葡萄糖苷酶能力。通过酪蛋白固体培养基筛选产蛋白酶菌株,不产蛋白酶的菌株周围呈黄色,而周围出现透明水解圈的菌株表示具备产生蛋白酶能力,结果(图1-B)从271株菌株中筛选出136株蛋白酶活性菌株。通过平板扩散法筛选出对溶珊瑚弧菌具有生长抑制活性的菌株,结果(图1-C)显示在涂布溶珊瑚弧菌的MA培养基上有28株菌株周围出现明显的透明抑菌圈,即这些菌株对溶珊瑚弧菌具有拮抗作用。紫色杆菌CV12614具有明显的群体感应现象,且伴有紫色素产生,因此可通过观察紫色素的生成筛选具有群体淬灭活性的菌株(Marti-nellietal.,2004),结果(图1-D)发现有20株菌株周围出现透明圈且伴有无色的紫色杆菌菌落,说明这些菌株具有群体淬灭活性。
2.2 16S rRNA序列鉴定结果
将筛选到的所有菌株16S rRNA扩增产物送至生工生物工程(上海)股份有限公司测序,测序结果进行BLAST比对分析,结果表明,136株蛋白酶活性菌株分别来自4门24属(表1),87株β-葡萄糖苷酶活性菌株分别来自3门21属(表2),28株对溶珊瑚弧菌有拮抗活性的菌株分别来自3门16属(表3),20株具有群体淬灭活性的菌株分别来自2门4属(表4)。
2.3益生菌选择及其致病风险预测结果
既能产生胞外酶又具有拮抗活性或群体淬灭活性的菌株有芽孢杆菌属、假芽孢杆菌属(Fictibacil-lus sp.)、微小杆菌属(Exiguobacterium sp.)、鲁杰氏菌属、鞘脂菌属(Sphingobium sp.)、假交替单胞菌属、弧菌属、微球菌属(Micrococcus sp.)、诺卡菌属(Nocardia sp.)和鞘氨醇单胞菌(Sphingomonas sp.)。其中,芽孢杆菌属、假芽孢杆菌属、微小杆菌属、假交替单胞菌属和弧菌属能同时产生2种胞外酶且具有拮抗活性或群体淬灭活性,尤其是芽孢杆菌属和弧菌属不仅产生2种胞外酶还具有拮抗活性和群体淬灭活性。选择芽孢杆菌属、假芽孢杆菌属、微小杆菌属、假交替单胞菌属和弧菌属作为候选益生菌,并基于Web of Science等数据库检索和评估其致病风险,结果(表5)发现,芽孢杆菌属和假交替单胞菌属具有巨大的益生菌潜力。尤其是芽孢杆菌属一直被视为益生菌,在医学、种植业、畜牧业、水产养殖业等领域均有广泛应用和深入研究(Cutting,2011),大约800种具有抗生素活性的代谢物(肽类抗生素)是由各种芽孢杆菌产生(Wiese et al.,2009)。Shnit-Orland等(2012)从珊瑚黏液层分离出的6株假交替单胞菌属菌株中有5株能抑制多种珊瑚病原体生长。由于弧菌属细菌如哈维弧菌(V.harveyi)(Luna et al.,2010)、溶珊瑚弧菌(Kimes etal.,2012)和施罗氏弧菌(Rubio-Portillo et al.,2014)等多次被报道对珊瑚有致病作用,假芽孢杆菌属和微小杆菌属与珊瑚疾病相关的研究较少,尚无法判断其致病风险,因此,仅选取芽孢杆菌属和假交替单胞菌属进行珊瑚潜在益生菌缸内验证试验。
2.4珊瑚益生菌抗病作用缸内验证试验结果
由于假交替单胞菌属直接导致受试珊瑚死亡,因此排除进一步试验。珊瑚益生菌缸内验证试验结果表明,空白对照组(C)和芽孢杆菌处理组(VT)的珊瑚触手自然伸展,色泽保持一致,无组织脱落,无白化现象;弧菌对照组(V)的珊瑚触手收缩,有明显白化和组织脱落现象(图2-A)。说明用于珊瑚益生菌缸内验证试验的芽孢杆菌属菌株具有益生菌潜在功能,将其暂命名为GXU-Z9。继续测定珊瑚的Fv/Fm,结果发现仅弧菌对照组(V)从第6d起Fv/Fm开始呈逐渐下降趋势,至第11 d达最低值;空白对照组(C)和芽孢杆菌处理组(VT)珊瑚的Fv/Fm保持稳定波动状态,无明显下降趋势(图2-B)。珊瑚样品的虫黄藻密度测定结果(图2-C)显示,芽孢杆菌处理组(VT)珊瑚的虫黄藻密度显著高于空白对照组(C)(P<0.05,下同),极显著高于弧菌对照组(V)(P<0.01),空白对照组(C)珊瑚的虫黄藻密度显著高于弧菌对照组(V)。综上所述,芽孢杆菌GXU-Z9株能促进珊瑚共生虫黄藻增殖,维持正常的光合效率,且能有效抵御溶珊瑚弧菌感染,维持珊瑚宿主健康。
2.5芽孢杆菌GXU-Z9株全基因组测序结果
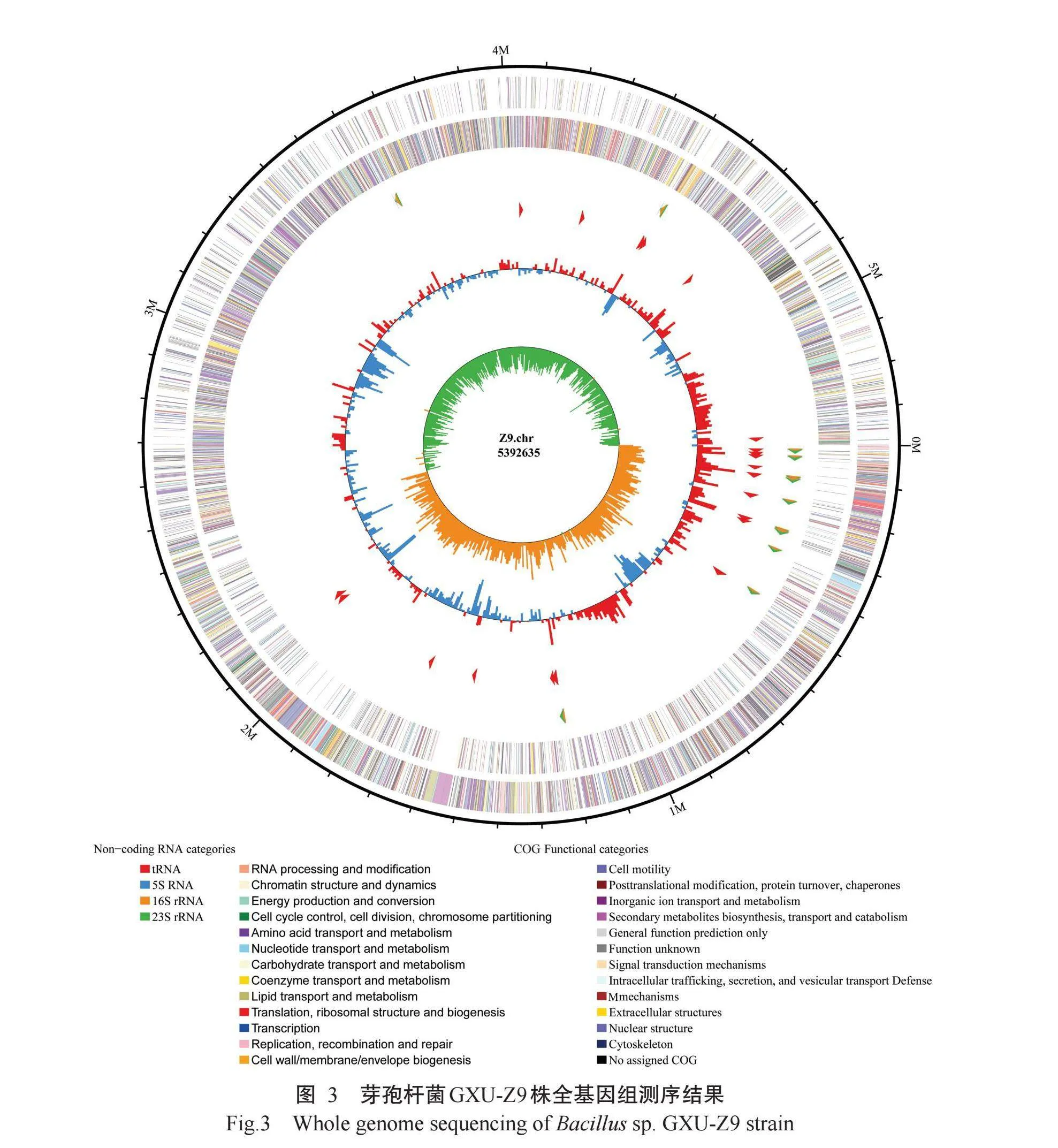
芽孢杆菌GXU-Z9株的染色体基因组呈单链环状(图3),组装后的基因组总长度为5392635bp,GC含量为41.45%。芽孢杆菌GXU-Z9株全基因组包含5796个蛋白编码基因(占全基因组总长度的85.9%),以及108个tRNAs基因和36个rRNAs基因。2.6芽孢杆菌GXU-Z9株益生功能分析结果
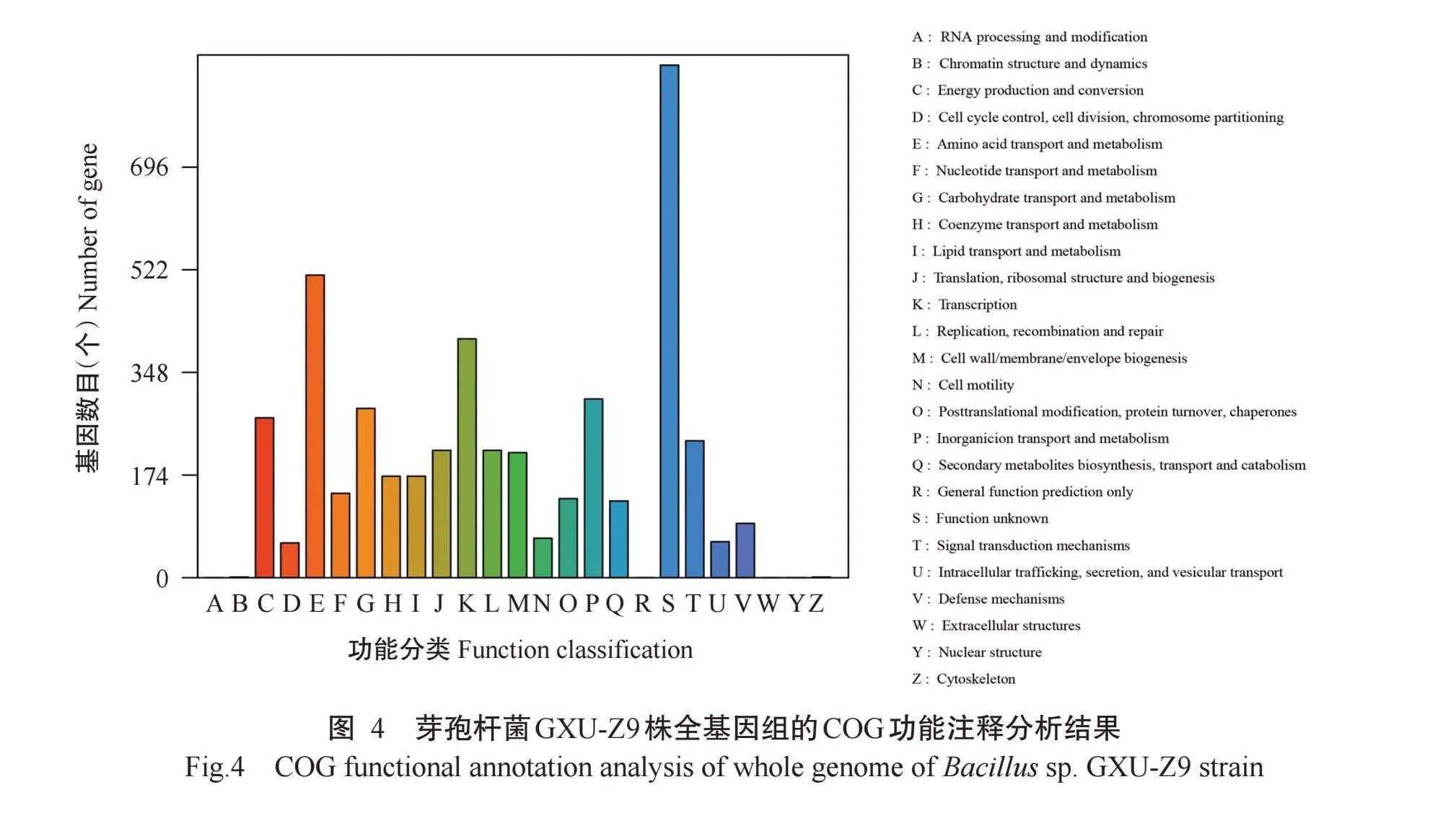
2.6.1基因组功能注释分析结果将测序获得的基因注释到COG数据库中并进行功能归类,结果(图4)发现:除了869个未知功能的基因外,注释基因数目最多的是氨基酸运输和代谢(Amino acidtransport and metabolism),共注释到513个基因;其次是转录(Transcription)、无机离子运输和代谢(Inor-ganicion transport and metabolism)、碳水化合物运输和代谢(Carbohydrate transport and metabolism)、能量生产和转换(Energy production and conversion)、信号转导机制(Signal transduction mechanisms)、翻译/核糖体结构和生物发生(Translation,ribosomal structure and biogenesis)、复制/重组和修复(Replica-tion,recombination and repair)、细胞壁/细胞膜/细胞膜的生物发生(Cell wall/membrane/envelope biogene-sis)等过程,注释到的功能基因数分别为405、303、287、271、232、216、216和212个。表明芽孢杆菌GXU-Z9株具有非常活跃的氨基酸代谢和运输能力,具备为珊瑚宿主提供必需和非必需氨基酸的潜力。
KEGG代谢通路富集分析结果(图5)显示:在一级分类中,所有基因富集在五大分支上,以代谢(Metabolism)所占的基因数最多(3550个);在二级分类中,富集基因数最多的是全局和总览图(Global and overview maps),有1941个,其次是碳水化合物代谢(Carbohydrate metabolism)和氨基酸代谢(Amino acid metabolism),分别富集到402和381个基因。此外,还富集到多种氨基酸和维生素的生物合成途径,如色氨酸/赖氨酸/酪氨酸生物合成(Bio-synthesis of tryptophan,lysine and tyrosine)、泛酸和辅酶A生物合成(Biosynthesis of pantothenic acid and coenzyme A)、叶酸生物合成(Folate biosynthesis)等。故推测芽孢杆菌GXU-Z9株是通过产生对珊瑚有益的化合物(抗生素等)而抵御弧菌感染。
2.6.2碳水化合物相关酶注释分析结果在CAZy数据库中,注释到基因数最多的酶类是糖酯酶类(CEs),有37个基因注释到该类;其次是糖苷水解酶类(GHs)、碳水化合物结合结构域(CBMs)和糖基转移酶类(GTs),分别注释到36、35和33个基因;氧化还原酶类(AAs)注释到10个(图6)。此外,芽孢杆菌GXU-Z9株含有几丁质酶及β-葡萄糖苷酶合成基因,编码参与碳水化合物代谢的重要酶类(表6)。珊瑚感染溶珊瑚弧菌后通常会失去虫黄藻,在缸内验证试验中表现出白化现象,说明此时珊瑚宿主失去了能量来源,需要其他共生体协助消化主动或被动捕获纤维类及蛋白类颗粒物质,以获得能量补充(Grottolietal.,2006)。芽孢杆菌GXU-Z9株含有β-葡萄糖苷酶等编码基因,因此可通过降解纤维类或蛋白类颗粒物质来补充珊瑚能量需求。
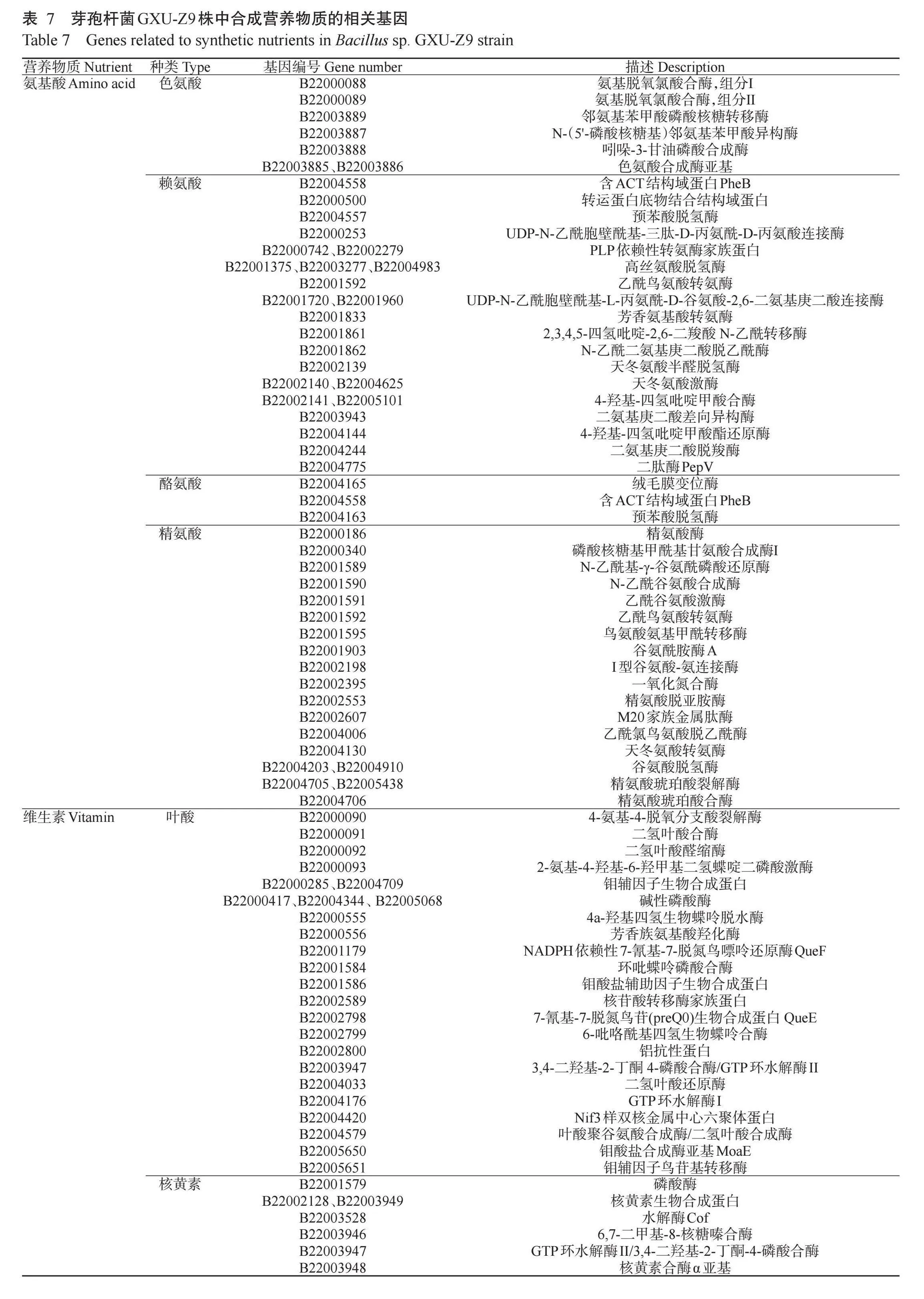
2.6.3参与营养物质合成相关基因分析结果产生营养物质也是益生菌的重要益生作用之一(Peixoto et al.,2017)。基于COG数据库,在芽孢杆菌GXU-Z9株中预测到53个与氨基酸生物合成相关基因,分别与色氨酸、赖氨酸、酪氨酸及精氨酸的生物合成有关;同时预测到32个与B族维生素合成相关基因,主要参与叶酸和核黄素的生物合成(表7)。
3讨论
珊瑚共附生微生物是最复杂的生物圈,由虫黄藻、古菌、细菌、原生动物和病毒共同组成,与珊瑚的相互作用对珊瑚礁生态系统具有重要意义(Tran,2022)。珊瑚共附生细菌群体随着环境的变化而改变,且没有任何一个细菌属是珊瑚特定的必需共生体。有学者提出,珊瑚可通过调控自身的细菌群落来适应环境变化,其共附生细菌群体在促进珊瑚健康和抵御疾病等方面可能发挥着重要作用(Reshef et al.,2006)。共附生微生物是珊瑚益生菌的重要来源,但由于种类繁多、培养难度大,至今尚未完全开发利用。为此,本研究采用原位模拟培养以提高菌株的存活率和可培养率,结果从7种珊瑚中共分离纯化获得271株珊瑚共附生细菌,且部分菌株显示出胞外酶活性、拮抗活性和群体淬灭活性,即具有潜在的珊瑚益生特性,研究结果扩大和丰富了珊瑚益生菌资源库。
芽孢杆菌属是常见的珊瑚共附生细菌。Nithy-anand和Pandian(2009)从马纳尔湾鹿角珊瑚组织和黏液中分离出相关细菌群落,并证实坚强芽孢杆菌(B.firmus)、枯草芽孢杆菌(B.subtilis)和堀越氏芽孢杆菌(B.horikoshii)同时存在于珊瑚组织和黏液中,对不同病原体具有拮抗活性;Elahwany等(2015)对软珊瑚Sarcophyton glaucum进行微生物分离及抗菌活性测试,结果发现芽孢杆菌属占分离细菌总数的60%,且芽孢杆菌属对病原菌和真菌表现出广泛的拮抗作用。假单胞菌属能产生抗菌物质,且对哈维弧菌(Radjasa et al.,2005)、溶珊瑚弧菌(Tang et al.,2020)具有较强的生长抑制作用。Devi等(2022)从鱼类肠道微生物群落中分离出的假交替单胞菌对14株致病菌具有拮抗作用,能产生胞外酶,同时具有清除自由基的能力。本研究分离纯化获得的271株珊瑚共附生细菌也展现出多种活性,根据相关文献和数据库追踪发现芽孢杆菌属和假交替单胞菌属的致病风险较小,被确定为珊瑚候选益生菌。此外,本研究筛选获得的大量珊瑚益生菌资源有望为后续的珊瑚益生菌研究提供丰富材料。
已有研究表明,益生菌的加入不仅能提高鹿角杯形珊瑚对溶珊瑚弧菌和高温的抵抗力,还能减轻溶珊瑚弧菌引起的珊瑚骨骼损伤,而维持珊瑚钙化率(Rosado et al.,2019;Moradi et al.,2023)。Zhang等(2021)通过对鹿角杯形珊瑚接种益生菌团,有效增加了潜在益生菌的丰度,进而提高了珊瑚的能量储备和钙化率。本研究的缸内验证试验也发现,益生菌处理组(VT)与空白对照组(C)的鹿角杯形珊瑚保持良好健康状态,而弧菌对照组(V)的鹿角杯形珊瑚出现明显疾病症状,表明芽孢杆菌GXU-Z9株具有抗溶珊瑚弧菌感染的功能。芽孢杆菌GXU-Z9株全基因组中含有合成几丁质酶和β-葡萄糖苷酶的相关基因。几丁质酶已被证实可协助珊瑚获得几丁质用于合成骨骼,且在防御真菌感染方面具有一定作用(Yoshioka et al.,2017);β-葡萄糖苷酶是一种纤维素酶,可水解纤维素内部的β-1,4键,产生纤维素酶以协助珊瑚宿主消化虫黄藻,而更好地维持共生关系(Su et al.,2021)。珊瑚主要通过共附生微生物获取必需氨基酸和维生素(Li etal.,2022)。芽孢杆菌GXU-Z9株全基因组中还包含多种氨基酸生物合成相关基因,包括色氨酸、赖氨酸、酪氨酸和精氨酸等,即具备为珊瑚宿主提供氨基酸以获得能量的潜力。此外,芽孢杆菌GXU-Z9株全基因组中拥有合成B族维生素(叶酸和核黄素)的相关基因,故推测芽孢杆菌GXU-Z9株是珊瑚维生素的重要来源。
虽然珊瑚益生菌的研究尚处于起步阶段,但其环境友好型的特点,将成为珊瑚疾病防治的重要研究方向。本研究建立的珊瑚益生菌筛选体系及其获得的珊瑚益生菌资源,为后续的珊瑚益生菌抗病研究提供了原创材料;但今后还应继续对候选益生菌开展抗病作用的浓度、温度及时间等验证试验,或通过多组学技术解析芽孢杆菌GXU-Z9株对珊瑚宿主肠道微生物与免疫的调控机理,同时进行海区原位试验以拓展益生菌抗病应用。
4结论
芽孢杆菌GXU-Z9株能有效维持鹿角杯形珊瑚的光合效率和虫黄藻密度,展现出良好的抗病功能;芽孢杆菌GXU-Z9株全基因组中含有编码几丁质酶和β-葡萄糖苷酶的相关基因,能大量合成珊瑚的必需氨基酸和维生素等营养物质,具有辅助珊瑚消化碳水化合物的酶类物质、提供氨基酸和维生素等益生特性。
参考文献(References):
蒋庆茹,柯才焕,虞晋晋,赵晶.2012.杂色鲍肠道益生菌的分离和鉴定[J].厦门大学学报(自然科学版),51(4):782-788.[Jiang Q R,Ke C H,Yu J J,Zhao J.2012.Isolation and characterisation of putative probiotics from Haliotisdiversicolour[J].Journal of Xiamen University(Natural Science),51(4):782-788.]
刘旭.2020.造礁石珊瑚对温度胁迫的响应机制研究[D].南宁:广西大学.[Liu X.2020.Response mechanisms of reef-building coral rocks to temperature stress[D].Nan-ning:Guangxi University.]doi:10.27034/d.cnki.ggxiu.2020.001544.
王为善,邹姗姗,尹守亮,官倩红,于文功.2009.具有群体感应抑制活性海洋放线菌的分离和鉴定[J].微生物学通报,36(9):1372-1377.[Wang W S,Zou S S,Yin S L,Guan Q H,Yu W G.2009.Isolation and identification of marine actinomycetes with quorum sensing inhibitory activity[J].Microbiology,36(9):1372-1377.]
Aeby G S,Kenyon J C,Maragos J E,Potts D C.2003.First record of mass coral bleaching in the Northwestern Ha-waiian Islands[J].Coral Reefs,22:256.doi:10.1007/s00338-003-0309-2.
Bourne D G,Garren M,Work T M,Rosenberg E,Smith G W,Harvell C D.2009.Microbial disease and the coral holo-biont[J].Trends in Microbiology,17(12):554-562.doi:10.1016/j.tim.2009.09.004.
Certner R H,Vollmer S V.2015.Evidence for autoinduction and quorum sensing in white band disease-causing microbes on Acropora cervicornis[J].Scientific Reports,5(1):11134.doi:10.1038/srep 11134.
Cutting S M.2011.Bacillus probiotics[J].Food Microbiology,28(2):214-220.doi:10.1016/j.fm.2010.03.007.
Davy S K,Allemand D,Weis V M.2012.Cell biology of cnidarian-dinoflagellate symbiosis[J].Microbiology and Molecular Biology Reviews,76(2):229-261.doi:10.1128/MMBR.05014-11.
Devi A A,Khan M I R,Choudhury T G,Kamilya D.2022.In vitro assessment of probiotic potential of an autochthonous bacterial isolate,Pseudomonas mosselii COFCAU_PMP5[J].Microbiology,91(2):207-214.doi:10.1134/S002626 1722020047.
Dong Y H,Xu J L,Li X Z,Zhang L H.2000.AiiA,an enzyme that inactivates the acylhomoserine lactone quorum-sensing signal and attenuates the virulence of Erwinia carotovora[J].Proceedings of the National Academy of Sciences of the United States of America,97(7):3526-3531.doi:10.1073/pnas.97.7.3526.
Elahwany AM D,Ghozlan HA,Elsharif HA,Sabry SA.2015.Phylogenetic diversity and antimicrobial activity of marine bacteria associated with the soft coral Sarcophytonglau-cum[J].Journal of Basic Microbiology,55(1):2-10.doi:10.1002/jobm.201300195.
Grottoli A G,Rodrigues L J,Palardy J E.2006.Heterotrophic plasticity and resilience in bleached corals[J].Nature,440(7088):1186-1189.doi:10.1038/nature04565.
Kimes N E,Grim C J,Johnson W R,Hasan N A,Tall B D,Kothary M H,Kiss H,Munk A C,Tapia R,Green L,Detter C,Bruce D C,Brettin T S,Colwell R R,Morris P J.2012.Temperature regulation of virulence factors in the patho-gen Vibrio coralliilyticus[J].The ISME Journal,6(4):835-846.doi:10.1038/ismej.2011.154.
Kitamura R,Miura N,Ito M,Takagi T,Yamashiro H,Nishi-kawa Y,Nishimura Y,Kobayashi K,Kataoka M.2021.Specific detection of coral-associated Ruegeria,a potential probiotic bacterium,in corals and subtropical seawater[J].Marine Biotechnology,23:576-589.doi:10.1007/s 10126-021-10047-2.
Krediet C J,Ritchie K B,Alagely A,Teplitski M.2013.Mem-bers of native coral microbiota inhibit glycosidases and thwart colonization of coral mucus by an opportunistic pathogen[J].The ISME Journal,7(5):980-990.doi:10.1038/ismej.2012.164.
Li J,Zou Y Y,Yang J,Li Q Q,Bourne D G,Sweet M,Liu C,Guo A J,Zhang S.2022.Cultured bacteria provide insight into the functional potential of the coral-associated micro-biome[J].mSystems,7(4):e00327-22.doi:10.1128/msys-tems.00327-22.
Li Y,Wu C Y,Zhou M Y,Wang E T,Zhang Z P,Liu W,Ning J C,Xie Z H.2017.Diversity of cultivable protease-producing bacteria in Laizhou Bay sediments,Bohai Sea,China[J].Frontiers in Microbiology,8:405.doi:10.3389/fmicb.2017.00405.
Luna G M,Bongiorni L,Gili C,Biavasco F,Danovaro R.2010.Vibrio harveyi as a causative agent of the White Syn-drome in tropical stony corals[J].Environmental Microbio-logy Reports,2(1):120-127.doi:10.1111/j.1758-2229.2009.00114.x.
Martinelli D,Grossmann G,Séquin U,Brandl H,Bachofen R.2004.Effects of natural and chemically synthesized fura-nones on quorum sensing in Chromobacteriumviolaceum[J].BMC Microbiology,4:25.doi:10.1186/1471-2180-4-25.
Moradi M,Magalhaes P R,Peixoto R S,Jonck C C A C,François D,Bellot A C F,Teixeira J B,Silveira C S,Duarte G,Evangelista H,Barbosa C F.2023.Probiotics mitigate pathogen-and thermal stress-driven impacts on coral skeleton[J].Frontiers in Marine Science,10:1212690.doi:10.3389/fmars.2023.1212690.
Morais J,Cardoso A P L R,Santos B A.2022.A global synthe-sis of the current knowledge on the taxonomic and geo-graphic distribution of major coral diseases[J].Environ-mental Advances,8:100231.doi:10.1016/j.envadv.2022.100231.
Ng W L,Bassler B L.2009.Bacterial quorum-sensing network architectures[J].Annual Review of Genetics,43(1):197-222.doi:10.1146/annurev-genet-102108-134304.
Nithyanand P,Pandian S K.2009.Phylogenetic characteriza-tion of culturable bacterial diversity associated with themucus and tissue of the coral Acropora digitifera from the Gulf of Mannar[J].FEMS Microbiology Ecology,69(3):384-394.doi:10.1111/j.1574-6941.2009.00723.x.
Okwadha G D O,Li J.2010.Optimum conditions for microbial carbonate precipitation[J].Chemosphere,81(9):1143-1148.doi:10.1016/j.chemosphere.2010.09.066.
Peixoto R S,Rosado P M,de Assis Leite D C,Rosado A S,Bourne D G.2017.Beneficial microorganisms for corals(BMC):Proposed mechanisms for coral health and resi-lience[J].Frontiers in Microbiology,8:341.doi:10.3389/fmicb.2017.00341.
Radjasa O K,Martens T,Grossart H P,Sabdono A,Simon M,BachtiarT.2005.Antibacterial property of a coral-associated bacterium Pseudoalteromonasluteoviolace against shrimp pathogenic Vibrio harveyi(In vitro study)[J].HAYATI Journal of Biosciences,12(2):77-81.doi:10.1016/S1978-3019(16)30329-1.
Reshef L,Koren O,Loya Y,Zilber-Rosenberg I,Rosenberg E.2006.The coral probiotic hypothesis[J].Environmental Mi-crobiology,8(12):2068-2073.doi:10.1111/j.1462-2920.2006.01148.x.
Rosado P M,Leite D C A,Duarte G A,Chaloub R M,Jospin G,da Rocha N U,Saraiva J P,Dini-Andreote F,Eisen J A,Bourne D G,Peixoto R S.2019.Marine probiotics:Increa-sing coral resistance to bleaching through microbiome manipulation[J].The ISME Journal,13(4):921-936.doi:10.1038/s41396-018-0323-6.
Rubio-Portillo E,Yarza P,Peñalver C,Ramos-EspláA,Antón J.2014.New insights into Oculina patagonica coral di-seases and their associated Vibrio spp.communities[J].The ISME Journal,8(9):1794-1807.doi:10.1038/ismej.2014.33.
Rudrappa T,Bais H P.2008.Curcumin,a known phenolic from Curcuma longa,attenuates the virulence of Pseudomonas aeruginosa PAO1 in whole plant and animal pathogenicity models[J].Journal of Agricultural and Food Chemistry,56(6):1955-1962.doi:10.1021/jf072591j.
Shnit-Orland M,Sivan A,Kushmaro A.2012.Antibacterial activity of Pseudoalteromonas in the coral holobiont[J].Microbial Ecology,64(4):851-859.doi:10.1007/s00248-012-0086-y.
Spadafore R,Fura R,Precht W F,Vollmer S V.2021.Multi-variate analyses of coral mortality from the 2014-2015 stony coral tissue loss disease outbreak off Miami-Dade County,Florida[J].Frontiers in Marine Science,8:723998.doi:10.3389/FMARS.2021.723998.
Su H F,Xiao Z L,Yu K F,Zhang Q,Lu C R,Wang G H,Wang Y H,Liang J Y,Huang W,Huang X Y,Wei F.2021.High diversity ofβ-glucosidase-producing bacteria and their genes associated with scleractinian corals[J].International Journal of Molecular Sciences,22(7):3523.doi:10.3390/ijms22073523.
Tait K,Hutchison Z,Thompson F L,Munn C B.2010.Quorum sensing signal production and inhibition by coral-associated vibrios[J].Environmental Microbiology Reports,2(1):145-150.doi:10.1111/j.1758-2229.2009.00122.x.
Tang K H,Zhan W E,Zhou Y Q,Xu T,Chen X Q,Wang W Q,
Zeng Z S,Wang Y,Wang X X.2020.Antagonism between coral pathogen Vibrio coralliilyticus and other bacteria in the gastric cavity of scleractinian coral Galaxeafascicu-laris[J].Science China Earth Sciences,63(1):157-166.doi:10.1007/s 11430-019-9388-3.
Tang K H,Zhang X H.2014.Quorum quenching agents:Resources for antivirulence therapy[J].Marine Drugs,12(6):3245-3282.doi:10.3390/md 12063245.
Tran C.2022.Coral-microbe interactions:Their importance to reef function and survival[J].Emerging Topics in Life Sciences,6(1):33-44.doi:10.1042/ETLS20210229.
Ushijima B,Meyer J L,Thompson S,Pitts K,Marusich M F,Tittl J,Weatherup E,Reu J,Wetzell R,Aeby G S,Häse C C,Paul V J.2020.Disease diagnostics and potential coin-fections by Vibrio coralliilyticus during an ongoing coral disease outbreak in Florida[J].Frontiers in Microbiology,11:569354.doi:10.3389/fmicb.2020.569354.
Walton C J,Hayes N K,Gilliam D S.2018.Impacts of a regional,multi-year,multi-species coral disease outbreak in Southeast Florida[J].Frontiers in Marine Science,5:323.doi:10.3389/fmars.2018.00323.
Ward J R,Lafferty K D.2004.The elusive baseline of marine disease:Are diseases in ocean ecosystems increasing?[J].PLoS Biology,2(4):e120.doi:10.1371/journal.pbio.002 0120.
Wegley L,Edwards R,Rodriguez-Brito B,Liu H,Rohwer F.2007.Metagenomic analysis of the microbial community associated with the coral Porites astreoides[J].Environ-mental Microbiology,9(11):2707-2719.doi:10.1111/j.1462-2920.2007.01383.x.
Wiese J,Thiel V,Nagel K,Staufenberger T,Imhoff J F.2009.Diversity of antibiotic-active bacteria associated with the brown alga Laminaria saccharina from the Baltic Sea[J].Marine Biotechnology,11:287-300.doi:10.1007/s 10126-008-9143-4.
Williams G J,Knapp I S,Work T M,Conklin E J.2011.Out-break of Acropora white syndrome following a mild bleaching event at Palmyra Atoll,Northern Line Islands,Central Pacific[J].Coral Reefs,30(3):621.doi:10.1007/s00338-011-0762-2.
Yamamura H,Hayakawa M,Iimura Y.2003.Application of sucrose-gradient centrifugation for selective isolation of Nocardia spp.from soil[J].Journal of Applied Microbio-logy,95(4):677-785.doi:10.1046/j.1365-2672.2003.020 25.x.
Yoshioka Y,Tanabe T,Iguchi A.2017.The presence of genes encoding enzymes that digest carbohydrates in coral ge-nomes and analysis of their activities[J].PeerJ,5:e4087.doi:10.7717/peerj.4087.
Zhang Y,Yang Q S,Ling J,Long J L,Huang H,Yin J P,Wu M L,Tang X Y,Lin X C,Zhang Y Y,Dong J D.2021.Cor-rection to:Shifting the microbiome of a coral holobiont and improving host physiology by inoculation with a potentially beneficial bacterial consortium[J].BMC Micro-biology,21(1):1-14.doi:10.1186/s 12866-021-02219-w.
(责任编辑兰宗宝)
