河北省土著鳜鱼种质资源鉴定
2024-10-12王江江高晓田赵春龙于琪赵欣孙砚峰吴成宾




摘要:为探明河北省内的鳜属鱼类现状,更好地为全国鳜鱼种质资源保护提供可供参考的资料,以线粒体COX1和全基因组范围SNP标记为支撑对鳜属群体进行了分子水平的种质资源鉴定和遗传多样性水平相关分析。研究结果显示,获得40条全长为697 bp的COX1同源序列和SSH01、SSH02两个单倍型序列。最大似然法构建的系统发育树显示,两个单倍型先聚为一支,后又与斑鳜聚为一支。在地理种群系统发生树中,两个单倍型与参考的韩国Changju斑鳜聚为一支。群体的观测杂合度Ho均值为0.330,PIC均值为0.269,近交系数均值约为0,SSH01和SSH02在观测杂合度上差异不显著(P>0.05)。群体中共祖系数为0的占总体的71.41%,存在较多亲缘关系近的个体。PCA分析结果发现,群体为可分为3个遗传结构,Cluster0、Cluster1、Cluster2;Admixture显示最有可能存在2个祖先;连锁不平衡分析的结果为,在95%置信区间下群体的Ne范围为14.8~27.6。河北省斑鳜存在两个单倍型,群体遗传多样性较低,与韩国Changju斑鳜有较高的亲缘性,可通过人为引种和增殖放流的方式保护好省内的鳜鱼种质资源。
关键词:斑鳜(Siniperca scherzeri);线粒体COX1;SNP;遗传多样性
鳜鱼俗名桂花鱼,隶属于硬骨鱼纲(Osteichthyes),鲈形目(Perciformes),暖鲈科(Percichthyidae),鳜亚科(Siniperinae),是东亚地区特有的经济鱼类之一,在中国、日本、俄罗斯、朝鲜半岛等均有分布[1]。目前普遍认同的分布于我国境内的鳜鱼有鳜(S. chuatsi)、斑鳜(S. scherzeri)、大眼鳜(S. kneri)等。不同种类的鳜鱼在生长特性、繁殖能力、营养价值等方面存在差异。陈军等[2]发现同塘饲养下鳜的生长速度是大眼鳜的4~5倍,并且鳜卵巢发育的绝对怀卵量大于大眼鳜。宓国强等[3]发现斑鳜的肌肉蛋白质含量显著高于鳜。经过多年的选择培育、品种杂交,我国已培育出了多个鳜鱼新品种,如翘嘴鳜“华康1号”“秋浦杂交斑鳜”“长珠杂交鳜”等,在生长性能、抗病性能、适应能力等方面表现良好[4]。此外,我们积极通过分子选育手段开发与鳜鱼各种生长性状潜在相关的基因组区域和候选基因。2022年Zhou等[5]通过特定位点扩增片段测序和SNP基因分型,进行了全基因组关联研究(GWAS),在相关QTL内鉴定出65个与骨分化、生长和发育、细胞分裂和神经发生有关候选基因。
种质资源的多样性,不仅与改良生物性状,培育优质品种密切相关,还对研究物种起源、进化等理论有着极为重要的作用[6]。然而近年来,由于环境污染、竭泽而渔式的捕捞等原因,鳜鱼野生资源在逐步衰退。Yang等[7]的调查显示,鳜鱼是长江各水系的优势种之一。河北省拥有丰富的渔业资源,有104种淡水鱼类,包括鲫、乌鳢、鳜鱼、细鳞鱼等。河北省已建立了19个国家级水产种质资源保护区,其中位于安新县的白洋淀国家水产种质资源保护区和位于兴隆县和承德县境内的柳河特有鱼类国家级水产种质资源保护区都将鳜鱼作为主要保护对象[8]。
分子标记是研究鱼类遗传多样性的重要方法之一,鱼类常用的分子鉴定手段有Cytb、D-loop、线粒体12S rRNA等[9]。赵金良等[10]在细胞色素b基因序列的基础上,初步构建了东亚鳜类种间的系统发育关系,建议将长体鳜归入鳜鱼属,鳜类可分为鳜鱼群和少鳞鳜群两支;范世琦[11]利用筛选出的10对微卫星引物对重庆地区三个养殖场鳜群体的遗传结构和多态性进行了分析评价,发现三个鳜养殖群的香农威纳指数平均值在1.378~1427,群体遗传异质性较高;余科[12]利用线粒体DNA Cytb基因分析出了不同稻田鲤群体的遗传分化情况,发现从江等6个稻田鲤群体由欧洲鲤(Cyprinus carpio carpio)、远东鲤(C. carpio haematopterus)和华南鲤(C. carpio rubrofuscus)3个亚种组成;李龑[13]利用COI-Cytb-D-loop序列对鲤群体进行了遗传多样性分析,发现大头鲤、荷包红鲤抗寒品系和蓝鳞鲤之间的遗传分化较大。除此之外,近年来DNA条码技术也越来越受到人们关注,DNA条码指的是能够用来区分不同物种的,有一定标准的,并且足够变异的DNA片段[14]。于潇[15]利用PCR技术扩增出的线粒体cox 1基因,成功对50条软骨鱼类进行了鉴定,并验证了传统分类学中软骨鱼类的在目阶元上的分类。程汉良等[16]基于线粒体细胞色素c氧化酶亚基Ⅰ基因序列对帘蛤科贝类进行了系统发育研究,成功解决了帘蛤科传统分类上的难题。陈治等[17]使用12S和COI对145种常见海洋鱼类进行了鉴定,结果表明,COI对物种鉴定的准确率更高。线粒体DNA因其高拷贝的特点,也常被选为eDNA宏条形码的遗传标记,如12S、Cytb和COI[18]。Feng等[19]利用eDNA条形码来监测雅鲁藏布江中上游地区稀有和入侵鱼类的情况并对多样性进行了评估,其分析结果与传统的捕获法相印证。因此,为了解河北省内的鳜属鱼类现状,并探究其遗传多样性情况,我们利用线粒体COX1基因对鳜属鱼类进行了种质鉴定,并在全基因组范围SNP标记的基础上分析了其遗传结构和多样性,以期为鳜属鱼类资源保护和可持续发展提供参考。
1材料和方法
1.1材料来源
采集到的40尾鳜属鱼类,取自河北省承德市兴隆县柳河特有鱼类国家级水产种质资源保护区,如图1所示。每尾鱼在测量完基础生物数据后,取其背鳍后部1 cm长的鳍条,用95%乙醇固定保存,并存放在4 ℃的冰箱中用于DNA提取。
1.2DNA提取
待检样本DNA采用常规的酚-氯仿法提取,并通过1%浓度的琼脂糖凝胶电泳检测提取的样本DNA的完整性,部分结果如图2所示。
1.3PCR扩增
基于DNA条形码技术,以鳜属(Siniperca)鱼类线粒体基因组COX1基因5’端697 bp的一段序列作为种质鉴定的条形码序列,其扩增的上下游引物分别为:F primer:5′-TCGACCAATCACAAAGACATC-3′,R primer:5′-GGTGGCCAAAGAATCAGAAT-3′。PCR 反应体系总体积为 50 μL,其中 2×Taq PCR Master Mix 25 μL,上下游引物各 1.25 μL(5 μmol/L),DNA模板100 ng。PCR反应程序为:94 ℃预变性5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,35 个循环;最后72 ℃延伸1 min。PCR产物采用上述的测序引物进行测通。
1.4数据分析
采用DNASP、Arlequin和MEGA软件,分析测通的40个待检鳜属样本序列,通过比对和单倍型分析,获得待检样本的全部单倍型。应用MEGA软件将上述单倍型与鳜属鱼类线粒体参考序列进行比对,并提取鳜属鱼类线粒体参考序列的目标单倍型序列。最后,通过MEGA构建系统发生树,结合系统发生树的拓扑关系和遗传距离,对待检样本开展基于分子生物学的种质鉴定。种质鉴定后,探究送检样本与我国不同地理区系鳜属鱼类在线粒体基因水平上的遗传变异关系。
基于全基因组范围SNP标记对送检样本开展种质遗传多样性及遗传结果分析工作,采用准确且具有高精度的高密度SNP标记对整个基因组进行评估,SNP过滤参数为maf=0.05,hwe=10-5,SiteMissingRatio=0,均匀度=20K。利用软件计算待检群体个体杂合度和近交系数。通过共祖系数分析群体内部个体之间的亲缘关系组成,利用派诺森云作图构建亲缘关系热图。通过主成分分析来探究群体遗传结构,通过Admixture软件估算40个样本的祖先组成情况,并利用Origin输出图像。基于对连锁不平衡参数LD值的无偏估计算法,对该种群以及线粒体单倍型亚群的有效群体的大小进行评估。
2结果与分析
2.1种质鉴定
使用引物扩增得到的目的条带单一清晰,符合标准。经过对待检样本PCR扩增产物测序(双向测通),获得40条全长为697 bp的目的片段,通过比对和单倍型分析在待检样本中共获得2个单倍型序列,分别为SinSample HB Hap01(简称SSH01)和SinSample HB Hap02(简称SSH02)。与此同时,以待检样本的2个单倍型序列、7个鳜属鱼类的8个单倍型序列和外群物种中国少鳞鳜,Coreoperca whiteheadi单体型序列,分别为SinSample HB Hap01,SinSample HB Hap02,Siniperca scherzeri DTH NC 015815.1,Siniperca oulei KP710957.1,Siniperca fortis NC 047290.1,Siniperca obscura EF143390.1,SinSample HLJ Hap01,Siniperca chuatsi NC015822.1,Siniperca knerii NC 015987.1,Siniperca undulata EF143393.1,Coreoperca whiteheadi KJ149811构建基于最大似然法的系统发生树,拓扑关系和遗传距离如图3所示。
通过系统发生树可以看出,7个鳜属参考物种先聚为一支,随后与同为鳜科鱼类的少鳞鳜属鱼类中国少鳞鳜(Coreoperca whiteheadi)聚为一支,该结果表明本研究采用的种质鉴定方法正确有效;待检样本的2个单倍型聚为一支,表明40个待检样本均为同一物种;随后待检样本与参考物种斑鳜(S. scherzeri)聚为一支,而且与常见的鳜属物种鳜(S. chuatsi)和大眼鳜(S. kneri)存在较大的遗传差异,表明待检样本为斑鳜(S. scherzeri)的可能性最大。
2.2不同地理种群系统发生分析
经过种质鉴定后,我们可在分子水平上判断送检样本为斑鳜(S. scherzeri)。斑鳜在我国分布广泛,北至鸭绿江水系、南至珠江水系,我国不同地理区系斑鳜在线粒体基因水平上也会有一些差异。为探清送检的斑鳜样本与我国不同地理区系斑鳜在线粒体基因水平上的遗传变异关系,收集了在公开数据库中发表的斑鳜线粒体COX1基因序列,包括分布在我国长江及长江以南水系的斑鳜线粒体COX1基因序列11条,来源于韩国Changju的斑鳜单倍型序列1条,以及外群物种(鳜)的2个序列,分别为Siniperca scherzeri ZJ02 OP050807.1,Siniperca scherzeri ZJ03 OP08431,Siniperca scherzeri ZJ01 OP0508031,Siniperca scherzeri XJ JQ0109881,Siniperca scherzeri ML01 MW6807251,Siniperca scherzeri ML02 MW6807581,Siniperca scherzeri DTH JQ010986.1,Siniperca scherzeri LiuJ MW680745.1,Siniperca scherzeri PYH JQ010985.1,Siniperca scherzeri CD EF1433911,Siniperca scherzeri LiJ JQ0109871,SinSample HB Hap02,SinSample HB Hap01,Siniperca scherzeri SK EF143392.1(韩国Changju斑鳜),Siniperca chuatsi NC 015822.1,SinSample HLJ Hap01。由于公开数据库中斑鳜线粒体COX1基因序列长短不一,所以经过比对和序列对齐后,提取到可用于下游分析线粒体单倍型的序列长度为647 bp。随后利用待检样本的2个单倍型序列、12个斑鳜不同地理区系单倍型序列以及外群物种(鳜)的2个序列,构建基于最大似然法的系统发生树,拓扑关系和遗传距离如图4所示。
通过系统发生树可以看出,14个斑鳜单倍型序列首先聚为一支,随后与同属的鳜(S. chuatsi)聚为一支,结果表明该系统发生方法正确有效。随后,14个斑鳜单倍型分为两个明显的分支,分别为长江及长江以南水系的分支、以及待检样本和韩国Changju斑鳜的分支,该结果表明待检样本与长江及长江以南水系的斑鳜存在一定的遗传差异,而与同为北方水系的韩国Changju斑鳜存在较小的遗传差异。
2.3遗传多样性与近交水平
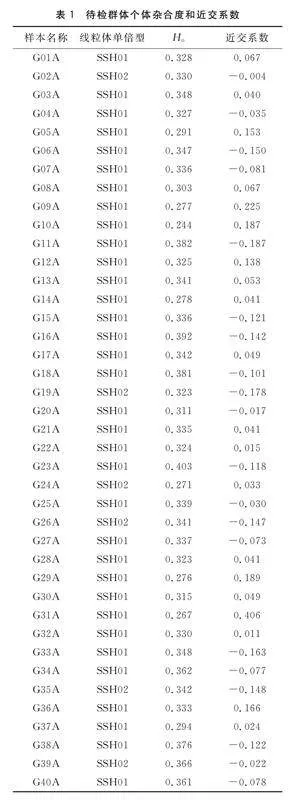
该鳜属鱼类群体共40个个体,其个体观测杂合度在0.244至0.403之间,整个群体的均值为0.330,其中由线粒体单倍型为SSH01组成的亚群平均值为0.330,由线粒体单倍型为SSH02组成的亚群均值为0.329,在观测杂合度方面不存在差异,如表1和表2所示。
该群体个体近交系数在-0.187至0.406之间,整个群体平均的近交水平约为0,但是群体内部有部分个体近交水平偏高,如表1和表2所示。群体中近交系数大于0.25的个体有1个,占群体的2.5%;近交系数大于0.125小于0.25的个体有5个,占群体的12.5%;近交系数大于0.062 5小于0.125的个体有2个,占群体的5%。
2.4基因组亲缘关系
共祖系数(coefficient of coancestry)表示任意两个个体之间的亲缘关系水平,亲缘关系越近,共祖系数(CC)的值越大。通过群体内个体之间的亲缘关系分析,能够了解群体内部个体之间的亲缘关系组成,可较为直观地反映出当前群体内潜在交配个体之间产生具有较高近交水平后代的可能性。通过分析亲缘关系热图结果(见封三图5)可以发现该群体内部具有较多的亲缘关系较近的个体对,其中共祖系数(CC)大于0.25的个体对共有7对,共祖系数范围在0.271至0.360之间共涉及两组8个样本,分别为G05A、G10A、G32A、G36A和G12A、G14A、G20A、G31A,占总数的0.90%;其中共祖系数大于0.125的个体对共有22对,占总数的2.8%;其中大于0.062 5的个体对共有46对,占总数的5.90%;其中大于0小于0.062 5的个体对共有148对,占总数的1897%;无亲缘关系的个体对共计557对,占总数的71.41%。
2.5群体遗传结构分析
主成分分析(PCA)结果如封三图6所示。PC1能够解释4.07%的遗传变异,PC2能够解释3.53%的遗传变异,PC3能够解释2.14%的遗传变异。通过PCA分析可以发现该群体内部可以分成3个遗传结构,分别由Cluster0(24个样本)、Cluster1(10个样本)、Cluster2(6个样本)三个聚类群组成。
通过Admixture软件估算40个样本的祖先组成情况,经过CV error评估最优K值,发现当K=2时错误率最小,说明该群体存在两个祖先的概率最大。从个体祖先组成柱形图(封三图7)可以发现,该群体样本中主要分为三类,分别为绝大部分为绿色的样本、绝大部分为橙色的样本以及两者比例适中的个体。
2.6有效群体规模评估
有效群体大小是水产种质资源保存与利用的重要参数,本研究基于对连锁不平衡参数LD值的无偏估计算法,对该种群以及线粒体单倍型亚群的有效群体大小进行评估,经过评估后群体有效规模和95%的置信区间如表3所示。
3讨论与结论
鳜,作为东亚地区特有的经济鱼类之一,在我国分布地区广泛,根据李思忠[20]的研究,鳜亚科鱼类在长江水系、珠江水系、黄河水系、海河水系、黑龙江水系等均有分布。周才武等[21]认为,长江流域以南低纬度的华南区是鳜亚科鱼类的分布中心。关于鳜的种类,Liu等[22]认为鳜的种类有12种,支持将麻鳜(S. fortis)作为有效种。不同种的鳜地理分布地区也有所差异,生活在我国境内的鳜鱼有9种,主要包括鳜(S. chuatsi)、麻鳜(S.fortis)、大眼鳜(S. kneri)、暗鳜(S. obscura)、长体鳜(S. roulei)、斑鳜(S. scherzeri)、波纹鳜(S. undulata)等,其中鳜和斑鳜的分布范围最广,甚至在朝鲜半岛也有记录[20]。而对于鳜类的分属划分,曾引起了学者们的广泛讨论。Fang等[23]认为鳜亚科只设1个鳜属(Siniperca)即可;周才武等[21]主张将鳜亚科划分为少鳞鳜属(Coreoperca)、鳜属(Siniperca)、长身鳜属(Coreosiniperca);Liu等[22]认为鳜亚科下可设有少鳞鳜属(Coreoperca)、鳜属(Siniperca)2个属。目前,第三种观点有较多分子学证据支撑。赵金良等[10]通过分析鳜类的细胞色素b序列并结合NJ树和MP法的结果,支持:长体鳜不单独设属而应归入鳜鱼属,鳜亚科可分为少鳞鳜属(Coreoperca)和鳜属(Siniperca)。章群等[24]对采集到的7种鳜类的12个个体的Cytb基因进行了全序列分析,结果支持了Liu的观点。宋书莉等[25]通过靶基因富集技术和二代测序的方法构建了鳜类的系统演化关系,结果支持鳜类包括两个属:鳜属(Siniperca)和少鳞鳜属(Coreperca)。而我们经过此次的鉴定,发现从河北省获得的鳜属鱼类应为同一物种,且有极大可能为斑鳜,同时系统发生树的结果也从一定程度上支持了Liu、宋书莉、章群等人的观点,长体鳜可归入鳜属,鳜亚科下设鳜属、少鳞鳜属即可。
DNA是遗传物质的载体。利用DNA中的一些重复序列,我们能获得有关物种鉴别和遗传学鉴定的重要信息。例如,由于在DNA中多次出现,而在不同个体间表现出多态性的简单重复序列。就如Schlotterer等[26]所指出的那样,微卫星标记在遗传多态性分析、种群遗传学研究和亲缘关系推断中得到了广泛的应用,已成为遗传学研究和物种鉴定中的重要工具。2017年Tian等[27]从8 923个含SSR的序列(转录本>1 kb)中鉴定了总共991个SSR引物对并成功开发了能有效区分翘嘴鳜、斑鳜和大眼鳜的18个种特异性微卫星标记。但与核基因相比,线粒体DNA为裸露的环状双链DNA分子,结构简单,进化速度快,被广泛应用在鱼类群体遗传分化和种群结构特征分析研究当中[9]。地理隔离是影响物种遗传分化的重要因素,麻智芳等[28]发现清水江斑鳜下游群体的Cytb基因序列遗传多样性高于上游群体。通过分析不同地理种群的斑鳜线粒体基因,我们发现待检样本与韩国Changju斑鳜的遗传差异较小,而与长江及长江以南水系的斑鳜存在一定的遗传差异,这可能受到了地理隔离的影响。我国境内水系可划分为七大水系,河北省鳜属鱼类种质资源保护区主要位于白洋淀和柳河,白洋淀所在的大清河与柳河所属的滦河从水系划分上属于海河水系;鸭绿江是中朝两国的界河,在我国它流至辽宁省丹东市后汇入黄海[29]。从地理距离上看,在自然基因交流方面,海河水系和松花江水系存在交流的可能性比和长江水系要更大一些。从地质史上,鸭绿江和海河同属于古黄河水系,较晚时期才发生分化,因而两个斑鳜群体的亲缘关系要更近一些。与之相似的是,荔波漳江的野生斑鳜群体虽然生活在珠江水系,但是与长江水系的陆水江斑鳜群体和洞庭湖斑鳜群体的亲缘关系更近,麻智芳等[28]推测虽然苗岭山脉崛起形成了地理屏障,但早更新世时两区域的祖先斑鳜生活在一起,母系祖先单倍型得到了保留,因而在系统发生树中两者聚为了一支。周文漪[30]研究测定了中国7条水系10个地理群体176尾斑鳜的线粒体细胞色素 b 基因全序列,将其划分为5个谱系。其中鸭绿江丹东斑鳜与海河淇县斑鳜共同组成了谱系4。另外,王伟伟等[31]利用AFLP分子标记了不同地理种群的斑鳜,其聚类分析结果显示,鸭绿江的斑鳜群体与海河群体聚为一支,长江、钱塘江、闽江和西江四个群体聚为另一支,推测可能是秦岭山脉的形成导致了斑鳜的南北群体分化。本次的研究结果恰恰与之前周文漪、王伟伟等的研究相印证。
遗传多样性指的是群体之间以及群体内部的遗传差异,与物种的生存、适应能力以及进化的潜能密不可分,是分析种群资源现状的重要指标。杂合度是评估群体遗传变异的重要参数,杂合度越大,群体的遗传变异越大。PIC可以用来衡量标记的多态性,它的大小由等位基因的个数和等位基因的频率共同决定。通常情况下,当PIC<0.25时,该位点的多态性低;0.25≤PIC<0.5时,该位点多态性适中;PIC≥0.5时,该位点多态性高[32]。Ke等[33]利用10对微卫星标记评估了珠江流域不同地理群体泥鳅的遗传多样性,发现PIC=0.478 1,H0=0.389 2,遗传分化程度较高。罗慧等[34]利用SNP基因分型技术分析了青海湖水域6个地理群体的青海湖裸鲤的遗传多样性,结果发现H0为0.459 4~0.482 3,遗传多样性水平较高。在本研究中,整个群体的近交系数平均值约为0,整个群体的观测杂合度H0平均值为0.330,PIC平均值为0.269;SSH01亚群H0平均值为0.330,PIC平均值为0.270;SSH02亚群H0平均值为0.329,PIC平均值为0.230,SSH01和SSH02在观测杂合度方面不存在差异。这表明,此斑鳜群的遗传多样性水平较低,推测可能与人为或自然的干扰有关。曾可为等[32]分析了武汉、顺德、韶关等12个群体的遗传多样性,结果显示顺德的H0和He均为0.30,但PIC均值为0.53~0.64,与我们的结果相差较大,可能是分析方法不同的原因。另外,还可能与采集地点有关。
共祖系数,又名亲缘系数,指的是两个个体间加性基因效应间的相关。虽然近交能够产生出具有优良性状的纯种后代,但是也会产生有害基因的纯合个体。当群体规模较小且个体间亲缘较近,又不与外界进行基因交流时,就极易发生近交衰退。栾培贤等[35]开发了一套水产动物基因组近交分析软件工具包,能利用多种统计基因组学分析方法提供出基因组共祖系数、血缘同源、状态同源、基因组近交系数等数据。本研究中,各个体间共祖系数在-0.117 4~0.360之间,其中共祖系数为0的占总体的71.41%,可通过跨区域引种或者增殖放流的方法改善目前遗传多样性较低的现状。
PCA分析后进行的聚类分析显示,该群体可分为3个遗传结构,Cluster0、Cluster1、Cluster2各占比60%、25%、15%,并且Cluster0和Cluster1的遗传相似度较高。Admixture分析显示,该群体最有可能存在2个祖先,同时样本可分为3类,正好与之前PCA的聚类结果互相验证。2013年Cao等[36]对珠江、钱塘江、鸭绿江和长江的三个支流采集到的斑鳜进行了基于7个微卫星位点的聚类分析,发现在排除基因座CBD120时,所有种群都能形成两个聚类,并且与特定地理区域相关联。
连锁不平衡分析是指分属两个或两个以上基因座位的等位基因同时出现在一条染色体上的几率,高于随机出现的频率,呈现出一种相互关联的现象。连锁不平衡分析的结果表明,在95%的置信区间下,群体两个单倍型SSH01和SSH02的Ne大小均值分别是17.7和32.0,与单倍型群体的数量成反比,符合遗传漂变的规律。
总的来说,从研究结果来看,目前河北省内的鳜科鱼类大概率为斑鳜,存在两个单倍型SSH01和SSH02,与韩国Changju斑鳜亲缘性更近。研究群体的遗传多样性较低,有极大可能存在两个祖先,个体间的亲缘关系较近,应采取建立保护区和增殖放流的方式以防止近交退化,保持遗传的多样性。鳜的肉质鲜美,无肌间刺,养殖价值高,是我国重要名贵的淡水养殖鱼类。据2022年渔业统计年鉴,2020及2021年我国鳜鱼养殖总量均在30万t以上,其中广东、湖北、安徽、江西等省份是养殖的主产地,为各省的经济发展作出了重要贡献[37]。虽然鳜鱼并不是河北省主要养殖的经济水产动物之一,但是为了更好地保存水产动物种质资源,发展水产种业,探清河北省内鳜属鱼类的种质资源状况,分析其群体遗传结构和遗传多样性对全国斑鳜种质资源保护和开发利用意义重大。
参考文献:
[1] 范士琦,冯婧昀,苗晓敏,等.重庆养殖场鳜群体微卫星遗传多样性研究[J].水产养殖,2023, 44 (7): 18-23.
[2] 陈军,郑文彪,伍育源.等.鳜鱼和大眼鳜鱼年龄生长和繁殖力的比较研究[J].华南师范大学学报(自然科学版), 2003 (1): 110-114.
[3] 宓国强,陈建明,练青平,等.杂交鳜与鳜鱼、斑鳜肌肉营养成分和氨基酸含量比较[J].水产养殖, 2009, 30 (4): 35-37.
[4] 国家特色淡水鱼产业技术体系.中国鳜鱼产业发展报告[J].中国水产, 2021 (4): 23-32.
[5] ZHOU Y, FU H C, WANG Y Y, et al. Genome-wide association study reveals growth-related SNPs and candidate genes in mandarin fish (Siniperca chuatsi) [J].Aquaculture,2022,550: 737879.
[6] 刘洪军,宋爱环.加强水产种质资源保护利用[J].中国水产, 2020 (9): 31-32.
[7] YANG H L,SHEN L,HE Y F,et al.Status of aquatic organisms resources and their environments in Yangtze River system (2017–2021) [J]. Aquaculture and Fisheries, 2024, 9 (5): 833-850.
[8] 薛振宏,石敏,武艳丽,等.河北省水产种质资源保护区现状调查与研究报告[J].河北渔业, 2020(11): 41-45.
[9] 邱实.鱼类种质资源鉴定技术[J].黑龙江水产, 2018 (4):35-37.
[10] 赵金良,李思发,蔡完其,等.基于细胞色素b基因序列的东亚鳜类系统发育关系[J].动物学报, 2006(4): 676-680.
[11] 范士琦.重庆三个养殖场鳜群体的种质资源测定与分析[D].重庆:西南大学,2022.
[12] 余科.从江稻田鲤种质资源现状的鉴定与评估[D].贵阳:贵州大学, 2021.
[13] 李龑.基于线粒体、饲料蛋白需求、呼吸与排泄鲤种质资源鉴定与评价[D].大连:大连海洋大学, 2023.
[14] 陈信忠,郭书林,龚艳清.鱼类DNA条形码技术的应用进展[J].水产科学,2017,36(6): 834-842.
[15] 于潇.部分软骨鱼类线粒体基因组特征分析和cox1基因的鉴定应用[D].济南:山东大学, 2017.
[16] 程汉良,彭永兴,董志国,等.基于线粒体细胞色素c氧化酶亚基Ⅰ基因序列的帘蛤科贝类分子系统发育研究[J].生态学报,2013,33 (9):2744-2753.
[17] 陈治,高天翔.线粒体12S与COI条形码对海洋鱼类的鉴定差异[J].海南热带海洋学院学报, 2023,30(2):10-16.
[18] 舒璐,林佳艳,徐源,等.基于环境DNA宏条形码的洱海鱼类多样性研究[J].水生生物学报,2020,44(5):1080-1086.
[19] FENG X,LI B,CHEN Y F,et al.Species-level monitoring of rare and invasive fishes using eDNA metabarcoding in the middle and upper Yarlung Zangbo River, Tibet [J].Water Biology and Security,2023,2(1):100089.
[20] 李思忠.鳜亚科鱼类地理分布的研究[J].动物学杂志,1991(4):40-44.
[21] 周才武,杨青,蔡德霖.鳜亚科SINIPERCINAE鱼类的分类整理和地理分布[J].动物学研究,1988(2):113-125.
[22] LIU H Z,CHEN Y Y.Phylogeny of the sinipercine fishes with some taxonomic notes[J].Zoological Research,1994,15:1-12.
[23] FANG P W,CHONG L T.Study on the fishes referring to Siniperca of China [J].Sinensia,1932,2 (12):137-200.
[24] 章群,任岗,钱开诚,等.鳜类系统发育的线粒体Cytb基因全序列分析[J].生态科学,2006 (5):430-432+436.
[25] 宋书莉,赵金良,李晨虹.鳜属鱼类的进化和遗传多样性研究[C]//中国水产学会.2017年中国水产学会学术年会论文摘要集. 上海水产大学水产与生命学院,2017:146.
[26] SCHLOTTERER C,RITTER T,HARR H H.Microsatellites revisited [J].Journal of Molecular Ecology,1998,7(5):769-779.
[27] TIAN C X,GUO W J,LIANG X F,et al.Identification of species-specific microsatellite markers in three Siniperca species by RNA-Seq[J].Biochemical systematics and ecology,2017,70:126-131.
[28] 麻智芳,潘秋芝,安苗,等.基于mtDNA D-Loop区和Cytb基因的清水江斑鳜群体遗传多样性分析[J].海洋渔业,2022,44(6):657-669.
[29] 我国七大水系[J].中国水能及电气化,2022(2):69-70.
[30] 周文漪.基于线粒体细胞色素b的7水系斑鳜遗传多样性分析[D].广州:暨南大学,2014.
[31] 王伟伟,赵金良,李思发,等.斑鳜不同地理群体遗传变异的AFLP分析[J].水生生物学报,2009,33(2):304-309.
[32] 曾可为,宋文,王青云,等.基于微卫星标记的鳜种质遗传多样性与群体遗传结构分析[J].华中农业大学学报,2019,38(6):104-115.
[33] KE X L,LIU J,GAO F Y,et al.Analysis of genetic diversity among six dojo loach (Misgurnus anguillicaudatus) populations in the Pearl River Basin based on microsatellite and mitochondrial DNA markers[J].Aquaculture Reports,2022,27:101346.
[34] 罗慧,方弟安,何苗,等.基于SNP标记的青海湖裸鲤遗传多样性及种群结构研究[J].南方水产科学,2023,19(1):86-96.
[35] 栾培贤,张晓峰,户国.水产动物基因组近交分析软件的开发和应用[J].水产学杂志,2021,34(4):79-84.
[36] CAO L,LIANG X F,DU Y Q,et al.Genetic population structure in Siniperca scherzeri (Perciformes: Siniperca) in China inferred from mitochondrial DNA sequences and microsatellite loci[J].Biochemical Systematics and Ecology,2013,51:160-170.
[37] 农业农村部渔业渔政管理局,全国水产技术推给总站,中国水产学会.2022中国渔业统计年鉴[M].北京:中国农业出版社,2023:2.
Identification of germplasm resources of indigenous
Mandarin fish in Hebei Province
WANG Jiangjiang GAO Xiaotian ZHAO Chunlong
YU Qi2, ZHAO Xin1, SUN Yanfeng1, WU Chengbin1
(1. Ocean College, Hebei Agricultural University, Qinhuangdao 066003, China;
2. Hebei Academy of Marine and Fishery Sciences, Qinhuangdao 066003, China)
Abstract:In order to investigate the status quo of mandarin fishes in Hebei Province and provide better reference data for the protection of mandarin fishes germplasm resources in China, the mandarin fishes populations were identified at the molecular level and the correlation analysis of genetic diversity level was carried out with the support of mitochondrial COX1 and genome-wide SNP markers. The results showed that 40 homologous sequences of COX1 with a total length of 697 bp and two haplotype sequences of SSH01 and SSH02 were obtained. The phylogenetic tree constructed by the maximum likelihood method showed that the two haplotypes clustered first into one, and then clustered A0i15RXv0ct1fz4jX9G35Q==with Siniperca scherzeri. In the phylogenetic tree of geographic population, two haplotypes were clustered together with the reference to Changju S. scherzeri in Korea . The mean of observed heterozygosity (Ho) was 0.330, the mean of PIC was 0.269, and the mean of inbreeding coefficient was about 0. There was no significant difference in observed heterozygosity between SSH01 and SSH02 (P>005). The coancestry coefficient was 0 in 71.41% of the population, and there were more closely related individuals. PCA analysis resnlts showed that the population can be divided into 3 genetic structures, i.e., Cluster0, Cluster1 and Cluster2; The Admixture showed that two ancestors were most likely present; The result of linkage disequilibrium analysis was that the Ne range of the population was 14.8~27.6 under 95% confidence interval. S. scherzeri in Hebei Province has two haplotypes with low population genetic diversity. S. scherzeri is closely related to Changju S. scherzeri in Korea. S. scherzeri in Hebei Province can be protected by artificial introduction and breeding and release.
Key words:Siniperca scherzeri; mitochondrial COX1; SNP; genetic diversity
(收稿日期:2024-06-13)
