美味猕猴桃MYB家族鉴定及其对水淹胁迫的响应
2024-06-25乐有章王建军戢小梅迟文超林之希
乐有章 王建军 戢小梅 迟文超 林之希
摘要:基于美味猕猴桃(Actinidia deliciosa)二代转录组测序数据,通过多种生物信息学手段鉴定和分析了AcMYB家族基因。结果表明,在美味猕猴桃转录组数据中共获得64个具有完整ORF序列的AcMYB家族成员,将其分成4个亚族,其中亚族Ⅰ共34个成员,亚族Ⅱ共18个成员,亚族Ⅲ共10个成员,亚族Ⅳ共2个成员。水淹处理前后表达分析显示,AcMYB_214187、AcMYB_25788等AcMYB成员美味猕猴桃根中的表达水平明显降低,快速响应水淹胁迫,而叶中的表达水平差异不明显。AcMYB_19450、AcMYB_30021等AcMYB成员根中的表达水平差异不明显,但叶中的表达水平明显降低,可能原因是根接收到低氧胁迫信号后,将其传递到地上部并发挥作用,通过转录水平调控美味猕猴桃对低氧胁迫的响应。研究结果为后续开展美味猕猴桃MYB转录因子研究、促进抗逆境育种、提高产量提供了依据。
关键词:美味猕猴桃(Actinidia deliciosa);AcMYB;鉴定;水淹胁迫;响应
中图分类号:S663.4 文献标识码:A
文章编号:0439-8114(2024)05-0215-08
DOI:10.14088/j.cnki.issn0439-8114.2024.05.037 开放科学(资源服务)标识码(OSID):
Identification of the MYB family of Actinidia deliciosa and its response to flooding stress
YUE You-zhang1,WANG Jian-jun2, JI Xiao-mei1, CHI Wen-chao2,LIN Zhi-xi2
(1.Wuhan Academy of Agricultural Sciences,Wuhan 430075,China;
2. College of Horticulture,Nanjing Agricultural University,Nanjing 210095,China)
Abstract: Based on the second-generation transcriptome sequencing data of Actinidia deliciosa, AcMYB family genes were identified and analyzed using various bioinformatics methods. The results showed that a total of 64 AcMYB family members with complete ORF sequences were obtained from the transcriptome data of Actinidia deliciosa, which were divided into 4 subgroups. Among them, subgroup I had 34 members, subgroup II had 18 members, subgroup III had 10 members, and subgroup IV had 2 members. Expression analysis before and after waterlogging treatment showed that the expression levels of AcMYB members such as AcMYB214187 and AcMYB25788 were significantly reduced in the roots of Actinidia deliciosa, responding quickly to waterlogging stress, while there was no significant difference in expression levels in the leaves. The expression levels in the roots of AcMYB members such as AcMYB19450 and AcMYB30021 were not significantly different, but the expression levels in the leaves were significantly reduced. This might be due to the fact that the roots, upon receiving hypoxic stress signals, transmitted them to the aboveground parts and exerted their effects, regulating the response of Actinidia deliciosa to hypoxic stress at the transcriptional level. The research results provided a basis for further research on MYB transcription factors in Actinidia deliciosa, promoting stress resistance breeding, and improving yield.
Key words: Actinidia deliciosa; AcMYB; identification; flooding stress; response
收稿日期:2023-07-29
作者简介:乐有章(1966-),男,湖北通山人,高级农艺师,主要从事猕猴桃资源搜集、评价及猕猴桃栽培育种工作,(电话)13986282616(电子信箱)390086056@qq.com;通信作者,王建军(1969-),男,浙江嵊州人,副教授,硕士,主要从事园艺作物遗传与育种工作,(电话)13951709767(电子信箱)1991009@njau.edu.cn。
乐有章,王建军,戢小梅,等. 美味猕猴桃MYB家族鉴定及其对水淹胁迫的响应[J]. 湖北农业科学,2024,63(5):215-222.
MYB是植物中最大的转录因子家族之一,且广泛存在与真核生物中,功能丰富,参与植物多种生物学途径的转录调控[1]。MYB转录因子N端存在高度保守的DNA结合结构域(DNA-binding domain,DBD),该结构域通常由4个约52个氨基酸的不完全氨基酸重复序列(R)组成,这些重复序列被重命名为R1、R2和R3,在植物中,MYB转录因子包含1~4个DNA结合重复序列。然而,大多数MYB蛋白包含2个重复序列,属于R2R3-MYB亚家族[2]。许多植物中的MYB家族成员已被鉴定,拟南芥中共鉴定到196个MYB成员,其中包含126个R2R3-MYB亚家族,水稻中共鉴定到185个MYB成员,包含109个R2R3-MYB亚家族[3]。
多项研究表明,MYB转录因子在植物胁迫响应中发挥关键作用。过表达OsMYB48-1增强了水稻的耐旱性和耐盐性,且通过ABA途径参与干旱和盐胁迫的调控[4]。R2R3类型的MYB成员OsMYB2可以调控水稻的耐盐性、耐旱性和耐冷性[5]。过表达猕猴桃中AcMYB3R增强了拟南芥转基因植株的耐旱性和耐盐性[6]。
猕猴桃因富含高维生素C而享誉全球,广受消费者的喜爱。近年来,随着中国猕猴桃产业的不断发展,其栽培面积和产量居世界前列。猕猴桃对涝害胁迫敏感[7],同时由于南方年均降雨较多,北方短期内强降雨导致积水,在猕猴桃种植过程中易发生涝害,严重阻碍了猕猴桃稳定生产[8]。通过深入研究猕猴桃耐涝性的生理和分子机制,可以减少涝害造成的损失[9]。因此,通过生物信息学分析手段挖掘猕猴桃对水淹胁迫的响应基因,对培育耐涝猕猴桃品种具有重要意义。本研究基于美味猕猴桃(Actinidia deliciosa)的转录组分析鉴定AcMYB家族成员,并分析水淹处理后AcMYB的逆境响应模式,对后续开展AcMYB转录因子功能研究,推进猕猴桃抗逆境育种,提高猕猴桃产量具有重要意义。
1 材料与方法
1.1 材料
猕猴桃品种为美味猕猴桃;对美味猕猴桃水淹处理2周左右,收集根和叶片组织,置于液氮快速冷冻,-80 ℃中保存用于RNA提取。
1.2 RNA-Seq测序
按照Trizol试剂盒说明书提取美味猕猴桃各组织的总RNA,经检测合格后等量混合,基于Illumina公司 Hiseq 4000(Illumina, San Diego, USA)平台进行转录组测序,并采用Trinity软件[10]拼接获取Unigenes。
1.3 AcMYB转录因子基因筛选
利用EMBOSS软件[11]预测美味猕猴桃Unigenes的开放阅读框(ORF),获取美味猕猴桃Unigenes编码蛋白质数据。从EMBL-EBI网站(https://pfam.xfam.org/)下载MYB保守域种子序列(序列号PF000249),利用HMMER 3.0软件建立隐马可夫模型(Profile HMM),检索美味猕猴桃Unigenes编码蛋白质数据库。根据MYB保守结构域序列覆盖度和E值等参数,利用Perl脚本筛选获得覆盖度高、置信度高的AcMYB转录因子序列。
1.4 AcMYB转录因子的系统进化分析
运用MEGA-X对筛选到的AcMYB转录因子蛋白序列进行多重比对,采用邻接法(Neighbor-joining method)构建系统进化树(参数设置:Poisson model、Pairwise deletion和Bootstrap,1 000次重复)。
1.5 AcMYB蛋白序列保守结构域分析
利用DNAMAN 7.0软件对筛选到的AcMYB蛋白序列进行多序列比对,获得保守结构域区域。利用MEME(http://meme-suite.org/)和WebLogo 3(http://weblogo.threeplusone.com/)对AcMYB保守结构域序列标签进行分析。
1.6 AcMYB理化性质分析
AcMYB氨基酸序列长度、分子大小和理论等电点等信息采用ProtParam tool(https://web.expasy.org/protparam/)分析。二级结构采用SOPMA(htttp://npsa-prabi.ibcp.fr/cgi-bin/nps)获取相关信息。
1.7 AcMYB基因的逆境响应模式分析
利用Bowtie 2.0软件(http://bowtie-bio.sourceforge.net/bowtie2/index.shtml)将下载的转录组reads数据与筛选到的AcMYB基因进行映射分析。使用Trinity包的Perl脚本语言,计算每个AcMYB基因的FPKM(Reads per kilobase of exon model per million mapped reads),分析AcMYB基因家族在水淹处理前后对水淹胁迫响应的表达水平。
2 结果与分析
2.1 RNA-Seq测序及组装
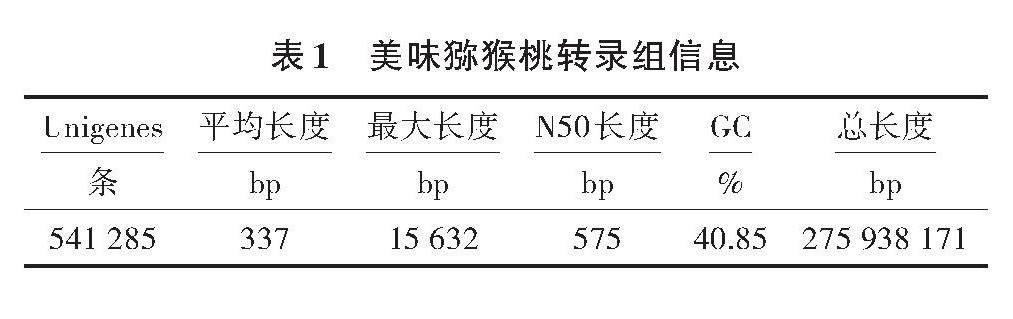
通过Illumina HiSeq?对混合RNA样品进行二代转录组测序,经组装、聚类后得到美味猕猴桃转录组数据,其中Unigenes为541 285条,总长度为275 938 171 bp,如表1所示。
2.2 AcMYB家族成员鉴定与系统进化分析
541 285条美味猕猴桃Unigenes经预测获取了170 578条蛋白序列(长度≥150 aa),利用Pfam获取MYB转录因子保守结构域序列,序列号为PF00249。共筛选到64条AcMYB蛋白序列。
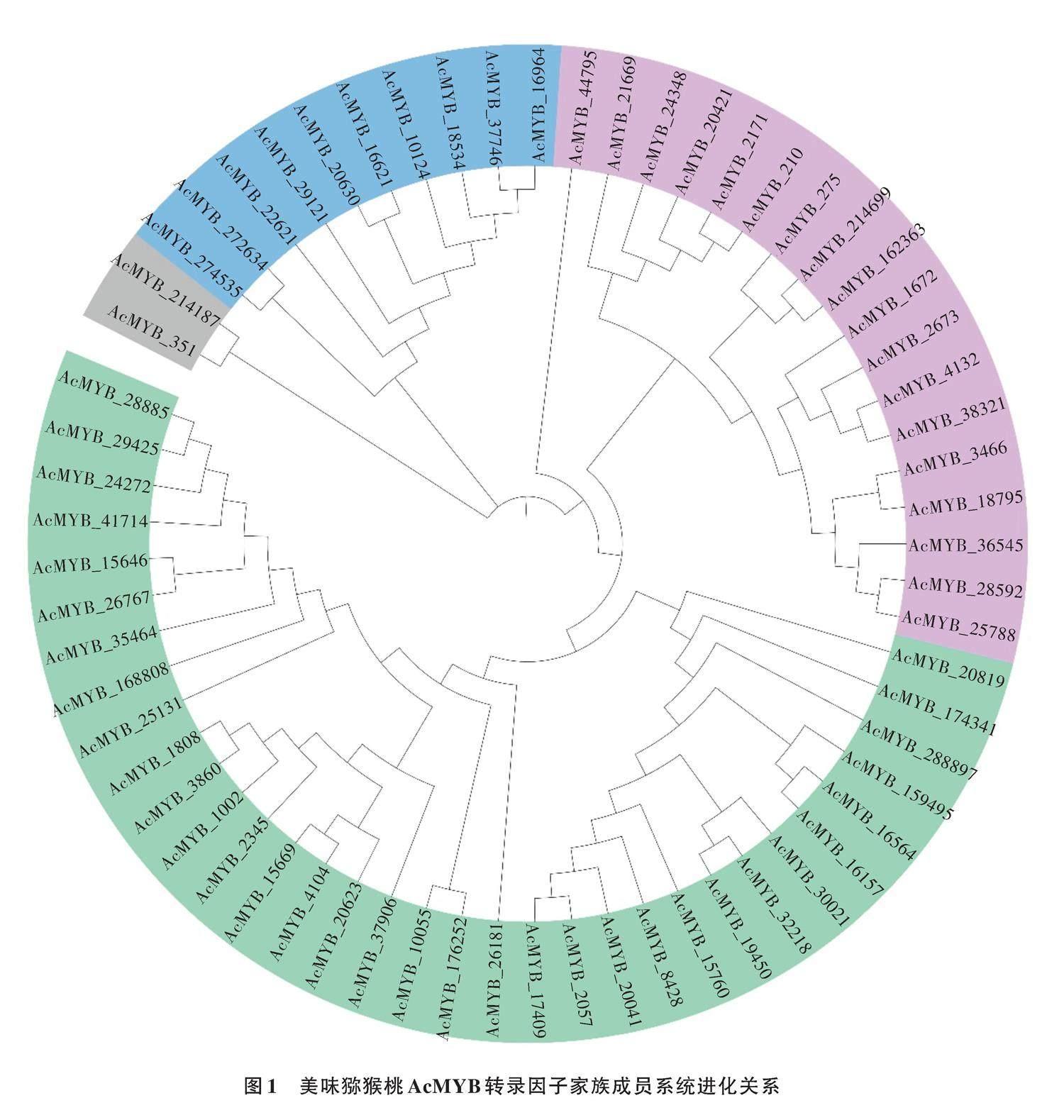
利用MEGA-X对AcMYB蛋白序列进行多序列比对,使用Neighbor Joining法构建系统进化树。根据AcMYB家族成员的亲缘关系和保守结构域将64个AcMYB成员分成4个亚族。亚族Ⅰ共34个成员,亚族Ⅱ共18个成员,亚族Ⅲ共10个成员,亚族Ⅳ共2个成员,如图1所示。
2.3 AcMYB保守结构域分析
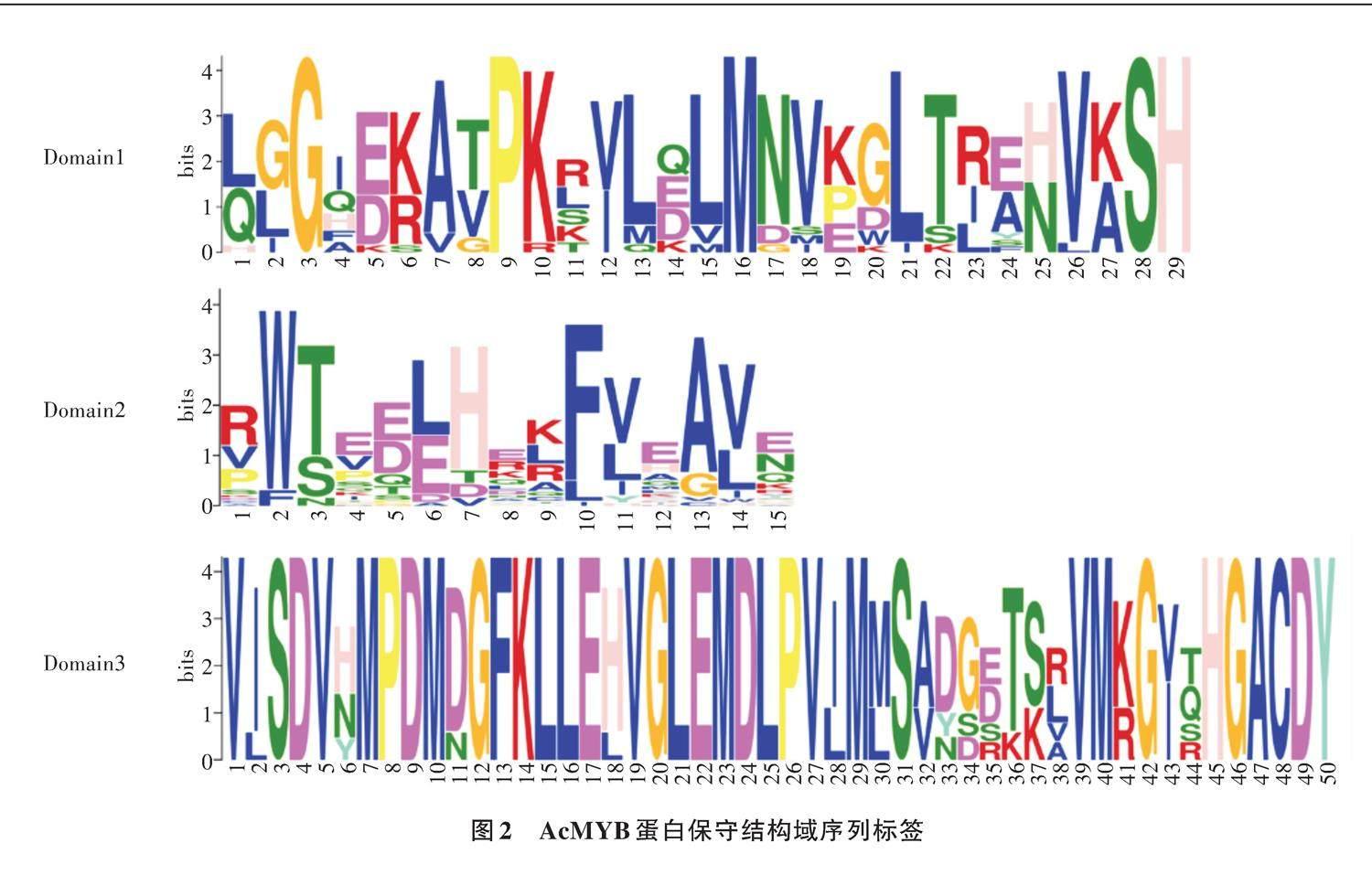
对美味猕猴桃AcMYB转录因子进行保守结构域分析,共预测到3个保守结构域,分别为AcMYB domain1、AcMYB domain2、AcMYB domain3。AcMYB domain1成员共27个,该结构域由29个氨基酸组成,其中有非常保守的甘氨酸(G)、脯氨酸(P)、甲硫氨酸(M)、丝氨酸(S)、组氨酸(H),以及较保守的赖氨酸(K)、亮氨酸(L)、缬氨酸(V);AcMYB domain1主要存在于亚族Ⅰ。AcMYB domain2成员共29个,由15个氨基酸组成,其中色氨酸(W)和苯丙氨酸(F)较保守;AcMYB domain2主要存在于亚族Ⅰ和亚族Ⅱ。AcMYB domain3成员共6个,该结构域由50个氨基酸组成且大部分氨基酸极为保守,如图2所示。
随后对AcMYB家族中不同成员的保守基序(motif)进行分析,共预测到19个motif,其中27个AcMYB成员都包含motif5,17个成员都包含motif2,13个成员都包含motif8(图3),表明motif5、motif2、motif8对AcMYB的功能起较为关键的作用。
2.4 AcMYB蛋白序列的理化性质分析
对AcMYB蛋白序列的理化性质进行分析,结果(表2)表明,不同亚族之间的等电点、相对分子质量和氨基酸长度等理化性质存在差异。亚族Ⅲ与其他亚族之间的差异较大,其等电点较低、相对分子质量较大、氨基酸长度较长、α-螺旋占比较高、β-折叠占比较低。亚族Ⅰ和亚族Ⅱ的蛋白序列具有较高的相似性。
2.5 AcMYB家族对逆境胁迫的响应模式
为了探究AcMYB家族基因对水淹胁迫的表达模式,对美味猕猴桃水淹处理前后的根、叶进行转录组分析。美味猕猴桃中大多数AcMYB转录因子在水淹处理后均显示差异表达,说明AcMYB家族作为上游调节因子在水淹低氧胁迫中发挥重要功能。对差异表达基因进行聚类分析,结果(图4)显示,聚类Ⅰ中AcMYB转录因子主要在根中高表达,主要包括AcMYB_288897、AcMYB_159495、AcMYB_29425等亚族Ⅰ成员,AcMYB_44795、AcMYB_38321等亚族Ⅱ成员和少部分AcMYB_37746、AcMYB_22621等亚族Ⅲ成员。通过水淹处理后,聚类Ⅰ中AcMYB成员在根中表达明显降低。
在水淹处理后,聚类Ⅱ中AcMYB_1672/AcMYB_2171等AcMYB成员根、叶的表达水平明显升高,可能在低氧胁迫中发挥正调节作用。在水淹处理后,聚类Ⅲ中AcMYB_1002、AcMYB_20041、AcMYB_15669等AcMYB成员根中的表达水平明显降低,而在叶中的表达水平明显升高;在水淹处理后,聚类Ⅲ中AcMYB_214187、AcMYB_25788等AcMYB成员根中的表达水平明显降低,而叶中的表达水平差异不明显。在水淹处理后,聚类Ⅳ中AcMYB_37906、AcMYB_17409、AcMYB_28885等AcMYB成员根、叶中的表达水平明显降低;聚类Ⅳ中AcMYB_19450、AcMYB_30021等AcMYB成员根中的表达水平差异不明显,但叶中的表达水平明显降低,可能原因是根接收到低氧胁迫信号后,将其传递到地上部并发挥作用,通过转录水平调控猕猴桃对低氧胁迫的响应。
以上结果表明,美味猕猴桃中的大部分AcMYB转录因子均参与水淹胁迫响应,有些AcMYB成员在根中快速响应水淹胁迫,有些AcMYB成员在叶中接受到来自根的胁迫信号后产生相应的响应。
3 小结与讨论
水淹胁迫本质是根系缺氧诱发的一系列生理变化,如光合作用抑制、无氧呼吸[12,13]。长时间的厌氧呼吸会导致有害物质(如乙醛、乙醇和重金属离子)在根际中积累,导致根部损伤甚至植物死亡[14,15]。拟南芥中VII族乙烯响应因子(ERF-VIIs)参与水淹胁迫信号传递的内在机制逐渐被解析[16]。同时,SR1-WRKY33-RAP2.2模块开关与N端降解因子(N-degron)途径存在串扰,调节拟南芥对水淹胁迫的响应[17,18]。猕猴桃中耐涝相关基因的鉴定集中于ERF家族。通过转录组分析,猕猴桃KR5中鉴定了131个ERF基因,发现ERF-VIIs中6个AvERF基因在水淹处理12 h后表达显著上升[19]。有研究发现ERF-VIIs亚家族的2个成员AcERF75和AcERF1直接调控乙醇脱氢酶AcADH74的表达[20]。然而关于MYB参与水淹胁迫响应的研究较少。
本研究基于转录组数据从美味猕猴桃中筛选鉴定出了64个AcMYB家族成员。通过生物信息学分析,明确了64个AcMYB基因家族成员的基本信息,包括保守结构域、蛋白理化特性等,并初步获得了参与水淹胁迫响应的成员,为进一步研究这些响应基因的功能奠定了基础。转录组测序的深度、拼接效果等诸多原因可能会导致MYB家族成员鉴定的缺失,其准确性还有待进一步的验证。
参考文献:
[1] WANG X, NIU Y, ZHENG Y. Multiple functions of MYB transcription factors in abiotic stress responses[J]. International journal of molecular sciences,2021, 22(11): 6125.
[2] MILLARD P S, KRAGELUND B B, BUROW M. R2R3 MYB transcription factors—Functions outside the DNA-Binding domain[J]. Trends in plant science,2019, 24(10): 934-946.
[3] CHEN Y H,YANG X Y,HE K, et al. The MYB transcription factor superfamily of arabidopsis: Expression analysis and phylogenetic comparison with the rice MYB family[J]. Plant molecular biology,2006, 60: 107-124.
[4] XIONG H, LI J, LIU P, et al. Overexpression of OsMYB48-1, a novel MYB-related transcription factor, enhances drought and salinity tolerance in rice[J]. PLoS one,2014, 9(3): e92913.
[5] YANG A, DAI X, ZHANG W. A R2R3-type MYB gene, OsMYB2, is involved in salt, cold, and dehydration tolerance in rice[J]. Journal of experimental botany,2012, 63(7): 2541-2556.
[6] LI X, GUO C, AHMAD S, et al. Systematic analysis of MYB family genes in potato and their multiple roles in development and stress responses[J]. Biomolecules,2019, 9(8): 317.
[7] XU P, LIU Z, FAN X, et al. De novo transcriptome sequencing and comparative analysis of differentially expressed genes in Gossypium aridum under salt stress[J]. Gene,2013, 525(1): 26-34.
[8] 白丹凤,李志,齐秀娟,等. 4种基因型猕猴桃对淹水胁迫的生理响应及耐涝性评价[J]. 果树学报,2019, 36(2): 163-173.
[9] BUTSAYAWARAPAT P, JUNTAWONG P, KHAMSUK O, et al. Comparative transcriptome analysis of waterlogging-sensitive and tolerant zombi pea (Vigna vexillata) reveals energy conservation and root plasticity controlling waterlogging tolerance[J]. Plants (Basel),2019, 8:264.
[10] GRABHERR M G, HAAS B J, YASSOUR M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature biotechnology, 2011, 29(7): 644-652.
[11] RICE P, LONGDEN I, BLEASBY A. EMBOSS: The European molecular biology open software suite[J]. Trends in genetics,2000, 16(6): 276-277.
[12] PAN J, SHARIF R, XU X, et al. Mechanisms of waterlogging tolerance in plants: Research progress and prospects[J]. Frontiers in plant science,2021, 11: 627331.
[13] YAN K, ZHAO S, CUI M, et al. Vulnerability of photosynthesis and photosystem I in Jerusalem artichoke (Helianthus tuberosus L.) exposed to waterlogging[J]. Plant physiology and biochemistry,2018, 125: 239-246.
[14] ZHANG Z, FU D, SUN Z, et al. Tonoplast-associated calcium signaling regulates manganese homeostasis in Arabidopsis[J]. Molecular plant,2021, 14(5): 805-819.
[15] QI X, LI Q, MA X, et al. Waterlogging induced adventitious root formation in cucumber is regulated by ethylene and auxin through reactive oxygen species signalling[J]. Plant, cell & environment,2019, 42(5): 1458-1470.
[16] HINZ M, WILSON I W, YANG J, et al. Arabidopsis RAP2.2: An ethylene response transcription factor that is important for hypoxia survival[J]. Plant physiology,2010, 153(2): 757-772.
[17] LIU B, JIANG Y, TANG H, et al. The ubiquitin E3 ligase SR1 modulates the submergence response by degrading phosphorylated WRKY33 in Arabidopsis[J]. The plant cell,2021, 33(5): 1771-1789.
[18] TANG H, BI H, LIU B, et al. WRKY33 interacts with WRKY12 protein to up-regulate RAP2.2 during submergence induced hypoxia response in Arabidopsis thaliana[J]. The new phytologist,2021, 229(1): 106-125.
[19] BAI D, LI Z, HU C, et al. Transcriptome-wide identification and expression analysis of ERF family genes in Actinidia valvata during waterlogging stress[J]. Scientia horticulturae,2021, 281: 109994.
[20] LIU J, CHEN Y, WANG W, et al. Transcription factors AcERF74/75 respond to waterlogging stress and trigger alcoholic fermentation-related genes in kiwifruit[J]. Plant science,2022, 314: 111115.
