小麦新种质“普冰3228”穗下节长度QTL定位与候选基因分析
2024-04-24王健胜王二伟马爱锄程世平
王健胜,王二伟,马爱锄,程世平
(1. 平顶山学院,河南 平顶山 467000;2. 河南省生态经济型木本植物种质创新与利用重点实验室, 河南 平顶山 467000;3. 平顶山市农业科学院,河南 平顶山 467001)
【研究意义】茎秆是小麦植株形态的主要组成部分,也是小麦育种实践中重点考察的指标。小麦茎秆具有多种重要功能,包括水分及营养物质的输导和贮藏,对穗部的支撑,同时也具有一定的光合作用[1-2]。茎秆最突出的功能是其与小麦的倒伏性密切相关。育种实践表明,抗倒伏是决定小麦最终产量的关键因素,倒伏性差可能导致小麦减产甚至绝产。小麦茎秆是由多个茎节组成,其中处于小麦穗部下的第一个节(穗下节)的作用尤为突出[1],目前穗下节已作为小麦种质筛选及新品种培育的重要指标,因此,开展小麦穗下节遗传机制分析对其遗传改良具有重要意义。【前人研究进展】目前,国内外有关小麦穗下节的相关研究非常有限,只有少数学者针对小麦穗下节进行了初步探索。项超等[1]利用91份小麦材料研究了穗下节性状与灌浆速率及产量相关因素的关系,结果发现,穗下节长度、粗度、体积与千粒重均呈正相关线性回归显著关系,穗下节长度最长类型灌浆特性综合表现最好,穗下节粗度与中等类型灌浆特性无明显差异,穗下节长度最长类型具有较高千粒重,因此,该研究认为,在西南地区选择穗下节最长、粗细中等的材料有助于选育灌浆特性、产量表现优异的小麦新品种。王瑞清等[2]探讨了穗下节对小麦粒重的影响,研究发现,在对14个小麦品种穗下节遮光后,小麦千粒重均下降,下降幅度为4.88%~23.92%,表明穗下节不仅有支持和输导作用,也是重要的光合器官,对小麦千粒重提高具有重要影响。Liu等[3]以5个重组自交系群体为材料,在构建其高密度分子遗传连锁图谱的基础上,利用该图谱对小麦穗下节直径及其相关性状进行QTL定位分析,结果在1A、1D、2B、2D、3B、3D、4A、4B、4D、5A、5B、6B和7D染色体上共发现25个相关QTL,其中主效且稳定的QTL有5个,QUid.sau-2CN-1D.1、QUid.sau-2SY-1D和QUid.sau-SC-3D是首次发现的穗下节直径QTL;同时,研究开发了与部分穗下节QTL连锁的KASP标记,这些标记可以有效用于目标QTL近等基因系的构建;另外,基于发现的主效且稳定的QTL,研究预测分析了穗下节直径候选基因。Yu等[4]利用2个重组自交系群体开展小麦穗下节长度QTL定位并对其在小麦株高方面的效应做分析,共检测到穗下节长度相关QTL 18个,该QTL主要位于1B、1D、2A、2B、3A、3B、4A、4D、5A、5B、6B、6D和7B染色体上,其表型解释变异率为2.50%~20.10%,该QTL中4个来自亲本‘Opata85’,而其余14个均来自人工合成小麦‘W7984’。Sang等[5]利用2个中国小麦优异品种构建的双单倍体对小麦穗下节的总维管束、大维管束、小维管束及大维管束/小维管束进行QTL分析,检测到11个加性QTL和1对上位性QTL。【本研究切入点】虽然前人针对小麦穗下节开展了部分研究,但主要围绕穗下节直径、穗下节维管束等相关性状展开,而专门针对小麦穗下节长度的研究极少,目前小麦穗下节长度遗传机制仍不清楚。本研究材料“普冰3228”是普通小麦与冰草通过远缘杂交获得的遗传稳定新种质[6-8],除具有突出的高产、抗病等优异性状外,其穗下节长度表现也很突出,而有关该种质穗下节长度的研究鲜有报道。【拟解决的关键问题】本研究将以“普冰3228”与“京4839”构建的重组自交系群体为材料,对“普冰3228”穗下节长度进行QTL定位及候选基因分析,以期为小麦穗下节遗传机制解析及“普冰3228”在育种实践中的有效利用提供一定依据。
1 材料与方法
1.1 研究材料
本研究材料是小麦新种质“普冰3228”与“京4839”杂交产生的重组自交系群体(RIL),该群体通过单籽粒法构建,包括210个株系,均由中国农业科学院作物科学研究所李立会课题组提供。
1.2 小麦田间种植及其穗下节长度测定
在2019—2020年和2020—2021年将“普冰3228”ד京4839”RIL群体分株系在河南平顶山和陕西杨凌2种环境下种植,株系间采用随机区组设计,每个株系种植4行,3个重复。种植行长保持2 m,行距20 cm,株距6.7 cm,单籽粒点播。试验地周围均种植保护行,按照普通产田水平进行田间统一管理。待小麦成熟后,在每个株系中随机选择5~6株,利用直尺对小麦穗下节长度进行测定,最后取其平均值作为该株系穗下节长度。
1.3 基因型检测
采用SDS法提取基因组DNA[9],取亲本和RIL群体幼苗叶片提取DNA,提取的DNA保存至TE中,并用琼脂糖凝胶电泳法检测其纯度。然后将遗传群体检测质量合格的样品DNA送往北京博奥京典生物技术有限公司,由该公司基于Illumina SNP Genotyping技术测试平台使用微珠芯片技术检测,其多态性使用Genomestudio v1.0软件分析,最终获得研究遗传群体的有效基因型。
1.4 遗传图谱构建及QTL定位
基于遗传定位群体的SNP基因型,利用QTL IciMapping 4.0 软件构建分子标记遗传连锁图谱,利用Mapmarker3.0软件绘制遗传连锁图谱。结合穗下节长度表型性状,利用IciMapping 4.0软件(http://www.isbreeding.net/)中的完备区间作图法(ICIM-ADD)对穗下节长度进行QTL检测[10-11],设置参数为步移距离1 cM,PIN 0.001,LOD阈值为2.5,并计算每个QTL的加性效应。QTL按照通用规则命名,具体为QTL+性状英文缩写+染色体+环境。
1.5 候选基因分析
以与穗下节长度性状紧密连锁SNP 标记序列为探针,以QTL定位置信区间作为候选基因的预测区间,在小麦基因组数据库(http://wheatomics.sdau.edu.cn/)上获得穗下节长度初步候选基因,然后将初步候选基因在小麦基因表达数据库(http://www.wheat-expression.com)中进行表达分析,获得穗下节长度相关候选基因,并对部分候选基因进行功能注释。
2 结果与分析
2.1 “普冰3228” × “京4839” RIL群体穗下节长度表型性状分析
“普冰3228” × “京4839” RIL群体穗下节长度存在较丰富的遗传差异(表1,图1)。在2020年平顶山地区,定位群体穗下节长度分布于34.00~117.50 cm,平均77.63 cm,而在同一地区的2021年,群体穗下节分布于16.00~77.00 cm,平均52.28 cm,由此可知,不同年度遗传群体在平顶山地区均具有较长的穗下节。2020年和2021年在平顶山地区变异系数分别为20.95%、16.01%,可见遗传群体在同一环境下的不同年度均具有丰富的遗传差异。在杨凌环境下,群体穗下节长度分布于19~53 cm,平均37.40 cm,由此可知,与平顶山环境相比,杨凌环境下遗传群体穗下节相对较短,同时该环境下的变异系数也较小,只有15.88%,表明杨凌环境下遗传群体的遗传差异相对较小。对该群体在3个环境下穗下节综合比较发现,编号为2、3、8、10、41、89、98、104和126株系穗下节表现均较好。研究也对该遗传群体穗下节长度的遗传力进行估算,发现该群体的遗传力表现较好,为86.39%。此外,研究对“普冰3228” × “京4839” RIL群体在不同环境下穗下节长度进行相关性分析,由表2可知,不同环境间该群体穗下节长度均呈正相关,在平顶山环境下的2020年和2021年其穗下节呈极显著正相关。从遗传群体穗下节长度在不同环境及不同年际的分布(图1)可以看出,其均表现为正态或近正态分布,说明该性状是受多基因控制的数量性状。
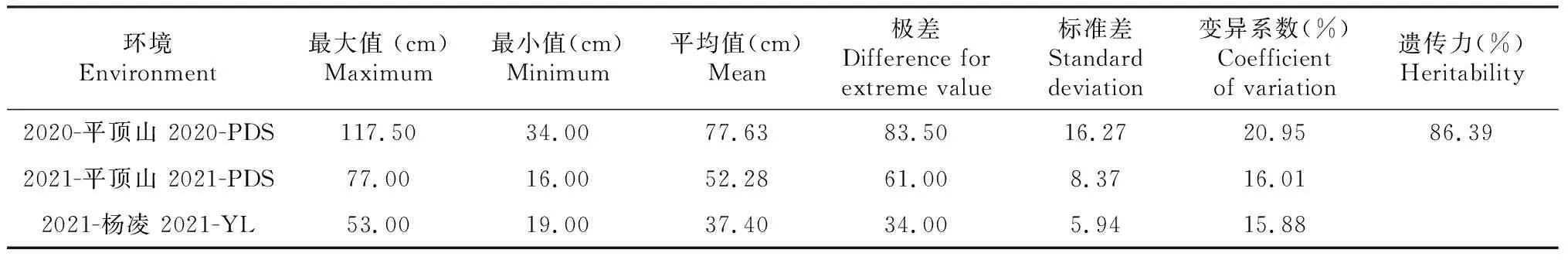
表1 “普冰3228” × “京4839” RIL群体穗下节长度差异及遗传力分析Table 1 The difference and heritability analysis for uppermost-internode length in the RIL population of ‘Pubing3228’בJing4839’

表2 “普冰3228” × “京4839” RIL群体穗下节长度在不同环境下的相关系数Table 2 The correlation coefficient for uppermost-internode length of the ‘Pubing3228’בJing4839’ RIL population under the different environments
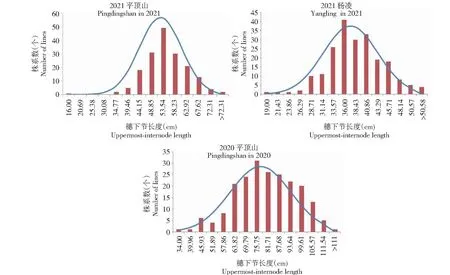
图1 “普冰3228” × “京4839” RIL群体穗下节长度分布Fig.1 The distribution of the uppermost-internode length in the RIL population of ‘Pubing3228’בJing4839’
2.2 “普冰3228” × “京4839” RIL群体穗下节长度QTL定位
研究共检测到小麦穗下节QTL 6个,该QTL主要分布于2B、4B、4D、5B和6D染色体上(表3,图2)。这些QTL的LOD值介于2.55~7.88,解释变异率分布于3.43%~15.84%。在发现的QTL中,定位于4B染色体上AX-109373490~AX-111540511区间和定位于4D染色体上AX-110984743~AX-109230716区间的2个QTL的表型解释变异率均超过10%,说明其为穗下节的主效QTL。多环境下稳定QTL对小麦穗下节遗传机制深入研究更为重要,本研究检测到1个穗下节稳定QTL,其位于4D染色体上的AX-110984743~AX-109230716区间,该QTL在平顶山和杨凌环境下均被检测到,同时从上述分析可知,该稳定QTL也是穗下节主效QTL,因此该QTL应作为穗下节重点基因位点在后期研究中予以关注。从QTL的加性效应来看,除4B、6D染色体上发现的2个QTL为负值外,其余QTL均为正值,表明绝大多数QTL均来自母本“普冰3228”。从不同环境下QTL检测效果来看,本研究在杨凌和平顶山均分别发现了3个QTL。
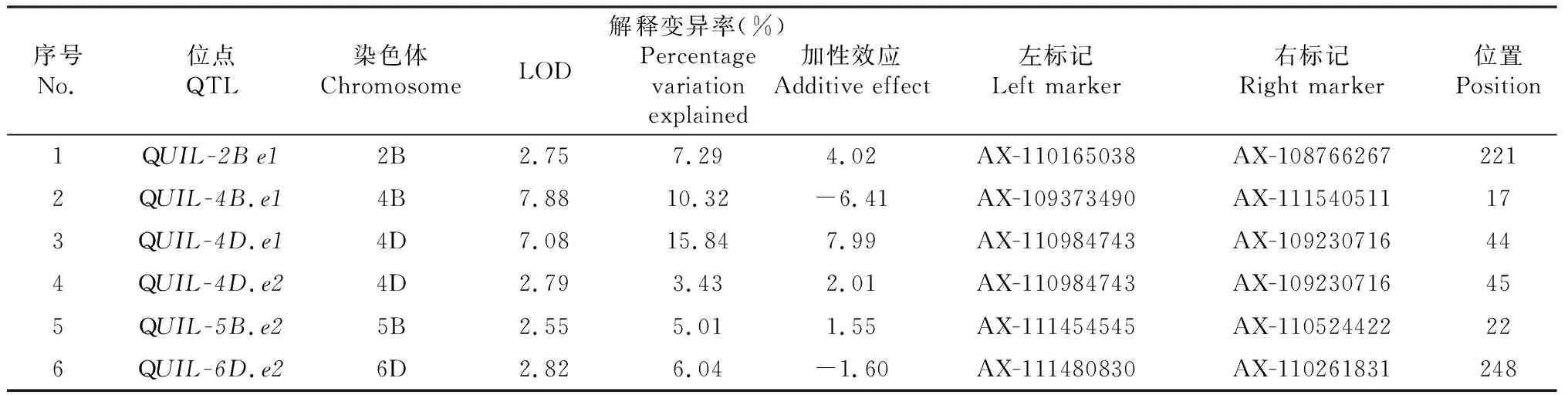
表3 “普冰3228” × “京4839” RIL群体穗下节长度QTL定位Table 3 QTL mapping for uppermost-internode length in the ‘Pubing3228’בJing4839’RIL population
2.3 小麦穗下节候选基因分析
基于上述穗下节长度QTL定位结果,在目标QTL定位遗传区段内获得穗下节初步候选基因674个。将这些候选基因分别在小麦根、茎、叶、籽粒等主要组织器官内进行模拟表达,筛选出只在小麦茎组织特异表达量大的基因作为穗下节长度候选基因,最终获得穗下节长度候选基因15个(表4),这些基因分布在小麦4D染色体上9994582~36839260 bp区段内,其中与AX-110984743位点关联的基因有9个,包括TraesCS4D01G014100、TraesCS4D01G019600、TraesCS4D01G035600、TraesCS4D01G026200、TraesCS4D01G039200、TraesCS4D01G040300、TraesCS4D01G041200、TraesCS4D01G044400、TraesCS4D01G045400,与AX-109230716位点关联的基因有6个,分别是TraesCS4D01G040300、TraesCS4D01G041200、TraesCS4D01-G044400、TraesCS4D01G045400、TraesCS4D01G058000、TraesCS4D01G061100。
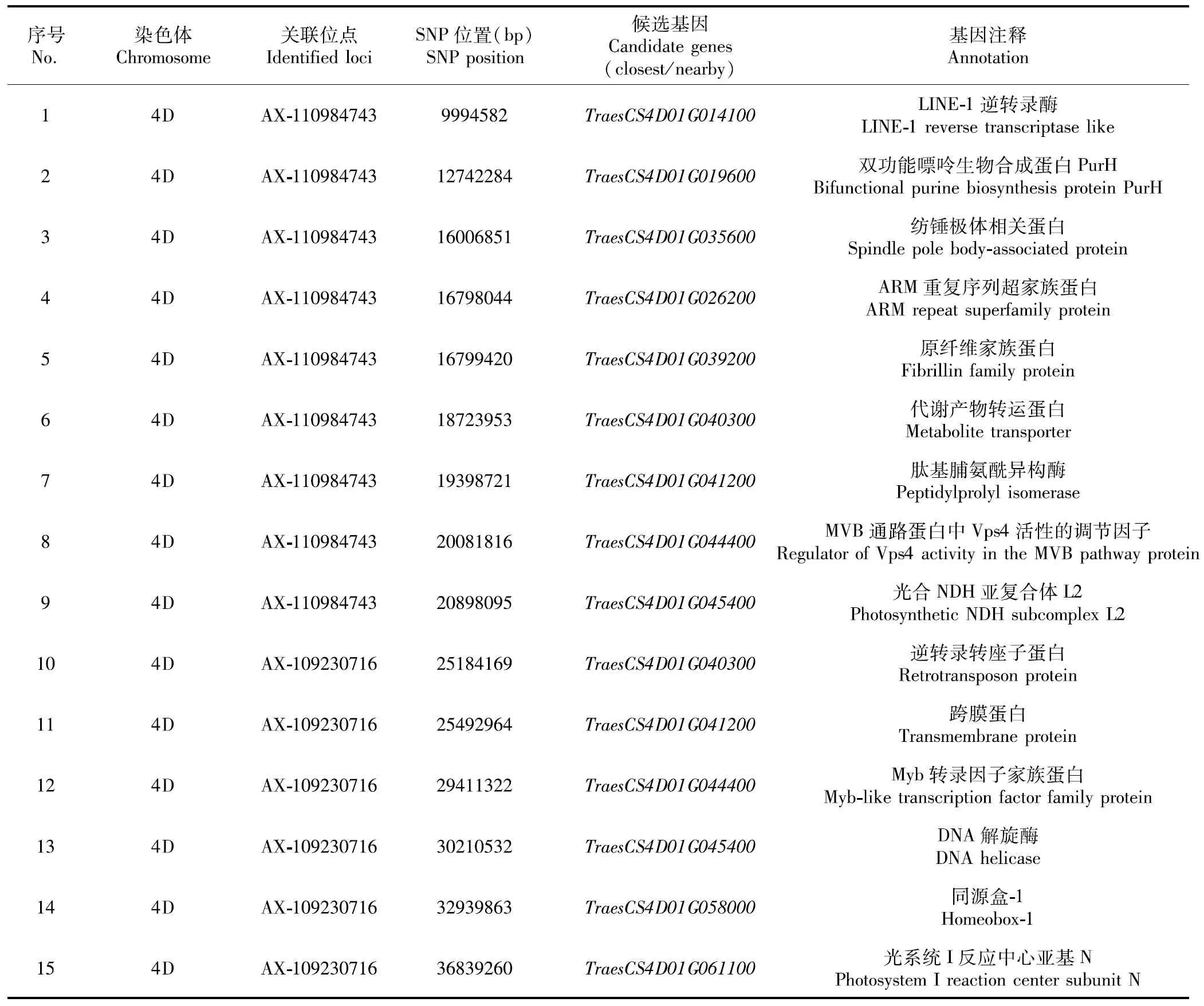
表4 本研究预测的小麦穗下节长度候选基因Table 4 The candidate genes and their information for wheat uppermost-internode length identified in the study
由表4可知,这些候选基因功能多样,其中大部分候选基因编码一些功能蛋白,例如,TraesCS4D01G019600可能编码一种双功能嘌呤生物合成蛋白,TraesCS4D01G035600可能编码一种纺锤极体相关蛋白,TraesCS4D01G026200可能编码一种ARM重复序列超家族蛋白,TraesCS4D01G040300可能编码一种逆转录转座子蛋白,TraesCS4D01G044400可能编码一种Myb类转录因子家族蛋白。部分候选基因编码一些功能酶,例如,TraesCS4D01G014100可能编码一种逆转录酶,TraesCS4D01G045400可能编码一种DNA解旋酶。也有部分候选基因编码一些与光合作用相关的复合体或亚基,例如,TraesCS4D01G045400可能编码光合NDH亚复合体L2,TraesCS4D01G061100可能编码光系统I反应中心亚基N。个别候选基因编码生物活性相关的调节因子,TraesCS4D01G044400可能编码一种MVB通路蛋白中Vps4活性的调节因子。
3 讨 论
与其它农艺性状相比,目前有关小麦穗下节的研究极少,已有研究主要集中于穗下节的解剖及与产量的关系等方面[1-2, 12]。前人研究与育种实践表明,穗下节长度对小麦株高、株型和产量等均有重要影响,一般来说,在株高相同的情况下,穗下节在合理范围内相对较长,其株型和产量表现更好,因此在育种实践中已将穗下节长度作为小麦新品种筛选的主要指标之一。本研究材料“普冰3228”是穗下节长度表现突出的小麦新种质,为探讨该种质穗下节长度的遗传机制并为其后期利用提供有效基础,研究利用“普冰3228” 与 “京4839” 构建RIL群体,田间性状调查发现,该群体穗下节长度表现突出,其遗传差异也较丰富,在2020年平顶山环境下其穗下节长度分布于34.00~117.50 cm,平均达77.63 cm,Yu等[4]对Opata85×W7984和SHW-L1×Chuanmai32构建的2个重组自交系群体穗下节长度也进行了测定,发现其穗下节长度分布于27.23~50.00和24.14~59.06 cm,穗下节平均长度为40.80和42.67 cm。与Yu等[4]利用的2个遗传群体相比,本研究定位群体穗下节长度表现更突出,这可能与本研究利用新种质材料“普冰3228”作为亲本材料有关,该材料由普通小麦与冰草通过远缘杂交获得,很可能含有控制穗下节长度的外缘优异基因。
前人有关小麦穗下节长度QTL定位研究报道较少。为探讨“普冰3228”穗下节长度的遗传机制,本研究基于构建了“普冰3228” × “京4839” 重组自交系群体,利用55K SNP芯片构建高密度分子遗传连锁图谱,结合多环境下穗下节长度表型数据对小麦穗下节长度进行QTL定位,结果在2B、4D、5B、6D染色体上检测到控制穗下节长度QTL,Yu等[4]在2B、4D、5B染色体上也发现控制穗下节长度的QTL,表明2B、4D、5B染色体可能是小麦穗下节QTL存在的主要区域。本研究在4B染色体上发现控制穗下节的QTL,而Yu等[4]在该染色体并未发现目标性状QTL,表明该QTL可能是控制穗下节长度新的QTL,进一步分析发现,该QTL的LOD值达7.88,表型解释变异率为10.32%,说明其也是控制穗下节长度的主效QTL,应引起后期研究的重视。从现有文献报道来看,目前只有本研究和Yu等[4]开展了有关穗下节长度的QTL分析。其他学者也针对小麦穗下节性状开展了相关QTL研究,但该研究是针对穗下节茎秆直径[3]和维管束组成[5]等其它性状开展。
穗下节长度候选基因分析对穗下节遗传机制深入解析及其基因资源挖掘十分重要,目前有关此方面的研究未见报道。本研究在QTL定位的基础上,通过进一步分析共获得与小麦穗下节长度相关的候选基因15个,从这些候选基因的功能来看,其主要为一些相关蛋白和功能酶,它们通过调节或影响植物代谢活动进而对小麦穗下节长度产生间接影响[13-15]。例如,TraesCS4D01G019600可能编码双功能嘌呤生物合成蛋白,该蛋白是许多具有调节代谢功能的同工酶的重要组成因子[16]。TraesCS4D01G039200可能编码原纤维家族蛋白,已有学者发现这类基因在植物茎秆中表达[17-18]。TraesCS4D01G040300可能编码代谢产物转运蛋白,其可能通过调节代谢产物在穗下节区域的分配影响穗下节的长度。TraesCS4D01G045400和TraesCS4D01G061100可能编码与植物光合作用相关的因子[19]。TraesCS4D01G044400可能编码Myb转录因子家族蛋白,该蛋白具有调节次生代谢的功能[20]。当然,本研究只是对穗下节长度候选基因的初步分析,这些基因仍需更为深入的研究。本研究结果将为小麦穗下节长度的遗传改良及“普冰3228”的有效利用提供一定科学依据。
4 结 论
本研究以“普冰3228”ד京4839”构建的重组自交系群体为材料,利用55K SNP芯片对穗下节长度进行QTL定位及候选基因分析。研究共检测到与小麦穗下节相关QTL 6个,这些QTL主要分布于2B、4B、4D、5B和6D染色体上,其中QUIL-4B.e1、QUIL-4D.e1为主效QTL,QUIL-4D为多环境下稳定QTL。预测分析获得与穗下节长度相关的候选基因15个。本研究不仅有助于小麦穗下节长度遗传机制解析,也将为其遗传改良提供新材料和新候选基因。
致 谢:感谢中国农业科学院作物科学研究所李立会研究员为本研究提供材料支持,感谢中国农业科学院作物科学研究所李立会研究员和张锦鹏研究员对本研究的技术指导。
