玉米叶片硝酸盐积累性状的全基因组关联分析
2024-04-08邬奇孙扬名桑俊伟周玲赵涵
邬奇 孙扬名 桑俊伟 周玲 赵涵

摘要:为鉴定筛选调控玉米氮素吸收代谢相关位点和基因,以遗传来源广泛的350份玉米自交系为材料,利用玉米55K SNP芯片,对玉米叶片硝酸盐积累性状进行基于Q+K混合线性模型的全基因组关联分析。结果表明,350份玉米自交系构成的自然群体具有广泛的遗传多样性,3次重复测量的叶片硝酸盐含量均表现出良好的正态分布特性。叶片硝酸盐的全基因组关联分析显示,共有27个稳定的SNP位点与玉米叶片硝酸盐积累显著相关,这27个位点共分布在玉米6条染色体上,其中16个集中在4号染色体上。对9个极显著关联[-lgP≥5.5]位点基因进行氮响应相对表达分析,有8个基因表现出显著氮响应特性,其中3个基因响应达到极显著水平,分别较对照提高3.54、2.74、2.02倍,对应已注释的基因分别为NAC79、GA20ox7和PREP2,表明它们可能在氮素吸收代谢的调控中发挥作用。相关研究结果可作为玉米硝酸盐吸收代谢研究的重要候选基因进行功能验证,对开发氮高效分子标记及育种利用具有重要意义。
关键词:玉米;硝酸盐;全基因组关联分析;SNP;候选基因
中图分类号:S513.01 文献标志码:A
文章编号:1002-1302(2024)03-0047-06
氮素作为植物生长的必需营养元素,对作物产量和品质性状形成起重要作用。目前,全球农业生产的氮肥年使用量已经超过1.2亿t,但氮肥利用率很低,氮素损失率达70%[1-2]。氮肥的大量施用,在支撑作物增产的同时,也引起了土壤酸化、水体富营养化、温室效应加剧等一系列环境问题[3-4]。因此,筛选和培育氮高效型作物种质是协同解决作物产量、资源效率和环境安全的关键。
玉米是全球栽培面积最大的粮食作物[5],其产量对氮素的依赖非常显著。开发和培育氮高效玉米种质资源和新品种,对减少氮肥消耗及降低氮肥引发的环境问题具有重要意义。为解析玉米氮素吸收和代谢相关性状的遗传机制,前人做了大量的研究。ZmDof1是最早被发现与玉米氮素吸收调控相关的转录因子。在低氮环境下,ZmDof1在水稻中的表达可以提高水稻对碳氮的吸收和同化[6]。Ge等利用泛转录组学(pan-transcriptome)手段,对503个不同玉米自交系氮響应转录组数据进行分析,构建了氮响应基因表达调控网络,筛选出处于关键节点的ZmNLP5转录因子,该调控因子在玉米氮饥饿状态下的氮吸收和同化过程中起重要作用[7]。MADS-box转录因子在水稻和拟南芥中被证明与氮素的吸收和同化密切相关。在玉米中,第一个被发现的MADS-box转录因子Zmm28被证明与氮素利用密切相关,过表达Zmm28可显著提高玉米的产量[8]。Liu等鉴定了1个局部氮响应的MADS-box转录因子ZmTMM1,该转录因子主要在根中表达,调控不同氮素水平下的根系构型并影响根对硝酸盐的吸收[9]。Du等通过基因共表达网络分析玉米和高粱氮响应保守模块,鉴定了玉米和高粱进化保守的氮调控转录因子ZmNIGT1,该转录因子是一个负调控因子,在优化氮素营养代谢中发挥重要作用[10]。上述氮素吸收代谢调控因子的发掘一般基于组学和反向遗传研究,利用正向遗传学手段挖掘更多玉米中尚未被发现的调控因子亟待进一步探索。
全基因组关联分析(GWAS)具有效率高、通量大、适用性强、遗传多样性覆盖度高等优点[11]。目前,基于GWAS分析已鉴定到多个水稻氮高效基因位点,如OsTCP19、OsNAC42、OsNPF61、OsNLP4、OsNiR等[12]。上述基因位点的鉴定为氮高效利用分子调控网络的构建奠定了基础,其中多个基因已在现代栽培稻氮高效遗传改良育种中获得成功应用。到目前为止,通过GWAS在玉米中开展氮高效基因或调控因子的鉴定和挖掘鲜有报道。
本研究使用来自温带、热带以及亚热带且具有丰富遗传多样性的350 份玉米自交系组成的关联群体为试验材料,对其叶片中的硝酸盐积累进行表型评价并进行GWAS分析,旨在调查玉米硝酸盐积累情况及鉴定可能影响硝酸盐含量的位点及候选基因,研究结果可为玉米氮素吸收代谢、相关调控网络的构建及育种应用提供理论基础和科学依据。
1 材料与方法
1.1 试验材料
试验材料为350份来源广泛的优良玉米自交系组成的自然群体。群体材料遗传背景丰富,形态差异显著,包含我国温带5个杂种优势群的代表种质及其衍生系、热带和亚热带种质及来源国外的种质材料。
1.2 试验设计
350份自交系于2022年种植在江苏省农业科学院人工智能温室。为控制统一的氮素浓度,采用水培法培育玉米苗,水培溶液为含2 mol/L KNO3的改良Hoagland营养液。水培容器为50 cm×80 cm×20 cm中转箱,采用泡沫板悬浮法培养,每盆培养不同玉米自交系幼苗40株。每隔10 d更换1次营养液以保证相对充足的营养环境。试验共重复3次,分别于2022年4月、5月和9月开展。待玉米长到4叶1心时收获植物地上部所有叶片,烘干研磨至粉末用于硝酸盐含量的测定。
1.3 玉米叶片硝酸盐含量的测定
试验于玉米生长至4叶1心时取样,直接剪取植株全部叶片,快速放入烘箱于105 ℃杀青30 min后转为80 ℃烘干至恒质量,使用粉碎仪将样品磨成粉末,玉米植株硝酸盐含量的测定参考王学奎等的方法[13],采用水杨酸-硫酸法测定,具体方法如下:准确称取0.1 g烘干的叶片样品于10 mL离心管中,加入5 mL超纯水充分混匀,水浴锅100 ℃水浴30 min,期间每隔10 min充分摇晃均匀,待冷却至室温后离心,离心机于室温15 000 r/min离心10 min,离心结束后吸取100 μL上清于新的10 mL离心管中,加入质量分数为5%的水杨酸-浓硫酸溶液 400 μL,混匀静置反应20 min,再向离心管中加入9.5 mL质量分数为8%的NaOH溶液,反应冷却至室温后,使用紫外-可见光分光光度计在410 nm处测吸光度,根据硝酸盐标准曲线计算样品硝酸盐含量。
1.4 群体结构分析
利用Alexander 等开发的Admixture 1.3软件进行最佳亚群数的计算[14],对每个K值进行10次独立重复运算,当交叉检验误差(CV Error)最小时,此时对应的K值即为最佳亚群个数,基于此选用Structure 2.3.4 软件绘制群体结构图。
1.5 全基因组关联分析
全基因组关联分析选用TASSEL 5.2.28 软件,联合群体结构(Q矩阵)及亲缘关系(K矩阵)采用混合线性模型(MLM)对基因型数据及硝酸盐含量进行关联分析,P值的显著阈值选择为10-5,结合R程序进行曼哈顿图及QQ散点图的可视化。
1.6 候选基因功能注释
基于关联分析结果找到于硝酸盐含量显著相关的SNP位点,选用SNP eff工具在玉米V3 (以B73自交系为模板)参考基因组中(https://www.maizegdb.org/)根据显著SNP位点的物理位置找到其所在的基因或结构域,以LD(r2≤0.1)为范围筛选硝酸盐含量性状的候选基因,将玉米V3参考基因组作为候选基因预测的依据并进行候选基因的功能注释。
1.7 候选基因氮响应表达分析
参照已发表的RNA-seq数据,对定位到的显著关联位点进行表达分析和统计[7]。试验材料为B73自交系,统计数据为10 mmol/L KNO3处理0、30 min后的表达变化。
2 结果与分析
基于Admixture 1.3软件计算最佳亚群数量,当交叉检验误差(Cross-Validation Error)最小时,其对应的K 值即为最佳亚群数。由图1-A可见,当K值为14时,交叉检验误差最小。基于此,将350份玉米自交系划分为14个亚群(图1-B),分别为黄改群Ⅰ、黄改群Ⅱ、IDT群、改良IDT群、兰卡群Ⅰ(Mo17)、兰卡群Ⅱ(OH43)、糯玉米群、P群热带群、瑞德群Ⅰ、瑞德群Ⅱ、改良瑞德群Ⅰ、改良瑞德群Ⅱ及改良瑞德群Ⅲ(图1-C)。
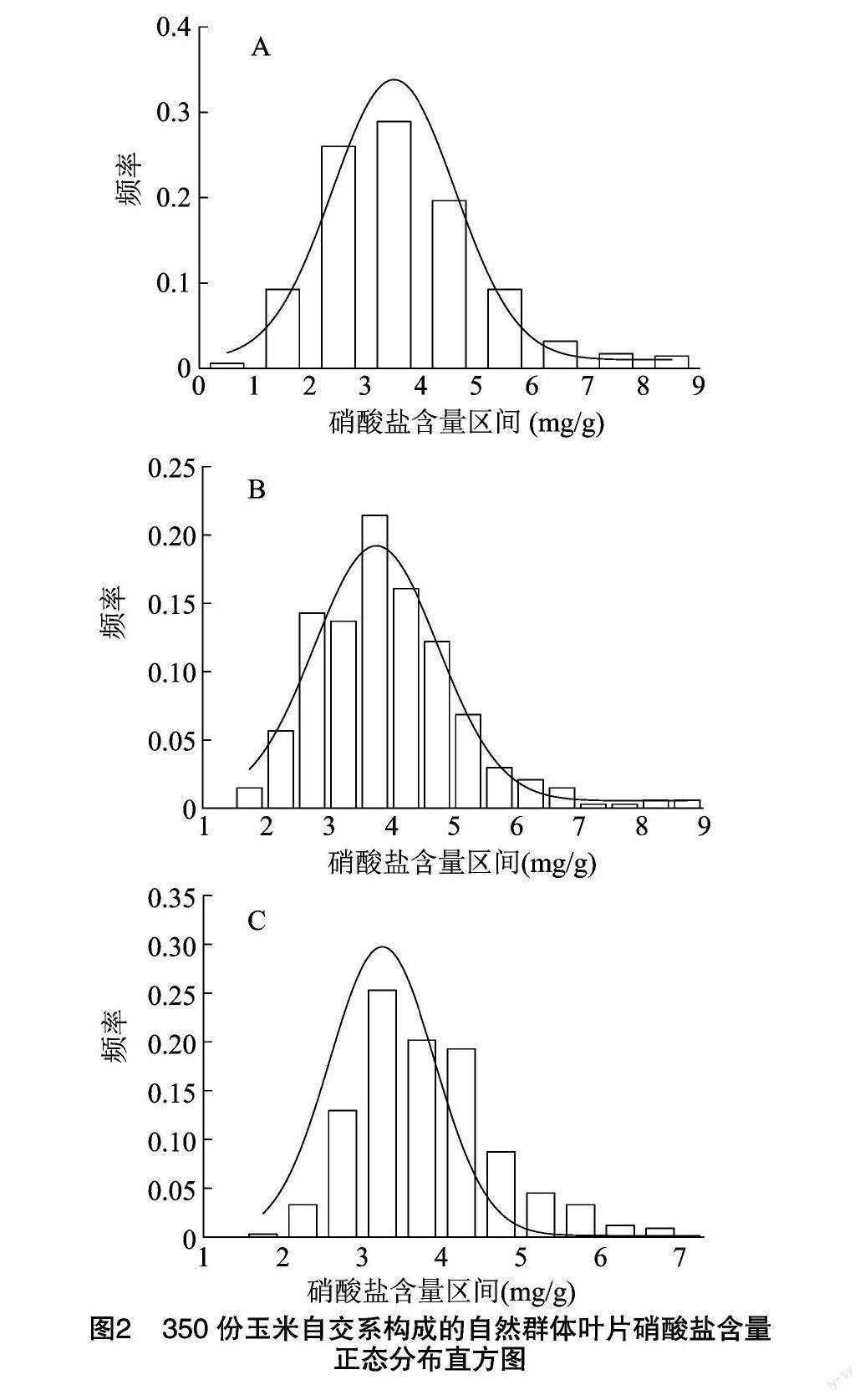
由图2可见,对350份玉米自交系开展了3次水培试验,对收集到的玉米叶片开展硝酸盐含量测定,正态分布分析结果显示,3组试验重复中玉米叶片苗期硝酸盐含量均呈现出中间高两端低的分布特点,表现出良好的正态分布特征,表明GWAS群里叶片硝酸盐积累符合数量性状的遗传规律,并存在丰富的遗传变异。
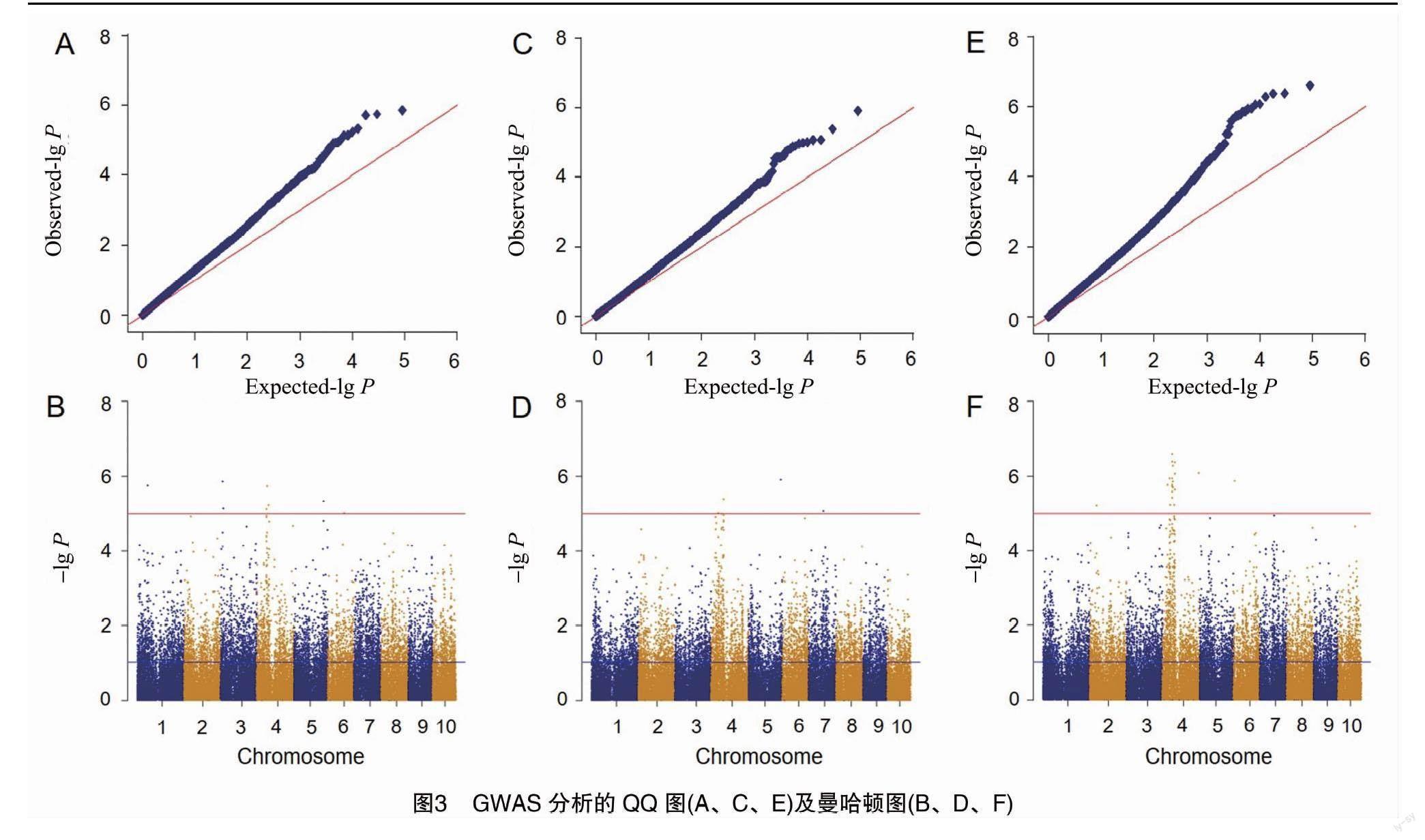
基于45 299个高质量SNP标记对3个不同环境350 份玉米自交系苗期硝酸盐含量性状进行全基因组关联分析。由图3-A、图3-C、图3-E的QQ图可见,每组重复样品点分布的实际值与理论值表现出较好的拟合特性,反映了各重复组样品点具有较好的正态分布特征。
以-lgP≥5为标准统计与硝酸盐含量性状显著关联位点,共鉴定到27个显著关联位点。由图3-B、图3-D、图3-F及表1可见,这27个显著关联位点分布在6条染色体上。其中在4号染色体上分布最多,共有16个显著关联位点。其他位点分布分别为:1号染色体1个,2号染色体2个,5号染色体3个,6号染色体3个,7号染色体2个。对-lgP≥6的极显著位点进行统计,结果发现4个与硝酸盐可能存在极显著关联的位点,对应的基因分别为GRMZM2G101271、GRMZM2G478034、GRMZM2G010097和GRMZM2G478034(表1)。
为进一步确定鉴定位点及其对应基因是否对氮素存在响应,对-lgP≥5.5的9个关联位点及对应基因进行相对表达分析。由图4可见,与对照(Con:1 mmol/L KNO3)相比,玉米幼苗在高氮(HN:10 mmol/L KNO3)处理15 min后,除GRMZM2G478034以外,其他8个基因均表现出不同程度的氮响应特性,且均达到显著响应水平,其中基因GRMZM2G033884、GRMZM2G0002704、GRMZM2G101271和GRMZM2G153722的氮响应水平最为显著,分别为对照的3.54、2.74、2.02、2.51倍。
3 讨论与结论
群体结构体现了一个群体中不同亚群的分化程度[15],在进化研究、筛选并展开性状与分子标记的关联分析中发挥重要作用。本研究基于交叉检验误差(CV Error)来确定最佳亚群数,将350份玉米自交系划分成14个亚群,各亚群之间基因交流广泛,重组变异充足。然而,群体结构会引起分布在不同染色体上的位点间的连锁不平衡增加,这会导致目的性状与真实情况并不相關的位点关联,从而使结果产生假阳性[16]。因此,在进行关联分析时,必须要考虑到群体结构对GWAS分析结果的影响,然后引入其他协变量或算法加以解决[17]。本研究同时考虑亲缘关系与群体结构对最终结果的影响,因而采用混合线性模型(MLM)将2个协变量(Q+K或PCA+K)导入统计计算模型中以此来控制假阳性结果。尽管采用双矩阵进行质控导致了统计方法过于严格、有相关性的位点因其不显著而被筛除,但保证了本研究鉴定的显著性关联位点的可靠性。
采用遗传力高、科学稳定、遗传变异丰富、简单经济的指标来衡量作物的氮效率是选育氮高效种质的关键。在全基因组关联中,选择合适的氮素吸收表型指标与自然群体基因型进行关联分析,对成功挖掘氮素吸收利用相关的功能基因或调控因子至关重要。Liu等通过调查分析低、中、高N条件下产量相关农艺性状,发现分蘖氮响应(tillering response to nitrogen,TRN)是衡量水稻N利用效率的理想指标,并通过GWAS成功定位一个与氮素利用显著相关的候选基因OsTCP19[18]。 本研究选择硝酸盐含量作为GWAS分析的表型指标,原因如下:一是玉米是旱地作物,主要利用的氮素形态是硝酸盐;二是硝酸盐通常被用来作为反映作物氮素积累差异的重要指标,在叶片的积累能够很好地反映植物本身的硝酸盐的吸收情况。尽管代谢同化等其他过程也会影响硝酸盐形态在植物体的积累,但不能完全消除不同植物对硝酸盐吸收的差异性。本研究的结果显示,350个玉米自交系叶片硝酸盐含量存在差异,表现出良好的正态分布特性,表明以硝酸盐作为GWAS分析的表型指标具有较好的科学性和代表性。
本研究通过3次试验共定位到了27个显著位点,在关联的相关基因组共发现了3个转录因子,分别是MYB49(GRMZM2G032655)、GNAT18 (GRMZM2G055141)、NAC79(GRMZM2G033884)。目前为止,关于MYB和GNAT家族转录因子在植物氮响应及调控中的研究尚未见报道。在水稻中,NAC家族转录因子OsNAC42是重要的氮高效转录因子,它与NPF6.1的信号级联在水稻氮利用及产量提高中发挥重要作用[19]。在玉米中,OsNAC7是调控玉米持绿的关键调控因子,在调节叶绿素降解和蛋白质周转中发挥重要作用,可能与玉米氮素代谢密切相关[20]。本研究中,NAC79的表达受氮素诱导,高氮诱导后其表达提高了3.54倍(图4),推测可能是玉米氮素吸收或代谢调节的重要转录因子,相关表型性状和机理仍需进一步研究。除此以外,本研究还发现一个重要的功能基因ZmGA20ox7(GRMZM2G0002704)可能与氮素吸收代谢有关。已有报道表明,玉米GA20ox5突变体在低氮条件下叶绿素含量显著高于野生型,对氮素的敏感性显著降低[21]。ZmGA20ox7作为同源基因,在氮诱导后其表达上升了2.74倍,表明其可能在调控氮响应及代谢过程中发挥重要功能。
综上所述,本研究中获得的显著关联 SNP 位点及基因在调控氮素吸收代谢过程中可能具有重要作用。然而,对鉴定的基因进行功能验证及育种利用需要进一步探索,相关深入研究将为通过分子辅助育种提升玉米的氮素利用效应有重要参考价值。
参考文献:
[1]Fowler D,Steadman C E,Stevenson D,et al. Effects of global change during the 21st century on the nitrogen cycle[J]. Atmospheric Chemistry and Physics,2015,15(24):13849-13893.
[2]Kanter D R,Bartolini F,Kugelberg S,et al. Nitrogen pollution policy beyond the farm[J]. Nature Food,2020,1(1):27-32.
[3]Zhang X,Davidson E A,Mauzerall D L,et al. Managing nitrogen for sustainable development[J]. Nature,2015,528(7580):51-59.
[4]申建波,白 洋,韦 中,等. 根际生命共同体:协调资源、环境和粮食安全的学术思路与交叉创新[J]. 土壤学报,2021,58(4):805-813.
[5]Hochholdinger F,Yu P,Marcon C.Genetic control of root system development in maize[J]. Trends in Plant Science,2018,23(1):79-88.
[6]Kurai T,Wakayama M,Abiko T,et al. Introduction of the ZmDof1 gene into rice enhances carbon and nitrogen assimilation under low-nitrogen conditions[J]. Plant Biotechnology Journal,2011,9(8):826-837.
[7]Ge M,Wang Y C,Liu Y H,et al. The NIN-like protein 5 (ZmNLP5) transcription factor is involved in modulating the nitrogen response in maize[J]. The Plant Journal,2020,102(2):353-368.
[8]Wu J R,Lawit S J,Weers B,et al. Overexpression of zmm28 increases maize grain yield in the field[J]. Proceedings of the National Academy of Sciences of the United States of America,2019,116(47):23850-23858.
[9]Liu Y,Jia Z T,Li X L,et al. Involvement of a truncated MADS-box transcription factor ZmTMM1 in root nitrate foraging[J]. Journal of Experimental Botany,2020,71(15):4547-4561.
[10]Du H Y,Ning L H,He B,et al. Cross-species root transcriptional network analysis highlights conserved modules in response to nitrate between maize and Sorghum[J]. International Journal of Molecular Sciences,2020,21(4):1445.
[11]楊 彬,周嘉润,沈玉婷,等. 水稻种子活力性状全基因组关联分析研究进展[J]. 江苏农业科学,2023,51(3):16-21.
[12]Hu B,Wang W,Chen J J,et al. Genetic improvement toward nitrogen-use efficiency in rice:lessons and perspectives[J]. Molecular Plant,2023,16(1):64-74.
[13]王学奎,黄见良. 植物生理生化实验原理与技术[M]. 3版.北京:高等教育出版社,2015:55-57.
[14]Alexander D H,Novembre J,Lange K. Fast model-based estimation of ancestry in unrelated individuals[J]. Genome Research,2009,19(9):1655-1664.
[15]陈 斐,魏臻武,李伟民,等. 基于SSR标记的苜蓿种质资源遗传多样性与群体结构分析[J]. 草地学报,2013,21(4):759-768.
[16]吴永升. 玉米谷氨酰胺合成酶基因Gln1-3、Gln1-4氮利用效率关联性分析[D]. 北京:中国农业科学院,2009:17-18.
[17]Song M S,Hao W,Storey J D.Testing for genetic associations in arbitrarily structured populations[J]. Nature Genetics,2015,47(5):550-554.
[18]Liu Y Q,Wang H R,Jiang Z M,et al. Genomic basis of geographical adaptation to soil nitrogen in rice[J]. Nature,2021,590(7847):600-605.
[19]Tang W J,Ye J,Yao X M,et al. Genome-wide associated study identifies NAC42-activated nitrate transporter conferring high nitrogen use efficiency in rice[J]. Nature Communications,2019,10:5279.
[20]Zhang J,Fengler K A,van Hemert J L,et al. Identification and characterization of a novel stay-green QTL that increases yield in maize[J]. Plant Biotechnology Journal,2019,17(12):2272-2285.
[21]張肖逢. 玉米ga20ox5矮化突变体和转ZmGRF1基因玉米的鉴定及氮素利用率研究[D]. 北京:中国农业科学院,2021:62-68.
