AM FRel:一种中文电子病历实体关系联合抽取方法
2024-03-19余肖生李琳宇周佳伦马洪彬
余肖生,李琳宇,周佳伦,马洪彬,陈 鹏
(1.三峡大学湖北省水电工程智能视觉监测重点实验室,湖北 宜昌 443002;2.三峡大学计算机与信息学院,湖北 宜昌 443000)
0 引言
医疗文本的实体关系抽取是医学自然语言处理研究中非常重要的一环[1]。早期的关系抽取是基于流水线(pipeline)的方法,首先从文本中抽取出所有的实体,然后判断所有可能的实体对之间的关系。其中具有代表性的模型有基于卷积神经网络(convolutional neural networks,CNN)的关系抽取模型[2]、将注意力机制引入了CNN模型中的Attention CNNs模型[3]等。Pipeline方法易于实现且实体模型和关系模型可以灵活使用,但在抽取实体与关系时,忽略了2个任务之间的内在联系,容易造成误差传播。而联合抽取可以加强实体模型和关系模型之间的联系,一定程度上缓解误差传播的问题[4]。早期联合抽取方法也是基于特征工程的[5-6],严重依赖手工制作的特征。随着深度学习技术的不断发展,双向长短期记忆网络(Bi-directional long short term memory network,BiLSTM)、循环神经网络(recurrent neural network,RNN)等模型层出不穷,它们在关系抽取领域有广泛的应用。例如,Zheng等[7]通过运用神经网络设计了一种新型的标注机制,用于对序列进行标注;Zeng等[2]使用CNN来提取句子和词汇信息,将2个特征拼接进行关系抽取。
目前现有工作在通用领域的文本中有着不错的效果,但在医疗领域的文本中,实体往往是专业名词,这些实体的密度大且实体之间的关系比通用领域的文本更为复杂,使用简单的神经网络方法无法充分地捕捉文本特征[1]。针对这些问题,本文中提出了基于对抗学习与多特征融合的中文电子病历实体关系联合抽取模型AMFRel。模型通过信息融合模块来丰富文本结构特征,并且还加入对抗训练增加扰动,提高了模型抽取三元组的性能。该模型在2个医疗文本关系抽取数据集上进行了实验,与现有方法相比,其在F1值等方面有明显提升。
1 相关工作
1.1 关系抽取
医疗文本的实体关系抽取最初是基于模板匹配与字典驱动的方法[8],这种方法主要是依靠医学专家人工制定相关的规则,虽然具有一定的效果,但采用这种方法非常耗时且制定的相关规则可移植性差。
随后是基于机器学习的方法,包括支持向量机、最大熵等,利用各种特征向量进行关系分类。如Zhai等[9]将句子结构信息与支持向量机相结合进行电子病历实体关系提取。该方法和基于模板匹配与字典驱动的方法相比有更好的移植性,但过度依赖特征选择。
近年来,深度学习技术逐渐进步,各种神经网络也得到了广泛应用。该技术在医疗文本实体关系抽取领域也有着不错的发展,能更好地从医疗文本中抽取出所需信息。Kim[10]使用CNN提取文本的局部特征,最后连接Softmax层,得到各关系类别的概率。Li等[11]使用BiLSTM和CNN来提取文本特征,并整合最短路径用于医疗实体关系抽取。Zhang等[12]提出了一种方法,通过融合BiLSTM和多跳自注意力机制,增强模型对文本特征的捕捉能力。这种融合策略使模型能够更准确地获取文本的向量表示,进而提升实体关系抽取的性能。2018年,随着预训练模型BERT(bidirectional encoder representations from transformers,BERT)[13]的出现,自然语言处理的子任务,如命名实体识别、关系抽取等都呈现出了更好的效果。Wei等[14]使用BERT模型来获得文本的向量表示,连接Softmax在医疗文本中进行关系抽取,产生了良好的效果。李天昊等[15]融合ERNIE模型和注意力机制进行中文文本的关系抽取。
上述基于深度学习的关系抽取方法在处理简单文本的关系抽取方面表现良好,但在识别包含重叠关系的句子时存在一些问题。为解决关系重叠问题,Zeng等[16]提出了一个序列到序列的框架,并且采用复制机制直接生成实体关系三元组,该方法可以缓解实体间关系重叠的问题,但没有考虑到实体间具有多种关系的重叠情况。Wei等[17]使用BERT模型进行句子编码,提出了将主语实体映射为宾语实体的二进制标注框架CASREL。该模型一定程度上解决了关系重叠问题,但在宾语和相关关系抽取时,仅将主语信息和文本特征融合,忽略了实体和关系之间的联系,导致一些结构特征丢失。此外,该模型使用指针网络标注实体的首尾,在标注较长实体时该网络对边界的识别比较敏感,容易出现实体识别不稳定的问题[18]。
1.2 对抗学习
对抗学习一般都应用于计算机视觉领域[19]。随着自然语言处理的发展,2016年,Papernot等[20]开始研究在文本中应用对抗样本的问题,他们采用快速梯度标志法(fast gradient sign method,FGSM)来寻找对抗样本,并在多个场景下对梯度生成对抗样本的可行性进行了研究。Samanta等[21]使用插入、删除、替换3个修改策略生成对抗样本,应用于分类结果。Chen等[22]将对抗训练应用到关系抽取模型中,在多个数据集上都取得了不错的效果。
基于上述研究,本文中提出了AMFRel模型,其中构建了信息融合模块来减轻结构特征有限的问题,并且在识别医疗实体时,加入对抗训练来缓解医疗实体长度过长造成的识别不稳定问题,从而提高了模型在医疗文本中抽取三元组的性能。
2 基于对抗学习与多特征融合的中文电子病历实体关系联合抽取模型
为了解决中文医疗文本实体密度大、关系复杂所导致的三元组抽取效果不佳的问题,提出了基于对抗学习与多特征融合的中文电子病历实体关系联合抽取模型(AMFRel)。该模型利用ERNIE-Health模型的医疗实体掩码策略和对抗学习来提高模型辨别实体边界的能力,并通过融合中文电子病历文本特征、主语特征和关系特征3个提示信息,抽取与关系特征相关联的宾语,进而构建医疗三元组。本文模型由以下几部分组成:嵌入模块、主语抽取模块、信息融合模块、关系及相应宾语抽取模块,如图1所示。首先,通过预训练模型对医疗文本进行编码,以获取文本的特征向量,同时利用文本的词性信息来辅助主语抽取任务。然后将主语特征、文本特征和关系特征送入信息融合模块进行特征融合,以预测特定关系下的宾语,最后得到医疗文本的三元组表示。

图1 AMFRel模型
2.1 嵌入模块
2.1.1 文本特征
2019年,受Bertmask策略的启发,Sun等[23]提出了一个新的语言模型ERNIE(enhanced representation through knowledge integration),旨在改进BERT在中文自然语言处理领域表现不佳的问题。与BERT的token屏蔽策略不同,该模型提出了Knowledge Masking策略,它除了token级别的屏蔽策略外,还拥有短语级别和实体级别的屏蔽策略,能够更加全面地考虑到句子中的先验信息,提高模型对字词和短语的理解。本文采用的预训练模型是ERNIE-Health[24],它以ERNIE模型为基础,运用医疗实体掩码策略来学习医疗文本中的专业术语。通过该策略,ERNIE-Health能够有效地掌握医疗领域中丰富的实体信息。此外,它还利用医疗问答数据获取实体间的内在联系,提升了对医疗文本的理解和建模能力。该模型的屏蔽策略如图2所示,例如句子“糖尿病肾病造成下肢浮肿”,除了随机选择的token级别的屏蔽策略外,还有实体级别的“下肢浮肿”和短语级别的“造成”。

图2 ERNIE-Health的屏蔽策略
具体过程如图1嵌入模块所示,将文本句子S={s1,s2,s3,…,sN}和实体间关系G={g1,g2,g3,…,gM}输入到ERNIE-Health模型,其中,si代表文本句子的第i个字符,gi代表实体间关系的第i个字符,N和M分别代表文本句子和实体间关系的长度,最后得到输出序列和
2.1.2 词性特征
词性是一种常见的语言学术语,通过对词的特征进行分类,可以获取更深层次的语义特征。观察发现,在医疗三元组中,主语、宾语及两者之间的关系通常都是名词或动词,获取词性信息对于三元组的抽取有一定的帮助[25]。因此,本文在模型中引入了词性特征,首先使用jieba分词软件对医疗文本进行分词与词性标注,并将文本中出现的词性类别整合为一个字典。然后,构建与文本信息对应的词性类别编码序列C={c1,c2,c3,…,cN},其中ci表示第i个词对应的词性类别编码。最后,对离散的词性信息进行处理,将其送入预训练模型中,通过训练得到融合了文本特征和词性特征的编码序列A={a1,a2,a3,…,aN}。
2.1.3 对抗训练
医疗文本实体密度大,使用指针网络进行实体标注时,如果漏标,容易导致实体边界识别出现误差。所以模型中使用对抗学习的方法,利用噪声数据进行对抗训练,提高模型辨别实体边界的能力。对抗训练的范式形式是通过内部损失最大化来寻找对抗扰动,当扰动固定时,外部进行网络优化得到内部对抗样本的损失函数最小化[26]。计算方法如式(1)所示。
式中:θ代表模型参数;x代表输入的文本;y代表输入文本对应的标签;Δx是指对原始文本的对抗扰动;s是指扰动的空间。首先,在原始文本中加入扰动,使得现有模型尽可能多地出错,也就是使Loss(x+Δx,y;θ)的值尽可能大,以获得最适宜的扰动。然后,利用外部的minθE(x,y)使扰动固定后的损失最小化,进一步优化模型的参数,提高模型辨别实体边界的能力,进而增强关系三元组的抽取性能。
2.2 主语抽取模块
主语抽取就是抽取文本中的主语信息。医疗文本中存在关系重叠的问题,本文采用了指针网络标注策略,利用首尾指针来标注文本中主语的起始位置。具体过程:首先将嵌入模块获得的编码序列A={a1,a2,a3,…,aN}输入到全连接层进行特征处理,然后将其送入用于二分类任务的sigmoid函数中,得到token的概率值输出。如果概率值比设置的阈值大,则将其位置的token标记为1,否则标记为0。计算方法如式(2)、式(3)所示。
完成阈值判断和0/1标注后,可以得到主语的头尾token,然后采用“邻近原则”,先找出主语的头位置,也就是概率值大于设置阈值的位置,随后寻找头token之后最近的概率值大于设置阈值的位置,最终得到主语。
2.3 信息融合模块
医疗实体间关系复杂,如果仅仅使用嵌入模块得到的编码向量进行后续的关系与宾语的抽取,则获取的结构特征非常有限。针对该问题,在AMFRel模型中构建了信息融合模块。首先,将融合词性特征的文本编码向量与关系向量进行拼接,将其输入到BiLSTM中获取更深层次的语义信息。然后,将主语编码与深层语义信息进行融合得到多结构特征的编码向量。最后,将其送入多头自注意力机制[27],该机制通过引入多个注意力头,使得模型能够学习到不同的注意力模式,从而更好地捕捉输入序列中的关键信息。这一信息融合模块通过对多种结构特征进行融合,提高了医疗三元组的抽取性能。
将融合词性特征的文本编码向量A与关系特征Hg进行拼接,在拼接前对关系特征Hg进行padding操作。这个过程是为了使关系词与医疗文本之间产生交互,通过这种方式可以使模型区分出医疗文本中的中文字符与关系词的关联程度,从而利用不同的关系特征识别相应的宾语。接着将拼接后的编码向量H输入到BiLSTM中,该网络可以从2个方向对信息进行编码,学习到更丰富的上下文内容,从而获得更深层次的融合序列L={l1,l2,l3,…,lN}。计算公式如式(4)—式(8)所示。
丰富的结构特征能够提升后续的关系及相应宾语抽取性能。本文还将主语的编码信息ai和拼接关系特征词的文本向量li进行融合,得到多种结构特征向量。该融合方式采用条件批处理规范化层(conditional layer normalization,CLN),通过2个变换矩阵进行维度变换,并将变换结果融合到了α和β中。计算公式如式(9)—式(10)所示。
式中:O为特征融合后的向量;μ和σ2表示L的均值和方差;α和β是指缩放和平移的参数值;Wα和Wβ是变换矩阵。
医疗文本中医疗实体分布密集,且在进行后续操作时实体的重要程度不一。因此,在进行特征融合后,本文采用多头自注意力机制对融合特征进行处理,该机制能够捕捉句子中实体之间的关联关系,从而提高模型抽取三元组的准确率。
2.4 关系及相应宾语抽取模块
在该模块中,使用2.3节中得到的融合向量进行关系及相应宾语抽取。与抽取主语的方法相同,首先将得到的编码向量P={p1,p2,p3,…,pN}输入到一个全连接层进行特征处理,然后将输出结果输入到sigmoid函数中,以获取每个token的概率值输出。如果概率值比设定的阈值大,则标记该token为1,反之为0。计算方法如式(12)、式(13)所示。
2.5 损失函数
本文在抽取主语和宾语时使用的都是二分类标注的方法,因此在计算损失函数时采用二值交叉熵损失函数(binary cross entropy loss,BCELoss)[28]。计算方法如式(13)、式(14)所示。
3 实验
3.1 数据集
AMFRel模型在2个数据集上进行了实验,一个是CHIP2020中文医疗文本实体关系抽取数据集,由郑州大学自然语言处理实验室和北京大学计算语言学教育部重点实验室一起构建。该数据集包括了53个预定关系类别,常见疾病的训练语料有上百种,都来源于专业医生编写的教材[29]。部分关系三元组schemas如表1所示。

表1 CHIP2020数据集部分关系三元组schemas
第2个数据集来源于某市疾控中心,已对其进行脱敏处理。该数据集主要包含糖尿病的多种发病症状与相关治疗手段,其中包括1 130条医疗文本,5 774个三元组数据和13种实体间关系。这些实体间的关系涵盖了手术-并发症、药物-疾病、手术-疾病等。部分关系三元组schemas如表2所示。

表2 糖尿病数据集部分关系三元组schemas
本次实验将在这2个数据集上进行,数据集详情如表3所示。

表3 数据集信息
3.2 评价指标
本文评价指标采用准确率(Precision)、召回率(Recall)和F1值。计算公式如式(15)—式(17)所示。
式中:TP为正样本判为正的个数;FP为正样本判为负的个数;FN为负样本判为正的个数。
3.3 实验环境与参数设置
为找到最适合模型的学习率,在CHIP2020关系抽取数据集上使用了不同的学习率进行训练和评估,Batch_size大小设置为4。具体来说,我们尝试了1×10-5、1×10-6、1×10-6、2×10-64种不同的学习率,结果如表4所示。

表4 CHIP2020数据集不同学习率下结果
根据表4的结果可以看出,在不同学习率下的F1值不同。通过观察可以发现在CHIP2020关系抽取数据集中,学习率为0.000 01时模型表现最佳,F1值为60.418%。因此,在模型训练过程中,设置学习率为0.000 01。
本次实验模型参数设置见表5,其中BERT编码器版本为chinese-bert-wwm-ext,ERNIE编码器版本为ERNIE-Health。

表5 模型参数设置
3.4 实验结果分析
为了验证AMFRel模型的效果,在CHIP2020关系抽取数据集和糖尿病数据集上进行了实验,并将其与基线模型进行对比。这些基线模型有PRGC[30]:使用token-pair进行分类,是一个基于潜在关系和全局指针网络的实体关系联合抽取框架;SPN[31]:将关系抽取看作一个seq2seq问题,属于联合抽取中的多任务学习方法;GRTE[32]:使用表格填充的方式,并考虑全局信息来进行三元组抽取;CASREL[17]:采用级联二进制方法,首先识别出主语,再识别出特定关系下的宾语,最后抽取出三元组。
表6展示了CHIP2020关系抽取数据集的基线、PRGC、SPN、GRTE、CASREL、CASRELERNIE模型在CHIP2020关系抽取数据集上的实验结果。
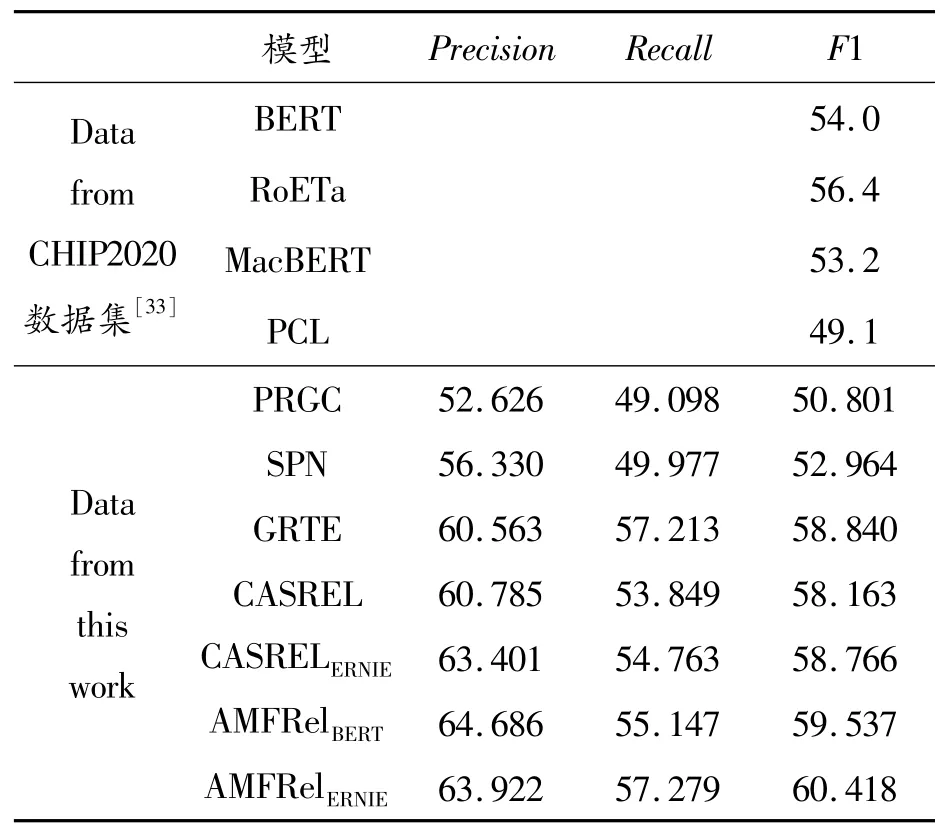
表6 CHIP2020数据集上各模型结果%
表7展示了糖尿病数据集在PRGC、SPN、GRTE、CASREL和CASRELERNIE模型上的结果。
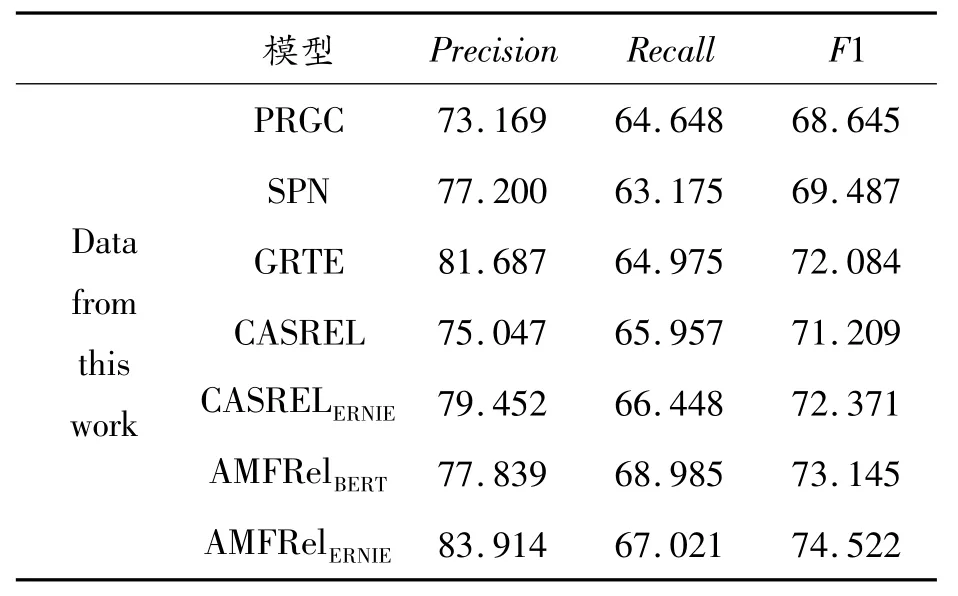
表7 糖尿病数据集上各模型结果%
表6和表7的结果表明,AMFRelBERT模型在CHIP2020关系抽取数据集中取得了59.537%的F1值,超越该数据集提供的基线。此外,将AMFRelBERT模型与基于潜在关系和全局指针网络的关系抽取模型PRGC、基于seq2seq的关系抽取模型SPN、基于表格填充的关系抽取模型GRTE和基于级联二进制结构的关系抽取模型CASREL进行了对比,在以Chinese-bert-wwm-ext作为预训练模型的前提下,AMFRelBERT模型在CHIP2020关系抽取数据集和糖尿病数据集上的准确率、召回率以及F1值这3个指标均有所提升。
具体而言,在CHIP2020关系抽取数据集上,与CASREL模型相比,AMFRelBERT在精确度、准确率和F1值上分别提高了3.901%、1.298%和1.374%;在糖尿病数据集上,AMFRelBERT相对于CASREL模型在精确度、准确率和F1值上分别提升了2.792%、3.028%和1.936%。这表明,在模型中融入多结构特征并引入对抗扰动对提升三元组的抽取性能是有效的。
此外,相较于AMFRelBERT,AMFRelERNIE在2个数据集上的F1值分别提升了0.881%和1.377%。这是因为ERNIE-Health采用了与传统BERT不同的屏蔽策略,并且该预训练模型使用医疗相关的数据进行训练,从而更适用于医疗文本的关系抽取。这也进一步证明了AMFRel模型在医疗文本关系抽取方面具有较好的表现。
3.5 消融实验
为了验证本文模型中各个模块的有效性,在CHIP2020关系抽取数据集和糖尿病数据集上进行了消融实验,实验结果如表8、表9所示。
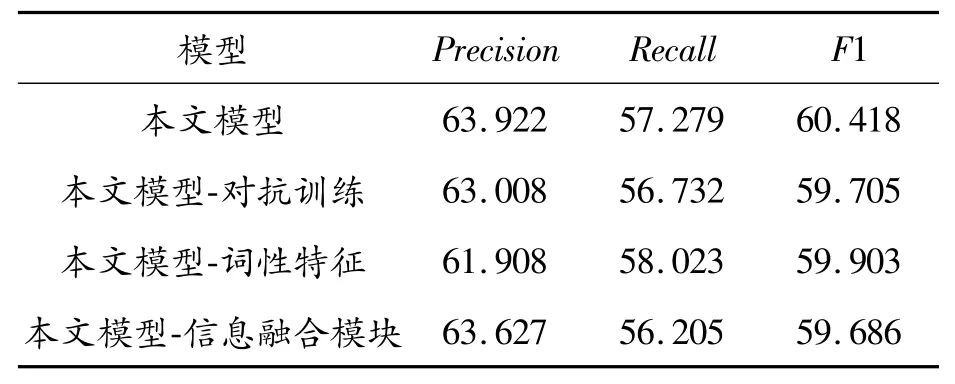
表8 CHIP2020数据集的消融实验结果%

表9 糖尿病数据集的消融实验结果%
从表中结果可以看出,在移除本文模型中的对抗训练后,CHIP2020关系抽取数据集和糖尿病数据集上的F1值分别下降了0.713%和1.084%。这表明移除对抗训练会降低模型抽取三元组的性能。另外,移除词性特征后,CHIP2020关系抽取数据集和糖尿病数据集上的F1值分别下降了0.515%和0.704%。这是由于医疗三元组中的实体及关系基本都是名词或动词,去掉词性信息会导致模型无法获取更深层次的语义特征,从而导致性能下降。最后,移除信息融合模块后,CHIP2020关系抽取数据集和糖尿病数据集上的F1值分别下降了0.732%和1.356%。这说明信息融合模块能够提升模型在医疗文本三元组抽取方面的性能,验证了该模块的有效性。
4 结论
提出了一种基于对抗学习与多特征融合的中文电子病历实体关系联合抽取模型AMFRel,旨在解决关系抽取时因医疗文本实体密度大且实体间关系复杂而导致医疗名词识别不准确的问题。该模型利用融合了词性特征的文本编码向量抽取主语,并通过信息融合模块获取更加丰富的文本结构特征,从而更有效地进行关系与相应宾语的抽取,最终得到了医疗文本的三元组信息。此外,模型还利用对抗训练缓解指针网络进行标注时带来的实体边界识别不稳定问题。实验结果表明,该模型在F1值上的表现优于其他模型,证明了AMFRel模型能够有效识别中文医疗文本中的复杂关系。在未来的研究中将尝试引入更加丰富的结构特征,并进行深入探索,进一步增强关系抽取效果。
