p53 活性四聚体全原子分子动力学分析*
2024-03-19周晗耿轶钊晏世伟3
周晗 耿轶钊 晏世伟3)
1) (北京师范大学物理学系,北京 100875)
2) (河北工业大学理学院,天津 300131)
3) (北京师范大学文理学院,珠海 519085)
p53 是一种肿瘤抑制蛋白,对阻碍癌症发展、维持遗传完整性起着至关重要的作用.在细胞核内,4 个p53 分子通过高度协同的方式、通过DNA 结合域与DNA 结合,形成稳定的四聚体活性结构,并转录激活或抑制其靶向基因.然而,大多数肿瘤细胞中存在大量p53 的突变,其中绝大部分突变发生在p53 的DNA 结合域,而p53 的DNA 结合域又是p53 形成四聚体活性结构、调控下游靶基因转录的重要区域.本文通过全原子分子动力学模拟,研究了野生型p53 四聚体内分子间的相互作用机制.结果表明,位于DNA 两侧的对称二聚体是一个稳定的二聚体,在与DNA 结合前后都能维持稳定的结构.位于DNA 同侧的两个单体依靠两个接触面提供的蛋白-蛋白相互作用和DNA 的骨架作用使四聚体活性结构保持稳定,这些相互作用为四聚体的形成机制提供了重要支撑.该工作厘清了p53 四聚体在动力学过程中的内部相互作用机制和关键残基,揭示了四聚化过程中各个相互作用界面的关键位点,对于理解p53 的抑癌机制、探索有效治癌策略、发展治癌药物具有重要意义.
1 引言
p53 肿瘤抑制基因是最常见的癌变相关基因之一[1].在应激环境下,p53 蛋白主要作为一种转录因子,促进下游靶基因的转录[2],从而引起细胞周期停滞、损伤修复或细胞凋亡[3,4].在人类癌症中超过50%的肿瘤细胞中发现有p53 突变[5,6],其中错义突变广泛发生在p53 的核心结合域,约占所有p53突变的95%[7].这种突变不但直接影响了p53 与DNA 的结合亲和力,也会不同程度地影响其正确折叠[8],这对靶基因的转录和细胞凋亡水平有很大的影响.因此,p53 是肿瘤治疗中的关键靶点.在针对p53 的靶向治疗中,有两种治疗策略: 一种是针对野生型p53,利用化合物占据MDM2 蛋白的活性位点,抑制MDM2 蛋白介导的p53 泛素化和核输出[9–11];另一种是针对发生突变的p53,利用化合物结合,诱导突变型p53 的重新激活,恢复野生型功能[12].无论哪一种治疗策略,都需要对p53 野生型结构的分子特征有清晰的认识.
稳定的p53-核心结构域(p53 DNA binding domain,p53-DBD)在细胞核内形成四聚体[13](见图1),与特异性靶DNA 结合并调节基因转录,诱导下游信号通路的改变.p53 核心结构域的四聚化是p53 作为一个转录因子的生物学功能核心.对于p53-DBD,各种结晶实验已经得到了p53 识别DNA 的分子机制[14].p53-DBD 与DNA 的结合主要包括两个结合槽,其中,环-片-螺旋基序(loopsheet-helix motif,LSH)与主槽对接,通过关键残基K120,C277,R280,R285,A276,S241和R273 与DNA 形成氢键.来自环L3 的残基R248 与小凹槽相互作用形成氢键.这些相互作用使得p53 可以与DNA 稳定的结合.核心结构域与DNA 的复合物晶体结构也解释了各种突变体如何使其失活,并在结晶实验和分子动力学模拟中得到了大量的证实[8,15,16].然而,游离的p53-DBD 主要以单体形式存在于溶液中[17],不易与DNA 的单一四分之一位点结合.显然,它需要一种更稳定的结合方式,才能够使p53 发挥其转录激活功能.
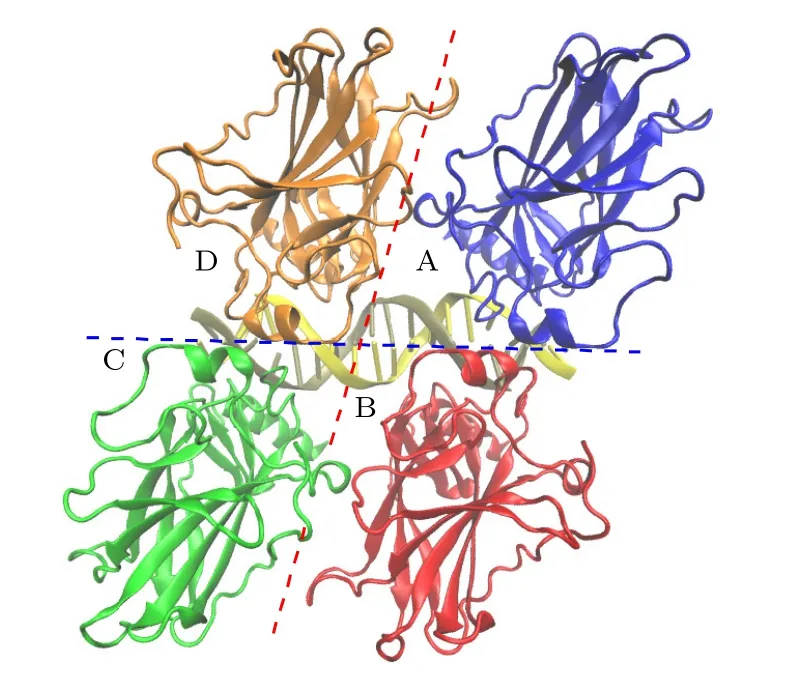
图1 p53 核心结合域四聚体结构: A 链(蓝色);B 链(红色);C 链(绿色);D 链(橙色).蓝线表示对称二聚体界面,红线表示二聚体-二聚体界面Fig.1.The p53 core binding domain tetramer structure:Chain A (blue);Chain B (red);Chain C (green);Chain D(orange).Blue lines represent symmetric dimer interfaces,while red lines represent dimer-dimer interfaces.
实验结果表明,p53-DBD 主要以一种高度协同的方式结合DNA 一致序列[18].两个p53-DBD相对于DNA 以对称的形式结合DNA 的半位点形成对称二聚体结构(symmetrical dimer structure).Nicholls 等[19]在研究p53 的体外生物发生实验中进一步证实,p53 二聚体是通过共翻译形成的,这种二聚体是位于DNA 两侧的二聚体.实验上对对称二聚体的结构已经有了比较清楚的描述: 对称二聚体(图1 中的A-B 和C-D)中的蛋白接触界面,被称为对称界面.它由每个p53 分子的H1 螺旋残基、Zn 配位体以及L2 和L3 环区域形成环-片-螺旋基序.疏水作用和由水介导的极性相互作用稳定了对称二聚体[20].然而有实验表明,对称二聚体仍可能在一定时间后从DNA 上脱离[21,22].
为了保持p53-DBD 与DNA 的稳定结合,两个对称二聚体(A-B 和C-D)通过蛋白-蛋白相互作用(protein-protein interaction)和碱基堆叠作用,在一个完整的DNA 响应元件上构成一个四聚体[13,20,23].随着两个对称二聚体之间接触点的增加,四聚体与DNA 的配合物更加稳定,半衰期与亲和度有很大提高[24].Weinberg 等[22]进行了野生型p53 四聚体和一个只能形成二聚体的L344A 突变体的对比实验.结果表明,四聚体对DNA 的亲和力是二聚体的6 倍.由此可见,核心结合域与DNA的四聚体配合物是p53 行使功能的关键,而二聚体-二聚体相互作用则在二聚体组装成四聚体的过程中起着十分重要的作用.
迄今为止,相继出现了一系列关于p53-DBD四聚体结构的实验结果[14,20,24–26],着重分析了四聚体分子中A-DNA,B-DNA 和A-B 之间的结构和相互作用.对于DNA 同侧A-D 和B-C 之间的蛋白-蛋白相互作用却鲜有分析.目前,对这些蛋白-蛋白相互作用的细节和结构的认识还相当匮乏.Cho 等[14]得到的不对称的p53-DBD 三体结构中,由于处于DNA 同侧的B,C 链结合的是非一致序列,导致B-C 之间的相互作用非常微弱.此外,在Kitayner 等[20]描述的p53 四聚体中,由于DNA不连续导致的错位,使得两个对称二聚体之间几乎没有蛋白-蛋白相互作用.然而,如前所述,在四聚体的结构中,二聚体-二聚体之间的相互作用是不可或缺的.
为了分析和研究p53 四聚体中位于DNA 同侧和对侧的两种蛋白-蛋白相互作用,利用全原子分子动力学模拟方法分别对这两种相互作用进行了系统分析.晶体结构选择Chen 等[23]提出的一种自组装的p53 四聚体结构(PDB ID: 3KMD).这一结构能够保证p53 四聚体可以结合到一个连续的同源DNA 位点,且DNA 不会弯曲.如图1所示,结构中每个p53-DBD 单体都与DNA 的一致序列结合.p53 四聚体内包含以下3 类接触面:1) p53-DNA 接触面;2) p53 对称二聚体接触面(A-B,C-D,蓝色线);3)二聚体-二聚体接触面(AD,B-C,红色线).由于DNA 两侧的p53 是对称分布,即A-B 和C-D 接触面一致,A-D 和B-C 接触面一致,所以本文将这个体系分为两部分进行模拟:1)由单体A,B 和DNA 组成的对称二聚体模型;2)由A,D 和DNA 组成的二聚体-二聚体界面模型.本文还分别计算了在没有DNA 结合时,两种模型的二聚作用,分析两种二聚模式是否都需要DNA才能稳定,以及DNA 在两种二聚体中的作用.
在本文模拟计算中,使用Amber14SB 和OL15力场分别模拟p53-DBD (残基92-291)和DNA.Zn 离子与4 个残基配位体(C176,C238,C242 和H179)结合.采用Lu 等[32]研究工作中的参数,对Zn 离子的四配位体进行描述.将整个结构在TIP3P 水分子的立方盒子中溶剂化,采用所有原子与水盒边缘之间最小距离为12 Å (1 Å=10–10m)的原则,防止周期性边界条件造成不同盒子内的分子有相互作用.通过添加0.15 mol/L 的Na+和Cl–来中和系统.使用Gromacs2018[33]来执行分子动力学模拟.静电相互作用使用离子网格Ewald(PME)方法来处理,实际空间截止距离为1.2 nm.范德瓦耳斯相互作用(van der Waals interaction)计算的截止距离为1.2 nm.使用 V-rescale 方法的外部温度浴使溶质和溶剂保持恒温,压力浴使用Parrinello-Rahman 方法维持恒压,温度和压力保持在310 K 和1 Pa.使用Verlet 缓冲区每10 步更新一次,截止距离为1.2 nm.首先采用共轭梯度法最小化5000步,以消除空间冲突.并先后在NVT和NPT 条件下进行0.1 ns 的模拟,以平衡系统.文中所有结构均采用VMD 软件[34]进行可视化处理.
本文首次揭示了四聚体中位于DNA 对侧和同侧的两种二聚形式在“有”和“无” DNA 结合时的构象差别和相互作用变化,得到各个p53 单体间协同结合的分子机制,并且证明这两种二聚形式的模拟,可以复现p53-DBD 四聚体中的两种蛋白-蛋白相互作用.系统地分析p53-DBD 四聚体的内禀属性,有助于开发针对p53 的抗癌性药物,例如恢复错误折叠的突变型p53 的野生型功能,促进其重新折叠成野生型p53 等.
2 对称二聚体内的相互作用
p53-DBD 的四聚体结构是通过两个对称二聚体的蛋白-蛋白相互作用形成的.因此,首先分析对称二聚体中分子之间的作用机制,分别对p53 对称二聚体在“有”和“无”DNA 结合的体系进行200 ns的分子动力学模拟,以研究p53 对称二聚体在动力学过程中的相互作用.如图2 所示,选择晶体结构(PDB ID: 3KMD)中的A,B 链和DNA 构成的对称二聚体作为模拟的初始结构(后文称为“有”DNA 结构).为了分析DNA 的作用,在图2 的结构中剪去DNA 分子,构成“无”DNA 对称二聚体的初始结构(图3).

图2 p53 对称二聚体与DNA 复合物的初始结构(a)由A,B 链和DNA 组成的p53 对称二聚体结构;(b) Zn 离子的配位结构;(c) DNA 轴垂直于图平面的对称二聚体Fig.2.Initial structure of the p53 symmetric dimer-DNA complex: (a) The p53 symmetric dimer structure composed of chains A,B and DNA;(b) coordination structure of Zn ions;(c) symmetric dimer with the DNA axis perpendicular to the plane of the figure.
图3 “无”DNA 的p53 对称二聚体初始结构Fig.3.Initial structure of the p53 symmetric dimer without DNA.
2.1 对称二聚体结构的稳定性
首先,计算了这两组模拟中p53 对称二聚体的主链碳原子相对于其初始时刻的均方根偏差(root mean square deviation,RMSD),以检验模拟的收敛性以及DNA 的存在对二聚体结构稳定的影响.如图4 所示,经过20 ns 的模拟后,两个p53 对称二聚体都达到稳定.因此,选择20—200 ns 的稳定轨迹进行分析.与有DNA 结合时的二聚体的RMSD (约(0.1791±0.0159) nm)相比,没有结合DNA 时二聚体的RMSD 平均值和涨落(约为(0.2935±0.0576)nm)较大.所以,没有DNA结合的情况下,p53 对称二聚体表现出更大的构象变化和更低的稳定性.这说明,与DNA 的结合可以限制二聚体的构象变化,增强二聚体稳定性.另外还通过计算A,B 两个蛋白的溶剂可及表面积,得到了A,B 两个蛋白单体之间的埋没表面积,即二聚体接触面积.在有DNA 的对称二聚体中,接触面积为425.76 Å2.在除去DNA 之后,接触面积为416.38 Å2,说明DNA 的结合不会对二聚接触界面的大小有太大影响,即对称二聚界面是一个比较稳定的界面.

图4 对称二聚体在“有”和“无”DNA 结合时的RMSD 演化Fig.4.Evolution of RMSD for the symmetric dimer with and without DNA.
为了更深入地理解DNA 结合对对称二聚体内相互作用的影响,对两个p53 单体之间的相互作用进行分析,如图5 所示.以供体原子X与受体原子Y之间的距离小于3.5 Å,并且X-氢-Y三个原子形成的角度小于30°,作为判定原子X-Y形成氢键的标准[35].当带负电荷的天冬氨酸(Asp)或谷氨酸(Glu)侧链COO–[36]与带正电荷的精氨酸(Arg)或赖氨酸(Lys)侧链或原子之间的最小距离小于4 Å时,判定两残基间形成盐桥.本文的结论是持续度大于20%的氢键和盐桥.A-B单体间的相互作用分析表明,两单体之间没有形成氢键,静电相互作用是由E180 和R181 间的盐桥构成.与DNA 结合时,来自E180(A)-R181(B)和R181(A)-E180(B)的两个盐桥,持续度分别为82.07%和50.53%.在除去DNA后,这两个盐桥的持续度有所变化,持续度分别为43.59%和70.51%.这些结果表明,在DNA 结合前后,位于A-B 单体H1 螺旋上的E180 和R181 间形成两个盐桥,一个稳定性较强,一个稳定性较弱,两个盐桥的叠加对二聚体中的静电相互作用有很大的贡献.
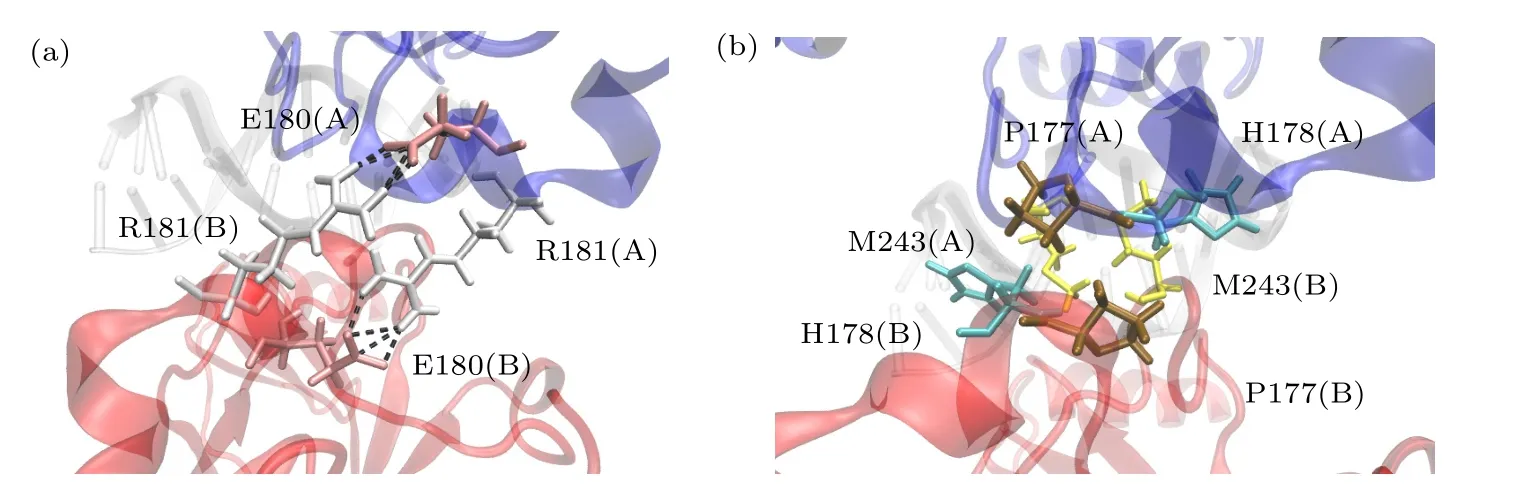
图5 A-B 单体间的相互作用残基(a) A-B 单体间形成盐桥的残基;(b) A-B 单体间参与范德瓦耳斯作用的残基Fig.5.Residues involved in interactions between A and B monomers: (a) Residues forming salt bridges between A and B monomers;(b) residues involved in van der Waals interaction between A and B monomers.
为了分析与DNA 结合前后对称二聚体的亲和力变化,采用分子力学/泊松-玻尔兹曼表面积(molecular mechanics/Poisson Boltzmann surface area,MM/PBSA)算法[37]计算对称二聚体的结合自由能.在无DNA 结合的情况下,二聚体的结合能为–23.84 kcal/mol.在与DNA 结合后,二聚体的结合能为–22.60 kcal/mol.表明在有无DNA 结合时,对称二聚体的亲和力是相似的,这与我们的静电相互作用分析结果一致.2.2 节将说明,这种结合能差异来源于DNA 分子的支撑作用.通过对A-B 间结合能的残基分解,得到了二聚作用的残基贡献分布.图6 列出了两个体系中残基贡献绝对值大于1 kcal/mol 的残基,一般认为对复合物结合的能量贡献大于1 kcal/mol 的残基是关键残基.由于A-B 单体在DNA 两侧对称分布,所以两个p53 单体的残基贡献差别很小.

图6 与DNA 结合前(a)后(b)对称二聚体的残基能量贡献分布Fig.6.Residue energy contribution distribution of symmetric dimers with (a) and without (b) DNA.
由图6 可以发现,在与DNA 结合前后,对称二聚体结合的关键残基完全一致,共有6 个关键残基,分别为来自H1 短螺旋的P177,H178,H179,E180,R181 和来自L3 环的M243.E180 和R181 参与了静电相互作用.P177,H178 和M243 虽未参与静电相互作用,但P177(A)-P177(B),H178(A)-M243(B)和M243(B)-H178(A)之间紧密接触,参与了范德瓦耳斯相互作用(VDW).两个H179 残基各自包含一个Zn 离子,提供了静电相互作用.在这6 个残基中,有4 个残基在A-B 二聚体中的能量贡献都很接近.但是,来自两个p53 单体中的E180 和R181 的贡献有明显的差别.这是由于DNA 的结合导致的构象差别造成的.在DNA 结合时,E180(A)-R181(B)盐桥更稳定.而除去DNA后,R181(A)-E180(B)盐桥更稳定.二聚作用的关键残基中,有4 个残基位于Zn 离子的配位体附近(P177,H178,H179 和M243),所以Zn 离子的稳定将会影响二聚体的稳定性.如果在Zn 离子周围的残基发生了突变[38],将会对二聚体的稳定性产生影响.
1.3.5 体表光学监测 经1.3.4行误差校正后,启动OSMS软件,再次扫描面部,静态获取新的体表参考影像Vision RT影像,在机架及影像臂不遮挡摄像头条件下采集与参考影像比对后获得的影像偏差值,3次采集监测数值均值记录为当次影像偏差值。按照治疗计划参数进行出束治疗,同时启动OSMS软件监控面部位置变化,以Vision RT影像监测当次分次内治疗,位置变化超过误差允许值2 mm,加速器出束治疗被暂停,否则继续出束治疗,采集3次监测数值均值记录为当次影像偏差值,治疗结束再次重复CBCT位置验证,得到治疗靶区位置误差并记录。
本文结果表明,除去DNA后,A-B 二聚体的结合状态几乎没有发生改变,短时间内不会由于DNA 的缺失而变构.前期的很多研究工作都表明,二聚体是通过疏水和极性相互作用来稳定的[20].NMR 结果显示H1 螺旋(P177-C182)在二聚过程中起到重要作用[39].通过X 射线晶体学确定的小鼠p53 二聚体结构中[26],由R181 和E180 形成了两对盐桥,M243 与水介导形成氢键,R177 与H178广泛参与了范德瓦耳斯接触.这些实验结果与我们的模拟结果是一致的.然而,目前实验上尚没有得到无DNA 结合情况下的对称二聚体结构[18].
2.2 DNA 分子的作用
对p53-DNA 间的相互作用分析表明,A,B 单体与DNA 结合的关键残基基本一致.如图7 所示,来自环片-螺旋基序的残基K120,R280,A276,R273 和S241 与主槽结合,环L3 上的R248 与次凹槽结合.唯一不同的残基是S121,它在单体A 中与DNA 主链结合形成氢键,而在单体B 中没有与DNA 形成氢键.
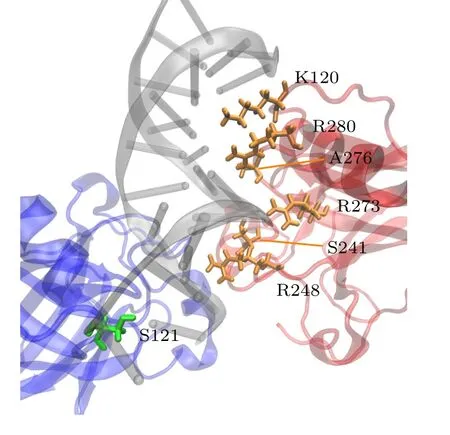
图7 对称二聚体与DNA 结合的关键残基.黄色残基表示A,B 单体与DNA 结合的一致残基,绿色表示与DNA结合不一致的残基Fig.7.Key residues involved in the binding of the symmetric dimer to DNA.Yellow residues represent consistent residues between monomers A and B and DNA binding,while green residues represent inconsistent residues with DNA binding.
下面分别计算了p53 单体、对称二聚体与DNA的结合自由能.对称二聚体与DNA 的结合能(–147.64 kcal/mol)约为p53 单体与DNA 结合能(–70.47 kcal/mol)的两倍,正是这种能量贡献,使得p53 不是以单体与DNA 结合的方式进行转录激活.同时还发现,相比于蛋白-蛋白之间的结合,p53 蛋白与DNA 间的相互作用是更加牢固的.所以,DNA 是对称二聚体形成和稳定的关键因素.如前所述,实验上尚未发现无DNA 结合下的对称二聚体的稳定结构[18],这里给出结合能的计算结果,提供了一种可能的解释,我们推测对称二聚体的形成应该是一种接受到转录信号后的协同作用.
通过能量分解,能量贡献最大的3 个残基是R273,R248,R280 (残基结合贡献大于 6 kcal/mol).而这3 个残基正是p53 突变的热点残基(R248Q,R248W,R273H,R273C,R280K).这些突变型属于接触型突变,即这些残基位于DNA 结合位点上,突变会导致p53-DBD 失去结合DNA 的能力、p53-DBD 不同程度的构象变化以及DNA 结合表面的结构重排[40,41].与此同时,这些突变的p53 蛋白具有致癌潜力.它们可以以显著负性的方式抑制任何剩余的野生型p53,也可以获得促进肿瘤生长、侵袭、转移和化疗耐药性的新功能[42].
3 二聚体-二聚体界面的相互作用
迄今为止,实验上得到的p53 核心结构域包括野生型和突变型单体、二聚体,以及各种四聚物结构.然而,位于DNA 同侧的二聚体结构还没有发现.为了探究位于DNA 同侧的二聚体在生理环境下的构象特征,从p53 四聚体中提取出单体A,D 和DNA 的结构(图8)作为初始结构,对“有”和“无”DNA 结合的A-D 二聚体分别进行200 ns 的分子动力学模拟.由于在四聚体中DNA 两侧的p53 是对称的,两个蛋白-蛋白相互作用界面也完全一致,所以选择DNA 一侧的A-D 二聚体就可以很好地描述二聚体-二聚体相互作用.
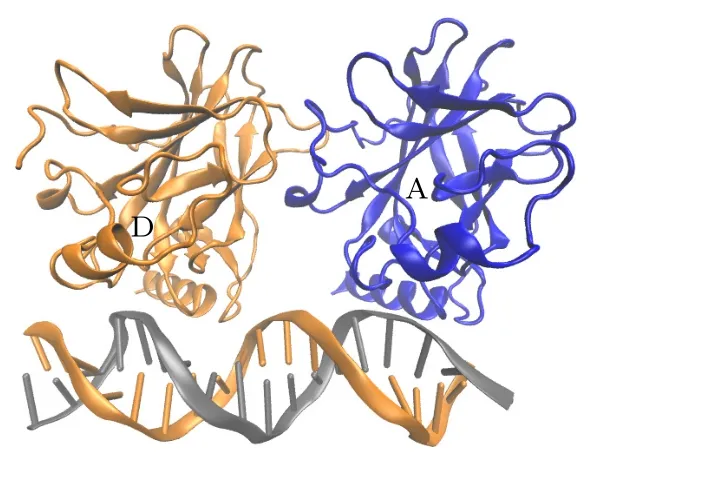
图8 由 A,D 和DNA 组成的复合物初始结构Fig.8.Initial structure of composite molecule including A,D and DNA.
3.1 无DNA 结合的A-D 二聚体不是一种合理的结构
首先对有/无DNA 结合时A-D 二聚体的结构构象进行对比.通过动力学轨迹可以发现,与DNA结合时,A-D 二聚体接触界面与晶体结构相似,而无DNA 结合时,A-D 接触界面由原来的两个接触面变成一个接触面,如图9(c)和图9(d)所示.通过计算A-D 两单体质心间距,发现当没有DNA结合时,两个单体质心间距变小.在初始时刻,A-D单体的质心间距为35.524 Å.通过计算两个体系稳定后A-D 的质心间距的概率密度分布(图9(a))可以看出,与初始时刻相比,无DNA 结合时,AD 单体的质心间距更小(最可几值为33.925 Å).与DNA结合后,两单体的质心间距有微小的增大(最可几值为36.623 Å).图9(b)显示了两个体系稳定后二聚界面接触面积的概率密度分布.从图9(c),(d)可以明显看到,在与DNA 结合的A-D二聚体内,二聚体的接触面积较小(380.2544 Å2),这是由于DNA 结合下的A-D 二聚体共形成两个接触面.而除去DNA后,单体由于相互作用逐渐靠近,两个接触面间的空腔逐渐消失变为一个接触面,接触面积增加了2.1 倍(830.1783 Å2).这种构象上的变化会使得相互作用区域发生改变,从而影响A-D 二聚体的构象.

图9 有/无DNA 结合时,A-D 二聚体构象上的差别(a) A,D 两个单体质心间距的概率密度分布;(b) A,D 分子间接触面积的概率密度分布;(c)有DNA 的A,D 分子间二聚接触面;(d)无DNA 的A,D 分子间二聚接触面Fig.9.Differences in the A-D dimer conformation with/without DNA binding: (a) Probability density distribution of the distance between the centers of mass of A and D monomers;(b) probability density distribution of the interfacial contact area between A and D molecules;(c) dimeric contact area between A and D molecules in the presence of DNA;(d) dimeric contact area between A and D molecules in the absence of DNA.
如图10(a)所示,从RMSD 的时间演化曲线可以看出,在没有DNA 结合的A-D 二聚体中,两单体在120 ns 后逐渐达到稳定,RMSD 值在(0.5634±0.0391) nm 处稳定.单体A,D 的RMSD 分别稳定于(0.2983±0.0177) nm 和(0.2177±0.0143) nm附近.与对称二聚体的结果(图4)相比,无DNA结合的A-D 二聚体的稳定需要更长的时间,且AD 二聚体的构象变化更大.在120 ns 附近A-D 二聚体的RMSD 有明显的升高.分别计算了105—120 ns 和120—135 ns 轨迹的平均结构并进行重叠,结果如图10(b)所示,可以看出,120 ns 后单体A 整体向左上方移动,单体D 向右后方移动,这会导致分子间接触面相互作用发生改变.单体间的移动使得单体D 的L6 区域和单体A 的L7,L5'区域更加靠近,从而形成了更多的相互作用.例如,在120 ns后,位于L6 的G224,S227分别与L5'上的N210 形成新的氢键,位于L6 的V225 分别与位于L7 上的N263 和L264 形成新的氢键.
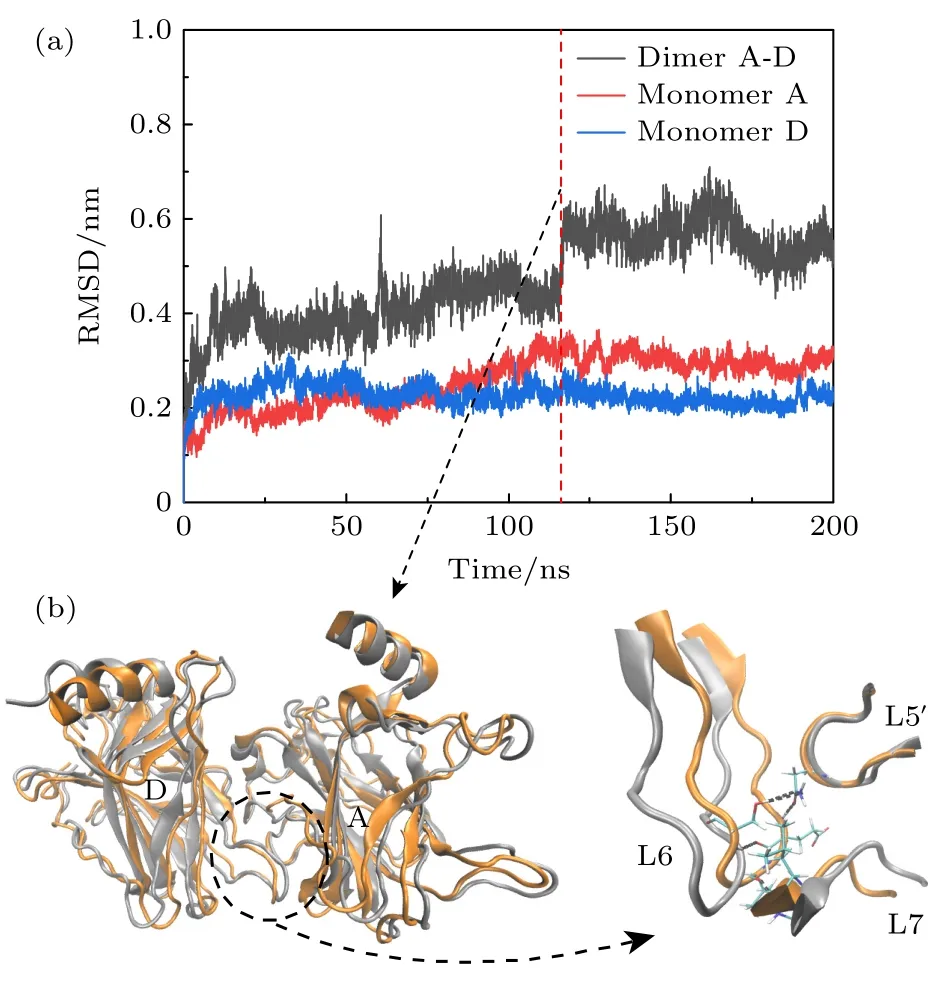
图10 (a)无DNA 结合时A-D 二聚体 的RMSD演化;(b) 120 ns 前后A-D 二聚体的构象重叠,两单体的构象发生了明显的偏移,使得A-D 之间的相互作用增加.120 ns前后的构象分别用银色和橙色表示Fig.10.(a) RMSD evolution of the A-D dimer in the absence of DNA binding;(b) at around 120 ns,there is an overlap in the conformation of the A-D dimer,with noticeable deviations in the conformations of the two monomers,leading to an increased interaction between A and D.Conformations before and after 120 ns are represented in silver and orange,respectively.
对接触界面的相互作用分析表明,稳定后的A-D 二聚界面大约有8 个氢键和2 个盐桥,稳定性分析见表1.这些氢键和盐桥分别来自R267-D228(盐桥),R209-E224 (盐桥),S94-L201,N210-D228,S94-G199,L264-V225,S99-D228,S96-T231,N263-V225 和N210-S227 (左侧为单体A 的残基号,右侧为单体D 的残基号).这与晶体结构中A-D 单体间的相互作用完全不同.从结构和相互作用分析上来看,无DNA 结合的A-D 二聚体变为一种结合更为紧密的二聚形式.然而,这种结合更紧密的A-D二聚体却不是一种合理的结构.将无DNA 结合时的模拟结果与有DNA 的结构进行重叠,如图11所示,除了A-D 接触界面的变化外,上述形成的无DNA 结合的稳定的A-D 二聚复合物(图9(d))已经不能与连续的同源DNA 序列结合.因此,在生理环境下,没有DNA 结合时,位于DNA 同侧的二聚体不能保持合理的构象,这不利于p53-DBD 四聚体的形成.这也解释了为什么在四聚体的形成过程中,优先形成对称二聚体.
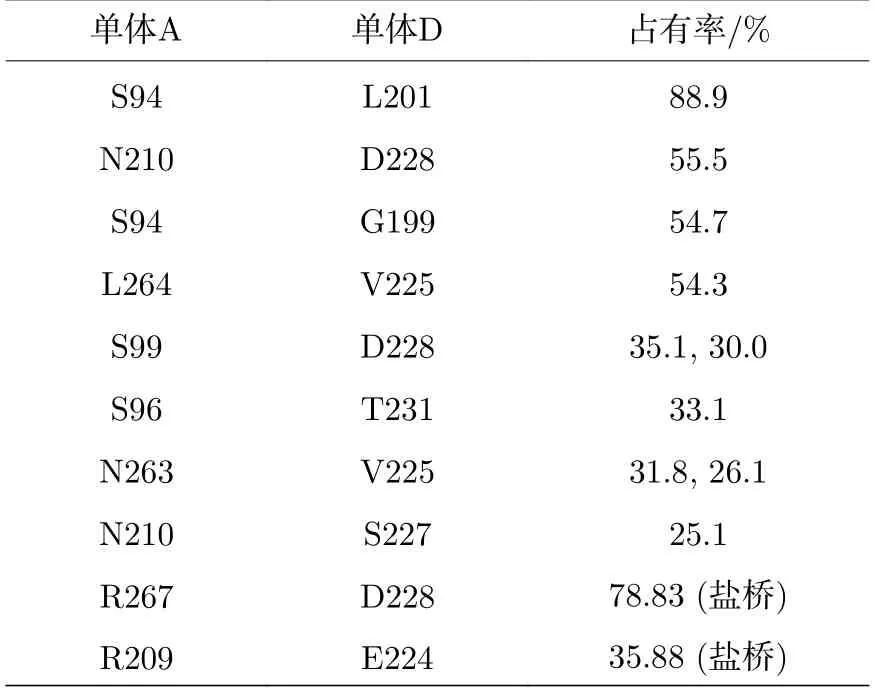
表1 氢键、盐桥稳定性Table 1.Hydrogen bond and salt bridge stability.

图11 无DNA 时的A-D 二聚体不能与DNA 结合Fig.11.A-D dimer can’t bind to DNA in the absence of DNA.
那么,这是否说明游离的p53-DBD 会产生很多不合理的寡聚呢?答案是否定的.因为在体内,p53-DBD 除了与DNA 形成四聚体的有效功能外,还会和其他蛋白之间存在蛋白-蛋白相互作用[43–45],这些相互作用使得p53 能够通过多种途径调控肿瘤发展.与此同时,A-D 二聚体与DNA 的复合物能够保持与晶体结构相似的构象状态,说明DNA在其中起到了重要作用,下面将详细分析DNA 在A-D 二聚体中的作用.
3.2 DNA在A-D 二聚体中的骨架作用
3.1 节计算了有/无DNA 结合时A-D 单体间的质心距离,发现在没有DNA 结合时,A-D 单体质心间距相对较小;而有DNA 结合时A-D 单体间质心距离相对较大.为了分析产生这一现象的原因,在有DNA 结合时的A-D 二聚体 200 ns 得到的分子结构中剪去DNA 分子,采用伞形取样方法[46],得到应用在A-D 分子上的弹性应力,此力原则上与DNA 分子的作用相同,抵消了A-D 分子间的静电力和范德瓦耳斯力,从而保持A-D 分子间一定的空间距离不变.具体来说,在与DNA 结合的A-D 二聚体中,平衡后两单体的平均质心间距为3.6109 nm,以此平衡结构作为初始结构,剪去DNA分子,对A-D 二聚体采用伞形取样的方法,设置A-D 质心的参考距离为 3.6109 nm,进行 10 ns 的模拟,得到相应的弹性应力,如图12(a)所示.在模拟初期,由于两单体质心间距大于参考距离,所以两单体之间的弹性应力的方向沿着质心轴向内,而在 4.5 ns后,质心距离小于参考距离,所以弹性应力的方向沿着质心轴向外,最终弹性应力稳定在一个确定的大小.

图12 (a)二聚体上的弹性应力;(b)稳定后,弹性应力作用下A-D 二聚体的结构Fig.12.(a) Elastic stress on the dimer;(b) structure of the A-D dimer under the action of elastic stress after stabilization.
如图12(b)所示,在此情况下,A-D 单体间形成两个清晰的接触面.接触面1 上有两条稳定氢键,分别为T170(A)-G199(D)和S166(A)-H233(D).接触面2上,S99(A),R267(A)与V225(D)之间形成稳定性较弱的氢键.与实验得到的结晶结构相比,虽然相互作用残基有所差别,但是相互作用区域是一致的.本文的结果表明,与DNA 结合时,A-D单体各自与DNA 的一致序列结合,DNA 分子提供骨架的支撑作用,使A,D 单体保持一定的空间距离.DNA 分子的支撑作用是DNA 同侧二聚体稳定的一个关键机制.
3.3 与DNA 结合的A-D 二聚体
A-D 二聚体可以在与DNA 结合时,保持合理的构象状态,对这个体系的稳定性和相互作用进行细致分析,探究在DNA 同侧的单体在动力学过程中的相互作用特点.首先,对与DNA 结合的A-D 二聚体的稳定性分析表明,p53 A-D 二聚体的RMSD在 120 ns 后达到稳定(约(0.3231±0.0194)nm),如图13(a)所示.此外,单体D在150 ns 后才达到稳定,单体D 的RMSD 稳定在(0.3505±0.011) nm,且在90—150 ns 期间有明显上升,如3.1 节中的分析,说明在这个时间前后单体D 存在构象变化.
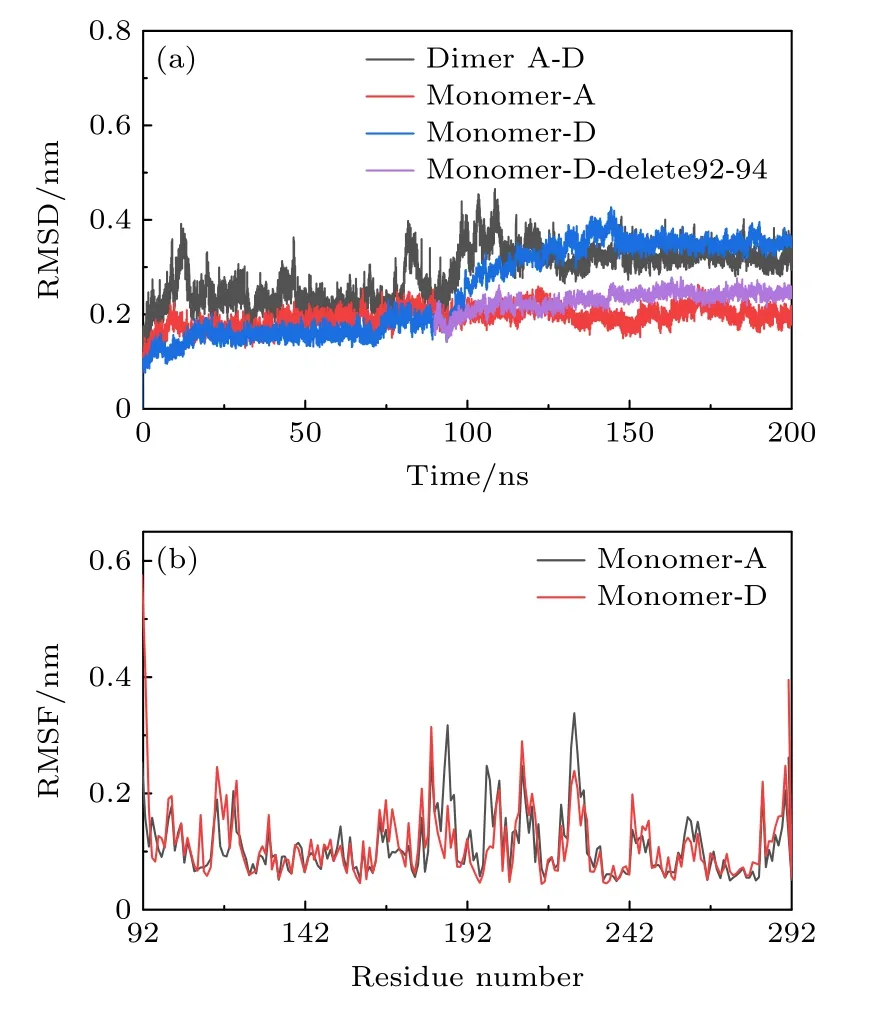
图13 有DNA时,A-D 二聚体 的稳定 性(a) RMSD;(b) RMSFFig.13.Stability of A-D dimer in the presence of DNA:(a) RMSD;(b) RMSF.
通过计算残基的均方根波动(root mean square fluctuation,RMSF),可以得到各个残基的灵活性情况.如图13(b)所示.A,D 两单体的RMSF 趋势总体上是一致的,但单体D 的RMSF 在残基92—94 处明显增大.这是由于此区域正处于p53-DBD区域的结构边缘,此模型忽略了p53 的1—91 号残基(N 端)和292-393 (C 端)残基的影响.在除去92—94 这3 个残基的影响后,单体D 也在 20 ns后一直保持稳定(图13(a)紫色线).这表明92—94段残基的灵活性增强并不会影响单体D 其他区域的稳定.而在单体A中,92—94 残基区域正处于p53 A-D 二聚界面上,参与了蛋白-蛋白相互作用,所以,单体A 的N 端区域没有灵活性增强的情况.此外,在186—225 残基区间,发现A,D 单体的个别残基(186,187,198 和225 等)灵活性有差别,这是由于这些残基位于p53 A-D 二聚接触界面的环路上,由于两个单体是平行分布的,所以位于边缘的环路受到的约束更少,导致残基的灵活性更大.
对p53 A-D 二聚体界面的相互作用分析表明,A-D 单体之间存在两个接触面.在两个接触面上存在盐桥和丰富的氢键网络,所有氢键和盐桥的稳定性分析见表2.如图14 所示,位于接触面1 上的T170(A)-G199(D)形成一条稳定氢键,P92(A)-D186(D)之间形成两条稳定性很弱的氢键.位于接触面2 上的K101(A),分别与D 链的E224 形成稳定盐桥,与P222,P223 形成不稳定的氢键.接触面2 上还有一条不稳定氢键,来自Q100(A)-P223(D).结果表明,与接触面2 上的复杂相互作用相比,接触面1 上的氢键更稳定,更简单.而接触面2 上的残基参与的相互作用更复杂.P222,P223 和E224位于L6环,P92,K101 位于N 端无序区域,这些区域的灵活性较强,导致接触面2 的相互作用稳定性也较差,这也促使某些残基参与了更多的相互作用(如K101).此外,T170 和D186 位于L2 环上,G199 位于L5 环上,表明L2,L5,L6 以及N 端无序区域是A-D 二聚体内静电相互作用的关键区域.

表2 与DNA 结合时A-D 二聚体间的氢键、盐桥稳定性Table 2.Stability of hydrogen bonds and salt bridges between A-D dimers when binding to DNA.
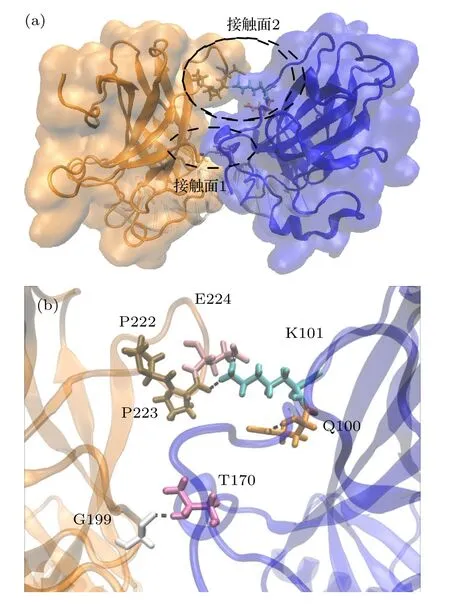
图14 与DNA 结合的A-D 二聚体间的(a)接触面和(b)相互作用残基Fig.14.Contact surface (a) and residues involved in the interaction (b) between the A-D dimer bound to DNA.
p53 的每个单体都与DNA 间形成广泛的氢键网络.单体A 和单体D 与DNA 间平均有12 和11条氢键.如图15 所示,对于单体A,K120,A276,R283,R280,R273,N239,N288 和S241 与主凹槽相互作用,其中R280 深深地插入主凹槽,形成2 条稳定的氢键.R283,S241 和K291 只在单体A 中与DNA 形成氢键,而在单体D 中没有.R248 和K291与次凹槽形成稳定的氢键,但R248 并未插入到凹槽内部,而是与DNA 主干形成3 条氢键.对于单体D,K120,A276,N239,R280,R273,N288 和S121与主凹槽相互作用.与单体A 不同的是与主凹槽相互作用的残基增加了S121,与主凹槽形成两条氢键.R248 与次凹槽形成稳定的氢键.结果表明,A-D 两单体与DNA 形成的氢键网络和各氢键的稳定性是有差别的,单体A 中S241 贡献了一条稳定性最强的氢键,在单体D 中没有出现.相反,单体D 中由S121 贡献了两条稳定的氢键,在单体A中则没有.这些相互作用确保了A,D 单体与DNA的稳定结合,表明A-D-DNA 的复合物分子也是一个稳定的结构,可以正确描述四聚体中的结构特点.

图15 单 体A,D 与DNA 间相互作用的 关键残基.黄色残基表示A,D 单体与DNA 结合的一致残基,绿色表示与DNA 结合不一致的残基Fig.15.Key residues involved in the interaction between monomers A and D with DNA.Yellow residues represent consistent residues between monomers A and D and DNA binding,while green residues represent inconsistent residues with DNA binding.
通过结合自由能计算,在DNA 结合状态下,A-D单体间的结合能为–17.17 kcal/mol,与2.2 节给出的对称二聚体的结合能(–22.60 kcal/mol)相比较小.因此,相对于对称二聚体,A 与D 单体之间相对结合较弱,这一结果与实验给出的结论是一致的.结合能的残基分解如图16 所示,A-D 二聚体的关键残基包括来自单体A 的L93,K101,Q167和来自单体D 的D186,L201.如前所述,K101(A)和D186(D)分别参与了静电相互作用.L93(A)-G199(D),L93(A)-E171(A),Q167(A)-H233(D),S94(A)-L201(D)和S95(A)-L201(D)之间形成范德瓦耳斯相互作用.

图16 与DNA 结合的A-D 二聚体的能量分解Fig.16.Energy decomposition of the A-D dimer binding to DNA.
在结晶实验中得到的p53-DBD 四聚体结构[23],DNA 同侧的两单体间有大量的范德瓦耳斯接触和氢键网络.二聚体-二聚体界面的氢键网络组成主要包括: 接触面1 由T170(A)-G199(D)形成氢键;接触面2 的氢键网络由K101,Q100-E224,以及K101-V225 组成.文中与结晶实验有差别的氢键位于接触面2.如图13(a)所示,通过观察动力学轨迹发现,接触面2 两侧残基间距略有增加,且K101 残基的灵活性较大,会利用其灵活性大的特性与E224 形成盐桥.于此同时,还与距离较近的P222 和P223 形成较弱的氢键.本文的结果表明,位于DNA 同侧的A-D 二聚体可以形成稳定的结合界面,虽然A-D 二聚体的亲和力弱于对称二聚体,但是它介导的蛋白-蛋白相互作用是四聚体形成过程中的必要条件,它能够促进两个对称二聚体的协同结合,进而形成稳定的p53 四聚体.
4 结构域之间的协同作用
早期对DNA 与p53 蛋白家族(包括p53,p63和p73)结合的比较研究表明,p53 的H1 螺旋界面二聚化对协同结合至关重要[47].为了得到二聚体中两个单体之间的关联作用,计算了体系中所有主链碳原子(共400 个)的动态互相关图(dynamics cross correlation map,DCCM)[48].图17 提供了在模拟过程中蛋白质残基之间的相关运动的总体图像,图中的正高峰(深红色)代表对应的两个残基的运动有很强的相关性,而负值(蓝色)区域则代表两个残基向相反的方向运动(anticorrelated motion).这两种不同的相关性,特别是结构中相距较远的蛋白质之间的长程关联,都与蛋白质的生物功能密切相关[49].图中左上角和右下角的信息是完全对称的,对角线上的两个正方形中的信息代表单体内部的残基间的关联作用.而我们重点关注的是结构域之间的残基相关性.
在不含DNA 的体系中,可以观察到对称二聚体的A-B 单体之间有很强的正相关性.单体A 的176—181 区域分别与单体B 的123—125,137—139,176—181,237—245 以及276—277 区域的残基有较强的正相关性,单体A 的242—243 区域与单体B 的178—179 区域也发现了强相关性.其中A (176—181)与B (176—181)的强相关区域最大,并且与A (242—243)-B (178—179)都是参与蛋白-蛋白相互作用的区域,这与第2 节所述不含DNA 对称二聚体系的结论一致.在与DNA 结合的对称二聚体中,只在A (176—181)-B (176—181)和A (242—243)-B (178—179)区域有较强的正相关性.这是由于DNA 分子的支撑作用,使得A-B分子之间的相关性降低,而且,通过DNA 分子的支撑,A-B-DNA 复合分子更加稳定.这一点与RMSD 结果一致,在图4中,A-B结合DNA后,系统表现出很强的稳定性.综上所述,通过动态互相关网络图得到了对称二聚体协同结合DNA 的直接证据,各分子单体之间有明显的协同效应.除了参与蛋白-蛋白相互作用的区域之外,还发现了其他残基的长程正相关性.
四聚体结构研究表明[24,50],对称二聚体中两个p53 单体之间的作用在很大程度上强于A-D 二聚体中两个p53 单体之间的作用.在无DNA 结合时,A-D 二聚体由于没有DNA 的支撑,两单体的质心逐渐减小,变为一种生物学上不合理的结构.从动态相关图上可以看出,即使在这种情况下,两个p53 单体的动态相关性仍然相对很弱.结合了DNA 后的A-D 二聚体,相比对称二聚体,A-D 二聚体中p53 单体间残基的相关性较弱,在A (105—108,148)和D (180—184)的区间发现了较弱的正相关性.这一结论与A-D 二聚体中两个p53 单体之间的相互作用较弱有关.
5 总结
了解蛋白质的结构和内部相互作用有多种途径.通过X 射线单晶衍射、核磁共振、电子衍射等实验手段可以确定蛋白质、多糖、核酸、病毒等生物大分子的晶体结构,这些结构通常是静态的.在体外培养实验中观察蛋白相互作用,常常受到体外环境以及温度的限制,而且缺乏对结构细节的描述和理解.分子动力学模拟可以在合适的力场和参数设定下计算动力学过程中晶体结构的演化,同时可以设定分子处于目标环境(如生理环境)下.因此,分子动力学模拟能够从原子层次上提供生物大分子结构和动力学的充足信息.为了理解p53-DBD四聚体中的两种蛋白-蛋白相互作用在四聚体形成过程中的作用,本文分别对位于DNA 对侧和同侧的p53 蛋白进行了分子动力学模拟,观察在动力学稳定后蛋白-蛋白相互作用的演化,并分析DNA 在两个模型中的重要性.应该指出,采用全原子分子动力学方法,对p53 活性四聚结构中分子间相互作用的动力学行为和稳定性进行系统的分析和讨论,迄今为止未见报道.
本文的研究表明,在活性p53-DBD 四聚体中,对称二聚体是一种稳定的结构.在与DNA 结合中,对称二聚体主要依靠H1 短螺旋和L3 环上的残基贡献的非键相互作用,使对称二聚体保持稳定的构象和很强的二聚亲和力.此外,对称二聚体还表现出很强的协同作用,这与对称二聚体协同结合DNA 的实验事实一致.位于DNA 同侧的A-D 二聚体与DNA 结合时,通过L2,L5,L6 环路以及N端无序区域组成的两个接触面形成相互作用,并维持稳定的结合状态.但除去DNA后,A-D 二聚体会演化成一种不合理的结构,此结构不利于p53-DBD 四聚体的形成.对DNA 的作用分析表明,DNA 在A-D 二聚体中起到了重要的骨架支撑作用.以上结果中各个p53 单体与DNA 的相互作用与实验上的结果一致,且蛋白-DNA 间的相互作用远强于两种蛋白-蛋白相互作用,即与DNA 结合的二聚体受益于DNA 和二聚体的稳定接触.本工作对p53 核心结构域的四聚体形成机制提供了见解,也表明了A-D 二聚体与DNA 的复合物是一个稳定的系统,能够正确地反映四聚体内的相互作用.
值得指出,p53-DNA 四聚体的结构取决于它所结合的DNA 序列,p53-DBD 四聚体在DNA 上的二聚体-二聚体接触在体内未必是固定的[24],而可能是根据不同的细胞条件适应不同的接触.本文所研究的结构是4 个p53 亚基与DNA 一致序列结合的结构,而在一些实验中,发现了DNA 弯曲的四聚体[25].而且,由于计算资源所限,本文仅对对称和同侧两个二聚结构做出了分析.今后,我们还将探索不同的四聚体中蛋白-蛋白相互作用,对p53 核心四聚体全原子结构内的相互作用做出更系统的分析和讨论.
