我国华东与华南地区养殖鱼类迟缓爱德华氏菌分离株的多样性分析
2024-03-15李素一池洪树张晓佩许斌福
李素一,池洪树,陈 斌,张晓佩,许斌福
爱德华氏菌属(Edwardsiella)为肠杆菌科革兰氏阴性杆菌,是人兽共患的重要致病菌,传统上分为迟缓爱德华氏菌(Edwardsiellatarda)、鲶鱼爱德华氏菌(Edwardsiellaictaluri)和保科爱德华氏菌(Edwardsiellahoshinae)3个种[1-2]。其中,迟缓爱德华氏菌和鲶鱼爱德华氏菌是水产动物的主要致病菌,保科爱德华氏菌是鸟类和爬行动物的致病菌。鲶鱼爱德华氏菌只感染鱼类,可引起鲶鱼的肠道败血症。而迟缓爱德华氏菌 (E.tarda) 具有广泛的宿主适应性,能感染淡水海水养殖鱼类、爬行动物、两栖动物和哺乳类等多种动物;流行性广、易暴发、危害大,是水产上常见的致病菌[3-4]。鱼类迟缓爱德华氏菌是大菱鲆、鳗鲡等多种重要经济鱼类的首要病原之一,一旦暴发感染往往导致鱼类的大量死亡,危害极其严重[5-6];同时该菌也可感染人类,引起细菌性胃肠炎、腹泻、败血症等,威胁着人类的公共卫生安全[7-8]。爱德华氏菌分布广泛,宿主多样,因此多样性分析对了解爱德华氏菌菌群的种属关系或变异情况、掌握其流行规律、研发疫苗、制定精准的免疫防控策略具有重要的指导意义。迟缓爱德华氏菌的分型传统上以生理生化和血清分型为主,随着基因测序技术的产生,16S rRNA、hsp60等基因测序分析的方法逐步应用到迟缓爱德华氏菌的鉴定与分型中。传统的血清型分析主要是基于菌体表面抗原与特异性血清抗体发生凝集反应来进行鉴定分型,具有特异、灵敏、快速、简便的优点,但存在无法精准对迟缓爱德华氏菌进行鉴定与分型的局限。例如陈翠珍[9]用菌体-血清凝集的方法对130株分离自牙鲆的迟缓爱德华氏菌分离株进行血清分型,结果表明均为同种血清型。研究表明,16S rRNA在漫长的进化史中高度保守,被称为是细菌进化的分子钟,联合hsp60等基因测序对迟缓爱德华氏菌进行分子层面分型分析的方法具有更高的种间与种内分辨率,能补充利用血清学分型方法的不足。本研究对本室近年来分离保存和收集的来源于我国华东地区(福建、山东、浙江)与华南的广东地区的17株迟缓爱德华氏菌进行多样性分析,以期为迟缓爱德华氏菌的区系划分提供参考,为迟缓爱德华氏菌疫苗的开发设计和免疫防控策略制定提供依据。
1 材料与方法
1.1 材料
1.1.1 爱德华氏菌菌株 本研究所用的爱德华氏菌菌株信息如表1所示,其中17株由本室保存的迟缓爱德华氏菌分离株来自我国华东地区(福建、浙江、山东)及华南地区(广东),其中12株迟缓爱德华氏菌分离自鳗鲡(包括欧洲鳗鲡、日本鳗鲡、花鳗鲡),4株分离自大菱鲆,1株分离自鳜鱼。
1.1.2 主要试剂及材料 细菌培养基TSB、TSA购自广东环凯微生物科技有限公司,细菌DNA提取试剂盒、琼脂糖凝胶DNA回收试剂盒及质粒小量提取试剂盒购自天根生化科技有限公司,PCR引物由上海生工有限公司合成,PCR克隆试剂盒(Ex Taq酶、dNTP等)、PMD18-T克隆载体购自大连宝生物工程有限公司(Takara),宿主菌Escherichiacoli(DH5α)由本室保存。
1.2 方法
1.2.1 细菌复苏鉴定及基因组DNA提取 复苏保存于-80 ℃菌株,在TSA平板上划线,28 ℃培养36 h,挑取单菌落于10 mL TSB培养液,在28 ℃、200 r/min条件下振荡培养过夜。按1%接种于新的10 mL TSB培养液,培养至OD600≈1.0。利用API-20E生化鉴定系统再次对标本迟缓爱德华氏菌分离株进行生化鉴定,确定为迟缓爱德华氏菌后,用于后续试验。收集5 mL细菌培养液,用天根细菌DNA提取试剂盒(DP304-03)按操作说明提取菌株基因组DNA,作为PCR扩增的模板。其余5 mL培养液在10 000 r/min条件下常温离心5 min收集菌体,用1 mol/L PBS(pH7.0)洗涤,再次10 000 r/min条件下常温离心5 min后用1 mol/L PBS(pH7.0)重悬,制备浓度为3×108CFU/mL的菌液,用于菌体-血清凝集试验。
1.2.2 血清型分析 参考许斌福等[10]的方法分别制备了迟缓爱德华氏菌ATCC15947、ETY及鲶鱼爱德华氏菌EIV的兔抗O血清。参考陈斌[11]等的方法,将1.2.1所制备的迟缓爱德华氏菌菌液加入96孔微孔板,每孔25 μL,分别加入同体积倍比稀释的ATCC15947、EIV、ETY 3种爱德华氏菌兔抗O血清以及正常兔血清并混匀,试验设3组重复,在37 ℃条件下静置温育30 min后观察凝集结果。
1.2.3 PCR扩增16S rRNA与hsp60基因序列 利用细菌16S rRNA (16S small subunit ribosomal RNA)基因通用引物及参照NCBI基因库中的hsp60 (heat shock protein 60)基因序列,设计引物扩增迟缓爱德华氏菌的16S rRNA及hsp60基因部分序列。根据Ex Taq(Takara)试剂盒使用说明配制PCR反应体系,进行PCR扩增。16S rRNA产物预期大小为1 508 bp,hsp60基因产物预期大小约1 099 bp。本研究使用的引物及产物大小如表2所示。

表2 16S rRNA及hsp60基因引物
PCR反应体系(50 μL):10×Ex Taq Buffer 5 μL,dNTP Mixture (10 mmol/L) 4 μL,Ex Taq DNA聚合酶(5 U/μL) 1 μL,上下游引物(10 mmol/L) 各1 μL,模板1 μL,ddH2O 37 μL。
16S rRNA的PCR反应程序为:94 ℃ 5 min;94 ℃ 1 min,55 ℃ 1 min ,72 ℃ 1 min,30个循环;72 ℃ 10 min。hsp60的PCR反应程序为:94 ℃ 5 min ;94 ℃ 1 min,51 ℃ 1 min ,72 ℃ 1 min,30个循环;72 ℃ 10 min。用1%琼脂糖凝胶电泳检测并回收PCR产物,与PMD18-T克隆载体连接后转化大肠杆菌E.coliDH5α,经PCR验证具有插入片段的阳性质粒送Takara公司测序,取3次平行测序结果一致的序列为最终序列,提交到GenBank,获得序列登录号(表4)。
1.2.4 序列分析与系统进化树的构建 将所得的16S rRNA及hsp60序列,在NCBI上BLAST比对分析。16S rRNA序列和hsp60序列集合分别使用MEGA11进行1 000次Bootstrap采样及多序列比对,并构建NJ系统发育树。
2 结 果
2.1 血清型分析 菌体凝集试验显示(表3),分离自浙江的日本鳗鲡迟缓爱德华氏菌ETY的抗O血清能与大部分鱼源迟缓爱德华氏菌发生凝集反应,其中包括分离自山东地区大菱鲆的所有迟缓爱德华氏菌分离株;与分离自福建地区日本鳗鲡的ET080511的凝集效价最高,为212,但不与人源迟缓爱德华氏菌ATCC15947凝集。ATCC15947作为人源迟缓爱德华氏菌标准株,其抗O血清除了能凝集本菌外,仅与鱼源迟缓爱德华氏菌WY37产生效价较低的凝集反应。鲶鱼爱德华氏菌EIV的抗O血清只能与自身EIV菌株发生凝集,无法与其他任何鱼源或人源的迟缓爱德华氏菌发生凝集。
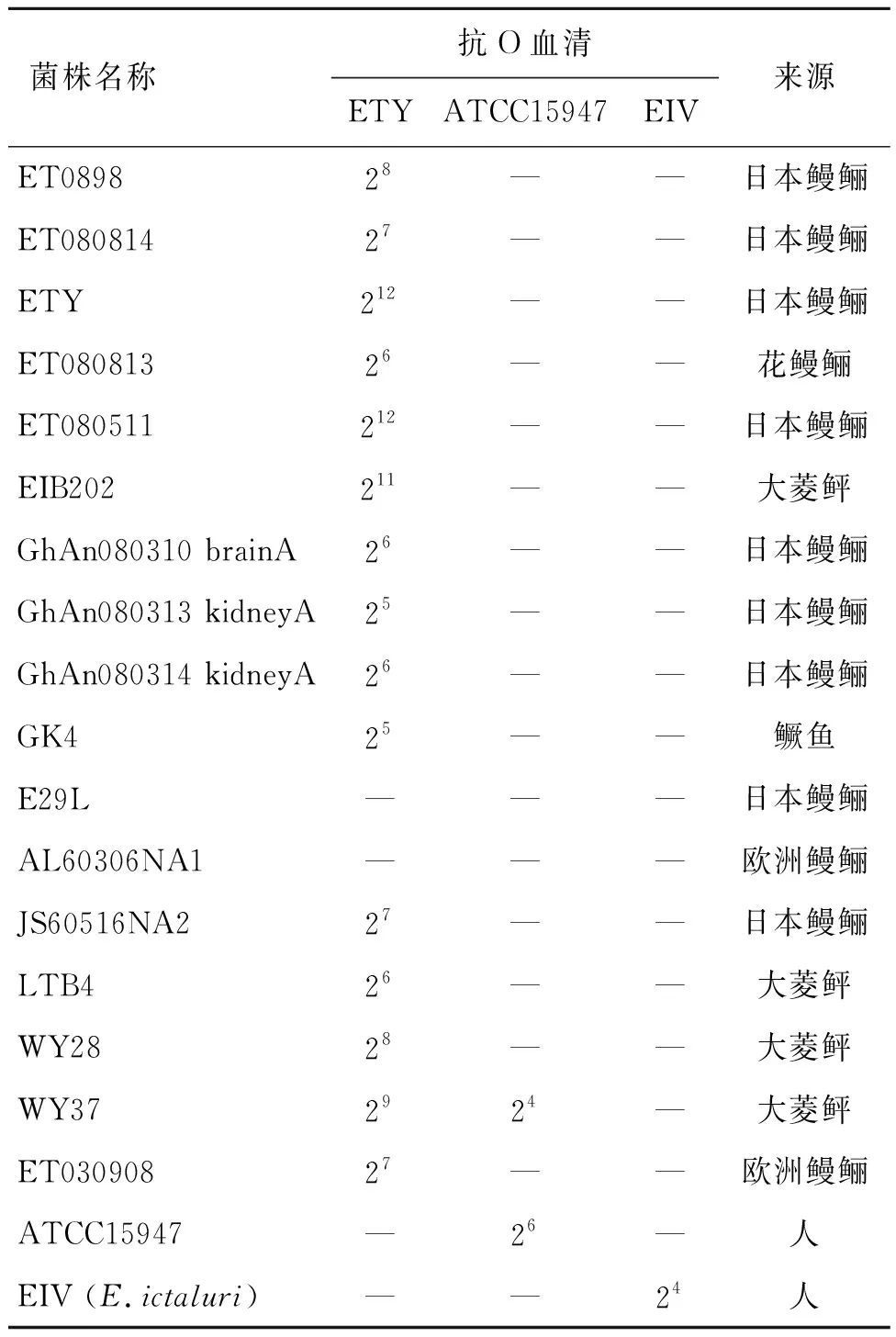
表3 各菌株与3种抗O血清的凝集结果
2.2 基于16S rRNA及hsp60序列的遗传进化分析 将PCR扩增获得的ET0898等17株迟缓爱德华氏菌及鲶鱼爱德华氏菌EIV的16S rRNA序列及hsp60序列上传至NCBI,获得基因序列号,并在GenBank中与已知的相关序列进行比对分析,同时从NCBI下载保科爱德华氏菌属(E.hoshinae)ATCC35051、及大肠杆菌BCE049的16S rRNA与hsp60序列作为构建系统进化树的参照(表4)。一共21个16S rRNA及hsp60序列用于构建系统进化树。
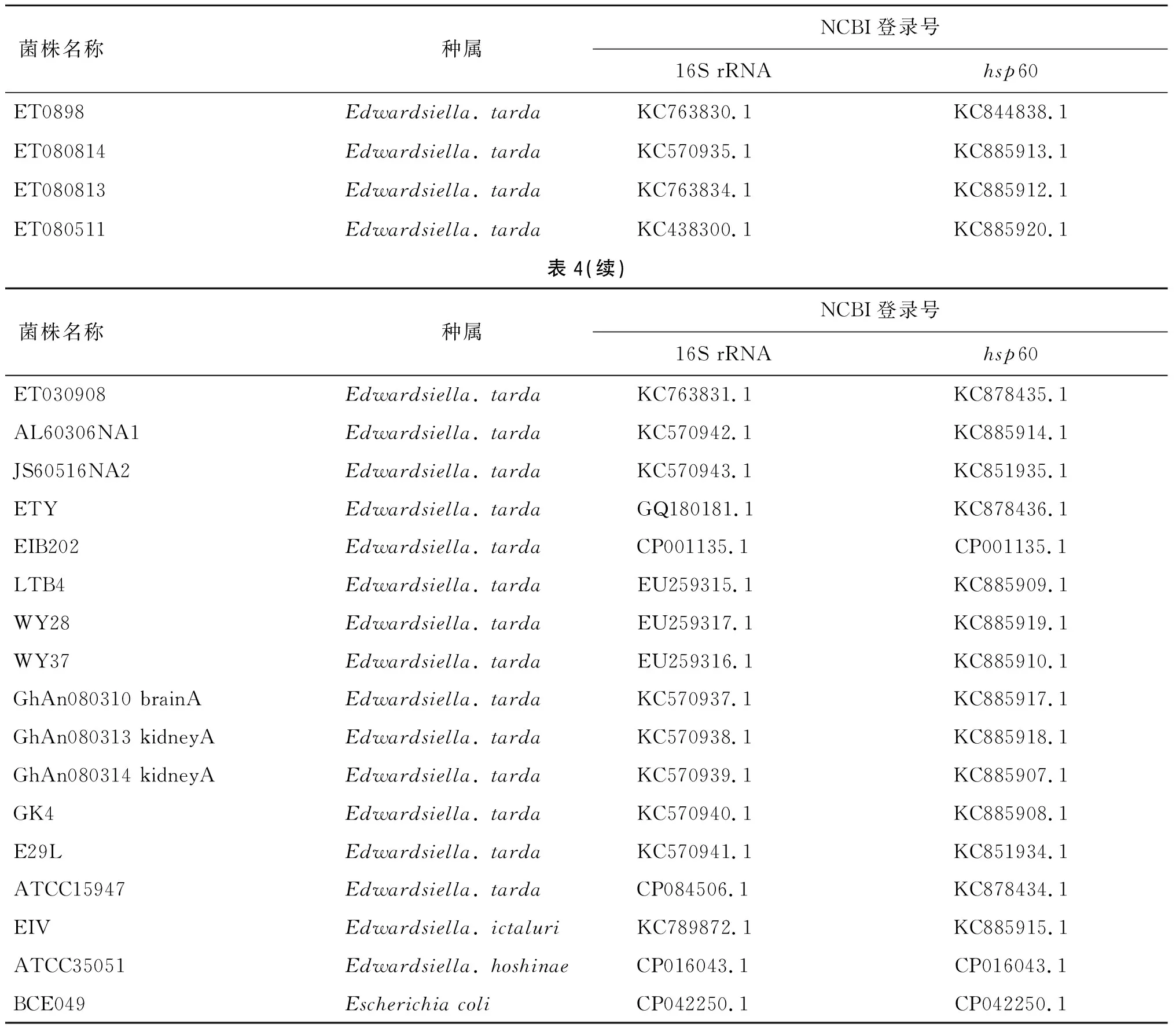
表4 用于构建系统进化树的菌株16S rRNA及hsp60
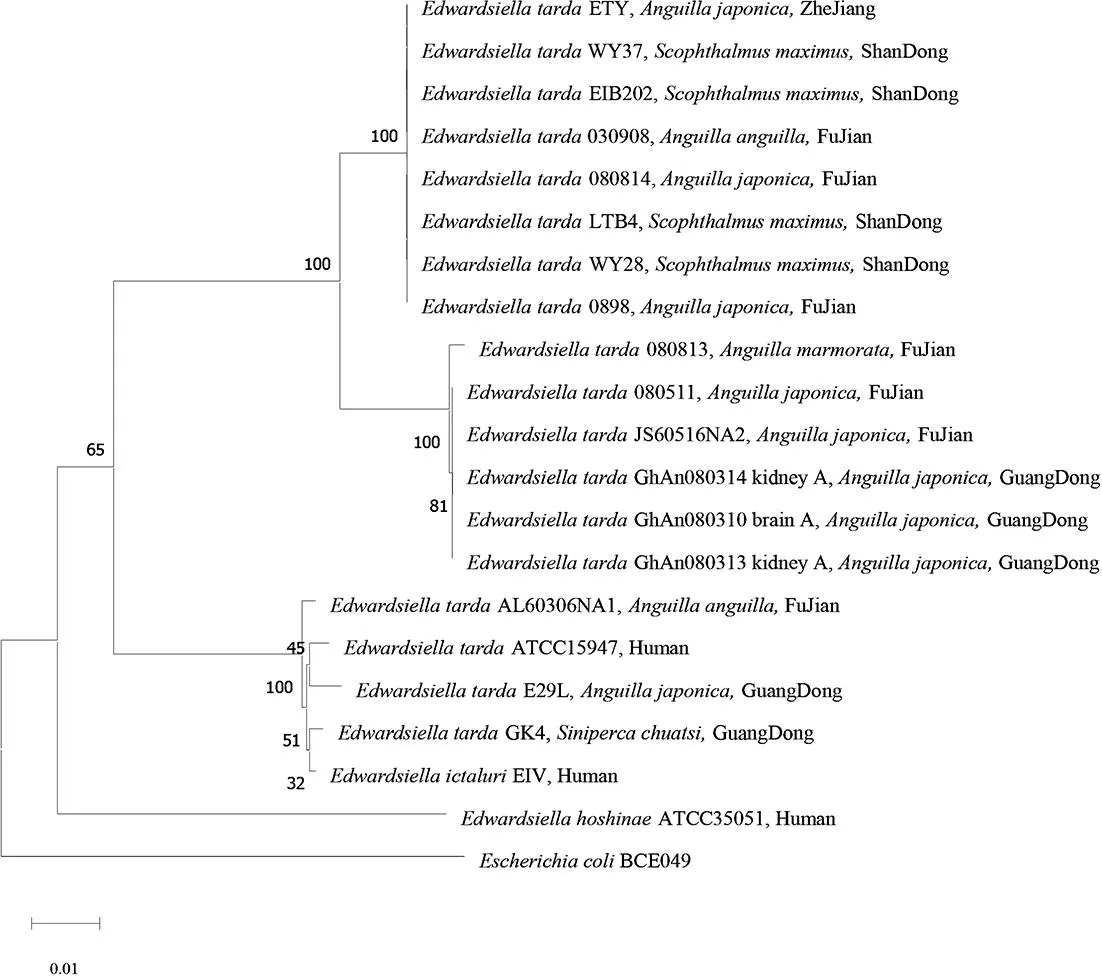
基于16S rRNA序列构建的系统进化树如图1显示,绝大部分鱼源迟缓爱德华氏菌进化同源性较高,其中来源于山东大菱鲆的迟缓爱德华氏菌分离株聚为一支;来源于福建的迟缓爱德华氏菌ET0898、ET080814、ET080813、ET080511、ET030908、JS60516NA2、来源于浙江的迟缓爱德华氏菌ETY、来源于山东的迟缓爱德华氏菌EIB202、LTB4、WY28、WY37及来源于广东的迟缓爱德华氏菌GhAn080310 brainA、GhAn080313 kidneyA、GhAn080314 kidneyA相聚类;来源于福建的AL60306NA1与来源于广东的E29L、GK4及人源迟缓爱德华氏菌ATCC15947相聚,而鲶爱德华氏菌EIV及保科爱德华氏菌ATCC35051与鱼源迟缓爱德华氏菌有一定的进化距离。

注:从左到右排列依次是菌株名称、宿主来源、分离地区。
图2显示,基于hsp60部分序列构建的菌株系统发育进化树结果显示:6株来源于福建的迟缓爱德华氏菌(ET0898、ET080814、ET080813、ET080511、ET030908、JS60516NA2)、1株来源于浙江的迟缓爱德华氏菌ETY、4株来源于山东的迟缓爱德华氏菌(EIB202、LTB4、WY28、WY37)及3株来源于广东的迟缓爱德华氏菌(GhAn080310 brainA、GhAn080313 kidneyA、GhAn080314 kidneyA)相聚类。来源于福建的迟缓爱德华氏菌AL60306NA1与来源于广东的迟缓爱德华氏菌E29L、GK4及人源迟缓爱德华氏菌ATCC15947相聚。这两个结果与16S rRNA系统发育进化树一致。有差异的是,鲶鱼爱德华氏菌EIV与迟缓爱德华氏菌GK4聚为一支。此外,保科爱德华氏菌ATCC35051与所有的迟缓爱德华氏菌及鲶鱼爱德华氏菌显示出明显的进化差距。
注:从左到右排列依次是菌株名称、宿主来源、分离地区。
3 讨 论
迟缓爱德华氏菌是全球范围内水产养殖业最重要致病菌之一,具有广泛的表型和遗传多样性[12-13]。随着我国水产养殖行业的蓬勃发展,不同区域和宿主来源的迟缓爱德华氏菌分离株也在不断增加[14],对迟缓爱德化氏菌分离株进行多样性分析既有助于推进爱德华氏菌的流行病学研究,又为疫苗开发提供依据。
细菌菌体O抗原与血清的凝集反应操作简便、敏感性高,是常用于判定细菌血清型的基本分型方法[15]。细菌菌体O抗原是由多糖重复单元构成的多聚糖,具有多样性,基于O抗原的细菌血清凝集方法是划分细菌血清型的主要标准。本研究前期制备了迟缓爱德华氏菌ETY、ATCC15947及鲶鱼爱德华氏菌EIV的菌体O抗原及抗O血清,利用三种抗爱德华氏菌抗O血清与标本迟缓爱德华氏菌进行凝集反应,初步判断迟缓爱德华氏菌分离株的血清学类型。凝集试验结果显示,分离自我国华东与华南地区的的大部分迟缓爱德华氏菌O抗原血清型相似,但迟缓爱德华氏菌福建株AL60306NA1和广东株E29L与三种菌体O抗原均不发生凝集,说明鱼源迟缓爱德华氏菌存在不同的血清型。鱼源迟缓爱德华氏菌(ETY)兔抗O血清可与大部分鱼源迟缓爱德华氏菌菌体发生凝集反应,不与人源迟缓爱德华氏菌(ATCC15947)菌体发生凝集反应,人源迟缓爱德华氏菌(ATCC15947)兔抗O血清与大部分的鱼源迟缓爱德华氏菌菌体不发生凝集反应,说明鱼源和人源的迟缓爱德华氏菌存在血清型差异。鲶鱼爱德华氏菌兔抗O血清不与迟缓爱德华氏菌菌体发生凝集反应,说明爱德华氏菌种间差异明显。
16S rRNA由于高度保守、易于分析,常用于细菌的分子鉴定与分型,以补充常规细菌鉴定或分类的不足。蒋德明等[16]通过对15株粘细菌的16S rRNA序列的系统进化关系的分析表明,在形态上差异较小的菌株,在16S rRNA上的同源性差异却较大,最大差异达到了4.4%;另外根据形态归为同一个属不同种的菌株,它们的16S rRNA同源性却在99%以上。目前一些重要的水产致病菌例如创伤弧菌、副溶血弧菌、嗜水气单胞菌、假单胞菌等也常用16S rRNA序列的聚类分析来进行分子鉴定和分型[17-18]。热休克蛋白hsp60是另一类高度保守的持家基因,同时具有比16S rRNA更丰富的多变性,已被证明在分类学上的分辨率鉴定及近缘细菌间的系统发育分析优于16S rRNA[19]。因此学者们通常联合16S rRNA及hsp60聚类结果,进行细菌的亲缘进化关系及分类分析[20-21]。为进一步对标本爱德华氏菌进行分子系统进化性的分析,本研究克隆并测序标本迟缓爱德华氏菌的16S rRNA与热休克蛋白基因hsp60部分基因序列,构建了系统进化树。
基于16S rRNA及hsp60部分基因序列构建的系统进化树结果一致显示,绝大部分鱼源迟缓爱德华氏菌分离株进化同源性较高。人源的迟缓爱德华氏菌分离株ATCC15947与以上鱼源迟缓爱德华氏菌有一定的进化距离,但与来源于福建的鱼源迟缓爱德华氏菌AL60306NA1(16S rRNA系统进化树中)或与来源于广东的鱼源迟缓爱德华氏菌E29L(hsp60系统进化树中)相聚,说明不同种属来源的迟缓爱德华氏菌存在一定的亲缘关系。同时,AL60306NA1与E29L在菌体-血清凝集试验中显示不与3种血清中的任何一种凝集,说明AL60306NA1与E29L血清型与其他鱼源迟缓爱德华氏菌不同。
另外,在16SrRNA系统进化树中,来源于广东的鱼源迟缓爱德华氏菌GK4、E29L及来源于福建的鱼源迟缓爱德华氏菌AL60306NA1与人源迟缓爱德华氏菌ATCC15947聚类;在hsp60系统进化树中,GK4、E29L、AL60306NA1显示与人源迟缓爱德华氏菌ATCC15947及人源鲶鱼爱德华氏菌EIV聚类。但在菌体-血清凝集实验中,E29L与AL60306NA1均不与3种抗O血清反应,而GK4与迟缓爱德华氏菌ETY的抗O血清有凝集反应。以上结果说明作为人兽共患菌株,爱德华氏菌菌株可能通过食物链等在水产动物与人之间交叉传染[22-23]。同时说明迟缓爱德华氏菌的血清分型与分子系统进化分析结果不具有绝对的相关性。这提示要更好地防控爱德华氏菌,不仅要进行分子层面的聚类分析,还应结合对菌体表面抗原的免疫原性等进行必要分析。
利益冲突:无
引用本文格式:李素一,池洪树,陈斌,等.我国华东与华南地区养殖鱼类迟缓爱德华氏菌分离株的多样性分析[J].中国人兽共患病学报,2024,40(2):147-153. DOI:10.3969/j.issn.1002-2694.2024.00.022
