珍稀濒危植物越南小花金花茶的叶绿体基因组特征分析
2024-03-10邓永彪张进蓝伦礼赵博
邓永彪 张进 蓝伦礼 赵博


摘 要: 越南小花金花茶(Camellia minima) 是一种珍稀濒危的金花茶组植物,目前尚未有其叶绿体基因组的相关研究。該研究利用Illumina HiSeq 2000平台对越南小花金花茶进行了叶绿体基因组序列的测序、组装、注释和分析。结果表明:(1) 越南小花金花茶叶绿体基因组全长156 961 bp,为典型的四分体结构,共注释到136个基因,其中包含87个蛋白编码基因、41个tRNA基因和8个rRNA基因。(2) 分析鉴定出66个SSR位点和39个重复序列。(3) 密码子偏好使用以A/U结尾的密码子,综合有效密码数(ENC)绘图、PR2-plot和中性分析推测自然选择是影响密码子使用模式的主导因素。(4) 边界分析显示ycf1基因的长度和位置在不同金花茶组植物间存在差异。(5) 对已发表的金花茶组植物叶绿体基因组的系统发育分析发现越南小花金花茶与小花金花茶聚为一支,亲缘关系最近。该研究结果为探索物种进化和提高外源基因表达提供了重要的参考信息,同时为后续的金花茶组植物的保护和利用提供了理论基础。
关键词: 越南小花金花茶, 叶绿体基因组, 特征分析, 系统发育分析, 密码子偏好性
中图分类号: Q943
文献标识码: A
文章编号: 1000-3142(2024)01-0030-13
Analysis of chloroplast genome features of endangered
and rare plant Camellia minima
DENG Yongbiao, ZHANG Jin, LAN Lunli, ZHAO Bo*
( Pharmacy School, Guilin Medical University, Guilin 541199, Guangxi, China )
Abstract: Camellia minima, a rare and endangered species of sect. Chrysantha, has not been previously explored in terms of its chloroplast genome. Utilizing the Illumina HiSeq 2000 platform, the chloroplast genome sequence of C. minima was sequenced, assembled, annotated, and analysed. The results were as follows: (1) The chloroplast genome of C. minima was 156 961 bp in length, embodied a typical tetrad structure, and contained 136 annotated genes, including 87 protein-coding genes, 41 tRNA genes, and 8 rRNA genes. (2) The analysis identified 66 SSR loci and 39 repetitive sequences. (3) Codons prefered to use codons ending in A/U. Comprehensive effective number of codons (ENC) mapping, PR2-plot, and neutral analyses suggested natural selection as a primary factor shaping codon usage patterns. (4) Boundary analysis showed variation in the length and position of the ycf1 gene among different species of yellow Camellia. (5) Phylogenetic analysis of the chloroplast genomes of published sect. Chrysantha species revealed that C. minima was most closely related to C. micrantha. This study provides crucial references for exploring species evolution and enhancing exogenous gene expression, establishing a theoretical foundation for the conservation and utilization of species of sect. Chrysantha in the future.
Key words: Camellia minima, chloroplast genome, characteristic analysis, phylogenetic analysis, codon preference
金花茶组(Sect. Chrysantha )隶属于山茶科(Theaceae)山茶属(Camellia L.),主要分布在我国的广西、云南以及越南部分地区(赛璇,2018)。其生长环境为人类活动踪迹较少的喀斯特地貌和潮湿山地等(李桂娥等,2022)。所有金花茶组植物都被列入我国国家重点保护野生植物名录,保护等级为二级(国家林业和草原局 农业农村部,2021)。金花茶组植物被称为“植物界的大熊猫”“茶中皇后”,因为该组植物是山茶属唯一拥有金黄色花瓣的类群(张蕾等,2019; 吴丽君等,2020)。金花茶组植物的花瓣和叶片中含有丰富的黄酮,常被用作保健食品与饮品(刘青等,2021; 赛璇,2018)。金花茶植物由于频繁的种间杂交和多倍化导致其分类研究十分困难(Zhang et al., 2019)。Wei等(2022)基于双酶切位点相关的简化基因组测序(ddRAD)、转录组和核糖体内部转录间隔区(nrITS)等数据对20个中国金花茶物种进行系统发育重建,结合形态学证据确定了物种之间的亲缘关系。研究表明,强烈的杂交/渗入信号表明网状进化是造成核基因数据和叶绿体基因组数据构建的系统发育树不一致的主要原因。本研究利用基因内容较少的叶绿体基因组进行有关信息的研究,更多完整的叶绿体基因组数据的积累将为今后金花茶属植物的系统发育和网状进化关系的研究提供更多的分子证据,也可以为金花茶组植物的保护与综合利用提供基础理论支持。
四分体结构是叶绿体基因组的典型特征,由一个大单拷贝(large single-copy, LSC)区、一个小单拷贝(small single-copy, SSC)區和两个倒置重复序列(inverted repeats, IRs)显示和编码110~130个基因,大小范围为120~180 kb(Li DM et al., 2021)。相较于核基因组,叶绿体基因组在基因结构、基因内容、基因序列方面更加稳定,较慢的进化速率,因此被广泛应用于系统发育、DNA条形码、基因工程和种群间亲缘关系等研究中(Dong et al., 2018)。近年来叶绿体基因组测序的成本大大降低,越来越多的叶绿体基因组数据被成功应用于植物系统发育和进化研究。同时,叶绿体基因组中包含大量的重复序列是研究物种进化进程以及遗传特征的重要依据(Hui et al., 2014),其中简单重复序列(SSRs)又称微卫星序列可以作为有效的分子标记来检测种群多态性,广泛应用于分子辅助育种和物种保护等方面 (Cavalier, 2002)。密码子是连接核酸和蛋白质的纽带,在遗传信息的传递过程中具有重要作用(Liu et al., 2012)。研究物种密码子使用模式并确定最优密码子,有助于设计基因表达载体来提高目的基因的表达量,在品种改良方面具有重要应用价值(Qi et al., 2015; 胡晓艳等,2019)。
越南小花金花茶(C. minima)为金花茶组植物之一,可做观赏植物和嫁接砧木(George & Anthony, 2015)。目前,尚未有关于越南小花金花茶叶绿体基因组的相关研究,限制了对其遗传特征和系统发育关系的理解。为此,本研究利用高通量测序技术对越南小花金花茶进行了叶绿体全基因组测序组装,拟探讨以下科学问题:(1) 越南小花金花茶叶绿体基因组图谱和序列特征;(2)越南小花金花茶叶绿体基因组密码子使用的偏好性及影响密码子使用模式主导因素;(3)越南小花金花茶与其近缘物种在IR和SC边界区域碱基分布的差异;(4)越南小花金花茶在金花茶组植物系统发育中的位置。本研究不仅为后续开展越南小花金花茶物种鉴定、遗传多样性分析、叶绿体基因工程以及分子育种等研究提供了重要的理论基础,也为金花茶组植物的系统发育研究提供了丰富的叶绿体基因组数据。
1 材料与方法
1.1 植物材料
越南小花金花茶采集于中国广西壮族自治区南宁市金花茶公园(108°21′43″ E、 22°49′30″ N),采集单株新鲜幼嫩的叶片,装进有硅胶的自封袋内低温保存,用于后续提取总DNA,标本保存在广西壮族自治区中国科学院广西植物研究所标本馆,标本凭证为CSF2020003。
1.2 DNA提取和测序
使用改良的CTAB法从越南小花金花茶干燥的叶片中提取总DNA,叶绿体基因组测序服务由北京格致博雅生物科技有限公司提供。使用Fastp软件处理测序获得的原始数据,除去过多的N序列、接头序列和过短序列,输出大小为1 005.18 Mb的clean data,用于后续的叶绿体基因组组装(Gu et al., 2018)。
1.3 基因组组装注释和序列表征
使用getOrganelle v1.7.6.1组装clean data,参考数据库为embplant_pt(陆生植物叶绿体),最大扩充循环数为20,调用Spedas的K-mer值为21、45、65、85、105、127;拼接完成后用Bandage 软件可视化(Jian et al., 2018)。使用叶绿体基因组在线注释网站CPGAVAS2 (http://47.96.249.172:16019/analyzer/home),以C. parvipetala (NC_067089.1)为参照基因组进行注释。使用GB2sequin检查序列分区是否存在颠倒并手动调整准确位置,得到完整的注释结果(Pascal & Stephan, 2018; Shi et al., 2019)。最后使用在线网站CPGview (http://www.1kmpg.cn/cpgview/)绘制了叶绿体基因组圈图(Liu et al., 2023)。注释完成的叶绿体基因组提交至GenBank数据库,登录号为NC_069310.1,相应的SRA、BioProject和BioSample编号分别为SRR20648317、PRJNA861872和SAMN29930849。
1.4 密码子使用偏好性分析
1.4.1 密码子相关参数计算[HTSS] 在剔除重复基因、含有终止密码子和长度小于300 bp的编码序列(coding sequence, CDS)后,选取52条CDS序列进行密码子分析。使用Codon W 1.4.2软件对相对同义密码子使用度(relative synonymous codon usage,RSCU)进行分析。利用EMBOSS工具包中的CUSP程序分析总GC含量以及第1、第2和第3个碱基位置(GC1、GC2、GC3)的GC含量,CHIPS程序用于分析有效密码子数(effective number of codons,ENC)(朱伟垚等,2022)。采用R软件中的cor()函数计算相关性。
1.4.2 密码子中性绘图、ENC-plot及PR2-plot绘图[HTSS] 在中性绘图分析中,以GC12和GC3做散点图,研究密码子3个位置碱基之间的相关性,从而分析突变压力和自然选择对于密码子偏好性的影响(Wei et al., 2014)。GC12为GC1和GC2的平均值。如果GC12和GC3之间显著相关,即R2越大且趋于1时,GC12与GC3的相关程度越高,表明突变压力是密码子使用偏向的决定因素;相反,如果相关性不显著,则回归曲线斜率偏低甚至接近于零,表明密码子偏好性由自然选择主导(Sueoka, 1988)。
ENC-plot绘图进一步分析了密码子偏好性受碱基组成对的影响,以GC3为横坐标、有效密码子数为纵坐标进行绘制散点图,根据下式计算:ENC =2+ GC3+29/ [GC32+(1-GC3)2] (杨祥燕等,2021;辛雅萱等,2021)。当密码子偏好性仅受突变影响时,基因将沿着或接近标准曲线分布,而当基因落在标准曲线以下时则说明自然选择等因素是影响密码子偏好性的主要因素(Chakraborty et al., 2020)。
奇偶偏好性分析(parity rule 2 plot, PR2-plot)用于计算各密码子第3位A、T、C、G的含量,通常用于估计突变压力和自然选择对密码子偏好性的影响(Xiang et al., 2015)。以A3 /(A3+T3)为y轴,G3 /(G3+C3)为x轴绘制PR2-plot图。中心点(A=T, G=C)意味着自然选择和突变压力之间没有偏差。如果基因均匀分布在中心点周围,则认为密码子偏好性可能完全是由突变压力造成的,否则,密码子的使用可能受到自然选择和其他因素的影响(Xiang et al., 2015)。
1.5 重复序列分析
使用在线网站REPuter对散在重复序列进行计算。参数设置:最大计算重复次数=200; 最小重复序列>30 bp;重复序列相似度>90%;汉明距离(Hamming Distance)= 3 (Kurtz et al., 2001)。简单重复序列使用在线网站MISA进行鉴定和统计分析。各重复单元(unit size) 对应的最少重复次数分别为1~10、2~5、3~4、4~3、5~3、6~3,相邻SSR之间的最小单位间隔距离设置为100 bp (Beier et al., 2017)。
1.6 叶绿体基因组IR边界分析
基于系统发育分析的结果,以簇蕊金花茶(C. fascicularis)叶绿体基因组为参考,使用本地部署的IRscope对越南小花金花茶及其近缘物种叶绿体基因组的LSC/IRb/SSC/IRa边界进行可视化分析(Amiryousefi et al., 2018)。
1.7 系统发育分析
将测序组装得到的越南小花金花茶以及从NCBI数据库中获得的23个为金花茶组植物的叶绿体基因组全长序列,以Polyspora penangensis为外类群,构建系统发育树。具体流程:使用MAFFT(version 7.505)进行多序列比对,使用trimAI(V1.4)去除多序列比对结果中的低质量区域,以提高比对结果的质量和准确性;使用IQ-TREE2软件在TVM+F+I+I+R6模型下,以最大似然法(Maximum Likelihood, ML)构建系统发育树(Katoh & Standley, 2013; Minh et al., 2021)。
2 结果与分析
2.1 叶绿体基因组特征
越南小花金花茶叶绿体基因组包含136个功能基因(表1),包括87个蛋白质编码基因,41个tRNA基因和8个rRNA基因,在这些基因中,与自我复制相关的基因有78个,与光合作用有关的基因有45个,其他功能未知的基因共有13个。基因组中有16个双拷贝基因 (ndhB、rpl2、rpl23、rps7、rps12、rrn4.5S、rrn5S、rrn16S、rrn23S、trnI-CAU、trnL-CAA、trnN-GUU、trnR-ACG、trnV-GAC、ycf2、ycf15), 2个四拷贝基因(trnI-GAU, trnA-UGC)。在越南小花金花茶的叶绿体基因组中共有10个基因含有内含子,分别是petB、petD、rps16、rpl16、rpl12、atpF、rpoC1、clpP、ndhB、ndhA,其中rpl12和ndhB基因都含有2个内含子。
越南小花金花茶叶绿体基因组为环状双链四分体结构(图1),长度为156 961 bp,其中有两个长度为26 047 bp的反向重复(IR)区域被一个86 600 bp的大单拷贝(LSC)区域和一个18 267 bp的小单拷贝(SSC)区域分开。叶绿体基因组的GC总含量为37.32%,其中LSC区为35.33%,SSC区为30.60%,IR区为42.98%。
2.2 重复序列分析
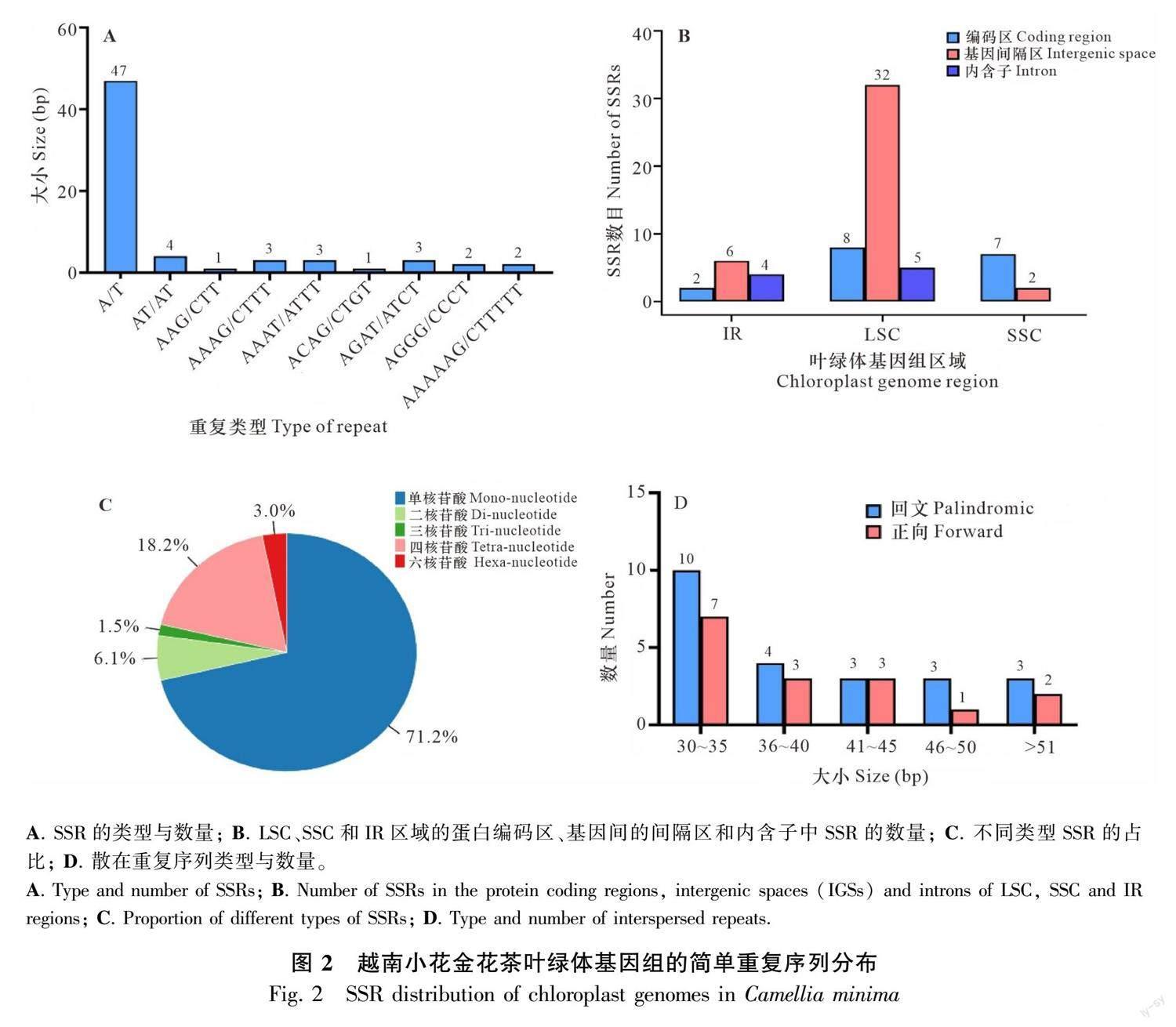
通过MISA在线网站分析,发现越南小花金花茶的叶绿体基因组中含有的SSR位点数目为66个,类型包括单核苷酸、二核苷酸、三核苷酸、四核苷酸和六核苷酸且大多数为单核苷酸重复(图2:A,C)。这些类型的SSR有17个分布在蛋白编码区、40个分布在基因间隔区、9个分布在内含子区(图2:B)。通过REPuter软件分析了越南小花金花茶叶绿体基因组中的散在重复序列,共检测到39个散在重复序列,分别是16个正向重复序列(forward)和23个回文重复序列(palindromic),但没有检测到反向(reverse)和互补(complement)重复序列的存在。这些散在重复序列的长度在30~64 bp之间,且大部分位于IR区的ycf2基因中(图2:D)。
2.3 密码子偏好性分析
2.3.1 同义密码子相对使用度分析[HTSS] 对越南小花金花茶叶绿体基因组的52个大于300 bp的蛋白质编码序列进行密码子偏好性分析发现,其中相对同义密码子使用度(RSCU)值大于1的密碼子有30个,其中13个以A (30个中13个)或U (30个中16个)结尾,以G结尾的数目仅有1个;RSCU值小于1的32个密码子多以C (32个中16个)或G (32个中13个)结尾 ,表明密码子偏好使用以A/U结尾的密码子(图3)。
2.3.2 密码子中性绘图、ENc-plot及PR2-plot分析[HTSS] 对越南小花金花茶叶绿体基因进行中性图(GC12 vs GC3)分析。由图4:A可知,叶绿体基因分布相对集中,但并不密集。GC12和GC3的平均值分别为43.10%和27.49%。GC12和GC3的相关系数为r=0.118(R2=0.014),回归曲线斜率为0.107,表明GC12与GC3无显著相关性。由此推断密码子使用偏好性受突变压力影响较小,而自然选择等因素的贡献更大。
为了评估越南金花茶叶绿体基因组中蛋白编码基因的密码子偏好程度,计算并分析了ENC值。如图4:B所示,大部分基因的ENC值低于预期值,位于标准曲线下方,表明密码子使用偏好性主要受自然选择等因素的影响,而突变压力仅起次要作用。为了进一步反映差异,分析了越南小花金花茶叶绿体基因的ENC频数分布(表2)。ENC比值在-0.05~0.45之间,其中9个(17.00% )基因分布在-0.05~0.05范围内,表明这部分基因分布在标准曲线周围,而剩余的大部分基因都距离标准曲线较远,以上结果说明了自然选择对越南小花金花茶叶绿体基因组密码子偏好的影响更大。
如果密码子偏好性完全受突变影响,则密码子第3位核苷酸A、T、C、G的使用频率相同。在本研究中,PR2平面的4个区域并非均匀分布。由图4:C可知,大部分基因位于平面的下半部分,尤其是右下方。因此,从碱基的使用频率来看,T>A,G>C,表明叶绿体基因组中A/T和G/C的密码子使用偏好性不平衡,推断越南小花金花茶叶绿体基因组的密码子偏好性同时受到突变压力和自然选择等因素的影响。
2.4 IR边界分析
叶绿体基因组在LSC、IRb、IRa和SSC之间有4个边界,即LSC/IRb边界、IRb/SSC边界、SSC/IRa边界和IRa/LSC边界。本研究对越南小花金花茶及6个近缘金花茶组植物的叶绿体基因组边界进行了比较分析。由图5可知,在所有比较的叶绿体基因组中,IR區仅有轻微的扩张和收缩,长度从25 996 bp(四季花金花茶)到26 096 bp(毛瓣金花茶)。在LSC/IRb边界处,所有物种的rps19基因都跨越了LSC/IRb边界,向IRb区域延伸了46 bp。在IRb/SSC边界处,7个金花茶组植物的边界均位于ycf1假基因和ndhF基因的重叠处,在越南金花茶和毛瓣金花茶中,ndhF基因跨越IRb/SSC边界,分别有39、25 bp位于IRb区域中。在SSC/IRa边界处,ycf1基因跨越该边界,包含在IRa 1 034~1 055 bp区域内。在IRa/LSC边界处,所有物种的边界均位于rps19拷贝基因和trnH基因之间,trnH距离IRa/LSC边界1 bp。
2.5 系统发育分析
基于25种山茶科植物(包括24种金花茶组植物和1种大头茶属物种)叶绿体全基因组序列构建系统发育树,以P. penangensis 作为外类群,利用最大似然法构建系统发育树。由图6可知,24种金花茶组植物主要分为2个分支,即Clade Ⅰ分支和Clade Ⅱ分支。Clade Ⅰ分支中,越南小花金花茶(C. minima)与小花金花茶(C. micrantha)以100%的支持率聚为了一个单支,表明这两个物种的亲缘关系最近。
3 讨论与结论
金花茶组植物均为国家二级保护植物(国家林业和草原局 农业农村部,2021),具有较高的观赏和药用价值。其中,越南小花金花茶是一种原产于越南北部的金花茶组植物,生长于潮湿荫蔽的山谷,具有较强的适应能力,被视为优良的嫁接砧木,近年来受到了园艺界的广泛关注(李桂娥等,2022)。越南小花金花茶叶绿体基因组为典型的环状双链四分体结构,长度为156 961 bp,GC含量为37.32%,叶绿体全基因组注释到了136个基因,包括87个蛋白编码基因,41个转运RNA基因和8个核糖体RNA基因, 共有10个基因含有内含子,其中rpl12和ndhB基因含有2个内含子,与金花茶组其他植物的特征一致,可能与叶绿体基因组的特殊结构和复制方式有关(Henry et al., 2016)。此外,越南小花金花茶的叶绿体基因组长度、基因的类型和排列顺序、GC含量等与山茶属的其他已发表物种 (如山茶、油茶、凹脉金花茶等) 相似(Li et al., 2019)。这表明越南小花金花茶叶绿体基因组的进化可能保守而缓慢(Sophiarani et al., 2019; 丁祥青等,2022a)。
简单重复序列(SSRs)是由DNA链的滑移而产生,在基因组和叶绿体基因组中都有较广分布。与其他中性DNA区域相比,SSRs通常具有更高的突变率(Li et al., 2018; Zhang, 2019)。由于其具有非重组、单倍体和单亲遗传的特性,因此常被用作遗传标记,为植物群体遗传学和生态学及进化研究提供有价值的信息(Aii et al., 1997; Gui et al, 2020)。本研究在越南小花金花茶叶绿体基因组中共鉴定了66个SSR位点,主要位于大单拷贝区(LSC),其中所有的单核苷酸重复都是由A/T组成,类似的结果在金花茶组叶绿体基因组(丁祥青等,2022b)和其他被子植物中均有发现(Hui et al., 2014),可能归因于polyA和polyT相比polyC和polyG具有更高的结构稳定性(Jin et al., 2023)。然而,我们检测到的SSR位点数量与Hui等(2014)的结果存在差异,可能是由于检索 SSR 位点的参数设定不同,导致输出的结果不同。在越南小花金花茶的叶绿体基因组中,cp-SSRs含量丰富,可用于检测群体的遗传多态性。此外,在越南小花金花茶叶绿体基因组中鉴定出的散在重复序列主要以正向和回文重复为主,与抱茎金花茶叶绿体基因组序列特征类似(丁祥青等,2022b)。这些重复序列是重要的遗传资源,在系统发育研究中具有重要作用(Wei et al., 2022)。
密码子使用偏好性对于研究基因的分子进化和外源表达具有重要意义(Li et al., 2022)。本研究首次系统分析了越南小花金花茶叶绿体基因组的密码子使用模式,发现精氨酸、异亮氨酸、甘氨酸是密码子编码的最丰富的氨基酸,由6个密码子编码,相比之下,色氨酸、甲硫氨酸仅有一个密码子编码,叶绿体基因倾向于使用A/U密码子,与耿晓姗等(2022)对金花茶的密码子分析研究结果一致。通过进行中性绘图分析,本研究发现GC12和GC3之间并无显著相关性且基因分布相对集中,表明自然选择是影响密码子偏好的主要因素(Zhang et al., 2012)。进一步结合ENC绘图、PR2-plot分析结果,表明越南小花金花茶的密码子使用偏好性受多个因素影响,包括突变压力、碱基组成和基因长度,其中自然选择是主导影响因素,这与丁祥青等(2023)和李清等(2022)的研究结果一致。自然选择是叶绿体基因组进化的主要驱动力,这一发现加深了我们对越南小花金花茶进化历史的理解,特别是与自然选择有关的进化历史。对密码子偏好性的分析结果有利于密码子优化,可为今后金花茶组植物转基因技术提供理论依据(段义忠和张凯,2020; Zhao et al., 2016)。
在叶绿体基因组中,IR的收缩和扩张时常发生,这些差异会导致假基因的产生、基因重复和基因缺失,进一步造成IR/SC连接处的位置变化,高等植物叶绿体基因组长度变异就是由这种位置变化而造成(Li DM et al., 2021)。本研究对越南小花金花茶及6个近缘金花茶组植物的叶绿体基因组边界进行比较分析,结果表明7个物种IR区长度(25 996~26 096 bp)基本一致,未检测到基因丢失,表明IR区的高度保守可能对于维持其长度和结构稳定至关重要。通过边界收缩和扩张分析,本研究发现ycf1基因的长度和位置在不同金花茶组植物间存在差异,可能是潜在的突变热点区域。Li L等(2021)研究表明,ycf1基因因其高度的多态性而被推荐作为植物的核心DNA条形码,然而这一潜在的高变区是否可以作为金花茶组植物有效的DNA条形码还需要进一步的验证。此外,越南小花金花茶及6个近缘金花茶组植物的叶绿体基因组边界相对保守,表明其亲缘关系较近,之后的系统发育研究也支持这一推论。
迄今为止,许多学者已经采用诸如随机扩增多态性DNA (random amplified polymorphic DNA, RAPD )、叶绿体DNA trnL-trnF、扩增片段长度多态性(amplified fragment length polymorphism, AFLP)、ISSR以及核糖體内转录间隔区(nuclear ribsomal internal transcribe spacer, nrITS )等多样化的分子标记,以阐明山茶属之间的亲缘关系(Ju et al., 2021)。 Wei等(2022)整合了ddRAD、转录组、nrITS和SSC等分子标记研究金花茶组系统发育关系,但基于叶绿体基因组SSC区的系统发育树支持率极低。本研究基于叶绿体全基因组序列,对越南小花金花茶和其余23种金花茶组植物的系统发育关系进行了分析。结果表明,依据系统发育的结果可将系统发育树分为两个大的分支,其中越南小花金花茶与小花金花茶聚为高支持率的分支,亲缘关系最近。各分支支持率较高,表明完整的叶绿体基因组数据可为重建金花茶组系统发育关系提供重要的数据支持。
综上所述,本文首次完成了越南小花金花茶叶绿体基因组的序列测序、组装注释和基础信息分析,揭示了越南小花金花茶叶绿体基因组基本特征。在此基础上,通过剖析密码子使用模式以及影响密码子偏好的各类因素,确定了越南小花金花茶叶绿体基因组的高频密码子,为后续山茶属金花茶组植物的转基因研究提供了理论支持。此外,基于全叶绿体基因组构建的系统发育树明确了越南小花金花茶在金花茶组植物中的系统位置。本研究为后续越南小花金花茶和其他金花茶组植物的保护和合理开发利用研究提供了理论基础。
参考文献:
AI I J, KISHIMA Y, MIKAMI T, et al., 1997. Expansion of the IR in the chloroplast genomes of buckwheat species is due to incorporation of an SSC sequence that could be mediated by an inversion [J]. Curr Genet, 31: 276-279.
AMIRYOUSEFI A, HYVNEN J, POCZAI P, 2018. IRscope: an online program to visualize the junction sites of chloroplast genomes [J]. Bioinformatics, 34(17): 3030-3031.
BEIER S, THIEL T, MNCH T, et al., 2017. MISA-web: a web server for microsatellite prediction [J]. Bioinformatics, 33(16): 2583-2585.
CAVALIER ST, 2002. Chloroplast evolution:secondary symbiogenesis and multiple losses [J]. Curr Biol, 12: 62-64.
CHAKRABORTY S, YENGKHOM S, UDDIN A, 2020. Analysis of codon usage bias of chloroplast genes in Oryza species [J]. Planta, 252: 1-20.
DING XQ, CHEN SY, CHEN JT, et al., 2023. Codon bias analysis of 11 yellow Camellia chloroplast genome [J]. J Fujian Agric For Univ (Nat Sci Ed), 52(4): 1-9. [丁祥青, 陈丝雨, 陈佳婷, 等, 2023. 11种金花茶叶绿体基因组密码子偏好性分析 [J]. 福建农林大学学报(自然科学版), 52(4): 1-9.]
DING XQ, LI WF, WU JL, et al., 2022a. Chloroplast genome characteristics and genetic relationship of yellow Camellia [J]. J Fujian Agric For Univ (Nat Sci Ed), 52(3): 1-11. [丁祥青, 李文芳, 吴丽君, 等, 2022a. 4种金花茶叶绿体基因组的比较分析 [J]. 福建农林大学学报(自然科学版), 52(3): 1-11.]
DING XQ, BI YY, CHEN JT, et al., 2022b. Analysis of the chloroplast genome characteristics of Camellia tienii [J]. Agric Sci Jiangsu, 50(23): 33-40. [丁祥青, 畢远洋, 陈佳婷, 等, 2022b. 抱茎金花茶(Camellia tienii)的叶绿体基因组特征分析 [J]. 江苏农业科学, 50(23): 33-40.]
DONG W, XU C, WU P, et al., 2018. Resolving the systematic positions of enigmatic taxa: Manipulating the chloroplast genome data of Saxifragales [J]. Mol Phylogenet Evol, 126: 321-330.
DUAN YZ, ZHANG K, 2020. Comparative analysis and phylogenetic evolution of the complete chloroplast genome of Ammopiptanthus [J]. Acta Bot Boreal-Occident Sin, 40(8): 1323-1332. [段义忠, 张凯, 2020. 沙冬青属植物叶绿体基因组对比和系统发育分析 [J]. 西北植物学报, 40(8): 1323-1332.]
GENG XS, JIA W, CHEN JN, et al., 2022. Codon usage bias analysis of chloroplast genome in Camellia nitidissima [J]. Mol Plant Breed, 20(7): 2196-2203. [耿晓姗, 贾魏, 陈佳宁, 等, 2022. 金花茶叶绿体基因组密码子偏好性分析 [J]. 分子植物育种, 20(7): 2196-2203.]
GEORGE O, ANTHONY SC, 2015. In pursuit of hidden camellias 32 new Camellia species from Vietnam and China [M]. 2nd ed. Sydney: Theaceae Exploration Associates.
GUI L, JIANG S, XIE D, et al., 2020. Analysis of complete chloroplast genomes of curcuma and the contribution to phylogeny and adaptive evolution [J]. Gene, 732: 144355.
GU J, CHEN S, ZHOU Y, et al., 2018. Fastp: an ultra-fast all-in-one FASTQ preprocessor [J]. Bioinformatics, 34(17): i884-i890.
HENRY D, LIN CS, YU M,et al., 2016. Chloroplast genomes: diversity, evolution, and applications in genetic engineering [J]. Genome Biol, 17(134): 1-29.
HU XY, XU YQ, HAN YZ, et al., 2019. Codon usage bias analysis of the chloroplast genome of Ziziphus jujuba var. spinosa [J]. J For Environ, 39(6): 621-628. [胡晓艳, 许艳秋, 韩有志, 等, 2019. 酸枣叶绿体基因组密码子使用偏性分析 [J]. 森林与环境学报, 39(6): 621-628.]
HUI H, CHAO S, YUAN L, et al., 2014. Thirteen Camellia chloroplast genome sequences determined by high-throughput sequencing: genome structure and phylogenetic relationships [J]. BMC Evol Biol, 14(1): 151-168.
JIAN JJ, YU WB, YANG JB, et al., 2018. GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes [J]. Genome Biol, 21(1): 241.
JIN GZ, LI WJ, SONG F, et al., 2023. Comparative analysis of complete Artemisia subgenus Seriphidium (Asteraceae: Anthemideae) chloroplast genomes: insights into structural divergence and phylogenetic relationships [J]. BMC Plant Biol, 136: l-23.
JU NG, HOANG DKD, KIM CK, et al., 2021. Complete chloroplast genomes shed light on phylogenetic relationships, divergence time, and biogeography of Allioideae (Amaryllidaceae) [J]. Sci Rep, 11(1): 3262
KATOH K, STANDLEY DM, 2013. MAFFT mutiple sequence alignment software version 7:improvements in performance and usability [J]. Mol Biol Evol, 30(4): 772-780.
KURTZ JV, CHOUDHURI, OHLEBUSCH E, et al., 2001. REPuter: the manifold applications of repeat analysis on a genomic scale [J]. Nucl Acids Res, 29(22): 4633-4642.
LI DM, LI J, WANG DR, et al., 2021. Molecular evolution of chloroplast genomes in subfamily Zingiberoideae (Zingiberaceae) [J]. BMC Plant Biol, 21: 1-24.
LI GE, JIANG CJ, QI Y, et al., 2022. Morphological characteristics and identification points of Camellia minima and Camellia cucphuongensis from Vietnam [J]. S Hortic, 33(1): 54-60. [李桂娥, 蔣昌杰, 漆娅, 等, 2022. 越南小花金花茶和菊芳金花茶的形态特征及识别要点 [J]. 南方园艺, 33(1): 54-60.]
LI L, HU YF, HE M, et al., 2021. Comparative chloroplast genomes: insightsinto the evolution of the chloroplast genome of Camellia sinensis and the phylogeny of Camellia [J]. BMC Genomics, 22(138): 471-493.
LI Q, LUO RJ, GE R, et al., 2022. Analysis on codon usage bias of chloroplast genomein Ampelopsis grossedentata [J]. Guangdong Agric Sci, 49(11): 162-169. [李清, 罗永坚, 葛蓉, 等, 2022. 显齿蛇葡萄叶绿体基因组密码子使用偏好性分析 [J]. 广东农业科学, 49(11): 162-169.]
LI W, ZHANG CP, GUO XP, et al., 2019. Complete chloroplast genome of Camellia japonica genome structures, comparative andphylogenetic analysis [J]. PLoS ONE, 14(5): e0216645.
LI XP, MENG J, ZHANG N, et al., 2018. Comparative analysis of chloroplast genomes of Aconitum vilmorinianum and Aconitum vilmorinianum var. patentipilum [J]. J Chin Med Mat, 41(8): 1812-1820. [李雪佩, 孟静, 张琳娜, 等, 2018. 黄草乌与展毛黄草乌叶绿体全基因组结构的比较分析 [J]. 中药材, 41(8): 1812-1820.]
LIU H, HUANG Y, DU X, et al., 2012. Patterns of synonymous codon usage bias in the model grass Brachypodium distachyon [J]. Genet Mol Res, 11(4): 4695-4706.
LIU Q, LI Y, YANG RM, et al., 2021. Yellow Camellia: resource status and research progress in modern studies [J]. Mod Chin Med, 23(4): 727-733. [刘青, 李月, 杨润梅, 等, 2021. 金花茶组植物资源现状与现代研究进展 [J]. 中国现代中药, 23(4): 727-733.]
LIU S, NI Y, LI J, et al., 2023. CPGView: A package for visualizing detailed chloroplast genome structures [J]. Mol Ecol Resour, 23(3): 694-704.
MINH BQ, SCHMIDT HA, CHERNOMOR O, et al., 2021. IQ-TREE 2: New models and efficient methods for phylogenetic inference in the genomic era [J]. Mol Biol Evol, 37(5): 1530-1534.
National Forestry and Grassland Administration, Ministry of Agriculture and Rural Affairs, 2021. List of National Key Protected Wild Plants [EB/OL]. 2021(15). https://www.gov.cn/zhengce/zhengceku/2021-09/09/content_5636409.htm. [国家林业和草原局 农业农村部, 2021. 国家重点保护野生植物名录 [EB/OL] 2021年第15号. https://www.gov.cn/zhengce/zhengceku/2021-09/09/content_5636409.htm.]
PASCAL L, STEPHAN G, 2018. GB2sequin — A file converter preparing custom GenBank files for database submission [J]. Genomics, 111(4): 759-761.
QI YY, XU WJ, XING T, et al., 2015. Synonymous codon usage bias in the plastid genome is unrelated to gene structure and shows evolutionary heterogeneity [J]. Evol Biol Online, 11: 65-77.
SAI X, 2018. Anti-lung cancer effect of Camellia euphlbia flowers extract and its potential mechanism of action [D]. Dalian: Dalian University of Technology. [赛璇, 2018. 金花茶花提取物的肺癌防治作用及其机理初探 [D]. 大连: 大连理工大学.]
SHI L, CHEN H, JIANG M, et al., 2019. CPGAVAS2, an integrated plastome sequence annotator and analyzer [J]. Nucl Acids Res, 7(W1): W65-W73.
SOPHIARANI Y, ARIF U, SUPRIYO C, 2019. Deciphering codon usage patterns and evolutionary forces in chloroplast genes of Camellia sinensis var. assamica and Camellia sinensis var. sinensis in comparison to Camellia pubicosta [J]. J Inte Agric, 18(12): 2771-2785.
SUEOKA N, 1988. Directional mutation pressure and neutral molecular evolution [J]. Proc Natl Acad Sci USA, 85(8): 2653-2657.
WEI L, HE J, JIA X, et al., 2014. Analysis of codon usage bias of mitochondrial genome in Bombyx mori and its relation to evolution [J]. BMC Evol Biol, 14: 262.
WEI SJ, LIUFU YQ, ZHENG HE, et al., 2022. Using phylogenomics to untangle the taxonomic incongruence of yellow-flowered Camellia species (Theaceae) in China [J]. J Syst Evol, 61(5): 1-16.
WU LJ, ZHENG HC, CHEN WR, et al., 2020. Performance and thinking on introduction and cultivation of Camellia Sect.Chrysantha Chang in Fujian [J]. Fujian For Sci Technol, 47(2): 109-115. [吳丽君, 郑惠成, 陈文荣, 等, 2020. 福建金花茶组植物引种栽培现状与思考 [J]. 福建林业科技, 47(2): 109-115.]
XIANG H, ZHANG RZ, BUTLER RR,et al., 2015. Comparative analysis of codon usage bias patterns in Microsporidian genomes [J]. PLoS ONE, 10: e0129223.
XIN YX, LI RZ, LI X, et al., 2021. Analysis on codon usage bias of chloroplast genome in Mangifera indica [J]. J Centr S Univ For Technol, 41(9): 145-157. [辛雅萱, 黎若竹, 李鑫, 等, 2021. 杧果叶绿体基因组密码子使用偏好性分析 [J]. 中南林业科技大学学报, 41(9): 145-157.]
YANG XY, CAI YB, TAN QL, et al., 2021. Analysis of codon usage bias in the chloroplast genome of Ananas comosus [J]. J Trop Crop, 43(3): 439-446 [杨祥燕, 蔡元保, 谭秦亮, 等, 2021. 菠萝叶绿体基因组密码子偏好性分析 [J]. 热带作物学报, 43(3): 439-446.]
ZHANG L, NI S, LI JY, et al., 2019. Analysis of petal nutrition and bioactive components in different periods of Camellia nitidissima [J]. For Res, 32(2): 32-38. [张蕾, 倪穗, 李纪元, 等, 2019. 金花茶不同时期花瓣营养与生物活性成分分析 [J]. 林业科学研究, 32(2): 32-38.]
ZHANG JW, 2019. The complete chloroplast genome and phylogenetic analysis of the endangered species Syringa pinnatifolia (Oleaceae) [D]. Yangling: Northwest A & F University. [张靖雯, 2019. 濒危植物羽叶丁香叶绿体全基因组及系统发育研究 [D]. 杨凌: 西北农林科技大学.]
ZHANG W, ZHAO Y, YANG G, et al., 2019. Determination of the evolutionary pressure on Camellia oleifera on Hainan Island using the complete chloroplast genome sequence [J]. PeerJ, 7: e7210.
ZHANG Y, NIE X, JIA X, et al., 2012. Analysis of codon usage patterns of the chloroplast genomes in the Poaceae family [J]. Aust J Bot, 60: 461-470.
ZHAO YC, ZHENG H, XU AX, et al., 2016. Analysis of codon usage bias of envelope glycoprotein genes in nuclear polyhedrosis virus (NPV) and its relation to evolution [J]. BMC Genom, 17: 677-677.
ZHU WY, LONG Y, ZHENG S, et al., 2022. Chloroplast genome structure and characterization of Melaleuca bracteata [J/OL]. Mol Plant Breed: 1-14 [2023-08-12]. [朱伟垚, 龙宇, 郑叔召, 等, 2022. 黄金宝树叶绿体基因组测序与特征分析 [J]. 分子植物育种: 1-14 [2023-08-12]. http://ifgfy2b08d79e045e4fd4hpu0ow5n56x966nfnficg.res.gxlib.org.cn/kcms/detail/46.1068.S.20220428.1423.017.html.]
( 責任编辑 李 莉 )
收稿日期: 2023-08-12
基金项目: 国家自然科学基金(32060090); 广西科技基地和人才专项(桂科AD19110089); 广西自然科学基金(2021JJA130119)。
第一作者: 邓永彪(2000-),硕士研究生,研究方向为中药资源,(E-mail)dengyongbiao1@gmail.com。
* 通信作者: 赵博,博士,教授,研究方向为药用植物资源学,(E-mail)122017017@glmc.edu.cn。
