极小种群植物川柿叶绿体基因组特征与系统发育分析
2024-03-10杨楠王奥张子晨赵良成
杨楠 王奥 张子晨 赵良成

摘 要: 川柿(Diospyros sutchuensis)為极小种群和国家重点保护野生植物,分布范围狭窄,种群数量极少。目前,川柿基因组信息缺乏,在柿属(Diospyros)中的系统亲缘关系不明确。该研究通过Illumina平台对川柿叶绿体基因组进行测序,应用Getorganellev1.7.3.4和PGA软件对基因组进行组装和注释,使用DnaSP6.12.03软件进行多序列对比分析,并使用REPuter、Tandem Reapeats Finder和MISA软件进行重复序列分析,使用CodonW1.4和EasyCodemL软件分别进行密码子偏好性和选择压力分析。同时,基于4个不同的叶绿体基因组序列数据集,使用IQtree软件分析川柿与11个柿属物种的系统发育关系。结果表明:(1)川柿叶绿体基因组全长157 917 bp,包含1对26 111 bp的反向重复区、大单拷贝区(87 303 bp)和小单拷贝区(18 392 bp),GC碱基含量为37.4%。(2)川柿叶绿体基因组共注释到113个基因,包括79个蛋白编码基因、30个tRNA基因和4个rRNA基因;共检测到49个长重复序列、27个串联重复序列和34个简单重复序列;蛋白编码基因中高频密码子31个,多数密码子末位碱基为A或U,编码亮氨酸的密码子使用最多;基因组编码区比非编码区更为保守,10个高变热点区域可作为潜在的分子标记;蛋白编码基因中有8个基因(ndhB、ndhG、ndhI、rbcL、rpoB、petB、petD、rps12)受到正选择压力。(3)系统发育分析显示,川柿与老鸦柿(D. rhombifolia)和乌柿(D. cathayensis)亲缘关系最为密切,它们与海南柿(D. hainanensis)共同形成一个单系分支。该研究结果既为川柿及柿属种质资源鉴定、遗传多样性保护以及种群恢复等提供了叶绿体基因组资源,也为阐明川柿的系统进化提供了重要的分子信息。
关键词: 川柿, 叶绿体基因组, 重复序列, 选择压力, 系统发育
中图分类号: Q943
文献标识码: A
文章编号: 1000-3142(2024)01-0015-15
Characteristics of chloroplast genome and phylogenetic
analysis of Diospyros sutchuensis with
extremely small populations
YANG Nan1 , WANG Ao1 , ZHANG Zichen1 , ZHAO Liangcheng2*
( 1. School of Ecology and Nature Conservation, Beijing Forestry University, Beijing 100083, China;
2. Museum of Beijing Forestry University, Beijing 100083, China )
Abstract: Diospyros sutchuensis is a national protected wild plant species with narrow distribution and extremely small population in southwestern China. At present, the genomic information of this species is lacking and its phylogenetic relationships among Diospyros remain unclear. In this study, the chloroplast genome of D. sutchuensis was sequenced by Illumina platform, assembled and annotated by Getorganelle v1.7.3.4 and PGA, and analyzed by DnaSP 6.12.03 for sequence comparison, REPuter, Tandem Reapeats Finder, MISA for repetitive sequences, CodonW1.4 for codon usage bias, and EasyCodemL for selection pressure. Meanwhile, based on four different chloroplast genome sequence datasets, the phylogenetic relationships between D. sutchuensis and 11 Diospyros species were analyzed using IQtree. The results were as follows: (1) The chloroplast genome of D. sutchuensis was 157 917 bp in length, including two inverted repeats (IRs) of 26 111 bp, which was separated by large single copy (LSC) and short single copy (SSC) of 87 303 bp and 18 392 bp, respectively. The GC content was 37.4%. (2) The genome contained 113 genes, including 79 protein-coding genes, 30 tRNA genes, and 4 rRNA genes. A total of 49 long repeats, 27 tandem repeats, and 34 simple sequence repeats were identified. There were 31 high-frequency codons in protein-coding genes, most of them ended in A or U, and the most used codons were the ones encoding leucine. The coding regions were more conserved than the non-coding ones, and 10 regions were identified as most divergent hotspots for potential molecular markers. There had been positive selection on ndhB, ndhG, ndhI, rbcL, rpoB, petB, petD and rps12 among protein-coding genes. (3) Phylogenetic analyses showed that D. sutchuensis was closely related to D. rhombifolia and D. cathayensis. Together with D. hainanensis, these four species formed a monophyletic group. The phylogenetic tree constructed from the chloroplast genomes had the highest support values, indicating that the chloroplast genomes with most variable and informative sites are more suitable for phylogenetic studies of Diospyros. The results provide useful chloroplast genomic resources for germplasm identification, genetic diversity conservation, repopulation, and phylogenetic analysis of D. sutchuensis as well as Diospyros.
Key words: Diospyros sutchuensis, chloroplast genome, repeat sequence, selection pressure, phylogenetic analysis
柿科(Ebenaceae)是一个主要分布于热带和亚热带的木本植物科,包含3~4个属(Wallnfer, 2001; Duangjai et al., 2006)。柿属(Diospyros)是柿科内种类最多、分布最广的属,也是柿科在中国唯一的属。同时,柿属很多种类也是重要的经济树种,在食用、药用、木材、园艺等方面用途广泛(左大勋等, 1984; Rauf et al., 2017)。柿属的系统分类研究主要集中在形态学和孢粉学(Utsunomiya et al., 1998; Venkatasamy et al., 2006; Akinsulire et al., 2018; 胡超琼等, 2018; 邹璞等, 2022)。近年来,有学者陆续对柿属不同类群开展了分子系统学和基因组学研究(Yonemori et al., 2008; Turner et al., 2013; 梁晋军, 2014; 傅建敏等, 2015; 傅建敏等, 2017)。在国内,柿属包含的物种数量一直存有争议,很多种类的系统地位也不明确(左大勋等, 1984; 吴征镒和崔鸿宾, 1987; Li et al., 1996)。Tang等(2019)通过资料分析、标本查阅和实地调查,确认了45种中国特有的柿属种类,其中18种分布极为狭窄,川柿(Diospyros sutchuensis )即为分布范围狭窄的种类之一。
川柿由森林植物学家杨衔晋于1945年在《华西边疆研究学会杂志》上命名发表,模式标本于1941年采自四川盆地东部华蓥山海拔1 300 m的杂木林中(Yang, 1945)。但是,自川柿发表后,几十年来一直未在野外见到。研究人员曾去模式原产地调查,也未采到(方文培, 1985)。2017年,科研人员在重庆市江津区四面山林区再次调查到川柿,目前共发现6株个体,分散在3个分布点(刘燕琴等, 2021a,b)。川柿在野外分布范围狭窄,种群数量极少,生境受威胁严重,根据极小种群野生植物(PSESP)的定义(孙卫邦等, 2015; 杨文忠等, 2015),该种应属于极小种群野生植物。在2021年公布的《国家重点保护野生植物名录》中(http://www.gov.cn/zhengce/zhengceku/2021-09/09/content_5636409.htm),柿科共有2种被列为国家二级保护植物,分别为川柿和小萼柿(D. minutisepala)。目前,仅有刘燕琴等(2021a,b)对川柿的生物学特性和种子萌发进行了初步研究。在柿属已有的系统分类学研究中,由于所涉及的类群均没有包含川柿,因此该物种的系统亲缘关系尚不明确。
叶绿体作为植物细胞特有的细胞器,拥有一套半自主性遗传的基因组。与核基因相比,叶绿体基因组具有高度的保守性以及进化相对独立、基因组小、结构稳定、突变率低等特点,已被越来越多用于植物分子进化、系统发育以及物种鉴定等研究中(Dong et al., 2012; Daniell et al., 2016)。同时,叶绿体基因组还可为重要经济植物及珍稀濒危植物的保育提供分子信息(陆奇丰等, 2021)。在柿属中,目前共报道了11个物种的叶绿体基因组(https://www.ncbi.nlm.nih.gov/)。其中,Fu 等(2016)首次对4种和1品种柿属植物,即柿(Diospyros kaki)、君迁子(D. lotus)、油柿(D. oleifera)、粉叶柿(D. glaucifolia)和金枣柿(Diospyros‘Jinzaoshi)进行了叶绿体基因组测序和系统发育分析,结果显示柿与油柿的亲缘关系较近,而君迁子则与粉叶柿较近。Liu 等(2018)报道了琼岛柿(D. maclurei)和海南柿(D. hainanensis)两个海南特有种的叶绿体基因组序列,发现两者基因组特征相近,但亲缘关系却较远。
本文依托“中国西南地区极小种群野生植物调查与种质保存”项目对重庆市四面山川柿的调查结果,以川柿叶片为研究材料,通过叶绿体基因组高通量测序、组装和基因注释,结合已经报道的其他11种柿属植物叶绿体基因组数据进行系统发育分析,拟探讨:(1)川柿的叶绿体基因组特征,包括基因重复序列、变异位点、密码子偏好性和进化选择压力;(2)川柿与部分柿属种类的系统发育关系以及不同叶绿体基因组序列建立系统发育树的比较。本研究结果有助于增加对川柿系统发生关系的理解,并为后续开展川柿及柿属种质资源鉴定、遗传育种、遗传多样性保护以及种群恢复等研究提供分子信息。
1 材料与方法
1.1 植物材料与DNA提取
川柿的叶片材料采自重庆市江津区四面山大窝铺林场(28°34′34.48″ E、106°21′4.48″ N),用硅胶干燥保存,凭证标本(BJFUZLC022)存放于北京林业大学植物标本馆(BJFC)。利用改良CTAB法(Pahlich & Gerlitz, 1980)提取川柿叶片总DNA,试剂盒采用新型植物基因组DNA提取试剂盒(北京美吉桑格生物医药科技有限公司)。
1.2 叶绿体基因组拼装与注释
叶绿体全基因组的测序分為五步,即基因组DNA质检、DNA片段化、文库构建、桥式PCR、Illumina Hiseq 4000 测序仪进行双端测序。共得到5.44 Gb原始数据(raw data),去除其中测序质量较低的序列后得到clean data,利用Getorganelle v1.7.3.4软件(Jin et al., 2020),以柿的叶绿体全基因组作为参考,对所获得的clean data进行从头拼接。
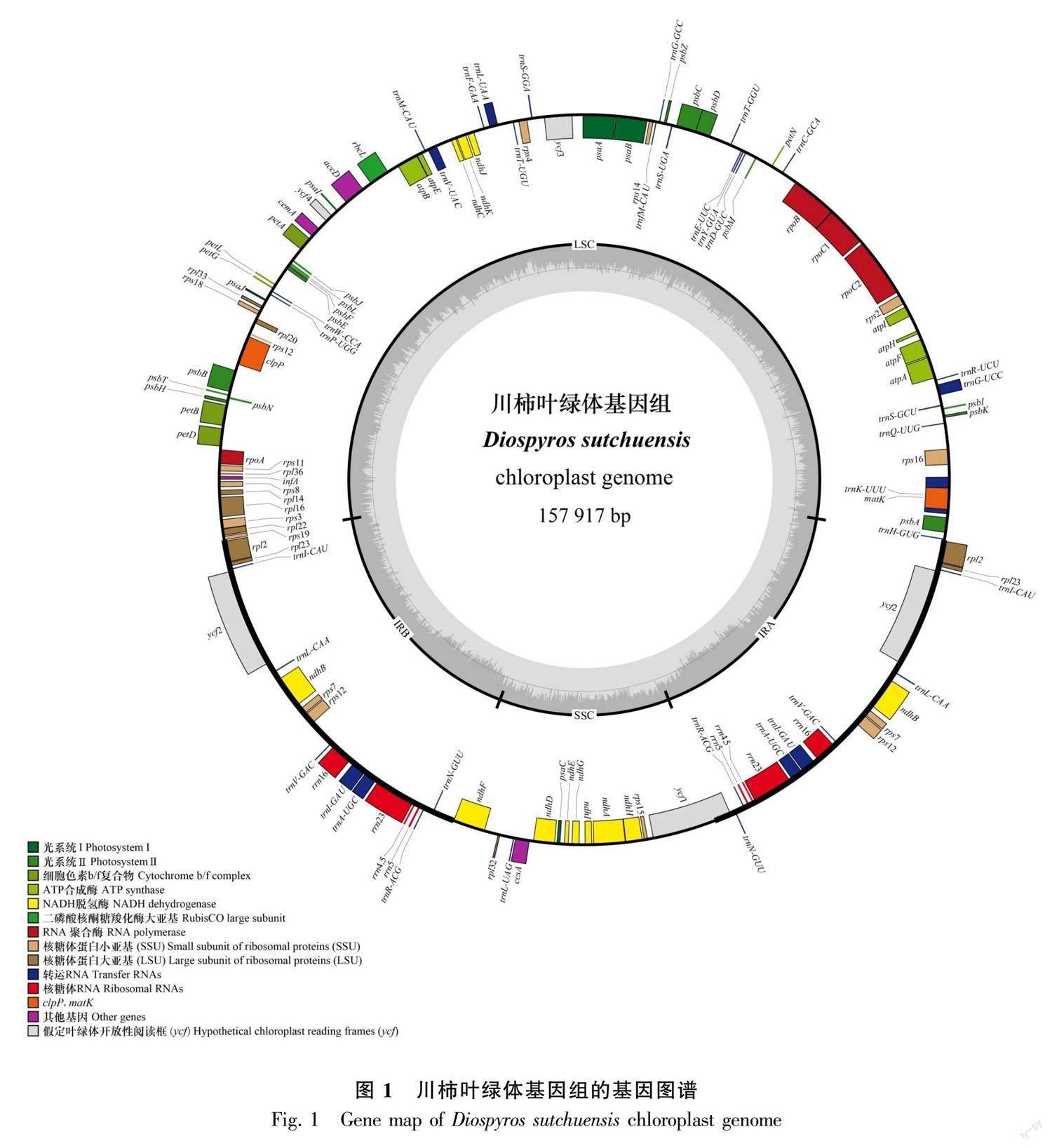
将所获得的川柿叶绿体全基因组序列使用软件PGA(Plastid Genome Annotator)进行注释,参照柿的叶绿体全基因组注释信息,得到注释结果。注释结果经过NCBI中Blast比对,比对正确后提交至GenBank,得到基因序列号OK641590。使用在线网站OG DRAW (https://chlorobox.mpimp-golm.mpg.de /OGDraw.html),参数为默认值,绘制川柿叶绿体全基因组图谱。
1.3 多序列对比分析
使用DnaSP6.12.03软件(Librado & Rozas, 2009)对11个柿属物种的叶绿体基因组进行滑动窗口分析,计算核苷酸多样性(Pi)。步长设定为100 bp,窗口长度为300 bp。使用MAFFT 7软件(Katoh & Standley, 2013)分别对编码区与非编码区进行同源位点比对,计算每段序列的变异位点百分比。
1.4 重复序列与SSR分析
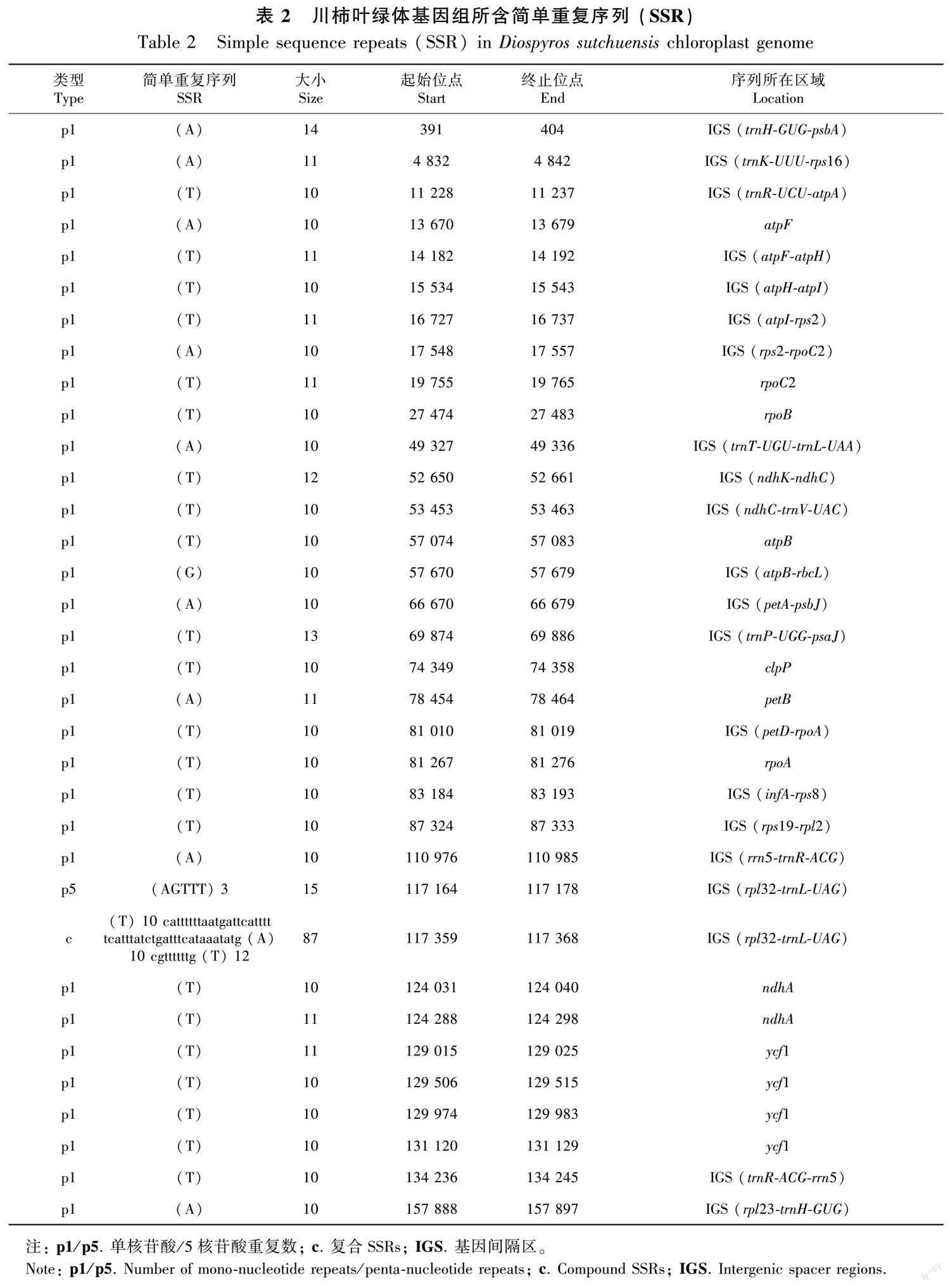
使用REPuter软件(Kurtz et al., 2001)查找川柿叶绿体全基因组中的散在重复序列 (dispersed repeats),海明距离(Hamming distance)设置为3。串联重复序列(tandem repeats)通过Tandem Reapeats Finder(Benson, 1999)进行分析。简单重复序列(simple sequence repeats, SSR)的检测利用MISA软件(Beier et al., 2017),参数设置为单核苷酸重复≥10,二核苷酸重复≥6,三核苷酸重复≥5,四核苷酸重复≥4,五和六核苷酸重复≥3。
1.5 密码子偏好分析
使用CodonW1.4软件分析川柿叶绿体基因组中蛋白编码基因同义密码子的相对使用度(relative synonymous codon usage, RSCU),通过RSCU值判断密码子使用偏好。当RSCU>1时,说明该密码子被高频使用;当RSCU=1时,说明密码子没有使用偏好;当RSCU<1时,说明该密码子使用频率较低。软件参数设置为默认,并根据所获得的参数绘制相关堆积柱形图。
1.6 选择压力分析
选择川柿以及与川柿亲缘关系较近的老鸦柿(D. rhombifolia)、乌柿(D. cathayensis)、海南柿,对其叶绿体全基因组的78个共有蛋白编码基因进行选择压力计算与分析。利用EasyCodemL软件(Gao et al., 2019),选择预置模式(preset mode)与位点模型(site model),对这些基因的非同义突变率(dN)与同义突变率(dS)及其比值(ω=dN/dS)进行计算。为了检验统计的显著性,进行LRT检验(liklihood ratio tests)。此外,选取PamlX 软件(Xu & Yang, 2013)中的YN00模块计算4个物种两两对比后每个蛋白编码基因的dN、dS及ω值,所得结果与EasyCodeml所得结果进行比较,以确认结果的准确性。
1.7 系统发育分析
在NCBI(https://www.ncbi.nlm.nih.gov/)数据库中下载柿属已经过验证的11个物种的叶绿体全基因组序列,包括分布于中国的柿(NC_030789.1)、君迁子(NC_030786.1)、乌柿(NC_039554.1)、粉叶柿(NC_030784.1)、油柿(NC_030787.1)、海南柿(NC_042160.1)、琼岛柿(NC_042161.1)、老鸦柿 (NC_039556.1),以及分布于东南亚的D. blancoi (NC_033502.1)、D. celebica(NC_046040.1)和分布于北美的美洲柿(D. virginiana)(NC_039555.1),与本研究川柿的叶绿体全基因组序列一起进行系统发育分析。由于安息香科与柿科存在较近的亲缘关系,选用中华安息香(Styrax chinensis)(MT648752.1)作为外类群。使用Phylosuite 软件(Zhang et al., 2020)筛选出编码区序列与非编码区序列,并进行序列串联,用于后续系统发育关系分析。
为比较不同基因组序列,本研究对川柿的系统发育关系分析采用了4个不同的数据集:(1)叶绿体全基因组序列;(2)编码区序列;(3)非编码区序列;(4)非编码区20个变异热点区域的一致性序列。使用MEGA X软件(Kumar et al., 2018)对序列进行比对,每个数据集的最适模型选择采用Model-Finder(Kalyaanamoorthy et al., 2017)进行计算。利用IQtree (Nguyen et al., 2015)构建基于最大似然法(maximum-likelihood)的系统发育树。5 000 次自展分析检验,计算各节点的靴带支持率(bootstrap values, BS),同时采用贝叶斯方法(Bayesian inference)(Posada et al., 2004),重复1 000次,通过贝叶斯后验概率(Bayesian posterior probabilities, PP)对系统发育树进行检验。
2 结果与分析
2.1 川柿叶绿体全基因组基本特征
川柿葉绿体全基因组呈现闭合环状四分体结构,包含一个大单拷贝区(LSC)、一个小单拷贝区(SSC)和一对反向重复区IR(IRa、IRb)(图1)。基因组序列全长157 917 bp,其中LSC区、SSC区、IR区长度分别为87 303、18 392、26 111 bp。基因组总GC碱基含量为37.4%,LSC区、SSC区、IR区GC含量分别为35.4%、30.9%、43.0%。
川柿叶绿体全基因组中共注释到113个基因,包括79个蛋白编码基因、30个tRNA基因和4个rRNA基因。IR区重复的基因分别为7个蛋白编码基因(rpl2、rpl23、ycf15、ndhB、rps7、rps12、ycf2),7个tRNA (trnV-GAC、trnI-GAU、trnA-UGC、trnR-ACG、trnN-GUU、trnI-CAU、trnL-CAA)、4个rRNA(rrn16、rrn4.5、rrn23、rrn5)。含有1个内含子的基因有15个(rpl16、rpl2、ndhB、rps16、atpF、rpoC1、trnA-UGC、trnI-GAU、trnK-UUU、trnL-UAA、trnV-UAC、rps12、trnG-UCC、petB、petD),含有2个内含子的基因有2个(ycf3、clpP)。根据基因功能的不同,川柿叶绿体基因组基因可分为与表达有关的基因(59个)、光合作用相关基因(46个)以及其他基因(8个)(表1)。川柿与已报道的其他11个柿属物种叶绿体全基因组特征比较见附表1。
2.2 重复序列和SSR分析
通过鉴定得到川柿叶绿体基因组中的长重复序列共49个,长度在18~30 bp之间,包括正向重复序列(forward)18个、反向重复序列(reverse)7个、回文重复(palindromic)24个,未检测到互补序列(complement)。共检测到串联重复序列27个,长度在4~32 bp之间,其中19个序列重复了2次、3个序列重复了3次、2个序列重复了6次、2个序列重复了4次、1个序列重复了12次、1个序列重复了5次。17个串联重复发生在基因间隔区(IGS)、7个发生在基因ycf2上、1个位于基因ndhF、其余2个分布在基因ndhA的内含子上。在川柿叶绿体基因组共检测到34个简单重复序列SSR,其中33个为单核苷酸重复序列,另外1个为复合SSR(表2)。单核苷酸重复序列的重复单元大部分为碱基A或碱基T,只在LSC区出现了一个G碱基的重复单元。五核苷酸重复序列1个,重复单元为AGTTT。
2.3 序列变异分析
通过计算比较了12个柿属物种叶绿体全基因组序列间的核苷酸多样性 (Pi)。图2结果显示,Pi值区间为0~0.053 59,平均值为0.006 99。各分区中,LSC区Pi值为0~0.053 59,SSC区Pi值0~0.038 63,IR区Pi值为0~0.015 7。LSC区的高变区域(Pi>0.03)包括trnH-GUG-psbA、trnT-UGU-trnL-UAA、petA-psbJ、psbE-petL、ycf2,SSC区的高变区域(Pi>0.02)为ndhF-rpl32、rpl32-trnL-UAG、ccsA-ndhD、ycf1,IR区的高变区域(Pi>0.01)为rpl2。
此外,还分别统计了12个柿属物种叶绿体全基因组、编码区、非编码区,以及LSC区、SSC区、IR区变异位点的数量和百分比。叶绿体全基因组共发现3 980个变异位点,占比2.43%;编码区变异位点1 141个,占比1.23%;非编码区变异位点2 444个,占比3.09%。在4个分区中,LSC区变异位点数量最多,为1 588个,占比1.78%;其次是SSC区,为863个,占比4.60%;IR区变异位点最少,仅有119个,占比0.45%。
在12种柿属植物叶绿体基因组中,编码区共发现含有变异位点的基因21个,不同基因变异位点从高到低占比为25.94%~1.15%(附表2)。其中,psbK变异位点百分比最高,为25.94%。在21个基因中,16个位于LSC区、3个位于IR区、2个位于SSC区。非编码区共发现含变异位点的基因序列118条,不同序列变异位点从高到低占比为49.30%~0.18%。其中,rps12-2-rps12-1变异位点百分比最高,为49.30%。非编码区变异位点百分比最高的20个基因序列如表4所示。
2.4 密码子偏好性分析
川柿叶绿体全基因组序列中的蛋白编码基因共包含52 639个密码子,高频密码子(RSCU>1)共有31个。其中,以A结尾的13个、以U结尾的12个、以C结尾的3个、以G结尾的3个(图3)。密码子Aug、Trp的RSCU值为1,表示其无偏好性。在这些密码子所编码的蛋白质中,亮氨酸(Leu)所占数量最多,含5 096个密码子,占比9.68%;色氨酸(Trp)所占数量最少,含689个密码子,占比1.31%。
2.5 选择压力分析
对川柿、老鸦柿、乌柿、海南柿叶绿体基因组的78个共有蛋白编码基因进行选择压力检测(图4)。图4结果表明,在兩两物种的比较中,各基因的dN平均值为0.470 0,dS平均值为0.652 1,dN最高的基因为petD(2.054 9),dS最高的基因为rpoC1(3.352 4),ω(dN/dS)平均值为0.720 8。共有8个基因(ndhB、ndhG、ndhI、rbcL、rpoB、petB、petD、rps12)的ω平均值大于1,41个基因的ω等于0,其余基因的ω平均值均小于1。对于川柿而言,两两物种比较中,基因rps12的ω最大,为3.957 3,出现在川柿与海南柿的比较中。基因ndhG和ndhI的ω较大,分别为2.589 0和2.563 9,出现在川柿与乌柿及川柿与海南柿的比较中。
2.6 系统发育分析
分别以叶绿体全基因组、非编码区序列、非编码区20个变异热点区域的一致性序列以及编码区序列4个不同的数据集,进行包括川柿在内的12种柿属植物的系统发育分析(图5:a-d)。全基因组、非编码区序列、非编码区变异热点序列3个系统树(图5:a-c)均强烈支持川柿与乌柿和老鸦柿亲缘关系最为接近,乌柿与老鸦柿互为姐妹种,川柿与这2个种形成姐妹群(支持率均为PP/BS: 1/100)。这3个种又与海南柿形成姐妹群,共同构成一个单系分支。在以上4个物种构成的分支中,海南柿位于基部,较先分化。海南柿与其他3个种的姐妹关系在全基因组系统树上表现为强支持(图5:a),但在非编码区及其变异热点系统树上的靴带支持率却较弱(图5:b, c)。
在编码区序列构建的系统树上,川柿的近缘类群与以上3个数据集的系统树相比有一些差异(图5:d)。老鸦柿先与美洲柿(Diospyros virginiana)形成姐妹群,二者再与乌柿形成姐妹群,川柿则是这3个种的姐妹群。海南柿与川柿、乌柿、老鸦柿及美洲柿共同构成一个单系分支。
3 讨论与结论
3.1 叶绿体基因组特征分析
本研究中,川柿叶绿体全基因组为典型的四分体结构,基因组全长157 917 bp,共编码113个基因,没有发生基因重排现象。经比较,在基因组结构、大小及基因类型和排列顺序等方面,川柿与柿属其他11个物种的叶绿体全基因組非常相似。周晓君等(2020)研究表明基因组的GC含量是判断物种间亲缘关系的重要指标,本研究川柿叶绿体基因组的GC含量与其他11个物种一致,均为37.4%。这表明柿属物种的叶绿体基因组结构较为保守。
基因组中存在的重复序列在序列变异中发挥着重要作用,其长度影响基因组的结构和功能(Weng et al., 2014)。在许多已知的被子植物中都发现叶绿体基因组具有不同类型的重复序列,特别是在基因间隔区中(Daniell et al., 2016)。本研究川柿叶绿体基因组的长重复序列中,63.0%的重复序列位于非编码区,在串联重复序列中也得到了类似的结果,70.0%的序列位于非编码区。检测到的3种重复序列类型中,回文序列占比最高,为49.0%。这一结果与柿属其他一些种类相似,在柿、油柿、金枣柿、粉叶柿、君迁子中,重复序列同样主要分布于非编码区,并且回文序列占比最高 (Fu et al., 2016)。
简单重复序列SSR具有含量丰富、多态性高、共显性等优点,被广泛用作分子标记(罗冉等, 2010)。戚建锋等(2018)已将SSR成功应用于柿地方品种的鉴定中。在所有SSR类型中,A和T是最常用的碱基,很少包含碱基G或C重复,这可能是因为碱基A/T比碱基G/C更容易改变(Kuang et al., 2011)。本研究川柿叶绿体基因组鉴定出的34个SSR中,仅一个位点由碱基C、G组成。此外, 非编码区SSR位点出现的数量占比61.8%,远多于编码区。川柿SSR以上特点与其他很多被子植物叶绿体基因组SSR序列组成的规律基本一致(Sun et al., 2021),这可能是导致叶绿体基因组具有较强保守性的原因之一。这些SSR序列为川柿的遗传多样性研究提供了候选的分子标记。
密码子的使用偏好性与物种长期的进化过程密切相关,能够对基因功能及其编码蛋白的表达产生影响(冉然等, 2022)。川柿叶绿体基因组密码子具有明显的A/U偏好性,此特征与其他柿属植物及很多双子叶植物相似(Lin et al., 2016; 傅建敏等, 2017)。与已报道的5种柿属(柿、油柿、君迁子、金枣柿、粉叶柿)叶绿体基因组密码子比较发现(傅建敏等, 2017),在共有密码子中,有8个密码子的使用频率不同:在GGU、AGU、CGU中,这5个柿属植物的RSCU>1,而川柿RSCU<1;在AUA、GGG、AGG、CCC、ACC中,这5个柿属植物的RSCU<1,而川柿RSCU>1,显示川柿对这些密码子的使用频率有一定的独特性。
本研究中,对川柿及其他11种柿属植物叶绿体基因组序列的变异分析显示,LSC 区包含最多核苷酸变异值高的序列,表明LSC 区具有更高的序列多样性水平。在编码区与非编码区的比较中,非编码区变异位点数量和比例明显高于编码区,表明编码区更加保守,这与大多数被子植物的结果相一致(Daniell et al., 2016)。通过比较共选定了10个高变热点区域,其中LSC区5个、SSC区4个、IR区1个。这些高变热点区域提供了非常有价值的变异信息,可作为潜在的分子标记用于柿属物种鉴定和系统发育研究。
不同的环境会对物种产生不同的选择压力,蛋白编码基因中非同义突变率(dN)与同义突变率(dS)的比值ω是一个衡量分子进化选择压力的重要指标(Dos reis, 2015)。ω=1表示基因受到中性选择,ω>1表示基因受到正选择,ω<1表示基因受到负选择。本研究对川柿、老鸦柿、乌柿、海南柿4个物种共有的78个叶绿体蛋白编码基因进行两两比较,发现41个基因的ω平均值为0,意味着在适应性进化过程中,多数基因并没有发生同义和非同义替换,在一定程度上表明了柿属基因的稳定性。对于川柿而言,rps12、ndhG和ndhI的ω>2,显示这几个基因受到较强烈的正选择压力。rps12基因在所有叶绿体基因中结构极为特殊且高度保守(平晶耀等, 2021)。Martín和Sabater(2010)的研究表明ndh基因可能参与叶绿体对光氧化胁迫的保护,同时植物抗氧化胁迫能力与其对其他环境条件的抗性密切相关。因此,rps12和ndh基因对研究川柿和柿属植物叶绿体基因组适应性进化过程以及进化速率具有重要价值。
3.2 系统发育分析
近年来,对于柿属的部分种类,国内外基于叶绿体全基因组、叶绿体基因片段以及核基因片段,已进行了一些分子系统学研究,为了解柿属的系统发育提供了重要线索。虽然川柿早在1945年即已发表,但直到近年才在野外被再次发现,目前对川柿的研究极少,其系统亲缘关系也不明确。
本研究中,叶绿体全基因组、非编码区以及非编码区变异热点构建的系统发育树均显示川柿与老鸦柿和乌柿亲缘关系最为接近,并且支持率均为最高。老鸦柿与乌柿的形态特征非常相似,以至于过去老鸦柿的标本常被误定为乌柿(吴征镒和崔鸿宾, 1987)。本研究中,二者的果实均较小,直径1.5~3 cm,比川柿果实略小,其他形态特征与川柿很相似。在地理分布上,川柿、老鸦柿和乌柿在中国均分布于亚热带山地,在生境上非常相似。这3种柿树进而与分布于海南热带常绿阔叶林的海南柿组成一个单系,显示有最近的共同祖先。海南柿果实直径3~4 cm且幼时密被毛,与川柿果实大小相近。
在编码区构建的系统树中,美洲柿与老鸦柿聚为同一分支,与乌柿形成姐妹群,进而与川柿汇于同一分支。然而,美洲柿的染色体为6倍体(2n=90),而老鸦柿染色体为4倍体(2n=60)(Tamura et al., 1998)。其他叶绿体和核基因片段分子证据显示,美洲柿与柿、油柿和君迁子关系密切,而与老鸦柿关系较远(Yonemori et al., 2008; Turner et al., 2013)。张永芳等(2016)和胡超琼等(2018)对部分柿属植物花粉形态的观察显示,美洲柿、柿、油柿、粉叶柿在花粉表面纹饰上非常相似,而乌柿花粉表面呈现较多的褶皱,与这些种类有较大差异。编码区系统树的不一致可能是因葉绿体基因组编码区序列较为保守、变异信息位点较少而导致。
另外,由分布于东南亚的D. blancoi和D. celebica以及中国海南的琼岛柿(D. maclurei)组成的分支,与其他国内种类分支的拓扑关系在4个系统树中并不完全一致,并且支持率均很低,显示叶绿体基因组数据尚不能阐明这3种柿树的系统发育关系,需进一步深入研究。
综上所述,基于叶绿体全基因组、非编码区及其变异热点所得到的川柿系统发育研究结果与其他已有证据的相一致。因此,在这11个柿属物种中,与川柿亲缘关系最近的是乌柿和老鸦柿。在全基因组、非编码区及其变异热点建立的3个系统树中,全基因组系统树的支持率最高,显示应该优先选择信息位点最多的叶绿体全基因组进行柿属的系统发育研究。
参考文献:
AK INSULIRE OP, OLADIPO OT, ABDULRAHEEM OA, et al., 2018. Taxonomic significance of epidermal and venation characters in the genus Diospyros L. (Ebenaceae) in Nigeria [J]. Braz J Biol Sci, 5(10): 459-514.
BEIER S, THIEL T, MNCH T, et al., 2017. MISA-web: a web server for microsatellite prediction [J]. Bioinformatics, 33(16): 2583-2585.
BENSON G, 1999. Tandem repeats finder: a program to analyze DNA sequences [J]. Nucleic Acids Res, 27(2): 573-580.
DANIELL H, LIN CS, YU M, et al., 2016. Chloroplast genomes: diversity, evolution, and applications in genetic engineering [J]. Genome Biol, 17(1): 134.
DONG WP, LIU J, YU J, et al., 2012. Highly variable chloroplast markers for evaluating plant phylogeny at low taxonomic levels and for DNA barcoding [J]. PLoS ONE, 7(4): e35071.
DOS REIS M, 2015. How to calculate the non-synonymous to synonymous rate ratio of protein-coding genes under the Fisher-Wright mutation-selection framework [J]. Biol Lett, 11(4): 20141031.
DUANGJAI S, WALLNFER B, SAMUEL R, et al., 2006. Generic delimitation and relationships in Ebenaceae sensu lato: evidence from six plastid DNA regions [J]. Am J Bot, 93(12): 1808-1827.
FANG WP, 1985. Flora of Sichuan: Vol. 3 [M]. Chengdu: Sichuan Peoples Publishing House: 282. [方文培, 1985. 四川植物志: 第3卷 [M]. 成都: 四川人民出版社: 282.]
FU JM, LIU HM, HU JJ, et al., 2016. Five complete chloroplast genome sequences from Diospyros: genome organization and comparative analysis [J]. PLoS ONE, 11(7): e0159566.
FU JM, LIANG JJ, WUYUN TN, et al., 2015. Sequence analysis of ITS regions and ndhA gene for determining phylogenetic relationship of Diospyros kaki(persimmon) with other related wild Diospyros (Ebenaceae) species [J]. Bull Bot Res, 35(4): 515-520. [傅建敏, 梁晋军, 乌云塔娜, 等, 2015. 基于叶绿体DNA ndhA和nrDNA ITS序列变异分析栽培柿及其近缘种亲缘关系 [J]. 植物研究, 35(4): 515-520.]
FU JM, SUO YJ, LIU HM, et al., 2017. Analysis on codon usage in the chloroplast protein-coding genes of Diospyros spp. [J]. Nonwood For Res, 35(2): 38-44. [傅建敏, 索玉静, 刘慧敏, 等, 2017. 柿属植物叶绿体蛋白质编码基因密码子用法 [J]. 经济林研究, 35(2): 38-44.]
GAO F, CHEN C, ARAB DA, et al., 2019. EasyCodeML: A visual tool for analysis of selection using CodeML [J]. Ecol Evol, 9(7): 3891-3898.
HU CQ, WANG RZ, GUAN CF, et al., 2018. Pollen morphology observation and palynological studies of Diospyros [J]. J Fruit Sci, 35(9): 1067-1078. [胡超琼, 王仁梓, 关长飞, 等, 2018. 柿属植物花粉形态观察及孢粉学研究 [J]. 果树学报, 35(9): 1067-1078.]
JIN JJ, YU WB, YANG JB, et al., 2020. GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes [J]. Genome Biol, 21(1): 241.
KALYAANAMOORTHY S, MINH BQ, WONG TKF, et al., 2017. ModelFinder: fast model selection for accurate phylogenetic estimates [J]. Nat Methods, 14(6): 587-589.
KATOH K, STANDLEY DM, 2013. MAFFT multiple sequence alignment software version 7: improvements in performance and usability [J]. Mol Biol Evol, 30(4): 772-780.
KUANG DY, WU H, WANG YL, et al., 2011. Complete chloroplast genome sequence of Magnolia kwangsiensis (Magnoliaceae): implication for DNA barcoding and population genetics [J]. Genome, 54(8): 663-673.
KUBITZKI K, 2004. The families and genera of vascular plants: Vol. 6 [M]. Berlin: Springer.
KUMAR S, STECHER G, LI M, et al., 2018. MEGA X: molecular evolutionary genetics analysis across computing platforms [J]. Mol Biol Evol, 35(6): 1547-1549.
KURTZ S, CHOUDHURI JV, OHLEBUSCH E, et al., 2001. REPuter: the manifold applications of repeat analysis on a genomic scale [J]. Nucleic Acids Res, 29(22): 4633-4642.
LI N, LI YY, ZHENG CC, et al., 2016. Genome-wide comparative analysis of the codon usage patterns in plants [J]. Genes Genomics, 38(8): 723-731.
LI S, MICHAEL S, FRANK W, 1996. Ebenaceae [M]//Flora of China: Vol 15. Beijing: Science Press St. Louis: Missouri Botanical Garden Press: 215-234.
LIANG JJ, LIANG YQ, FU JM, 2014. Genetic relationships of Diospyros kaki and related Diospyros species using chloroplast DNA PCR-RFLP markers [J]. Acta Hortic Sin, 41(12): 2474-2480. [梁晉军, 梁玉琴, 傅建敏, 2014. 基于叶绿体DNA PCR-RFLP分析柿及其近缘种亲缘关系 [J]. 园艺学报, 41(12): 2474-2480.]
LIBRADO P, ROZAS J, 2009. DnaSP v5: a software for comprehensive analysis of DNA polymorphismdata [J]. Bioinformatics, 25(11): 1451-1452.
LIU WW, TAN XH, ZHAO KK, et al., 2018. Complete plastome sequences of Diospyros maclurei Merr. and Diospyros hainanensis Merr. (Ebenaceae): two endemic species in Hainan Province, China [J]. Mitochondrial DNA Part B, 3(2): 1205-1207.
LIU YQ, LIU ZY, CAO M, et al., 2021a. Status of field resources of Diospyros sutchuensis with extremely small population [J]. S Chin Fruits, 50(2): 148-150. [刘燕琴, 刘正宇, 曹敏, 等, 2021a. 极小种群川柿野外资源现状 [J]. 中国南方果树, 50(2): 148-150.]
LIU YQ, LIU ZY, LIU CL, et al., 2021b. Study on biological characteristics and techniques of seed germination of endangered species of Diospyros sutchuensis Yang [J]. S Chin Fruits, 50(4): 91-95. [刘燕琴, 刘正宇, 刘春雷, 等, 2021b. 极小种群植物“川柿”生物学特性及种子萌发方法研究 [J]. 中国南方果树, 50(4): 91-95.]
LU QF, HUANG ZH, LUO WH, 2021. Characterization of complete chloroplast genome in Firmiana kwangsiensis and F. danxiaensis with extremely small populations [J]. Biodivers Sci, 29(5): 586-595. [陸奇丰, 黄至欢, 骆文华, 2021. 极小种群濒危植物广西火桐、丹霞梧桐的叶绿体基因组特征 [J]. 生物多样性, 29(5): 586-595.]
LUO R, WU WL, ZHANG Y, et al., 2010. SSR marker and its application to crop genetics and breeding [J]. Genom Appl Biol, 29(1): 137-143. [罗冉, 吴委林, 张旸, 等, 2010. SSR分子标记在作物遗传育种中的应用 [J]. 基因组学与应用生物学, 29(1): 137-143.]
MARTN M, SABATER B, 2010. Plastid ndh genes in plant evolution [J]. Plant Physiol Biochem, 48(8): 636-645.
NGUYEN LT, SCHMIDT HA, VON HAESELER A, et al., 2015. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies [J]. Mol Biol Evol, 32(1): 268-274.
PAHLICH E, GERLITZ ChR, 1980. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Phytochemistry, 19(1): 11-13.
PING JY, FENG PP, HAO J, et al., 2021. Molecular evolution pattern of rps12 gene in gymnosperms [J]. Chin Sci Bull, 66(24): 3182-3202. [平晶耀, 冯佩沛, 郝静, 等, 2021. rps12基因在裸子植物中的分子进化式样 [J]. 科学通报, 66(24): 3182-3202.]
POSADA D, BUCKLEY TR, 2004. Model selection and model averaging in phylogenetics: advantages of akaike information criterion and bayesian approaches over likelihood ratio tests [J]. Syst Biol, 53(5): 793-808.
QI JF, LI XP, WANG WR, et al., 2018. Identification of 72 persimmon landraces by using SSR markers-based MCID method [J]. J Plant Genet Resour, 19(5): 895-903. [戚建锋, 李晓鹏, 王文然, 等, 2018. 利用基于SSR标记的MCID法鉴定72个柿地方品种 [J]. 植物遗传资源学报, 19(5): 895-903.]
QU XJ, MOORE MJ, LI DZ, et al., 2019. PGA: a software package for rapid, accurate, and flexible batch annotation of plastomes [J]. Plant Methods, 15: 50.
RAN R, ZHANG XM, WAN T, et al., 2022. Method analysis of codon usage bias and its related research [J]. Grassland Pratac, 34(3): 5-10. [冉然, 张晓明, 宛涛, 等, 2022. 密码子偏好性的相关研究及方法分析 [J]. 草原与草业, 34(3): 5-10.]
RAUF A, UDDIN G, PATEL S, et al., 2017. Diospyros, an under-utilized, multi-purpose plant genus: A review [J]. Biomed Pharmacother, 91: 714-730.
SUN K, LIU QY, WANG A, et al., 2021. Comparative analysis and phylogenetic implications of plastomes of five genera in subfamily Amyridoideae (Rutaceae) [J]. Forests, 12(3): 277.
SUN WB, HAN CY, 2015. Researches and conservation for plant species with extremely small populations (PSESP) [J]. Biodivers Sci, 23(3): 426-429. [孙卫邦, 韩春艳, 2015. 论极小种群野生植物的研究及科学保护 [J]. 生物多样性, 23(3): 426-429.]
TAMURA M, TAO R, YONEMORI K, et al., 1998. Ploidy level and genome size of several Diospyros species [J]. J Jpn Soc Hortic, 67(3): 306-312.
TANG DL, ZHANG QL, XU LQ, et al., 2019. Number of species and geographical distribution of Diospyros L. (Ebenaceae) in China [J]. Hortic Plant J, 5(2): 59-69.
TURNER B, MUNZINGER J, DUANGJAI S, et al., 2013. Molecular phylogenetics of new caledonian Diospyros (Ebenaceae) using plastid and nuclear markers [J]. Mol Phylogenet Evol, 69(3): 740-763.
UTSUNOMIYA N, SUBHADRABANDHU S, YONEMORI K, et al., 1998. Diospyros species in Thailand: Their distribution, fruit morphology and uses [J]. Econ Bot, 52(4): 343-351.
VENKATASAMY S, KHITTOO G, NOWBUTH P, et al., 2006. Phylogenetic relationships based on morphology among the Diospyros (Ebenaceae) species endemic to the Mascarene Islands [J]. Bot J Linn Soc, 150(3): 307-313.
WALLNFER B, 2001. The biology and systematics of Ebenaceae: a review [J]. 103B: 485-512.
WENG ML, BLAZIER JC, GOVINDU M, et al., 2014. Reconstruction of the ancestral plastid genome in Geraniaceae reveals a correlation between genome rearrangements, repeats, and nucleotide substitution rates [J]. Mol Biol Evol, 31(3): 645-659.
WU ZY, CUI HB, 1987. Flora Reipublicae Popularis Sinicae: Vol. 60 [M]. Beijing: Science Press: 147. [吳征镒, 崔鸿宾, 1987. 中国植物志: 第六十卷 [M]. 北京: 科学出版社: 147.]
XU B, YANG Z, 2013. PAMLX: a graphical user interface for PAML [J]. Mol Biol Evol, 30(12): 2723-2724.
YANG WZ, XIANG ZY, ZHANG SS, et al., 2015. Plant species with extremely small populations (PSESP) and their significance in Chinas national plant conservation strategy [J]. Biodivers Sci, 23(3): 419-425. [杨文忠, 向振勇, 张珊珊, 等, 2015. 极小种群野生植物的概念及其对我国野生植物保护的影响 [J]. 生物多样性, 23(3): 419-425.]
YANG Y, 1945. New ligneous plants from eastern Szechuan [J]. J W Chin Border Res Soc (Ser. B), 15: 89-91.
YONEMORI K, HONSHO C, KANZAKI S, et al., 2008. Sequence analyses of the ITS regions and the matK gene for determining phylogenetic relationships of Diospyros kaki (persimmon) with other wild Diospyros (Ebenaceae) species [J]. Tree Genet Genomes, 4(2): 149-158.
ZHANG D, GAO FL, JAKOVLI C ' [KG1.5mm] I, et al., 2020. PhyloSuite: An integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies [J]. Mol Ecol Resour, 20(1): 348-355.
ZHANG YF, HU CQ, YANG Y, et al., 2016. Pollen morphology observation of eight resources in Diospyros [J]. Acta Hortic Sin, 43(6): 1167-1174. [张永芳, 胡超琼, 杨勇, 等, 2016. 柿属8种植物花粉形态观察 [J]. 园艺学报, 43(6): 1167-1174.]
ZHOU XJ, ZHANG K, PENG ZF, et al., 2020. Comparative analysis of chloroplast genome characteristics between Paeonia jishanensis and other five species of Paeonia [J]. Sci Silv Sin, 56(4): 82-88. [周晓君, 张凯, 彭正锋, 等, 2020. 矮牡丹与芍药属其他5个种叶绿体基因组特征的比较 [J]. 林业科学, 56(4): 82-88.]
ZOU P, LONG KL, TU TY, et al., 2023. Leaf epidermal morphology of Diospyros from China and its taxonomic significance [J]. J Trop Subtrop Bot, 31(1): 117-127. [鄒璞, 龙开莲, 涂铁要, 等, 2023. 国产柿属(Diospyros)的叶表皮形态研究及其分类学意义 [J]. 热带亚热带植物学报, 31(1):117-127.]
ZUO DX, LIU L, WANG XQ, 1984. Geographical distribution and utilization of Diospyros spp. resources in China [J]. Chin Fruits, 3(8): 27-34. [左大勋, 柳鎏, 王希蕖, 1984. 我国柿属植物资源的地理分布及利用 [J]. 中国果树, 3(8): 27-34.]
( 责任编辑 蒋巧媛 )
收稿日期: 2023-10-11
基金项目: 国家科技基础资源调查专项“中国西南地区极小种群野生植物调查与种质保存” (2017FY100100)。
第一作者: 杨楠(1999-),硕士研究生,研究方向为植物发育与进化,(E-mail)nanmuyi@163.com。
* 通信作者: 赵良成,博士,教授,主要从事植物系统进化生物学研究,(E-mail)lczhao@bjfu.edu.cn。
