甜瓜HDM基因家族鉴定及特性分析
2024-02-22张治张婧张瑾哈斯阿古拉郝金凤
张治,张婧,张瑾,哈斯阿古拉,郝金凤
研究报告
甜瓜HDM基因家族鉴定及特性分析
张治,张婧,张瑾,哈斯阿古拉,郝金凤
内蒙古大学生命科学学院,内蒙古自治区牧草与特色作物生物技术重点实验室,呼和浩特 010070
组蛋白去甲基化酶(histone demethylase,HDM)是生物生长过程中的重要调节因子之一,在植物的生长发育和环境适应过程中发挥着重要的调控作用。本研究通过生物信息学方法对甜瓜(L.) HDM基因家族进行了鉴定,并通过转录组数据检测了其成员在甜瓜组织的表达特性。结果显示,在甜瓜基因组中鉴定得到了20个基因,在各条染色体上非均匀分布;其成员可以分成LSD1和JmjC两大类,而JmjC类群又可划分为5个亚群,各类群成员数量不等;种内和种间的共线性关系显示,甜瓜基因仅存在一对片段重复,与番茄()基因共线性区域较多,亲缘关系较近;各成员所含保守结构域数量不等,且外显子和内含子在数量上有所差异;在启动子区域存在多种与激素和环境信号响应的顺式作用元件,表达特性分析显示各基因成员在甜瓜雄花、雌花、根、茎、叶、子房和成熟果实中均存在不同程度的表达。这些结果将有助于更好地理解基因的潜在功能以及它们在参与调节甜瓜生长发育和环境自适中可能发挥的作用。
甜瓜;HDM家族;基因鉴定;表达分析
在植物的生长和发育过程中,染色质对基因表达和环境的反应非常重要。染色质的调控是不涉及DNA序列改变情况下,通过改变DNA和组蛋白修饰来调节真核染色质结构以及基因表达的一种重要机制[1]。染色质由核小体组成,核小体由一条长约146 bp的DNA缠绕组蛋白修饰位点形成八聚体结构(H2A、H2B、H3和H4各有两个分子)[2],H1作为连接蛋白,将核小体逐个连接起来呈念珠状。组蛋白有许多共价修饰,常见的方式包括组蛋白乙酰化、甲基化、磷酸化、泛素化和ADP核糖基化等[3],并且组蛋白修饰具有多样性,相互之间能够形成一个调控网络来参与染色质介导的多种生命活动[4]。
组蛋白甲基化作为组蛋白修饰的主要方式之一,在基因组水平上对染色质构型和基因表达调控起着重要的作用。组蛋白甲基化主要发生在组蛋白H3、H4的赖氨酸和精氨酸残基上[5],其中赖氨酸残基可以被单、二、三甲基化修饰,而精氨酸残基只能被单甲基化、不对称或对称二甲基化修饰,这可能是受到了组蛋白甲基转移酶和去甲基化酶的双重调节[6~8]。在植物中,组蛋白赖氨酸甲基化在组蛋白H3的K4、K9、K27、K36、K79位点和H4K20位点均存在修饰作用[9~11]。一般来说,组蛋白H3相关位点的甲基化修饰作用存在着差异性,K4、K36、K79位点的甲基化修饰主要与转录激活密切相关,而K9、K27位点的甲基化修饰与转录抑制密切相关[12]。
目前,已知进化上保守的组蛋白赖氨酸去甲基化酶主要有赖氨酸特异性去甲基化酶1 (LSD1)和含有Jumonji C端(JmjC)结构域蛋白的去甲基化酶两种,并且这两类去甲基化酶通过不同的途径发挥其去甲基化作用[13,14]。LSD1需要黄素腺嘌呤二核苷酸为辅助因子,而JmjC结构域蛋白需要Fe(II)和α-酮戊二酸(αKG)为辅助因子,作用于组蛋白赖氨酸去甲基化修饰过程[15,16]。含有JmjC结构域的蛋白是一种很重要的组蛋白赖氨酸去甲基化酶,其在维持体内组蛋白甲基化的动态平衡中起着关键作用[17,18]。根据序列相似性和催化特异性,植物中JmjC类群进一步又可以被划分为5个亚群:KDM5/JARID1、KDM4/ JHDM3、KDM3/JHDM2、JMJD6和JmjC domain- only[19]。有研究报道,一些植物JmjC基因家族成员在多个发育过程中发挥着重要作用,包括影响花期[20,21]、叶片、花序及种子的形态[22,23]、生物节律[24]、芽的再生[25]、茎的伸长[26]等过程。另外,其在响应盐胁迫、热胁迫、干旱胁迫等逆境反应过程中也发挥着十分重要的作用[1,27,28]。
甜瓜(L.)是葫芦科最重要的二倍体作物之一,其果实含有可溶性糖、有机酸、矿物质和维生素[29]。甜瓜具有很重要的经济和园艺价值,在世界各地均有种植[30]。目前,通过生物信息学方法已完成了拟南芥()[19]、水稻()[19]、玉米()[5]、大豆()[31]和月季()[18]等组蛋白去甲基化酶(histone demethylases,HDMs)中JmjC基因家族成员的鉴定,但迄今为止对甜瓜基因尚未进行鉴定和功能研究。本研究通过生物信息学方法对甜瓜HDM基因家族成员进行搜索和鉴定,并对鉴定得到的家族成员的蛋白理化性质、基因结构、系统进化、保守功能域、染色体分布、基因表达等特性进行了分析,以期为后续阐明CmHDM的生物学功能奠定基础。
1 材料和方法
1.1 甜瓜HDM基因家族成员的鉴定与分类注释
从葫芦科数据库CuGenDB (http://www.cucurbitgenomics.org/)获取甜瓜(v3.6.1)全基因组序列和蛋白质相关信息。从Pfam数据库分别下载HDM中JmjC和LSD1蛋白保守结构域的隐马尔可夫(HMM)模型文件,并利用TBtools搜索甜瓜基因组蛋白序列,参数为默认值。同时,以拟南芥基因组数据库(25个成员) HDM氨基酸序列为参考序列,基于BioEdit7.2.0软件建立本地蛋白数据库,进行本地Blastp蛋白比对(值为10–3)以获取同源性较高的序列。通过以上两种方法获得CmHDM基因家族成员的潜在候选序列,并提交至SMART数据库进行保守结构域的鉴定,选择具有完整HDM中LSD1和JmjC基因家族特有保守域的成员进行后续分析,并参考成员所在的染色体以及在染色体上的位置进行命名。
1.2 甜瓜HDM基因家族蛋白理化特性和染色体定位
通过在线网页ExPASy预测CmHDM蛋白的氨基酸数目、分子量等理化性质。利用WoLF PSORT在线工具进行其成员亚细胞定位预测分析。根据基因在染色体的位置信息,使用TBtools绘制CmHDM家族染色体物理图谱。
1.3 甜瓜HDM基因家族结构、编码蛋白结构域和进化分析
根据CmHDM家族基因GFF注释文件信息,对其外显子及内含子等结构特征进行分析并绘制基因结构图。通过SMART数据库对提交的成员蛋白序列进行保守结构域分析,并利用TBtools软件进行可视化。在MEME(http://meme-suite.org/tools/meme)网站分析CmHDM蛋白序列的保守基序(motif)并对其进行可视化分析。采用MEGA 7.0通过邻接法(Neighbor-joining)构建甜瓜、拟南芥、番茄()HDM基因家族成员系统进化树,其中模式采用Poisson correction模型,数据缺失设置为Pairwise deletion,Bootstrap运行次数为1000,其他参数默认。
1.4 甜瓜HDM基因启动子顺式作用元件分析
从甜瓜全基因组序列中提取CmHDM家族成员起始密码子上游2000 bp序列,提交至Plant CARE (http://bioinformatics.psb.ugent.be/Webtools/plantcare/html/),对顺式作用元件进行预测和分析。
1.5 甜瓜HDM基因家族复制和共线性分析
为了研究甜瓜基因是否包含串联重复或片段复制,通过MCScanX软件对基因的重复事件进行分析。为了研究甜瓜基因和拟南芥、番茄物种之间的复制关系,利用TBtools软件进行共线性关系分析并绘制。
1.6 甜瓜HDM基因家族表达模式分析
为探究甜瓜HDM基因家族在不同组织器官中的表达差异,从NCBI SAR数据库下载甜瓜植株的根、茎、叶、雌花、雄花、子房(登录号:PRJNA803327)以及成熟果实组织(登录号:PRJNA543288)的转录组数据,将其中基因的相关数据筛选并提交至TBtools进行热图绘制,从表达模式推测其在甜瓜发育中可能发挥的作用。
2 结果与分析
2.1 甜瓜HDM基因的鉴定和蛋白理化性质分析
通过序列比对和保守结构域分析,共鉴定到20个甜瓜HDM基因家族成员,其中17个基因和3个基因。根据各基因所在染色体及染色体上的位置依次进行命名,即~和~(附表1)。蛋白理化特性分析发现,CmJMJ蛋白长度为413~1817 aa,蛋白分子量为52,175.9~208,706.01 Da,其中CmJMJ15蛋白分子量最大,CmJMJ04蛋白分子量最小。CmJMJ不稳定系数在37.95~60.48之间,等电点范围在4.57~8.84,其中酸性氨基酸占64.71%,碱性氨基酸占35.29%。而CmLSD1蛋白长度最长的是CmLSD02 (1934 aa),其次是CmLSD01 (906 aa),CmLSD03最短(886 aa)。蛋白分子量为98,220.41~ 211,756.59 Da,不稳定系数在38.31~45.20之间,CmLSD01和CmLSD03为碱性蛋白,CmLSD02为酸性蛋白。CmHDM基因家族成员均为亲水性蛋白,其大部分成员预测分布于细胞核,个别分布于细胞质、内质网和叶绿体。
2.2 甜瓜HDM基因结构、保守结构域和系统进化分析
为探究甜瓜基因的系统发育进化,本研究分析了甜瓜与拟南芥和番茄之间的同源进化关系。使用ClustalW对20个CmHDM、25个AtHDM和SlHDM的编码蛋白序列进行比对,借助MEGA7.0进行系统发育进化分析。参照拟南芥分类方法,甜瓜HDM成员被分成LSD1和JmjC两大类(图1),LSD1蛋白类群有3个成员(LSD01~03),在甜瓜JmjC域蛋白类群中,包括4个KDM3/JHDM2 (CmJMJ10、11、13和14)、4个KDM4/JHDM3 (CmJMJ05、07、12和17)、4个KDM5/JARID1 (CmJMJ01、09、15和16)、2个JMJD6 (CmJMJ06和08)和3个JmjC domain-only (CmJMJ02~04)。整体来说,甜瓜(20个)与拟南芥(25个)、番茄(25个) HDM基因家族成员在各亚族分布均匀,数量上相差不大,表明三者HDM基因家族成员在进化过程中具有一定相似性。
在基因表达和调控过程中,内含子的数量和位置非常重要。通过对甜瓜HDM基因家族基因结构的分析发现(图2A),各成员均含有内含子和外显子且彼此间含有的个数不相同,3个基因中内含子最多有7个(),最少2个();17个基因中内含子最多有35个(),最少1个()。为了进一步解析CmHDM家族基因的功能,本研究使用TBtools软件分析了CmHDM蛋白的保守motif和motif微标(图2,B和C),发现不同亚族之间保守基序组成有所差异,但各亚族间保守基序基本相同。其中,KDM3/JHDM2均存在motif4、motif5、motif7、motif10、motif11和motif12,KDM4/JHDM3和KDM5/JARID1均存在motif1、motif2和motif15,JMJD6和JmjC domain-only均存在motif8,LSD1类群蛋白均存在motif8、motif13和motif14,同时发现系统进化树中关系密切的成员往往具有相同的motif,表明CmHDM家族各亚族基因的结构域在进化上具有极高的保守性和相似性,可能具有相同的生物学功能。
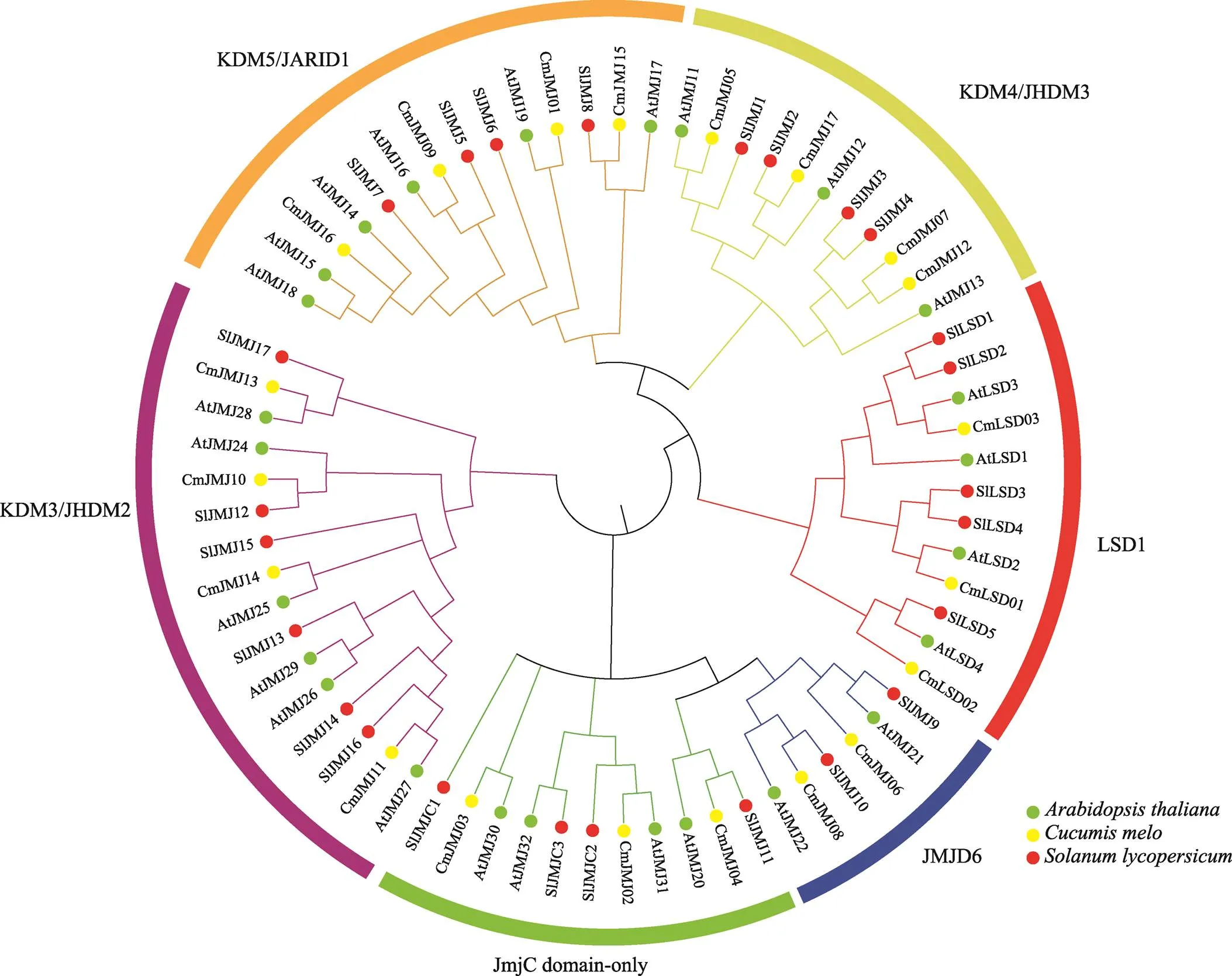
图1 甜瓜与拟南芥、番茄HDM蛋白家族的系统进化树
绿色圆点:(拟南芥);黄色圆点:(甜瓜);红色圆点:(番茄)。系统进化树外圈用6种不同颜色的弧线分别表示LSD1、KDM3/JHDM2、KDM4/JHDM3、KDM5/JARID1、JMJD6和JmjC domain-only亚家族。

图2 甜瓜HDM家族成员基因结构和保守motif分析
A:基因结构;B:保守motif;C:motif序列微标(每个字母的高度表示该位置氨基酸的出现频率)。UTR:untranslated region (非翻译区);CDS:coding sequence (编码序列)。
在编码蛋白的保守结构中发现(图3),LSD1蛋白类群N末端含有SWIRM结构域和C末端含有胺氧化酶活性的Amino oxidase结构域,它们共同发挥相应作用。在含JmjC域蛋白类群中,除了JmjC domain-only亚族成员仅有JmjC结构域,而其他亚族成员不仅包含JmjC结构域,还主要具有JmjN、F-box、FYRN、FYRC、ARID和RING等蛋白结构域,它们的去甲基化功能不仅依赖于JmjC结构域还依赖于其他的结构域,并且它们的作用方式存在特异性。
2.3 甜瓜HDM基因染色体定位、基因复制分析
在甜瓜12条染色体中(图4),20个HDM家族成员分布不均且有所差异,其成员主要分布在1、2、3、4、5、6、8、9和12号染色体上,而在7、10和11号染色体上没有成员分布,除8号染色体仅存在一个基因外,其他染色体均存在两个或两个以上基因。基因家族主要的扩增方式分为串联重复和片段重复,根据MCscan结果显示(图5),在20个组蛋白去甲基化酶基因(和)中,仅在JmjC家族存在一对片段重复(和),且均不存在串联重复,表明甜瓜组蛋白去甲基化酶基因在进化过程中相对独立。
2.4 甜瓜HDM基因启动子顺式调控元件分析
转录起始位点上游的顺式作用元件在调控植物对逆境胁迫应答过程中发挥了十分重要的作用。本研究通过Plant CARE软件对启动子区域的顺式作用元件分析发现(图6),甜瓜HDM家族启动子区域除了含有大量的核心元件CAAT-box和TATA-box以外,其他主要分为植物生长发育、激素和环境信号响应三类:与植物生长发育相关的元件包括分生组织表达CAT-box、胚乳表达GCN4-motif、生理节律调节Circadian等;与植物激素响应有关的元件包括生长素AuxRR-core、赤霉素GARE-motif、茉莉酸CGTCA-motif、TGACG-motif和脱落酸ABRE等;与环境信号相关的包括光应答Box4和AE-box、低温诱导LTR、干旱诱导MBS等应答元件。
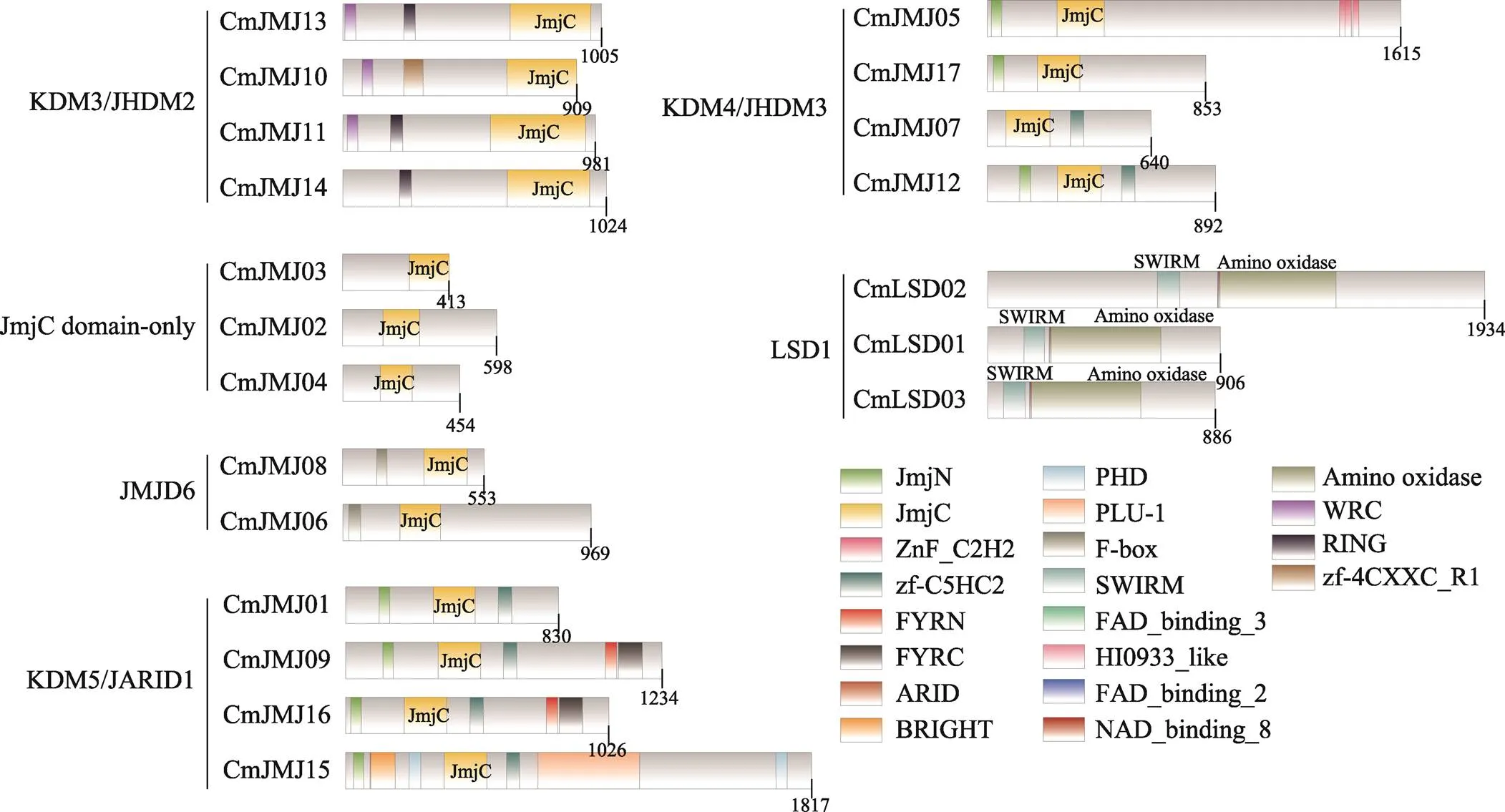
图3 甜瓜HDM家族成员保守结构域分析
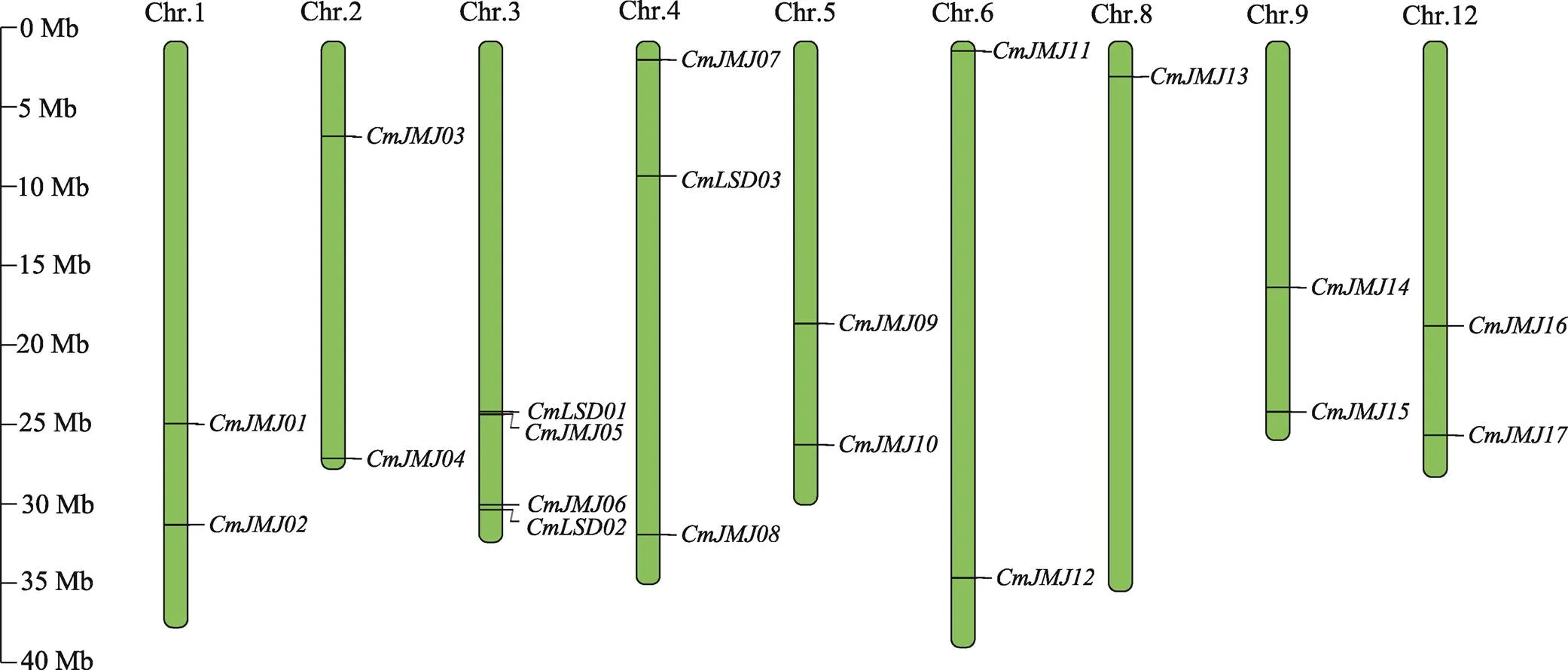
图4 甜瓜HDM家族成员染色体定位
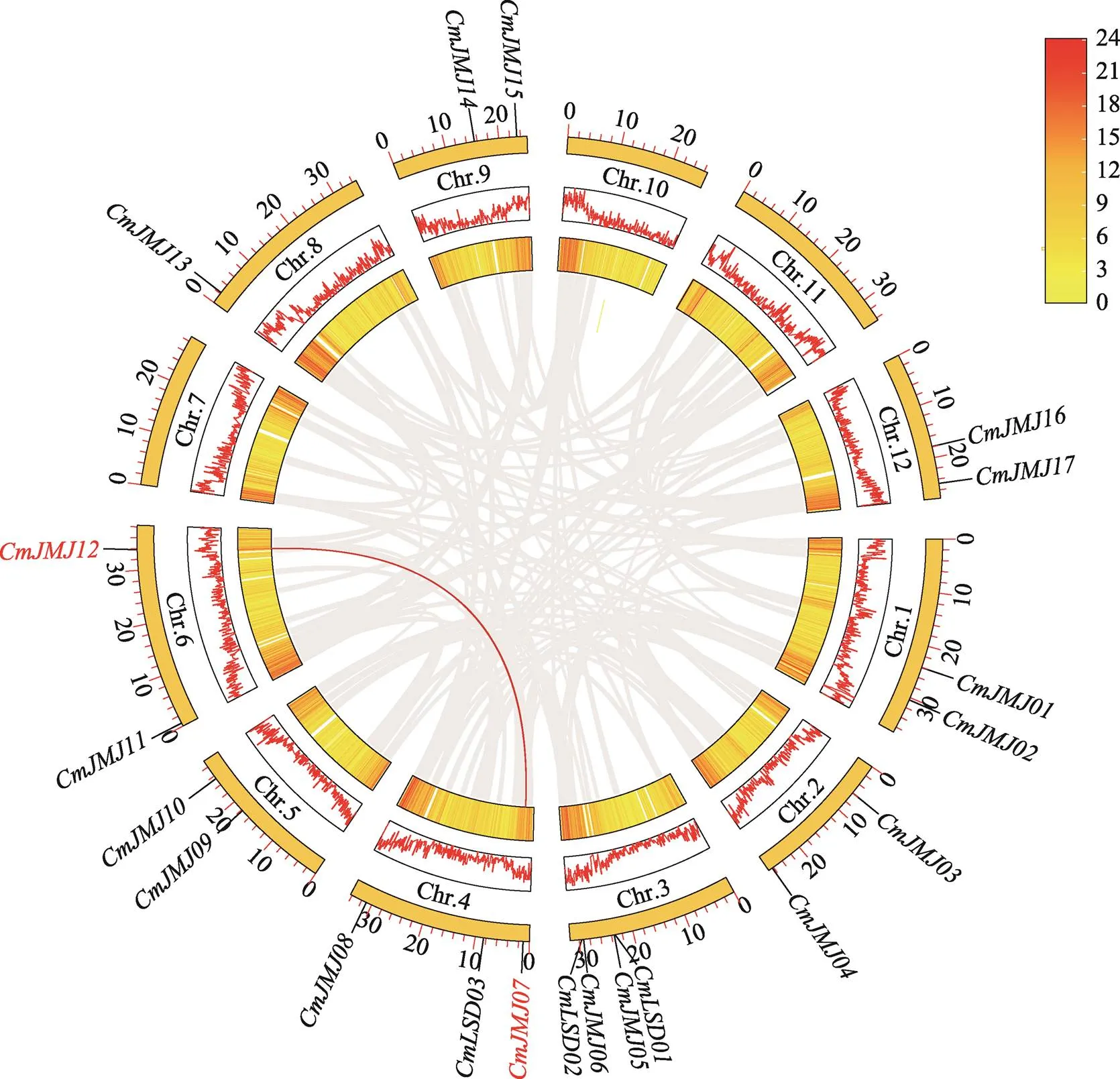
图5 甜瓜HDM家族成员共线性分析
红色线条表示甜瓜HDM家族成员间存在片段复制基因对。
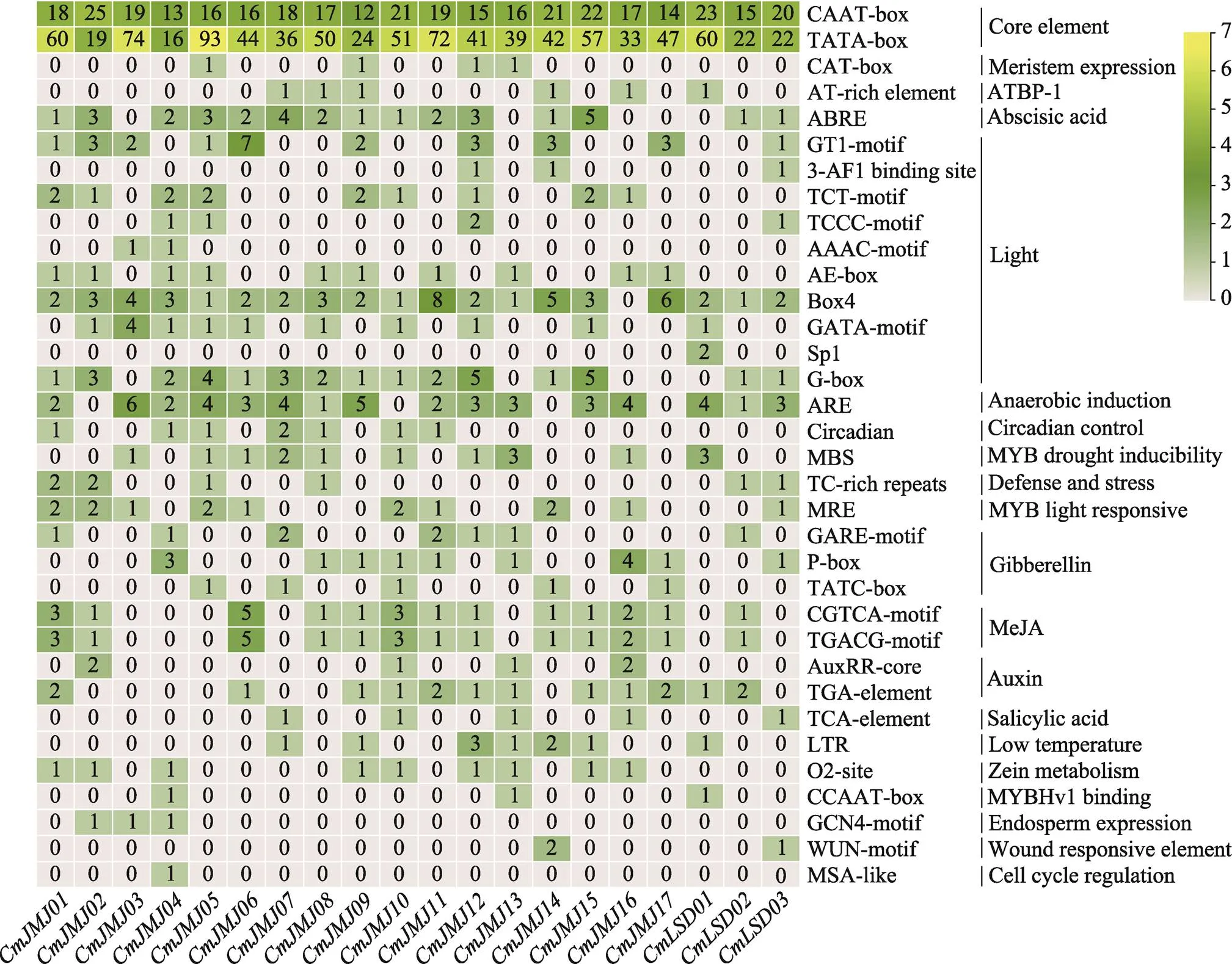
图6 甜瓜HDM家族成员启动子顺式元件分析
其中,环境信号光响应元件在20个基因中均存在,尤其是低温诱导、干旱诱导元件较多;植物激素生长素和茉莉酸元件占65%,脱落酸和赤霉素元件占75%此;外45%成员有玉米胶蛋白代谢调节元件O2-site。存在细胞周期调节元件MES-like,和存在创伤响应元件WUN-motif等特殊顺式作用元件,表明这类基因可能在参与调控植物生长发育和逆境胁迫应答过程中发挥了关键作用。
2.5 甜瓜HDM基因与不同物种间共线性分析
为了更好的理解组蛋白去甲基化酶基因在不同物种之间的进化关系,本研究选取拟南芥、番茄及甜瓜的基因组进行了共线性分析(图7)。分析结果表明,甜瓜与番茄基因存在14对共线性关系,而与拟南芥基因存在12对共线性关系,两者数量上区别不大,番茄相对于拟南芥,与甜瓜亲缘关系更近,共线性区域较多。
在20个甜瓜基因中,有4个基因(、、和)和1个基因()与拟南芥和番茄都不存在共线性关系,其他基因均与拟南芥、番茄同源基因存在共线性关系。同时,还发现有一些基因至少与2个同线基因对有关,如和能够与番茄同源基因形成2个同线基因对,猜测这些基因可能在植物的进化过程中起着非常重要的作用,然而这些基因在拟南芥基因组并未出现一对多的共线性关系,表明甜瓜和拟南芥在分化之后并没有发生基因家族复制事件,在进化过程中还是处于较保守的状态。此外,在甜瓜、拟南芥和番茄中发现了8个共线基因对,包括、、、、、、和,这可能是最早存在于植物中的组蛋白去甲基化酶基因,能够在不同的组织结构发挥相应的生物学功能。
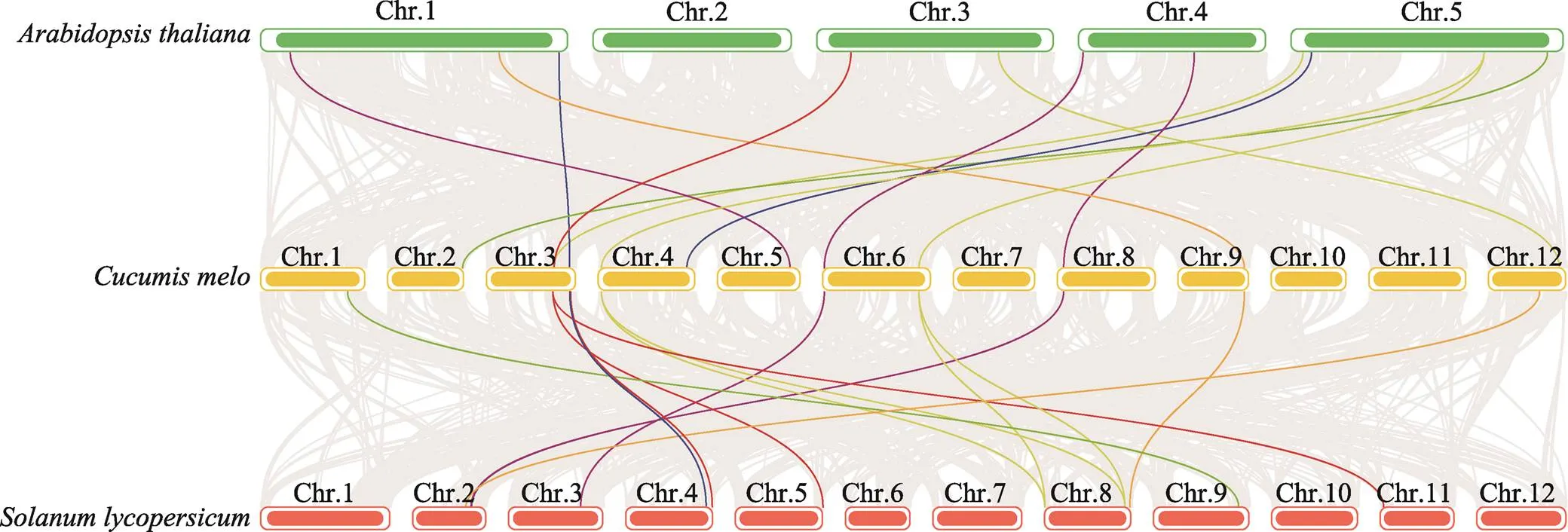
图7 甜瓜、拟南芥和番茄之间HDM基因共线性分析
浅灰色线条表示甜瓜基因组分别与拟南芥和番茄基因组之间的共线性区块;不同彩色线条表示共线性区块中LSD1和JmjC不同亚族基因的位置(各亚族基因所用的线条颜色与图1所用颜色对应)。
2.6 甜瓜HDM基因表达模式分析
基因在植物不同组织的表达特性能够很好的揭示其在生长发育过程中发挥的生物学功能,本研究通过转录组数据对甜瓜基因在雌花、雄花、成熟果实、根、茎、叶和子房等组织的表达模式进行分析(图8)。结果发现,20个基因在甜瓜的不同组织部位均存在不同水平的表达(附表2)。其中,、、、和在甜瓜的不同组织中均具有较高的表达水平,而在各个组织的表达水平相对较低。在雌花中的表达量最高,和次之,而在雄花中的表达量最高,和次之,这些基因在甜瓜的花器官发育过程中发挥功能,并且发现CmLSD1基因家族成员可能主要参与甜瓜花器官的调控过程。在甜瓜的茎和叶表达水平较高,仅在根中表达较高,在其他组织表达较低。、和在子房中有显著表达,推测这些基因在子房发育时期影响甜瓜果实的发育。和较其他基因在成熟果实中有较高的表达水平,推测它们在甜瓜果实成熟过程中起着重要作用。以上结果表明,基因在参与甜瓜的生长发育过程中发挥了不同程度的作用。
3 讨论
组蛋白甲基化和去甲基化修饰在基因表达的调控中起关键作用,主要受组蛋白甲基转移酶和组蛋白去甲基化酶的动态调控,能够通过修饰不同位点或不同程度的甲基化水平来调控相应基因的表达。本研究从甜瓜全基因组中鉴定到20个基因(3个和17个),这些基因在9条染色体上非均匀分布,相对于拟南芥(4个和21个)[32]、番茄(5个和20个)[6]成员数量较少。参照拟南芥HDM系统进化树分类,甜瓜HDM被分成LSD1和JmjC两大类,而JmjC类群又被划分为KDM3/JHDM2、KDM4/ JHDM3、KDM5/JARID1、JMJD6和JmjC domain-only五类亚群,JMJD6类群成员与拟南芥和番茄数量一致,而其他类群成员数量略有差异,这可能与JMJD6类群在进化中高度保守有关。在基因复制事件中,甜瓜HDM家族仅存在一对片段重复,而无串联重复事件,表明基因在进化过程中具有相对独立性。甜瓜基因结构稳定,LSD1蛋白类群均含有SWIRM和Amino oxidase结构域,JmjC蛋白亚家族都含有特定的JmjC结构域,其生物学功能的发挥还依靠于其他结构域,并且具有底物特异性。
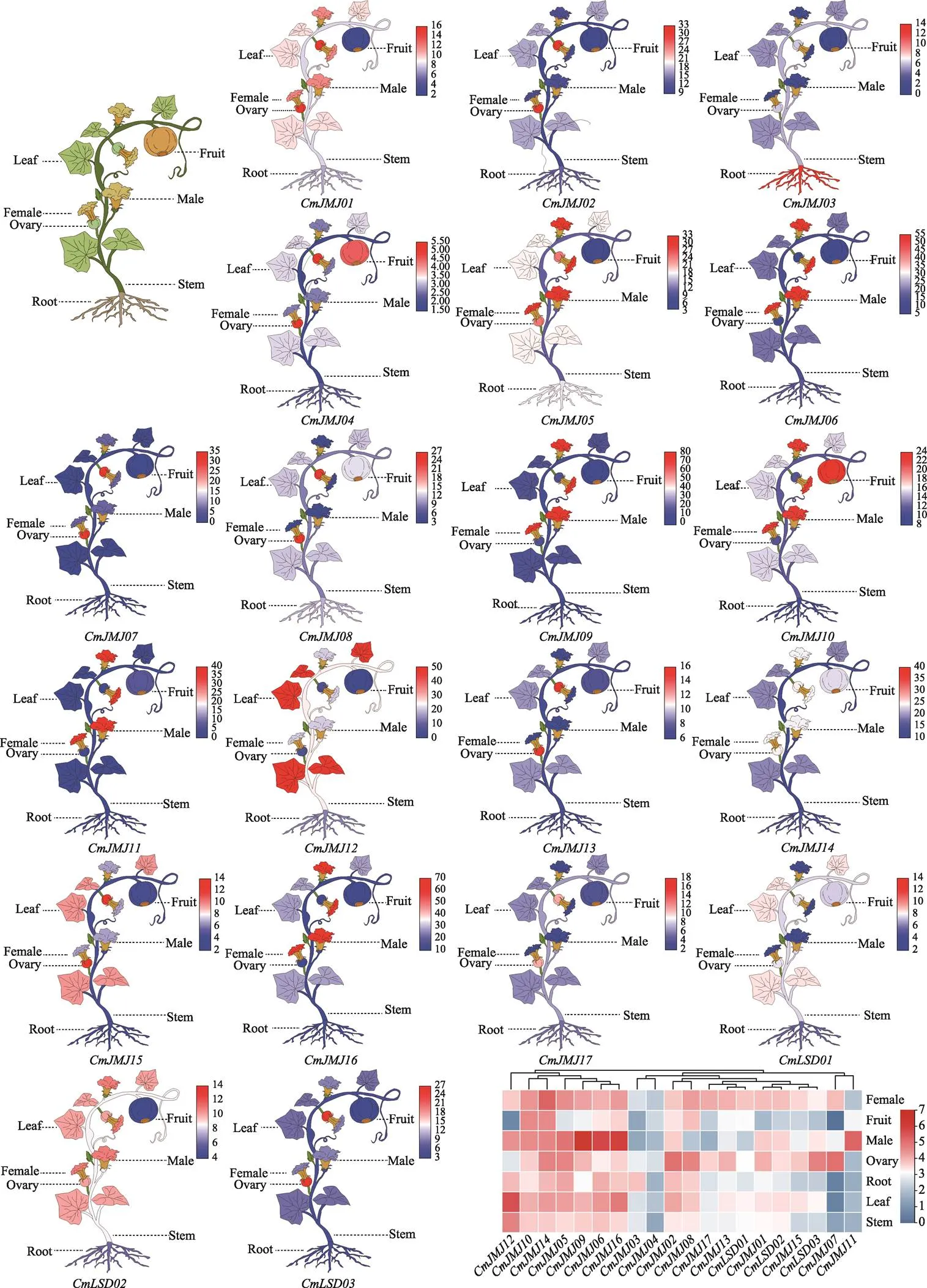
图8 甜瓜HDM基因在不同组织的表达分析
采用植株模拟卡通图和热图两种形式展示甜瓜基因在根、茎、叶、雌花、雄花、子房、成熟果实中的表达情况。红色表示表达量高,蓝色表示表达量低。
在植物生长和发育过程中灵活而稳健的基因表达和对环境的反应主要受到表观遗传的调控,而组蛋白去甲基化修饰在这个过程中扮演了重要的角色,主要影响植物花期、叶片的衰老、生物节律调控、体外芽的再生、茎的伸长等过程。启动子上游的顺式作用元件能够参与基因表达的调控,从而影响植物的生长发育以及植物对逆境(生物和非生物胁迫)的响应。本研究分析发现,甜瓜HDM基因家族具有一些与ABA、GA等激素及光、干旱等环境信号响应的元件,表明基因可能在参与调控甜瓜的非生物胁迫中发挥了不可或缺的作用,也为后续开展基因通过激素介导和环境信号胁迫调节甜瓜生长发育研究提供了思路。甜瓜HDM基因家族在根、茎、叶、雄花、雌花、子房以及果实中均有不同程度表达,说明在不同组织部位中发挥着不同的功能。CmLSD1基因家族成员在花组织表达比较集中,可能参与对甜瓜花器官的调控,这与拟南芥LSD1家族的同源蛋白LDL1和LDL2的功能相似,具有H3K4去甲基化酶催化活性[33]并能够影响植株开花。、等基因在花中表达量较高,推测在甜瓜的花器官发育中发挥重要作用,如水稻能参与调控花的形态发育和花器官数目的变化[34]。在叶和茎中表达较高,而拟南芥能够正向调节叶片衰老[22],水稻可以通过H3K4me1/2/3去甲基化调节茎的伸长和植株的生长[35],推测基因可能具有相似的功能。
肉质果实成熟是一个独特的发育过程。果实成熟过程是由内源激素(如乙烯)和遗传调控因子(如转录因子)驱动完成,然而,目前对肉质果实成熟的关键和早期调节因子还知之甚少。2020年,Li等[6]首次报道,是一种专一性的H3K27me3组蛋白去甲基化酶,通过对一些成熟相关基因的去甲基化,从而正向调控番茄果实成熟。随后,Ding等[36]发现是番茄果实成熟的负调控因子,通过H3K4me3去甲基化直接调控一组关键的成熟相关基因,包括乙烯生物合成基因(、和)、转录调控因子(RIN和NOR)、品质形成相关基因(、、、、和等),从而调控果实成熟。同样,本研究在甜瓜转录组数据中发现、和在子房组织中的表达量显著高于其他组织,猜测这些基因可能在子房发育时期影响甜瓜果实的发育。、较其他基因在成熟果实中有较高的表达水平,可能在参与甜瓜果实成熟中发挥重要的作用。
HDM在参与调控植物营养生长和环境自适中发挥了关键作用,但在不同类型植物的生长发育和抗逆抗病的功能方面还有待研究。目前,关于HDM在植物生长发育调控和抗逆抗病的分子机制及途径尚未明朗,因而解析这些机理过程对植物生长发育和环境自适尤为必要。通过对甜瓜HDM基因家族的特性分析,将为后期研究基因如何动态调控甜瓜生长发育以及抗逆胁迫响应机制奠定基础。
附加材料见文章电子版www.chinagene.cn。
[1] Qian SZ, Wang YX, Ma H, Zhang LS. Expansion and functional divergence of Jumonji C-containing histone demethylases: significance of duplications in ancestral angiosperms and vertebrates., 2015, 168(4): 1321–1337.
[2] Luger K, Richmond TJ. The histone tails of the nucleosome., 1998, 8(2): 140–146.
[3] Chang YN, Zhu C, Jiang J, Zhang HM, Zhu JK, Duan CG. Epigenetic regulation in plant abiotic stress responses., 2020, 62(5): 563–580.
[4] Suganuma T, Workman JL. Crosstalk among histone modifications., 2008, 135(4): 604–607.
[5] Qian YX, Chen CL, Jiang LY, Zhang J, Ren QY. Genome-wide identification, classification and expression analysis of the JmjC domain-containing histone demethylase gene family in maize., 2019, 20(1): 1–16.
[6] Li ZW, Jiang GX, Liu XC, Ding XC, Zhang DD, Wang XW, Zhou YJ, Yan HL, Li TT, Wu KQ, Jiang YM, Duan XW. Histone demethylase SlJMJ6 promotes fruit ripening by removing H3K27 methylation of ripening-related genes in tomato., 2020, 227(4): 1138–1156.
[7] Zhang Y, Reinberg D. Transcription regulation by histone methylation: interplay between different covalent modifications of the core histone tails., 2001, 15(18): 2343–2360.
[8] Chang BS, Chen Y, Zhao YM, Bruick RK. JMJD6 is a histone arginine demethylase., 2007, 318(5849): 444–447.
[9] Liu CY, Lu FL, Cui X, Cao XF. Histone methylation in higher plants., 2010, 61(1): 395–420.
[10] Allis CD, Berger SL, Cote J, Dent S, Jenuwien T, Kouzarides T, Pillus L, Reinberg D, Shi Y, Shiekhattar R, Shilatifard A, Workman J, Zhang Y. New nomenclature for chromatin-modifying enzymes., 2007, 131(4): 633–636.
[11] Strahl BD, Ohba R, Cook RG, Allis CD. Methylation of histone H3 at lysine 4 is highly conserved and correlates with transcriptionally active nuclei in tetrahymena., 1999, 96(26): 14967–14972.
[12] Martin C, Zhang Y. The diverse functions of histone lysine methylation., 2005, 6(11): 838–849.
[13] Tsukada Y, Fang J, Erdjument-Bromage H, Warren ME, Borchers CH, Tempst P, Zhang Y. Histone demethylation by a family of JmjC domain-containing proteins., 2006, 439(7078): 811–816.
[14] Shi YJ, Lan F, Matson C, Mulligan P, Whetstine JR, Cole PA, Casero RA, Shi Y. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1., 2004, 119(7): 941–953.
[15] Forneris F, Binda C, Vanoni MA, Mattevi A, Battaglioli E. Histone demethylation catalysed by LSD1 is a flavin- dependent oxidative process., 2005, 579(10): 2203–2207.
[16] Accari SL, Fisher PR. Emerging roles of JmjC domain-containing proteins., 2015, 319 (2015): 165–220.
[17] Klose RJ, Kallin EM, Zhang Y. JmjC domain-containing proteins and histone demethylation., 2006, 7(9): 715–727.
[18] Dong YW, Lu J, Liu JY, Jalal A, Wang CQ. Genome-wide identification and functional analysis of JmjC domain- containing genes in flower development of., 2020, 102(4–5): 417–430.
[19] Lu FL, Li GL, Cui X, Liu CY, Wang XJ, Cao XF. Comparative analysis of JmjC domain-containing proteins reveals the potential histone demethylases inand rice., 2008, 50(7): 886–896.
[20] Yang HC, Han ZF, Cao Y, Fan D, Li H, Mo HX, Feng Y, Liu L, Wang Z, Yue YL, Cui SJ, Chen S, Chai JJ, Ma LG. A companion cell-dominant and developmentally regulated H3K4 demethylase controls flowering time invia the repression of FLC expression., 2012, 8(4): 1002664.
[21] Lu FL, Cui X, Zhang SB, Liu CY, Cao XF. JMJ14 is an H3K4 demethylase regulating flowering time in., 2010, 20(3): 387–390.
[22] Wang XL, Gao J, Gao S, Song Y, Yang Z, Kuai BK. The H3K27me3 demethylase REF6 promotes leaf senescence through directly activating major senescence regulatory and functional genes in, 2019, 15(4): 1008068.
[23] Liu P, Zhang SB, Zhou B, Luo X, Zhou XF, Cai B, Jin YH, Niu D, Lin JX, Cao XF, Jin JB. The histone H3K4 demethylase JMJ16 represses leaf senescence in., 2019, 31(2): 430–443.
[24] Lu SX, Knowles SM, Webb CJ, Celaya RB, Cha C, Siu JP, Tobin EM. The Jumonji C domain-containing protein JMJ30 regulates period length in thecircadian clock., 2011, 155(2): 906–915.
[25] Li W, Liu H, Cheng ZJ, Su YH, Han HN, Zhang Y, Zhang XS. DNA methylation and histone modifications regulateshoot regeneration inby modulating WUSCHEL expression and auxin signaling., 2011, 7(8): 1002243.
[26] Chen QF, Chen XS, Wang Q, Zhang FB, Lou ZY, Zhang QF, Zhou DX. Structural basis of a histone H3 lysine 4 demethylase required for stem elongation in rice., 2013, 9(1): 1003239.
[27] Yamaguchi N, Matsubara S, Yoshimizu K, Seki M, Hamada k, Kamitani M, Kurita Y, Nomura Y, Nagashima K, Inagaki S, Suzuki T, Gan ES, To T, Kakutani T, Nagano AJ, Satake A, Ito T. H3K27me3 demethylases alter HSP22 and HSP17.6C expression in response to recurring heat in., 2021, 12(1): 3480.
[28] Song T, Zhang Q, Wang HQ, Han JB, Xu ZQ, Yan SN, Zhu ZG. OsJMJ703, a rice histone demethylase gene, plays key roles in plant development and responds to drought stress., 2018, 132: 183–188.
[29] Shin AY, Kim YM, Koo N, Lee SM, Nahm S, Kwon SY. Transcriptome analysis of the oriental melon (Lvar.) during fruit development., 2017, 5: 2834.
[30] Wu B, Wang L, Pan GY, Li T, Li X, Hao JH. Genome- wide characterization and expression analysis of the auxin response factor (ARF) gene family during melon (L) fruit development., 2020, 257(3): 979–992.
[31] Han YP, Li XY, Cheng L, Liu YC, Wang H, Ke DX, Yuan HY, Zhang LS, Wang L. Genome-wide analysis of soybean JmjC domain-containing proteins suggests evolutionary conservation following whole-genome duplication., 2016, 7: 1800.
[32] Gu TT, Han YH, Huang RR, McAvoy RJ, Li Y. Identification and characterization of histone lysine methylation modifiers in.,2016, 6: 23581.
[33] Jiang DH, Yang WN, He YH, Amasino RM.relatives of the human lysine-specific demethylase1 repress the expression of FWA and FLOWERING LOCUS C and thus promote the floral transition., 2007, 19(10): 2975–2987.
[34] Sun QW, Zhou DX. Rice jmjC domain-containing gene JMJ706 encodes H3K9 demethylase required for floral organ development., 2008, 105(36): 13679–13684.
[35] Cui XK, Jin P, Cui X, Gu LF, Lu ZK, Xue YM, Wei LY, Qi JF, Song XW, Luo M, An G, Cao XF. Control of transposon activity by a histone H3K4 demethylase in rice., 2013, 110(5): 1953–1958.
[36] Ding XC, Liu XC, Jiang GX, Li ZW, Song YB, Zhang DD, Jiang YM, Duan XW. SlJMJ7 orchestrates tomato fruit ripening via crosstalk between H3K4me3 and DML2- mediated DNA demethylation., 2022, 233(3): 1202–1219.
Identification and characterization analysis of the HDM gene family in melon (L.)
Zhi Zhang, Jing Zhang, Jin Zhang, Agula Hasi, Jinfeng Hao
Histone demethylase (HDM) play crucial roles in regulating plant growth and environmental adaptation. In this study, the HDM gene family in melon was identified by bioinformatics methods and the expression patterns of theCmHDM family members in different melon tissues were analyzed using transcriptome data. The results showed that 20genes were identified in the melon genome, which were unevenly distributed across each chromosome. These members fall into two major categories: LSD1 and JmjC. The JmjC group could be further divided into five subgroups with different numbers. The results of collinearity analysis of intraspecific and interspecific relationships showed that there were only one pair of segmental duplication in melongenes, and more collinearity in genetic relationship ofgenes between melon and tomato. The numbers of conserved domains, exons and introns in each member vary and various-acting elements responding to hormones and environmental signals existed in the respective promoter regions. Expression analysis showed that the respective gene members were expressed at different levels in male flowers, female flowers, roots, stems, leaves, ovary, and mature fruits of melon. These results will contribute to the understanding on the potential functions of thegenes and their potential functions in regulating melon growth and environmental adaptation.
melon; HDM family; gene identification; expression analysis
2023-08-23;
2023-09-25;
2023-10-25
内蒙古自然科学基金项目(编号:2020MS03025)资助[Supported by the Inner Mongolia Natural Science Foundation (No.2020MS03025)]
张治,硕士研究生,专业方向:植物学。E-mail: 1819296047@qq.com
郝金凤,博士,副研究员,硕士生导师,研究方向:植物分子生物学及基因工程。E-mail: haojindd@163.com
10.16288/j.yczz.23-226
(责任编委: 许勇)