荧光RNA及其生物传感技术研究进展
2024-02-22左方婷张雅强杨慧敏杨弋陈显军
左方婷,张雅强,杨慧敏,杨弋,陈显军
特邀综述
荧光RNA及其生物传感技术研究进展
左方婷1,2,3,张雅强1,2,3,杨慧敏1,2,杨弋1,2,陈显军1,2
1. 华东理工大学光遗传学与合成生物学交叉学科研究中心,生物反应器工程国家重点实验室,上海 200237 2. 华东理工大学药学院,上海市细胞代谢光遗传学技术前沿科学研究基地,上海 200237 3. 华东理工大学生物工程学院,上海 200237
荧光RNA技术是一种新兴的RNA标记技术,可用于活细胞RNA的原位实时标记与成像,对于人们理解RNA的功能和调控机制发挥着至关重要的作用。基于荧光RNA的生物传感技术可用于活细胞内小分子代谢物以及蛋白质等靶标的实时动态检测,为生命科学基础研究以及生物医学传感技术开发提供极具价值的工具。本文对遗传编码的荧光RNA的发展历程、荧光RNA技术在活细胞RNA成像,以及基于荧光RNA的生物传感技术在活细胞代谢物检测等方面的应用进行了介绍和总结,并对该领域的发展现状和未来发展方向展开讨论和展望,以期为该技术的进一步发展和在相关领域的应用提供参考。
荧光RNA;荧光标记;RNA成像;生物传感
光遗传学可被视作研究光信号与生物信号交互作用的学科,而遗传编码荧光标记技术可帮助人们解析活细胞中生命的动态过程。绿色荧光蛋白(green fluorescent protein, GFP)是人类发现的第一个天然的荧光蛋白,1962年由日本化学家、海洋生物学家Osamu Shimomura从维多利亚水母()中分离提取[1]。1992年,美国分子生物学家Douglas C. Prasher等[2]完成对其基因序列的测定。1994年,美国科学家Martin Chalfie等[3]在大肠杆菌()、线虫()中成功实现了GFP的异源表达,首次证明GFP可以作为遗传编码的荧光标签。此后,研究者也发现了来源于其他物种的典型荧光蛋白,包括俄罗斯科学院生物有机化学研究所Sergey A. Lukyanov课题组发现的来源于香菇珊瑚()的红色荧光蛋白DsRed[4]等。美国加州大学钱永健课题组进一步优化荧光蛋白的生化和荧光性质,拓宽了荧光蛋白的应用范围[5~7]。荧光蛋白的出现,为活细胞蛋白质标记带来革命性突破,已经成为当代生命科学研究中最重要的工具之一。近年来也涌现出一些基于黄素单核苷酸(flavin mononucleotide, FMN)、藻蓝胆素(phycocyanobilin)、胆绿素(biliverdin, BV)、胆红素(bilirubin, BR)等配体结合的新型非典型荧光蛋白,如与BV结合后荧光增强的近红外荧光蛋白(infrared-fluorescent proteins, IFP)系列[8~10],与BR结合后产生非氧依赖性绿色荧光的UnaG[11]等。此外,基于蛋白质标签和激活型荧光染料的类荧光蛋白如SNAP-tag[12]、Halo-tag[13]、Y-FAST-tag[14]也发展迅速,它们结合了蛋白质可遗传编码以及小分子荧光染料在荧光亮度、光稳定性等方面的优势,进一步拓宽了遗传编码荧光标记技术的应用范围。
与蛋白质相比,细胞内的RNA种类更多、功能更为复杂。发展可以对活细胞RNA特异标记与成像的技术,将允许人们实时监测RNA在活细胞中各种代谢行为,如生成、运输、翻译、降解等,这是深入研究RNA功能机制极为有用的工具和所需要解决的重要技术挑战。RNA研究领域也迫切需要类似于荧光蛋白这样操作方便、简单可行的活细胞RNA标记技术。自然界中存在天然的荧光蛋白,但是人们尚未在自然界中发现天然存在的荧光RNA。受荧光蛋白和类荧光蛋白的启发,研究者发展了荧光RNA(fluorescent RNA)技术,其工作原理为RNA适配体与荧光团分子高特异性、高亲和力结合,当将RNA适配体与目标RNA融合,在细胞中表达并加入荧光团孵育,通过特定波长的光激发后发出明亮的荧光实现对目标RNA的成像。RNA适配体可以利用指数富集的配体系统进化技术(systematic evolution of ligands by exponential enrichment, SELEX)[15~17]筛选获得。相比于传统遗传编码的荧光蛋白-RNA结合蛋白技术,荧光RNA技术具有标记更加直接、对靶标RNA干扰小以及成像信噪比高的优势,将为人们理解活细胞中RNA的功能和作用提供实用工具。本文将对遗传编码的荧光RNA技术及基于荧光RNA的生物传感技术进行重点介绍,包括荧光RNA的发展历程、在活细胞RNA成像及代谢物检测方面的应用,同时还将探讨该技术未来的发展方向以及面临的挑战,以期为该技术的进一步发展和在相关领域的应用提供参考。
1 荧光RNA的发展历程
荧光蛋白可在基因水平上进行遗传编码,并在自身生化反应机制下产生荧光。经典的绿色荧光蛋白GFP由238个氨基酸组成,三个氨基酸残基Ser65-Tyr66-Gly67自催化形成生色团HBI,被包裹在由11个β折叠环绕而成的桶状结构中[18](图1A)。此外,科学家们也发现含外源生色团的荧光蛋白,如来源于鳗鱼以胆红素BR为生色团的UnaG[11](图 1B)等。无论是蛋白自身催化产生生色团还是外源生色团,它们本质上都属于分子转子型荧光团,被蛋白质骨架固定住(经高温加热或其他方式变性后的荧光蛋白不再具有荧光[19]),从而限制分子内的电荷转移,进而产生荧光。因此,科学家们基于类似的原理,利用RNA适配体的结构骨架来固定分子转子型荧光团,发展了系列荧光RNA (图1, C~E)。
1.1 概念期的荧光RNA
2003年,因发展荧光蛋白技术而获得诺贝尔奖的钱永健教授,开发了首个基于小分子染料祖母绿(malachite green, MG)的“荧光团-RNA适配体”复合物(图2A)。他们对MG荧光团进行SELEX筛选,获得了可以特异性结合MG的RNA适配体。MG是一种分子转子型荧光团,当处于自由溶液中受激发时,获得的能量主要通过围绕结构中的乙炔桥进行类似于“呼啦圈旋转”的运动方式非辐射衰减,所以其受激发时不会发出荧光。当MG嵌合在RNA适配体形成的空间结构中时,MG中的乙炔桥被固定,因而只能通过发出荧光辐射衰减。不过MG是一种三苯甲烷染料,荧光辐照后会产生活性氧(reactive oxygen species, ROS)自由基对细胞有明显的毒性[20,21],因此该复合物并不能应用于活细胞研究。
一些研究者利用核酸染料进行荧光RNA的开发,如基于Hoechst开发的II-mini3-4-Hoechst 1c蓝色荧光RNA[22]、基于二甲基吲哚红(dimethylindoline, DIR)开发的DIR-Apt1红色荧光RNA[23]、DIR2s-Apt系列荧光RNA[24]等。由于核酸染料嵌入DNA双螺旋结构后发出荧光,这导致该类荧光RNA存在很高的背景荧光,难以用于活细胞RNA的标记与成像。另外,加拿大西蒙菲莎大学Peter J. Unrau课题组基于花菁类核酸染料噻唑橙(thiazole orange, TO)开发了Mango系列[25~27]和Peach[28]荧光RNA。TO1-Biotin荧光团及衍生物由于分子量大,需要借助显微注射等方式导入细胞内。此外,核酸染料还存在一定的致癌风险。因此,该类荧光RNA并不适用于活细胞RNA标记。
荧光团-猝灭基团偶联物也被用于发展荧光RNA,RNA适配体对偶联物中荧光团或猝灭基团的特异性结合会解除猝灭基团对荧光团的猝灭作用,进而使其恢复荧光(图2B)。基于该设计思路,2011年,日本京都大学Motonari Uesugi课题组开发了基于BHQ1猝灭基团的A1系列荧光RNA[29]。德国海德堡大学Andres Jäschke课题组基于类似思路,陆续发展了DNB系列荧光RNA[30]、SRB-2-TMR-DN荧光RNA[31]和SR-DN-SRB-2荧光RNA[32]。2018年,美国科罗拉多大学Amy E. Palmer课题组报道了另一类基于钴胺素(Cbl, 也称维生素B12)核糖开关的Cbl系列荧光RNA[33]。2020年,法国斯特拉斯堡大学Michael Ryckelynck课题组基于荧光团二聚反应自猝灭的原理,开发了基于SRB二聚体染料Gemini-561的荧光RNA[34]。不过,该类型荧光团配体由于分子量大,很难自由进入细胞,如Cbl荧光团需要借助微珠碾压的方式破坏细胞膜结构导入,因此该类型荧光RNA并不便于活细胞RNA研究。
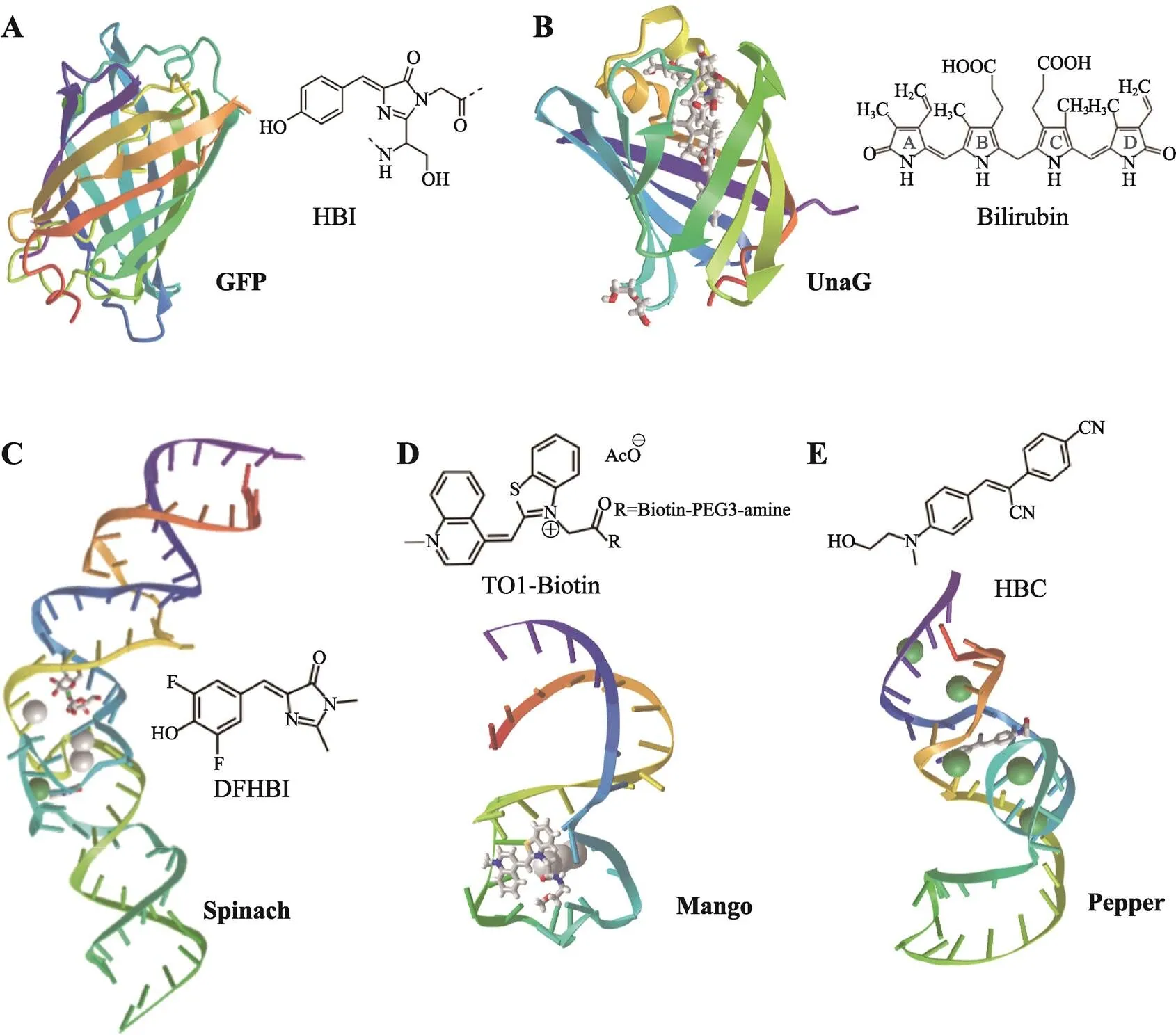
图1 荧光蛋白与荧光RNA晶体结构
A:GFP荧光蛋白晶体结构,氨基酸残基Ser65-Tyr66-Gly67自催化形成生色团HBI。B:UnaG荧光蛋白晶体结构,胆红素(bilirubin, BR)为生色团。C:Spinach荧光RNA晶体结构,DFHBI为荧光团。D:Mango荧光RNA晶体结构,TO1-Biotin为荧光团。E:Pepper荧光RNA晶体结构,HBC为荧光团。晶体结构示意图来源于美国国家生物技术信息中心(national center for biotechnology information, NCBI)网站,PDB ID依次为4OGS、4I3B、4TS2、6C63和7SZU (https://www.ncbi.nlm.nih.gov/Structure/icn3d)。
2011年,美国康奈尔大学Samie R. Jaffrey课题组以GFP生色团HBI为基础进行结构修饰获得DMHBI荧光团,并筛选优化获得Spinach适配体。Spinach-DFHBI荧光RNA在受激发时可发出绿色荧光[35]。这是第一例可以应用于活细胞RNA成像的荧光RNA,后续的研究也表明其可应用于不同类型的活细胞,包括细菌[36]、酿酒酵母[37,38]、哺乳动物细胞[39]、衣藻叶绿体[40]中RNA的标记与成像。随后,他们对荧光团优化获得DFHBI-1T[41]、DFHBI-2T[41],对RNA适配体优化获得“超折叠”的Spinach2[42]、Broccoli[43]、dBroccoli[43],提高了该类荧光RNA在活细胞中的荧光强度和稳定性。此外,还有一些研究者完成对Spinach-DFHBI复合物晶体结构的解析,并优化得到结构更为小巧的Baby Spinach RNA适配体[44];或通过微流控辅助的随机突变方法获得离子依赖性更低的iSpinach RNA适配体[45,46]。2017年,Jaffrey课题组以红色荧光蛋白生色团DsRed为基础开发了DFHO荧光团,并筛选获得二聚化的Corn-DFHO荧光RNA[47]。此后,他们优化获得Red Broccoli-OBI[48],筛选获得与DFHBI-1T和DFHO都能结合的Squash RNA适配体[49],基于DFHO优化获得DFAME荧光团并筛选获得与Corn具有相似结构的Beetroot[50]。2019年,德国维尔茨堡大学Claudia Höbartner课题组对HBI修饰获得DMHBI- Imi、DMHBI+和DMHBO+,它们均可被类似于Spinach结构的RNA适配体Chili结合并激活[51]产生大斯托克斯位移(large Stokes shift)荧光。该类荧光RNA的荧光团分子量小、柔性大,因此具有跨膜容易、无细胞毒性、背景荧光低等优点,但这种线性结构的荧光团很难被RNA适配体牢牢固定,导致它们与RNA适配体的亲和力较低,需要使用高浓度的荧光团才能实现有效标记。更值得注意的是,这些荧光RNA的活细胞亮度较低,部分还会因发生快速光异构化而具有较差的光稳定性。上述这些不足大大限制了这类荧光RNA的广泛应用。
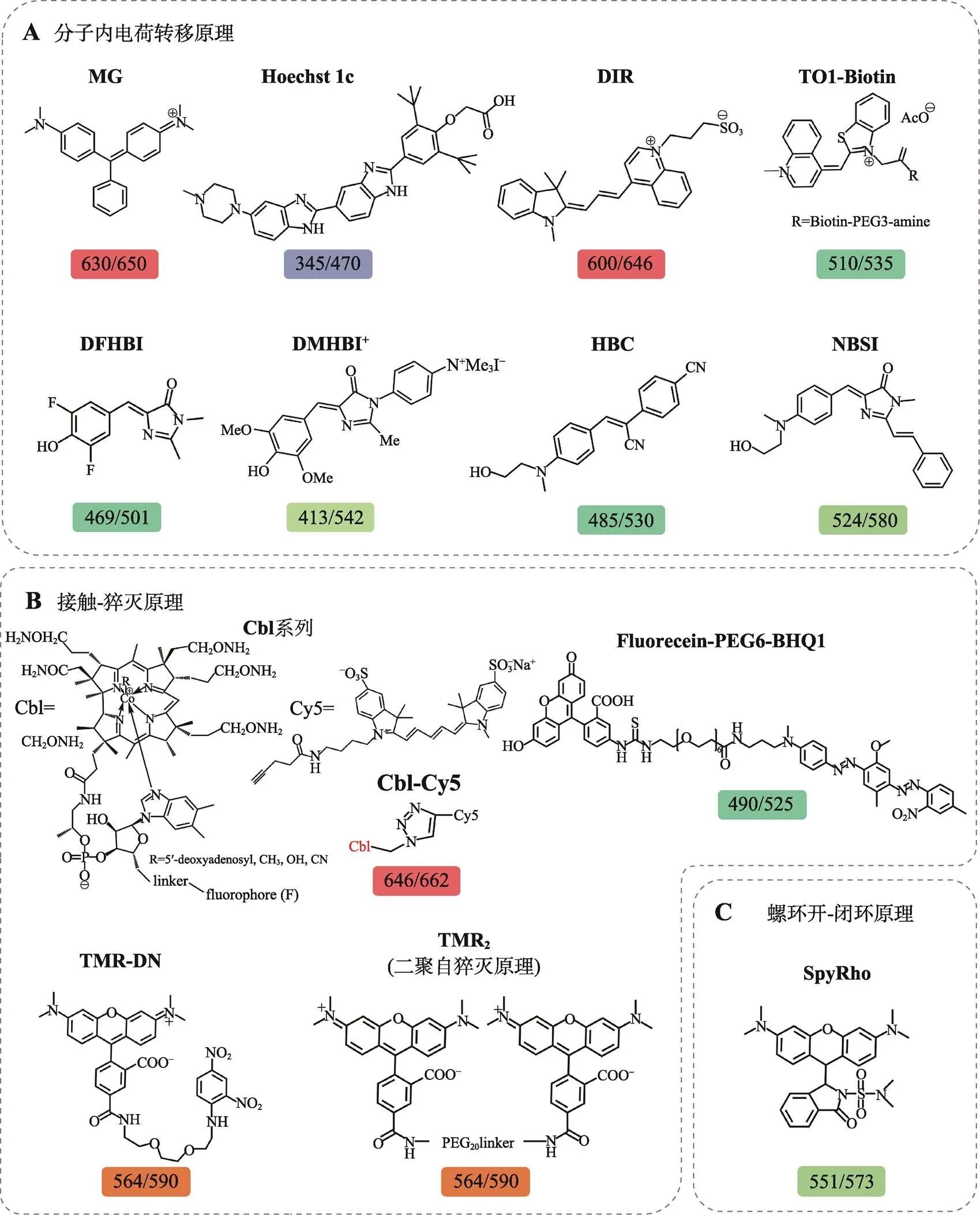
图2 不同荧光RNA识别的荧光团结构及荧光RNA光谱信息
A:基于分子内电荷转移原理。B:基于接触-猝灭原理。C:基于螺环开-闭环原理。方框标注“RNA适配体-荧光团复合物”的最大激发波长和发射波长,单位nm。
此外,近些年还发展了一类基于螺环结构开-闭环原理(spirolactonization)的荧光RNA(图2C)。通过将开环-闭环的平衡态转化为开环的荧光状态,从而增加螺旋内酯化硅罗丹明染料的荧光。德国海德堡大学Andres Jäschke课题组以硅罗丹明SiR为荧光团筛选优化获得了SiRA RNA适配体,并利用SiRA-SiR复合物实现中RNA的受激发射损耗(stimulated emission depletion microscopy, STED)超分辨成像[52]。此后,该课题组成员发展了光谱可调谐的BeCA-BC系列荧光RNA[53]。不过该类荧光RNA由于背景荧光较高,因此在应用到高等生物细胞时仍然存在较大挑战。
总体上看,基于核酸染料和荧光团-猝灭基团偶联物的荧光RNA存在染料进细胞困难、背景荧光高、明显的细胞毒性等缺点,而基于荧光团衍生物的荧光RNA存在亲和力弱、细胞亮度低、稳定性差、缺乏生物正交性和/或多聚体等不足,这些问题限制了概念期荧光RNA的应用。
1.2 高性能实用荧光RNA
相比于蛋白质,活细胞内的RNA具有显著缩短的半衰期(细菌中mRNA的平均半衰期只有2.5~ 8.0 min[54~56],哺乳动物细胞中mRNA的平均半衰期也只有2~10 h[57]),导致活细胞中RNA的丰度远低于蛋白质,这就对RNA的标记和成像工具提出了更高的要求。理想的荧光RNA应该具备以下特点:(1) RNA适配体为单体且不含G-四链体。RNA的二聚化可能会改变目标RNA的代谢,而RG4结构可被活细胞解旋酶靶向[58],导致RNA的解旋或降解;(2) 荧光团具有背景荧光低、结合RNA适配体后荧光激活倍数高、进细胞容易、细胞毒性低等特点;(3) RNA适配体在高等动物细胞中能够正确表达折叠,RNA适配体与荧光团亲和力强,产生明亮且稳定的荧光,以满足对目标RNA的高亮度和长时间动力学追踪。荧光RNA只有在各方面的性能均取得突破性进展后,才可能在活细胞内对各类RNA进行简单方便的标记与成像,实现荧光RNA技术的普遍应用(表1)。
1.2.1 Pepper-HBC系列荧光RNA
2019年,本团队基于全新的分子设计理念开发了一类创新的HBC荧光团分子,它们结构简单、分子量小,同时具有树枝状、刚性结构,理论上会相对容易被RNA结合固定。HBC荧光团在自由溶液中不发光,具备荧光蛋白生色团无背景的优势。研究者基于HBC荧光团筛选获得与之高度特异结合的RNA适配体,对其优化最终获得长度为43 nt的Pepper RNA适配体;对HBC荧光团进行分子结构上的光谱调谐,获得光谱涵盖青、绿、橙、红等不同颜色的8种系列荧光RNA[59](因其具有多种颜色与辣椒类似,因此被命名为Pepper)。进一步结果表明,Pepper以单体且非G-四链体的结构形式、以纳摩尔级的高亲和力与荧光团分子结合,且具有高温度稳定性、强pH耐受性,以及对外源序列融合插入的高度兼容性。Pepper的这些优良性质使得它在细胞中的荧光亮度、激活倍数比其他荧光RNA高出至少一个数量级,亲和力与稳定性高出两到三个数量级。此外,研究团队发现将多个Pepper以串联方式融合,这种串联体的单分子荧光强度会随拷贝数的增加呈等比例上升,因此可将成像检测灵敏度又提高一个数量级。Pepper系列荧光RNA的出现,应该说是荧光RNA从概念到实用的一个突破。
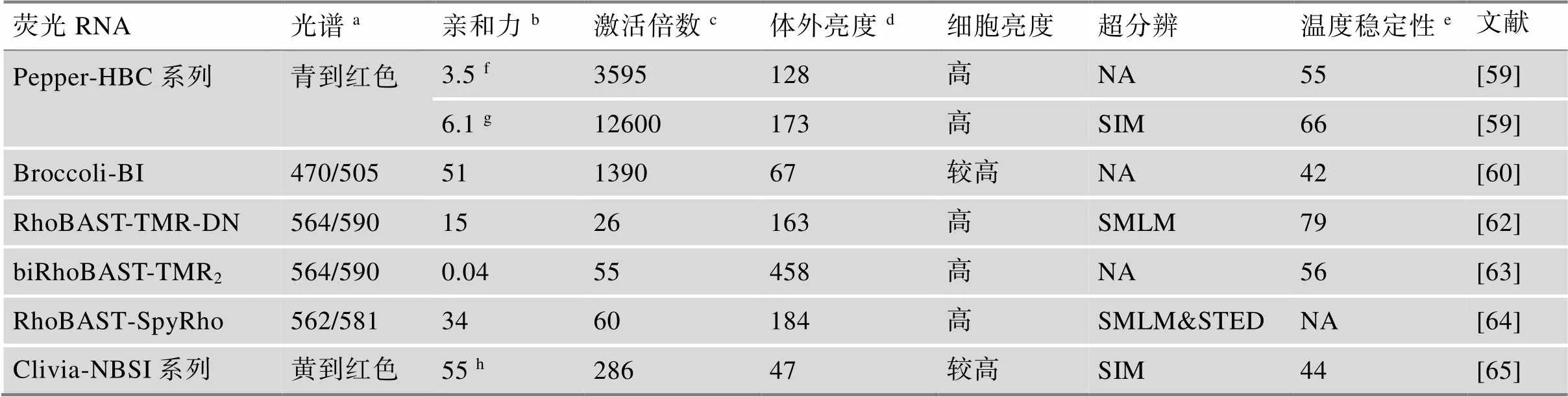
表1 哺乳动物细胞实用的荧光RNA
a光谱单位nm;b亲和力单位nmol/L;c体外激活倍数;d体外亮度[量子产率×摩尔消光系数]并根据EGFP为100校正;e温度稳定性单位为℃;f指Pepper530 (485/530);g指Pepper620 (577/620);h指Clivia580 (524/580);NA,not available,未获得相应数据。
Pepper应用于活细胞RNA成像时,研究者可将Pepper融合到不同类型的RNA分子序列中,实现活细胞中对目标RNA的荧光标记与示踪。其中,Pepper不仅可被用于细胞中表达丰度较高的5S、7SK等非编码RNA的活细胞成像(图3A),还可用于表达水平更低的mRNA的无背景成像(图3B),而不影响这些RNA的转录、定位、翻译、降解等正常代谢。利用Pepper荧光RNA标记活细胞中不同种类目标RNA,与利用经典的荧光原位杂交(fluorescencehybridization, FISH)技术标记固定细胞中相同目标RNA的结果一致,证明荧光RNA标记技术的准确性。得益于Pepper具有活细胞RNA无背景定量检测的特性,研究团队在国际上首次利用Pepper对单细胞中mRNA的翻译过程进行了大规模定量研究。结果显示癌细胞中mRNA量与其编码蛋白质量之间存在较低相关性,这表明癌细胞的翻译调控显著失调,这为癌症的诊疗提供一种全新的思路。此外,Pepper还可与CRISPR技术结合起来,通过sgRNA展示的方法利用Pepper对基因组特定位点进行活细胞成像。由于Pepper与荧光团分子非共价结合,因此可以通过加入不同荧光团的方法,灵活改变细胞内荧光RNA的颜色,对于多色荧光标记稳定细胞株和转基因动物的构建十分有利。得益于良好的光稳定性,Pepper620荧光RNA首次实现了红色荧光RNA用于活细胞内的结构光照明(structure illumination microscopy, SIM)超分辨成像,成像分辨率显著提高。Pepper系列荧光RNA为活细胞中研究目标RNA的功能提供了极具价值的工具。
1.2.2 Broccoli-BI系列荧光RNA
2020年,美国康奈尔大学Samie R. Jaffrey课题组在DFHBI-1T基础上优化获得BI[60]、TBI[61]等荧光团,大幅度提高了该类荧光RNA在活细胞中的光稳定性和亮度。他们利用优化的Broccoli-BI荧光RNA实现了活细胞中的单分子成像[60]。不过,Broccoli适配体结构中含有G-四链体。而且,受限于荧光蛋白生色团固有的特点,该类荧光RNA在活细胞中仍然存在荧光猝灭的现象,光稳定性和荧光强度仍有待提升。
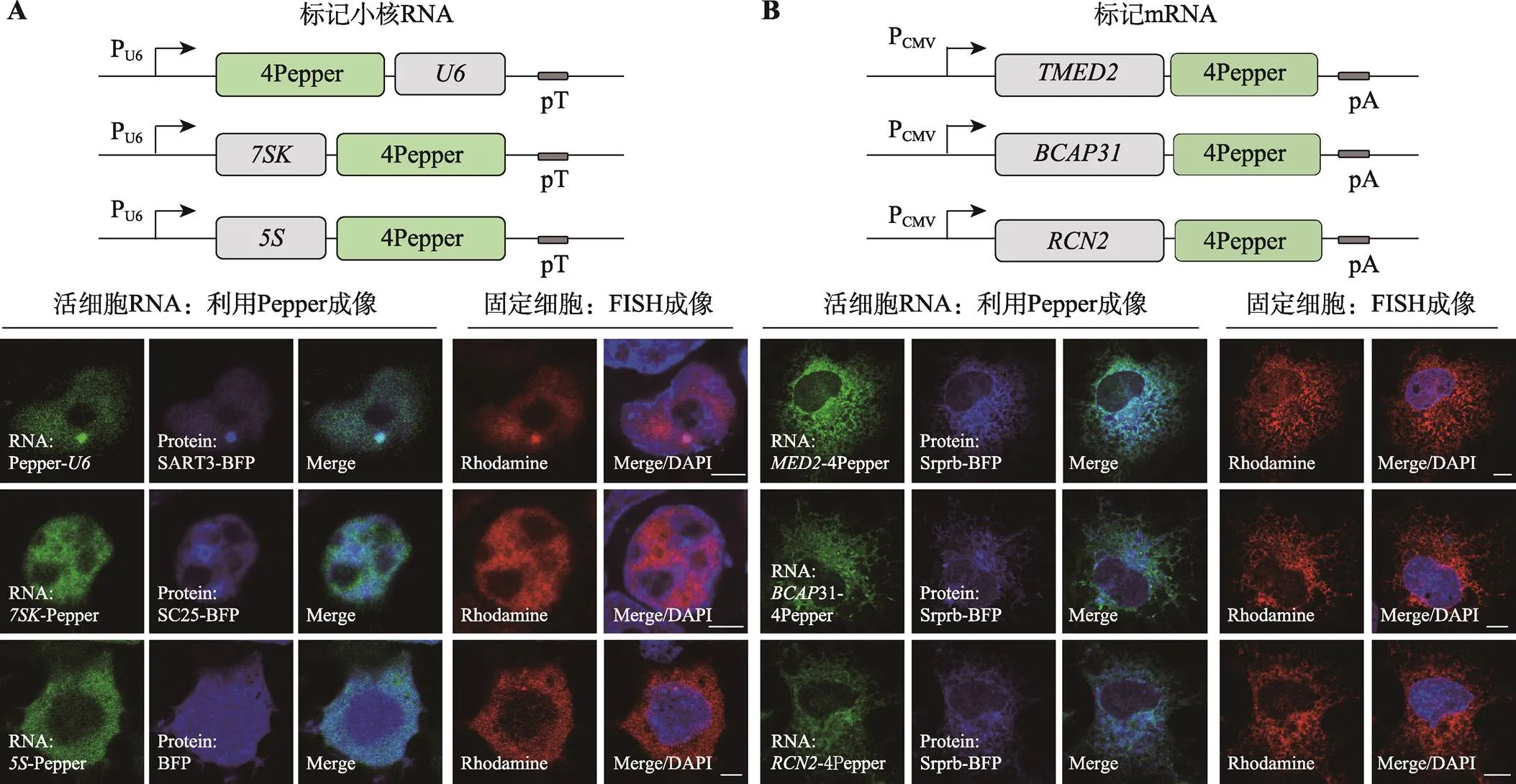
图3 利用Pepper荧光RNA实现活细胞中不同RNA的荧光成像
A:利用Pepper530标记活细胞内各类小核RNA。B:利用Pepper530标记活细胞内各类mRNA。标尺:10 μm。根据文献[59]修改绘制。
1.2.3 SiR系列荧光RNA
随着近几年的发展,SiR系列荧光RNA通过RNA适配体优化、二聚化、荧光团螺旋内酯化等技术手段,逐步降低荧光团的高背景荧光。所开发的荧光RNA在活细胞中表达后荧光强度高、光稳定性好,可实现活细胞RNA超分辨成像、单分子成像,进一步丰富了荧光RNA工具箱。2021年,德国海德堡大学Andres Jäschke课题组优化获得RhoBAST- TMR-DN荧光RNA,具有快速光转换性质,可实现细胞中RNA的单分子定位(single-molecule localization microscopy, SMLM)超分辨成像。然而,TMR-DN与RhoBAST亲和力低且结合后荧光激活倍数偏低,导致成像信噪比较低[62]。2023年,他们发展了二聚化的biSiRA-SiR2和biRhoBAST-TMR2[63],它们的亲和力显著提升达到皮摩尔级。研究者利用8个biRhoBAST重复串联序列,实现对活细胞中单分子水平的mRNA成像[63]。同年,该课题组成员改造得到SpyRho (spirocyclic rhodamine, 螺旋环罗丹明)荧光团,RhoBAST-SpyRho荧光RNA可实现活细胞中RNA的SMLM和STED超分辨成像[64]。然而,这类荧光团的分子量相对较大,显著影响了其进细胞的速度。此外,这些荧光团会被细胞内源组分非特异性结合并激活,进而产生较高的背景荧光。
1.2.4 Clivia-NBSI系列荧光RNA
在大斯托克斯位移(large Stokes shift)荧光RNA开发方面,2023年,本团队在原创的分子转子型荧光团的骨架结构上,引入更多的转动位点获得NBSI荧光团,使得其发射光谱大幅度红移,斯托克斯位移显著增加。研究团队基于NBSI荧光团筛选获得与之高度特异结合的RNA适配体,对其优化获得长度为30 nt的Clivia RNA适配体[65]。Clivia以纳摩尔级的高亲和力结合不发光的NBSI,激活产生高亮度橙色荧光(因其光谱与君子兰类似,因此被命名为Clivia)。同时,Clivia结构中不含G-四链体,对Mg2+的依赖性极低。研究团队通过对NBSI荧光团进行分子结构上的光谱调谐获得光谱从黄、橙到红的14种Clivia系列荧光RNA,这些荧光RNA的斯托克斯位移最大可达108 nm,高出传统荧光染料至少4倍。与此前报道的唯一大斯托克斯位移荧光RNA Chili相比,Clivia的活细胞成像亮度高出1~2个数量级,体现了大斯托克斯位移荧光RNA从概念到实用的突破。
基于Clivia独特的光谱特性,研究者结合Clivia和Pepper两种荧光RNA,首次实现了活细胞RNA和基因位点的“单激发-双发射”双色荧光成像。与此前基于CRISPR-dCas (clustered regularly interspaced short palindromic repeats, 成簇规则间隔短回文重复序列)(dead Cas, 失活的Cas蛋白)和MS2-MCP(MS2 coating protein,MS2衣壳蛋白)的基因位点标记方法相比,基于荧光RNA的方法更加直接和简单高效。通过嵌合sgRNA的颜色组合,该方法还可以实现不同染色体上三个基因位点的同时标记与成像。研究者利用该系统首次在活细胞水平直接对染色体两端的端粒序列拷贝数进行测定。得益于Clivia的小巧结构,研究者将Clivia插入到多种小核RNA序列中,实现这些小核RNA的高信噪比原位实时标记与动态成像,而不影响它们本身的定位与功能。基于此,研究者利用Clivia标记的U1小核RNA对细胞应激情况下U-小体的形成过程进行了实时监测。
此外,由于Clivia574的激发光谱和NLuc荧光素酶(NanoLuc luciferase)的发射光谱接近(图4A),研究者还将Clivia与NLuc结合起来,开发了基于生物发光共振能量转移(bioluminescence resonance energy transfer, BRET)的RNA-蛋白质相互作用检测技术。研究者将MS2发夹RNA插入到Clivia的环区,由于MS2与二聚化的MCP结合从而带动Clivia靠近NLuc(图4B)。通过设计NLuc和ddMCP (directed dimer MCP)连接处的氨基酸序列,以及Clivia和MS2连接处的碱基序列,优化后的系统其BRET效率超过90% (图4C),可实现活细胞中对MS2和MCP相互作用的BRET成像检测(图4D)。研究者进一步构建工程化的HEK293T细胞,并移植到小鼠体内(图4E),实现了活体水平的MS2和MCP相互作用的BRET成像检测(图4F)。此外,该系统不仅可以检测RNA与蛋白质是否发生相互作用,还可以检测相互作用的强弱。研究团队利用该系统检测了多种哺乳动物细胞内源RNA-蛋白质间的相互作用,为解析活细胞中RNA-蛋白质的相互作用提供了极具价值的实用工具。

图4 基于Clivia荧光RNA的生物发光成像技术检测RNA-蛋白相互作用
A:NLuc的发射光谱和Clivia574的激发和发射光谱。B:BRET融合蛋白与Clivia-MS2报告RNA结合模型示意图。C:表达Clivia和NLuc的HEK293T细胞与荧光团和底物孵育后的发射光谱。D:对图C成像结果,标尺:10 μm。E:表达Clivia和NLuc的移植小鼠皮下组织示意图。F:对图E成像结果。根据文献[65]修改绘制。
大斯托克斯位移荧光蛋白已被广泛应用于活细胞与活体蛋白质的多色荧光标记与成像,而Clivia则代表了大斯托克斯位移荧光RNA的原创性突破。Clivia具有高稳定性、高信噪比、高亮度等性质,是目前唯一可用于活细胞分析的大斯托克斯位移荧光RNA,也是唯一可在活体上对RNA动态进行测量的荧光RNA。Clivia系列荧光RNA为实现活细胞多个RNA的单激发多发射多色成像,活细胞中RNA与蛋白相互作用的动态监测,以及在时间和空间尺度深入探究RNA功能和相互作用、RNA功能与调控机制研究提供极具价值的创新研究工具,也有望为活细胞与活体生物传感、即时诊断甚至实时诊断技术的发展提供新机遇。
1.3 基于荧光RNA的活细胞内源性RNA标记
荧光RNA不仅可用于活细胞中外源RNA的标记与成像,还可检测内源RNA,包括miRNA[66]、mRNA[67,68]等。利用碱基互补配对原理对目标RNA直接杂交是最简单易行的方法,可实现对活细胞中内源性RNA的标记和成像(图5A)。2022年,武汉大学周翔课题组基于本团队开发的Pepper荧光RNA,实现对哺乳动物细胞中内源性β-肌动蛋白mRNA的标记,以及对mRNA所产生应激颗粒的检测与成像[68]。
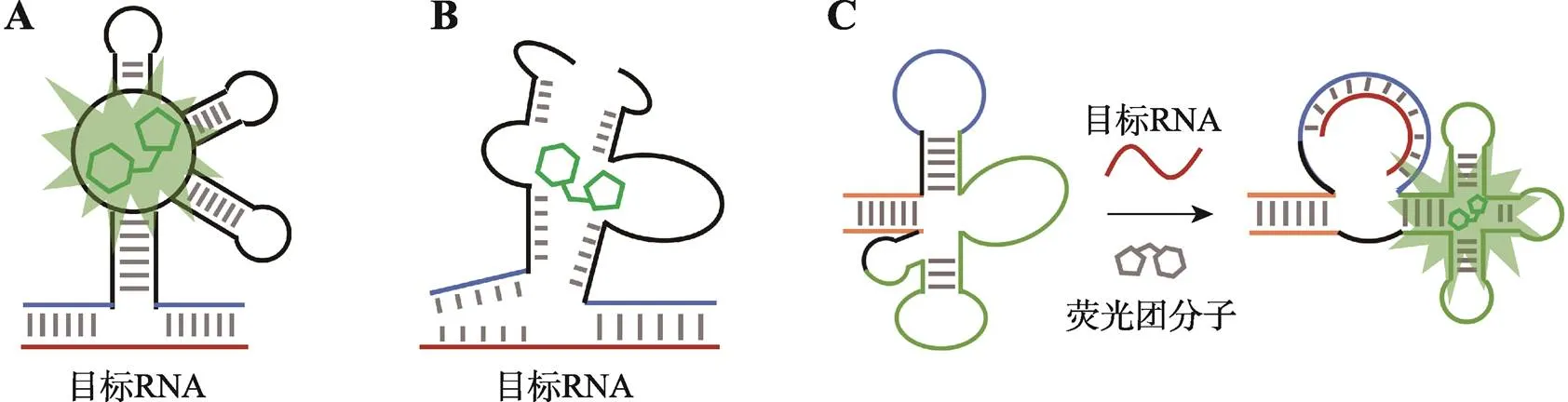
图5 基于荧光RNA的活细胞内源性RNA标记原理示意图
A:基于碱基互补配对原理。B:基于双分子荧光互补原理。C:基于链置换原理。
研究者还可将RNA适配体进行“Split”裂解设计,开发类似于荧光蛋白中的双分子荧光互补技术,进一步降低荧光RNA在未结合目标时产生的本底荧光,实现对活细胞中内源RNA的高信噪比成像 (图5B)。2018年,中国科学院上海应用物理研究所樊春海课题组发展了基于荧光RNA的适配体启动荧光互补(aptamer-initiated fluorescence complementation, AiFC)方法,实现HeLa细胞中β-肌动蛋白mRNA高对比度的实时成像[69]。美国马萨诸塞大学尤明旭课题组发展了基于荧光RNA的基因编码的催化发夹组装RNA回路(catalytic hairpin assembly RNA circuit that is genetically encoded, CHARGE)系统[70]和遗传杂交扩增技术(genetic hybridization amplification technique, INSIGHT)[71],实现活细胞中各种目标RNA的定位成像。此类生物传感器还可用于活细胞内相互作用的研究,如在活细胞中对RNA自组装过程及siRNA干扰过程监测[72]、对病毒自组装过程监测[73]、对内含子自剪接过程监测[74]、对核糖体构象变化和下游基因实时翻译监测[75]、对RNA共转录折叠、RNA-纳米粒复合物形成过程监测[76]等。
此外,研究者还可以基于链置换原理发展内源RNA标记技术,在RNA适配体的核心结构中添加与目标RNA互补的序列,通过结合目标RNA改变RNA适配体的二级结构,即形成荧光团的结合口袋实现荧光激活,从而实现对活细胞内源RNA的检测(图5C)。2017年,湖南大学蒋健晖课题组利用荧光RNA实现在多种肿瘤活细胞中对癌症生物标志物miR-21的成像检测[77]。同年,美国密苏里大学Blake C. Meyers课题组利用同样原理开发出FASTmiR,实现活细胞中对miR-122水平差异的定量检测[78]。
2 基于荧光RNA的生物传感技术
荧光蛋白技术为活细胞蛋白质成像带来了革命性突破。研究者基于荧光蛋白发展了多种遗传编码的荧光探针[79,80](图6A),包括本团队开发的Frex[81]、SoNar[82]、iNap[83]、FiNad[84]、FiLa[85]等,实现了对体外、细胞甚至活体内多种代谢物及生物过程的实时原位监测与成像。不过,由于多数靶标缺乏对应的结合蛋白,研究者难以构建基于荧光蛋白的生物传感器。此外,该类传感器还可能存在信噪比低的问题,限制了它们的应用。

图6 基于荧光蛋白和荧光RNA的生物传感器示意图
A:基于荧光蛋白的生物传感器示意图。示意图中为环化重排荧光蛋白(cyclic rearrangement of fluorescent proteins, cpFP)和青霉素结合蛋白(penicillin binding proteins, PBPs)。B:基于荧光RNA的生物传感器示意图。
在过去十余年的发展中,荧光RNA技术使得活细胞内相关生物过程的监测变得更加简单可行(图6B)。相较于基于荧光蛋白的生物传感器,基于荧光RNA的生物传感器理论上更容易被设计和构建,且具有更大的动态检测范围。除荧光RNA技术本身的RNA适配体组分可遗传编码、荧光团组分灵活多样、信噪比高、荧光信号生成速度快等优势外,研究者可通过SELEX技术筛选获得与目标分子结合的“变构RNA”,将荧光RNA的RNA适配体核心区域外的茎环结构替换成变构RNA。当检测体系中目标分子不存在时,RNA适配体呈现松散结构不能结合荧光团,不产生荧光;目标分子存在时可诱导变构RNA识别靶标并形成结合口袋,从而传感到RNA适配体正确折叠形成结合荧光团的结构,激活荧光团发出荧光,从而实现活细胞代谢物小分子等的生物传感与检测。
自荧光RNA技术问世以来,多个团队不断发展了基于荧光RNA的生物传感技术。研究者将变构RNA分别替换为识别S-腺苷甲硫氨酸(s-adenosine methionine, SAM)和腺苷-5′-二磷酸(adenosine 5′-diphosphate, ADP)的RNA适配体,开发出SAM探针和ADP探针[86]。基于类似原理,基于荧光RNA的MS2衣壳蛋白(MS2 coating protein, MCP)探针[86]、硫胺素焦磷酸(thiamine 5′-pyrophosphate, TPP)探针[87]、环二鸟苷酸(cyclic diguanylate monophosphate, c-di-GMP)探针[88]、环鸟苷酸-腺苷酸(cyclic AMP- GMP, c-GMP-AMP)探针[88]、环二腺苷酸(cyclic diadenosine monophosphate, c-di-AMP)探针[89]、S-腺苷-L-高半胱氨酸(s-adenosyl-l-homocysteine, SAH)探针[90]等陆续被报道。此外,多个团队继续优化探针,通过添加脚手架发展更为稳定的SAM探针[91],更换荧光RNA发展红色SAM探针[48]等。其中,比率型探针由两种不同波段的荧光RNA以及变构RNA组成,其具备自校准功能可最大限度减少转染和表达效率的差异,实现活细胞中对目标RNA的定量成像。多个团队也陆续发展了SAM比率型探针[49]、miRNA比率型探针[77]、mRNA比率型探针[92]、四环素及c-di-GMP比率型探针[93,94]等。不过由于早期的荧光RNA性质并不稳定,在活细胞中表达时荧光弱、光稳定性差,因此基于这些概念期的荧光RNA而开发的生物传感器性能并不出众,并且很多较难应用于活细胞中。
2023年,本团队发展了基于Pepper的系列可特异性识别不同小分子代谢物与蛋白质的遗传编码荧光探针[95]。研究者设计了不同碱基组成和数量的SAM适配体与Pepper连接处的传导模块(图7A),获得的Pepper-SAM探针可实现药物刺激下活细胞中SAM水平动态变化的实时标记与成像(图7, B和C)。此外,研究者还设计构建了Pepper-MCP探针,其可实现光遗传学技术操纵的蛋白质易位的实时监测。得益于Pepper荧光RNA优异的性质,基于Pepper荧光RNA构建的探针在哺乳动物细胞中显示出更高的折叠效率和细胞亮度,并且不同颜色的探针具有良好的适配性,能够与其他成像工具进行组合使用,为生命科学基础研究提供实用的工具。此外,中国科学院北京生命科学研究院李幸课题组于同年也基于Pepper荧光RNA开发出系列生物传感器[96]。受益于Pepper在活细胞中的优异表现,这些基于Pepper荧光RNA的生物传感器实现了对活细胞中代谢物、外源药物、蛋白与金属离子等靶标的监测,展现出高通量、高内涵药物筛选的潜力。
3 结语与展望
RNA最初作为生物体遗传信息传递的中间体而被人们熟知。然而,近年来的研究表明,RNA种类繁多,功能复杂,广泛参与生命过程的各个阶段,包括感知细胞内外信号、控制基因表达等。活细胞中的RNA具有高度的时空动态特性,而时空分布的异常往往会引起多种疾病的发生与发展。因此,发展可以对活细胞内RNA进行原位实时标记与成像的技术将会极大促进人们对RNA功能与调控机制的解析。荧光RNA技术在过去的十余年发展迅猛。凭借适配体组分可遗传编码、更快的荧光信号生成速率、更优异的光学性质、对目标RNA更小的干扰,荧光RNA技术为活细胞RNA标记成像提供极具价值的实用工具。此外,基于荧光RNA的生物传感技术,在保留荧光RNA上述优势外,还可应用于活细胞中小分子代谢物以及蛋白质等靶标的实时动态监测。但是目前的荧光RNA技术仍然存在筛选技术落后、具有高稳定性与高荧光强度的荧光RNA偏少、光谱范围无法涵盖到近红外波段、以及应用范围较窄等不足。本文针对这些不足提出以下展望。
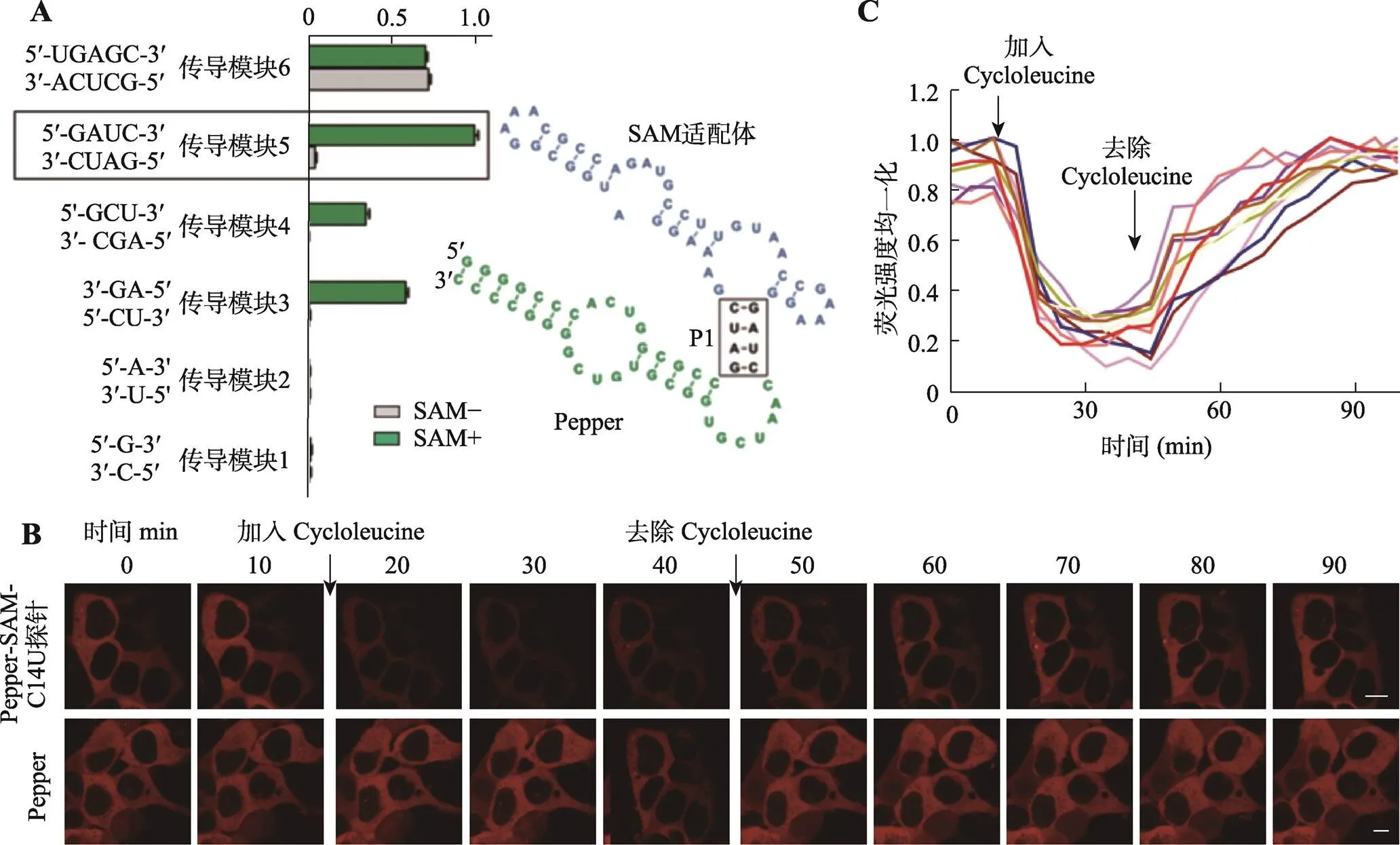
图7 利用Pepper-SAM-C14U探针实现活细胞内源性SAM的动力学检测
A:SAM探针设计原理及其对SAM的响应。B:SAM探针对活细胞内SAM浓度变化的实时监测与成像。标尺:10 μm。C:对图B中细胞荧光信号的统计结果。根据文献[95]修改绘制。
首先,更加高效的荧光RNA筛选方法的开发。相较于传统仅依靠SELEX技术筛选获得的荧光RNA,Broccoli-DFHBI[43]、Mango II/III/IV-TO1- Biotin[27]分别借助流式分选与SELEX技术、微流控与SELEX技术筛选获得,性质有一定的提升。2023年,美国哥伦比亚大学Milan N. Stojanovic课题组发展了一种官能团引导的小分子适配体筛选方法,该方法还可用于计算机辅助适配体设计的机器学习[97]从而提升筛选效率。同年,美国宾夕法尼亚州立大学王勇课题组改善了筛选介质,报道了一种以三维大孔聚乙二醇(polyethylene glycol, PEG)水凝胶作为适配体筛选基质的方法,可显著缩短筛选周期从而提高筛选效率[98]。
其次,荧光RNA稳定性和荧光强度的进一步提高。由于细胞中核酸酶对RNA的水解作用,以及部分荧光团膜通透性差、RNA适配体缺乏良好的折叠能力、复合物亲和能力低等问题,目前能真正应用于哺乳动物细胞RNA标记与成像的荧光RNA种类和数量仍很少。研究者采取了多种方式,如优化折叠效率更高的RNA适配体获得Spinach2[42]、通过定向SELEX筛选RNA获得Broccoli[43]、通过官能团的修饰改善荧光团性能获得BI[60]和TBI[61]、使用类似天然核糖开关结构的RNA文库进行SELEX筛选获得Squash[49]、使用F30[93,99]或tRNA[100]支架作为侧翼序列表达RNA、在活细胞中表达环状的RNA适配体[101]以及构建以二价方式结合的荧光RNA[63]等方法提高荧光RNA在活细胞中的稳定性、折叠效率、表达丰度以及荧光强度等性能。
最后,荧光RNA技术应用范围的拓宽。进一步发展基于荧光RNA的多色成像技术、超分辨成像技术、单分子RNA成像技术、活体RNA成像技术等。目前具有生物正交性的活细胞实用荧光RNA数量仍然十分有限,已报道的Pepper497可与Clivia系列联合使用,Pepper530可与Clivia618、RhoBAST- TMR-DN或RhoBAST-SpyRho联合使用,Pepper620可与Broccoli-BI联合使用。研究者需要开发荧光团种类更为丰富、RNA适配体与荧光团之间亲和力更强、且具有良好生物正交性的新型荧光RNA,以实现活细胞中对多种目标RNA的多色成像;发展荧光强度高、光稳定性好的荧光RNA实现超分辨成像,以揭示活细胞RNA中更为精细的空间结构;发展基于荧光RNA的单分子RNA成像技术,该技术可以避免传统的荧光蛋白-RNA结合蛋白技术中过大的荧光标签对目标RNA代谢与功能产生的潜在干扰;发展具有近红外光谱的荧光RNA,实现哺乳动物深层组织、甚至是活体中目标RNA的可视化。
综上所述,荧光RNA及其生物传感技术已经让人们对多种RNA的生物学功能和动态变化规律有了更加深入的了解。随着这些技术的不断发展,未来它们将继续为探究RNA更为复杂的功能与调控机制提供极具价值且不可或缺的工具。
[1] Shimomura O, Johnson FH, Saiga Y. Extraction, purification and properties of aequorin, a bioluminescent protein from the luminous hydromedusan,., 1962, 59: 223–239.
[2] Prasher DC, Eckenrode VK, Ward WW, Prendergast FG, Cormier MJ. Primary structure of thegreen-fluorescent protein., 1992, 111(2): 229–233.
[3] Chalfie M, Tu Y, Euskirchen G, Ward WW, Prasher DC. Green fluorescent protein as a marker for gene expression., 1994, 263(5148): 802–805.
[4] Matz MV, Fradkov AF, Labas YA, Savitsky AP, Zaraisky AG, Markelov ML, Lukyanov SA. Fluorescent proteins from nonbioluminescent anthozoa species., 1999, 17(10): 969–973.
[5] Miyawaki A. Green fluorescent protein glows gold., 2008, 135(6): 987–990.
[6] Zimmer M. Green fluorescent protein (GFP): Applications, structure, and related photophysical behavior., 2002, 102(3): 759–781.
[7] Aarthy M, George A, Ayyadurai N. Beyond protein tagging: Rewiring the genetic code of fluorescent proteins—a review., 2021, 191: 840–851.
[8] Shu XK, Royant A, Lin MZ, Aguilera TA, Lev-Ram V, Steinbach PA, Tsien RY. Mammalian expression of infrared fluorescent proteins engineered from a bacterial phytochrome., 2009, 324(5928): 804–807.
[9] Auldridge ME, Satyshur KA, Anstrom DM, Forest KT. Structure-guided engineering enhances a phytochrome- based infrared fluorescent protein., 2012, 287(10): 7000–7009.
[10] Yu D, Gustafson WC, Han C, Lafaye C, Noirclerc- Savoye M, Ge WP, Thayer DA, Huang H, Kornberg TB, Royant A, Jan LY, Jan YN, Weiss WA, Shu XK. An improved monomeric infrared fluorescent protein for neuronal and tumour brain imaging., 2014, 5: 3626.
[11] Kumagai A, Ando R, Miyatake H, Greimel P, Kobayashi T, Hirabayashi Y, Shimogori T, Miyawaki A. A bilirubin- inducible fluorescent protein from eel muscle., 2013, 153(7): 1602–1611.
[12] Mollwitz B, Brunk E, Schmitt S, et al. Directed evolution of the suicide protein6-alkylguanine-DNA alkyltransferase for increased reactivity results in an alkylated protein with exceptional stability. Biochemistry, 2012, 51(5): 986–994.
[13] Los GV, Encell LP, Mcdougall MG, Hartzell DD, Karassina N, Zimprich C, Wood MG, Learish R, Ohana RF, Urh M, Simpson D, Mendez J, Zimmerman K, Otto P, Vidugiris G, Zhu J, Darzins A, Klaubert DH, Bulleit RF, Wood KV. Halotag: a novel protein labeling technology for cell imaging and protein analysis., 2008, 3(6): 373–382.
[14] Plamont MA, Billon-Denis E, Maurin S, Gauron C, Pimenta FM, Specht CG, Shi J, Quérard J, Pan B, Rossignol J, Moncoq K, Morellet N, Volovitch M, Lescop E, Chen Y, Triller A, Vriz S, Le Saux T, Jullien L, Gautier A. Small fluorescence-activating and absorption- shifting tag for tunable protein imaging., 2016, 113(3): 497–502.
[15] Tuerk C, Gold L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase., 1990, 249(4968): 505– 510.
[16] Robertson DL, Joyce GF. Selectionof an RNA enzyme that specifically cleaves single-stranded DNA., 1990, 344(6265): 467–468.
[17] Ellington AD, Szostak JW.selection of RNA molecules that bind specific ligands., 1990, 346(6287): 818–822.
[18] Chudakov DM, Matz MV, Lukyanov S, Lukyanov KA. Fluorescent proteins and their applications in imaging living cells and tissues., 2010, 90(3): 1103–1163.
[19] Ward WW, Bokman SH. Reversible denaturation of aequorea green-fluorescent protein: physical separation and characterization of the renatured protein., 1982, 21(19): 4535–4540.
[20] Grate D, Wilson C. Laser-mediated, site-specific inactivation of RNA transcripts., 1999, 96(11): 6131–6136.
[21] Engelhart AE. RNA imaging: a tale of two G-quadruplexes., 2017, 13(11): 1140– 1141.
[22] Sando S, Narita A, Hayami M, Aoyama Y. Transcription monitoring using fused RNA with a dye-binding light-up aptamer as a tag: a blue fluorescent RNA., 2008(33): 3858–3860.
[23] Constantin TP, Silva GL, Robertson KL, Hamilton TP, Fague K, Waggoner AS, Armitage BA. Synthesis of new fluorogenic cyanine dyes and incorporation into RNA fluoromodules., 2008, 10(8): 1561–1564.
[24] Tan XH, Constantin TP, Sloane KL, Waggoner AS, Bruchez MP, Armitage BA. Fluoromodules consisting of a promiscuous RNA aptamer and red or blue fluorogenic cyanine dyes: selection, characterization, and bioimaging., 2017, 139(26): 9001–9009.
[25] Dolgosheina EV, Jeng SCY, Panchapakesan SSS, Cojocaru R, Chen PSK, Wilson PD, Hawkins N, Wiggins PA, Unrau PJ. RNA Mango aptamer-fluorophore: a bright, high-affinity complex for RNA labeling and tracking., 2014, 9(10): 2412–2420.
[26] Cawte AD, Unrau PJ, Rueda DS. Live cell imaging of single RNA molecules with fluorogenic Mango II arrays., 2020, 11(1): 1283.
[27] Autour A, Jeng SCY, Cawte AD, Abdolahzadeh A, Galli A, Panchapakesan SSS, Rueda D, Ryckelynck M, Unrau PJ. Fluorogenic RNA Mango aptamers for imaging small non-coding RNAs in mammalian cells., 2018, 9(1): 656.
[28] Kong KYS, Jeng SCY, Rayyan B, Unrau PJ. RNA Peach and Mango: orthogonal two-color fluorogenic aptamers distinguish nearly identical ligands., 2021, 27(5): 604–615.
[29] Murata A, Sato SI, Kawazoe Y, Uesugi M. Small- molecule fluorescent probes for specific RNA targets., 2011, 47(16): 4712–4714.
[30] Arora A, Sunbul M, Jäschke A. Dual-colour imaging of RNAs using quencher- and fluorophore-binding aptamers., 2015, 43(21): e144.
[31] Sunbul M, Jäschke A. SRB-2: a promiscuous rainbow aptamer for live-cell RNA imaging., 2018, 46(18): e110.
[32] Sunbul M, Jäschke A. Contact-mediated quenching for RNA imaging in bacteria with a fluorophore-binding aptamer., 2013, 52(50): 13401–13404.
[33] Braselmann E, Wierzba AJ, Polaski JT, Chromiński M, Holmes ZE, Hung ST, Batan D, Wheeler JR, Parker R, Jimenez R, Gryko D, Batey RT, Palmer A E. A multicolor riboswitch-based platform for imaging of RNA in live mammalian cells., 2018, 14(10): 964–971.
[34] Bouhedda F, Fam KT, Collot M, Autour A, Marzi S, Klymchenko A, Ryckelynck M. A dimerization-based fluorogenic dye-aptamer module for RNA imaging in live cells., 2020, 16(1): 69–76.
[35] Paige JS, Wu KY, Jaffrey SR. RNA mimics of green fluorescent protein., 2011, 333(6042): 642–646.
[36] Masuda I, Igarashi T, Sakaguchi R, Nitharwal RG, Takase R, Han KY, Leslie BJ, Liu CP, Gamper H, Ha T, Sanyal S, Hou YM. A genetically encoded fluorescent tRNA is active in live-cell protein synthesis., 2017, 45(7): 4081–4093.
[37] Ilgu M, Ray J, Bendickson L, Wang TJ, Geraskin IM, Kraus GA, Nilsen-Hamilton M. Light-up and FRET aptamer reporters; evaluating their applications for imaging transcription in eukaryotic cells., 2016, 98: 26–33.
[38] Guet D, Burns LT, Maji S, Boulanger J, Hersen P, Wente SR, Salamero J, Dargemont C. Combining Spinach- tagged RNA and gene localization to image gene expression in live yeast., 2015, 6: 8882.
[39] Han KY, Leslie BJ, Fei JY, Zhang JC, Ha T. Understanding the photophysics of the Spinach-DFHBI RNA aptamer-fluorogen complex to improve live-cell RNA imaging., 2013, 135(50): 19033– 19038.
[40] Guzmán-Zapata D, Domínguez-Anaya Y, Macedo- Osorio KS, Tovar-Aguilar A, Castrejón-Flores JL, Durán- Figueroa NV, Badillo-Corona JA. mRNA imaging in the chloroplast of chlamydomonas reinhardtii using the light-up aptamer Spinach., 2017, 251: 186–188.
[41] Song WJ, Strack RL, Svensen N, Jaffrey SR. Plug-and- play fluorophores extend the spectral properties of Spinach., 2014, 136(4): 1198–1201.
[42] Strack RL, Disney MD, Jaffrey SR. A superfolding Spinach2 reveals the dynamic nature of trinucleotide repeat-containing RNA., 2013, 10(12): 1219–1224.
[43] Filonov GS, Moon JD, Svensen N, Jaffrey SR. Broccoli: rapid selection of an RNA mimic of green fluorescent protein by fluorescence-based selection and directed evolution., 2014, 136(46): 16299–16308.
[44] Warner KD, Chen MC, Song WJ, Strack RL, Thorn A, Jaffrey SR, Ferré-D'amaré AR. Structural basis for activity of highly efficient RNA mimics of green fluorescent protein., 2014, 21(8): 658–663.
[45] Autour A, Westhof E, Ryckelynck M. iSpinach: a fluorogenic RNA aptamer optimized forapplications., 2016, 44(6): 2491– 2500.
[46] Fernandez-Millan P, Autour A, Ennifar E, Westhof E, Ryckelynck M. Crystal structure and fluorescence properties of the iSpinach aptamer in complex with DFHBI., 2017, 23(12): 1788–1795.
[47] Song WJ, Filonov GS, Kim H, Hirsch M, Li X, Moon JD, Jaffrey SR. Imaging RNA polymerase III transcription using a photostable RNA-fluorophore complex., 2017, 13(11): 1187–1194.
[48] Li X, Mo LT, Litke JL, Dey SK, Suter SR, Jaffrey SR. Imaging intracellular s-adenosyl methionine dynamics in live mammalian cells with a genetically encoded red fluorescent RNA-based sensor., 2020, 142(33): 14117–14124.
[49] Dey SK, Filonov GS, Olarerin-George AO, Jackson BT, Finley LWS, Jaffrey SR. Repurposing an adenine riboswitch into a fluorogenic imaging and sensing tag., 2022, 18(2): 180–190.
[50] Wu JH, Svensen N, Song WJ, Kim H, Zhang SL, Li X, Jaffrey SR. Self-assembly of intracellular multivalent RNA complexes using dimeric Corn and Beetroot aptamers., 2022, 144(12): 5471–5477.
[51] Steinmetzger C, Palanisamy N, Gore KR, Höbartner C. A multicolor large Stokes shift fluorogen-activating RNA aptamer with cationic chromophores., 2019, 25(8): 1931–1935.
[52] Wirth R, Gao P, Nienhaus GU, Sunbul M, Jäschke A. SiRA: a silicon rhodamine-binding aptamer for live-cell super-resolution RNA imaging., 2019, 141(18): 7562–7571.
[53] Zhang JY, Wang L, Jäschke A, Sunbul M. A color- shifting near-infrared fluorescent aptamer-fluorophore module for live-cell RNA imaging., 2021, 60(39): 21441–21448.
[54] Chen HY, Shiroguchi K, Ge H, Xie XS. Genome-wide study of mRNA degradation and transcript elongation in., 2015, 11(5): 808.
[55] Bernstein JA, Khodursky AB, Lin PH, Lin-Chao S, Cohen SN. Global analysis of mRNA decay and abundance inat single-gene resolution using two-color fluorescent DNA microarrays., 2002, 99(15): 9697–9702.
[56] Selinger DW, Saxena RM, Cheung KJ, Church GM, Rosenow C. Global RNA half-life analysis inreveals positional patterns of transcript degradation., 2003, 13(2): 216–223.
[57] Yang E, Van Nimwegen E, Zavolan M, Rajewsky N, Schroeder M, Magnasco M, Darnell JE. Decay rates of human mRNAs: correlation with functional characteristics and sequence attributes., 2003, 13(8): 1863–1872.
[58] Guo JU, Bartel, DP. RNA G-quadruplexes are globally unfolded in eukaryotic cells and depleted in bacteria., 2016, 353(6306): aaf5371–5378.
[59] Chen XJ, Zhang DS, Su N, Bao BK, Xie X, Zuo FT, Yang LP, Wang H, Jiang L, Lin QN, Fang MY, Li NF, Hua X, Chen ZD, Bao CY, Xu JJ, Du WL, Zhang LX, Zhao YZ, Zhu LY, Loscalzo J, Yang Y. Visualizing RNA dynamics in live cells with bright and stable fluorescent RNAs., 2019, 37(11): 1287–1293.
[60] Li X, Kim H, Litke JL, Wu JH, Jaffrey SR. Fluorophore-promoted RNA folding and photostability enables imaging of single Broccoli-tagged mRNAs in live mammalian cells., 2020, 59(11): 4511–4518.
[61] Li X, Wu JH, Jaffrey SR. Engineering fluorophore recycling in a fluorogenic RNA aptamer., 2021, 60(45): 24153–24161.
[62] Sunbul M, Lackner J, Martin A, Englert D, Hacene B, Grün F, Nienhaus K, Nienhaus GU, Jäschke A. Super- resolution RNA imaging using a rhodamine-binding aptamer with fast exchange kinetics., 2021, 39(6): 686–690.
[63] Bühler B, Schokolowski J, Benderoth A, Englert D, Grün F, Jäschke A, Sunbul M. Avidity-based bright and photostable light-up aptamers for single-molecule mRNA imaging., 2023, 19(4): 478–487.
[64] Englert D, Burger EM, Grün F, Verma MS, Lackner J, Lampe M, Bühler B, Schokolowski J, Nienhaus GU, Jäschke A, Sunbul M. Fast-exchanging spirocyclic rhodamine probes for aptamer-based super-resolution RNA imaging., 2023, 14(1): 3879.
[65] Jiang L, Xie X, Su N, Zhang DS, Chen XJ, Xu XC, Zhang BB, Huang KY, Yu JW, Fang MY, Bao BK, Zuo FT, Yang LP, Zhang R, Li HW, Huang XY, Chen ZD, Zeng QM, Liu RM, Lin QN, Zhao YZ, Ren AM, Zhu LY, Yang Y. Large Stokes shift fluorescent RNAs for dual-emission fluorescence and bioluminescence imaging in live cells., 2023, 20(10): 1563–1572.
[66] Ying ZM, Tu B, Liu L, Tang H, Tang LJ, Jiang JH. Spinach-based fluorescent light-up biosensors for multiplexed and label-free detection of microRNAs., 2018, 54(24): 3010–3013.
[67] Ong WQ, Citron YR, Sekine S, Huang B. Live cell imaging of endogenous mRNA using RNA-based fluorescence "turn-on" probe., 2017, 12(1): 200–205.
[68] Wang Q, Xiao F, Su HM, Liu H, Xu JL, Tang H, Qin SS, Fang ZT, Lu ZA, Wu J, Weng XC, Zhou X. Inert Pepper aptamer-mediated endogenous mRNA recognition and imaging in living cells., 2022, 50(14): e84.
[69] Wang ZJ, Luo Y, Xie XD, Hu XJ, Song HY, Zhao Y, Shi JY, Wang LH, Glinsky G, Chen N, Lal R, Fan CH.spatial complementation of aptamer-mediated recognition enables live-cell imaging of native RNA transcripts in real time., 2018, 57(4): 972–976.
[70] Karunanayake Mudiyanselage APKK, Yu QK, Leon-Duque MA, Zhao B, Wu R, You MX. Genetically encoded catalytic hairpin assembly for sensitive RNA imaging in live cells., 2018, 140(28): 8739–8745.
[71] Ren KW, Wu R, Karunanayake Mudiyanselage APKK, Yu QK, Zhao B, Xie YW, Bagheri Y, Tian Q, You MX.genetically cascaded amplification for imaging RNA subcellular locations., 2020, 142(6): 2968–2974.
[72] Rogers TA, Andrews GE, Jaeger L, Grabow WW. Fluorescent monitoring of RNA assembly and processing using the split-Spinach aptamer., 2015, 4(2): 162–166.
[73] Tsvetkova IB, Yi GH, Yi Y, Kao CC, Dragnea BG. Segmented GFP-like aptamer probes for functional imaging of viral genome trafficking., 2015, 210: 291–297.
[74] Furukawa A, Tanaka T, Furuta H, Matsumura S, Ikawa Y. Use of a fluorescent aptamer RNA as an exonic sequence to analyze self-splicing ability of agroup I intron from structured RNAs., 2016, 5(4): 43.
[75] Alam KK, Tawiah KD, Lichte MF, Porciani D, Burke DH. A fluorescent split aptamer for visualizing RNA-RNA assembly., 2017, 6(9): 1710–1721.
[76] Chandler M, Lyalina T, Halman J, Rackley L, Lee L, Dang D, Ke WN, Sajja S, Woods S, Acharya S, Baumgarten E, Christopher J, Elshalia E, Hrebien G, Kublank K, Saleh S, Stallings B, Tafere M, Striplin C, Afonin KA. Broccoli fluorets: split aptamers as a user-friendly fluorescent toolkit for dynamic RNA nanotechnology., 2018, 23(12): 3178.
[77] Ying ZM, Wu Z, Tu B, Tan WH, Jiang JH. Genetically encoded fluorescent RNA sensor for ratiometric imaging of microRNA in living tumor cells., 2017, 139(29): 9779–9782.
[78] Huang K, Doyle F, Wurz ZE, Tenenbaum SA, Hammond RK, Caplan JL, Meyers BC. FASTmiR: an RNA-based sensor forquantification and live-cell localization of small RNAs., 2017, 45(14): e130.
[79] Newman RH, Fosbrink MD, Zhang J. Genetically encodable fluorescent biosensors for tracking signaling dynamics in living cells., 2011, 111(5): 3614–3666.
[80] Zhang J, Campbell RE, Ting AY, Tsien RY. Creating new fluorescent probes for cell biology., 2002, 3(12): 906–918.
[81] Zhao YZ, Jin J, Hu QX, Zhou HM, Yi J, Yu ZH, Xu L, Wang X, Yang Y, Loscalzo J. Genetically encoded fluorescent sensors for intracellular NADH detection., 2011, 14(4): 555–566.
[82] Zhao YZ, Hu QX, Cheng FX, Su N, Wang AX, Zou YJ, Hu HY, Chen XJ, Zhou HM, Huang XZ, Yang K, Zhu Q, Wang X, Yi J, Zhu LY, Qian XH, Chen LX, Tang Y, Loscalzo J, Yang Y. SoNar, a highly responsive NAD+/NADH sensor, allows high-throughput metabolic screening of anti-tumor agents., 2015, 21(5): 777–789.
[83] Tao RK, Zhao YZ, Chu HY, Wang AX, Zhu JH, Chen XJ, Zou YJ, Shi M, Liu RM, Su N, Du JL, Zhou HM, Zhu LY, Qian XH, Liu HY, Loscalzo J, Yang Y. Genetically encoded fluorescent sensors reveal dynamic regulation of NADPH metabolism., 2017, 14(7): 720–728.
[84] Zou YJ, Wang AX, Huang L, Zhu XD, Hu QX, Zhang YN, Chen XJ, Li FW, Wang QH, Wang H, Liu RM, Zuo FT, Li T, Yao J, Qian YJ, Shi M, Yue X, Chen WC, Zhang Z, Wang CR, Zhou Y, Zhu LY, Ju ZY, Loscalzo J, Yang Y, Zhao YZ. Illuminating NAD+metabolism in live cells andusing a genetically encoded fluorescent sensor., 2020, 53(2): 240–252.e7.
[85] Li X, Zhang YN, Xu LY, Wang AX, Zou YJ, Li T, Huang L, Chen WC, Liu SN, Jiang K, Zhang XZ, Wang DM, Zhang LJ, Zhang Z, Zhang ZY, Chen XJ, Jia W, Zhao AH, Yan XF, Zhou HM, Zhu LY, Ma XR, Ju ZY, Jia WP, Wang CR, Loscalzo J, Yang Y, Zhao YZ. Ultrasensitive sensors reveal the spatiotemporal landscape of lactate metabolism in physiology and disease., 2023, 35(1): 200–211.e9.
[86] Song WJ, Strack RL, Jaffrey SR. Imaging bacterial protein expression using genetically encoded RNA sensors., 2013, 10(9): 873–875.
[87] You MX, Litke JL, Jaffrey SR. Imaging metabolite dynamics in living cells using a Spinach-based riboswitch., 2015, 112(21): E2756–E2765.
[88] Kellenberger CA, Wilson SC, Sales-Lee J, Hammond MC. RNA-based fluorescent biosensors for live cell imaging of second messengers cyclic di-GMP and cyclic AMP-GMP., 2013, 135(13): 4906–4909.
[89] Kellenberger CA, Chen C, Whiteley AT, Portnoy DA, Hammond MC. RNA-based fluorescent biosensors for live cell imaging of second messenger cyclic di-AMP., 2015, 137(20): 6432–6435.
[90] Su YC, Hickey SF, Keyser SGL, Hammond MC.andenzyme activity screening via RNA-based fluorescent biosensors for s-adenosyl-l-homocysteine (SAH)., 2016, 138(22): 7040–7047.
[91] Moon JD, Wu JH, Dey SK, Litke JL, Li X, Kim H, Jaffrey SR. Naturally occurring three-way junctions can be repurposed as genetically encoded RNA-based sensors., 2021, 28(11): 1569–1580.e4.
[92] Ying ZM, Yuan YY, Tu B, Tang LJ, Yu RQ, Jiang JH. A single promoter system co-expressing RNA sensor with fluorescent proteins for quantitative mRNA imaging in living tumor cells., 2019, 10(18): 4828–4833.
[93] Wu R, Karunanayake Mudiyanselage APKK, Shafiei F, Zhao B, Bagheri Y, Yu QK, Mcauliffe K, Ren KW, You MX. Genetically encoded ratiometric RNA-based sensors for quantitative imaging of small molecules in living cells., 2019, 58(50): 18271–18275.
[94] Wu R, Karunanayake Mudiyanselage APKK, Ren KW, Sun ZN, Tian Q, Zhao B, Bagheri Y, Lutati D, Keshri P, You MX. Ratiometric fluorogenic RNA-based sensors for imaging live-cell dynamics of small molecules., 2020, 3(5): 2633–2642.
[95] Fang MY, Li HW, Xie X, Wang H, Jiang Y, Li TY, Zhang BB, Jiang X, Cao YY, Zhang R, Zhang DS, Zhao YZ, Zhu LY, Chen XJ, Yang Y. Imaging intracellular metabolite and protein changes in live mammalian cells with bright fluorescent RNA-based genetically encoded sensors., 2023, 235: 115411.
[96] Chen ZY, Chen W, Reheman Z, Jiang HD, Wu JH, Li X. Genetically encoded RNA-based sensors with Pepper fluorogenic aptamer., 2023, 51(16): 8322–8336.
[97] Yang K, Mitchell NM, Banerjee S, Cheng ZZ, Taylor S, Kostic AM, Wong I, Sajjath S, Zhang YM, Stevens J, Mohan S, Landry DW, Worgall TS, Andrews AM, Stojanovic MN. A functional group-guided approach to aptamers for small molecules., 2023, 380(6648): 942–948.
[98] Singh NK, Wang YX, Wen C, Davis B, Wang XL, Lee K, Wang Y. High-affinity one-step aptamer selection using a non-fouling porous hydrogel., 2023, doi: 10.1038/s41587-023-01973-8.
[99] Filonov GS, Kam CW, Song WJ, Jaffrey SR. In-gel imaging of RNA processing using Broccoli reveals optimal aptamer expression strategies., 2015, 22(5): 649–660.
[100] Jepsen MDE, Sparvath SM, Nielsen TB, Langvad AH, Grossi G, Gothelf KV, Andersen ES. Development of a genetically encodable FRET system using fluorescent RNA aptamers., 2018, 9(1): 18.
[101] Litke JL, Jaffrey SR. Highly efficient expression of circular RNA aptamers in cells using autocatalytic transcripts., 2019, 37(6): 667–675.
Progress on fluorescent RNA and fluorescent RNA-based biosensing technology
Fangting Zuo1,2,3, Yaqiang Zhang1,2,3, Huimin Yang1,2, Yi Yang1,2, Xianjun Chen1,2
Fluorescent RNA is a kind of emerging RNA labeling technique that can be used forlabeling and imaging of RNA in live cells, which plays an important role in understanding the function and regulation mechanism of RNA. Biosensing technology based on fluorescent RNA can be applied in dynamic detection of small molecule metabolites and proteins in real time, offering valuable tools for basic life science research and biomedical sensing technology development. In this review, we introduce the development of genetically encoded fluorescent RNA, and the application of fluorescent RNA in RNA imaging and biosensing technology based on fluorescent RNA in biosensing in live cell. Meanwhile, we discuss the direction and challenge of future development of fluorescent RNA technology to provide valuable insights for further development and application of this technology in relevant fields.
fluorescent RNA; fluorescent labeling; RNA imaging; biosensing

杨弋,教授,博士生导师,长江学者特聘教授,国家杰出青年基金获得者,国家自然科学基金创新群体负责人,国家重点研发计划首席科学家。现任生物反应器工程国家重点实验室副主任,华东理工大学光遗传学与合成生物学交叉学科研究中心主任,中国生物化学与分子生物学会常务理事及酶学分会副主任、秘书长,中国生物物理学会理事。主要研究方向为光遗传控制方法、细胞代谢监控方法和生物大分子标记方法。系列前沿方法学研究成果发表于等国际重要期刊,已被国内外实验室广泛应用。
陈显军,教授,博士生导师,国家高层次青年人才。研究方向主要为新型光遗传学技术的开发与应用和生物大分子荧光标记技术开发与应用,近年来聚焦活细胞RNA实时动态监测与时空精确调控的方法学关键难题,创新发展了系列高性能荧光RNA与RNA代谢光控因子,实现了活细胞RNA高时空分辨成像与精密控制。相关研究成果以第一或通讯作者发表在等国际重要期刊,授权多项国内外发明专利。研究成果在国际同行中产生重要影响,所发展技术被300余个国内外一流机构实验室跟踪应用。主持包括十余项国家与省部级基金项目。
2023-12-12;
2024-01-25;
2024-01-29
国家自然科学基金项目 (编号: 32121005, 32150028, 21937004, 91857202, 32250009)和国家重点研发计划(编号: 2022YFC3400100)资助 [Supported by the National Natural Science Foundation of China (Nos. 32121005, 32150028, 21937004, 91857202, 32250009) and the National Key Research and Development Program of China (No. 2022YFC3400100)]
左方婷,博士后,研究方向:荧光RNA技术的开发与应用。E-mail: ftzuo@ecust.edu.cn
杨弋,博士,教授,博士生导师,研究方向:生命成像与调控。E-mail: yiyang@ecust.edu.cn
陈显军,博士,教授,博士生导师,研究方向:活细胞RNA成像与调控。E-mail: xianjunchen@ecust.edu.cn
10.16288/j.yczz.23-306
(责任编委: 史岸冰)