基于Y-SNP和Y-STR揭示汉族人群父系遗传关系
2024-02-22朱信金鑫刘俊杨澜邹丽馨李彩霞黄江江丽
朱信,金鑫,刘俊,杨澜,邹丽馨,李彩霞,黄江,江丽
研究报告
基于Y-SNP和Y-STR揭示汉族人群父系遗传关系
朱信1,2,金鑫3,刘俊2,4,杨澜2,4,邹丽馨2,5,李彩霞1,2,黄江1,江丽2
1. 贵州医科大学法医学院,贵阳 550004 2. 公安部鉴定中心,北京市现场物证检验工程技术研究中心,现场物证溯源技术国家工程实验室,北京 100038 3. 海南省公安厅,海口 570203 4. 山西医科大学,太原 030001 5. 江苏师范大学,徐州 221116
汉族是中国人口最多的民族,现有研究多集中于汉族人群的起源、迁徙、融合等遗传历史,以及局部地区汉族人群的父系遗传关系,鲜有全局视角下的汉族人群父系遗传结构研究。本研究检测了362份青海、四川和辽宁的汉族无关男性样本,整合已发表文献相关数据,最终获得了国内15个省份16个汉族人群1830人份样本,覆盖89个Y-SNP、16个Y-STR的数据。通过统计Y-SNP单倍群频率、Y-STR单倍型多样性,使用主成分分析(principal component analysis, PCA)、系统发育树、单倍型网络等分析,综合Y-SNP和Y-STR两个反映不同时间尺度的遗传标记,研究不同地区汉族人群之间的遗传分化、汉族人群与其周边少数民族的遗传关系。单倍群频率统计结果显示单倍群O-M175是汉族人群主体单倍群(青海汉族60.53%~广东汉族92.7%),其下游亚单倍群呈现地域差异化分布。单倍群O2-M122高频分布于各地汉族,总体分布趋势北高南低;单倍群O1b-M268分布频率由南向北递减,尤其在岭南地区汉族人群中分布显著;单倍群O1a-M119在中部汉族人群中分布频率较高。汉族人群遗传结构研究表明,其主要分为北部、中部及南部三个聚类簇,其中青海汉族与其他地区汉族存在一定的遗传分化。在合并少数民族的遗传关系研究中,汉族人群彼此之间遗传关系更紧密,但北部汉族与回族遗传关系更近,而南部汉族则与仡佬族、黎族遗传关系更近。总之,本文基于89个Y-SNP和16个Y-STR,系统地研究了中国不同地域的汉族人群的单倍群分布、遗传亚结构及其与周边少数民族的遗传关系,为群体遗传学、法医遗传学补充理论依据,为Y染色体的法医学应用提供数据支撑。Y-SNP单倍群结合Y-STR单倍型对于分析汉族人群遗传亚结构以及法医学应用具有重要作用。
群体遗传学;法医遗传学;Y-SNP;Y-STR
男性特异的Y染色体非重组区域(non- recombining Y,NRY)遵循严格的父系遗传[1],被广泛应用于进化人类学[2]、遗传系谱学[1,3]、医学遗传学[1,4]和法医遗传学[5,6]等研究领域。Y染色体上常用的遗传标记是短串联重复序列(Y-chromosome short tandem repeat,Y-STR)和单核苷酸多态性(Y-chromosome single nucleotide polymorphism,Y-SNP)。具有高突变率的Y-STR (3.78×10–4~7.44× 10–2次突变/代)随父系世代传递过程中累积突变产生了独特的单倍型[6~9]。然而,在实际应用中由于标记集的大小有限,来自不同父系的个体可能巧合地表现出相同的单倍型[10~13],Y-STR单倍型匹配有时并无法代表个体父系祖源的一致性。而突变速度相对缓慢的Y-SNP(1.0×10−9突变/代)能够通过突变发生时期建立系统发育树并定义Y-SNP单倍群类型[14,15],Y-SNP单倍群的分布具有一定地理及族群相关性。因此,Y-STR结合Y-SNP对分析人群父系遗传关系具有重要的意义[16,17],目前,在法医学实践中利用Y-STR推测男性犯罪嫌疑人的家系和来源群体,尚缺应用于法医学领域的Y-SNP单倍群人群分布作为参考数据,为弥补缺少适用于物证鉴定的Y-SNP复合扩增体系等问题,国内外不少研究尝试基于Y-STR推断Y-SNP单倍群[18~20],但其推断准确性有待进一步验证,因此开展Y-SNP单倍群人群验证研究十分必要。以往研究已开发了覆盖数十至数百个Y-SNP位点的检测体系[21~29],但随着测序技术的发展许多新发现及不断被更正的Y-SNP位点仍有待进一步人群数据验证。
汉族是世界上人数最多的民族,其母语——汉语,属于汉藏语系两大语族之一。据全国第七次人口普查数据,汉族人口约占中国总人口的91.11%[30],占全球人口的近18%。我国人口历史悠久、起源复杂,汉族主要集中在东部和中部地区,少数民族多分布在西南、西北及东北等地区,各民族间呈现“大杂居、小聚居”的分布格局。汉族在历史迁徙过程中与周边其他民族相互交流融合[31],受地理、经济和文化因素影响,不同地区的汉族人群产生了一定程度的遗传分化[32~34]。我国总体地势呈现西高东低,居住在东部平原、丘陵上以种植业为主的民族多形成大而集中或长条形的聚落,彼此交流更频繁;生活在西部高原山区的民族多呈小而分散的聚落,聚落间彼此交流相对较少[35]。基于全基因组的汉族人群遗传关系研究已有不少,如PGGHan、WBBC (Westlake BioBank for Chinese)、ChinaMap等[36~38],基于线粒体研究中汉族人群的覆盖也十分广泛[32],其都表现出汉族人群明显的地理分化。而汉族父系遗传结构的研究还存在采样的局限性,主要集中在中国东部,其表现出明显的南北分化[33,39~41],但对与少数民族混居的西部地区汉族研究仍较少,总体地域覆盖度不够。因此,本研究检测了362份汉族样本,并与收集的已发表文献中不同地域汉族Y染色体数据进行整合,得到15个省份16个汉族人群1830份样本的89Y-SNP、16Y-STR数据集[33,42],并与文献中收集的少数民族Y-SNP、Y-STR数据合并。拟通过不同地域的汉族人群Y-SNP单倍群分布、遗传分化,汉族人群与周边少数民族遗传关系等研究,对中国不同地区汉族父系遗传关系及汉族人群与周边少数民族的遗传交流进行综合解析。
1 材料与方法
1.1 样本采集与DNA提取
本研究采集了362份汉族无关男性个体静脉血样本,其中辽宁铁岭汉族80份,青海汉族114份,四川阿坝汉族76份,四川甘孜汉族91份。本研究已通过公安部鉴定中心伦理委员会审查(伦理批号:2023-021),所有参与志愿者均签署知情同意书。根据试剂盒说明书,采用QIAamp®DNABloodMidiKit(德国QIAGEN公司)进行DNA提取。使用Nano Drop 2000c (美国ThermoFisherScientific公司)进行DNA定量并稀释至5 ng/μL待检。
1.2 Y-SNP位点筛选及分型检测
使用AIYSNP89试剂盒(北京刑技技术有限责任公司)对上述样本在89个Y-SNP位点上的分型进行检测,操作步骤、体系配比及参数设置等均按照试剂盒说明书进行。应用GeneAmp PCR Systerm 9700热循环仪(德国Eppendorf公司)进行复合扩增,使用ABI 3500xL遗传分析仪(美国Thermo Fisher Scientific公司)对扩增产物进行毛细管电泳检测,利用GeneMapper ID v3.2软件分析Y-SNP分型。
1.3 Y-STR分型检测
使用PowerPlex®Y23试剂盒(美国Promega公司)检测76份四川阿坝汉族、91份四川甘孜汉族、80份辽宁铁岭汉族及90份青海汉族样本;使用DNATyper Y26试剂盒(北京刑技技术有限责任公司)检测另外25份青海汉族样本。应用GeneAmp PCR Systerm 9700热循环仪(德国Eppendorf公司)进行扩增,扩增产物用ABI 3500xL遗传分析仪(美国Thermo Fisher Scientific公司)进行毛细管电泳检测,使用GeneMapper ID-X 软件分析Y-STR分型。
1.4 数据整合
本研究使用的人群信息列表如表1所示。
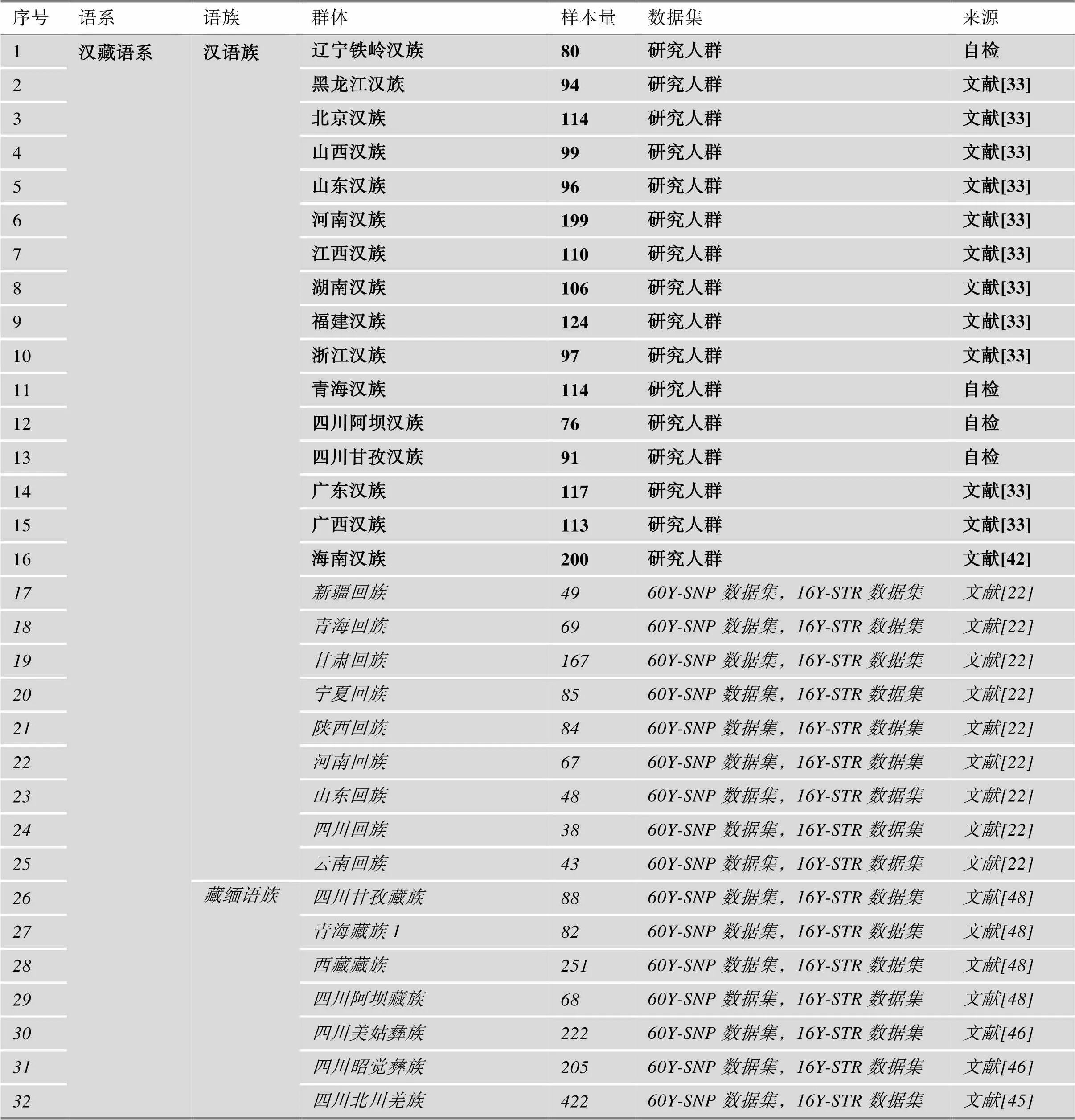
表1 中国人群信息列表
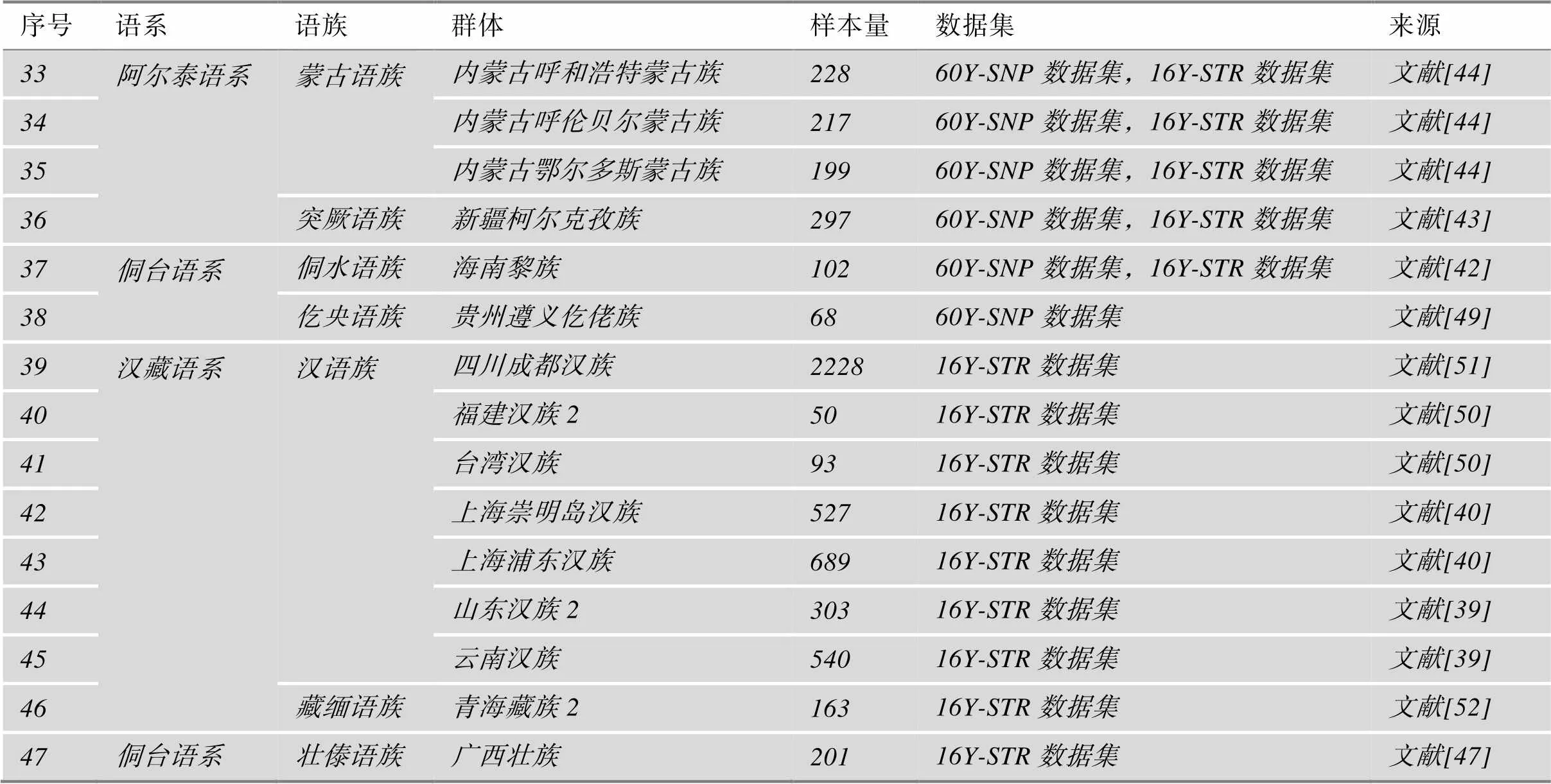
续表
主体研究人群用加粗字体表示,其他人群用斜体表示。人群尾缀1、2表示引自不同文献的人群。
从文献中收集了1468份汉族Y-SNP、Y-STR数据[33,42],与上述检测数据进行整合,得到覆盖16个汉族人群89个Y-SNP、16个Y-STR的研究人群数据集。
同时,为了了解不同地区汉族群体与周边少数民族之间的遗传关系,本研究还收集了文献中Y-SNP位点数相近的国内人群数据[22,33,39,40,42~49]与本研究人群进行合并后得到包含60个Y-SNP位点的38个中国人群数据集,下文简称为60Y-SNP数据集;收集了文献中的中国人群Y-STR数据[33,39,40,42~48,50,51]与本研究人群进行合并后得到包含16个Y-STR的46个中国人群数据集,下文简称为16Y-STR数据集。
1.5 数据分析
1.5.1 Y-SNP单倍群划分及在不同地区汉族中分布
基于研究人群数据集,根据国际Y染色体协会(Y chromosome consortium,YCC)的单倍群命名原则,依据Y染色体谱系树(ISOGG网站版本号14.177,更新日期2019年10月8日)进行Y染色体单倍群划分[53]。使用Excel直接计数统计16个汉族人群基于89个Y-SNP单倍群频率数据。
1.5.2 Y-STR的基因多样性与单倍型多样性
基于16个Y-STR基因座(DYS19、DYS389I、DYS389II、DYS390、DYS391、DYS392、DYS393、DYS437、DYS438、DYS439、DYS448、DYS456、DYS458、DYS533、DYS635和GATA-H4),使用Excel直接计数计算16个研究人群的等位基因频率和单倍型频率、单倍型多样性(haplotype diversity,HD)、单倍型多样性与基因多样性(genetic diversity,GD)、匹配概率(haplotype match probability,HMP)和鉴别能力(discrimination capacity,DC)[54~55]等法医学参数。
1.5.3 中国不同地区汉族人群父系遗传关系分析
基于研究人群数据集的Y-SNP单倍群频率,使用R v4.1.2软件prcomp函数进行PCA分析及ggplot2[56]包进行可视化。基于研究人群数据集的16个Y-STR数据,删除单倍型中单拷贝基因座二等位变异和空等位变异的样本。使用Arlequin v3.5.2.2[57]软件中的AMOVA参数,通过分子方差分析(analysis of molecular variance,AMOVA)[58]计算群体之间成对遗传距离R值分析遗传分化情况,用MEGA v11软件构建邻接树[59]。基于7个Y-STR基因座(DYS19、DYS392、DYS393、DYS437、DYS438、DYS448、YGATAH4)使用NETWORK v10.2软件[60]构建不同地域汉族人群的单倍型中位连接(median joining,MJ)网络图[11],为了更明显观察人群间个体的聚类关系,网络图使用最大简约树进行展示。
1.5.4 中国不同人群父系遗传关系分析
基于60Y-SNP数据集,用R v4.1.2软件prcomp函数进行PCA分析及ggplot2[56]包进行可视化。基于全部16Y-STR数据集,删除单倍型中单拷贝基因座二等位变异和空等位变异的样本。使用Arlequin v3.5.2.2[57]软件计算中国各人群间的R值,并基于人群R矩阵用R v4.1.2软件绘制热图展示人群聚类关系,分析汉族与周边少数民族遗传关系。基于同时覆盖60Y-SNP、16Y-STR的人群,对其7个Y-STR基因座(DYS19、DYS392、DYS393、DYS437、DYS438、DYS448、YGATA-H4)用NETWORK v10.2软件[60]进行周边少数民族特征单倍群的中位连接网络分析,观察特定单倍群下不同语系人群的单倍型共享情况[11],网络图使用最大简约树进行展示。
2 结果与分析
本研究将实验室检测的362份汉族与文献收集的1468份汉族样本Y-SNP、Y-STR数据进行整合得到16个汉族人群1830份样本的研究人群数据集,位点覆盖89个Y-SNP、16个Y-STR[33,42]。通过Y-SNP单倍群频率统计、Y-STR单倍型多样性计算、PCA、R值系统发育树、单倍型网络分析等方法研究不同地区汉族之间的遗传关系。此外,我们还将汉族研究人群数据集分别与文献报道的国内人群数据进行合并,分别得到了60Y-SNP数据集[22,33,39,40,42~49]和16Y-STR数据集[33,39,40,42~48,50,51](表1),从不同角度观察汉族人群与其周边人群的遗传交流。
2.1 Y-SNP单倍群在不同地区汉族人群中的分布差异
基于89个Y-SNP,16个汉族人群共观察到63个不同的末端单倍群。主干单倍群在不同地区汉族的分布频率如图1所示,东亚地区高频分布的单倍群O-M175、C2-M217、N-M231、D1-M174占汉族人群父系单倍群的88%以上。O-M175单倍群为汉族人群的主体单倍群(青海汉族60.53%~广东汉族92.7%),其中,青海汉族O-M175单倍群分布(60.53%)较其他汉族(67%~92.7%)低。O-M175下游的亚单倍群分布呈现出一定的地域差异性,O2-M122单倍群在各个地区汉族人群中分布最高(青海汉族46.49%-山西汉族65.5%),但广西汉族例外(35.7%),总体分布趋势呈现北高南低,但西北的青海汉族O2-M122分布较除广西汉族以外的其他汉族都低。O2-M122单倍群下游的三个大分支分别为O2a2b1*-M134+, M117-及其下游分支(广西汉族4.5%~黑龙江汉族17%)、O2a2b1a1-M117及其下游分支(青海汉族10.53%~山西汉族24.2%)和O2a1b-IMS-JST002611及其下游分支(广西汉族8%~黑龙江汉族22.2%)。单倍群O1b-M268在岭南地区汉族人群中分布明显高于其他地区(广西汉族38%、海南汉族21.5%),呈现由南向北递减趋势;单倍群O1a-M119在中部地区的汉族人群中分布频率明显较高(江西汉族30.6%、浙江汉族26.7%、湖南汉族23.6%、福建汉族16.9%)。单倍群C2-M217是汉族人群的第二大单倍群(福建汉族3.2%~河南汉族15.5%),在北部及西北部汉族人群中分布频率较高(河南汉族15.5%、青海汉族12.15%、黑龙江汉族12.8%、山西汉族12.1%、辽宁铁岭汉族11.25%),在南部汉族人群中分布频率均低于6%。单倍群N-M231的分布也呈现出一定由北向南递减的趋势(黑龙江汉族10.7%、青海汉族10.53%、山东汉族12.5%,南部汉族人群中均低于10%)。单倍群D1-M174在青藏高原周边地区及北部地区低频分布(甘孜汉族5.49%、广西汉族4.4%、辽宁铁岭汉族3.75%、北京汉族2.7%),在南部平原地区几乎不分布。其他零散分布的单倍群还有Q-M242 (0%~7.1%)、R-M207(0%~4.39%)、J-M304(0%~1.75%),分布趋势呈现北高南低。
2.2 Y-STR单倍型多样性和基因多样性
在16个单拷贝Y-STR基因座组成的单倍型中,1830份汉族样本共检测到1737个单倍型,其中1个单倍型重复了7次、1个单倍型重复了6次、1个单倍型重复了4次、8个单倍型重复了3次、63个单倍型重复了2次。16个单拷贝Y-STR基因座中汉族群体的单倍型多样性(HD)均高于0.99,HD及DC为1的汉族人群均分布于中国北部(分别为黑龙江汉族、辽宁铁岭汉族、山西汉族),且北部汉族HD值普遍高于其他地区(表2)。在1830份汉族样本中共检测到60个中间等位基因、2个空等位基因和9个拷贝数变异,16个Y-STR基因座在汉族人群中的GD值介于0.292(DYS391)到0.84(DYS458)之间(图2),其中DYS458、DYS635、DYS389II、DYS392、DYS19、DYS448、DYS390等基因座在各地汉族人群中基因多样性普遍较高,GD值均高于0.6。在部分基因座上不同地区汉族GD值表现出较大的差异。北部地区(辽宁铁岭、山东、山西、河南)汉族人群在DYS19、DYS389II、DYS437、DYS392等基因座上GD值较高,其中北京和黑龙江汉族在DYS389I、DYS390、DYS437、DYS438等基因座上变异较大,GD值较其他北部汉族低。中部(浙江、江西、福建、湖南)汉族人群在DYS389、DYS533、YGATA-H4等基因座上GD值较低。而南部地区(广东、广西、海南)汉族在不同基因座的GD值差异较大,在DYS389I、DYS393、DYS533、YGATA-H4基因座上广西汉族明显高于广东、海南汉族。

图1 中国16个汉族人群单倍群频率分布
红色字体代表其下游单倍群频率的加和。
2.3 不同地区汉族人群遗传关系及遗传分化
2.3.1 不同地区汉族人群Y-SNP遗传关系及遗传分化
基于不同地区汉族人群的Y-SNP单倍群频率的主成分分析(PCA)结果见图3A。在第一个主成分上显示了北部汉族(黑龙江、辽宁铁岭、山东、山西、北京和河南)、中部汉族(浙江、湖南、福建、江西)和南部汉族(广东、广西和海南)三个明显的遗传聚类簇,这与地理分布关系一致。北部汉族聚类紧密,而南部汉族聚类相对松散,表明南部汉族彼此之间的遗传分化程度比北部汉族大。西部汉族呈现明显的遗传差异,西南地区(四川阿坝、四川甘孜)的汉族人群与中部地区(湖南、福建、江西、浙江)汉族聚集在一起,西北地区的青海汉族与其他汉族人群显示出明显的遗传分化。
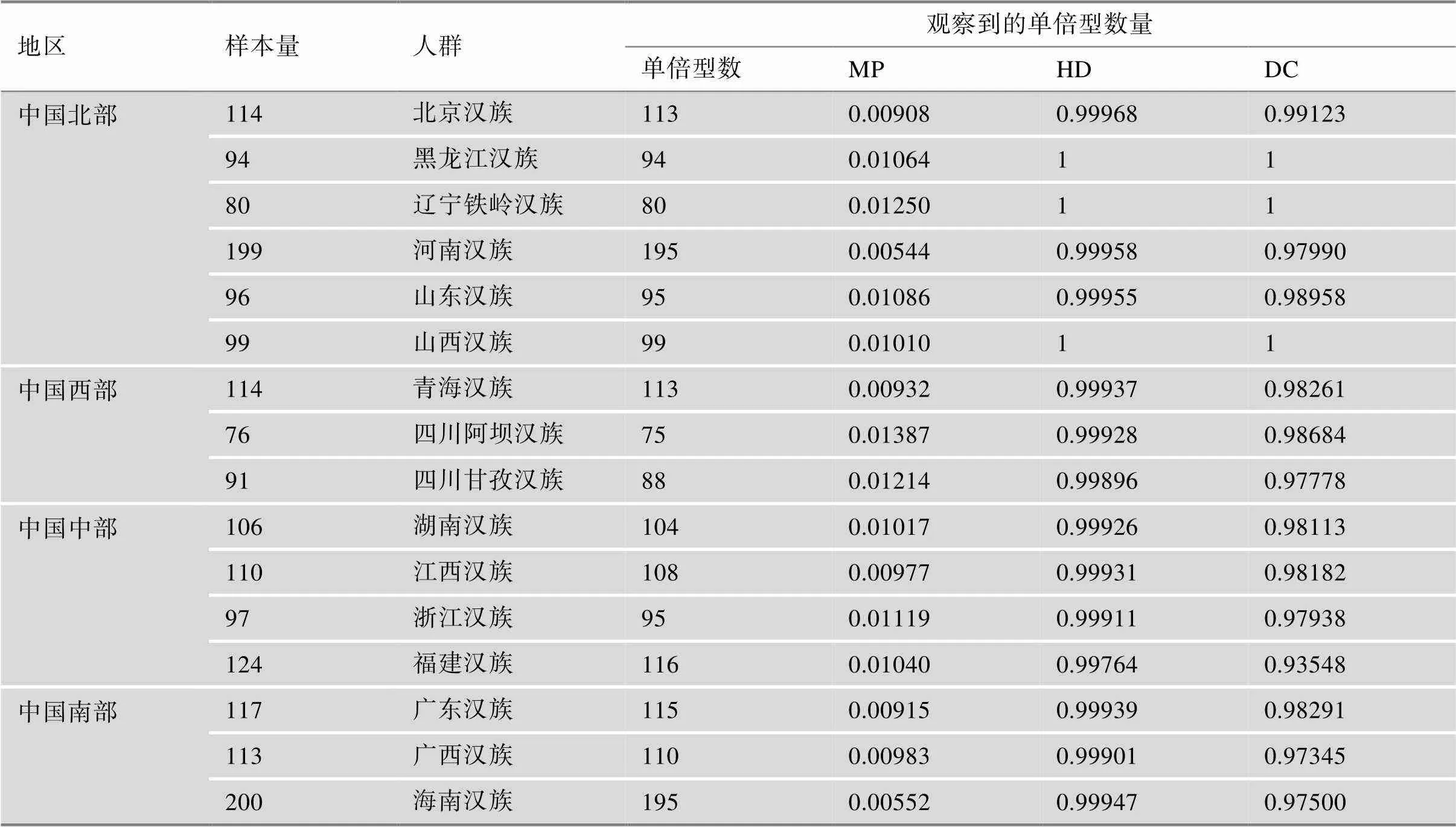
表2 基于16个Y-STR基因座的汉族人群法医学参数
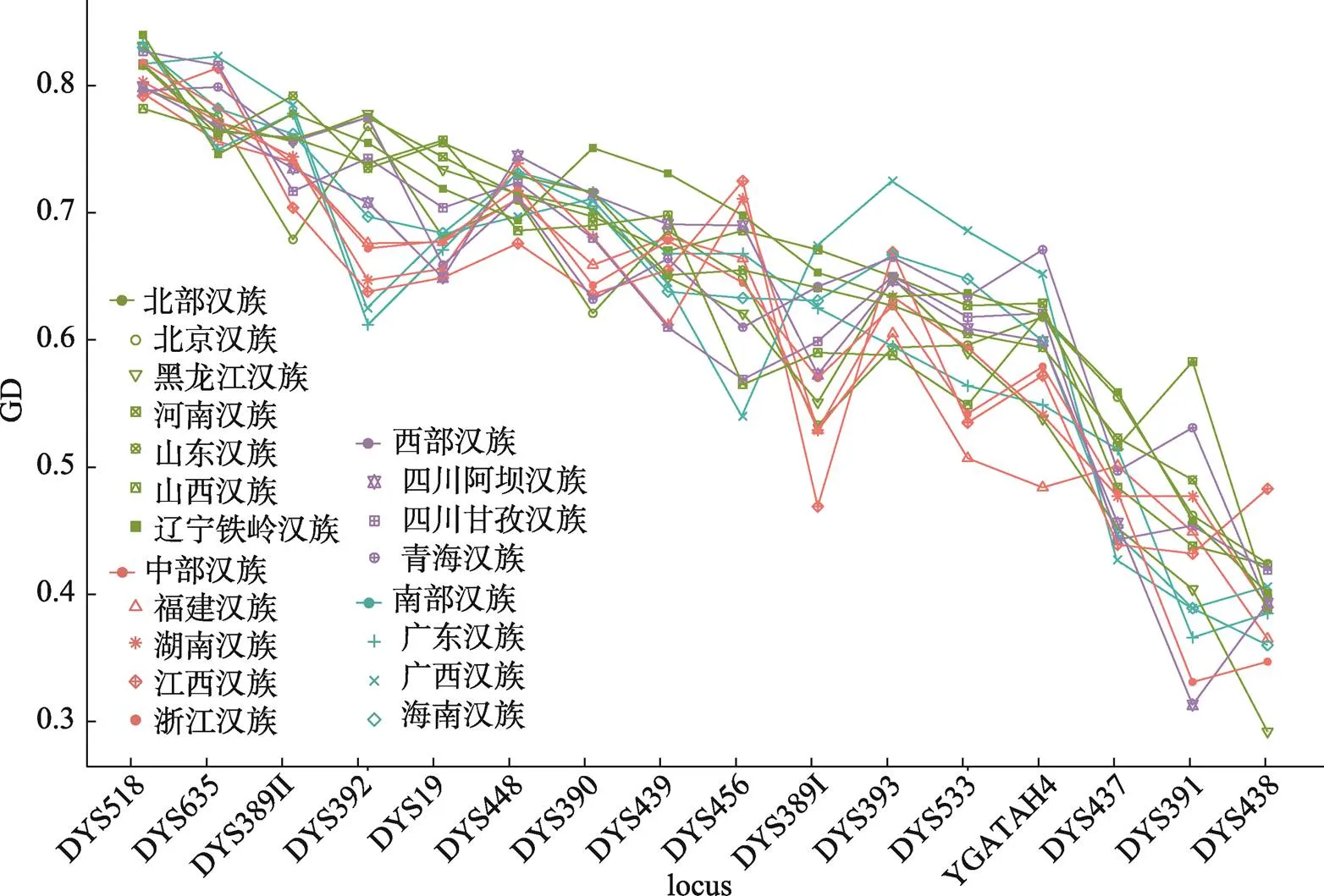
图2 16个Y-STR基因座在16个汉族人群中的基因多样性
2.3.2 不同地区汉族人群Y-STR遗传关系及遗传分化
为了进一步观察群体间的遗传分化关系,基于16个Y-STR的16个汉族人群遗传距离R值构建系统发育树见图3B,各地区汉族人群主要分成了三支,来自西北地区的青海汉族与北部地区(山东、山西、河南和辽宁铁岭)的汉族人群聚类成一簇形成北部汉族支系;中部地区(浙江、福建、江西和湖南)的汉族人群聚类在一起,同时西南地区的阿坝汉族与它们聚类较近形成中部汉族支系,表明它们的遗传关系更近;而甘孜汉族与受南部少数民族影响较深的华南地区汉族聚类较近形成南部汉族支系。黑龙江汉族和北京汉族在北部汉族和中部汉族之间。总之,不同地区的汉族人群遗传关系主要呈现出明显的北部、中部、南部汉族人群的遗传分化现象,遗传亲缘及分化关系与地理分布相关性强,其结果与图3A中PCA聚类结果相印证。
2.3.3 不同地区汉族人群单倍型网络分析
为了观察同一单倍群下Y-STR单倍型的个体关联,本研究选取了汉族人群主要分布的O2-M122单倍群的两大亚支及O1a-M119单倍群下的Y-STR进行了网络分析见图4。在网络图中观察到特定区域汉族的聚集分布,显示群体间单倍型共享的节点。在单倍群O2a2b1a1-M117、O2al-L127.1 (主要为O2a1b- IMS-JST002611)下的Y-STR单倍型网络图中,不同地区汉族个体随机分布在整个网络图中,且不同地区汉族人群共享中心单倍型,但南部(广东、广西、海南)汉族聚类在中心单倍型周围,北部(黑龙江、辽宁铁岭、山东、山西、北京和河南)、中部(浙江、湖南、福建、江西)汉族分散在末端单倍型的分布表明,北部(黑龙江、辽宁铁岭、山东、山西、北京和河南)、中部(浙江、湖南、福建、江西)汉族较南部(广东、广西、海南)汉族遗传分化程度高。
在单倍群O1a-M119下Y-STR单倍型主要以中部(浙江、湖南、福建、江西)及南部(广东、广西、海南)汉族人群共享单倍型为主,而北部汉族及西部汉族共享单倍型较少。此外在单倍群O1a-M119下Y-STR单倍型北部(黑龙江、辽宁铁岭、山东、山西、北京和河南)汉族聚类在中心单倍型周围,南部(广东、广西、海南)汉族及中部(浙江、湖南、福建、江西)汉族分散在末端单倍型的分布也表明,南部(广东、广西、海南)、中部(浙江、湖南、福建、江西)汉族较北部(黑龙江、辽宁铁岭、山东、山西、北京和河南)汉族遗传分化程度高。
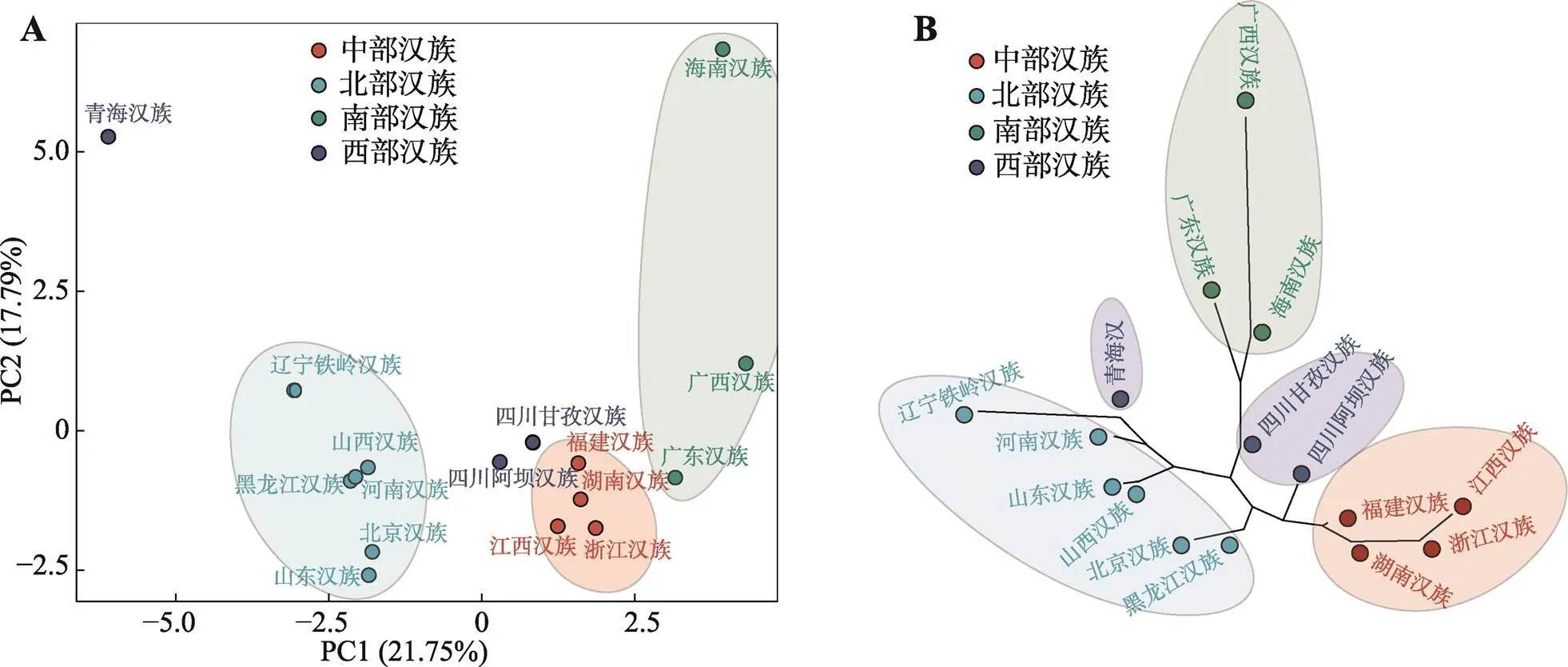
图3 汉族人群遗传聚类及遗传分化
A:不同地区汉族人群Y-SNP单倍群频率PCA图;B:不同地区汉族人群Y-STR系统发育关系图。
北部汉族:黑龙江汉族、辽宁铁岭汉族、北京汉族、山东汉族、山西汉族、河南汉族;中部汉族:湖南汉族、江西汉族、福建汉族、浙江汉族;西部汉族:四川甘孜汉族、四川阿坝汉族、青海汉族;南部汉族:广西汉族、海南汉族、广东汉族。
2.4 汉族人群与周边少数民族遗传关系
基于60Y-SNP数据集中38个中国人群的Y-SNP单倍群频率进行PCA分析,结果如图5A所示,中国人群分别形成了汉族人群聚类簇、藏缅语族(Tibeto-Burman)聚类簇、蒙古族聚类簇、回族及突厥语族(Turkic)聚类簇。遗传聚类与语系语族分类一致,同一语族人群聚类受地理因素影响呈现一定的遗传分化,如海南黎族和遵义仡佬族分别与地理关系相近的汉族聚集在一起,阿坝藏族与凉山彝族聚类较近,而与其他藏族聚类较远等。汉族人群彼此紧密聚集在一起,表明汉族人群彼此之间的遗传关系更近,另外观察到汉族表现出南部、中部、北部的遗传分化,与图3A的结果一致,南部汉族与侗台语系(Tai-Kadai)人群遗传交流更多,而北部汉族则与回族有一定的遗传交流,如南方汉族与黎族和仡佬族聚集在一起、四川回族及山东回族与北部汉族聚集在一起。基于该数据集中包含16Y-STR数据的样本网络分析结果显示:单倍群C2-M217下的北部汉族和西部的青海汉族与蒙古族人群共享单倍型,而南部汉族未观察到与蒙古族人群单独共享的单倍型支,其主要和各地区汉族人群共享(图5C),表明北部汉族C2-M217单倍群的分布受蒙古族人群影响,而南部汉族人群C2-M217单倍群的分布几乎不受其影响;单倍群O1b-M268下的汉族人群主要受侗台语人群影响,其中华南地区汉族影响最大,而北部汉族受影响较少(图5D)。
基于16Y-STR数据集的45个中国人群的群体间R值绘制热图,如图5B所示,各个人群遗传关系与语系语族分类有较强的相关性,同一语族人群倾向于聚类在一起,其结果与图3A和B中PCA和发育树聚类结果相印证。不同人群遗传关系还受地理分布影响,如广西汉族与广西壮族聚在一起,而北部汉族则与山东、河南、四川等地的回族聚集在一起,西北地区回族倾向于与阿尔泰语系人群聚在一支。总之,不同人群遗传关系同时受语系语族分类及地理隔离因素影响。
3 讨论
中国是东亚地区最大的国家,拥有丰富多样的语言体系,包括汉藏语系(Sino-Tibetan)、阿尔泰语系(Altaic)、侗台语系、苗瑶语系(Hmong-Mien)、南亚语系(Austro-Asiatic)、南岛语系(Austronesian)及印欧语系(Indo-European)[61]。这里有全世界人口最多的民族——汉族,汉族属于汉藏语系的汉语族人群,主要分布在中国东部,在西部主要与其他语系人群杂居在一起。大量的遗传学研究报道汉族人群起源于黄河流域的仰韶文化,并随着农业的驯化,仰韶文化由中原地区逐渐向西向南及向北迁移、定居,并与各地少数民族交流融合[31,62~64]。对现代不同地区汉族人群的遗传学研究也表现出汉族明显的遗传分化,如基于全基因组WBBC中将汉族分为北部、中部、南部及岭南四个地理亚群,ChinaMap中根据地理分区将中国汉族分了西北、北部、东部、中部、东南、南部和岭南七个地理亚群,基于线粒体的遗传研究中汉族人群依据水系划分了黄河流域、长江流域和珠江流域三个明显的亚群,Y染色体视角的研究也表明汉族人群存在明显的南北分化[34,36~38]。基于前人从地理角度及语系语族分类角度对汉族人群起源、迁移与演化的研究[31,65],本研究整合了1830份汉族人群的89Y-SNP、16Y-STR数据。结果显示89Y-SNP、16Y-STR位点在不同地区汉族人群中具有显著的遗传多态性。群体遗传学研究中表明不同地域汉族人群主要表现为北部、中部及南部三个聚类,其中青海汉族与其他地区汉族存在一定的遗传分化。合并中国其他人群的遗传关系研究显示,中国人群父系遗传关系受语系语族分类及地理因素影响,汉族人群彼此之间遗传关系更紧密,但北部汉族与回族遗传关系更近,而南部汉族则与仡佬族、黎族等南方少数民族的遗传关系更近。

A:汉族人群与其他中国人群单倍群频率PCA图;B:汉族人群与中国其他人群遗传距离热图;C:基于7个Y-STR基因座分型的不同地区汉族及蒙古族中单倍群C2-M217下Y-STR单倍型网络图;D:基于7个Y-STR基因座分型的不同地区汉族及蒙古族单倍群O1b-M268下Y-STR单倍型网络图。北部汉族:黑龙江汉族、辽宁铁岭汉族、北京汉族、山东汉族、山西汉族、河南汉族;中部汉族:湖南汉族、江西汉族、福建汉族、浙江汉族;西部汉族:四川甘孜汉族、四川阿坝汉族、青海汉族;南部汉族:广西汉族、海南汉族、广东汉族。
3.1 汉族人群Y-SNP的遗传多样性分析
本研究检测的89个Y-SNP,包含了CT-M168、D-M174、C-M130、J-M304、G-M201、N-M231、O-M175、Q-M242和R-M207等单倍群,在1830份样本中共观察到63个不同的末端单倍群(图1)。单倍群O2-M122是东亚地区最常见的父系单倍群[62,66,67],在本次研究的各个汉族人群(广西汉族除外)分布中占比最高(青海汉族46.9%-山西汉族65.5%)。单倍群O2-M122有三个主要的亚分支,分别为O2a1b- IMS-JST002611、O2a2b1-M134和O2a2b1a1-M117,分别占汉族人群的12%-17%[15,62,68,69],在本研究中这三个分支也是汉族人群的主要分支,其中O2a2b1a1-M117、O2a1b-IMS-JST002611在各地汉族人群中都分布较高,它们的网络分析显示Y-STR单倍型多样性普遍呈北部、中部向南下降(图4),这与O2-M122谱系在历史时期随农业的驯化由北向南迁移一致[67,69]。单倍群O2a2a-P201、O1a-M119是苗瑶语、侗台语及南岛语系人群的特征单倍群[70,71],本研究中中部地区汉族单倍群O1a-M119占比高于其他地区汉族,可能是受到周边少数民族的影响,如瑶族、畲族、侗族等,四川地区汉族人群单倍群O1a-M119分布频率也较高,这可能是受良渚文化携带O1a-M119西迁影响[31]。单倍群O1b-M268主要分布在东亚南部及东南亚地区,其中在东亚南部主要分布在侗台语系及南亚语系人群[72,73],本研究的汉族人群中单倍群O1b-M268分布也呈现由南向北递减趋势,尤其在岭南地区(广西汉族38%、海南汉族21.5%)汉族人群中分布频率较高,这可能是受当地侗台语系人群的影响,如黎族等,这与在单倍群O1b-M268的网络图中观察到的结果一致(图5D)。单倍群C2-M217是阿尔泰语系人群的主体单倍群,尤其在蒙古语族及通古斯语族人群中[74~76],单倍群C2-M217也是本研究中汉族人群的第二大单倍群(福建汉族3.2%~河南汉族15.5%),且其在北部及西北部汉族人群中分布较高(河南汉族15.5%、青海汉族12.15%、黑龙江汉族12.8%、山西汉族12.1%、辽宁铁岭汉族11.25%),可能是受北部和西北部地区的阿尔泰语系人群影响,这与在C2-M217单倍群构建的网络图中观察到的结果一致(图5C)。单倍群N-M231主要分布于北亚地区[77],在东亚地区分布也呈现出一定的由北向南递减的趋势(黑龙江汉族10.7%、青海汉族10.53%、山东汉族12.5%,在南部汉族人群中均低于10%)。单倍群D1-M174主要分布在藏缅语族人群[48,78,79],在本次研究中单倍群D1-M174在青藏高原周边地区及北部地区低频分布(甘孜汉族5.49%、广西汉族4.4%、辽宁铁岭汉族3.75%),在东南部平原地区几乎不分布,这可能是青藏高原周边地区的汉族与当地藏缅语族人群遗传交流造成的。源于欧亚西部的单倍群R-M207(0%~ 4.39%)、J-M304(0%~1.75%),在东亚地区主要是阿尔泰语系人群携带,本研究中零散分布于北部汉族,南部汉族极少,这可能是因为北部汉族受阿尔泰语系人群影响造成[66]。
3.2 汉族人群Y-STR的遗传多样性分析
16个单拷贝 Y-STR 基因座在1830个汉族个体中检测到1737个单倍型。北部汉族人群单倍型多样性(HD)普遍高于其他地区,总体呈现由北向南递减的趋势,这可能与汉族人群的北部起源而后向西向南迁徙有关[31]。在本研究中携带DYS448del的个体均归属到C2-M217单倍群,这与之前的研究一致[74]。DYS448位于无精子症基因c (Azoospermia factor c,AZFc)区域的近端,AZFc区域的缺失可能导致DYS448[80]处存在空等位基因,引物结合区单个碱基的变化也可能导致DYS448del。此外在DYS19基因座上,有75%存在二等位基因变异的个体属于C2a1a2-M48单倍群,这与之前的研究[81]一致。微变异可能是由单倍群特异性的祖先Y染色体突变引起的,一般来说,在某些Y-STR位点上发生的不同微变异通常与特定的单倍群相关,这可以为利用Y-STR单倍型[82,83]预测单倍群提供理论基础,同时这些Y-STR基因座的多样性也是形成单倍型多样性的基础,为推断个体家系来源提供数据支撑。然而本研究中涉及的Y-STR基因座数量较少且均为单拷贝基因座,而多拷贝和快速突变Y-STR基因座的多样性通常更高,增加Y-STR基因座数目及类型以观察到更高的单倍型多样性。
3.3 不同地区汉族人群遗传关系及遗传分化
在汉族人群间遗传关系的研究中,基于Y-SNP单倍群的PCA分析及基于Y-STR单倍型分析结果都表现出北部汉族、中部汉族和南部汉族三个明显的遗传聚类。同时,北部汉族人群聚类紧密,而南部汉族人群中形成了一个相对松散的聚类,这表明南部汉族间的遗传分化程度比北部汉族间大(图3)。例如:观察到处于秦岭淮河以南的湖南、江西、福建、浙江及四川的汉族与岭南地区的广西、海南等汉族呈现明显遗传分化,这种差异与长期的历史和地理因素以及东亚南部民族多样性更高、各人群间混居交流更多有关[15,23,64,65]。本研究发现西北、西南地区汉族人群间有一定的遗传分化,其中,西北地区的青海汉族与其他汉族表现出明显的遗传分化(图3,A和B),这可能由于仰韶文化一支早期西迁并在青海地区定居,与当地的少数民族混合,另外,仰韶文化及红山文化南迁沿东部不同纬度定居,在不同时期分别与大汶口文化、大溪文化、良渚文化混合[31],长时间的遗传交流使得青海汉族与其他汉族,北部、中部及南部汉族之间有着不同程度的遗传分化关系,这在全基因组研究中也得到了证实[36];而西南地区(四川阿坝、四川甘孜)的汉族表现出与中部(浙江、湖南、福建、江西)汉族更近的遗传关系,这可能受距今5300~4000年前后环钱塘江分布的良渚文化(O1a-M119)西迁的影响[31]。总之,不同地区的汉族人群遗传关系主要呈现出明显的北部、中部、南部的遗传分化现象,遗传亲缘及分化关系与地理分布相关性强。另外在基于单倍群频率的PCA分析中发现青海汉族与东部其他汉族人群呈现出一定的遗传分化,但本研究中西部地区汉族人群采样量有限,未来仍需更多的西部汉族人群样本进一步研究东西部汉族人群遗传分化关系。
3.4 汉族人群与周边少数民族之间的遗传关系
基于中国人群Y-STR和Y-SNP数据集获得的遗传关系表明,Y染色体视角可以很好地揭示语系语族分类/地理上不同群体之间的遗传关系。总体来说,中国人群父系遗传关系与语系语族分类联系紧密,各人群按照语系语族分类及地理分区很好的聚类,同一语族的各亚人群受地理分布影响表现出一定的遗传分化现象,这也与以往研究一致[42]。基于中国人群的遗传研究表明不同地区汉族人群受地理因素及与当地少数民族基因交流影响呈现出一定的遗传分化,南部汉族与侗台语系人群遗传关系更近,例如:PCA及O1b-M268的单倍型网络分析结果表明南部汉族受仡佬族、黎族遗传影响较多(图5,A和D),单倍群O1b-M268在起源于南部百越氏族的侗台语系人群占主体,南部汉族与南部少数民族的遗传交流是导致南北部汉族遗传分化的原因之一[27,42,47,73];而北部汉族与回族人群有一定的遗传交流,例如:山东回族和四川回族都表现出与北部汉族较近的遗传关系(图5,A和B),这可能是由于回族人群长期与汉族人群混居并不断发生遗传交流所致[84],这一点在全基因组相关研究中更为明显[85]。单倍群C2-M217下的Y-STR网络分析中北部汉族和西部的青海汉族和蒙古语族人群共享单倍型,而南部汉族未观察到单独与蒙古语族人群共享的单倍型,主要和其他汉族人群共享(图5C),这表明汉族人群单倍群C2-M217的分布起源不同,北部汉族C2-M217的分布受蒙古语族人群影响,而南部汉族C2-M217的分布则受蒙古语族影响较少,可能是C2-M217史前由南向北迁徙后未向南折返至中国南部[15,86]。在基于群体间R值的研究中还观察到藏缅语族的凉山彝族及北川羌族与北部汉族聚在一起,这可能与汉藏语系的共同起源有关[31]。总之,中国人群父系遗传关系受语系语族分类和地理因素影响。
综上所述,本研究检测了362个汉族样本89个Y-SNP、16个Y-STR数据,并与从文献中收集的1468名汉族样本89个Y-SNP和16个Y-STR数据进行整合,单倍群分类结果显示,汉族人群主要分布的单倍群包括O2-M122、O1a-M119、O1b-M268 、C2-M217、N-M231,低频分布的单倍群包括D1-M174、G-M201、I-M170、J-M304、Q-M242,各个汉族人群Y-SNP单倍群分布呈现出一定的地域差异性。同时检测到一些Y-STR基因座上的空等位变异及微变异与特定的Y-SNP单倍群相关,为通过Y-STR单倍型推断Y-SNP单倍群提供了理论依据。基于汉族人群Y-SNP单倍群频率的PCA及Y-STR的系统发育树结果显示汉族人群主要包括北部、中部及南部三个聚类簇,为汉族群体的父系遗传结构提供了遗传证据,另外发现来自青海的汉族与其他汉族在单倍群频率PCA上有明显的遗传分化。对汉族与中国其他人群父系遗传关系的研究表明,中国群体父系遗传结构与语系语族分类和地理划分高度一致,不同地区汉族人群与蒙古语族、突厥语族、藏缅语族、侗台语系人群和回族等少数民族人群相比,汉族人群之间遗传亲缘性更强,且可以观察到南部汉族与南部少数民族之间遗传交流更频繁。综上,中国族群的Y-SNP系统发育树结合Y-STR单倍型、地理来源不同的汉族群体遗传证据,对研究汉族群体的人群亚结构是必不可少的。未来可纳入更多地理区域的样本和更多的遗传标记集,进一步探索精细人群遗传结构并提高Y染色体遗传标记在法医学领域的应用价值。
[1] Jobling MA, Tyler-Smith C. Human Y-chromosome variation in the genome-sequencing era., 2017, 18(8): 485–497.
[2] Batini C, Jobling MA. Detecting past male-mediated expansions using the Y chromosome., 2017, 136(5): 547–557.
[3] King TE, Jobling MA. Founders, drift, and infidelity: the relationship between Y chromosome diversity and patrilineal surnames., 2009, 26(5): 1093–1102.
[4] Charchar FJ, Bloomer LD, Barnes TA, Cowley MJ, Nelson CP, Wang Y, Denniff M, Debiec R, Christofidou P, Nankervis S, Dominiczak AF, Bani-Mustafa A, Balmforth AJ, Hall AS, Erdmann J, Cambien F, Deloukas P, Hengstenberg C, Packard C, Schunkert H, Ouwehand WH, Ford I, Goodall AH, Jobling MA, Samani NJ, Tomaszewski M. Inheritance of coronary artery disease in men: an analysis of the role of the Y chromosome., 2012, 379(9819): 915–922.
[5] Khan K, Siddiqi MH, Abbas M, Almas M, Idrees M. Forensic applications of Y chromosomal properties., 2017, (26): 86–91.
[6] Kayser M. Forensic use of Y-chromosome DNA: a general overview., 2017, 136(5): 621–635.
[7] Santos FR, Epplen JT, Pena SD. Testing deficiency paternity cases with a Y-linked tetranucleotide repeat polymorphism., 1993, (67): 261–265.
[8] Corach D, Filgueira Risso L, Marino M, Penacino G, Sala A. Routine Y-STR typing in forensic casework., 2001, 118(2–3): 131–135.
[9] Pinto N, Gusmão L, Amorim A. Mutation and mutation rates at Y chromosome specific Short Tandem Repeat Polymorphisms (STRs): a reappraisal., 2014, (9): 20–24.
[10] Laouina A, Nadifi S, Boulouiz R, El Arji M , Talbi J , El Houate B , Yahia H , Chbel F. Mutation rate at 17 Y-STR loci in "Father/Son" pairs from moroccan population., 2013, 15(5): 269–271.
[11] Ballantyne KN, Goedbloed M, Fang RX, Schaap O, Lao O, Wollstein A, Choi Y, van Duijn K, Vermeulen M, Brauer S, Decorte R, Poetsch M, von Wurmb-Schwark N, de Knijff P, Labuda D, Vézina H, Knoblauch H, Lessig R, Roewer L, Ploski R, Dobosz T, Henke L, Henke J, Furtado MR, Kayser M. Mutability of Y-chromosomal microsatellites: rates, characteristics, molecular bases, and forensic implications., 2010, 87(3): 341–353.
[12] Claerhout S, Vandenbosch M, Nivelle K, Gruyters L, Peeters A, Larmuseau MHD, Decorte R. Determining Y-STR mutation rates in deep-routing genealogies: Identification of haplogroup differences., 2018, 34: 1–10.
[13] Wickenheiser RA. Expanding DNA database effectiveness., 2022, 4: 100226.
[14] Xue YL, Wang QJ, Long Q, Ng BL, Swerdlow H, Burton J, Skuce C, Taylor R, Abdellah Z, Zhao YL, Asan, MacArthur DG, Quail MA, Carter NP, Yang HM, Tyler-Smith C. Human Y chromosome base-substitution mutation rate measured by direct sequencing in a deep-rooting pedigree., 2009, 19(17): 1453– 1457.
[15] Wang CC, Li H. Inferring human history in East Asia from Y chromosomes., 2013, 4(1): 11.
[16] Yoshida Y, Kubo S. Y-SNP and Y-STR analysis in a Japanese population., 2008, 10(5): 243–252.
[17] Cortellini V, Verzeletti A, Cerri N, Marino A, De Ferrari F. Y-chromosome polymorphisms and ethnic group—a combined STR and SNP approach in a population sample from northern Italy., 2013, 54(3): 279–285.
[18] Song MY, Song F, Zhao CX, Hou YP. YHP: Y-chromosome Haplogroup Predictor for predicting male lineages based on Y-STRs., 2021.
[19] Yin CY, Sun H, Zhou HG, Jin L, Li SL. EA-YPredictor: one new software developed to predict pedigree haplogroup based on Y-STR haplotypes., 2020, 45(2): 117–124.殷才湧, 孙辉, 周怀谷, 金立, 李士林. EA-YPredictor:基于Y-STR数据的家系特异性单倍群归属判别分析软件. 刑事技术, 2020, 45(2): 117–124.
[20] Dogan S, Babic N, Gurkan C, Goksu A, Marjanovic D, Hadziavdic V. Y-chromosomal haplogroup distribution in the Tuzla Canton of Bosnia and Herzegovina: a concordance study using four different in silico assignment algorithms based on Y-STR data., 2016, 67(6): 471–483.
[21] Ralf A, Van Oven M, Zhong KY, Kayser M. Simultaneous analysis of hundreds of Y-chromosomal SNPs for high-resolution paternal lineage classification using targeted semiconductor sequencing., 2015, 36(1): 151–159.
[22] Xie MK, Song F, Li JN, Lang M, Luo HB, Wang Z, Wu J, Li CZ, Tian CC, Wang WZ, Ma H, Song Z, Fan YJ, Hou YP. Genetic substructure and forensic characteristics of Chinese Hui populations using 157 Y-SNPs and 27 Y-STRs., 2019, 41: 11–18.
[23] Wang MG, Wang Z, He GL, Liu J, Wang SY, Qian XQ, Lang M, Li JN, Xie MK, Li CT, Hou YP. Developmental validation of a custom panel including 165 Y-SNPs for Chinese Y-chromosomal haplogroups dissection using the ion S5 XL system., 2019, 38: 70–76.
[24] Yin CY, Ren YJ, Adnan A, Tian JZ, Guo KJ, Xia MY, He ZW, Zhai D, Chen XY, Wang L, Li X, Qin XJ, Li SL, Jin L. Title: developmental validation of Y-SNP pedigree tagging system: a panel via quick ARMS PCR., 2020, 46: 102271.
[25] Zhou ZH, Zhou YX, Yao YN, Qian JL, Liu BN, Yang QR, Shao CC, Li H, Sun K, Tang QQ, Xie JH. A 16-plex Y-SNP typing system based on allele-specific PCR for the genotyping of Chinese Y-chromosomal haplogroups., 2020, 46: 101720.
[26] Claerhout S, Verstraete P, Warnez L, Vanpaemel S, Larmuseau M, Decorte R. CSYseq: the first Y-chromosome sequencing tool typing a large number of Y-SNPs and Y-STRs to unravel worldwide human population genetics., 2021, 17(9): e1009758.
[27] Tao RY, Li M, Chai SY, Xia RC, Qu YL, Yuan CL, Yang GY, Dong XY, Bian YN, Zhang SH, Li CT. Developmental validation of a 381 Y-chromosome SNP panel for haplogroup analysis in the Chinese populations., 2023, 62: 102803.
[28] He GL, Wang MG, Miao L, Chen J, Zhao J, Sun QX, Duan SH, Wang ZY, Xu XF, Sun YT, Liu Y, Liu J, Wang Z, Wei LH, Liu C, Ye J, Wang L. Multiple founding paternal lineages inferred from the newly-developed 639-plex Y-SNP panel suggested the complex admixture and migration history of Chinese people., 2023, 17(1): 29.
[29] Liu J, Jiang LR, Zhao MY, Du WA, Wen YF, Li SY, Zhang SY, Fang FF, Shen J, He GL, Wang MG, Dai H, Hou YP, Wang Z. Development and validation of a custom panel including 256 Y-SNPs for Chinese Y-chromosomal haplogroups dissection., 2022, 61: 102786.
[30] 中华人民共和国国家统计局. 国家数据-2021年度第七次人口普查. (2021-11-26)[2023-10-11]. http://www. stats.gov.cn/sj/pcsj/rkpc/d7c/.
[31] Yu XE, Li H. Origin of ethnic groups, linguistic families, and civilizations in China viewed from the Y chromosome., 2021, 296(4): 783–797.
[32] Li YC, Ye WJ, Jiang CG, Zeng Z, Tian JY, Yang LQ, Liu KJ, Kong QP. River valleys shaped the maternal genetic landscape of Han Chinese., 2019, 36(8): 1643–1652.
[33] Lang M, Liu H, Song F, Qiao XH, Ye Y, Ren H, Li JN, Huang J, Xie MK, Chen SJ, Song MY, Zhang YF, Qian XQ, Yuan TX, Wang Z, Liu YM, Wang MG, Liu YC, Liu J, Hou YP. Forensic characteristics and genetic analysis of both 27 Y-STRs and 143 Y-SNPs in Eastern Han Chinese population., 2019, 42: e13–e20.
[34] Gu JQ, Jiang L, Xu JY, Wang H, Wei YL, Li CX. Genetic structure of East Asians based on high-density SNP data., 2023, 50(11): 1–14.顾佳琪, 江丽, 徐景怡, 王寒, 魏以梁, 李彩霞. 基于高密度SNP数据的东亚人群遗传结构研究. 生物化学与生物物理进展, 2023, 50(11): 1–14.
[35] Zhang XY, Lu L, Yu H, Zhang X, Deng HB. Research on the distribution characteristics and formation mechanisms of Chinese traditional villages., 2023, 32(4): 132–143.张晓瑶, 陆林, 虞虎, 张潇, 邓洪波. 中国传统村落分布特征与成因机制研究. 世界地理研究, 2023, 32(4): 132–143.
[36] Chiang CWK, Mangul S, Robles C, Sankararaman S. A comprehensive map of genetic variation in the world's largest ethnic group-Han Chinese., 2018, 35(11): 2736–2750.
[37] Xu SH, Yin XY, Li SL, Jin WF, Lou HY, Yang L, Gong XH, Wang HY, Shen YP, Pan XD, He YG, Yang YJ, Wang Y, Fu WQ, An Y, Wang JC, Tan JZ, Qian J, Chen XL, Zhang X, Sun YF, Zhang XJ, Wu BL, Jin L. Genomic dissection of population substructure of Han Chinese and its implication in association studies., 2009, 85(6): 762–774.
[38] Cao YN, Li L, Xu M, Feng ZM, Sun XH, Lu JL, Xu Y, Du PN, Wang TG, Hu RY, Ye Z, Shi LX, Tang XL, Yan L, Gao ZN, Chen G, Zhang YF, Chen LL, Ning G, Bi YF, Wang WQ, ChinaMAP Consortium. The ChinaMAP analytics of deep whole genome sequences in 10,588 individuals., 2020, 30(9): 717–731.
[39] Yin CY, Su KY, He ZW, Zhai D, Guo KJ, Chen XY, Jin L, Li SL. Genetic reconstruction and forensic analysis of Chinese Shandong and Yunnan Han Populations by co-analyzing Y Chromosomal STRs and SNPs., 2020, 11(7): 743.
[40] Zhang X, Tang Z, Wang B, Zhou XD, Zhou LM, Zhang GY, Tian JZ, Zhao YQ, Yao ZQ, Tian L, Zhang SH, Xia H, Jin L, Li CT, Li SL. Forensic analysis and genetic structure construction of Chinese Chongming Island Han based on Y Chromosome STRs and SNPs., 2022, 13(8): 1363.
[41] Zhang YL, Zhang R, Li M, Luo L, Zhang JY, Ding JD, Zhang SH, Li CT, Bian YN, Zhou CJ. Genetic polymorphism of both 29 Y-STRs and 213 Y-SNPs in Han populations from Shandong Province, China., 2020, 47:101738.
[42] Song MY, Wang Z, Zhang YQ, Zhao CX, Lang M, Xie MK, Qian XQ, Wang MG, Hou YP. Forensic characteristics and phylogenetic analysis of both Y-STR and Y-SNP in the Li and Han ethnic groups from Hainan Island of China., 2019, 39: e14–e20.
[43] Song F, Song MY, Luo HB, Xie MK, Wang XD, Dai H, Hou YP. Paternal genetic structure of Kyrgyz ethnic group in China revealed by high-resolution Y-chromosome STRs and SNPs., 2021, 42(19): 1892–1899.
[44] Wang MG, He GL, Zou X, Liu J, Ye ZW, Ming TY, Du WA, Wang Z, Hou YP. Genetic insights into the paternal admixture history of Chinese Mongolians via high- resolution customized Y-SNP SNaPshot panels., 2021, 54: 102565.
[45] Song MY, Wang ZF, Lyu Q, Ying J, Wu Q, Jiang LR, Wang F, Zhou YX, Song F, Luo HB, Hou YP, Song XB, Ying BW. Paternal genetic structure of the Qiang ethnic group in China revealed by high-resolution Y-chromosome STRs and SNPs., 2022, 61: 102774.
[46] Wang F, Song F, Song MY, Li JN, Xie MK, Hou YP. Genetic reconstruction and phylogenetic analysis by 193 Y-SNPs and 27 Y-STRs in a Chinese Yi ethnic group., 2021, 42(14–15): 1480–1487.
[47] Wang F, Song F, Song MY, Luo HB, Hou YP. Genetic structure and paternal admixture of the modern Chinese Zhuang population based on 37 Y-STRs and 233 Y-SNPs., 2022, 58: 102681.
[48] Wang XJ, Qian EF, Li Y, Song ZY, Zhao H, Xie HX, Li CX, Huang J, Jiang L. A genetic sub-structure study of the Tibetan population in Southwest China., 2020, 42(6): 565–576.王小娟, 钱恩芳, 李悦, 宋正阳, 赵慧, 谢何鑫, 李彩霞, 黄江, 江丽. 中国西南地区藏族人群遗传亚结构研究. 遗传, 2020, 42(6): 565–576.
[49] Wang XJ, Jiang L, Qian EF, Long F, Cui W, Ji AQ, Zhang FS, Zou K, Huang J, Li CX. Genetic polymorphisms and haplotypic structure analysis of the Guizhou Gelao ethnic group based on 35 Y-STR loci., 2020, 43: 101666.
[50] Trejaut JA, Poloni ES, Yen JC, Lai YH, Loo JH, Lee CL, He CL, Lin M. Taiwan Y-chromosomal DNA variation and its relationship with Island Southeast Asia., 2014, 15: 77.
[51] Wang H, Mao J, Xia Y, Bai XG, Zhu WQ, Peng D, Liang WB. Genetic polymorphisms of 17 Y-chromosomal STRs in the Chengdu Han population of China., 2017, 131(4): 967–968.
[52] Zhu BF, Wu YM, Shen CM, Yang TH, Deng YJ, Xun X, Tian YF, Yan JC, Li T. Genetic analysis of 17 Y-chromosomal STRs haplotypes of Chinese Tibetan ethnic group residing in Qinghai province of China., 2008, 175(2–3): 238–243.
[53] ISOGG.Y-DNA haplogroup tree. (2019-10-08)[2022- 11-22]. https://isogg.org/tree/index.html.
[54] Nei M. Analysis of gene diversity in subdivided populations., 1973, 70(12): 3321–3323.
[55] Nei M, Tajima F. DNA polymorphism detectable by restriction endonucleases., 1981, 97(1): 145–163.
[56] Ito K, Murphy D. Application of ggplot2 to pharmacometric graphics., 2013, 2(10): e79.
[57] Excoffier L, Lischer HE. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows., 2010, 10(3): 564–567.
[58] Excoffier L, Smouse PE, Quattro JM. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data., 1992, 131(2): 479–491.
[59] Kumar S, Stecher G, Li M, Knyaz C, Tamura K. MEGA X: molecular evolutionary genetics analysis across computing platforms., 2018, 35(6): 1547–1549.
[60] Bandelt HJ, Forster P, Röhl A. Median-joining networks for inferring intraspecific phylogenies., 1999, 16(1): 37–48.
[61] 蔡晓云. Y染色体揭示的早期人类进入东亚和东亚人群特征形成过程[学位论文]. 复旦大学, 2009.
[62] Yan S, Wang CC, Zheng HX, Wang W, Qin ZD, Wei LH, Wang Y, Pan XD, Fu WQ, He YG, Xiong LJ, Jin WF, Li SL, An Y, Li H, Jin L. Y chromosomes of 40% Chinese descend from three Neolithic super-grandfathers., 2014, 9(8): e105691.
[63] Sagart L, Jacques G, Lai YF, Ryder RJ, Thouzeau V, Greenhill SJ, List JM. Dated language phylogenies shed light on the ancestry of Sino-Tibetan., 2019, 116(21): 10317–10322.
[64] Wang LX, Lu Y, Zhang C, Wei LH, Yan S, Huang YZ, Wang CC, Mallick S, Wen SQ, Jin L, Xu SH, Li H. Reconstruction of Y-chromosome phylogeny reveals two neolithic expansions of Tibeto-Burman populations., 2018, 293(5): 1293–1300.
[65] Wang CC, Yeh HY, Popov AN, Zhang HQ, Matsumura H, Sirak K, Cheronet O, Kovalev A, Rohland N, Kim AM, Mallick S, Bernardos R, Tumen D, Zhao J, Liu YC, Liu JY, Mah M, Wang K, Zhang Z, Adamski N, Broomandkhoshbacht N, Callan K, Candilio F, Carlson KSD, Culleton BJ, Eccles L, Freilich S, Keating D, Lawson AM, Mandl K, Michel M, Oppenheimer J, Özdoğan KT, Stewardson K, Wen SQ, Yan S, Zalzala F, Chuang R, Huang CJ, Looh H, Shiung CC, Nikitin YG, Tabarev AV, Tishkin AA, Lin S, Sun ZY, Wu XM, Yang TL, Hu X, Chen L, Du H, Bayarsaikhan J, Mijiddorj E, Erdenebaatar D, Iderkhangai TO, Myagmar E, Kanzawa-Kiriyama H, Nishino M, Shinoda KI, Shubina OA, Guo JX, Cai WW, Deng QY, Kang LL, Li DW, Li DN, Lin R, Nini, Shrestha R, Wang LX, Wei LH, Xie GM, Yao HB, Zhang MF, He GL, Yang XM, Hu R, Robbeets M, Schiffels S, Kennett DJ, Jin L, Li H, Krause J, Pinhasi R, Reich D. Genomic insights into the formation of human populations in East Asia., 2021, 591(7850): 413–419.
[66] Zhong H, Shi H, Qi XB, Duan ZY, Tan PP, Jin L, Su B, Ma RZ. Extended Y chromosome investigation suggests postglacial migrations of modern humans into East Asia via the northern route., 2011, 28(1): 717–727.
[67] Shi H, Dong YL, Wen B, Xiao CJ, Underhill PA, Shen PD, Chakraborty R, Jin L, Su B. Y-chromosome evidence of southern origin of the East Asian-specific haplogroup O3-M122., 2005, 77(3): 408–419.
[68] Ning C, Yan S, Hu K, Cui YQ, Jin L. Refined phylogenetic structure of an abundant East Asian Y-chromosomal haplogroup O*-M134., 2016, 24(2): 307–309.
[69] Kang LL, Lu Y, Wang CC, Hu K, Chen F, Liu K, Li SL, Jin L, Li H, Genographic Consortium. Y-chromosome O3 haplogroup diversity in Sino-Tibetan populations reveals two migration routes into the eastern Himalayas.2012, 76(1): 92–99.
[70] Zhang XM, Kampuansai J, Qi XB, Yan S, Yang ZH, Serey B, Sovannary T, Bunnath L, Aun HS, Samnom H, Kutanan W, Luo X, Liao SY, Kangwanpong D, Jin L, Shi H, Su B. An updated phylogeny of the human Y-chromosome lineage O2a-M95 with novel SNPs., 2014, 9(6): e101020.
[71] Sun J, Li YX, Ma PC, Yan S, Cheng HZ, Fan ZQ, Deng XH, Ru K, Wang CC, Wang CC, Chen G, Wei LH. Shared paternal ancestry of Han, Tai-Kadai-speaking, and Austronesian-speaking populations as revealed by the high resolution phylogeny of O1a-M119 and distribution of its sub-lineages within China., 2021, 174(4): 686–700.
[72] Yan S, Wang CC, Li H, Li SL, Jin L, Genographic Consortium. An updated tree of Y-chromosome Haplogroup O and revised phylogenetic positions of mutations P164 and PK4., 2011, 19(9): 1013–1015.
[73] Chen H, Lin R, Lu Y, Zhang R, Gao Y, He YG, Xu SH. Tracing Bai-Yue ancestry in aboriginal Li People on Hainan Island., 2022, 39(10): msac210.
[74] Wei LH, Huang YZ, Yan S, Wen SQ, Wang LX, Du PX, Yao DL, Li SL, Yang YJ, Jin L, Li H. Phylogeny of Y-chromosome haplogroup C3b-F1756, an important paternal lineage in Altaic-speaking populations., 2017, 62(10): 915–918.
[75] Zhabagin M, Sabitov Z, Tazhigulova I, Alborova I, Agdzhoyan A, Wei LH, Urasin V, Koshel S, Mustafin K, Akilzhanova A, Li H, Balanovsky O, Balanovska E. Medieval super-grandfather founder of Western Kazakh Clans from Haplogroup C2a1a2-M48., 2021, 66(7): 707–716.
[76] Dulik MC, Osipova LP, Schurr TG. Y-chromosome variation in Altaian Kazakhs reveals a common paternal gene pool for Kazakhs and the influence of Mongolian expansions., 2011, 6(3): e17548.
[77] Chiaroni J, Underhill PA, Cavalli-Sforza LL. Y chromosome diversity, human expansion, drift, and cultural evolution., 2009, 106(48): 20174–20179.
[78] Qian EF, Deng P, Huang MS, Ma Q, Zhao H, Li CX, Huang J, Jiang L. Genetic polymorphism of Y chromosome haplogroup D-M174 in East Asian populations., 2019, 35(3): 308–313.钱恩芳, 邓盼, 黄美莎, 马泉, 赵慧, 李彩霞, 黄江, 江丽. 东亚族群中Y染色体D-M174单倍群的遗传多态性. 法医学杂志, 2019, 35(3): 308–313.
[79] Shi H, Zhong H, Peng Y, Dong YL, Qi XB, Zhang F, Liu LF, Tan SJ, Ma RZ, Xiao CJ, Wells RS, Jin L, Su B. Y chromosome evidence of earliest modern human settlement in East Asia and multiple origins of Tibetan and Japanese populations. BMC biology, 2008, 6: 45.
[80] Balaresque P, Bowden GR, Parkin EJ, Omran GA, Heyer E, Quintana-Murci L, Roewer L, Stoneking M, Nasidze I, Carvalho-Silva DR, Tyler-Smith C, de Knijff P, Jobling MA. Dynamic nature of the proximal AZFc region of the human Y chromosome: multiple independent deletion and duplication events revealed by microsatellite analysis., 2008, 29(10): 1171–1180.
[81] Malyarchuk B, Derenko M, Denisova G, Wozniak M, Grzybowski T, Dambueva I, Zakharov I. Phylogeography of the Y-chromosome haplogroup C in northern Eurasia., 2010, 74(6): 539–546.
[82] Tofanelli S, Ferri G, Bulayeva K, Caciagli L, Onofri V, Taglioli L, Bulayev O, Boschi I, Alù M, Berti A, Rapone C, Beduschi G, Luiselli D, Cadenas AM, Awadelkarim KD, Mariani-Costantini R, Elwali NE, Verginelli F, Pilli E, Herrera RJ, Gusmão L, Paoli G, Capelli C. J1-M267 Y lineage marks climate-driven pre-historical human displacements., 2009, 17(11): 1520– 1524.
[83] Myres NM, Ekins JE, Lin AA, Cavalli-Sforza LL, Woodward SR, Underhill PA. Y-chromosome short tandem repeat DYS458.2 non-consensus alleles occur independently in both binary haplogroups J1-M267 and R1b3-M405., 2007, 48(4): 450–459.
[84] Wang CC, Lu Y, Kang LL, Ding HQ, Yan S, Guo JX, Zhang Q, Wen SQ, Wang LX, Zhang MF, Tong XZ, Huang XF, Nie SJ, Deng QY, Zhu BF, Jin L, Li H. The massive assimilation of indigenous East Asian populations in the origin of Muslim Hui people inferred from paternal Y chromosome., 2019, 169(2): 341– 347.
[85] Wang QY, Zhao J, Ren Z, Sun J, He GL, Guo JX, Zhang HL, Ji JY, Liu YB, Yang MQ, Yang XM, Chen JW, Zhu KY, Wang R, Li YX, Chen G, Huang J, Wang CC. Male-dominated migration and massive mssimilation of indigenous East Asians in the formation of muslim Hui People in Southwest China., 2020, 11: 618614.
[86] Zhong H, Shi H, Qi XB, Xiao CJ, Jin L, Ma RZ, Su B. Global distribution of Y-chromosome haplogroup C reveals the prehistoric migration routes of African exodus and early settlement in East Asia., 2010, 55(7): 428–435.
Paternal genetic structure analysis of the modern Han populations based on Y-SNP and Y-STR
Xin Zhu1,2,Xin Jin3,Jun Liu2,4, Lan Yang2,4, Lixin Zou2,5,Caixia Li1,2, Jiang Huang1, Li Jiang2
The Han populations represent the largest ethnic group in China. Previous studies have primarily focused on investigating their genetic origins, migration and integration, as well as paternal genetic relationships within specific regional Han populations. However, a comprehensive analysis of the global paternal genetic structure of Han populations is lacking. In this study, we performed Y-chromosome sequencing on 362 unrelated male samples from Chinese Han individuals collected from Qinghai, Sichuan and Liaoning provinces. We then integrated relevant data from reported studies. Our final dataset comprised 1830 samples from 16 Han populations across 15 provinces in China, encompassing information on 89 Y-SNPs and 16 Y-STRs. Statistical analyses were conducted to assess Y-STR haplotype diversity (HD) and Y-SNP haplogroup frequencies. Additionally, we employed principal component analysis (PCA), phylogenetic tree and haplotype network to explore genetic differentiation within Han populations and the genetic relationships between Han populations and ethnic minorities surrounding them. Our results demonstrated that the O-M175 haplogroup represents the predominant paternal lineage in Han populations, with frequencies ranging from 60.53% (Qinghai Han) to 92.7% (Guangdong Han). Moreover, the subclades downstream of O-M175 showed distinct regional variations in their distribution patterns. The O2-M122 haplogroup was prevalent in all Han populations and demonstrated a gradual decline in frequency from north to south. Conversely, the distribution frequency of the O1b-M268 haplogroup decreased from south to north, particularly showed significant presence among Han populations in the Lingnan region. Haplogroup O1a-M119 distributed more frequently in the central Han populations. Our findings revealed that Chinese Han populations can be categorized into three subgroups: northern, central, and southern. Notably, there were significant differences among Han in Qinghai and other regions. Regarding the genetic relationships between Han populations and surrounding ethnic minorities, we observed a closer genetic affinity between different Han populations, but northern Han demonstrated a stronger relationship with the Hui ethnic group, while southern Han exhibited a closer connection with the Gelao and Li ethnic groups. In summary, this study presented a systematic analysis of haplogroup distribution, genetic substructure of Han populations and genetic relationships between Han populations and surrounding ethnic minorities based on 89 Y-SNPs and 16 Y-STRs systematically. Our research supplemented valuable insights into population genetics and forensic genetics, and provided data support for the forensic application of Y chromosome. The integration of Y-SNP haplogroups with Y-STR haplotypes offers enhanced understanding of the genetic substructure within Han populations, which holds significance for both population genetics research and forensic science applications.
population genetics; forensic genetics; Y-SNP; Y-STR
2023-12-11;
2024-01-24;
2024-01-26
国家重点研发计划项目(编号:2022YFC3341004),国家自然科学基金项目(编号:82171870),公安部鉴定中心基本科研业务费专项资金项目(编号:2022JB020)和现场物证溯源技术国家工程实验室开放课题(编号:2021FGKFKT01)资助 [Supported by the National Key R&D Program of China (No. 2022YFC3341004), the National Natural Science Foundation of China (No. 82171870), the Fundamental Research Funds for Institute of Forensic Science (No. 2022JB020), and the Open Project of the National Engineering Laboratory for Forensic Science (No. 2021FGKFKT01)
朱信,硕士研究生,专业方向:法医物证学。E-mail: 1987538437@qq.com
黄江,博士,教授,研究方向:法医物证学。E-mail: mmm_hj@gmc.edu.cn
江丽,博士,副主任法医师,研究方向:法医遗传学。E-mail: jl@mail.bnu.edu.cn
10.16288/j.yczz.23-260
(责任编委: 朱波峰)