辣椒遗传转化研究进展
2024-02-14张宏冠许晴赵红杰胡博文

摘要:辣椒(Capsicum annuum L.)作为一种重要的香料和蔬菜作物,具有巨大的经济价值。随着辣椒的需求量逐渐增大,育种目标也逐渐丰富,单纯依靠传统育种已无法满足人们的需求。分子育种是辣椒高效育种的有效途径,遗传转化技术是分子育种的核心,更是补充传统育种和加快辣椒改良的有力工具。由于辣椒具有较高的基因型依赖性和遗传转化顽拗性,只采用经典遗传转化策略获取阳性植株的概率渺茫。因此,基于经典遗传转化策略的拓展或是新策略的开发,对于推进辣椒遗传转化研究十分必要。本文重点围绕近5年来遗传转化策略的拓展开发,从辣椒功能解析的常用手段出发,就辣椒遗传转化的现有情况、可供参考的高效转化策略进行综述,并且针对辣椒遗传转化的难点总结相应的策略,包括植物发育调节因子策略、病毒载体递送策略、原生质体转化策略及其他物种中可供参考的转化策略。基于已有的研究,展望了辣椒遗传转化策略的进一步开发,为辣椒功能基因研究和分子育种提供理论支撑。
关键词:辣椒;植物再生;遗传转化;转化策略;功能验证
中图分类号:S641.303" 文献标志码:A
文章编号:1002-1302(2024)24-0017-06
收稿日期:2023-11-10
基金项目:湖南重点研发计划(编号:2023NK2006);湖南农业大学研究生科研创新项目(编号:2023CX102);国家自然科学基金(编号:32002040)。
作者简介:张宏冠(1998—),男,山东济宁人,硕士研究生,研究方向为辣椒分子设计育种。E-mail:871271896@qq.com。
通信作者:胡博文,博士,副教授,主要从事辣椒分子设计育种研究。E-mail:hubowen.cap@aliyun.com。
辣椒(Capsicum annuum L.)作为我国的重要蔬菜,年总产量可达6 400多万t,农业产值达2 500亿元,是我国经济价值最高的蔬菜,辣椒产业更是助农增收、乡村振兴的支柱产业[1]。种业是农业产业的 “芯片”,更是作物高产、高质的重要保障[2]。随着环境的变化,生物与非生物胁迫严重限制了辣椒的产量与质量,因此加强辣椒优良种质研究与创新已迫在眉睫[3-4]。传统的辣椒育种提高了辣椒产量与质量,但选育周期长、杂交不稔、遗传性状不稳定等因素使得该技术存在较大的局限性,阻碍了辣椒育种的进展,难以满足人民的需求[5]。分子育种为辣椒育种开辟了一条新的道路,通过植物遗传转化和以此为基础的基因编辑技术提高育种效率、创制传统育种方法难以实现的种质,可为优良品种的选育提供保障[6-7]。
遗传转化技术和以此为基础的基因编辑技术是分子育种的关键,通过该项技术可以实现目标基因的增加或改变,从而达到育种目的[8]。现如今,许多具有已知重要功能的辣椒基因,如果色相关基因、风味相关基因、对生物胁迫与非生物胁迫的抗性基因,被分离和表征,但因遗传转化体系限制,包括高度再生顽固、转化效率低等,辣椒分子育种进程缓慢[9-13]。遗传转化技术或是以此为基础的基因编辑技术,对已定位或是克隆到的基因进行鉴定和功能解析具有重要意义,因此高效遗传转化体系的建立对于功能基因研究和分子育种更是至关重要。本文针对辣椒遗传转化的难点如再生困难、转化效率低等问题与相应的解决策略进行了综述,并综述了可供辣椒遗传转化参考的高效转化策略,以期为辣椒高效遗传转化体系建立、分子育种效率提高提供理论支撑。
1 辣椒功能基因解析现状
辣椒基因组的公布与泛基因组的建立为优良基因的筛选提供了便利,随着测序技术的进步与成本的降低,辣椒基因被不断预测,如抗性基因、育性基因与辣椒素合成相关基因等。然而,辣椒基因功能验证的方法匮乏,主要原因在于高效遗传转化体系在辣椒中尚未建立,虽然有成功将外源基因导入的报道,但是转化效率低、重复性差等问题限制了其广泛的应用[14]。
目前,辣椒基因功能验证的方法主要集中在病毒诱导的基因沉默(virus induced gene silencing,VIGS)和在其他有成熟转化体系的物种中进行异源超表达(表1)。这些方法有自身的局限性,如烟草脆裂病毒(tobacco rattle virus,TRV)在辣椒中已被用作VIGS载体,可在辣椒中诱导系统性坏死,进而使得与防御和凋亡相关的应答基因难以被表征,相应的表型也难以分辨,同时,基因的异源超表达受到可转化物种的限制,导致表型与预期有偏差。辣椒的遗传转化壁垒是研究其基因组学的严重限制,使其基因组学研究落后于其他茄科植物[9,29] 。
2 辣椒遗传转化现状
2.1 植物发育调节因子促进辣椒再生
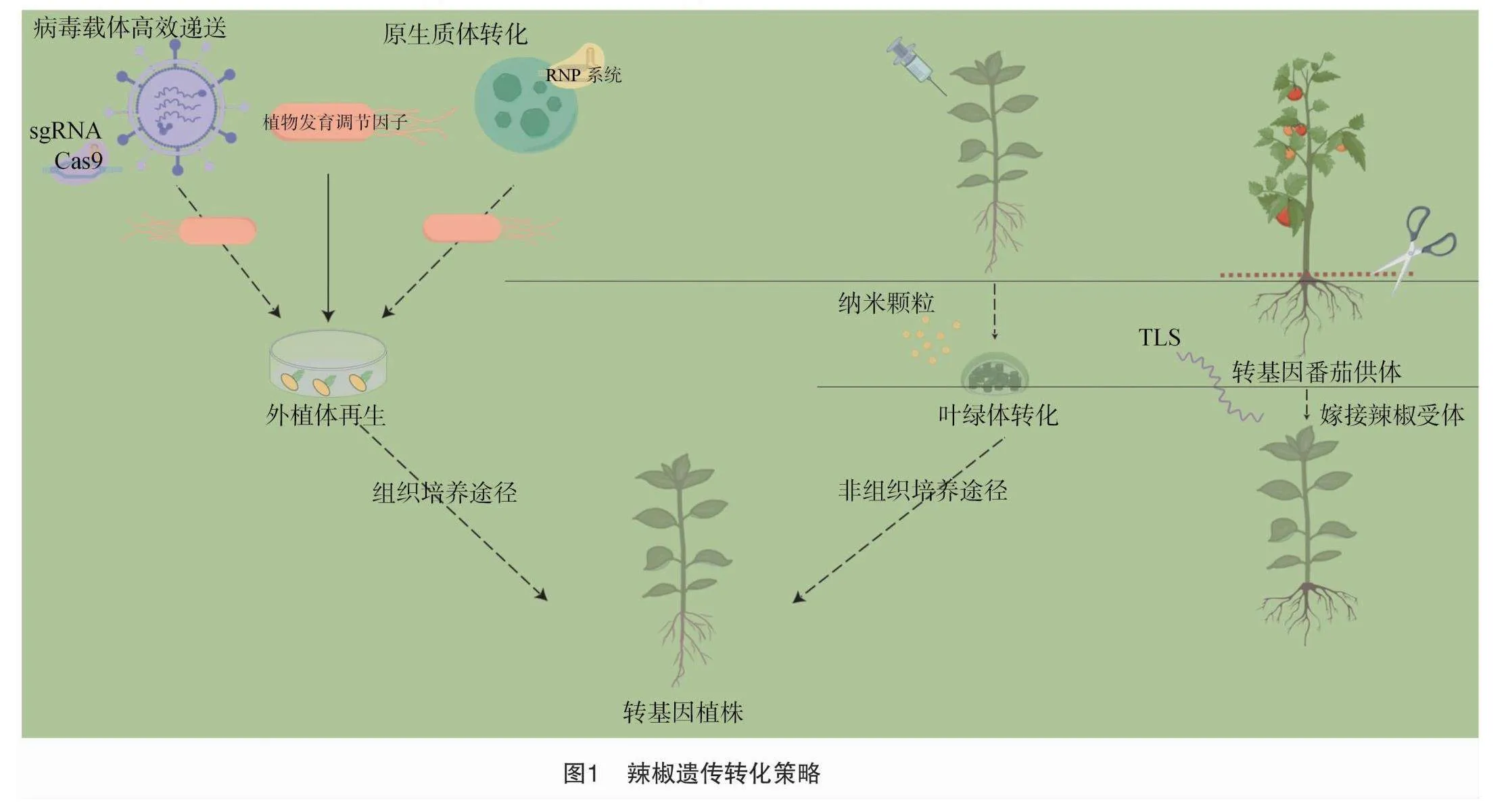
植物的离体再生能力制约了高效遗传转化体系的建立,常规的植物转化需要优化外部因素,如选择特定的植物基因型、调整植物生长调节剂比例 (主要是调整合适的生长素和细胞分裂素比例,用于改变植物生长)。植物发育调节因子,如Baby boom(BBM)、Wuschel(WUS)、PLETHORA(PLT)、GROWTH-REGULATING FACTOR4-GRF INTERACTING FACTOR1(GRF4-GIF1)、Wuschel-related homeobox(WOX),参与了生长素和细胞分裂素的合成途径,调控植物的再生过程,现已被证明能促进植物高效遗传转化并扩大可转化物种和基因型的范围[30-33]。2016年玉米BBM与WUS2的异位共表达促进了玉米、高粱、水稻、甘蔗体细胞胚的发生,并提高了农杆菌介导的遗传转化效率[34];2017年BBM与WUS2的异位共表达解决了顽拗性玉米B73的遗传转化问题[35];2020年GRF4-GIF1融合蛋白的表达扩大了小麦可转化基因型的范围,并提高了双子叶植物柑橘的再生效率[36];2021年GRF4-GIF1融合蛋白通过突变mi396位点,在西瓜中实现了高效遗传转化[37];2022年TaWOX5的过表达克服了小麦遗传转化的基因型依赖,并实现了高效遗传转化[38]。
辣椒具有基因型依赖和再生顽拗性,只采用经典遗传转化策略获得阳性植株的概率渺茫,植物发育调节因子的使用是促进辣椒高效遗传转化的有效途径。2011年,Heidmann等开发了一种经典遗传转化策略结合植物发育调节因子的转化再生系统,使用带有BBM基因的根癌农杆菌侵染甜椒子叶外植体,被转化甜椒在含有10 μmol/L 地塞米松(DEX)、1 mg/L 噻苯隆(TDZ)或10 μmol/L DEX+1 mg/L TDZ的MS培养基上能诱导出丰富的体细胞胚,最终生根并获得完整植株[39]。2022年,Lian等使用同样的策略,利用PLT5基因的表达提高了辣椒遗传转化效率,从0提升至3.8%[40]。
2.2 病毒递送系统提高递送效率
经典遗传转化在大多数植物中的转化效率低,其主要原因在于普通质粒载体递送效率低,因此如何将外源DNA高效递送至植物体内成为新的难题[41]。病毒载体介导的递送系统是一条有效途径,病毒载体的递送效率高,且具有靶向性和广适性,是基于各种宿主植物研究基因功能的有利工具。病毒诱导的基因编辑(virus induced genome editing,VIGE)和病毒诱导的基因沉默(virus induced gene silencing,VIGS)已经发展成为许多转化困难物种中基因功能验证的重要方法,其中最值得关注的就是VIGE[42-43]。2015年,甘蓝曲叶病毒(cabbage leaf curl virus,CaLCuV)通过VIGE技术系统感染了烟草,并编辑掉了八氢番茄红素脱氢酶(phytoene desaturase,PDS),新生叶产生了漂白表型[44]。2017年,小麦矮病毒(wheat dwarf virus,WDV)VIGE递送系统被开发,对水稻进行了高效基因编辑[45] 。值得注意的是,一些阳性RNA病毒,如烟草脆裂病毒(tobacco rattle virus,TRV)可以将sgRNA传递到生殖细胞或是分生组织中[46]。2020年,TRV-VIGE递送系统被开发,在烟草中实现了高效基因编辑和稳定遗传,在被病毒感染的植物后代中有30%在三靶中检测到突变[47]。同年,苦苣菜黄网病毒(sonchus yellow net virus,SYNV)VIGE递送系统被开发,在烟草中实现高效基因编辑并且实现了稳定遗传,在被感染病毒的植物后代中有57%存在突变,并且可绕过组织培养,进行稳定的机械传播[48]。
辣椒尚未建立高效遗传转化体系,导入或编辑基因的手段仍然局限于经典遗传转化,高效转化对于研究辣椒功能基因意义重大,基于病毒高效递送系统,绕过经典遗传转化的复杂操作从而提升转化效率,对加速辣椒分子设计育种及创制优良品种至关重要。2023年,番茄斑点枯萎病毒(tomato spotted wilt virus,TSWV)VIGE高效递送系统被开发,该系统具有宿主范围广、携带容量大且能够进行系统性感染等特点,对于一些难以转化的植物来说,TSWV克服了基因递送效率与基因型依赖的瓶颈,TSWV系统性地感染了辣椒的多种基因型,并且实现了40%的编辑效率[49]。TSWV在辣椒中克服了基因递送瓶颈问题,但仍然存在一定的局限性,如无法感染到生殖细胞、难以绕过离体再生,目前,VIGS仍是辣椒研究功能基因组学的常用技术。TRV介导的VIGS技术常用于辣椒中,但是沉默效率不稳定,仍需要被进一步优化。2021年,带有C2b基因的TRV载体被开发,TRV感染后 5个辣椒品种果实表型明显,随后的定量数据证明了基因沉默的高效性,TRV-C2b为VIGS技术提供了高效的载体,并有助于辣椒整个生命周期的基因功能研究[50]。接种TRV病毒后表现出的症状,如坏死、变黄、发育迟缓,可能会影响辣椒基因功能的系统分析[51],因此,蚕豆萎蔫病毒2号(broad bean wilt virus 2,BBWV2)被开发,BBWV2病毒载体介导的VIGS不会引起辣椒明显的病毒症状,且BBWV2与异源病毒抑制子共表达,增强了重组蛋白表达的稳定性[15]。
2.3 辣椒原生质体递送系统被开发
许多植物因遗传转化技术壁垒阻碍了其分子机理的研究,因此,一种分析分子过程的材料需要被开发。使用原生质体被认为是一种简便有效的办法,因其没有细胞壁且易吸收外源物质被广泛用于遗传转化和以此为基础的基因编辑[52-53],同时具有试验周期短、转化效率高、脱靶率低的优势[54-55]。在植物进行基因编辑之前,使用原生质体通过核糖核蛋白复合物(RNP)递送系统来确定目标基因的编辑效率,对于建立了再生系统的植物可以进一步产生基因编辑植物。2020年,使用RNP递送系统对橡胶树原生质体完成了基因编辑,编辑效率为70%[56]。2021年,使用RNP递送系统对辐射松原生质体完成了基因编辑,随后使用被编辑后的原生质体产生了再生苗[57]。2023年,使用RNP递送系统对葡萄原生质体进行了基因编辑,随后通过体细胞胚的发生途径获得了被编辑后的完整植株[58]。
辣椒遗传转化困难,以此为基础的基因编辑技术在之前未曾被报道,近几年有了突破。2020年,使用RNP递送系统对辣椒原生质体进行了基因编辑,并获得了19.3%的编辑效率,为辣椒创制了首个基因编辑系统[59]。然而,辣椒原生质体的再生系统尚未被报道,如何获得再生植株仍需进一步的探索。
3 高效转化策略为提升辣椒遗传转化效率提供参考
3.1 可移动RNA的应用
载体递送效率低与转化后再生困难是辣椒遗传转化的重要限制因素[9]。病毒递送系统的使用、植物发育调节因子共表达有效地解决了这些困难,但是这些方法仍不够完善,如病毒递送系统无法侵染到生殖器官,阳性苗的获得绕不开组织培养;植物发育调节因子WUS、BBM过表达产生的转基因植物表型出了异常的表型,如根尖卷曲、叶片扭曲或是下胚轴膨胀等[60]。因此,辣椒遗传转化策略开发工作依旧充满挑战。
尽管有新策略被开发,辣椒遗传转化仍然受限于组织培养,近几年其他物种的转化策略为辣椒遗传转化提供了参考(图1)。2023年,一种基于tRNA-like sequence(TLS)的基因编辑传递方法被开发,该方法使用砧木嫁接法将可移动的TLS从转基因供体转移到兼容的野生型受体植物中,从而在当代获得纯合编辑的植株[61]。这种绕过组织培养、省略杂交和自交步骤获得纯合编辑植株的方案,对加速优良品种培育意义重大。2022年,番茄与辣椒嫁接关键调控因子WOX4被鉴定且番茄有稳定的转化体系,该研究为辣椒嫁接携带TLS基因编辑的番茄供体提供了可能[62]。
3.2 叶绿体转化体系的应用
叶绿体转化与主流的核转化相比具有外源基因表达量高、无位置效应、无花粉漂移、有多基因共表达元件等优势[63]。2019年,一种具有广适性的叶绿体转化方法被开发,该方法使用纳米颗粒系统转化植物,研究人员在叶片表面下方使用注射法将颗粒通过气孔注入叶片,进入叶片内部后,纳米颗粒会穿过细胞壁、细胞膜,最终穿过叶绿体的双层膜,进入叶绿体后,叶绿体的弱酸性环境会促使 DNA从纳米颗粒中释放出来。随后,研究人员在菠菜、烟草、芝麻和拟南芥中进行了测试,利用黄色荧光蛋白进行验证,结果表明,47%的植物细胞可表达黄色荧光蛋白[64]。在高等植物中,茄科植物是进行叶绿体转化成功最多的,且该方案不需要组织培养,该研究为辣椒遗传转化新策略开发提供了参考[65]。
4 展望
目前辣椒基因功能验证的手段多为VIGS或是异源超表达,通过遗传转化手段的报道较少,近几年新开发的转化策略对辣椒基因组学研究、优良品种培育有巨大的价值,综合已有的研究,未来的研究可以在以下4个方面进一步延伸:(1)植物发育调节因子组合使用可能会有累加的作用进而促进外植体再生,如GRF-GIF促进单子叶与双子叶植物的再生、BBM-WUS促进单子叶植物体细胞胚发生[36,40] 。因此,可以利用多组学联合分析技术进一步挖掘与植物再生相关的基因,并单独或组合测试其在辣椒中的转化效率。(2)病毒递送系统提高了辣椒的转化,然而大多数病毒无法感染到生殖细胞或是分生组织无法稳定遗传至下一代,因此无法绕过组织培养,离体再生这一瓶颈依然存在[49]。植物发育调节因子共接种增加了被编辑外植体再生的可能,而使用病毒递送系统结合植物发育调节因子共接种,在提升递送效率的同时增加了再生的可能性[66]。(3)Flowering Locus T(FT) 融合sgRNA在TRV载体中被证明可移动到顶端分生组织和花组织[67]。这意味着通过病毒递送系统,可以获得无需组织培养的转基因苗。然而病毒的容载量(通常小于1 kb)限制了基因编辑组件的递送,如Cas9蛋白(大小为4 kb),TSWV的大容载量解决了这一问题,因此TSWV融合可移动RNA元件如FT,可能有助于基因编辑植物的获得[68]。(4)参考绕过组织培养的遗传转化策略,开发出新的方案如TLS、叶绿体转化等。
参考文献:
[1]吴永红,周书栋,李雪峰,等. 2019年辣椒科学研究进展[J]. 辣椒杂志,2020,18(2):1-7,13.
[2]林 巧,辛竹琳,孔令博,等. 我国辣椒产业发展现状及育种应对措施[J]. 中国农业大学学报,2023,28(5):82-95.
[3]Meng Y C,Zhang H F,Pan X X,et al. CaDHN3,a pepper (Capsicum annuum L.) dehydrin gene enhances the tolerance against salt and drought stresses by reducing ROS accumulation[J]. International Journal of Molecular Sciences,2021,22(6):3205.
[4]邹学校,马艳青,戴雄泽,等. 辣椒在中国的传播与产业发展[J]. 园艺学报,2020,47(9):1715-1726.
[5]Martins K C,Pereira T N S,Souza S A M,et al. Crossability and evaluation of incompatibility barriers in crosses between Capsicum species[J]. Crop Breeding and Applied Biotechnology,2015,15(3):139-145.
[6]Krishna H,Alizadeh M,Singh D,et al. Somaclonal variations and their applications in horticultural crops improvement[J]. 3 Biotech,2016,6(1):54.
[7]Karthik S,Pavan G,Prasanth A,et al. Improved in planta genetic transformation efficiency in bitter gourd (Momordica charantia L.)[J]. In Vitro Cellular amp; Developmental Biology-Plant,2021,57(2):190-201.
[8]张文斗,王 燕,徐 曼,等. 观赏桃分子育种研究进展与展望[J]. 农业与技术,2023,43(12):56-59.
[9]Kothari S L,Joshi A,Kachhwaha S,et al. Chilli peppers:a review on tissue culture and transgenesis[J]. Biotechnology Advances,2010,28(1):35-48.
[10]Lu B Y,Cheng G X,Zhang Z,et al. CaMYC,a novel transcription factor,regulates anthocyanin biosynthesis in color-leaved pepper (Capsicum annuum L.)[J]. Journal of Plant Growth Regulation,2019,38(2):574-585.
[11]Taller J. Gene functioning in pepper[J]. Acta Agronomica Hungarica,2006,54(2):233-269.
[12]Liu C Y,Peang H R,Li X Y,et al. Genome-wide analysis of NDR1/HIN1-like genes in pepper (Capsicum annuum L.) and functional characterization of CaNHL4 under biotic and abiotic stresses[J]. Horticulture Research,2020,7(1):93.
[13]Wu D,Ni M,Lei X,et al. Analyses of pepper cinnamoyl-CoA reductase gene family and cloning of CcCCR1/2 and their function identification in the formation of pungency[J]. Horticulturae,2022,8(6):537.
[14]Zhu Z S,Sun B M,Cai W,et al. Natural variations in the MYB transcription factor MYB31 determine the evolution of extremely pungent peppers[J]. New Phytologist,2019,223(2):922-938.
[15]Wang Y X,Wang Z,Geng S S,et al. Identification of the GDP-L-galactose phosphorylase gene as a candidate for the regulation of ascorbic acid content in fruits of Capsicum annuum L[J]. International Journal of Molecular Sciences,2023,24(8):7529.
[16]Zhang H F,Pei Y P,He Q,et al. Salicylic acid-related ribosomal protein CaSLP improves drought and Pst.DC3000 tolerance in pepper[J]. Molecular Horticulture,2023,3(1):6.
[17]Zhou Y,Li Y,Yu T,et al. Characterization of the B-BOX gene family in pepper and the role of CaBBX14 in defense response against Phytophthora capsici infection[J]. International Journal of Biological Macromolecules,2023,237:124071.
[18]Li Y,Ma X,Xiao L D,et al. CaWRKY50 acts as a negative regulator in response to Colletotrichum scovillei infection in pepper[J]. Plants,2023,12(10):1962.
[19]Mao L Z,Tian W F,Shen Y Y,et al. Auxin-related MYB (CaSRM1) is involved in leaf shape development and reproductive growth in pepper (Capsicum annuum L.)[J]. Scientia Horticulturae,2023,322:112383.
[20]Zhang L P,Wu D,Zhang W,et al. Genome-wide identification of WRKY gene family and functional characterization of CcWRKY25 in Capsicum chinense[J]. International Journal of Molecular Sciences,2023,24(14):11389.
[21]Baek W,Bae Y,Lim C W,et al. Pepper homeobox abscisic acid signalling-related transcription factor 1,CaHAT1,plays a positive role in drought response[J]. Plant,Cell amp; Environment,2023,46(7):2061-2077.
[22]Zhang J W,Xie M H,Yu G F,et al. CaSPDS,a spermidine synthase gene from pepper (Capsicum annuum L.),plays an important role in response to cold stress[J]. International Journal of Molecular Sciences,2023,24(5):5013.
[23]Zhang H F,Guo J B,Chen X Q,et al. Transcription factor CabHLH035 promotes cold resistance and homeostasis of reactive oxygen species in pepper[J/OL]. Horticultural Plant Journal,2023(2023-03-16)[2023-11-10]. https://www.sciencedirect.com/science/article/pii/S2468014123000468.
[24]Liu Y,Cao H Y,Ling J,et al. Molecular cloning and functional analysis of the pepper resistance gene Me3 to root-knot nematode[J]. Horticultural Plant Journal,2023,9(1):133-144.
[25]Dong J C,Hu F,Guan W D,et al. A 163-bp insertion in the Capana10g000198 encoding a MYB transcription factor causes male sterility in pepper (Capsicum annuum L.)[J]. The Plant Journal,2023,113(3):521-535.
[26]Ma X,Yu Y N,Li Y,et al. The CBL-interacting protein kinase CaCIPK7 enhances drought resistance in pepper[J]. Scientia Horticulturae,2023,310:111726.
[27]Hussain A,Khan M I,Albaqami M,et al. CaWRKY30 positively regulates pepper immunity by targeting CaWRKY40 against Ralstonia solanacearum inoculation through modulating defense-related genes[J]. International Journal of Molecular Sciences,2021,22(21):12091.
[28]Liang M M,Li H Y,Yong K,et al. Pepper autophagy related gene CaATG8e differentially regulates plant tolerance to heat and salt stress[J]. Scientia Horticulturae,2023,308(3):111559.
[29]Choi B,Kwon S J,Kim M H,et al. A plant virus-based vector system for gene function studies in pepper[J]. Plant Physiology,2019,181(3):867-880.
[30]Gordon-Kamm B,Sardesai N,Arling M,et al. Using morphogenic genes to improve recovery and regeneration of transgenic plants[J]. Plants,2019,8(2):38.
[31]Lee K,Wang K.Strategies for genotype-flexible plant transformation[J]. Current Opinion in Biotechnology,2023,79:102848.
[32]Nagle M,Déjardin A,Pilate G,et al. Opportunities for innovation in genetic transformation of forest trees[J]. Frontiers in Plant Science,2018,9:1443.
[33]Nalapalli S,Tunc-Ozdemir M,Sun Y J,et al. Morphogenic regulators and their application in improving plant transformation[J]. Methods in Molecular Biology,2021,2238:37-61.
[34]Lowe K,Wu E,Wang N,et al. Morphogenic regulators Baby boom and Wuschel improve monocot transformation[J]. Plant Cell,2016,28(9):1998-2015.
[35]Mookkan M,Nelson-Vasilchik K,Hague J,et al. Selectable marker independent transformation of recalcitrant maize inbred B73 and sorghum P898012 mediated by morphogenic regulators BABY BOOM and WUSCHEL2[J]. Plant Cell Reports,2017,36(9):1477-1491.
[36]Debernardi J M,Tricoli D M,Ercoli M F,et al. A GRF-GIF chimeric protein improves the regeneration efficiency of transgenic plants[J]. Nature Biotechnology,2020,38(11):1274-1279.
[37]Feng Q,Xiao L,He Y Z,et al. Highly efficient,genotype-independent transformation and gene editing in watermelon (Citrullus lanatus) using a chimeric ClGRF4-GIF1 gene[J]. Journal of Integrative Plant Biology,2021,63(12):2038-2042.
[38]Wang K,Shi L,Liang X N,et al. The gene TaWOX5 overcomes genotype dependency in wheat genetic transformation[J]. Nature Plants,2022,8(2):110-117.
[39]Heidmann I,de Lange B,Lambalk J,et al. Efficient sweet pepper transformation mediated by the BABY BOOM transcription factor[J]. Plant Cell Reports,2011,30(6):1107-1115.
[40]Lian Z Y,Nguyen C D,Liu L,et al. Application of developmental regulators to improve in planta or in vitro transformation in plants[J]. Plant Biotechnology Journal,2022,20(8):1622-1635.
[41]Kausch A P,Nelson-Vasilchik K,Hague J,et al. Edit at will:genotype independent plant transformation in the era of advanced genomics and genome editing[J]. Plant Science,2019,281:186-205.
[42]Oh Y,Kim H,Kim S G.Virus-induced plant genome editing[J]. Current Opinion in Plant Biology,2021,60:101992.
[43]Rssner C,Lotz D,Becker A. VIGS goes viral:how VIGS transforms our understanding of plant science[J]. Annual Review of Plant Biology,2022,73:703-728.
[44]Yin K Q,Han T,Liu G,et al. A geminivirus-based guide RNA delivery system for CRISPR/Cas9 mediated plant genome editing[J]. Scientific Reports,2015,5:14926.
[45]Wang M G,Lu Y M,Botella J R,et al. Gene targeting by homology-directed repair in rice using a geminivirus-based CRISPR/Cas9 system[J]. Molecular Plant,2017,10(7):1007-1010.
[46]Ali Z,Abul-faraj A,Li L X,et al. Efficient virus-mediated genome editing in plants using the CRISPR/Cas9 system[J]. Molecular Plant,2015,8(8):1288-1291.
[47]Ellison E E,Nagalakshmi U,Gamo M E,et al. Multiplexed heritable gene editing using RNA viruses and mobile single guide RNAs[J]. Nature Plants,2020,6:620-624.
[48]Ma X N,Zhang X Y,Liu H M,et al. Highly efficient DNA-free plant genome editing using virally delivered CRISPR-Cas9[J]. Nature Plants,2020,6(7):773-779.
[49]Liu Q,Zhao C L,Sun K,et al. Engineered biocontainable RNA virus vectors for non-transgenic genome editing across crop species and genotypes[J]. Molecular Plant,2023,16(3):616-631.
[50]Zhou Y J,Deng Y T,Liu D,et al. Promoting virus-induced gene silencing of pepper genes by a heterologous viral silencing suppressor[J]. Plant Biotechnology Journal,2021,19(12):2398-2400.
[51]Gleba Y,Klimyuk V,Marillonnet S.Viral vectors for the expression of proteins in plants[J]. Current Opinion in Biotechnology,2007,18(2):134-141.
[52]Wang Y Y,Zhang Y A,Dong Y X,et al. A highly efficient mesophyll protoplast isolation and PEG-mediated transient expression system in eggplant[J]. Scientia Horticulturae,2022,304:111303.
[53]Xia K K,Zhang D W,Liu G Y,et al. Efficient multiplex genome editing tools identified by protoplast technology in Phalaenopsis[J]. Biorxiv,2020,322:111368.
[54]Naing A H,Adedeji O S,Kim C K.Protoplast technology in ornamental plants:current progress and potential applications on genetic improvement[J]. Scientia Horticulturae,2021,283:110043.
[55]Zhang Q,Xing H L,Wang Z P,et al. Potential high-frequency off-target mutagenesis induced by CRISPR/Cas9 in Arabidopsis and its prevention[J]. Plant Molecular Biology,2018,96(4):445-456.
[56]Fan Y T,Xin S C,Dai X M,et al. Efficient genome editing of rubber tree (Hevea brasiliensis) protoplasts using CRISPR/Cas9 ribonucleoproteins[J]. Industrial Crops and Products,2020,146:112146.
[57]Poovaiah C,Phillips L,Geddes B,et al. Genome editing with CRISPR/Cas9 in Pinus radiata (D.Don)[J]. BMC Plant Biology,2021,21(1):363.
[58]Najafi S,Bertini E,DIncà E,et al. DNA-free genome editing in grapevine using CRISPR/Cas9 ribonucleoprotein complexes followed by protoplast regeneration[J]. Horticulture Research,2023,10(1):uhac240.
[59]Kim H,Choi J,Won K H.A stable DNA-free screening system for CRISPR/RNPs-mediated gene editing in hot and sweet cultivars of
Capsicum annuum[J]. BMC Plant Biology,2020,20(1):449.
[60]Maren N A,Duan H,Da K D,et al. Genotype-independent plant transformation[J]. Horticulture Research,2022,9:uhac047.
[61]Yang L,Machin F,Wang S F,et al. Heritable transgene-free genome editing in plants by grafting of wild-type shoots to transgenic donor rootstocks[J]. Nature Biotechnology,2023,41:958-967.
[62]Thomas H,van den Broeck L,Spurney R,et al. Gene regulatory networks for compatible versus incompatible grafts identify a role for SlWOX4 during junction formation[J]. The Plant Cell,2022,34(1):535-556.
[63]Ruf S,Forner J,Hasse C,et al. High-efficiency generation of fertile transplastomic Arabidopsis plants[J]. Nature Plants,2019,5(3):282-289.
[64]Kwak S Y,Lew T T S,Sweeney C J,et al. Chloroplast-selective gene delivery and expression in planta using chitosan-complexed single-walled carbon nanotube carriers[J]. Nature Nanotechnology,2019,14(5):447-455.
[65]Liu Y X,Li F,Gao L,et al. Advancing approach and toolbox in optimization of chloroplast genetic transformation technology[J]. Journal of Integrative Agriculture,2023,22(7):1951-1966.
[66]Maher M F,Nasti R A,Vollbrecht M,et al. Plant gene editing through de novo induction of meristems[J]. Nature Biotechnology,2020,38:84-89.
[67]Ellison E E,Nagalakshmi U,Gamo M E,et al. Author correction:multiplexed heritable gene editing using RNA viruses and mobile single guide RNAs[J]. Nature Plants,2021,7:99.
[68]Gao Q,Xu W Y,Yan T,et al. Rescue of a plant cytorhabdovirus as versatile expression platforms for planthopper and cereal genomic studies[J]. New Phytologist,2019,223(4):2120-2133.
