小麦脱镁叶绿酸加氧酶基因家族鉴定及其响应衰老特征分析
2024-01-26闫荣岳郝辉芳晋秀娟郭峰许成杰汤小莎王曙光范华孙黛珍
闫荣岳 郝辉芳 晋秀娟 郭峰 许成杰 汤小莎 王曙光 范华 孙黛珍


摘 要:脫镁叶绿酸a加氧酶(PaO)基因编码叶绿素降解过程中的关键酶。为了解小麦(Triticum aestivum L.)TaPaOs基因家族特征及其在衰老过程中发挥的作用,本试验利用已发布的小麦及其他物种的全基因组信息对TaPaOs家族成员的基因特性进行分析,并利用实时荧光定量PCR(qRT-RCR)技术初步分析其功能。结果表明:在小麦中共发现18个TaPaOs家族成员,分布在15条染色体上,其编码蛋白均具有亲水性,且大多数为不稳定蛋白,亚细胞定位预测均位于叶绿体中;18个TaPaOs家族成员启动子区包含大量响应元件,其中光响应元件(G-Box)和ABA响应元件(ABRE)最多。分析TaPaOs在不同组织中的表达模式发现,其家族成员表达模式为叶>茎>根,大多数家族成员在ABA诱导衰老、黑暗诱导衰老以及自然衰老时上调表达,尤其是TaPaO2、TaPaO5及TaPaO6,可作为小麦衰老研究的重点关注基因。这些研究结果为进一步研究TaPaOs基因家族的功能奠定基础。
关键词:小麦;TaPaOs基因家族;叶绿素;表达模式分析;衰老
中图分类号:S512.1;Q78 文献标志码:A DOI:10.3969/j.issn.1007-7146.2023.01.007
Familial Identification of Wheat Pheophorbide a Oxygenase Gene and Its Characteristic Analysis in Response to Aging
YAN Rongyue, HAO Huifang, JIN Xiujuan, GUO Feng, XU Chengjie, TANG Xiaosha, WANG Shuguang, FAN Hua*, SUN Daizhen*
(College of Agriculture, Shanxi Agricultural University, Taigu 030801, China)
Abstract: Pheophorbide being a oxygenase gene (PaO) encoded a key enzyme in the process of chlorophyll degradation. In this study, in order to understand the characteristics of the TaPaOs gene family in wheat (Triticum aestivum L.) and the role it plays in the aging process, the published genome-wide information of wheat and other species was used to analyze the genetic features of the TaPaOs family, preliminarily identified their functions by real-time fluorescence quantitative (qRT-PCR) technology. The results showed that a total of 18 TaPaOs family members were found in wheat, distributed on 15 chromosomes. The encoded proteins were hydrophilic, most of which were unstable proteins, and their subcellular localization was predicted in chloroplasts. The promoter regions of the 18 family members contained a large number of response elements, among which the light response element (G-Box) and ABA response element (ABRE) were the most. The expression patterns of the TaPaOs gene family in different tissues were analyzed and found that leaves were the most, followed by stems and lowest roots. Most of the family members upregulated expression during ABA-induced aging, dark induced aging, and natural aging, especially TaPaO2, TaPaO5, and TaPaO6 would be used as the focus of wheat aging research, which laid the foundation for further study of the TaPaoOs gene family function in wheat.
Key words: wheat; TaPaOs gene family; chlorophyll; expression analysis; aging
(Acta Laser Biology Sinica, 2023, 32(1): 043-054)
小麦(Triticum aestivum L.)是当前我国人民的主要粮食作物之一,能够提供人体所需要的多种养分,小麦的生产发展与我国粮食安全和社会稳定有着直接关系[1]。衰老是在细胞、组织、器官或生物体水平上的一种年龄依赖性退化过程,伴随着光合产物从叶片和茎秆向种子的迁移和积累,因此,衰老延缓将有利于提高光合速率、延长光合作用的时间,增加种子中营养物质的积累,对提高作物产量具有重要作用[2-4]。
叶绿素(chlorophyll,Chl)是地球上最丰富的色素,是植物进行光合作用必需的物质基础[5]。叶绿素的降解与衰老息息相关,随着叶绿素降解的加快,植物衰老也加快,反之亦然。研究表明,高等植物的叶绿素降解有两条途径:一是叶绿素a(chlorophyll a,Chl a)在叶绿素酶(chlorophyllase,CLH)作用下脱植基形成脱植基叶绿素a(chlorophyllide a,Chlide a),然后脱镁形成脱镁叶绿酸a(pheophorbide,Pheide a)[6];二是Chl a在脱镁螯合酶作用下形成脱镁叶绿素a(pheophytin a,Phein a),再在脱镁叶绿素酶(pheophytinase,PPH)作用下经脱植基形成Pheide a。随后,脱镁叶绿酸a加氧酶(pheophorbide a oxygenase,PaO)特异性切割Pheide a的卟啉环,氧化产生红色叶绿素分解产物[7]。其中PaO是叶绿素降解途径中的关键酶,是一种非血红素铁型单加氧酶,具有一个保守的Rieske铁硫中心、单核铁结合位点以及PaO结构域(cd03480)[8-9]。1997年,PaO基因首先在玉米(Zea mays L.)中被鉴定为致死叶斑点(lethal leaf spot 1,Lls1)基因[10];在拟南芥(Arabidopsis thaliana)中PaO被称为加快细胞死亡(accelerated cell death 1,ACD1)基因[11]。前人研究表明,PaO的表达是由植物的自然衰老和环境胁迫诱导的:在西兰花(Brassica oleracea L.)采后衰老过程中发现BoPaO表达显著增加,并与叶绿素降解相关[12];在油菜(Brassica napus L.)衰老过程中也发现PaO蛋白活性降低,会延迟叶绿素的降解[13];外源喷施脱落酸(abscisic acid,ABA)诱导衰老可以增强水稻(Oryza sativa L.)叶片中PaO的表达[14];ABA处理可以使大麦(Hordeum vulgare L.)叶片中的叶绿素迅速丧失,PaO活性增加[15];在黑暗诱导衰老期间,水稻OsPaO沉默后,延迟叶绿素降解[16],而拟南芥中AtPaO过表达的叶片在黑暗处理4 d后降低了叶片中叶绿素的含量[17]。以上研究均表明,PaO相关基因可能通过叶绿素降解途径参与叶片衰老的过程。
目前,小麦TaPaO已经被克隆[2],但TaPaOs家族成员的基因特性分析及功能未见报道。鉴于此,本研究利用生物信息学的手段确定TaPaOs家族成员,并分析其染色体(chromosome,chr)位置、蛋白质理化性质、二级结构、亚细胞定位、基因结构、保守基序和顺式作用元件等,预测并验证其在不同组织中及不同衰老阶段的表达情况,为后续进一步研究TaPaOs家族成员的功能奠定基础。
1 材料与方法
1.1 试验材料及处理
小麦品种Fielder作为供试材料,分别进行室内水培以及大田试验。
室内水培:挑选无病虫害且颗粒饱满的种子,用10%次氯酸钠消毒10 min,然后用蒸馏水冲洗5次后进行种子吸胀处理,放入人工气候培养箱(光照10 000 lx 14 h/黑暗0 lx 10 h,温度25℃/15℃,湿度60%)中过夜培养,之后挑选露白的种子放入育苗盘中培养,培养3 d后转入黑色水培盒中培养,待长至两叶一心期,取9株幼苗的根、茎、叶做TaPaOs家族成员在不同组织中的表达分析,剩余幼苗分两组进行不同处理,其中一组进行50 ?mol/L的ABA处理,并于处理后0、1、3、6、12、24、48、72 h取幼苗叶片;另一组进行黑暗处理,并于处理后0、0.5(12 h)、1、3、5、7 d取幼苗叶片。两个处理均设3个重复,每个重复各3株,储存于–80℃备用,用于ABA诱导衰老以及黑暗诱导衰老时TaPaOs家族成员的表达分析。
大田试验:于2022年3月11日,将小麦种植于山西农业大学农学院农作站小麦试验田(37.42°N,112.58°E)中,点播,行长2 m,行距10 cm,并于花后7、10、13、16、19、22、25、28 d每个时期均取3片主茎旗叶,用于自然衰老条件下TaPaOs家族成员的表达分析。
1.2 基因组序列的获取
从Ensembl Plants[18](http://plants.ensembl.org/info/data/ftp/index.html)数据库中获取中国春小麦、乌拉尔图小麦(Triticum urartu Thumanjan ex Gandilyan)、节节麦(Aegilops tauschii Coss.)、水稻、玉米及拟南芥的基因组数据、总蛋白序列、CDS序列以及GFF3文件。
1.3 TaPaOs基因家族成员鉴定
从Pfam[19](http://pfam-legacy.xfam.org/)数据库中下载PaO结构域(PF08417)和Rieske 2Fe-2S(PF00355)结构域的HMM模型,通过TBtools中的Simple HMM Search,筛选小麦总蛋白序列中含Rieske_RO_Alpha_PaO(cd03480)结构域的基因;从NCBI(https: //www.ncbi.nlm.nih.gov/)数据库中下载玉米和拟南芥PaO的蛋白序列,利用小麦多组学数据平台WheatOmics的Blastp[20](http://wheatomics.sdau.edu.cn/blast/blast.html)工具筛选TaPaOs家族的候选基因。然后,将上述两种方法获取的家族成员取交集,进而确定为TaPaOs家族成员,最后,利用NCBI中保守域数据库[21] (https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)檢查是否含有Rieske_RO_Alpha_PaO结构域,剔除冗余基因,最终获得小麦TaPaOs家族成员。使用TBtools[22]中的Fasta Extract(Recommended)提取TaPaOs家族成员氨基酸(amino acid,aa)序列,使用同样的方法筛选乌拉尔图小麦、节节麦、玉米、水稻和拟南芥的PaO家族成员。
1.4 染色体定位图绘制
通过小麦基因组GFF注释文件,获得TaPaOs家族基因的位置信息,并使用TBtools绘制TaPaOs家族成员的染色体定位图。
1.5 TaPaOs家族蛋白的理化性质、二级结构预测及亚细胞定位
通过Expasy[23](https://web.expasy.org/protparam/)在线软件获得TaPaOs蛋白的氨基酸数、相对分子质量、等电点、不稳定指数、脂肪指数、亲水性(grand average of hydropathicity,GRAVY);使用SOPMA(https://npsa-prabi.ibcp.fr/cgibin/npsa_automat.pl?page=npsa_sopma.html)在线网站预测TaPaOs蛋白的二级结构;通过PlantmPLoc[24] (http://www.csbio.sjtu.edu.cn/cgi-bin/PlantmPLoc.cgi)在线软件对TaPaOs蛋白序列进行亚细胞定位。
1.6 TaPaOs的系统进化分析
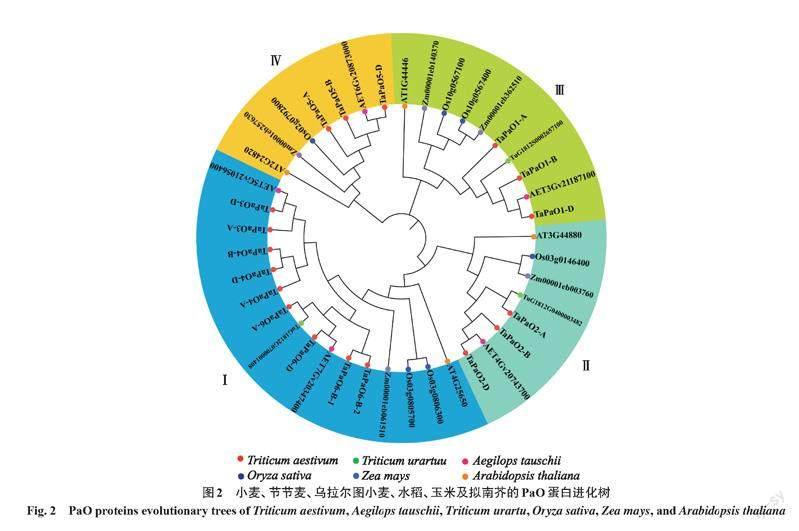
通过MEGA 7.0[25]将筛选到的TaPaOs蛋白序列与节节麦、乌拉尔图小麦、水稻、玉米及拟南芥的PaO家族蛋白序列进行多重序列比较,并采用邻接法(neighbor joining,NJ),设置Bootstrap重复数为1 000,构建进化树,分析小麦与麦类作物乌拉尔图小麦、节节麦;禾本科作物的玉米、水稻以及拟南芥之间的同源进化关系,使用iTOL[26](https://itol. embl. de/login. cgi)在线工具美化进化树。将进化树上位于小麦A、B、D亚基因组上并聚集在同一进化分支末端的基因定义为种内同源等位基因;节节麦、乌拉尔图小麦、玉米、水稻以及拟南芥的PaO基因与小麦基因在同一分支上聚合,定义为种间直系同源基因对[27]。
1.7 TaPaOs的基因结构和保守基序分析
使用TBtools分析TaPaOs的基因结构,通过MEME[28](https://meme-suite. org/)在线软件分析保守基序(motifs),并使用TBtools对结果可视化作图。
1.8 TaPaOs的顺式作用元件分析
使用TBtools软件从小麦基因组文件中提取TaPaOs起始密码子ATG上游2 000 bp序列,通过PlantCARE[29](https://bioinformatics.psb.ugent.be/webtool s/plantcare/html/)在线软件分析家族成员的顺式作用元件,并利用TBtools绘图。
1.9 TaPaOs的表达分析
在WheatOmics中下载并整理TaPaOs在不同组织中的转录组数据,包括根、茎、叶[30],以及在小麦抽穗期、花后0、15、25、30 d[31]的表达情况,使用TBtools软件绘制表达热图。
用Trizol法(Takara)提取不同处理下的小麦叶片RNA,并用反转录试剂盒(Takara)合成cDNA,使用WheatOmics的PrimerServer(http://wheatomics.sdau.edu.cn/PrimerServer/)在线软件设计小麦扩增引物(表1),采用qRT-PCR法分析其表达量。用2-ΔΔCt法计算各个基因在不同处理的不同时期下的相对表达量,以Actin为内参,设置3次生物学重复。
2 结果与分析
2.1 TaPaOs家族成员鉴定、蛋白质理化性质及其二级结构、亚细胞定位预测及进化分析
在小麦基因组中共找到18个TaPaOs,根据其在染色体上的位置以及进化树分类,将其分别命名为TaPaO1-A~TaPaO6-D,分布在小麦的3A、3B、3D、4A、4B、4D、5A、5B、5D、6A、6B、6D、7A、7B、7D染色体上(图1);根据TaPaOs家族成员在4个亚家族的位置及其在ABD亚基因组中的同源关系将其分为(TaPaO1~TaPaO6)(表2、图2);其家族成员大多有3个拷贝,TaPaO3有2个拷贝,TaPaO6有4个拷贝。
TaPaOs编码的氨基酸数介于470(TaPaO2-D)~571 aa(TaPaO6-A)之间,其平均值为527.94 aa;相对分子质量为52 866.92(TaPaO2-D)~63 745.33 Da(TaPaO6-A),平均值为59 085.01 Da;等电点(isoelectric point,pI)在6.70(TaPaO3-A)~9.53(TaPaO6-B-1)之间,平均值为8.28,除TaPaO1-B、TaPaO2-D、TaPaO3-A的pI值低于7.00外,其余15个蛋白的pI值均高于7.00,为碱性蛋白质;不稳定系数值在36.96(TaPaO1-D)~59.47(TaPaO4-A),平均值为48.77,说明TaPaOs家族蛋白大部分为不稳定蛋白质,但TaPaOs中TaPaO1-A、TaPaO1-B、TaPaO1-D、TaPaO2-D不稳定指数均低于40,为稳定蛋白质;脂肪族指数分布在72.00(TaPaO2-D)~83.82(TaPaO1-A),其平均值为79.32;亲水性分布在-0.368(TaPaO2-D)~-0.184(TaPaO6-B-2),其平均值为-0.263,均为亲水性蛋白质(表2)。蛋白质二级结构预测结果发现,TaPaOs家族蛋白均包含α-螺旋、延伸链、β-转角和无规则卷曲4种二级结构,其中无规则卷曲含量均最高,α-螺旋次之,β-转角最少(表2)。亚细胞定位预测结果显示,所有TaPaOs均定位于叶绿体(chloroplast)(表2)。
为了更加清晰直观地预测TaPaOs家族成员的分化情况以及其与节节麦、乌拉尔图小麦、水稻、玉米及拟南芥之间的同源关系,通过MEGA 7.0将41个PaO蛋白序列采用NJ构建进化树(图2),结果表明:18个小麦TaPaOs与5个节节麦、3个乌拉尔图小麦、6个水稻、5个玉米及4个拟南芥PaO可分为Ⅰ、Ⅱ、Ⅲ、Ⅳ共4个亚家族;其中,亚家族Ⅰ成员最多,含小麦TaPaO3(-A、-D)、TaPaO4(-A、-B、-D)、TaPaO6(-A、-B-1、-B-2、-D);亞家族Ⅱ中包括TaPaO2(-A、-B、-D);亚家族Ⅲ中包括TaPaO1(-A、-B、-D);亚家族Ⅳ中包括TaPaO5(-A、 -B、-D)。每个亚家族中均含有节节麦、乌拉尔图小麦、水稻、玉米及拟南芥的PaO家族蛋白,小麦PaO蛋白与麦类作物节节麦、乌拉尔图小麦亲缘关系最近,其次是禾本科作物玉米和水稻,与拟南芥亲缘关系最远。
2.2 TaPaOs的保守基序分析及基因结构分析
通过MEME数据库对TaPaOs的20个保守基序motif 1~motif 20(简称M1~M20)分析(图3)发现,M1、M2、M3、M5、M6、M10、M13、M15、M19为家族成员的共有motifs,除此之外,亚家族Ⅰ还均含有M7、M8、M9、M11、M20,亚家族Ⅱ还均含有M4、M7、M8、M9、M17、M20,亚家族Ⅲ还均含有M4、M12、M14、M17、M18,亚家族Ⅳ均含有M4、M14、M17。亚家族Ⅱ和亚家族Ⅲ成员间所有motifs种类及分布均相同,亚家族Ⅳ成员间所有motifs种类相同。
对TaPaOs的基因结构分析发现(图3),TaPaOs家族中含有3~9个外显子结构,亚家族Ⅰ的外显子在5~7个之间,亚家族Ⅱ均有7个外显子,亚家族Ⅲ均有9个外显子,亚家族Ⅳ均有3个外显子。亚家族Ⅰ中除TaPaO3-D、TaPaO4-B、TaPaO4-D、TaPaO6-B-1不含非编码区(untranslated regions,UTR)外,其余成员均具有完整的UTR;亚家族Ⅱ、亚家族Ⅲ、亚家族Ⅳ的所有成员均具有完整的UTR。亚家族Ⅱ、亚家族Ⅲ、亚家族Ⅳ成员基因结构相对保守,进一步说明该家族在进化上具有保守性。
2.3 TaPaOs的顺式作用元件分析
通过在线软件(PlantCARE)分析TaPaOs起始密码子上游2 000 bp序列,进而推测影响基因表达的可能因素及基因可能参与的调控途径。结果表明(图4),在TaPaOs起始密码子上游2 000 bp共发现50种响应元件,包括19種光响应元件、10种激素响应元件、6种逆境胁迫以及15种与生长发育有关的其他响应元件。除生长发育所必需的核心启动元件TATA-box和CAAT-box外,光响应元件最多,可能是由于叶绿素是光合作用中捕获光的主要成分。光响应元件中G-box响应元件共105个,占47.51%,除TaPaO2-B外其他成员均有该响应元件,还有AAAC-motif、ACA-motif、ACE、ATCT-motif、AE-box、Box 4、Box II、CAG-motif、chs-Unit 1 m1、GA-motif、GATA-motif、G-box、GT1-motif、GTGGC-motif、I-box、MRE、Sp1、TCCC-motif、TCT-motif等光响应元件。254个激素响应元件中包括39.76%的ABRE(脱落酸响应元件)、24.02%的TGACG-motif(茉莉酸甲酯响应元件)和24.02%的CGTCA-motif(茉莉酸甲酯响应元件),除TaPaO2-D外均包含ABRE,所有成员均含有TGACG-motif和CGTCA-motif,除此之外,还有生长素(TGA-element、AuxRE和AuxRR-core)、赤霉素(GARE-motif、TATC-box和P-box)、水杨酸TCA-element等响应元件。78个胁迫响应元件涉及厌氧诱导、缺氧特异性诱导、低温、干旱、防御和压力以及伤口响应元件(ARE、GC-motif、LTR、MBS、TC-rich repeats、WUN-motif)等。以上结果表明,TaPaOs在参与光响应、激素响应及各类非生物胁迫途径中发挥着重要作用。
2.4 TaPaOs的时空表达模式分析
在WheatOmics中下载并整理分析TaPaOs的时空表达模式。结果表明(图5),TaPaO3(-A、-D)、TaPaO5-D和TaPaO6-B-2在茎中表达量最高,TaPaO6-B-1在茎叶中几乎不表达,其余13个家族成员在叶中表达最高,TaPaOs在根中均表达量最低。TaPaO3(-A、-D)、TaPaO6-B-1在小麦抽穗期以及花后0、15、25、30 d各个时期表达量都很低,TaPaO1(-A、-B、-D)在抽穗期以及花后0、5、15、25 d的表达量较高,在花后30 d表达量较低,TaPaO2(-A、-B、-D)、TaPaO5(-A、-B、-D)、TaPaO6(-A、-B-1、-D)在花后25、30 d的表达最高,是在抽穗期以及花后0、5、15 d的表达量的二倍以上,TaPaO3(-A、-D)、TaPaO6-B-1在各时期几乎不表达,TaPaO4(-A、-B、-D)在各时期均有表达。
2.5 不同组织及胁迫处理下TaPaOs的表达特性
根据TaPaOs序列设计qRT-PCR引物(表1),分析TaPaOs(TaPaO1~TaPaO6)分别在幼苗根、茎、叶中的相对表达量(图6),结果表明,TaPaOs在根中几乎都不表达,表达模式可总结为:叶>茎>根,总体上与转录组数据结果一致。
为了分析TaPaOs在衰老过程中的表达模式,利用qRT-PCR分析TaPaOs(TaPaO1~TaPaO6)分别在ABA诱导衰老、黑暗诱导衰老以及自然衰老过程中的相对表达量(图7),结果表明,在三种处理下,TaPaOs表现出不同的衰老表达模式。在ABA诱导衰老过程中的表达模式略有差异,TaPaO1在处理24 h表达量最高,TaPaO3、TaPaO6在处理6 h表达量最高,TaPaO2、TaPaO5在处理48 h表达量最高,TaPaO4在处理后几乎不表达;在黑暗诱导衰老过程中,TaPaOs整体呈现出先升高后降低的趋势,可能由于在后期小麦适应了黑暗处理,导致表达降低,其中,TaPaO1、TaPaO3在黑暗处理12 h时表达量最高,TaPaO2、TaPaO4、TaPaO5、TaPaO6在黑暗处理3 d时表达量最高;在自然衰老时,各成员表达不一致,TaPaO1在花后16 d时,表达量达最高,TaPaO2、TaPaO5、TaPaO6在后期表达量较高,在花后22 d时表达最高,TaPaO3、TaPaO4在整个时期中均处于低水平的表达。
3 讨论
PaO是叶绿素分解代谢途径中的关键酶,在植物生长发育过程中具有重要作用。六倍体小麦在进化过程中发生了两次多倍体化现象[32],引起基因组中大规模基因的丢失和大量重复基因的产生[33]。本研究利用生物信息学方法在小麦全基因组中发现18个TaPaOs,分别分布于小麦的15条染色体上,其中发现TaPaO3缺少B基因组的同源基因,TaPaO6的B染色体有2个同源基因(TaPaO6-B-1和TaPaO6-B-2),推测其在小麦进化过程中发生了基因丢失和复制事件。前人研究表明,拟南芥[17]和黄瓜(Cucumis sativus L.)[34]PaO蛋白均定位于叶绿体中,与本研究亞细胞定位预测结果一致,叶绿体中的类囊体膜是植物进行光合作用光化学反应的场所[35],所以,TaPaOs可能与小麦叶片的光合作用有关。通过保守基序和基因结构分析基因家族具有重要意义[36],研究分析发现,TaPaOs家族基因分为4个亚家族,同一亚家族的外显子数量基本相同,保守基序相同或相似,说明TaPaOs在进化上保守,可能具有类似的功能。
PaO在不同植物中具有明显的组织特异性,其在花、种子及叶片发育中具有重要作用。例如:拟南芥AtPaO在花和角果中的表达高于其他组织[11];油菜BnPaO2在整个种子发育过程中均表达,BnPaO1在种子发育早期表达[13];水稻OsPaO在叶片中表达水平最高[16];辣椒(Capsicum annuum L.)CaPaO[37]在所有组织中均有表达,叶片中的表达水平高于其他组织。本研究采用转录组数据结合qRT-PCR方法,同样发现TaPaOs家族成员主要在叶中表达,在根中表达量较低。不同物种间组织的差异表达表明物种在进化过程中功能发生分化。
基因启动子区域的顺式作用元件决定了该基因的诱导表达模式[38]。本研究通过对TaPaOs起始密码子上游2 000 bp的顺式作用元件分析发现:除核心启动元件外,ABA响应元件(ABRE)以及光响应元件(G-Box)含量最高,说明TaPaOs可能受这些元件的调控。有研究表明,外源喷施ABA及黑暗诱导会引起PaO表达,PaO表达的增加与叶绿素降解有关[39],即PaO可能在转录水平上调控衰老。例如:外源施加ABA诱导衰老增加了拟南芥AtPaO[40]、水稻OsPaO[16]及辣椒CaPaO[37]的表达;在黑暗诱导的衰老和自然衰老过程中,水稻OsPaO[11]以及大豆[Glycine max (Linn.) Merr.]GmLlsl[41]随着衰老表达上调;在西兰花采后衰老过程中BoPaO的表达增加[12];在拟南芥整个生育期AtPaO表达逐渐升高,并在8周即自然衰老时达到最高峰[11]。本研究通过qRT-PCR分析也表明,TaPaOs响应ABA诱导衰老、黑暗诱导衰老以及自然衰老。前人研究发现:拟南芥AtACD1主要编码PaO,是叶绿素分解代谢途径中的关键酶[11];AtTic55编码蛋白转运过程中具有调节光合作用或氧化还原相关功能的前体蛋白,使叶绿体正常行使光合功能,并通过间接调节下游SAGs表达在拟南芥黑暗诱导的衰老中起作用[42-44];从叶绿体中分离的52 kD蛋白PTC52为原叶绿素a氧化酶,负向调控叶绿素的生物合成[45]。本研究发现,在3种衰老过程中TaPaO2、TaPaO5及TaPaO6的表达量最高,且分别与拟南芥AT3G44880(AtACD1)、AT2G24820(AtTic55)、At4G25650(AtPTC52)在同一亚家族,所以小麦TaPaOs基因家族中TaPaO2、TaPaO5及TaPaO6可能在叶绿素分解代谢中扮演着重要角色。
综上所述,本研究利用生物信息学方法在小麦中鉴定到18个基因家族成员,其编码蛋白大多数为不稳定蛋白,均具有亲水性,亚细胞定位预测均位于叶绿体中;表达模式分析发现,TaPaOs在不同组织中的表达模式主要为叶>茎>根;TaPaO2、TaPaO5及TaPaO6在ABA诱导衰老、黑暗诱导衰老以及自然衰老时表达量高于其他基因,表明这3个基因在小麦衰老过程中发挥着重要的作用,为衰老研究重点关注基因。
参考文献(References):
[1] 张浩. 关于中国小麦生产成本现状分析与展望[J]. 农业与技术, 2021, 41(23): 139-143.
ZHANG Hao. Analysis and prospect of wheat production cost in China[J]. Agriculture and Technology, 2021, 41(23): 139-143.
[2] 马娜. 小麦脱镁叶绿酸a加氧酶基因 (TaPaO)的克隆及表达分析[D]. 泰安: 山东农业大学, 2012.
MA Na. Cloning and expression analysis of wheat pheophorbide a oxygenase gene TaPaO [D]. Taian: Shandong Agricultural University, 2012.
[3] XIE Q, LIANG Y, ZHANG J, et al. Involvement of a putative bipartite transit peptide in targeting rice pheophorbide a oxygenase into chloroplasts for chlorophyll degradation during leaf senescence[J]. Journal of Genetics and Genomics, 2016, 43(3): 145-154.
[4] 王丹, 李升东, 冯波, 等. 不同耕作方式对小麦光合性能和产量形成的影响[J]. 山东农业科学, 2019, 51(10): 35-39.
WANG Dan, LI Shengdong, FENG Bo, et al. Effects of different tillage methods on photosynthetic performance and yield formation of wheat[J]. Shandong Agricultural Sciences, 2019, 51(10): 35-39.
[5] H?rtensteiner S. Update on the biochemistry of chlorophyll breakdown[J]. Plant Molecular Biology, 2013, 82(6): 505-517.
[6] H?rtensteiner S. Chlorophyll degradation during senescence[J]. Annual Review of Plant Biology, 2006, 57(1): 55-77.
[7] Schelbert S, Aubry S, Burla B, et al. Pheophytin pheophorbide hydrolase (pheophytinase) is involved in chlorophyll breakdown during leaf senescence in Arabidopsis[J]. The Plant Cell, 2009, 21(3): 767-785.
[8] Tang C, Wang X, Duan X, et al. Functions of the lethal leaf-spot 1 gene in wheat cell death and disease tolerance to Puccinia striiformis[J]. Journal of Experimental Botany, 2013, 64(10): 2955-2969.
[9] Reinbothe S, Bartsch S, Rossig C, et al. A protochlorophyllide (pchlide) a oxygenase for plant viability[J]. Frontiers in Plant Science, 2019, 10: 593.
[10] Gray J, Close P S, Briggs S P, et al. A novel suppressor of cell death in plants encoded by the Lls1 gene of maize[J]. Cell, 1997, 89(1): 25-31.
[11] Pruzinská A, Tanner G, Anders I, et al. Chlorophyll breakdown: pheophorbide a oxygenase is a Rieske-type iron-sulfur protein, encoded by the accelerated cell death 1 gene[J]. Proceedings of the National Academy of Sciences, 2003, 100(25): 15259-15264.
[12] Gomez-Lobato M E, Civello P M, Martínez G A. Effects of ethylene, cytokinin and physical treatments on BoPaO gene expression of harvested broccoli[J]. Journal of the Science of Food and Agriculture, 2012, 92(1): 151-158.
[13] Chung D W, Pruzinská A, H?rtensteiner S, et al. The role of pheophorbide a oxygenase expression and activity in the canola green seed problem[J]. Plant Physiology, 2006, 142(1): 88-97.
[14] Jiang H, L M, Liang N, et al. Molecular cloning and function analysis of the stay green gene in rice[J]. The Plant Journal, 2007, 52(2): 197-209.
[15] Rodoni S, Schellenberg M, Matile P. Chlorophyll breakdown in senescing barley leaves as correlated with phaeophorbidea oxygenase activity[J]. Journal of Plant Physiology, 1998, 152(2/3): 139-144.
[16] Tang Y, Li M, Chen Y, et al. Knockdown of OsPaO and OsRCCR1 cause different plant death phenotypes in rice[J]. Journal of Plant Physiology, 2011, 168(16): 1952-1959.
[17] Sakuraba Y, Schelbert S, Park S Y. STAY-GREEN and chlorophyll catabolic enzymes interact at light-harvesting complex II for chlorophyll detoxification during leaf senescence in Arabidopsis[J]. The Plant Cell, 2012, 24(2): 507-518.
[18] Yates A D, Allen J, Amode R M, et al. Ensembl genomes 2022: an expanding genome resource for non-vertebrates[J]. Nucleic Acids Research, 2022, 50(D1): D996-D1003.
[19] Mistry J, Chuguransky S, Williams L, et al. Pfam: the protein families database in 2021[J]. Nucleic Acids Research, 2021, 49(D1): D412-D419.
[20] International Wheat Genome Sequencing Consortium (IWGSC). Shifting the limits in wheat research and breeding using a fully annotated reference genome[J]. Science, 2018, 361(6403): eaar7191.
[21] Lu S, Wang J, Chitsaz F, et al. CDD/SPARCLE: the conserved domain database in 2020[J]. Nucleic Acids Research, 2020, 48(D1): D265-D268.
[22] Chen C, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202.
[23] Gasteiger E, Hoogland C, Gattiker A, et al. Protein identification and analysis tools on the ExPASy server[J]. The Proteomics Protocols Handbook, 2005: 571-607.
[24] Chou K C, Shen H B. Plant-mPLoc: a top-down strategy to augment the power for predicting plant protein subcellular localization[J]. PLoS One, 2010, 5(6): e11335.
[25] Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874.
[26] Letunic I, Bork P. Interactive tree of life (iTOL) v5: an online tool for phylogenetic tree display and annotation[J]. Nucleic Acids Research, 2021, 49(W1): W293-W296.
[27] Koonin E V. Orthologs, paralogs, and evolutionary genomics[J]. Annual Review of Genetics, 2005, 39(1): 309-338.
[28] Bailey T L, Elkan C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers[J]. Proceedings of the Second International Conference on Intelligent Systems for Molecular Biology, 1994, 2(1): 28-36.
[29] Lescot M, Déhais P, Thijs G, et al. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research, 2002, 30(1): 325-327.
[30] International Wheat Genome Sequencing Consortium (IWGSC). A chromosome-based draft sequence of the hexaploid bread wheat (Triticum aestivum) genome[J]. Science, 2014, 345(6194): 1251788.
[31] Gregersen P L, Holm P B. Transcriptome analysis of senescence in the flag leaf of wheat (Triticum aestivum L.)[J]. Plant Biotechnology Journal, 2007, 5(1): 192-206.
[32] 王艳珍. 普通小麦-十倍体长穗偃麦草衍生后代的分子细胞遗传学研究及特异标记开发[D]. 杨凌: 西北农林科技大学, 2021.
WANG Yanzhen. Molecular cytogenetic studies on wheat-Thinopyrum ponticum derivatives and development of specificmarkers[D]. Yangling: Northwest A&F University, 2021.
[33] 孫桑蓉. 麦类作物基因组结构解析与多倍化过程重构[D]. 唐山: 华北理工大学, 2017.
SUN Sangrong. Comparative genomics analysis of wheat genome structure and reconstruction of chromosome repatterning after polyploidizations[D]. Tangshan: North China University of Science and Technology, 2017.
[34] 程奕秋, 刘伟康, 陈广玲, 等. 黄瓜叶绿素降解关键酶基因CsPAO的克隆与分析[J]. 西北植物学报, 2021, 41(10): 1635-1642.
CHENG Yiqiu, LIU Weikang, CHEN Guangling, et al. Cloning and analysis of chlorophyll degradation key enzyme gene (CsPAO) in Cucumber sativus L.[J]. Acta Botanica Boreali-Occidentalia Sinica, 2021, 41(10): 1635-1642.
[35] 郑洁, 胡美君, 郭延平. 光质对植物光合作用的调控及其机理[J]. 应用生态学报, 2008, 19(7): 1619-1624.
ZHENG Jie, HU Meijun, GUO Yanping. Regulation of photosynthesis by light quality and its mechanism in plants [J]. Chinese Journal of Applied Ecology, 2008, 19(7): 1619-1624.
[36] 杨成兰, 祁存英, 马银花, 等. 青稞GPAT基因家族全基因鉴定及表达分析[J]. 植物生理学报, 2022, 58(10): 2006-2016.
YANG Chenglan, QI Cunying, MA Yinhua, et al. Identification and expression analysis of GPAT gene family in hulless barley[J]. Plant Physiology Journal, 2022, 58(10): 2006-2016.
[37] Xiao H J, Liu K K, Li D W, et al. Cloning and characterization of the pepper CaPAO gene for defense responses to salt-induced leaf senescence[J]. BMC Biotechnology, 2015, 15(1): 1-12.
[38] 王海波, 郭俊云. 小桐子低温诱导型启动子JcDnaJ20p的克隆及烟草转化功能鉴定[J]. 生物技术通报, 2021, 37(2): 24-31.
WANG Haibo, GUO Junyun. Molecular cloning of cold-induced JcDnaJ20p promoter from jatropha curcas and its functional identification in transgenic Tobacco[J]. Biotechnology Bulletin, 2021, 37(2): 24-31.
[39] Pruzinská A, Tanner G, Aubry S, et al. Chlorophyll breakdown in senescent Arabidopsis leaves. Characterization of chlorophyll catabolites and of chlorophyll catabolic enzymes involved in the degreening reaction[J]. Plant Physiology, 2005, 139(1): 52-63.
[40] Yang J, Worley E, Udvardi M. A NAP-AAO3 regulatory module promotes chlorophyll degradation via ABA biosynthesis in Arabidopsis leaves[J]. The Plant Cell, 2014, 26(12): 4862-4874.
[41] 李鹏丽, 马媛媛, 李小平, 等. 大豆GmLlsl基因的克隆及表达调控分析[J]. 科学通报, 2006, 9(53): 1034-1041.
LI Pengli, MA Yuanyuan, LI Xiaoping, et al. Cloning and expression regulation analysis of soybean GmLlsl gene[J]. Chinese Science Bulletin, 2006, 9(53): 1034-1041.
[42] 王琪, 李云洲, 须文, 等. 叶绿体TOC-TIC蛋白复合体转运机制研究进展[J]. 园艺学报, 2021, 48(4): 689-704.
WANG Qi, LI Yunzhou, XU Wen, et al. Research advance of protein transport mechanism of TOC-TIC complexes in plant chloroplast[J]. Acta Horticultural Sinica, 2021, 48(4): 689-704.
[43] Hauenstein M, Christ B, Das A, et al. A role for TIC55 as a hydroxylase of phyllobilins, the products of chlorophyll breakdown during plant senescence[J]. The Plant Cell, 2016, 28(10): 2510-2527.
[44] Chou M L, Liao W Y, Wei W C, et al. The direct involvement of dark-induced Tic55 protein in chlorophyll catabolism and its indirect role in the MYB108-NAC signaling pathway during leaf senescence in Arabidopsis thaliana[J]. International Journal of Molecular Sciences, 2018, 19(7): 1854.
[45] Bartsch S, Monnet J, Selbach K, et al. Three thioredoxin targets in the inner envelope membrane of chloroplasts function in protein import and chlorophyll metabolism[J]. Proceedings of the National Academy of Sciences, 2008, 105(12): 4933-4938.
收稿日期:2022-10-26;修回日期:2022-12-10。
基金项目:山西农业大学学术恢复科研专项项目(2020xshf02);國家自然科学基金项目(31671607)。
作者简介:闫荣岳,硕士研究生。
* 通信作者:范华,副教授,主要从事小麦遗传育种的研究,E-mail: jeanafan@163.com;
孙黛珍,教授,主要从事小麦遗传育种的研究,E-mail: sdz64@126.com。
