利用Markov模型研究Aβ突变体的构象转变过程
2024-01-15姚星宇刘璎锐韩葳葳万由衷
姚星宇, 刘璎锐, 韩葳葳, 万由衷
(1. 吉林大学 白求恩第三医院, 长春 130033; 2. 吉林大学 生命科学学院, 长春 130012)
阿尔兹海默症(AD)是一种进行性神经退行性疾病, 主要影响大脑的记忆、 思维和行为功能, 是老年人最常见的痴呆症之一[1]. 阿尔兹海默症的临床表现包括记忆力减退, 尤其是新近记忆受损. 阿尔兹海默症的发病机制尚不完全清楚, 但有几个关键因素被认为是导致疾病的原因之一[2-3], 其中一个因素是β-淀粉样蛋白(Aβ)的异常聚集[3]. 对阿尔兹海默症的治疗方法展开深入研究已引起人们广泛关注[4]. Aβ是一种在大脑中产生的蛋白质片段, 正常情况下会被清除掉. 而在阿尔兹海默症患者中, Aβ会形成斑块, 干扰神经元的正常功能[5]. 此外, 在阿尔兹海默症患者的大脑中, Tau蛋白会异常地聚集成纤维缠结, 干扰神经元之间的正常信号传递[6]. 炎症反应、 氧化应激和神经元死亡也与阿尔兹海默症的发病机制有关.
由于Aβ是由跨膜淀粉样前体蛋白(APP)经β-分泌酶与γ-分泌酶共同作用下水解, 并由39~43个残基结合而成. 因此存在一种淀粉质沉淀假说[7-8], 内容是Aβ聚集形成的寡聚物具有神经毒性, Aβ在大脑中的产生速率大于清除速率, 导致该神经毒性寡聚物聚集致病[9]. 因此, 对Aβ聚集过程进行抑制, 即可消除神经毒性物质的产生, 从而对阿尔兹海默症起到一定治疗作用[1,10].
研究表明, Aβ聚集的过程中, 有由α-螺旋或无规卷曲到β-折叠的构象转变现象, 且该现象被证实为聚集过程的关键步骤[11]. 因此, 通过残基突变抑制β-折叠的形成, 即可抑制Aβ聚集成为神经毒性物质[12-14]. 其中, 将4种甘氨酸(G25,G29,G33,G37)全部突变为丙氨酸, 甚至可以几乎完全消除β-折叠结构的产生[15-16]. 但该构象转变发生的十分迅速, 通过常规实验方法无法进行跟踪式检测, 难以对其机理进行更进一步研究. 因此, 本文通过使用计算生物学的方法进行实验, 结合Markov模型和分子动力学模拟, 对野生型、 A4型和D7N型Aβ的构象变化过程进行研究.
1 实 验
1.1 分子动力学模拟
计算生物学中的分子动力学模拟是将物理、 数学和化学结合的一门学科技术, 能使物质的宏观和微观性质相结合从而进行研究, 其主要原理是通过牛顿力学关系计算, 对分子体系的运动过程进行模拟仿真[17]. 分子动力学模拟的一般步骤包括:
1) 定义系统. 确定模拟的分子系统, 包括分子的种类、 初始位置和速度等.
2) 确定势能场. 选择适当的势能场描述分子之间的相互作用, 如分子力场.
3) 运动方程求解. 根据牛顿运动定律和势能场, 求解分子的运动方程, 通常使用数值积分方法.
4) 模拟时间演化. 通过迭代求解运动方程, 模拟分子在一定时间范围内的运动和相互作用.
5) 数据分析. 对模拟结果进行分析, 如计算物理量和构建分子轨迹等, 以获得关于分子系统的信息.
1.2 Markov模型
Markov模型是一种用于描述随机过程的数学模型, 它基于Markov性质, 即未来状态仅依赖于当前状态, 与过去的状态无关. 构建Markov模型的一般步骤包括: 定义状态空间, 确定系统的所有可能状态; 确定状态转移概率, 计算状态之间的转移概率, 即从一个状态转移到另一个状态的概率; 得到状态转移矩阵, 将状态和转移概率表示为一个矩阵, 其中每个元素代表从一个状态到另一个状态的概率; 进行状态预测, 利用状态转移矩阵进行状态预测, 可以通过给定的初始状态和转移概率计算未来状态的概率分布.
1.3 实验流程总述
图1为本文实验流程. 首先获取野生型Aβ、 两种突变体A4型Aβ和D7N型Aβ的轨迹, 用数组数据表示轨迹, 再用时滞独立组分分析对数据进行降维处理, 并用k-均值聚类获得准确的聚类结果, 然后确定时滞时间(lag time), 构建Markov模型, 最后将分子轨迹进行PCCA(perron cluster cluster analysis)聚类, 将上述获得的状态结果和转移概率矩阵用于通量分析.

图1 实验流程Fig.1 Flow chart of experiment
2 Aβ及突变体构象转变过程
2.1 3个体系的分子动力学模拟
为研究野生型Aβ(PDB id: 2M4J)[18]、 两种突变体A4型Aβ(G25,G29,G33,G37)和D7N型Aβ的动态构象变化过程, 先进行分子动力学(MD)模拟. 对每个体系进行10次1 000 ns的显式水模型模拟以探索其动态行为. 选择GROMACS 2018.3[19]作为MD软件包运行, 并采用Gromos54a7力场[20]描述分子间相互作用. 通过GROMACS自带的x2top组件, 生成3种Aβ的拓扑结构文件, 并将其嵌入到周期性边界模拟盒中, 盒子的尺寸为10 nm×10 nm×10 nm. 模拟温度设定为36 ℃. 为确保体系的稳定性, 先进行100 ps的预平衡MD模拟, 采用Berendsen恒温器[21]在微正则(NVT)系综下进行. 之后在等温等压(NPT)系综下, 保持体系的压强为100 kPa(1 bar), 温度为36 ℃, 对3个体系进行模拟. 范德华作用截断半径设为1 nm. 在所有模拟过程中, 采用2 fs的时间步长, 并每10 ps保存一次原子坐标, 共得到30条轨迹用于后续的分析和研究.
2.2 Aβ体系的时滞独立组分分析
应用pyemma软件包构建Markov模型的全过程. 首先计算野生型Aβ、 两种突变体A4型Aβ和D7N型Aβ的相互作用轨迹, 在平行条件下构建10份轨迹, 每份模拟时长为90 ns, 时间间隔为0.1 ns. 将得到的10份轨迹分别导入pyemma软件包. 导入时选择处残基骨架的phi角和psi角信息为轨迹信息, 并借此对轨迹的每帧进行量化, 将分子动力学模拟轨迹的信息用数据组表示, 实现从抽象化转为具体化. 10条轨迹被分别转化为10个二维数组. 之后, 分别对10个二维数组进行时滞独立组分分析降维, 降维结果至少保留95%的方差信息. 最终降维至82维度. 随后将降维结果进行k-均值聚类, 聚类结果如图2所示. 由图2可见, 野生型Aβ和A4型Aβ的轨迹前后变化较小, 仅有一小部分与整体分离. 其原因可能是野生型Aβ和A4型Aβ的α-螺旋基本不发生变化. 最终野生型Aβ和A4型Aβ选取合适的k值为30, D7N型Aβ选取合适的k值为45.
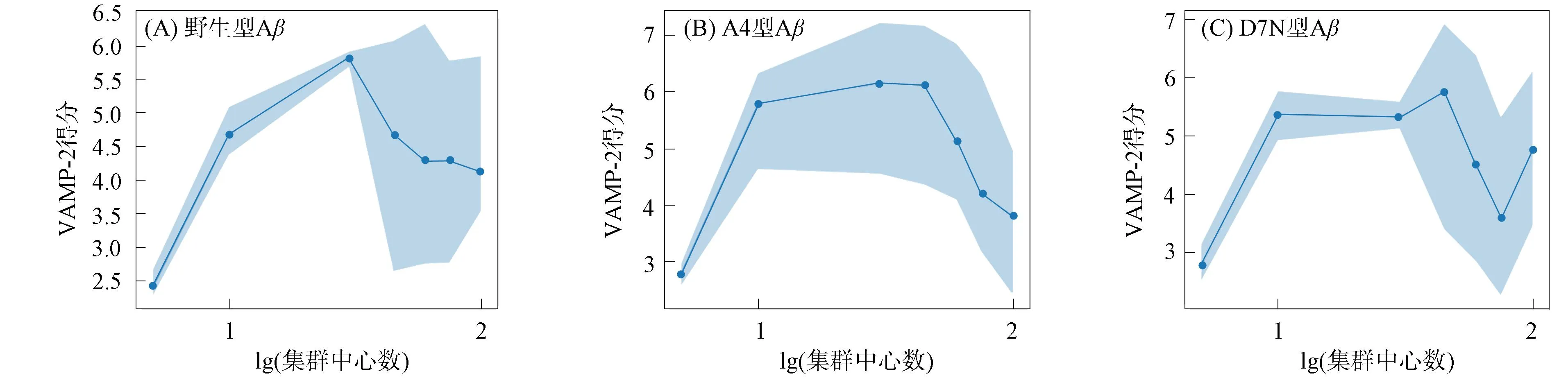
图2 k-均值聚类结果Fig.2 k-Means clustering results
2.3 Aβ体系时滞时间数值的确定
采用msm.its方法计算系统弛豫时间, 将系统弛豫时间标度与时滞时间进行绘图, 结果如图3所示. 由图3可见, 对于野生型Aβ, 当时滞时间约为0.2 ns时, 10个轨迹系统的弛豫时间尺度呈收敛趋势. 由于轨迹系统的间隔时间设定为0.1 ns, 因此弛豫时间为2 steps. 由于A4型Aβ和D7N型Aβ轨迹系统的间隔时间设定为0.1 ns, 10个轨迹系统的弛豫时间尺度呈收敛趋势, 时滞时间约为0.5 ns, 因此弛豫时间为5 steps. 明确时滞时间后, 开始构建Markov模型, 并对转移概率进行计算, 得到转移概率矩阵.
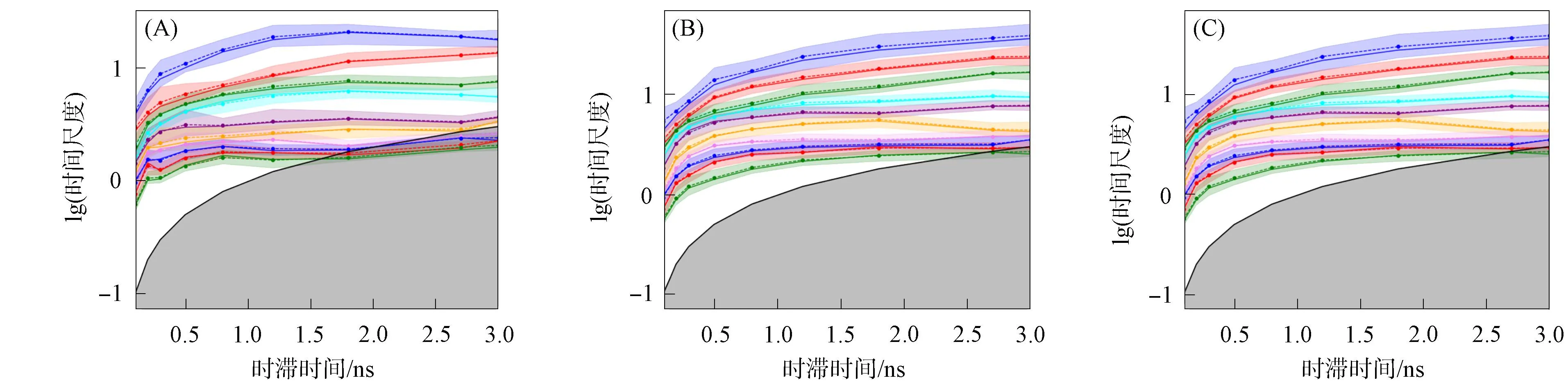
图3 野生型Aβ(A), A4型Aβ(B)和D7N型Aβ(C)时滞时间确定结果Fig.3 Results of determining delay time of wild type Aβ (A), A4 type Aβ (B) and D7N type Aβ (C)
2.4 构建Markov模型和结果分析
2.4.1 构建Markov模型和CK检测
根据获得的聚类结果和时滞时间构建Markov模型, 并计算转移概率矩阵. 为进一步验证所构建模型的Markov性质, 使用CK(Chapman-Kolmogorov test)进行检测, CK检测结果如图4所示. 在该过程中, 使用pyemma中的msm.cktest方法, 并将参数mlags设为4, 即CK检测算法中的倍数k, 同时将nstates设置为5, 表示后续PCCA聚类的宏观状态数. 由图4可见, 各坐标系的Markov模型预测与实验前估测都近于拟合, 表明时滞时间的选取及Markov模型构建均较合理, 说明构建的Markov模型具有良好的Markov性质, 可以用于开展后续研究.
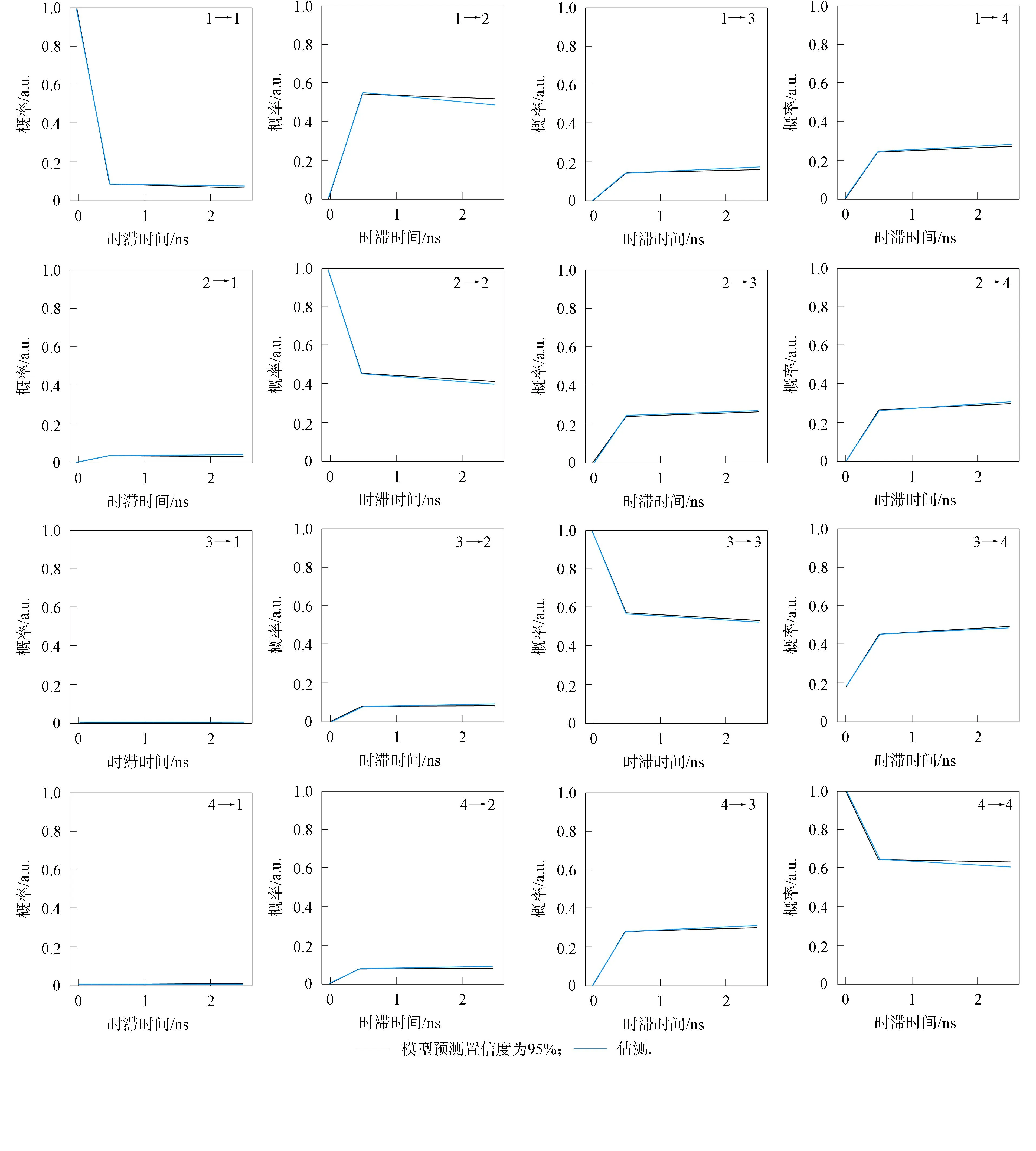
图4 Aβ的CK检测结果Fig.4 CK-test results of Aβ
2.4.2 野生型Aβ的β-折叠构象转变
使用PCCA聚类可对轨迹结果进行可视化分析, 并将分子轨迹系统用宏观状态进行描述. 采用msm.pcca方法对野生型Aβ的β-折叠构象转变进行研究. 在PCCA聚类结果中, SA表示模拟轨迹中构象的起始状态, SB表示模拟轨迹中构象的最终收敛状态, S1,S2,S3表示按时间顺序出现的中间状态. 各聚类状态如图5所示, 紫色标记区域为主要研究的α-螺旋区域. 野生型Aβ的通量分析结果列于表1.

表1 野生型Aβ通量分析结果
由表1可见, 在野生型Aβ的β-折叠构象转变过程中, SA→S3→SB概率为33.49%, 是最具有代表性的路径, 其次是SA→S3→S2→SB路径, 占总通量的28.71%. SA→S1→S3→SB构象转变路径占比也较大, 占总通量的15.79%. 分析其残基具体转变过程, 得出各状态代表构象帧的具体二级结构如下: SA→S3→S2→SB与SA→S1→S3→SB两种构象转变途径大致的二级结构转变与SA→S3→SB的结构转变基本类似, 都出现一个β-折叠的过程, 且通过观察各宏观状态的具体构象发现, 整个过程经历S3的一个中间微状态, 且S3在残基中出现了π-螺旋结构.
2.4.3 A4型Aβ的β-折叠构象转变
A4型Aβ宏观状态构象如图6所示. 由图6可见, A4型Aβ的各PCCA聚类宏观状态的特征分布极接近, 4个宏观状态间的构象差距较小, 且紫色强调处都保持着α-螺旋结构.
A4型Aβ的通量分析结果列于表2. 由表2可见, 在A4型Aβ的β-折叠构象转变过程中, SA→SB概率为43.96%, 是最具有代表性的路径. 通过观察宏观状态的构象和二级结构的计算结果可见, 宏观状态之间的差异非常小. 从S1到SB的二级结构基本没有发生变化, SB与初始状态SA存在一定差异, 但S1到SB仍保持螺旋结构, 表明螺旋只有一部分发生了解旋. 发生解旋的部分主要为该螺旋结构的N端, 而C端仍保持螺旋结构. 综上, A4型Aβ的4个微状态均未出现β-折叠, 说明A4型突变有显著抑制β-折叠构象的特性.

表2 A4型Aβ的通量分析结果
2.4.4 D7N型Aβ的β-折叠构象转变
D7N型Aβ宏观状态构象如图7所示. D7N型Aβ的通量分析结果列于表3. 由表3可见, 在D7N型Aβ的β-折叠构象转变过程中, SA→S1→SB概率为50.51%, 是最具有代表性的路径. 观察二级结构可知, S2和SB两个微状态均出现β-折叠. 由图7可见, 微状态SA代表的圆型面积很小, 说明符合微状态SA的构象很少, 可能意味着初始微状态SA发生变化的时间很短, 更进一步证明了D7N型突变对β-折叠转变过程的促进作用.

表3 D7N型Aβ的通量分析结果

图7 D7N型Aβ宏观状态构象Fig.7 Macroscopic state conformation of D7N type Aβ
综上可见, 在野生型Aβ的β-折叠构象转变过程中, SA→S3→SB路径出现概率为33.49%, 概率最大, 较具有研究意义. 整个过程经历S3一个中间微状态, 且微状态S3在残基中出现了π-螺旋结构, 此结构为野生型Aβ中发现的独有结构.
在A4型Aβ的β-折叠构象转变过程中, 4个微状态构象极相似, 几乎可认为没有发生变化, 完全由α-螺旋与无规卷曲组成. 其中SA→SB路径出现概率最大, 为43.96%, 说明该构象转变较大概率没有经过任何中间状态的改变, 更证实了构象几乎无变化. 因此, A4型突变可极大程度抑制β-折叠构象转变的发生, 通过抑制β-折叠抑制Aβ聚集为细胞毒性物质, 为开发抗阿尔兹海默症药物提供一定理论依据.
在D7N型Aβ的β-折叠构象转变过程中, SA→S1→SB概率为50.51%, 概率最大, 较具有研究意义. 其中, 最终构象SB的α-螺旋近乎完全解旋, 为β-折叠的形成提供有利条件. 相比于野生型Aβ只发生一个区域的β-折叠, D7N型突变体发生了两个区域的β-折叠, 说明D7N型突变可促进β-折叠特性. 而且D7N型Aβ中SA微状态的圆面积很小, 表明符合SA微状态的构象数量较少, 说明初始构象转变的速度非常快, 因此D7N突变可能会导致更快的β-折叠构象形成, 从而在阿尔茨海默症的发病机制中发挥重要作用. 该发现为深入理解D7N突变对蛋白质结构和功能的影响提供了有价值的线索.
