基于注意力机制和多模态特征融合的猕猴脑磁共振图像全脑分割
2024-01-12吴雪扬
吴雪扬,张 煜,张 华,钟 涛
南方医科大学生物医学工程学院//广东省医学图像处理重点实验室,广东 广州510515
对研究对象的脑部磁共振(MRI)图像开展精准的处理与分析,对推动神经科学研究的发展至关重要。猕猴作为一种被广泛应用于科学研究的动物模型,与人类在各方面有着高度的系统相似性。所以,针对猕猴大脑开展研究对了解人类大脑的发育和衰老机制,探索脑疾病的发病机制和验证临床技术以及药物的有效性等方面起到不可或缺的作用[1,2]。虽然人类脑影像学研究进展迅速,但猕猴脑影像学研究稍显落后,究其原因,一方面是猕猴的饲养需要较高成本和适宜的环境,导致猕猴医学影像数据较少。另一方面是数据分析方面的挑战,缺乏针对猕猴脑影像定制的分析工具。由于人类和猕猴的大脑在解剖结构和功能上存在一定差异,导致大部分根据人脑定制参数和校准的脑影像分析工具不适用于猕猴。因此,猕猴脑影像分析是一个需要迫切发展的领域。而全脑分割是脑影像分析的关键前置任务,通过对全脑的精细划分,可以进一步专注于特定目标或感兴趣区域,对研究与分析脑部解剖结构以及脑部疾病至关重要[3,4]。
目前,在猕猴脑影像分割研究领域,基于图谱的方法是猕猴全脑分割最常用的手段之一[5],其核心是利用配准算法把脑图谱的分割先验迁移到待处理样本上,然后使用标签转换等技术完成后续的全脑分割,例如图像配准软件(ANTs)[6]和近期发布的利用NIMH Macaque Template(NMT v2)[7]开发的猕猴脑影像处理工具CIVET-Macaque[8]和PREEMACS[9]。然而,一方面基于图谱配准的全脑分割手段较依赖图谱本身的精确度以及图谱和待处理样本之间的契合度。发育期猕猴大脑的低组织对比度和动态发育,包括不同组织的灰度强度和对比度、大脑尺寸等的剧烈变化,给图谱的选择以及配准算法的准确度带来了挑战;另一方面,图谱与MRI图像间的迭代配准,往往需要花费较长的计算时间。因此基于图谱的猕猴全脑分割速度和精度都存在较大的局限性。
全脑分割作为诸多医学图像分割任务中的一种,从数据输入角度可以简单的划分为单一模态输入和多模态输入两种。其中多模态输入可以利用不同模态之间丰富的特征为模型的决策提供更多的信息,降低信息的不确定性,从而提高模型预测结果的精准度。受文献[10-12]的启发,在本次研究中,我们使用T1-weighted(T1-w)图像和T2-weighted(T2-w)图像作为多模态输入,提出基于双编码器、双模态输入、注意力多模态特征融合的猕猴脑MRI图像全脑分割网络(DDAM)。与现有的图像分割网络相比,我们将U-Net[13]编码器部分改变为双编码器结构,并在解码器中加入注意力多模态特征融合模块,通过提取和融合更丰富的多模态特征,从而提升网络分割性能。
考虑到脑部MRI图像中可观察到的皮层区域和皮层下区域之间的不同位置和解剖结构差异,且二者都存在较多的标签类别(36 类cortical label 和34 类subcortical label),因此我们将全脑分割任务分离为皮层区域分割和皮层下区域分割两个任务,分别训练了专注于皮层区域的分割模型和专注于皮层下区域的分割模型,降低了任务复杂度和增强了网络适应训练数据的能力,进而提高分割的精确度。
1 材料和方法
1.1 实验资料
本文实验资料来自 UNC-Wisconsin Neurodevelopment Rhesus Database[14]公开的猕猴数据集。所使用的数据集由68例年龄均匀分布于13~36月的样本组成,每个扫描样本均采集T1-w图像和T2-w图像(图1A)。参数如下:T1-w图像矩阵为256×256,分辨率为0.5469 mm×0.5469 mm×0.8 mm;T2-w图像矩阵为256×256,分辨率为0.6016 mm×0.6016 mm×0.6 mm。在图像预处理方面,使用FSL(5.0版)[15]软件的图像配准工具(FLIRT)将每个T2-w图像与其对应的T1-w图像严格对齐,然后将所有图像重采样至各向同性分辨率为0.5469 mm。我们采用DIKA-Nets[16]方法去除脑颅骨,以及N4偏置性校正[17]对图像进行强度不均匀性校正。与此同时,为了获取T1-w图像和T2-w图像对应的标签,我们使用ANTs软件的SyN算法将NMT v2猕猴大脑模板[18,19]与当前所有实验样本进行配准得到对应的转换矩阵,并通过转换矩阵将NMT v2对应的全脑分割标签转换为每例样本对应的精细结构标签,包括皮层区域共36类和皮层下区域共34类(图1B),最后由经验丰富的影像专家使用ITK-SNAP软件[20]进行手工校正。
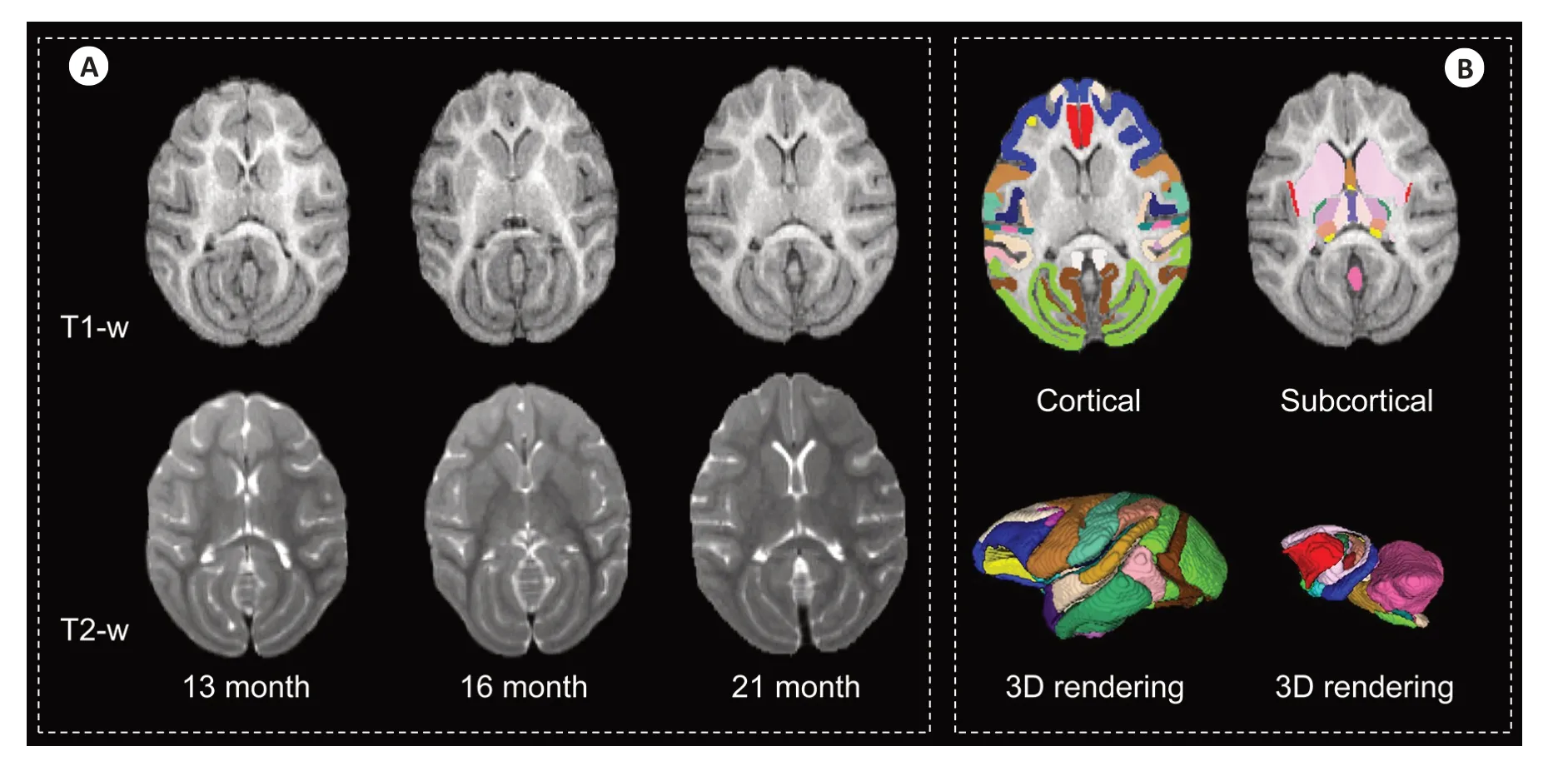
图1 猕猴脑不同年龄段MRI图像及分割真实标签Fig.1 MRI images of macaques at different ages (A) and parcellation ground truth and 3D rendering,including 36 categories of cortical labels and 34 categories of subcortical labels(B).
1.2 网络框架
本研究选用U-Net作为Backbone,包含编码器和解码器两部分,并且其输入模态分别为T1-w和T2-w。其中,编码器部分采用独立的双编码路径,每个编码器路径包含用于特征提取的4个由双卷积模块和最大池化层组成的下采样层。解码器部分为单解码路径,由4个上采样层和4个特征融合模块组成,通过对特征进行上采样并输出原尺寸大小的分割结果。对两个独立编码器的同一级下采样层进行特征拼接,并通过跳跃连接传入解码器的对应上采样层的特征融合模块。其中,特征融合模块可以有效地融合多模态的低级特征与高级特征并获取更多的特征信息,从而提高分割结果的准确度(图2)。
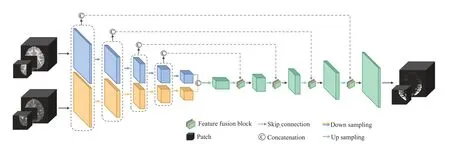
图2 网络框架Fig.2 Overview of the proposed 3D parcellation framework based on dual-encoder with dual-modality inputs and Attentional Multi-modality feature fusion block (DDAM).The blue and orange blocks represent the encoders of T1-w and T2-w,respectively.The green blocks represent the common decoder.The patches of preprocessed T1-w and T2-w are the input of the network.
1.3 注意力多模态特征融合模块
本研究提出注意力多模态特征融合模块(AMFF)。AMFF 可分为前后两部分:低级特征放大(LFA)(图3A)和深层特征融合(LFF)(图3B),二者均采用注意力机制。

图3 注意力多模态特征融合模块Fig.3 Architecture of the proposed Attentional Multi-modality Feature Fusion block(AMFF).Specifically,the input X2 is the up-sampling feature,and the input X1 is the down-sampling features after concatenation.A:LFA;B:LFF.
对于输入尺寸为C×H×W×D的特征图X1,X2,X1为跳跃连接传入的多模态拼接特征,X2为上采样特征,可转换为元素表示为:
tk压缩了Xn第k个通道内的所有空间信息。然后先让T通过一个全连接层和ReLU,此时再通过第二个全连接层复原T∈RC×1×1×1。最后通过sigmoid层将每个元素进行归一化得到通道权重系数σ(tk)并与特征图逐元素相乘得到d1,
σ(tk)代表了第k个通道的重要性。另一方面,通过压缩空间信息来获得空间权重系数。与LFA类似,先将Xn在元素重新表示为然后通过1×1×1卷积压缩空间信息得到q4,q4=Y4*Xn。最后将其归一化获得空间权重系数,并在空间区域上对特征图Xn重新校准,得到d2,
σ表示了空间元素的重要性。最后整合空间特征与通道特征,得到融合特征:
1.4 实验细节
本次实验共使用68例猕猴脑MRI样本,并采用4折交叉验证,所有实验均基于Pytorch1.13.0实现,并在Ubuntu 18.04 系统中使用24GB GPU(NVIDIA RTX 4090)进行。在模型训练前,将所有样本去除冗余背景,并经过裁剪使大小变为160×160×160,然后按步长32重采样成64×64×64的切块。模型训练时,采用Adam优化器,批大小为4,最大迭代为120,前60个迭代的学习速率固定0.0002,后60个迭代的学习速率逐渐递减至0。损失函数包括骰子相似(Dice)损失函数和交叉熵(CE)损失函数,可用公式表示为:
其中,N为每例样本中的元素集合,yi表示模型预测值的第i个元素,表示真实标签的第i个元素,平滑值ε=1e-5用于防止计算时出现分母为0的情况。C为每例样本中的类别数,yj表示第j个类别的模型预测值,表示第j个类别的真实标签的独热编码(onehot)。在模型测试阶段,将结果所得的切块进行滑动拼接,相邻切块之间的重叠部分取平均值,重建为160×160×160大小并进行结果评估。
为验证所构建模块对Backbone(即U-Net)的改进作用,我们基于单模态输入(T1-w)和双模态输入(T1-w、T2-w)对各组成部分进行消融实验分析。具体包括:U-Net+单模态输入(SS),U-Net+双模态输入(SD),双编码器U-Net+双模态输入(DD),双编码器U-Net+双模态输入+AMFF模块(DDAM)。为了客观对四个不同网络模型的分割结果进行评估,本文将分割结果与专家校正的真实标签进行比较,同时选用骰子相似系数(DSC)和平均表面距离(ASD)作为评估标准。
为了避免实验结果的偶然性,我们使用配对t检验比较了不同方法分割结果的Dice值,显著性水平设定为0.05。将各组分割结果的DSC分数作为独立样本分别计算t值,再通过t值得到P值并与设定的显著性水平0.05进行比较,当P<0.05时认为差异存在统计学意义。
2 结果
2.1 消融实验
针对本文方法的消融实验分析以及量化结果显示(表1)。在Backbone的基础上(SS)增加额外模态输入T2-w(SD)后并没有分割性能上的优势。在此基础上针对T2-w增加编码器(DD)后分割精度明显提升,皮层区域DSC分数提升约0.027且ASD分数降低约0.048,皮层下区域DSC 分数提升约0.038 且ASD 分数降低约0.075。在双编码器的基础上引入AMFF模块(DDAM)后,分割精度进一步提升。从DSC和ASD分数的表现可以看出,方法DDAM明显优于其他三种方法,证明了多编码器和AMFF模块对Backbone的有效改进。此外,图4展示了以上4个模型在同一样本的全脑分割的可视化结果。从图中可以清晰地看到,相较于其它3种方法,DDAM在皮层区域和皮层下区域分割结果都更接近与真实标签,特别是在皮层下区域,避免了常见的分割空洞问题,并保留了更多的解剖细节。
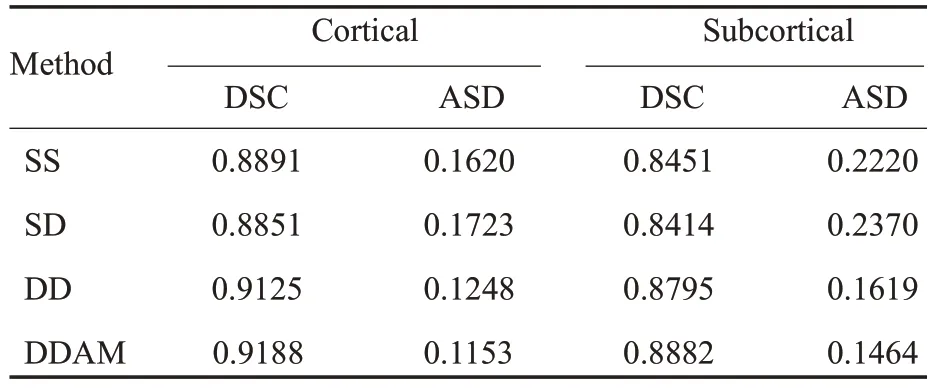
表1 多组消融实验结果对比Tab.1 Quantitative comparison of different ablation settings
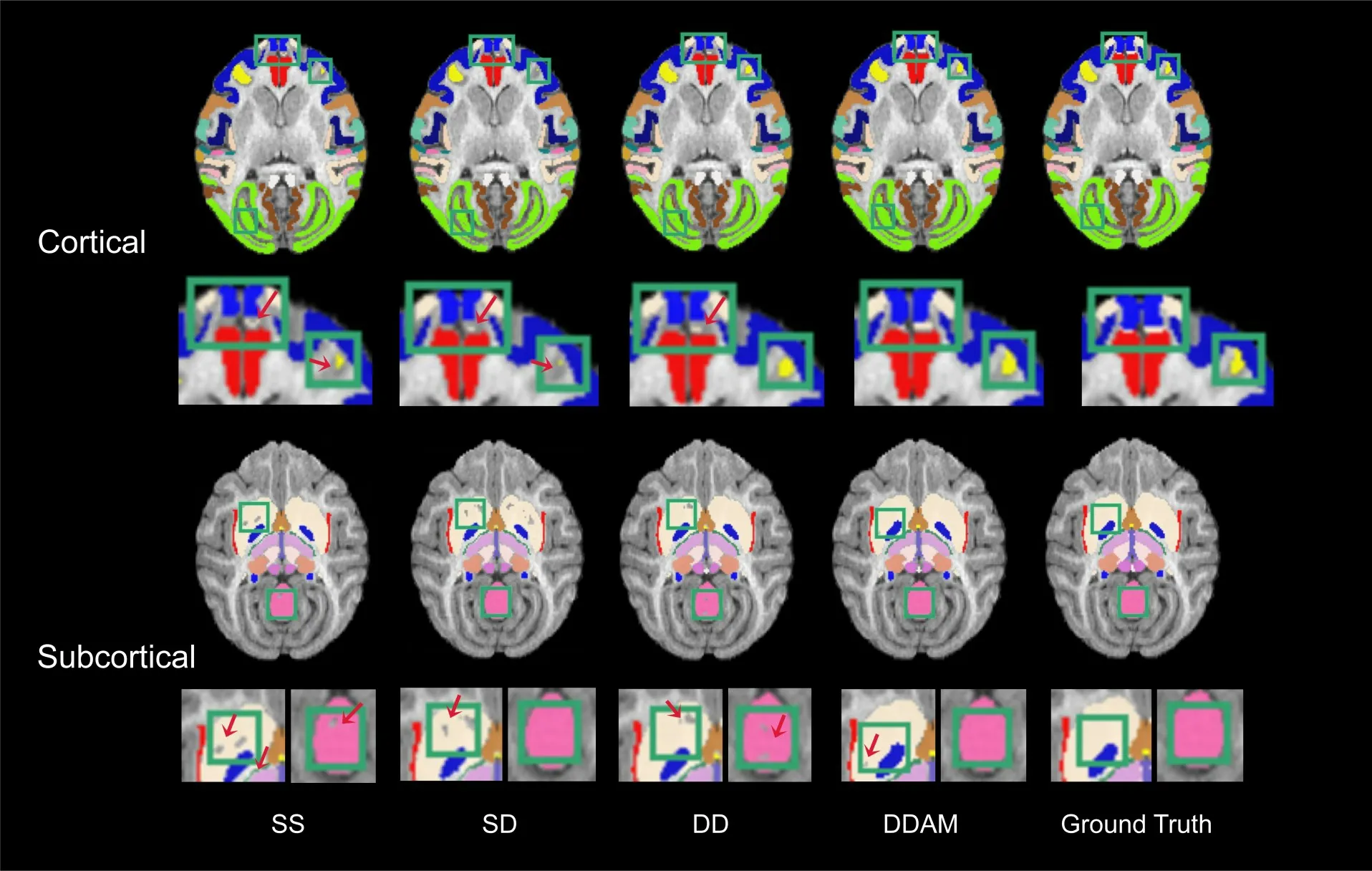
图4 皮层区域和皮层下区域分割可视化结果Fig.4 Representative results of parcellation of cortical and subcortical by DDAM and other settings.The green squares indicate some spots,where DDAM successfully segmented the true labels while other settings failed.Some inaccurate regions are indicated by arrows.SS:Single-encoder with Single-modality input.SD:Single-encoder with Dual-modality input.DD:Dual-encoder with Dual-modality input.DDAM:Dual-encoder with Dual-modality inputs and Attentional Multi-modality feature fusion block.
2.2 显著性检验
本研究将方法DDAM与其它3种方法的4折交叉验证结果的DSC分数进行配对t检验,得出每组配对t检验P<0.05。另外,汇总4折交叉实验结果,在任意年龄段中,代表方法DDAM的绿色曲线无论在皮层区域或是皮层下区域均表现出更高的分割精度(图5)。
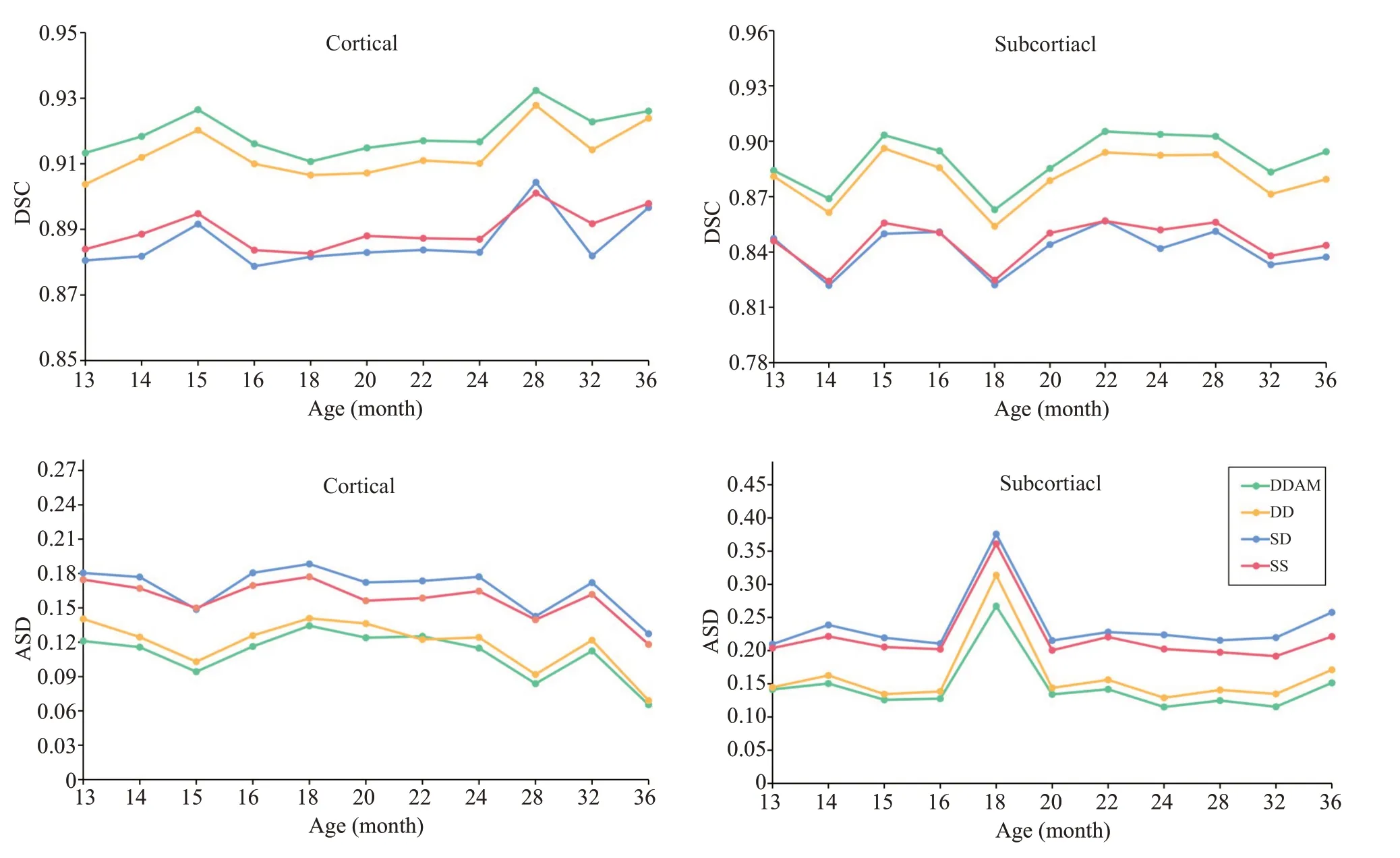
图5 不同年龄段测试结果Fig.5 Evaluation results of cortical and subcortical macaque brain at different month groups.The results of the 4 methods are represented by curves of different colors,respectively.
2.3 全脑结构量化评估
比较消融实验的4种方法结果(表2),方法DDAM在对全脑不同结构的分割方面,DSC分数和ASD分数仍优于其它3种方法。将方法DDAM与其它3种方法分别进行配对t检验,计算得出(P<0.05,表2)。
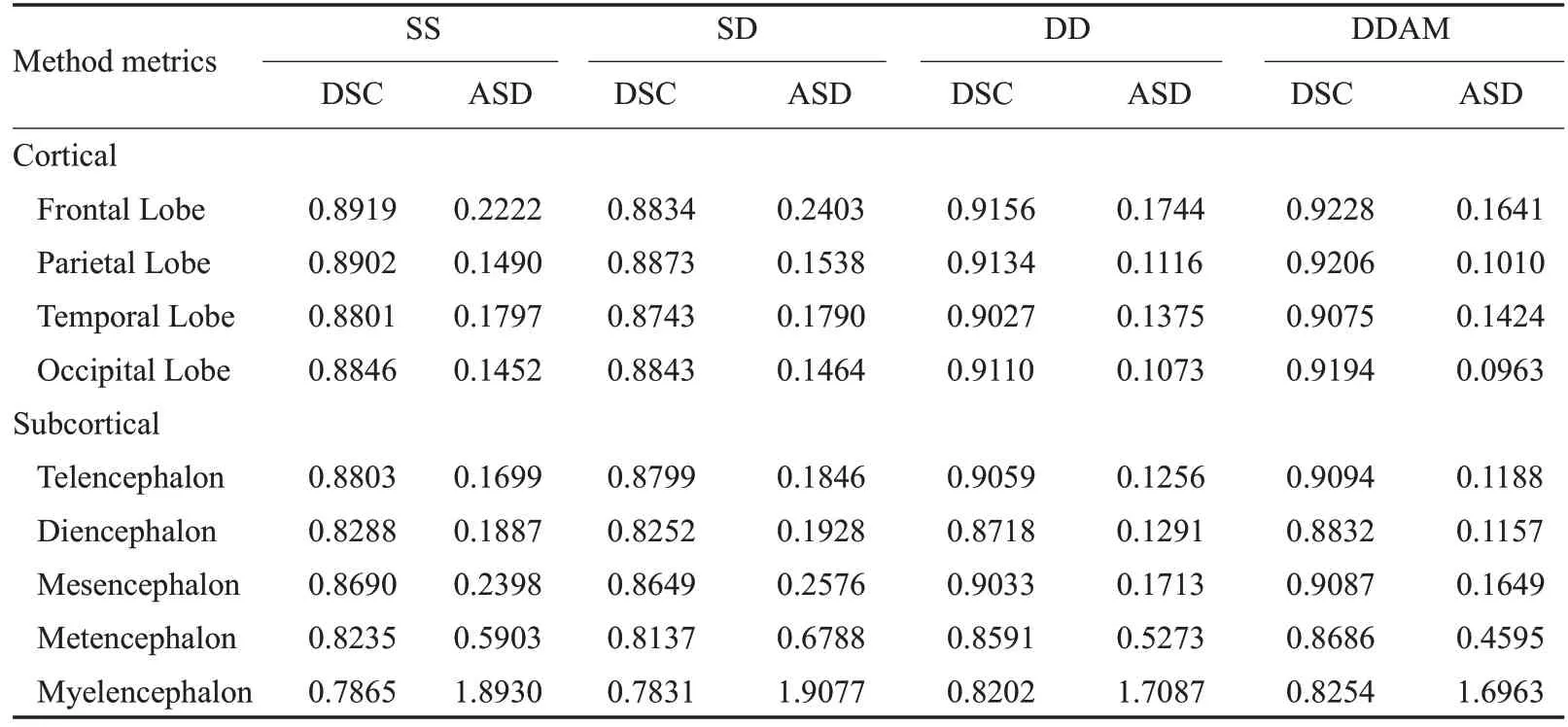
表2 全脑结构化细分结果对比Tab.2 Comparison results of macaque brain fine division
3 讨论
在神经科学研究中利用猕猴作为动物模型具有非常重要的意义,因其在生物学上与人类具有很高的相似性,特别是在大脑结构和认知功能方面。研究猕猴全脑分割为进行灵长类动物实验提供了独特的研究机会,能够揭示其与人类神经生物学潜在适用性之间的信息对比。尽管猕猴动物模型在促进人们对大脑功能的理解方面发挥着关键作用,但致力于猕猴全脑分割的相关工作仍然存在的空缺。同时,准确描绘大脑区域是神经科学研究的基础,缺乏全面的分割研究表明了该领域的一个重大局限性[21]。Wang等[22]提出全局到局部的端对端方法,准确地对低对比度、高噪声的人类婴儿大脑MRI图像进行全脑分割。但由于人类大脑与猕猴大脑在解剖结构、皮层分布、尺寸等方面存在较大的差异,人类大脑的分割方法并不适用于猕猴大脑。目前,关于猕猴的全脑分割方法绝大部分是通过模板进行配准[8,9,23],这存在一定的局限性。首先,对配准模板的精准度和适配性有很高的要求;其次,可能导致较低分辨率的图像在配准过程中丢失解剖学信息的部分内容;此外,配准过程通常需要较长的计算时间,这不利于对大规模图像数据的高效处理。
本次研究基于卷积神经网络开展全脑分割实验。我们从如何更有效地融合多模态特征的角度出发,对传统U-Net结构进行改进,提出嵌入AMFF模块的双编码器网络结构DDAM。其中,AMFF模块为方法的核心部分,由低级特征放大LFA和潜在特征融合LFF组成,前者能够将跳跃连接传入的低级特征的感兴趣区域进行放大;后者则结合空间与通道关系,充分利用模态间的互补性,捕捉融合特征更深层的信息。相比于Roy等[12]提出的结合空间和通道注意力机制的scSE模块,AMFF模块在其基础上进行改进并增加前置特征初步融合,用以筛选多模态特征的浅层信息,为后续更有效地融合深层特征做铺垫。
与仅使用单一模态数据相比,在生物医学研究中利用多模态数据的优势在于,可以为医学图像分析与处理提供更多有帮助的信息。具体而言,每种模态都具备独特的优缺点,将多模态数据集成有助于弥补单模态数据源的局限性[24,25]。然而,处理多模态数据可能会带来一些挑战,例如需要预处理、模态融合以及考虑不同模式之间复杂的关系等。因此,在本次实验中设计了消融实验,包括方法SS、SD以及DD。相较于单一T1-w输入的方法SS,方法SD将T1-w与T2-w进行通道拼接后作为输入,这样虽然增加了T2-w的语义信息,但从皮层区域和皮层下区域的分割结果来看,方法SD的DSC和ASD分数都要略低于方法SS。原因可能是单编码器结构对于提取多模态特征存在一定的困难,具体来说,可能是不同模态提供的特征信息存在错位或不一致,导致网络难以有效学习多模态特征信息;或是T2-w提供了不相关的信息,引入噪声阻碍了网络的学习[10]。此外,将方法DD与方法SD结果进行比较,得出双模态输入的双编码器结构对于双模态输入的单编码器结构,分割效果有着显著提升。分析原因可能是由于多编码器相较于单编码器,可以更有针对性地提取不同模态的特征信息,并且对于浅层信息的捕获也更为充分和高效,从而能有效地提高网络分割精度[25]。方法DDAM的分割精度高于DD,表明在多编码器的基础上,增加注意力机制来融合多模态特征,可以更深层地考虑模态间的复杂关系,使模态间的互补信息得到更充分地利用,并且通过在空间和通道上对多模态特征进行筛选,使得网络更加关注重要特征信息[11,12],从而提升网络性能。综上消融实验结果验证了本文方法的鲁棒性以及网络结构的优越性,可以实现全脑的精确分割。
为了进一步提升全脑分割网络模型的性能,在接下来的工作中,我们将进一步展开以下工作:尝试组合不同的注意力方法,并对解码器部分进行改进,通过增加对融合后特征的处理,使其得到更充分地还原,从而有效提升网络性能;针对婴儿期猕猴开展研究,增强网络对低组织对比度图像的处理能力;尝试加入更多的深度学习方法,如迁移学习、深监督学习等,提高网络的学习能力。
