基于测序分型方法开发日本沼虾微卫星分子标记
2024-01-06王宇希刘新轶
刘 凯,谢 楠,王宇希,刘新轶
(杭州市农业科学研究院水产研究所,浙江 杭州 310024)
日本沼虾(Macrobrachiumnipponense),又称青虾、河虾,是一种重要的经济性甲壳类动物,分类学上隶属于长臂虾科(Palaemonidae)、沼虾属(Macrobrachium)。它们适应性强、分布广、食性杂、生长快、养殖效益高,因此成为中国淡水虾类养殖中的重要资源之一[1-2]。近年来,随着人们对健康饮食需求的不断增加,沼虾的市场需求也在逐渐增长。同时,科学家们也在探索如何更好地利用沼虾的营养和药用价值,为人类带来更多的健康福利。微卫星DNA(Microsatellite)是一种由少量核苷酸(1~6个)为基本单位串联重复形成的一段序列,也被称为短串联重复序列(Short tandem repeat,STR)或简单重复序列 (Simple sequence repeat,SSR)。SSR多态性分析技术已被广泛应用于种群遗传结构分析、种群遗传多样性检测、遗传图谱的构建以及生产性状位点的连锁分析和QTL分析等方面[2-6]。目前有关日本沼虾SSR分子标记开发的报道相对较少。本研究利用测序分型方法开发日本沼虾的SSR分子标记,以期为其种质选育、遗传多样性的检测等提供基础。
1 材料与方法
1.1 基因组测序文库构建
试验所用日本沼虾包括钱塘江群体(QT)、武义群体(WY)、高塘湖群体(GT)、肇庆群体(ZQ)和南宁群体(NN),均于2021年采自国内5个日本沼虾野生区域,分别采集样本6尾进行测序文库构建,取其尾部肌肉后,用无水乙醇保存备用。基因组DNA的提取采用常规的酚氯法[7]。对基因组DNA进行定量后,用Covaris M220超声波仪将DNA剪切成片段,并通过以下步骤纯化以制备测序文库[8]:对DNA片段进行末端过滤、加A,并与测序接头连接,进而进行Illumina测序。最后,构建的文库通过双末端测序过程进行测序。上海派森诺生物科技有限公司完成了测序过程。对原始读数的数据质量控制采用Fastp ver.0.20.1预处理程序[9],使用默认参数。
1.2 多态性SSR序列的筛选
参照本实验室开发的无参考基因组SSR开发方法[10]筛选出含多态性SSR的序列。开发方法简述如下:利用WY_4和NN_5两个样本的测序数据,通过ebwt2InDel软件[11]从这两个样本的测序数据中寻找包含Insertion/Deletion (InDel)标记的序列。基于包含InDel标记的序列,利用SSR筛选软件MISA[12]搜索包含SSR的序列并删除非SSR型InDel标记的序列,得到最终序列。最后基于得到的最终序列,利用Primer3 ver.2.5.0设计SSR引物[13],以满足PCR验证的需要。MISA搜索设置:2个碱基重复6次及6次以上,3~6个碱基重复4次及4次以上。
1.3 SSR的测序分型
利用SSRgenotyper软件[14],对30尾日本沼虾进行了SSR的基因分型,对原程序的部分代码进行了改动,以直接读取MISA搜索的结果,修改后的代码可在https://github.com/zergger/SSRgenotyper地址中获取。参数设置为Q=30、S=1、M=0.1、B=30、m=3,输出格式设置为genepop格式,以自行筛选的含SSR的序列作为参考序列。分型之前,利用软件BWA-MEM ver.1.0.5[15]将fastp过滤后获得的clean reads比对到包含SSR的序列上,使用软件SAMtools ver.1.6[16]删除重复、低质量的clean reads后,获得用于基因分型的SAM文件。基于获得的SAM文件,利用SSRgenotyper软件获得最终的SSR分型结果。采用TouchDown PCR,使用HotStart MasterMix(购自康为世纪生物科技股份有限公司)及5~10个样本对其中3对引物进行了PCR验证。PCR扩增条件:95 ℃预变性5 min;98 ℃10 s,退火5 s,72 ℃15 s,起始退火温度60 ℃,每个循环降低1 ℃,共10个或12个循环;98 ℃10 s,48 ℃或50 ℃退火5 s,72 ℃15 s,共30个循环;72 ℃延伸5 min,4 ℃保存。采用3%琼脂糖凝胶进行常规电泳。
1.4 数据处理
微卫星的等位基因数、观测杂合度、期望杂合度以及 Hardy-Weinberg平衡检验,利用GenAlEx ver.6.51b2进行处理[17]。其中,多态性信息含量(Polymorphic information content,PIC)的计算采用Cervus ver.3.0.7进行处理[18]。Hardy-Weinberg平衡利用PopGene ver.1.32软件[19]进行检验,采用Likelihood ratio方法,同时采用Bonferroni修正进行显著性判别[20]。
2 结果与分析
通过ebwt2InDel软件从WY_4和NN_5两个样本的测序数据中寻找包含InDel标记的序列779条。通过本实验室开发的无参考基因组SSR开发方法从779条含InDel标记的序列中筛选出含多态性SSR的序列137条,去除包含复合型SSR的序列26条,最终得到包含完美型SSR的序列111条。以包含完美型SSR的111条序列为模板,利用SSRgenotyper软件对5个群体共30个样本进行SSR分型。测序分型结果显示,111条含SSR的序列中,共有19个SSR可以成功分型,分型成功率为17.12%。分型成功的19个SSR均为3碱基重复4~6次,其中以ATC/ATG为单位的重复序列占比最多,为6个(31.58%)。利用引物设计软件Primer3针对分型成功的19个SSR序列设计了引物19对(表1)。采用TouchDown PCR对其中3对引物进行了PCR验证。由于Cluster813下游引物的GC含量少,Tm值较低,因此PCR验证中将退火温度降低至48 ℃。由图1可见,3对引物均可扩增,扩增产物大小与预期大小基本一致。
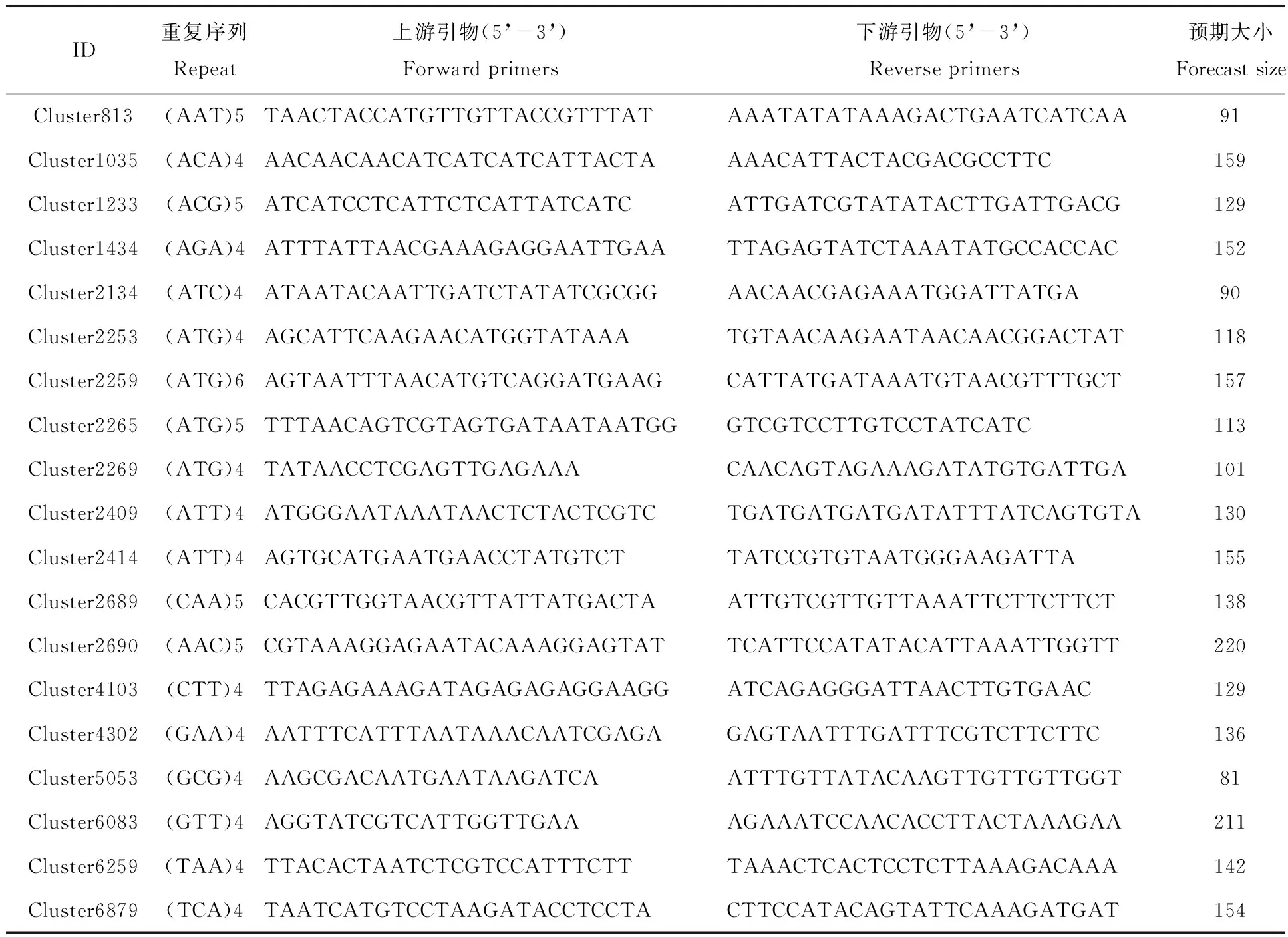
表1 测序分型法获得的日本沼虾SSR多态性位点引物的基本信息Tab.1 Basic information of microsatellite primers of M.nipponense obtained by genotyping by sequencing

有关每个位点的遗传多样性,包括每个基因座的等位基因数(Na)、观察到的杂合性(Ho)、期望杂合度(He)、香侬信息指数(SI)、多态性信息含量(PIC)和Hardy-Weinberg平衡检验的概率(PHW)等分型数据如表2所示。Hardy-Weinberg平衡检验表明,本研究筛选的19个SSR标记中7个偏离了Hardy-Weinberg平衡,计算出的PHW值小于Bonferroni修正后的P值0.002 6。根据Botstein D等[21]基于PIC的分类,19个SSR分为低度信息含量的标记2个、中度信息含量的标记17个。
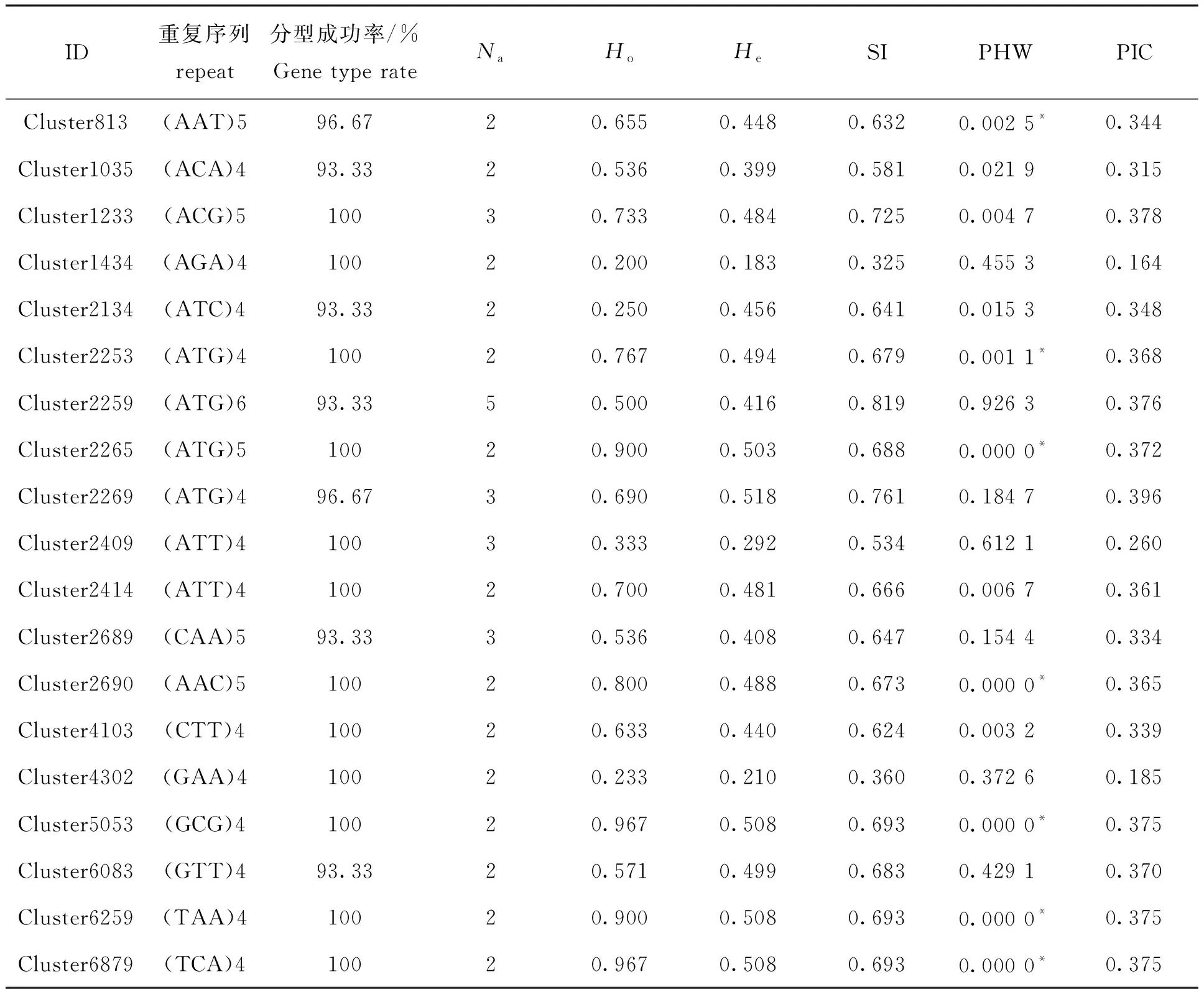
表2 测序分型法获得的日本沼虾SSR多态性位点的特征分析Tab.2 Characteristic of microsatellite primers of M.nipponense obtained by genotyping by sequencing
3 讨论
SSR在许多遗传应用中是首选的标记类型,包括基因组作图、保护遗传学、多倍体亲子鉴定、系统地理学和群体遗传学[22]。作为分子标记,SSR具有广泛的优势,包括高度多态性、共显性遗传以及在各种体外条件下的可重复性[23-24]。然而,传统的基于PCR、凝胶电泳或毛细管电泳的SSR开发方法存在劳动强度大、成本高、耗时长且效率低等问题。随着测序技术的进步和成本的下降,对整个群体进行SSR基因分型测序正在成为相对于传统的基于PCR/电泳方法的一种具有吸引力的替代方案。本研究使用SSRgenotyper从自行开发的共111个微卫星中鉴定出19个多态性SSR位点,基因分型效率超过90%(超过90%的个体都获得了分型结果)。
为验证SSR位点的多态性,本研究从5个地理群体中选择了30个样本,以确保验证样本的多样性;设计了19对引物来扩增筛选出19个多态性SSR位点,并对其中的3对引物进行了PCR和电泳分析。电泳结果验证了基于测序方法进行基因分型的准确性,凝胶电泳得到的扩增产物大小与预期大小基本一致。实际上,基于测序的SSR基因分型方法早已有之。在鱼类验证中,基于测序的基因分型方法的准确率超过90%[10]。Lewis D H等[14]将其应用在植物上,验证结果显示二核苷酸和四核苷酸SSR的不一致性最高(分别为5.7%和5.0%),重复次数超过15次的SSR最不一致(20.3%),但它们仅占总SSR数的不到0.4%。三核苷酸SSR是最常见的类型,占所有基因分型SSR位点的72.9%。Han J等[25]进行了基因组测序分型中SNP和SSR的比较研究,结果表明在群体遗传分析中,这两种方法具有高度一致性。此外,基于SSR的遗传分析还揭示了一些与SNP分析不同的结果[25]。
SSR核心序列的突变率相对较高(10-5~10-3),这会导致SSR长度的变化,从而产生SSR多态性[26]。SSR寡核苷酸的重复数在同一物种的不同基因型间差别很大,一般很少用于多态性分析。根据Weber J L[27]的研究,只有当双碱基重复序列的重复次数大于10次时,SSR标记才有可能表现出较高的PIC值。当重复次数大于16时,可提供的PIC值在0.5以上。这也是本实验中设置MISA搜索条件的依据:2个碱基重复6次及6次以上,3~6个碱基重复4次及4次以上。基于无参考基因组SSR方法筛选并经测序分型验证后获得的SSR标记都是三碱基重复,其重复次数都在4~6次之间,没有检测到具有多态性的双碱基重复,这可能与分型方法有关,也可能与样本或物种有关。而Lewis D H等[14]在植物上验证的结果也表明,三核苷酸SSR是最常见的类型。此外,在日本沼虾中,使用无参考基因组SSR筛选方法获得的多态性SSR标记的有效率较低,这也可能与物种有关。在三角鲂(Megalobramaterminalis)中,采用无参考基因组SSR筛选方法筛选的SSR标记中,多态性标记的比例可以达到90%以上[10]。将验证多态性的样本限定到WY和NN两个群体上,筛选出的多态性SSR标记数量可以提升到27个(文中未列出)。这表明作为参考的包含SSR的序列保守性不强造成比对率低,或者是由日本沼虾样本的序列多样性较高造成比对率低。
一些研究表明,重复次数较少的SSR标记往往难以检测出多态性。Valdes A M等[28]的研究结果显示,人类(Homosapiens)中重复次数低于5的SSR,几乎检测不出多态性。而Smulders M J M等[29]则认为,重复次数多的SSR既能在种间又能在种内产生多态性,但重复次数少的SSR仅能在种间产生多态性。Xu Z等[30]对斑节对虾(Penaeusmonodon)进行的研究也表明,核心序列重复次数较少的SSR标记往往是单态性的,或等位基因数目非常少,PIC值也偏低。因此,传统实验中设计SSR标记往往需要仔细选择重复次数较多的核心序列,以提高SSR标记的多态性和可靠性。但随着测序技术的发展,SSR基因分型测序正在成为相对于传统的基于PCR/电泳方法的一种具有吸引力的替代方案。由于SSR基因分型测序的高分辨率,即使传统实验中难以检测到的低重复次数SSR标记,其多态性也可检测到。本实验所获得的SSR序列重复数大部分在4~5次,但使用SSR基因分型测序依然可以检测到多态性,PIC值大多达到了中度信息含量,这对遗传多样性分析也是有利的。
