miRNAs-MUC13调控体系中与仔猪腹泻有关的基因与通路
2024-01-02路伏增华卫东褚晓红戴丽荷陈晓宇章利丰王志刚徐如海
路伏增,华卫东,褚晓红,戴丽荷,陈晓宇,章利丰,王志刚,徐如海
(浙江省农业科学院 畜牧兽医研究所,浙江 杭州 310021)
仔猪腹泻不仅会降低仔猪的成活率,还会降低仔猪对疾病的抵抗力,从而降低生产效益,因此,提高仔猪对腹泻的抗病性能是猪育种的目标之一[1]。编码黏蛋白13(Mucin 13)的MUC13基因,是决定仔猪ETEC F4ac腹泻易感/抗性的主效基因之一,种猪选育研究进一步验证了MUC13基因的抗性纯合基因型能有效降低断奶前仔猪腹泻的发生率[2-3]。microRNA(miRNA)在仔猪腹泻调控中起着不可缺少的微调作用,越来越多的证据表明,miRNA异常表达参与仔猪腹泻发病调控[4-5]。仔猪腹泻受到众多基因调控,目前研究大多关注于个别miRNA对mRNA的调节作用,大多数miRNA与mRNA之间的相互作用关系仍不清楚,围绕miRNAs-MUC13调控体系构建仔猪腹泻基因调控网络的系统研究还未见报道。因此,本研究围绕miRNAs-MUC13调控体系构建仔猪腹泻基因互作网络,对网络中有关基因与通路进行分析发掘,并以大肠埃希菌菌株200和仔猪肠道上皮细胞系(IPEC-1)作为试验材料,通过设计体外细胞试验进行验证,研究miRNAs-MUC13调控体系中与仔猪腹泻相关的分子调控机制,以期为靶向治疗仔猪腹泻提供理论参考和科学依据。
1 材料与方法
1.1 miRNA预测和靶基因筛选
用NCBI的blastn (http://blast.ncbi.nlm.nih.gov/Blast.cgi)工具比对小鼠MUC13基因(Musmusculus,NM_010739.2)和猪(Susscrofa)基因库中的核酸序列。采用miRDB数据库(http://www.mirdb.org/)、mirTarBase数据库(https://mirtarbase.cuhk.edu.cn/)、TargetScan数据库(https://www.targetscan.org/mmu_80/)预测调控MUC13基因的有关miRNA。由于数据库“物种”选项没有“猪”选项,基于核酸序列的同源性,选择小鼠为模型进行预测。然后选择starBase数据库(http://starbase.sysu.edu.cn/clipSeq.php)预测这些miRNA的靶基因,用starBase数据库整合多个数据库的结果,并将不同数据库预测的miRNA靶基因交集作进一步分析。
1.2 基因互作网络构建
将MUC13基因和筛选的miRNA靶基因名称提交到软件Cytoscape 2.7.0的插件Agilent Literature Search 2.7.1(美国Agilent Technologies公司)中进行文本挖掘,经过检索,去除假阳性相互作用信息后,将确定的基因相互作用关系读入Cytoscape 2.7.0软件中,构建基因相互作用网络图谱[6]。
1.3 生物信息学分析
由于网络非常庞大,本研究用Cytoscape 2.7.0软件的插件Clusterviz 1.2中的MCOMD算法,通过关联积分来评估该网络中存在的分子复合物集团,分子复合物中的基因一般具有相同的生物学功能,可以发现未知基因的功能或新的分子功能集团[7]。根据关联积分值,可获得整个网络中可能形成的分子复合物集团,并在Cytoscape中显示;采用David分析平台对分值比较高的分子复合物进行通路分析,从而获得一些未经研究或研究较少的与仔猪腹泻相关的调控机制[8]。
1.4 仔猪腹泻模型验证
通过体外细胞试验设计仔猪腹泻模型进行验证。以IPEC-1作为模型,用大肠埃希菌200(F4ac,C83907,O149:K91,猪源,来自中国兽医药品监察所)作为该细胞的黏附材料。IPEC-1培养条件为杜氏改良Eagle培养基/营养混合物F-12培养基(Dulbecco’s Modified Eagle Medium/Nutrient Mixture F-12, DMEM/F-12)中添加10%胎牛血清(FBS)、1%青霉素和链霉素双抗(P/S)和1%谷氨酰胺(glutamine),放置在含体积分数5% CO2的二氧化碳培养箱中培养,培养温度37 ℃,共传代25~30代。
黏附试验:分为对照组和试验组,对照组不做任何处理,试验组用大肠埃希菌200黏附到IPEC-1引起炎症反应。IPEC-1常规培养,试验之前将细胞接至6孔板培养,已长至单层(加盖玻片)的细胞用灭菌磷酸缓冲盐溶液(PBS,pH值7.4)漂洗2次,每孔中加入1 mL大肠埃希菌200菌悬液(细菌浓度为1×108~2×108mL-1)和l mL新鲜的DMEM基础液,于37 ℃、含体积分数5% CO2的二氧化碳培养箱中孵育1 h,漂洗细胞5次,以除去未黏附的细菌,用甲醇固定,用革兰氏染色,显微镜下观察大肠埃希菌黏附到IPEC-1的结果。
1.5 实时荧光定量PCR(qRT-PCR)检测
采用miRNeasy Mini Kit (QIAGEN)试剂盒提取1.4节对照组和试验组IPEC-1的总RNA,反转录成cDNA。每组5个样本重复数,-20 ℃保存。
根据GenBank中猪的MUC13、SRF、IL-6、MAPK、STAG2、NF-κB、TLR7和GAPDH基因序列设计引物,目的基因引物序列见表1。采用Applied Biosystems 7500 Real-Time PCR System进行检测,总反应体系为20 μL,包括9.2 μL Platinum SYBR Green qPCR SuperMix-UDG (Invitrogen), 8.7 μL ddH2O, 0.5 μL diluted cDNA,上、下游引物各0.8 μL。反应条件:95 ℃ 2 min;95 ℃ 15 s,60 ℃ 30 s,40个循环。

表1 目的基因引物序列Table 1 Primer sequence of target gene
1.6 数据分析
qRT-PCR检测结果采用相对定量2-ΔΔCT方法计算,用SPSS16.0软件进行数据统计分析,结果以平均值±标准误表示,P<0.05表明各组差异显著。
2 结果与分析
2.1 MUC13基因的miRNA和靶基因
在NCBI中用blastn比对小鼠MUC13基因和猪基因库中的核酸序列,两者的相似性在74%~87%,同源性较高。基于同源性分析,本研究以物种“小鼠”为对象,利用miRDB、mirTarBase、TargetScan在线数据库进行MUC13基因的miRNA预测,取得交集后得到2个保守性高的miRNA(miR-466d-5p和miR-466k),碱基序列分别为UGUGUGUGCGUACAUGUACAUG和UGUG-UGUGUACAUGUACAUGUGA。选择starBase数据库预测这2个miRNA的靶基因,并取其交集,共得到36个基因交集,基因集合如表2所示。
表2 MUC13基因的miRNA靶基因交集Table 2 miRNA target gene intersection of MUC13 gene
2.2 miRNAs-MUC13调控体系有关的仔猪腹泻基因互作网络
运用Cytoscape软件中的Agllent Literature search插件,在Terms对话框中输入MUC13基因与上述获得的36个靶基因名称,在Context对话框中输入检索的关键词“piglet diarrhoea”,经过文本挖掘,得到围绕仔猪腹泻建立起来的基因互作网络图谱(图1),其中输入的基因中有19个基因处于同一结构复杂的多中心基因互作网络中,该网络包含264个节点、441条边(相互作用关系)。
2.3 基因互作网络图谱中的分子复合物集团
由于仔猪腹泻基因互作网络非常庞大,本研究用Cytoscape 2.7.0软件的插件Clusterviz 1.2中的MCOMD算法,通过关联积分来评估该网络中存在的分子复合物集团,对构建的生物学网络中的区域进行关联度分析,结果显示,关联积分值不小于3的分子复合物共4个,其中分子复合物1的分值为3.219,其他的分子复合物分值均为3。这4个分子复合物总共包含5个输入的靶基因,分子复合物1包含SRF、STAG2基因,分子复合物2包含PRKACB基因,分子复合物3包含RTN1基因,分子复合物4包含IER5基因(图2)。
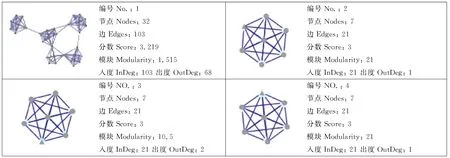
图2 通过基因互作网络图谱关联度获得的分子复合物集团Fig.2 The molecular complex groups obtained through the correlation analysis of gene interaction network map
2.4 分子复合物集团的基因和通路富集分析
通过DAVID平台分析上述获得的4个分子复合物集团中的每个基因集合,得到所属通路,在这些通路里共包含2个输入的靶基因,分别是分子复合物集团1里的SRF和STAG2,其中SRF参与MAPK和IL 6信号通路,STAG2参与Toll-like receptor信号通路(表3)。
2.5 仔猪腹泻模型验证结果
常规培育的IPEC-1细胞形态正常,如图3-A所示;大肠埃希菌200黏附的IPEC-1如图3-B所示,IPEC-1已经感染大肠埃希菌200并引起炎症反应,表明通过体外细胞试验设计的此仔猪腹泻模型可用于下一步有关基因的差异表达验证。
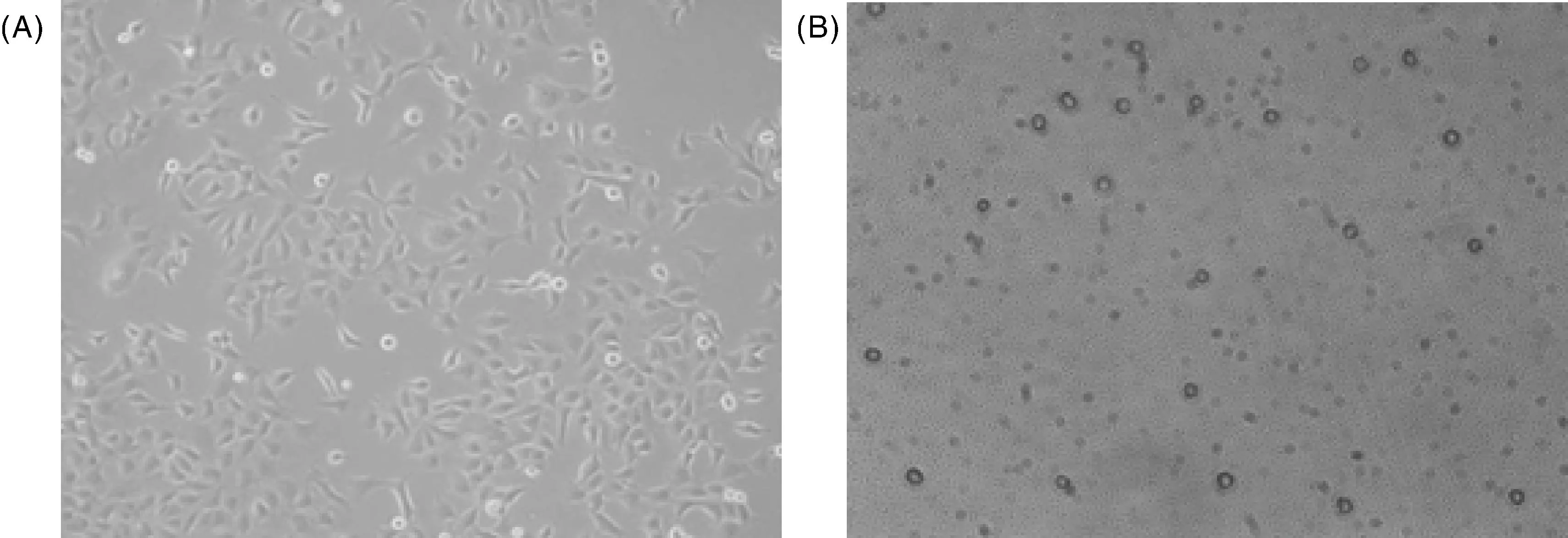
A,对照组,常规培育的仔猪肠上皮细胞系(IPEC-1);B,试验组,大肠埃希菌200黏附的IPEC-1。A, Control group, conventionally cultured IPEC-1; B, Test group, IPEC-1 adhered with Escherichia coli 200.图3 大肠埃希菌黏附仔猪肠上皮细胞IPEC-1结果Fig.3 Result of Escherichia coli adhered to IPEC-1
2.6 qRT-PCR检测结果
qRT-PCR检测结果(图4)显示:大肠埃希菌200黏附的IPEC-1中MUC13、SRF、IL-6、MAPK、STAG2、NF-κB、TLR7基因的相对表达水平均显著高于常规培育的IPEC-1。SRF参与MAPK和IL6信号通路,STAG2参与Toll-like receptor 信号通路,推测与SRF相关的基因是MAPK和IL6,与STAG2相关的基因是TLR7和NF-κB。
3 讨论
MUC13基因作为仔猪腹泻的候选基因越来越受到重视,对MUC13基因进行研究非常具有商业价值,其中调控该基因的miRNA在其中起着不可缺少的微调作用[4-5]。miRNA广泛存在于真核生物体内,在不同物种间具有高度的保守性,miRNA的序列、数量和功能在生物体内的诸多方面均有关键调节作用[9]。采用生物信息学方法可预测任何一个物种的miRNA,最初猪miRNA的发现均来自同源搜索[10]。2008年,Kim等[11]鉴定了58种新的猪miRNA,同源分析发现,其中23个miRNA与小鼠同源,21个miRNA与人同源,16个miRNA与人、鼠均同源。截至2023年1月26日,以Swine和miRNA组合为关键词在NCBI数据库PUBMED中检索,可获得1 084篇已经发表的文章,其中2019-2023年有523篇(占比48.2%);2009年miRBase数据库中收录猪miRNA的数量达77个,2013年增长到339个,2023年达到457个,这些数据表明,围绕猪miRNA的研究近年来处于一个相对快速发展的初期阶段。目前,关于调控靶基因的miRNA预测,大多采用2种或2种以上在线数据库进行预测,然后求交集,目的是提高准确率、减少单个数据库预测的假阳性。鉴于目前有关预测数据库并未注释到猪这一种属,根据其成熟体序列的保守性,一般是搜索同源性较高的其他物种(例如“人”和“小鼠”)的数据库进行预测。王伟等[12]选择人的miRNA数据库(TargetScan、miRDB、PicTar)预测猪miR-204的靶基因;朱静静等[13]通过TargetScan在线预测和分析靶向猪内质网应激通路中关键基因的miRNA;楚丹等[14]利用miRDB、miRWalk、Target Scan、ENCORI 4种在线软件预测可能与猪miR-139结合的靶基因;张名媛等[15]利用miRDB、miRWalk 2.0和TargetScan 7.2数据库预测广西巴马小型猪miR-148b的靶基因:这些有关“猪”的研究均是建立在其他物种基础上的。本研究分析发现,小鼠和猪的MUC13基因核酸序列同源性较高,为74%~87%,这表明利用小鼠物种的预测数据库进行猪该基因miRNA的预测是可行的。因此,本研究利用miRDB、mirTarBase、TargetScan在线数据库,以“小鼠”为对象进行了MUC13基因的miRNA预测,取得交集后得到了保守性高的miRNAs(miR-466d-5p和miR-466k),基于StarBase数据库预测这2个miRNA的靶基因并获取二者的交集,得到36个基因。为在物种“猪”上得到证实,运用Cytoscape软件基于关键词“piglet diarrhoea”构建仔猪腹泻基因互作网络,选择物种“Susscrofa”通过DAVID平台分析基因和通路富集分析;最后以IPEC-1作为模型,采用猪的有关基因序列设计引物进行体外细胞试验验证,验证结果进一步证明本研究的分析方法是合理的。
猪的疾病发生受到众多基因调控,目前围绕miRNAs-MUC13调控体系构建仔猪腹泻基因调控网络的系统探索还未见报道,至于其具体机制、信号通路尚不清楚。本研究基于miRNAs-MUC13调控体系,构建了仔猪腹泻基因互作网络,该网络包含有19个筛选到的靶基因,通过关联度分析获得4个分子复合物集团,包含5个输入的靶基因,分别是分子复合物1(靶基因SRF[16-18]、STAG2[19-20)、分子复合物2(靶基因PRKACB[21])、分子复合物3(靶基因RTN1[22])、分子复合物4(靶基因IER5[23]),这5个靶基因与腹泻有关的调控通路研究可参考相应的文献。分子复合物中的基因一般具有相同的生物学功能,可以发现未知基因的功能或新的分子功能集团[6-7]。据文献报道,这些基因与MUC13[24]基因功能涉及细胞凋亡、增殖失控、分化异常,免疫功能失调,细胞因子和黏附分子信号通路,以及与凋亡有关的通路、炎症介质的释放、炎症反应等。
SRF是高度保守的磷酸化DNA结合蛋白,是重要的细胞转录调控因子[16],也是上皮细胞中血管内皮生长因子(VEGF)通路和VEGF诱导的血管生成的关键因子,其中VEGF促进SRF表达与核转位,并增加SRF通过Rho-actin、MEK-ERK通路与DNA结合的活性[17]。SRF还是许多途径的下游靶基因,例如可通过与三元复合因子(TCFs)作用,参与胞外信号调节激酶途径(MAPK途径)[18]。SRF还与胃肠疾病有着密切的关系,目前有报道认为,在兔的胃溃疡中注射SRF反义表达的质粒可以抑制体内肉芽组织的血管发生[16]。幽门螺杆菌(HP)感染导致严重的肠胃炎症,HP诱导的胃肠上皮化生与ERK-1和SRF诱导的villin密切相关[16,18]。STAG2可编码生成cohesin复合物的一种亚基[19-20]。STAG2能够激活TLR7的活性,经一系列信号传导分子最终促进NF-κB的激活,NF-κB的激活可导致炎症介质如IL-6、IL-12的大量释放[25-26]。有研究表明,溃疡性结肠炎患者核内NF-κB的DNA结合活性明显升高,NF-κB被活化并进入核内,具有较强的与基因κB位点相结合的能力,是细胞因子转录与释放的前提和关键[27-28]。从信号传导通路的角度看,天然免疫的TLRs、NF-κB信号通路,细胞因子和黏附分子信号通路,以及与凋亡有关的通路在仔猪腹泻症患者中均可表现紊乱[29-30]。本研究分析显示,SRF主要涉及MAPK、IL6等信号通路。体外细胞试验验证显示,试验组SRF、MAPK、IL-6等基因表达量均显著高于对照组,推断SRF通过MAPK和IL6信号通路参与调控仔猪腹泻。另外,试验组STAG2、NF-κB、TLR7等基因表达量均显著高于对照组,推断STAG2通过Toll-like receptor信号通路参与调控仔猪腹泻。
综上所述,本研究通过对miRNAs-MUC13调控体系中与仔猪腹泻有关的基因与通路进行发掘与验证,发现了4个分子复合物集团和2个核心基因(SRF和STAG2),这为靶向治疗仔猪腹泻提供了理论参考和科学依据。
