菜心响应高温胁迫的转录组分析与基因挖掘
2024-01-01江定李光光袁凡崇雷世康张华戴修纯郑岩松





摘要:【目的】探索菜心响应高温胁迫的转录组分析及基因挖掘,为筛选响应高温胁迫的菜心耐热基因及耐热菜 心分子育种提供参考依据。【方法】以菜心耐热品种四九-19号和热敏品种特青迟心4号2个耐热性差异明显的菜心品 种为试验材料,经高温(42℃)胁迫处理后,对植株地上部和地下部共24个样本基因进行转录组测序,筛选出差异表 达基因(DEGs),利用COG、GO、KEGG、KOG、Pfam、Swiss-Prot、eggNOG和Nr生物数据库中对DEGs进行功能注释,筛 选响应高温胁迫的热激转录因子和热激蛋白,同时利用实时荧光定量PCR验证重要基因表达情况,并分析其启动子 区顺式作用元件。【结果】通过RNA-Seq在24个样本中共获得207.18 Gb的Clean bases和47462个DEGs;8个功能注释 数据库均得到注释的仅2663个DEGs,Nr数据库中得到注释的DEGs最多,为47324个,占比99.71%;4个样本比较组中剔除重复DEGs,获得15761个DEGs。GO功能注释结果显示,DEGs富集到生物过程、细胞组分和分子功能三大类;KEGG信号通路富集结果表明,植物激素信号转导、碳代谢和氨基酸的生物合成信号通路最为富集。在|log2Fold Change|≥8且Plt;0.001的高DEGs筛选标准下,4个比较组有141个DEGs共表达,38个基因在四九-19号菜心特有表达,54 个基因在特青迟心4号菜心特有表达;采用Nr、Pfam和Swiss-Prot等数据库对2个菜心品种共有表达和特有表达的共233个DEGs进行功能注释分析,获得响应高温胁迫9个热激转录因子基因和25个热激蛋白基因;最终筛选出10个与响 应高温胁迫高度相关的DEGs;10个DEGs的实时荧光定量PCR验证结果显示,基因表达趋势与RNA-Seq数据结果一致;通过启动子区顺式作用元件分析结果显示,10个DEGs存在多种响应环境和激素信号的顺式作用元件。【结论】筛 选获得10个与响应高温诱导相关且显著上调的重要候选基因,其启动子区域含有多种响应环境和激素信号的顺式作 用元件。
关键词:菜心;高温胁迫;转录组测序;差异表达基因(DEGs);基因挖掘
中图分类号:S634.5
文章编号:2095-1191(2024)03-0766-18
文献标志码:A
Transcriptome analysis and gene mining of flowering Chinese cabbage in response to high temperature stress
JIANG Ding, LI Guang-guang, YUAN Fan-chong, LEI Shi-kang, ZHANG Hua,
DAI Xiu-chun, ZHENG Yan-song*
(Guangzhou Academy of Agricultural Sciences, Guangzhou, Guangdong 510335, China)
Abstract:【Objective】The purpose of the study was to explore the transcriptome analysis and gene mining of flowe- ring Chinese cabbage in response to high temperature stress, and to provide a reference for screening heat-resistant genes of flowering Chinese cabbage in response to high temperature stress and molecular breeding of heat-resistant flowering Chinese cabbage. 【Method】Heat-resistant variety Sijiu-19 flowering Chinese cabbage and heat-sensitive variety Teqing- chixin No.4 flowering Chinese cabbage, two greatly different heat-resistant varieties of flowering Chinese cabbage, were used as experimental materials. After high temperature (42 ℃) stress treatment, transcriptome sequencing (RNA- Seq) technology was 766 conducted on genes of 24 samples in the shoots and roots of the plants. The differential expres- sion genes(DEGs)were screened, and functional annotation to DEGs was performed using COG, GO, KEGG, KOG, Pfam, Swiss-Prot, eggNOG and Nr biological databases, in order to screen heat shock transcription factors and heatshock proteins in respond to high temperature stress. At the same time, real-time fluorescence quantitative PCR was used to verify important genes of expression and to analyze the cis acting elements of the promoter region. 【Result]A total of 207.18 Gb of clean bases and 47462 DEGs were obtained from 24 samples using RNA-Seq. Only 2663 DEGs were anno- tated in all 8 functional annotation databases, while the Nr database had the highest number of annotated genes, which were 47324 DEGs, accounting for 99.71% of all genes. Employing eliminating duplicate DEGs from four comparison groups, 15761 DEGs were obtained. The GO functional annotation results showed that DEGs were enriched into three ma- jor categories: biological processes, cellular components, and molecular functions. The KEGG signaling pathway enrich- ment results indicated that the plant hormone signal transduction, carbon metabolism and biosynthesis of amino acids sig- naling pathways were the most enriched. Under high screening criteria for DEGs with |log2 Fold Change| 28 and Plt;0.001, a total of 141 DEGs were shared expression in the four comparison groups; 38 genes were specific expression in the Sijiu- 19 flowering Chinese cabbage; and 54 genes were specific expression in the Teqing-chixin No.4 flowering Chinese cab- bage. Using databases such as Nr, Pfam and Swiss-Prot, functional annotation analysis was performed on a total of 233 DEGs shared expression and specific expression in two flowering Chinese cabbage varieties, which obtained 9 heat shock transcription factors genes and 25 heat shock proteins genes responsive to high temperature stress. Finally, 10 DEGs highly correlated with response to high temperature stress were selected. The validation results on real-time fluorescence quantitative PCR analysis of 10 DEGs showed that the gene expression trend was consistent with the RNA-Seq data re- sults. The analysis of cis acting elements in the promoter region showed 10 DEGs had multiple cis acting elements that re- sponded to environmental and hormone signals. 【Conclusion】 Ten important candidate genes related to response to high temperature induction and significantly up-regulated are screened, and they contain multiple cis acting elements that re- spond to environmental and hormone signals in the promoter regions.
Key words: flowering Chinese cabbage; high temperature stress; transcriptome sequencing; differential expression genes(DEGs); gene mining
Foundation items: Guangdong Science and Technology Plan Project (2022B0202080001); Guangzhou Science and Technology Plan Project(202201010854)
0 引言
【研究意义】菜心(Brassica rapa var. parachinen- sis)是十字花科(Brassicaceae)芸薹属(Brassica)白菜 中易抽薹类型的一个变种,是华南地区特色蔬菜品 种,主要食用部位是花薹与嫩叶,其品质脆嫩甘甜, 营养丰富,深受华南地区消费者喜爱(张华和刘自珠, 2010)。菜心生长周期较短,一年可种植多茬,实现 周年生产供应(张华和刘自珠,2010;陈汉才等, 2021)。菜心最适生长温度为20~30℃,超过30°℃ 会减缓生长速度,降低产量及风味品质(曹毅等, 2010;李光光等,2012a)。华南地区地处热带与亚热 带季风气候区,夏季气候炎热,田间温度可高达 40℃以上,且雨水多、病虫害发生严重,导致菜心种 植产量及质量不高。2006年以来夏季菜心生产种植 逐步转移至我国北方和西南夏菜种植区(陈汉才等, 2021),随着全球气候变暖,厄尔尼诺现象加剧,导致 夏季这些地区菜心种植难度逐渐加大。目前耐热菜 心品种较少,仅有四九-19号、碧绿粗薹菜心等品质 一般的菜心品种(张衍荣,1997;陈汉才等,2010)。 因此进行菜心响应高温胁迫的转录组分析及基因挖 掘,对筛选响应高温胁迫的菜心耐热基因及耐热菜 心分子育种具有重要意义。【前人研究进展】随着全 球年平均气温逐渐升高,植物耐热性研究已成为国 内外研究热点之一,持续高温不仅会对植物外部造 成明显损伤,还会损害叶绿体结构、破坏光合系统、诱导细胞内活性氧积累、损伤细胞膜系统、引起代 谢功能和生理过程发生紊乱等,从而影响植物正 常生长甚至导致其枯萎死亡(Li et al.,2018;Zhao et al.,2020;周娜等,2022)。暴露在高温下的植物通 过迅速感知、传递热信号,诱导热响应基因如转录因 子、蛋白激酶、热激蛋白等基因的表达调节细胞及代 谢稳态,进而抵抗高温胁迫(Mittler et al.,2012;王坤 等,2023)。目前有关菜心高温胁迫的研究主要集中 在高温胁迫对菜心生长发育、农艺性状、生理指标的 影响及相应耐热性鉴定指标筛选(李光光等,2012b; 陈连珠等,2020;刘畅,2020;庞强强等,2020a, 2020b),从分子水平解析菜心高温响应机理的研究 报道较少(郭培国等,2015;Ahmed ct al.,2021;庞强强等,2023)。转录组测序(RNA-Seq)技术可将mRNA从总RNA反转录成cDNA,从而快速、全面地 获取特定样本中几乎所有cDNA的序列信息和含 量,可用于研究基因组功能元件的差异表达、转录分 类和应激条件等(彭国颖等,2022)。RNA-Seq技术 因为测序快速、准确、大量数据可用等优点,已成为 研究生物基因转录表达的重要工具(韦丽君等, 2020)。目前利用RNA-Seq技术研究菜心耐热方面 的研究较少,陈静芳(2018)建立了转录组数据库,并 开发出菜心EST-SSR标记。而利用RNA-Seq解析 其他农作物耐热基因的研究报道较多,徐海(2019) 利用RNA-Seq鉴定出6个与不结球白菜耐热相关的基因;王少波(2019)从水稻RNA-Seq数据中挖掘到 3个受热胁迫诱导表达的重要耐热基因;Wang等 (2019)通过辣椒RNA-Seq分析发现,谷胱甘肽代谢 途径在耐热品种幼苗响应高温胁迫中发挥关键作 用,推测其相关基因表达和代谢物积累是辣椒耐热 品种具有较高耐热性的主要原因之一。陈雪倩 (2020)结合RNA-Seq数据与生物信息学方法,对黄瓜中的Hsf基因进行分析,最终筛选鉴定出CsHsf-7 可能是黄瓜热激反应中的重要调节因子;蒋玲玉 (2020)利用RNA-Seq鉴定出167个可能与玉米响应高温胁迫有关的转录因子;Zhang等(2020)通过茄子RNA-Seq分析发现,高温胁迫下大量与抗氧化酶 系统、植物激素和转录因子相关的基因差异表达。 王辉等(2023)对高温胁迫下陆地棉转录组差异性分 析,鉴定出39个转录因子,主要是WRKY和MYB两大家族,鉴定出Gohir:A08G104100是响应高温胁迫的 主要基因。【本研究切入点】目前,有关白菜、辣椒、番 茄、黄瓜等大宗蔬菜的耐热研究已较为深入,而有关 高温胁迫下菜心的转录组分析及基因挖掘的研究鲜 见报道。【拟解决的关键问题】本研究以耐热性差异 明显的耐热四九-19号菜心和热敏特青迟心4号菜 心为试验材料(李光光等,2012b),利用RNA-Seg技 术对2个菜心品种的地上部分和地下部分高温胁迫处理前后的共24个样本进行分析,筛选出差异表达基因(Differentially expressed genes,DEGs),对DEGs进行功能注释分析,从中筛选出与菜心响应高 温胁迫相关的重要候选基因,为今后开展菜心耐热 分子育种研究提供理论基础。
1材料与方法
1.1试验材料及样本处理
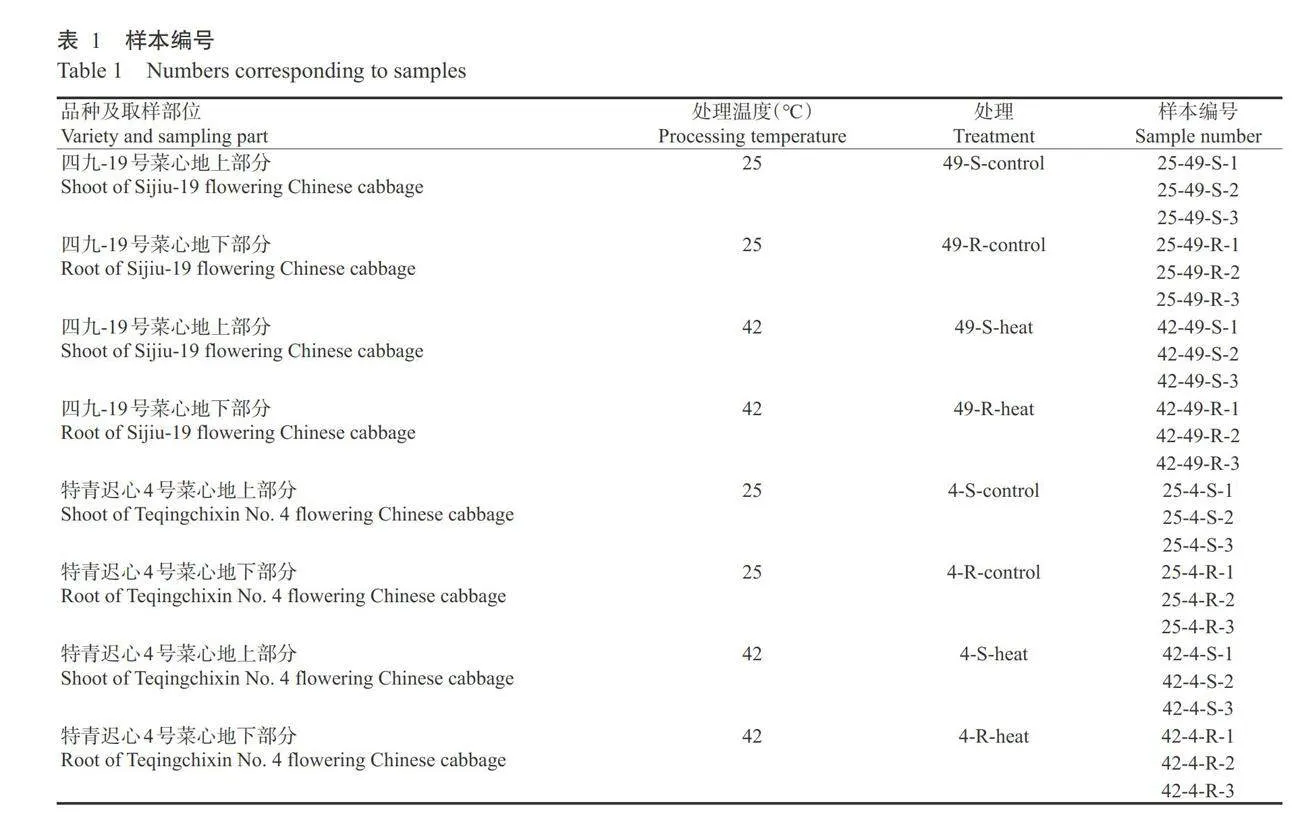
供试菜心品种为耐热四九-19号和热敏特青迟 心4号(广州市农业科学研究院选育),2019年8月 种植于广州市农业科学研究院重点实验室光照 培养室,昼、夜温度为25和18°℃,湿度为70%,黑暗 12h/光照12h,光照强度为8000lx,待幼苗生长到 5~6片真叶时,于人工气候培养箱(湿度70%,光照 强度8000lx)中42℃高温条件下处理6h,以25℃ 条件下培养6h为对照,取各品种的地上部分和地下 部分,每份样本设3个生物学重复,共24个样本(表1), 液氮速冻,-80℃保存备用。主要试剂:植物总RNA 快速抽提试剂盒购自生工生物工程(上海)股份有限 公司;HiSeript ⅢⅢ 1st Strand cDNA Synthesis Kit (+gDNA wiper)和 ChamQ Universal SYBR qPCR Master Mix(Q711-02)试剂盒均购自南京诺唯赞生物科技股份有限公司。主要仪器设备:CFX384C1000Touch实时荧光定量PCR仪(美国Bio-Rad公司)。
1.2 RNA-Seq与数据分析
参照植物总RNA快速抽提试剂盒说明书提取 总RNA,委托北京百迈客生物科技有限公司进行RNA-Seq,测序数据由测序平台进行转化处理,将长度小于50bp、低质量的Reads(序列质量在Q20以下)删除,将获取的有效序列(Clean reads)与参考基因组(Brassica rapa v3.0)进行序列比对,分析测序结果,并对基因进行功能注释,计算转录本的FPKM值。
1.3DEGs分析
利用DESeq2软件进行24个样本组间的DEGs 分析,以|log2Fold Change|≥2且Plt;0.01作为筛选DEGs 标准;将筛选出的DEGs在Nr、GO、KEGG、Pfam、 eggNOG、Swiss-Prot、COG和KOG数据库中进行比 对及功能注释;运用R语言ggplots2对4个比较组(49-S-heat vs 49-S-control、49-R-heat vs 49-R-control、 4-S-heat vs 4-S-control和4-R-heat vs 4-R-control)的 DEGs进行火山图分析。提高DEGs的筛选阈值为 llog2Fold Change|≥8且Plt;0.001,将4个比较组(49- S-heat vs 49-S-control、 49-R-heat vs 49-R-control、 4-S-heat vs 4-S-control和4-R-heat vs 4-R-control)进行韦恩图分析,从RNA测序数据中提取响应高温胁迫相关基因的FPKM值作表达热图分析,筛选响应高 温胁迫相关候选基因。
1.4DEGs的实时荧光定量PCR验证
为验证菜心RNA-Seq数据的可靠性,选取从 2个菜心品种不同部位筛选出的10个与响应高温胁 迫相关重要候选DEGs进行实时荧光定量PCR验证,选用甘油醛-3-磷酸脱氢酶基因(GAPDH)作为内参基因(Wang et al.,2023),利用Primer 5.0设计特 异性引物(表2)。使用HiScript III 1st Strand cDNA Synthesis Kit(+gDNA wiper)试剂盒将24个样本的总 RNA反转录合成cDNA,以ChamQ Universal SYBR qPCR Master Mix(Q711-02)作为荧光染料,采用 CFX384 C1000 Touch实时荧光定量PCR仪进行 检测。反应体系20.0pL:2×ChamQ Universal SYBR qPCR Master Mix 10.0 uL,10 umol/L上、下游引物 各0.4μL,cDNA模板1.0μL,无菌水补足至20.0μL。扩增程序:95℃预变性1min;95℃10s,60℃15s, 72°℃30s,进行40个循环。采用2-AAC法计算基因的相对表达量,试验数据采用Excel2016和SPSS22.0 作图并分析差异显著性。
1.5DEGs的启动子区顺式作用元件分析
基于参考基因组相关信息,利用TBtools-II(Chen et al.,2023)提取10个与响应高温胁迫相关重 要候选DEGs的起始密码子上游长度为2000bp碱 基序列,将提取的序列提交至Plant Care,预测和分 析启动子区顺式作用元件。
2结果与分析
2.1RNA-Seq与基因功能注释结果
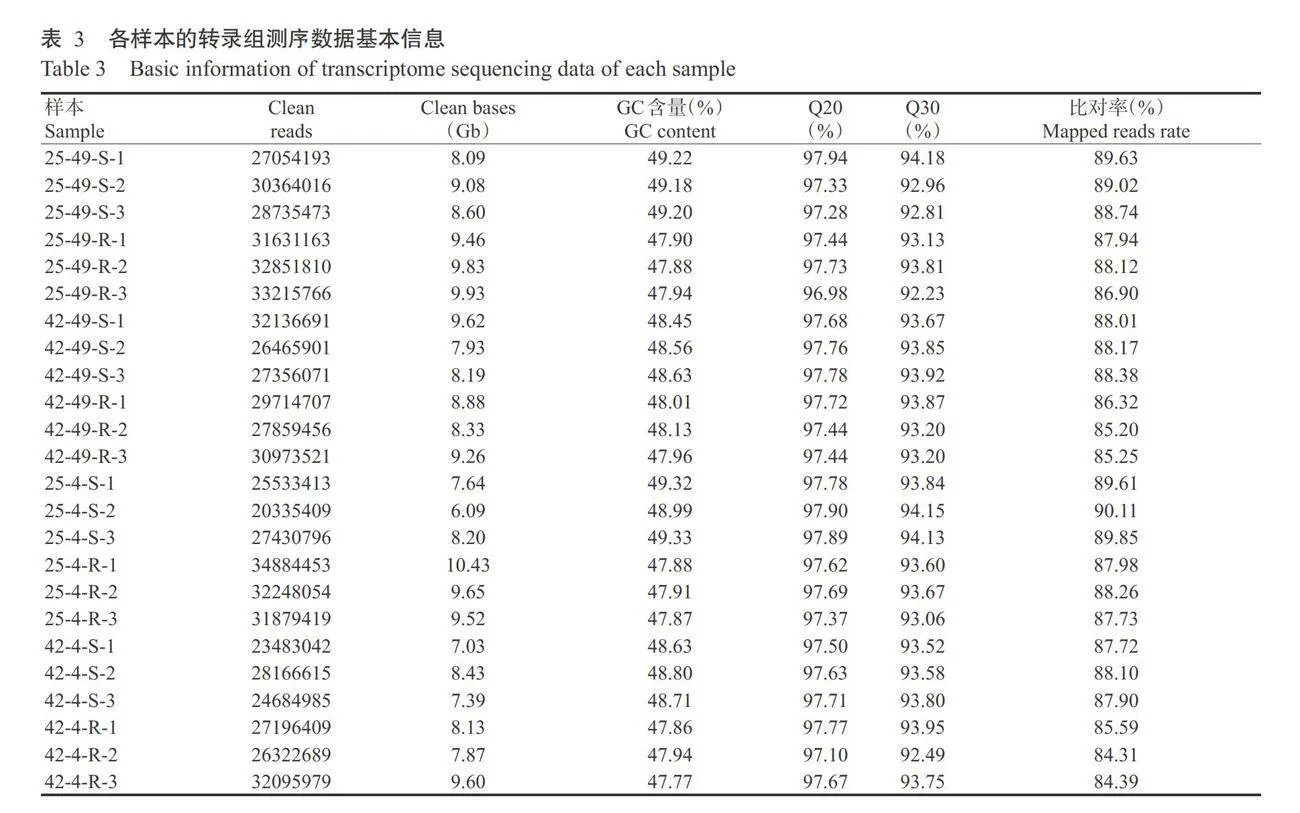
24个菜心样本的转录组分析共获得207.18Gb的 Clean bases(表3),各样本的Clean reads为20335409~ 34884453条,Clean bases为6.09~10.43 Gb,GC含量为47.77%~49.33%,Q20为96.98%~97.94%、Q30为92.23%~94.18%,说明测序数据质量合格。将这24 个样本的Clean reads分别与参考基因组进行序列比 对,最终的比对率为84.31%~90.11%。
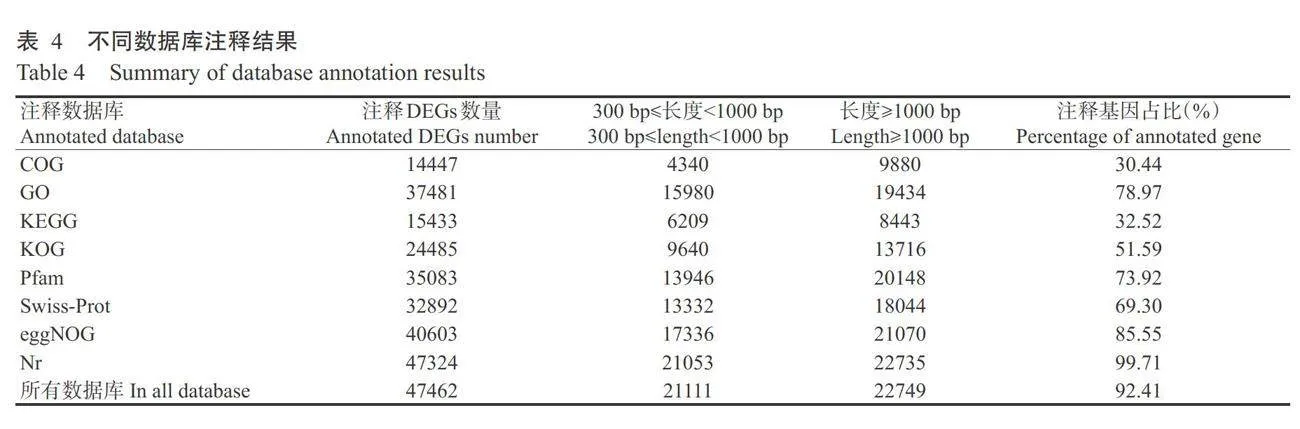
转录组数据中共检测到47462个DEGs,分别在COG、GO、KEGG、KOG、Pfam、Swiss-Prot、eggNOG和Nr生物数据库中进行功能注释(表4),其中在Nr数据库中获得注释率最高,有47324个基因得到注释,注释率为99.71%;其次为eggNOG数据库,注 释率为85.55%;在GO、Pfam、Swiss-Prot、KOG、KEGG 和COG数据库注释率依次分别为78.97%、73.92%、 69.30%、51.59%、32.52%和30.44%;而只有6698个 DEGs在这8个数据库中被共同得到注释,占总DEGs数的14.11%。
2.2新基因发掘与功能注释结果
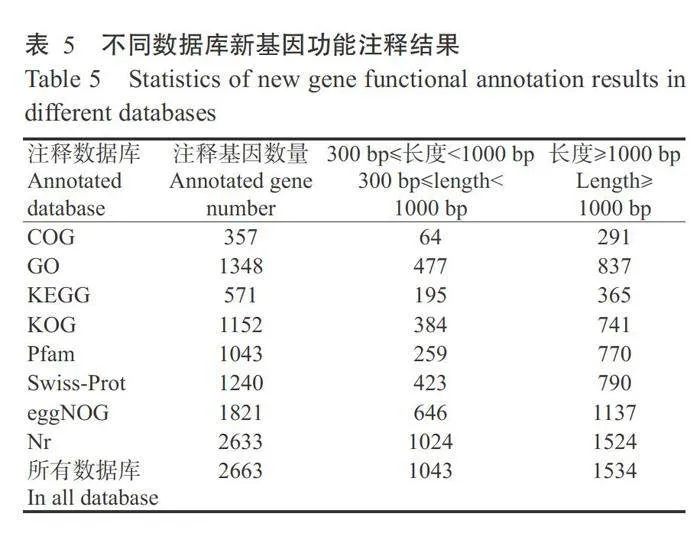
使用StringTie比对到参考基因组上的Reads (Mapped reads)进行拼接,根据参考基因组的注释信 息,比较寻找未被注释的转录区域,从而发掘新基因 或新转录本。同时,对编码少于50个氨基酸残基的 短肽或只包含单个外显子的序列进行删除,最终共 获得3298个新基因。将发掘的新基因在COG、GO、KEGG、KOG、Pfam、Swiss-Prot、eggNOG和Nr生物数据库中进行功能注释,最终注释到2663个新基 因(表5)。
2.34个比较组DEGs分析结果
在|log2Fold Change|≥2且Plt;0.01筛选标准下,24个样本的DEGs筛选结果如表6所示。49-S-heat
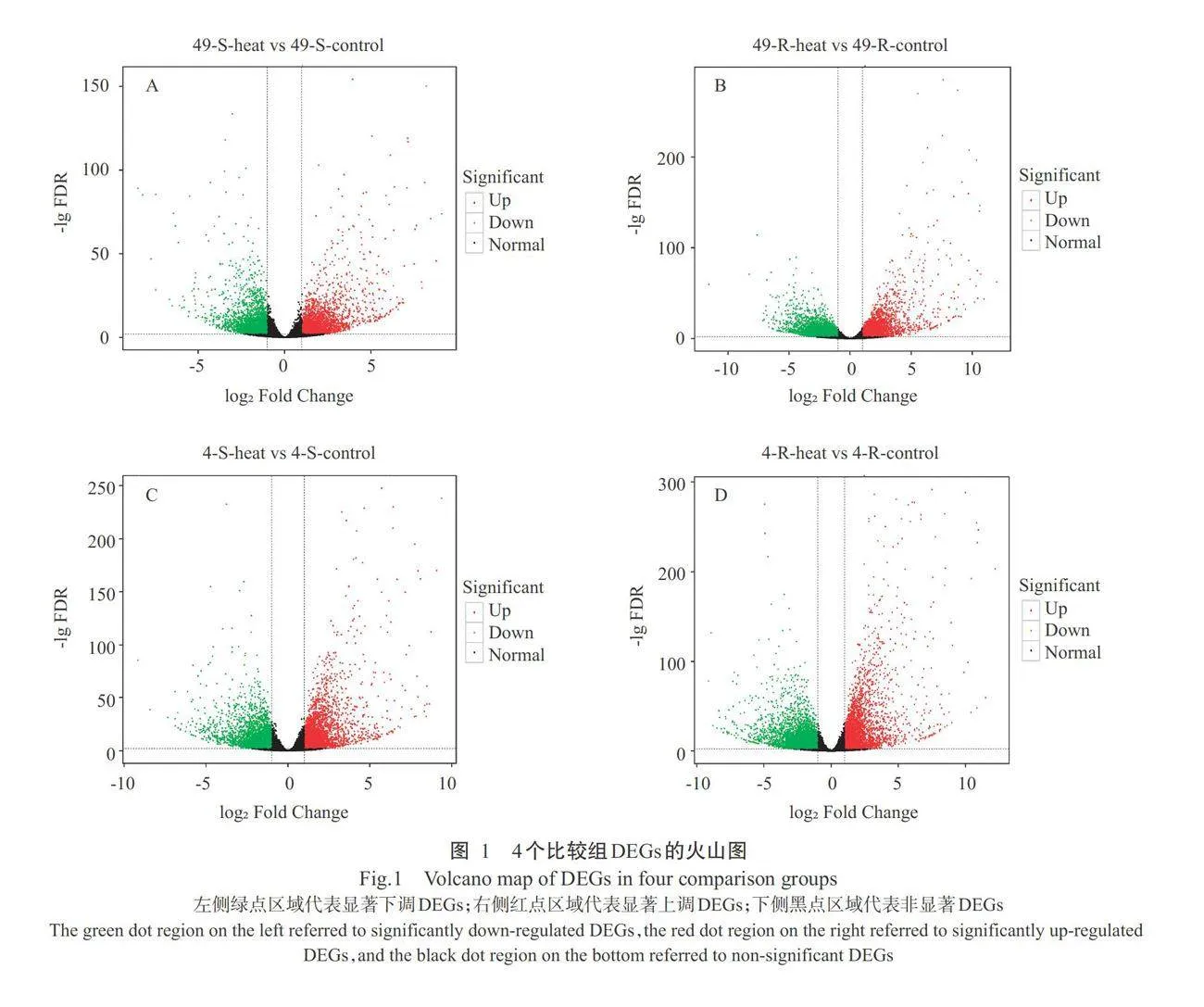
vs 49-S-control比较组中DEGs共5547个,其中显著上调2882个,显著下调2665个;49-R-heat vs 49-R-control比较组中DEGs共8197个,其中显著上调 3895个,显著下调4302个。4-S-heat vs 4-S-control比较组中,DEGs共6630个,其中显著上调3223个,显 著下调3407个;4-R-heat vs 4-R-control比较组中,DEGs共9509个,其中显著上调4401个,显著下调5108个。4个比较组中剔除重复DEGs,共筛选获得15761个DEGs。2个菜心品种地下部分响应高温胁迫的DEGs数量多于地上部分,热敏品种响应高温胁迫DEGs数量均多于耐热品种。由4个比较组 DEGs火山图(图1)可知,DEGs的log2Fold Change 主要集中于-5~5,说明2个菜心品种在高温胁迫下的 DEGs具有相似的表达趋势。
2.4DEGs的GO功能注释分析结果
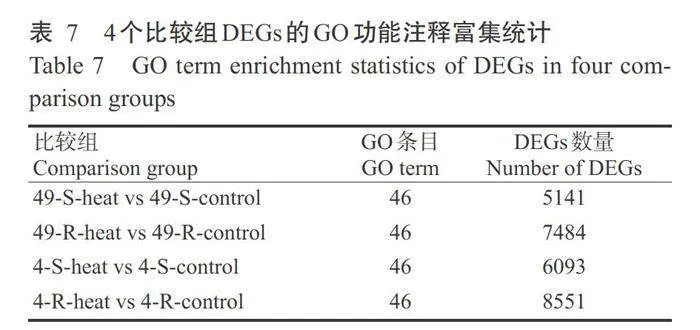
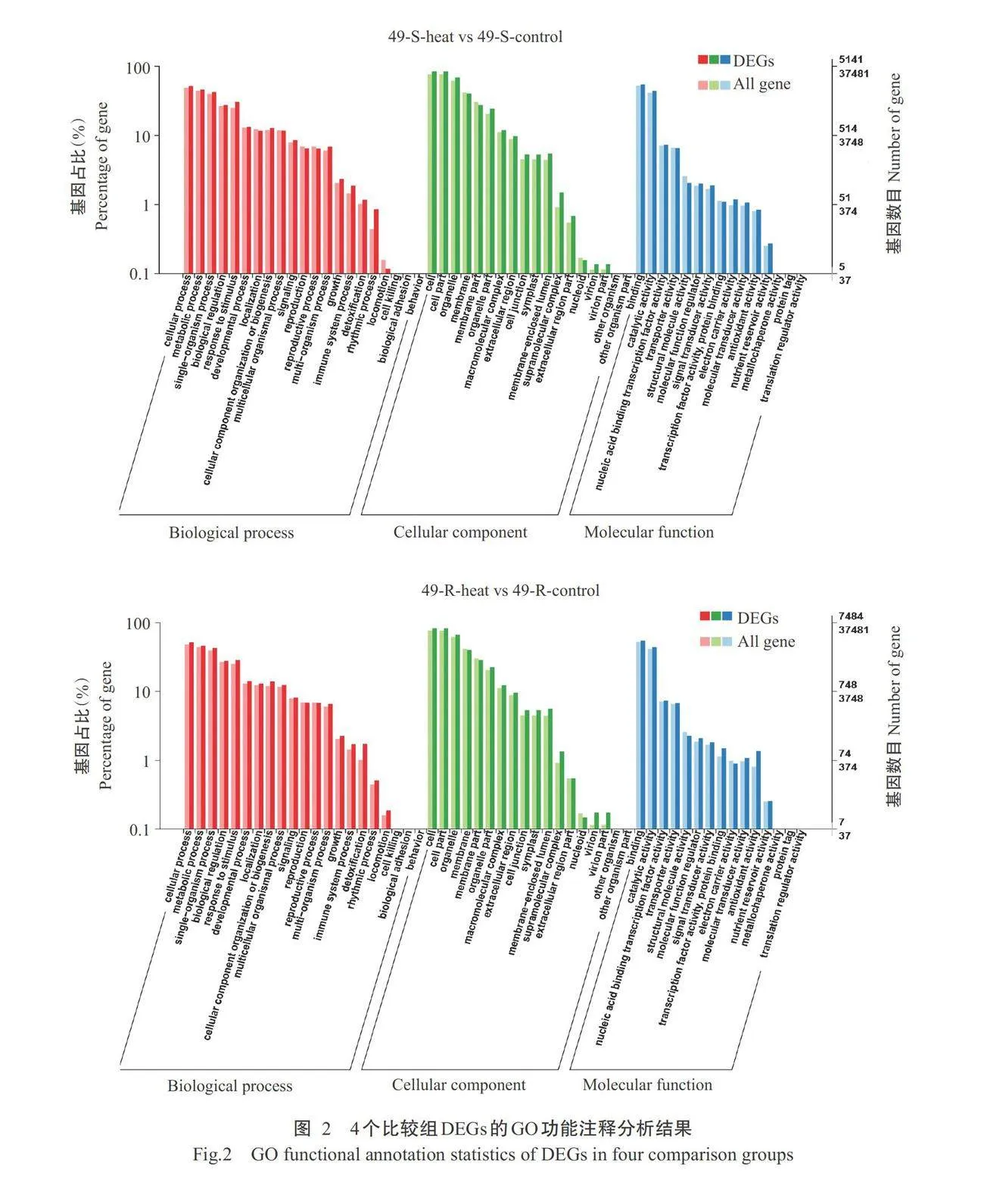
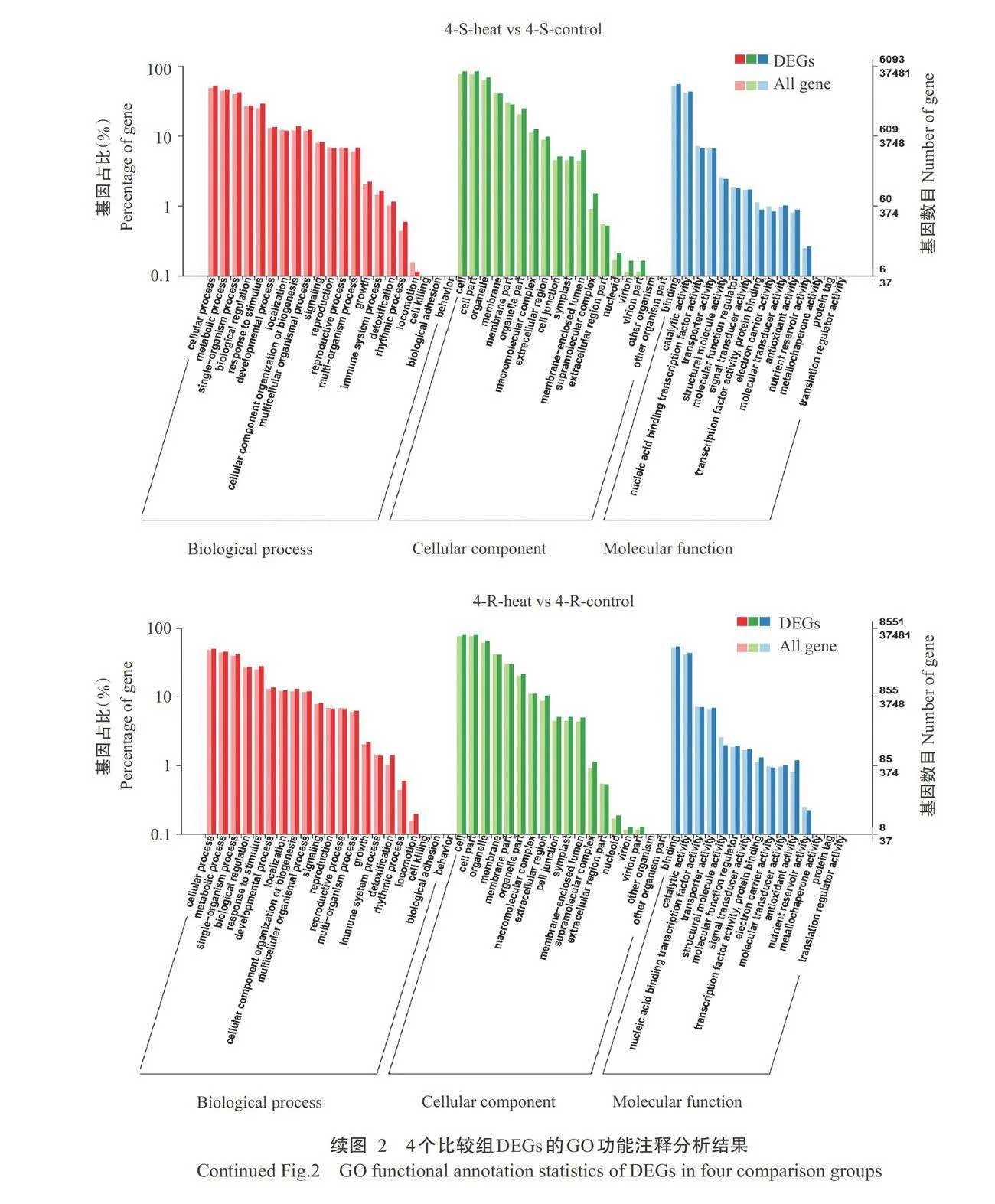
由DEGs的GO功能注释分析结果(图2)可知,共注释到生物过程(Biological process)、细胞组分 (Cellular component)和分子功能(Molecular func-tion)三大类。4个比较组DEGs中,属于生物过程类 有细胞过程(Cellular process)、代谢过程(Metabolic process)、单组织过程(Single-organism process)、应 激反应(Response to stimulus)和生物调节(Biologi- cal regulation);属于细胞组分类有细胞(Cell)、细胞 部分(Cell part)、细胞器(Organelle)、膜(Membrane)、 膜部分(Membrane part)和细胞器部分(Organelle part);属于分子功能类有结合(Binding)和催化活性 (Catalytic activity)。热敏特青迟心4号菜心经高温胁迫后的生物过程、细胞组分和分子功能富集的 DEGs数量明显高于耐热四九-19号菜心,同一菜心品种地下部分的DEGs数量明显高于地上部分(表7)。
2.5DEGs的KEGG信号通路富集分析结果
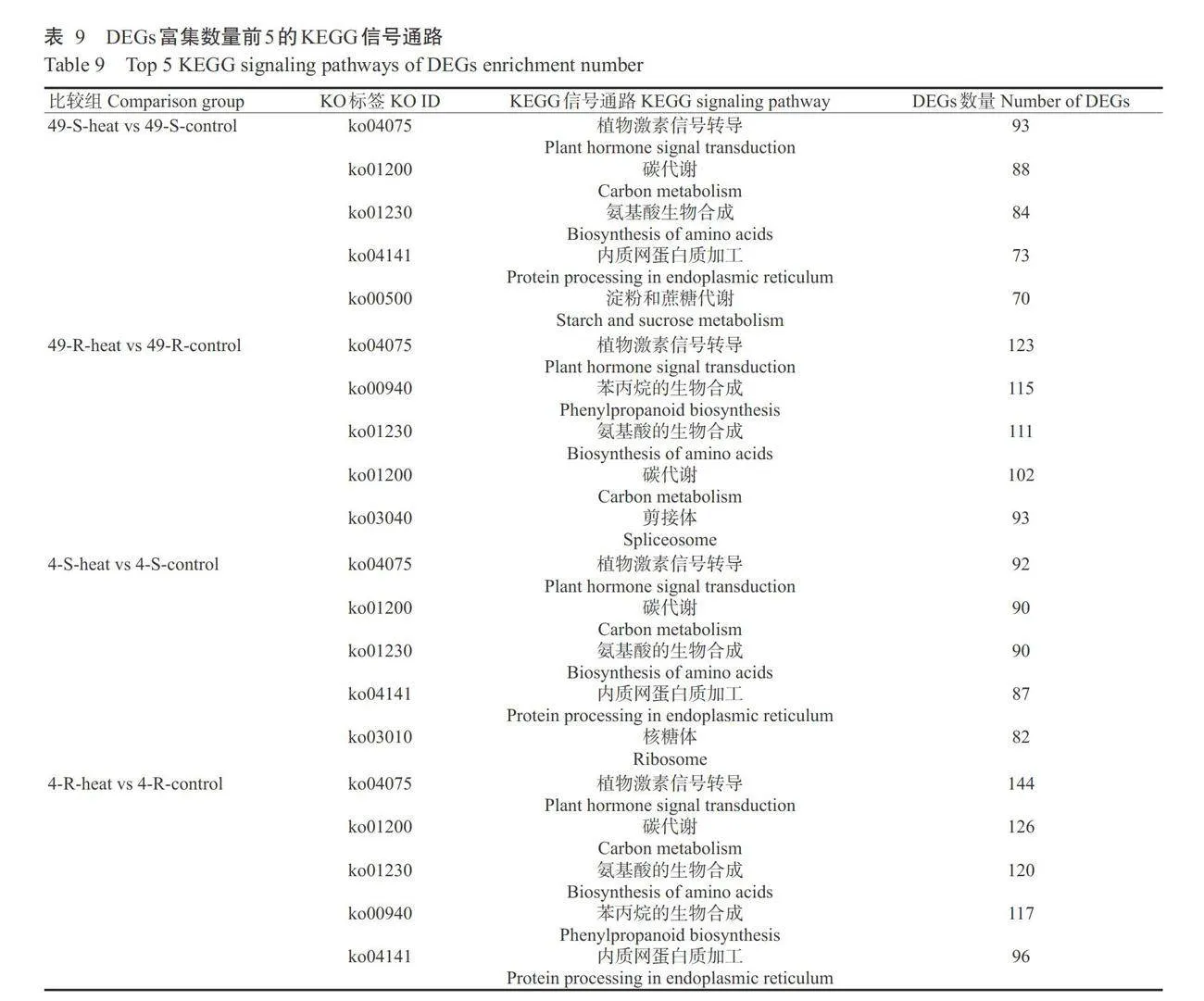
KEGG信号通路富集分析结果表明,由4个比较组DEGs信号通路(表8)及排名前5的信号通路 (表9)可知,49-S-heat vs 49-S-control 比较组中共1297个DEGs富集在122条信号通路中,主要有植物 激素信号转导、碳代谢、氨基酸生物合成、内质网蛋白质加工、淀粉和蔗糖代谢等。49-R-heat vs 49-R- control比较组共1829个DEGs富集在123条信号通 路中,主要涉及植物激素信号转导、苯丙烷的生物合 成、氨基酸的生物合成、碳代谢和剪接体等。4-S-heatvs4-S-control比较组中共1503个DEGs富集在122 条信号通路中,主要涉及植物激素信号转导、碳代 谢、氨基酸生物合成、内质网蛋白质加工和核糖体等。4-R-heat vs 4-R-control比较组中共1970个DEGs 富集在123条信号通路中,主要涉及植物激素信号 转导、碳代谢、氨基酸的生物合成、苯丙烷的生物合 成和内质网蛋白质加工等。综上可知,不同耐热性 菜心品种在响应高温胁迫时,产生的DEGs主要富 集于植物激素信号转导、碳代谢、氨基酸的生物合 成、内质网蛋白质加工等信号通路,说明这些信号通 路在菜心应对高温胁迫上具有重要作用。
2.6响应高温胁迫相关重要候选基因的筛选与分析结果
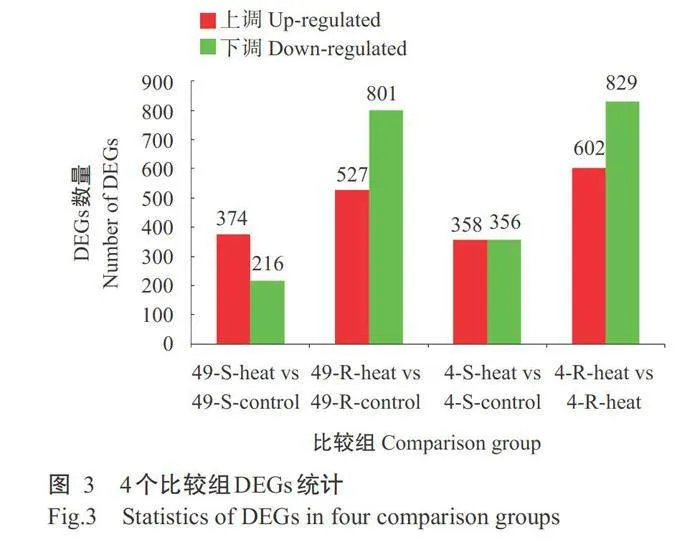
在|log2Fold Change|≥8且Plt;0.001的DEGs高标准筛选下,四九-19号菜心地上部分和地下部分响应 高温胁迫的DEGs分别为590和1328个,特青迟心 4号菜心分别为714和1431个,2品种菜心地下部分 高温胁迫响应的DEGs数量显著下调基因高于显著 上调基因,地下部分DEGs数量均高于地上部分(图3)。剔除4个比较组中重复出现的DEGs,共筛选获得2681个DEGs。
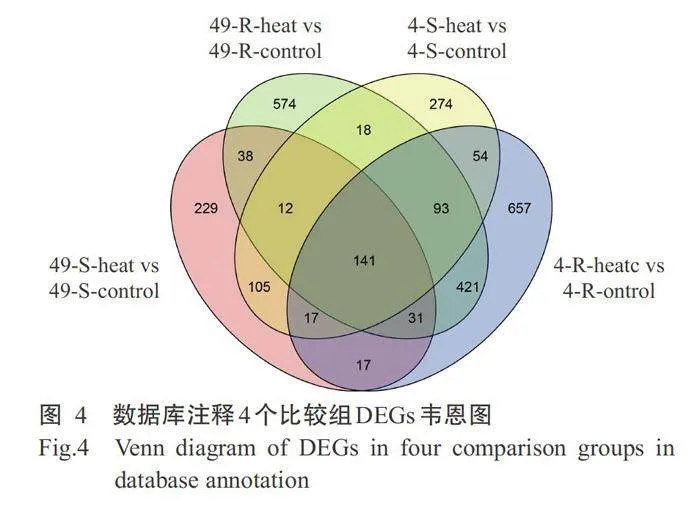
由4个比较组DEGs构建的韦恩图(图4)可知, 高温胁迫后,49-S-heat vs 49-S-control和49-R-heat vs 49-R-control2个比较组共有222个DEGs,4-S-heat vs 4-S-control和4-R-heat vs 4-R-control 2个比较组共有305个DEGs。4个比较组共有141个DEGs,耐热四九-19号菜心中特有的DEGs为38个,热敏特青迟心4号菜心中特有的DEGs为54个。利用Nr、Pfam和Swiss-Prot等对2个菜心品种共有表达和特有表达的233个DEGs进行基因功能注释分析,筛选获得 9个DEGs 被注释为热激转录因子(Heat shock tran- scription factors,HSFs),25个DEGs被注释为热激蛋 白(Heat shock proteins,HSPs)。而HSFs和HSPs是植物热胁迫反应中重要两大类基因家族(韩利红等, 2019;李广隆等,2019),因此,34个DEGs(表10)可 作为响应高温胁迫相关的重要候选基因进行分析 研究。
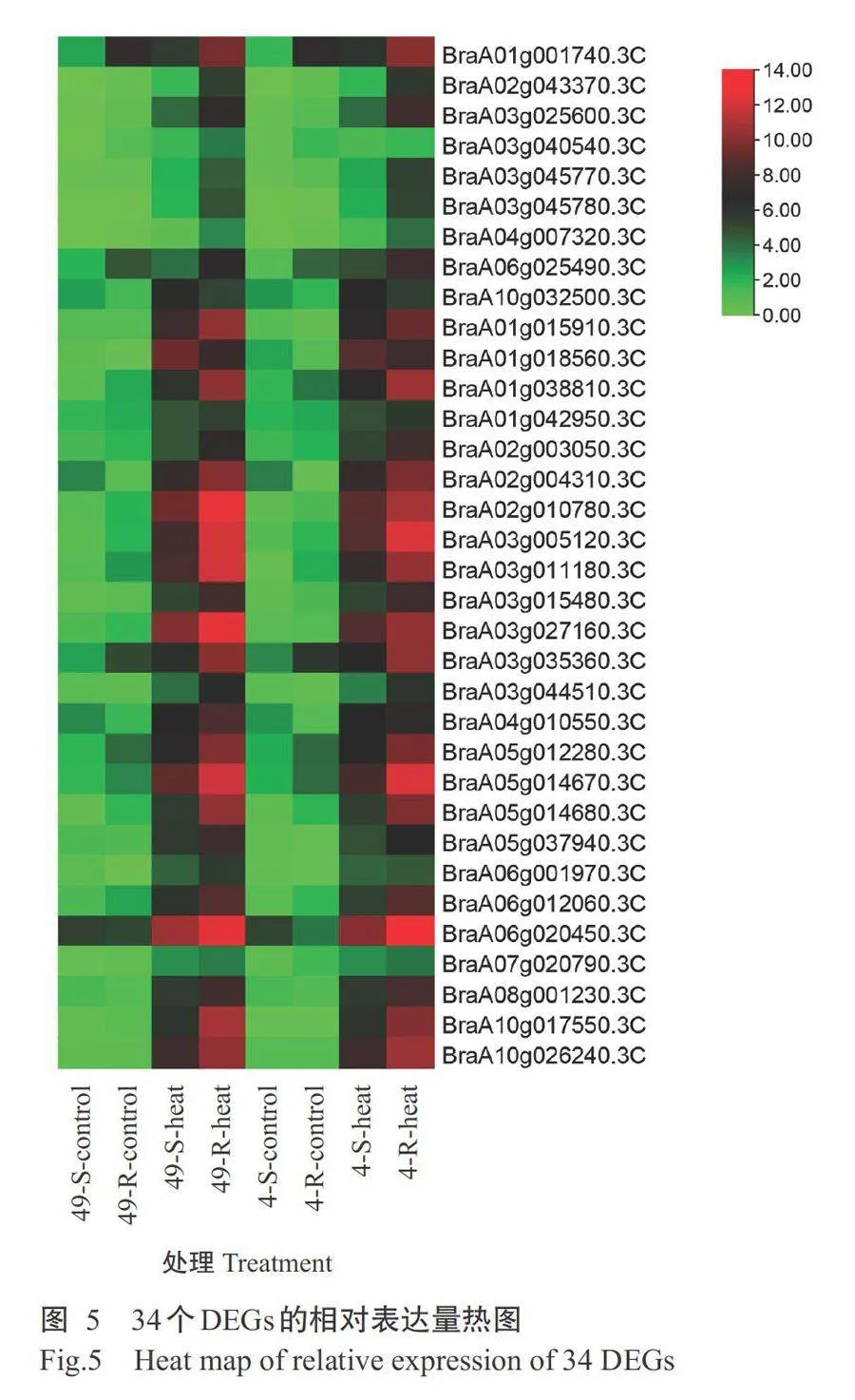
对34个DEGs的FPKM平均值进行归一化处 理,即取log2(FPKM+1)并绘制基因的相对表达量热 图(图5)可知,经过高温胁迫后,这34个基因在耐热 四九-19号菜心和热敏特青迟心4号菜心呈多种表 达模式,不同菜心品种的同一基因表现出相似的表 达模式,2个菜心品种基因表达量在受到高温胁迫后 均表现为显著上调,进一步证明这些基因可响应高 温胁迫。筛选高温胁迫后地上和地下部分中均显著
性上调倍数较大的DEGs,通过同源基因比对及查阅 文献综合筛选出10个响应高温胁迫的重要候选基 因(表11)。
2.7实时荧光定量PCR验证结果
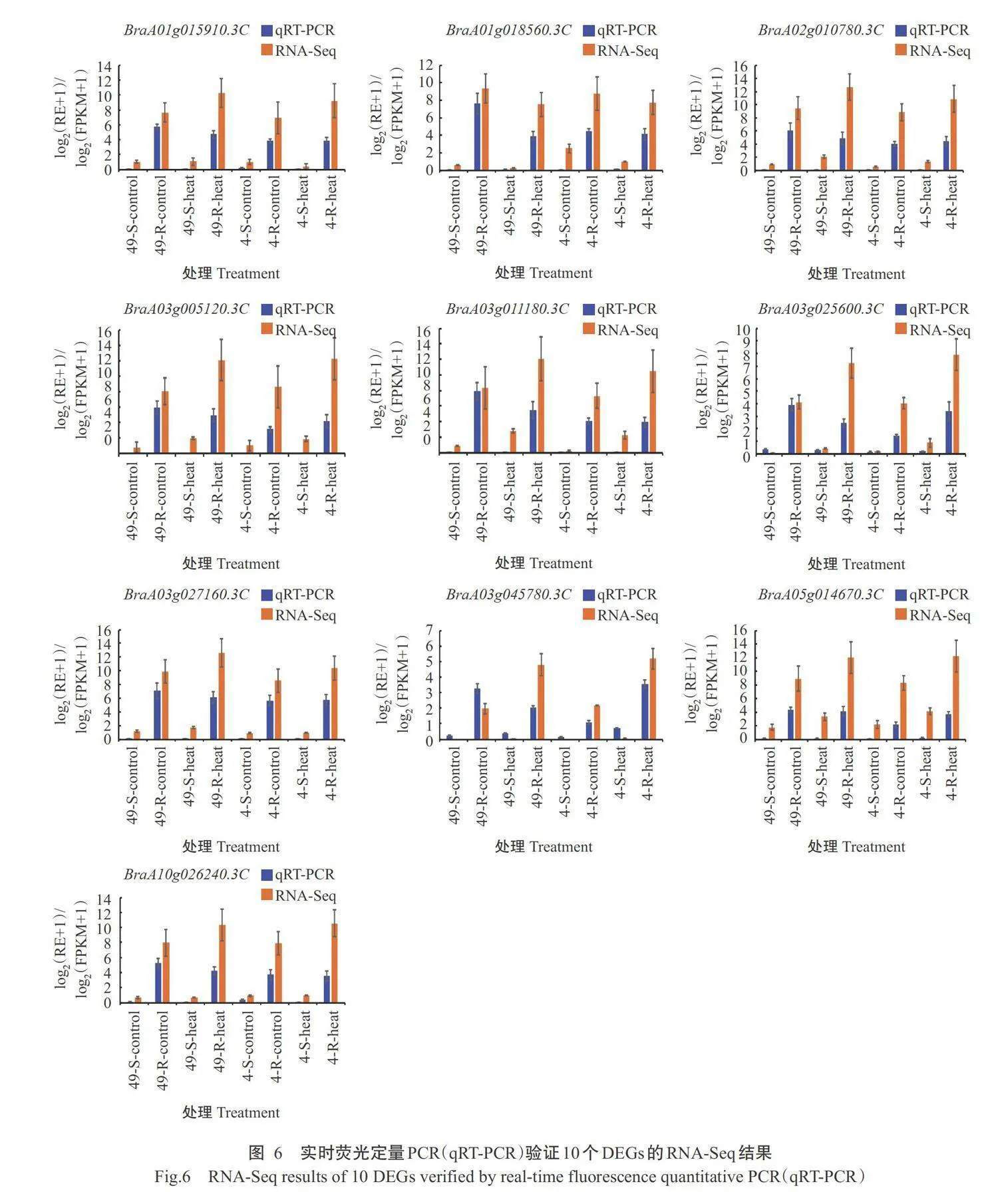
为验证高温胁迫下菜心RNA-Seq数据的准确 性和可靠性,将获得的10个与响应高温胁迫相关的 DEGs进行实时荧光定量PCR检测验证。由图6可知,经过高温胁迫后RNA-Seq与实时荧光定量PCR上调表达的趋势一致,说明RNA-Seq数据结果 可靠。
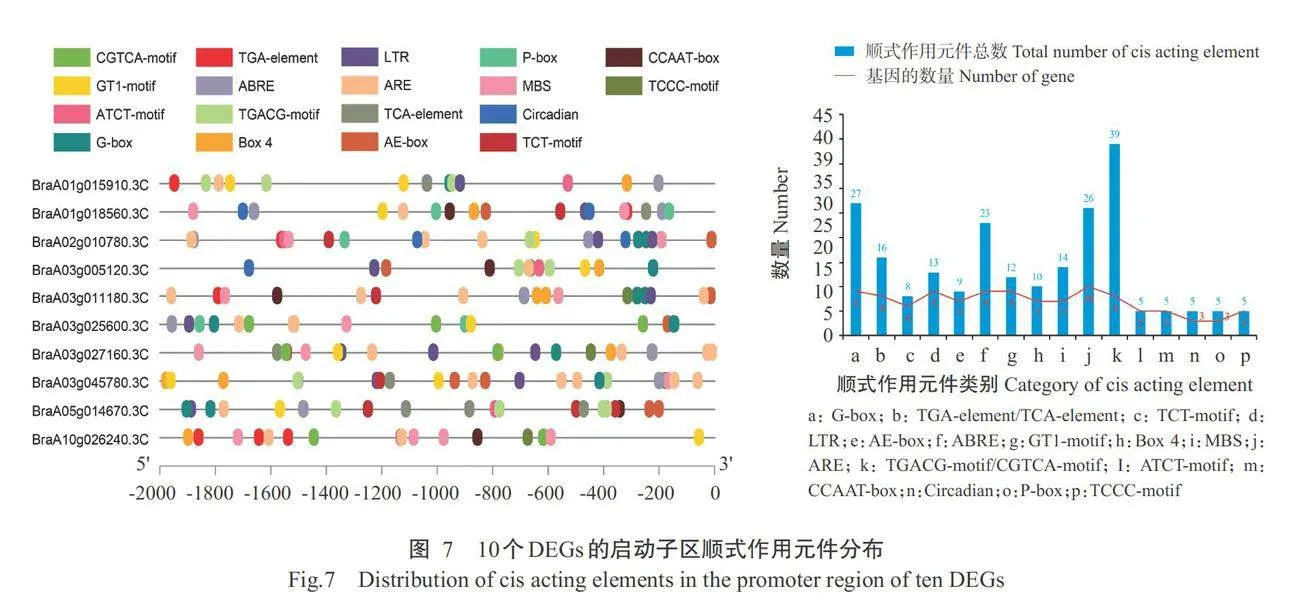
2.810个DEGs启动子区顺式作用元件分析结果
由图7可知,10个DEGs含有多种响应环境和激素信号的顺式作用元件,9个DEGs中的顺式作用元 件LTR与低温响应有关;7个DEGs的MBS与干旱 诱导有关,在;10个DEGs中的ARE与厌氧信号响应 有关;9个DEGs中的ABRE与脱落酸信号响应元有 关;8个DEGs中的CGTCA-motif或TGACG-motif 与茉莉酸响应有关;8个DEGs中的TGA-element或TCA-element与生长素响应有关;5个DEGs中P-box 与赤霉素响应有关;3个DEGs中的Circadian与昼夜节律响应有关;5个DEGs中的CCAAT-box与MYB结合位点有关;有9、7、9、6、7、5和5个DEGs中含有G-box Box 4、GT1-motif、TCT-motif、AE-box、ATCT- motif和TCCC-motif,均为光响应元件。除鉴定到低 温、干旱、厌氧、光、昼夜节律等信号响应顺式作用元 件外,大量激素(脱落酸、茉莉酸、生长素和赤霉素) 响应元件被鉴定,推测植物激素在调节热激反应中 有重要作用。
3讨论
随着全球气候变暖,高温气候导致世界各地的 种植业面临严峻挑战,高温严重抑制农作物的生长 发育,并造成产量与品质下降(王改妮等,2020)。因 此,选育耐热品种是当前农作物育种方向的一个重 要目标,而高温胁迫响应分子机制与挖掘耐热基因能 为耐热新品种的选育提供基础。本研究利用RNA- Seq技术构建不同耐热性菜心在生长发育期高温胁 迫的转录组数据库。转录组数据共获得207.18Gb的Clean bases,组装拼接得到47462个基因;在8个生物数据库中,Nr数据库中注释到的基因数量最 高,有47324个基因得到注释,占总基因数的99.71%; 同时发掘出3298个新基因,为挖掘菜心响应高温胁 迫重要功能基因提供了基因资源基础。
前人研究表明,植物耐热性强弱取决于物种的 基因型,不同基因型在应对高温胁迫上存在遗传上差异(Driedonks et al.,2016;Shi et al.,2017;Jagadish et al.,2021)。通过对耐热性差异极显著的2个材料 进行高温胁迫处理,从中筛选DEGs是寻找响应高 温胁迫候选基因的重要手段之一。本研究通过对耐 热性差异明显的2个菜心品种在高温胁迫条件下产 生的DEGs进行统计,共获得15761个DEGs。分析发现2个菜心品中DEGs表达情况有差异,在高温胁 迫处理前后,耐热四九-19号菜心地上部分、地下部 分中上调DEGs和下调DEGs数量均少于热敏特青 迟心4号菜心,进一步说明热敏感品种对高温胁迫 更敏感,进而触发级联反应,致使更多基因差异表 达。这一结果验证了高温胁迫对热敏型材料转录表 达水平上的影响更大,与陈静芳(2018)在菜心上的 研究结果不同,猜测原因与高温胁迫温度、试验材料 和筛选阈值不同有关。在水稻中发现,40℃高温处 理后,耐热品种996的DEGs数量明显少于热敏感品 种4628(周佳等,2022),与本研究结果相类似,而随 着高温胁迫时间延长,辣椒耐热株系P19055的DEGs数量多于热敏株系P17087(Song et al.,2023), 说明植物体自身耐热性的强弱,取决于生长发育进 程中基因的转录表达调控能力。当植物遭受高温胁 迫时,会激发不同基因的诱导表达或表达沉默,促使 植物外部形态和内部生物学功能进行调整,从而调 控植物生长(王改妮等,2020)。而在高温胁迫处理 前后,同一品种中地下部分响应高温胁迫的DEGs 数量远高于地上部分,猜测地下部分在植物响应高 温胁迫中发挥更大作用,因此,在研究植物响应高温 胁迫时需要考虑地下部分的变化情况。
将DEGs按GO功能注释分为生物过程、细胞组 分和分子功能三大类,其中在细胞过程、代谢过程、 细胞、细胞部分、催化活性和结合蛋白等方面大量富 集,与陈静芳(2018)发现的菜心叶片受高温胁迫的 响应结果相似。说明菜心响应高温胁迫是复杂的生 理生化过程,与辣椒(Li et al.,2015)、芥蓝(雷妙, 2018)、黄瓜(陈雪倩等,2021)、中国南瓜(鲁秀萍,2021)和萝卜(周娜等,2022)等蔬菜作物一样,需要 生物过程、细胞组分和分子功能基因相互协同去抵 抗高温胁迫带来的伤害。KEGG信号通路富集分析发现,菜心受到高温胁迫后DEGs主要富集在植物激素信号转导、碳代谢和氨基酸生物合成等信号通 路,同时在淀粉和蔗糖代谢、内质网蛋白质加工、剪 接体、核糖体、苯丙烷的生物合成等信号通路也受到 高温胁迫的影响,与周娜等(2022)在萝卜幼苗中 KEGG信号通路功能DEGs主要富集在内质网蛋白 质加工、光合作用、光合生物碳固定等研究结果有所 不同,差异原因可能与试验材料、高温胁迫的温度与 时间等方面不同有关。目前,已有研究报道植物激 素信号转导内质网蛋白质加工(Wang et al.,2018;陈 雪倩等,2021)、氨基酸的生物合成(王静,2021)、光 合作用(孙永江等,2023)等信号通路与植物响应高 温胁迫相关,但其发挥的具体功能尚未明确,有待于 进一步研究。徐海(2019)的研究也表明,不结球白 菜在受到高温胁迫后,在细胞膜、叶绿体、内质网等 细胞器中的大量基因发生不同程度的表达变化,参与植株体内蛋白质和其他代谢途径。
通过提高筛选DEGs阈值筛选与菜心响应高温 胁迫密切相关的候选基因,本研究筛选获得与响应 高温相关的9个热激转录因子基因和25个热激蛋白 基因。转录组表达谱分析表明34个DEGs的转录表 达呈多种表达模式,不同菜心品种的同一基因表现出相似的表达模式,同时这2个菜心品种地上部分 和地下部分基因相对表达量在受到高温胁迫后均呈显著上调,证明这些基因响应高温环境的变化,与陈 静芳(2018)研究菜心叶片受高温胁迫的响应相似。 本研究从34个DEGs中综合筛选获得10个与响应 高温胁迫密切相关的重要候选基因,这些基因在芸 薹属或拟南芥物种中的同源基因均被注释或证实与 响应高温胁迫相关。不同物种间的同源基因有相似的基因功能,因此推测这10个基因与响应高温胁迫密切相关,且BraA03g025600.3C和BraA03g045780.3C编码的蛋白是响应高温胁迫重要的正调控因子,而BraA01g015910.3C、BraA01g018560.3C、BraA02g010780.3C、BraA03g005120.3C、BraA03g011180.3C、BraA03g027160.3C、BraA05g014670.3C和BraA10g026240.3C编码的蛋白是响应高温胁迫重 要的结构蛋白。当植株在受到高温胁迫后,正调控 因子向结构蛋白发出信号,结构蛋白基因便大量表 达抵抗高温胁迫环境,但这些蛋白的具体功能有待 进一步验证。10个基因的启动子区顺式作用元件分 析结果显示,除鉴定到低温、干旱、厌氧、光、昼夜节 律等信号响应顺式作用元件外,还发现诸多激素响 应元件,与司修洋等(2020)研究甜瓜热激转录因子 启动子区域分析结果相似,说明植物激素可能在调 节热激反应中起着至关重要的作用,具体响应机制 有待进一步研究。总之,菜心响应高温胁迫是一个 复杂的生理生化过程(彭国颖等,2022),植物在受到 非生物胁迫时,需要多基因共同协调运作才能维持 植物自身的正常生长,因此需要挖掘更多的耐热基 因才能更为全面了解菜心响应高温胁迫的分子 机制。
4结论
从34个基因中筛选获得10个与响应高温诱导 相关且显著上调的重要候选基因,10个基因的启动 子区域鉴定到多种响应环境和激素信号的顺式作用元件,为今后开展菜心耐热分子机制和耐热新品种 的选育提供参考依据。
参考文献(References):
曹毅,李春梅,邓燏,刘永聪.2010.不同菜心品种耐热性研究 [J].西南师范大学学报(自然科学版),35(5):128-131.
[Cao Y,Li C M,Deng Y,Liu Y C. 2010. Study on heat tolerance of different Brassica campestris L. ssp.chinensis var. utilis Tsen et Lee varieties[J]. Journal of Southwest China Normal University (Natural Science Edition) , 35
(5):128-131.] doi:10.13718/j.cnki.xsxb.2010.05.037.
陈汉才,李桂花,宋钊,张艳,曹健,陈琼贤.2010.菜薹新品种 碧绿粗薹菜心[J].园艺学报,37(12):2039-2040.[Chen H C, Li G H,Song Z,Zhang Y,Cao J,Chen Q X. 2010. A new flowering Chinese cabbage cultivar ‘Bilü Cutai Caixin' [J]. Acta Horticulturae Sinica, 37 (12) : 2039-2040.] doi:
10.16420/j.issn.0513-353x.2010.12.013.
陈汉才,吴增祥,林悦欣,沈卓,黎庭耀,杨易,周轩,张艳.
2021.广东菜心、芥蓝研究现状与展望[J].广东农业科学,48(9):62-71.[Chen HC,Wu Z X,Lin Y X,Shen Z, Li T Y, Yang Y, Zhou X, Zhang Y. 2021. Research status and prospect of flowering Chinese cabbage and Chinese kale in Guangdong [J]. Guangdong Agricultural Sciences,48 (9) : 62-71. ] doi : 10.16768/j.issn.1004-874X.2021.09.007.
陈静芳.2018.热胁迫下菜心转录组分析与SSR分子标记开发[D].广州:广州大学.[ChenJF.2018.Transcriptome analysis of flowering Chinese cabbage under heat stress and development of SSR molecular markers[J]. Guang- zhou:Guangzhou University.]
陈连珠,张雪彬,杨小锋.2020.根际高温对菜心生长及光合 生理的影响[J].北方园艺,(14):50-55.[Chen LZ,Zhang X B, Yang X F. 2020. Effects of rhizosphere high tempera- ture on growth and photosynthetic physiology of Chinese flowering cabbages[J]. Northern Horticulture, (14) : 50-55.] doi: 10.11937/bfyy.20193738.
陈雪倩,韩静,任仲海.2021.黄瓜响应高温胁迫转录组分析 [J].分子植物育种,19(12):3905-3914.[Chen XQ,Han J, Ren Z H. 2021. Transcriptome analysis of cucumber in response to high temperature stress[J]. Molecular Plant Breeding, 19 (12) : 3905-3914.] doi: 10.13271/j.mpb.019.003905.
陈雪倩.2020.黄瓜耐热相关基因的鉴定及其热响应作用的 初步分析[D].泰安:山东农业大学.[Chen XQ.2020. Identifcation of cucumber heat-resistant genes and prelimi-nary analysis of their role in thermal response[D]. Tai'an:Shandong Agricultural University.] doi: 10.27277/d. cnki.gsdnu.2020.000088.
郭培国,王直亮,夏岩石,许兰桂,李荣华,黄红弟,张华,郑岩 松.2015.高温胁迫下菜心SSHcDNA文库的建立[J].
广州大学学报(自然科学版),14(1):43-49.[GuoPG, Wang Z L,Xia Y S,Xu L G,Li R H,Huang H D,Zhang H,Zheng Y S. 2015. Preliminary study for investigation of heat-tolerant related genes in flowering Chinese cabbage [J]. Journal of Guangzhou University (Natural Science Edition),14(1):43-49.]
韩利红,刘潮,张维维,李发,邓发虎,阮子恒.2019.铁皮石斛 热激转录因子(Hsf)基因家族鉴定及生物信息学分析
[J].南方农业学报,50(4):677-684.[Han LH,Liu C, Zhang W W, Li F, Deng F H,Ruan Z H. 2019. Gene fami- ly identification and bioinformatics analysis of heat shock transcription factors (Hsf) in Dendrobium officinale[J].
Journal of Southern Agriculture, 50(4) :677-684.] doi: 10.3969/j.issn.2095-1191.2019.04.01.
蒋玲玉.2020.玉米响应高温胁迫的转录组测序及关键调控 基因的鉴定与分析[D].芜湖:安徽师范大学.[Jiang L Y. 2020. Transcriptome sequencing and identification and analysis of key regulatory genes in response to heat stress in maize[D]. Wuhu:Anhui Normal University.]
雷妙.2018.芥蓝苗期对高温胁迫的生理响应和转录组测序 分析[D].广州:华南农业大学.[Lei M.2018.Physiologi- cal responses and transcriptome analysis of Chinese kale under high temperature stress[D]. Guangzhou: South China Agriculture University.]
李光光,张财顺,郑岩松.2012b.不同菜心品种耐热性鉴定指 标的筛选[J].北方园艺,(18):1-5.[LiG G,Zhang CX, Zheng Y S. 2012b. Screening of assessment indexes on heat tolerance in different Chinese flowering cabbage cultivar[J]. Northern Horticulture, (18): 1-5.]
李光光,张华,黄红弟,张财顺.2012a.菜心高温半致死温度 与耐热适应性[J].华北农学报,27(S1):202-204.[LiG
G,Zhang H,Huang H D,Zhang C S. 2012a. The LTso and heat tolerance adaptability of flowering Chinese cabbage in high temperature [J]. Acta Agriculturae Boreali-Sinica,
27(S1):202-204.] doi: 10.3969/j.issn.1000-7091.2012.z1.040.
李广隆,刘思言,鲁中爽,么梦凡,李远强,关淑艳,姚丹,曲 静.2019.植物热激蛋白响应非生物胁迫研究进展[J].
广东农业科学,46(3):24-30.[LiGL,Liu SY,LuZS, Yao M F, Li Y Q, Guan S Y, Yao D, Qu J. 2019. Research progress of plant heat shock protein response to abiotic stress[J]. Guangdong Agricultural Sciences,46(3) :24-30.]
doi:10.16768/j.issn.1004-874X.2019.03.004.
刘畅.2020.高温涝渍对菜心农艺性状和生理特性影响的研
究[D].广州:广州大学.[Liu C.2020.Effect of high tem- perature and waterlogging on agronomic and physiological traits in flowering Chinese cabbage[D]. Guangzhou:Guang- zhou University.]
鲁秀萍.2021.中国南瓜获得性耐热的转录组分析及相关基 因家族鉴定[D].青岛:青岛科技大学.[LuXP.2021.
Transcriptome analysis of acquire thermotolerance in Cucurbita moschata and identification of related gene families [D]. Qingdao: Qingdao University of Science amp; Technology.] doi: 10.27264/d.cnki.gqdhc.2021.000541.
庞强强,孙晓东,周曼,蔡兴来,张文,王亚强.2023.菜心 BrHsfA3基因克隆及其对高温胁迫的响应[J].生物技术 通报,39(2):107-115.[Pang QQ,Sun XD,Zhou M,Cai X L,Zhang W,Wang Y Q. 2023. Cloning of BrHsfA3 in Chinese flowering cabbage and its responses to heat stress [J]. Biotechnology Bulletin, 39 (2) : 107-115.] doi: 10.
13560/j.cnki.biotech.bull.1985.2022-0568.
庞强强,周曼,孙晓东,陈贻诵,蔡兴来.2020a.不同菜心品种 萌发期和苗期耐热性分析及其鉴定指标筛选[J].西北农业学报,29(2):295-305.[Pang QQ,Zhou M,Sun XD, Chen Y S, Cai X L. 2020a. Comprehensive evaluation and indexes screening of heat tolerance at germination and seedling stages in different cultivars of Chinese flowering cabbage[J]. Acta Agriculturae Boreali-Occidentalis Sinica,
29 (2) : 295-305. ] doi : 107606/j.issn.1004-1389.2020.02.016.
庞强强,周曼,孙晓东,张文,蔡兴来.2020b.菜心耐热性评价 及酶促抗氧化系统对高温胁迫的响应[J].浙江农业学报,32(1):72-79.[Pang QQ,Zhou M,Sun XD,Zhang W, Cai X L. 2020b. Evaluation of heat tolerance and response of enzymatic antioxidant system to heat stress in Brassica parachinensis L. [J]. Acta Agriculturae Zhejian- gensis,32(1):72-79.] doi: 10.3969/j.issn.1004-1524. 2020.
01.09.
彭国颖,胡亮,黄超,杨坤,万玮,黄长干.2022.紫鸭跖草根组 织应答铜胁迫的转录组分析[J].生物技术通报,38(2): 83-94. [Peng G Y, Hu L,Huang C, Yang K, Wan W, Huang C G. 2022. Transcriptome analysis of response to heavy metal copper stress in Setcreasea purpurea root tis- sue [J]. Biotechnology Bulletin, 38 (2) : 83-94.] doi: 10.
13560/j.cnki.biotech.bull.1985.2021-0664.
司修洋,梁文杰,罗澜,汪宇欣,刘大伟.2020.甜瓜热激转录 因子(Hsf)基因家族鉴定及生物信息学分析[J].中国蔬菜,(11):23-32.[SiXY,Liang WJ,Luo L,Wang YX, Liu D W. 2020. Identification and bioinformatics analysis of heat shock transcription factor (Hsf) gene family in melon[J]. China Vegetables, (11) : 23-32.] doi: 10.19928/ j.cnki.1000-6346.2020.11.004.
孙永江,王琪,邵琪雯,辛智鸣,肖辉杰,程瑾.2023.高温胁迫 对植物光合作用的影响研究进展[J].植物学报,58(3):
486-498. [Sun Y J, Wang Q, Shao Q W,Xin Z M, Xiao H J, Cheng J. 2023. Research advances on the effect of high temperature stress on plant photosynthesis[J]. Chinese Bulletin of Botany,58(3):486-498.] doi:10.11983/CBB22079.
王改妮,白万鹏,王锁民.2020.植物响应高温胁迫的信号转 导和转录调控机制研究进展[J].分子植物育种,18(24):8109-8118. [Wang G N, Bai W P, Wang S M. 2020.
Advances in research of signal transduction and transcriptional regulatory mechanism of plants in response to heat stress [J]. Molecular Plant Breeding, 18 (24) : 8109-8118.] doi:10.13271/j.mpb.018.008109.
王辉,董永梅,郭伟锋,曹新川,郭金成,谢宗铭,何良荣.
2023.高温胁迫下陆地棉转录组差异性分析[J].新疆农业科学,60(11):2618-2626.[WangH,DongY M,Guo W F, Cao X C, Guo J C,Xie Z M, He L R. 2023. Analysis of the difference of transcription groups of upland cotton under heat stress[J]. Xinjiang Agricultural Sciences,60(11):
2618-2626.] doi:10.6048/j.Issn.1001-4330.2023.11003.
王静.2021.辣椒种质资源耐热性评价及其对高温胁迫响应 机制的研究[D].长沙:湖南大学.[WangJ.2021.Evalua- tion of heat resistance of pepper resources and its response mechanism to heat stress[D]. Changsha:Hunan University.]
doi: 10.27135/d.cnki.ghudu.2021.002120.
王坤,杨淑华,丁杨林.2023.植物应答高温胁迫的机制研究 进展[J].植物生理学报,59(4):759-772.[Wang K,Yang S H, Ding Y L. 2023. Advances in uncovering mechanisms of plant responses to heat stress[J]. Plant Physiology Jour- nal,59(4):759-772.] doi:10.13592/j.cnki.ppj.400007.
王少波.2019.水稻耐热胁迫基因的挖掘及其表达分析[D].
福州:福建农林大学.[Wang S B.2019.Mining and analysis genome heat resistance genes in rice (Oryza sativa L.) [D]. Fuzhou: Fujian Agriculture and Forestry University.]
韦丽君,俞奔驰,宋恩亮,郑华,卢赛清,付海天.2020.基于转录组测序的木薯性别决定相关基因挖掘[J].南方农业学
报,51(8):1785-1796.[Wei LJ,Yu B C,Song EL,Zheng H, Lu S Q, Fu H T. 2020. Gene mining for sex determina- tion in cassava (Manihot esculenta Crantz) based on tran- scriptome sequencing[J]. Journal of Southern Agriculture,
51(8) : 1785-1796. ] doi : 10.3969/j.issn.2095-1191.2020.08.001.
徐海.2019.不结球白菜热胁迫响应生理机制及相关基因鉴 定研究[D].南京:南京农业大学.[Xu H.2019.Identifi- cation of physiological mechanism and related genes asso- ciated with heat stress response in non-heading Chinese cabbage [D]. Nanjing: Nanjing Agricultural University.]
doi:10.27244/d.cnki.gnjnu.2019.000157.
张华,刘自珠.2010.菜薹(菜心)的市场需求与育种现状[J].
中国蔬菜,(3):10-12.[Zhang H,Liu Z Z.2010.Market demand and breeding status of flowering Chinese cabbage [J]. China Vegetables, (3) : 10-12. ] doi : 10.19928/j.cnki.1000-6346.2010.03.005.
张衍荣.1997.菜心育种现状与展望[J].广东农业科学,(3):
14-16. [Zhang Y R. 1997. Current status and prospect of flowering Chinese cabbage breeding [J]. Guangdong Agricultural Sciences, (3) : 14-16.] doi: 10.16768/j.issn. 1004-874x.1997.03.006.
周佳,彭艳,陈信波.2022.高温胁迫下不同耐热性水稻品种 的差异表达基因分析[J].湖南农业大学学报(自然科学版),48(5):536-542.[ZhouJ,Peng Y,Chen X B.2022.
Analysis of differentially expressed genes in rice varieties with different heat tolerance under high temperature stress [J]. Journal of Hunan Agricultural University (Natural Sciences ) , 48 (5) : 536-542. ] doi : 10.13331/j.cnki.jhau.2022.05.005.
周娜,郑阳,陆景伟,胡燕,陶伟林,雷开荣,潘晓雪.2022.高 温胁迫下萝卜苗期的转录组分析[J].南方农业学报,53
(3) :665-675. [Zhou N,Zheng Y,Lu J W, Hu Y, Tao W L, Lei K R, Pan X X. 2022. Transcriptome analysis of Raphanus sativus seedlings in response to high temperature stress[J]. Journal of Southern Agriculture, 53 (3) :665675.] doi:10.3969/j.issn.2095-1191.2022.03.008.
Ahmed W,Xia Y S, Li R H,Zhang H,Siddique K H M,Guo P
G. 2021. Identification and analysis of small interfering RNAs associated with heat stress in flowering Chinese cabbage using high-throughput sequencing[J]. Frontiers in Genetics, 12: 746816. doi: 10.3389/fgene.2021.746816.
Charng Y Y,Liu H C,Liu N Y,Chi W T,Wang C N,Chang S H, Wang T T. 2007. A heat-inducible transcription factor, HsfA2, is required for extension of acquired thermotolerance in Arabidopsis[J]. Plant Physiology,143(1):251-262.
doi: 10.1104/pp.106.091322.
Chen C J,Wu Y, Li J W,Wang X,Zeng Z H,Xu J,Liu Y L, Feng J T, Chen H, He Y H, Xia R. 2023. TBtools-IⅡ: A \"one for all, all for one\" bioinformatics platform for bio-logical big-data mining[J]. Molecular Plant, 16(11) : 17331742. doi: 10.1016/j.molp.2023.09.010.
Driedonks N, Rieu I, Vriezen W H. 2016. Breeding for plant heat tolerance at vegetative and reproductive stages [J].
Plant Reproduction,29(1):67-79. doi 10.1007/s00497-016-0275-9.
Handa H, Itan K, Sato H. 2002. Structural features and expression analysis of a linear mitochondrial plasmid in rapeseed (Brassica napus L.)[J]. Molecular Genetics and Genomi-
cs,267(6):797-805. doi:10.1007/s00438-002-0711-4.
Handa H. 2003. The complete nucleotide sequence and RNA editing content of the mitochondrial genome of rapeseed (Brassica napus L.) : Comparative analysis of the mitochondrial genomes of rapeseed and Arabidopsis thaliana [J]. Nucleic Acids Research, 31 (20) : 5907-5916. doi: 10.1093/nar/gkg795.
Jagadish S V K, Way D A, Sharkey T D. 2021. Plant heat stress: Concepts directing future research[J]. Plant Cell amp; Environment,44(7):1992-2005. doi:10.1111/pce.14050.
Li B J, Gao K, Ren H M, Tang W Q. 2018. Molecular mechanisms governing plant responses to high temperatures[J].
Journal of Integrative Plant Biology, 60(9) : 757-779. doi: 10.1111/jipb.12701.
Li C G,Chen Q J,Gao X Q,Qi B S,Chen N Z,Xu S M, Chen J, Wang X C. 2005. AtHsfA2 modulates expression of stress responsive genes and enhances tolerance to heat and oxidative stress in Arabidopsis[J]. Science in China. Series C:Life Sciences,48(6):540-550. doi:10.1360/062005-119.
Li T, Xu X W, Li Y, Wang H M, Li Z X. 2015. Comparative transcriptome analysis reveals differential transcription in heat-susceptible and heat-tolerant pepper (Capsicum annum
L.) cultivars under heat stress [J]. Journal of Plant Bio-
logy,58(6):411-424. doi:10.1007/s12374-015-0423-z.
Mittler R, Finka A, Goloubinoff P. 2012. How do plants feel the heat?[J]. Trends in Biochemical Sciences, 37(3): 118-125.
doi:10.1016/j.tibs.2011.11.007.
Nishizawa A, Yabuta Y, Yoshida E, Maruta T, Yoshimura K, Shigeoka S. 2006. Arabidopsis heat shock transcription factor A2 as a key regulator in response to several types of environmental stress [J]. The Plant Journal, 48 (4) : 535547. doi:10.1111/j.1365-313X.2006.02889.x.
Nover L, Bharti K, Doring P, Mishra S K, Ganguli A, Scharf K
D. 2001. Arabidopsis and the heat stress transcription factor world:How many heat stress transcription factors do we need? [J]. Cell Stress amp; Shaperones,6(3): 177-189. doi: 10.1379/1466-1268(2001)006lt;0177:aathstgt;2.0.co;2. Parkin I A, Koh C, Tang H B, Robinson S J, Kagale S, Clarke W E, Town C D,Nixon J, Krishnakumar V,Bidwell S L, Denoeud F, Belcram H, Links M G, Just J, Clarke C, Bender T, Huebert T, Mason A S, Pires J C, Barker G, Moore J, Walley P G, Manoli S, Batley J, Edwards D, Nelson M N, Wang X Y, Paterson A H,King G, Bancroft I, Chalhoub B, Sharpe A G. 2014. Transcriptome and methy-
lome profiling reveals relics of genome dominance in the mesopolyploid Brassica oleracea[J].Genome Biology, 15(6):R77. doi:10.1186/gb-2014-15-6-r77.
Riechmann J,Heard J,Martin G,Reuber L,Jiang C,Keddie J, Adam L, Pineda O, Ratcliffe O J, Samaha R R, Creelman R, Pilgrim M, Broun P, Zhang J Z, Ghandehari D, Sherman B K, Yu G. 2000. Arabidopsis transcription factors: Genome-wide comparative analysis among eukaryotes [J].
Science, 290(5499): 2105-2110. doi:10.1126/science.290.5499.2105.
Shi J, Yan B Y, Lou X P, Ma H S, Ruan S L. 2017. Comparative transcriptome analysis reveals the transcriptional alterations in heat-resistant and heat-sensitive sweet maize (Zea mays L.) varieties under heat stress [J]. BMC Plant Biology,17(1):26. doi:10.1186/s12870-017-0973-y.
Sloan D B, Wu Z Q, Sharbrough J. 2018. Correction of persistent errors in Arabidopsis reference mitochondrial genomes [J]. The Plant Cell, 30 (3) : 525-527. doi: 10.1105/tpc. 18.
00024. Song Y P,Zhu Z W,Liu K G,Zhao Y L,Nie Z X,Zhang L L, Fahim A M, Yang X D. 2023. Comparative transcriptome analysis reveals differential gene expression pattern associated with heat tolerance in pepper (Capsicum annuum L.) [J]. Horticulturae , 9(7): 801. doi: 10.3390/horticulturae
9070801.
Wang J,Lv J H,Liu Z B,Liu Y H,Song J H,Ma Y Q,Ou L J, Zhang X L, Liang C L,Wang F,Juntawong N,Jiao C H, Chen W C, Zou X X. 2019. Integration of transcriptomics
and metabolomics for pepper (Capsicum annuum L.) in response to heat stress[J]. International Journal of Molecular Sciences,20(20) :5042. doi: 10.3390/ijms20205042.
Wang R H,Mei Y,Xu L,Zhu X W,Wang Y,Guo J,Liu L W.
2018. Genome-wide characterization of differentially expressed genes provides insights into regulatory network of heat stress response in radish (Raphanus sativus L.) [J].
Functional amp; Integrative Genomics, 18(2) : 225-239. doi:10.1007/s10142-017-0587-3.
Wang X W,Wang H Z,Wang J,Sun R F,Wu J,Liu S Y,Bai Y Q, Mun J H, Bancroft I, Cheng F, Huang S W, Li X X, Hua W,Wang J Y,Wang X Y,Freeling M,Pires J C,Paterson A H, Chalhoub B, Wang B, Hayward A, Sharpe A G, Park B S, Weisshaar B, Liu B H, Li B, Liu B, Tong C B, Song C,Duran C,Peng C F,Geng C Y,Koh C,Lin C Y, Edwards D, Mu D S, Shen D, Soumpourou E, Li F,Fraser F, Conant G, Lassalle G, King G J,Bonnema G, Tang H
B, Wang H P,Belcram H,Zhou H L, Hirakawa H, Abe H, Gou H, Wang H,Jin H Z,Parkin,I A P,Batley J,Kim J S, Just J,Li J W,Xu J H, Deng J,Kim J A, Li J P,Yu J Y, Meng J L, Wang J P, Min J M, Poulain J, Wang J, Hatakeyama K,Wu K,Wang L,Fang L, Trick M, Links M G,Zhao M X,Jin M N,Ramchiary N,Drou N,Berkman P
J, Cai Q L, Huang Q F, Li RQ, Tabata S, Cheng S F, Zhang S,Zhang S J,Huang S M,Sato S,Sun S L,Kwon S J,Choi S R,Lee T H,Fan W,Zhao X,Tan X,Xu X,Wang
Y,Qiu Y,Yin Y, Li Y R,Du Y C,Liao Y C,Lim Y,Narusaka Y,Wang Y P,Wang Z Y,Li Z Y,Wang Z W,Xiong Z Y, Zhang Z H. 2011. The genome of the mesopolyploid crop species Brassica rapa [J]. Nature Genetics,43 (10) : 1035-1039. doi: 10.1038/ng.919.
Wang Y D, Song S W,Hao Y W,Chen C M,Ou X,He B, Zhang J W,Jiang Z H,Li C M,Zhang S W,Su W,Chen R Y. 2023. Role of BraRGL1 in regulation of Brassica rapa bolting and flowering [J]. Horticulture Research, 10 (8) :
uhad119. doi: 10.1093/hr/uhad119.
Zhang A D, Zhu Z W,Shang J,Zhang S M,Shen H B, Wu X X, Zha D S. 2020. Transcriptome profiling and gene expression analyses of eggplant (Solanum melongena L.) under heat stress [J]. PLoS One, 15 (8) : e0236980. doi: 10.1371/journal.pone.0236980.
Zhang L,Cai X,Wu J, Liu M,Grob S,Cheng F,Liang J L,Cai
C C,Liu Z Y,Liu B,Wang F,Li S,Liu F Y,Li X M,Cheng
L, Yang W C,Li M H, Grossniklaus U,Zheng H K, Wang X W. 2018. Improved Brassica rapa reference genome by single-molecule sequencing and chromosome conformation capture technologies[J]. Horticulture Research, 5: 50.
doi:10.1038/s41438-018-0071-9.
Zhao J G, Lu Z G, Wang L,Jin B. 2020. Plant responses to heat stress: Physiology, transcription, noncoding RNAs, and epigenetics[J]. Internation Journal of Molecular Sciences,
22(1):117. doi:10.3390/ijms22010117.
(责任编辑李洪艳)
