丝状真菌Podospora anserina AA11家族裂解多糖单加氧酶基因的鉴定和功能研究
2023-12-20杜文珍李元敬吴佳玲陈思羽姜亮刘刚谢宁
杜文珍,李元敬,吴佳玲,陈思羽,姜亮,刘刚,谢宁
研究报告
丝状真菌AA11家族裂解多糖单加氧酶基因的鉴定和功能研究
杜文珍1,李元敬2,吴佳玲1,陈思羽1,姜亮1,刘刚1,谢宁1
1. 深圳大学生命与海洋科学学院,深圳市微生物基因工程重点实验室,深圳 518060 2.华南师范大学材料与新能源学院,汕尾 516622
辅助活性蛋白家族(auxiliary activity family,AA family)中的裂解多糖单加氧酶(lytic polysaccharide monooxygenase, LPMO)能催化纤维素、几丁质和淀粉等多种难降解碳水化合物的氧化解聚。尽管目前对LPMO的酶学研究较多,但对基因失活的研究却鲜有报道。本研究利用同源重组方法定点敲除丝状真菌中AA11家族的5个基因()、()、()、()和(),分别构建了单突变体ΔPaLPMO11A (ΔA)、ΔPaLPMO11B (ΔB)、ΔPaLPMO11C (ΔC)、ΔPaLPMO11D (ΔD)和ΔPaLPMO11E (ΔE),然后通过遗传杂交构建所有多基因突变体。通过在不同碳源培养基上的表型分析、DAB和NBT染色以及纤维素酶活测定分析野生型菌株与突变型菌株在生长速率、有性生殖、氧化应激和纤维素降解能力等方面的差异,揭示基因在菌株的生长发育和木质纤维素降解过程中的作用。实验结果表明,在不同纤维素碳源上,ΔBΔCΔE、ΔAΔBΔCΔE、ΔAΔCΔDΔE和ΔAΔBΔCΔDΔE突变型菌株的有性生殖能力降低,其余突变型菌株的孢子萌发效率、生长速率和生殖能力几乎没有差异。PaLPMO11家族5个基因的同时缺失,会导致菌株利用各种碳源的能力明显降低、生长速率降低、孢子萌发率降低、子实体数减少、部分子实体发育异常、寿命缩短和降解纤维素的能力显著下降,但仍有野生型45%以上的总纤维素酶活力。上述结果表明,基因可能参与的生长发育、有性生殖、衰老和纤维素降解过程。本研究为系统阐述丝状真菌中木质纤维素降解的调控机制提供参考。
丝状真菌;;;基因敲除;木质纤维素降解
人类使用的大多数燃料和化学品资源都来源于煤炭、石油和天然气等不可再生的化石能源。随着全球经济的快速发展和工业化规模的不断扩大,化石能源的过度消耗,引发了不可再生化石能源储量枯竭、环境污染和全球变暖等一系列问题,给全球环境保护和可持续发展带来了严峻的挑战。此外,不可再生化石燃料资源预计在未来40~50年内耗尽[1],因此,勘探和开发可持续、生态友好和储量大的可再生生物质资源已成为近年来的热点问题,有望克服因消耗化石燃料带来的环境污染和能源短缺等问题。木质纤维素生物质是地球上最丰富的可再生资源之一,它的年产量约为2×1011吨[2],是获得生物乙醇和替代化石燃料的理想可再生候选者之一。然而,迄今为止,大部分木质纤维素资源尚未得到有效地利用,木质纤维素资源的利用率甚至未能达到其总量的5%[3]。木质纤维素主要由木质素、纤维素和半纤维素组成,其中纤维素含量占比通常最高[4]。纤维素是生物圈中含量最丰富的有机多糖,是获取洁净能源如基于乙醇的生物燃料和高附加值化学品的主要原料[5]。因而,提高纤维素的有效利用率是提高木质纤维素资源利用率的重点。在自然界中,许多生物都能有效地降解利用木质纤维素生物质,其中能分泌碳水化合物活性酶的丝状真菌被认为是关键的初级降解者[6]。
裂解多糖单加氧酶(lytic polysaccharide monooxygenase,LPMO)是铜依赖的氧化还原酶,该酶通过氧化裂解顽固多糖链表面,使多糖易于处理以便进一步酶促作用并最终降解[7]。LPMO广泛存在于真菌、细菌、病毒和昆虫中,能催化纤维素、几丁质和淀粉等多种难降解顽固碳水化合物的氧化解聚。其在碳水化合物活性酶数据库(carbohydrate-active enzymes database,CAZy) (http://www.cazy.org/)中被划分到 AA9~AA11和AA13~AA16这7个不同的辅助活性家族(auxiliary activity family,AA family)中[8]。不同家族LPMO的氨基酸序列差异较大,但所有LPMO整体拓扑结构相似,催化结构域是免疫球蛋白样的反向平行β-三明治核心结构,活性位点位于该较为平坦的核心区域上,由两个高度保守的组氨酸与金属铜离子螯合形成的T型“组氨酸支架(histidine brace)”构成,其中一个组氨酸是氨基末端残基,另一个为侧链组氨酸[9,10]。Hemsworth等[11]发现来自米曲霉()AA11家族的LPMO(AoAA11)对晶体几丁质具有活性,表明AoAA11具有与AA9和AA10的LPMO相似的三维结构,包括核心的反向平行β-三明治结构和具有保守“组氨酸支架”的铜活性位点。Wang等[12]从富士镰刀菌()中获得截断形式的LPMO11新型酶,称为FfAA11。该酶对α-几丁质和β-几丁质以及龙虾壳均表现出氧化活性。近年来,Rieder等[13]发现烟曲霉()中的AA11B不具有假定的几丁质活性,而对N-乙酰葡糖胺的可溶性低聚物具有催化活性。Støpamo等[14]发现AfAA11A虽然缺少碳水化合物结合模块,但对α-和β-几丁质仍具有相当大的催化能力。结合以上研究可知,尽管LPMO11被假定具有几丁质酶活性,但AA11家族的部分成员对几丁质却没有催化活性。因此,LPMO11可能在真菌生理学中发挥着除几丁质酶催化活性外的其他作用[14]。
迄今为止,虽然已经有大量关于LPMO酶学的研究,但对于真菌中基因失活的研究却鲜有报道。此外,尚未有利用缺失基因的突变体进行复杂多糖如纤维素、淀粉或几丁质的降解研究,因此其功能尚未得到充分的研究。通过比对41种常见真菌的基因组发现,是含有最多降解木质纤维素蛋白酶类基因的子囊真菌[15],又因其生命周期短,易于培养和进行分子遗传操作,因此它是用来研究木质纤维素降解的良好模式生物[16]。尽管LPMOs在真菌界中广泛存在,但属于AA11家族的LPMO尚未得到充分研究。中5个基因功能仍有待进一步被揭示与研究,本研究以期为阐明LPMO11在真菌生理学中发挥的不同作用提供一定的参考价值。
本研究以为实验菌株,利用同源重组方法分别构建5个基因和缺失单突变型菌株,通过遗传杂交获得所有多基因突变体。通过突变型菌株与野生型菌株在生长速率、有性生殖、氧化应激和纤维素降解等方面的差异性分析,揭示AA11家族的基因在中的明确作用与功能,为系统阐述丝状真菌中木质纤维素降解的调控机制提供参考。
1 材料与方法
1.1 材料
(1) 菌株和质粒:野生型菌株(WT)Δmus51::phleoR菌株(用腐草霉素抗性基因代替基因编码序列所得到的菌株。基因具有修复真菌DNA突变的功能,敲除可显著提高同源重组效率[17])、Δmus51:: HygroR菌株、Δmus51:: NourR菌株和Δmus51:: GeneR菌株。质粒pBC-Geneticin、pBC-Hygromycin、pBC- Phleomycin和pBC-Nourseothricin分别用于突变体构建。
(2) 培养基:菌株培养条件以及M2基础培养基等常用培养基的配置方法可在Genome Project (http://podospora.i2bc.paris-saclay.fr/ methods.php)检索得到。木质纤维素培养基:在M2培养基的基础上,分别用木质素(lignin powder)、木屑(wood shaving)和微晶纤维素(avicel)等量取代糊精作为碳源,制备3种不同成分的木质纤维素培养基。
(3) 主要试剂和仪器:抗生素遗传霉素(geneticin)和腐草霉素(phleomycin)购自南京都莱生物技术有限公司;诺尔斯菌素(nourseothricin)购自上海吉至生化科技有限公司;潮霉素(hygromycin)、羟甲基纤维素钠(CMC-Na)和其他常用试剂均购自上海生工生物工程股份有限公司;Go Taq®DNA Polymerase、Ex Taq®DNA Polymerase、PrimeSTAR®Max DNA Polymerase均购自TaKaRa;2×Phanta®Max Master Mix(Dye Plus)和Plasmid Mini Kit均由南京诺唯赞生物科技股份有限公司生产;Gel Extraction Kit和Cycle-Pure kit购自OMEGA;溶壁酶购自Sigma;M5 Fungal Genomic DNA Kit购于北京聚合美生物科技有限公司;二氨基联苯胺(diaminobenzidine,DAB)显色试剂盒和氯化硝基四氮唑蓝(nitro blue tetrazolium,NBT)试剂购自Solarbio,4-硝基苯基-β-D-吡喃葡萄糖苷(pNPG)和4-硝基苯基-β-D-纤维二糖(pNPC)均由Aladdin生产。
1.2 PaLPMO11生物信息学分析
从基因组数据库和CAZy数据库中获得5个假定的基因、、、和,分别命名为和,对应的氨基酸序列为PaLPMO11A (XP_001906381.1)、PaLPMO11B (XP_001912736.1)、PaLPMO11C (XP_001911580.1)、PaLPMO11D (XP_001911666.1)和PaLPMO11E (XP_001909751.1)。在CAZy数据库和NCBI基因组数据库(https://www.ncbi.nlm.nih.gov/)中检索得到7条来自真菌子囊菌和轮虫的LPMO11蛋白序列。分别为:(BAE61530.1)、(XP 748042.1)、(CCT67099.1)、(EAA31701.1)、(XP 750980.1)、(EAA35129.1)和(UJR20201.1)。通过分子进化遗传学分析软件MEGA 11.0对12个蛋白质序列进行分析建树,使用muscle算法对编译后的序列进行比对,并手动调整,采用MEGA 11.0中的邻近法(neighbor-joining method)构建基于这些序列的系统进化树,利用自展法(bootstrap)设置1000次重复检验。使用I-TASSER在线网站(https://zhanglab. ccmb.med.umich.edu/I-TASSER/)和UCSF Chimera等软件对5个目的基因的蛋白三级结构和保守氨基酸位点进行预测。
1.3 构建PaLPMO11家族突变体
运用Split-marker[18]方法在WT菌株中分别敲除和五个目的基因。构建AA11家族5个单突变型菌株技术原理如图1所示。常规PCR分别扩增出目的基因上下游的同源臂片段(约1000 bp)以及相对应的抗性标记基因片段。其中选用nourseothricin标记,选用hygromycin B标记,和选用geneticin标记,选用phleomycin标记。融合PCR对常规PCR纯化后得到的目的基因上下游的同源臂片段和抗性标记基因片段进行融合连接,构建敲除表达盒(引物序列如表1所示)。将融合片段转化至ΔPaMus51菌株的原生质体后,分别使用含有nourseothricin、hygromycin、geneticin或phleomycin抗性的抗性平板筛选抗性稳定的转化子。转化子与WT菌株杂交,筛选出具有目的基因抗性而不具有原生质体抗性的小孢子,即获得可稳定遗传的突变型菌株。PCR和基因测序验证5个单突变型菌株是否构建成功。使用相应的不同交配型菌株进行遗传杂交,杂交后筛选同时具有相应抗性的小孢子,以此构建AA11家族的10个双重突变型菌株、10个三重突变型菌株、5个四重突变型菌株和五重突变型菌株,PCR验证具有相同抗性双重突变型菌株ΔAΔD和五重突变体菌株是否构建成功。
1.4 表型分析
(1) M2培养基培养:将不同交配型的WT和31个AA11家族突变型菌株接种在M2固体培养基上,并于光照条件下27℃恒温培养1周。在培养过程中观察并记录各个菌株的生长速率、菌落大小、菌落形态、色素沉着、子实体的形成以及子囊孢子的产生、喷发和萌发情况,进行显微观察并记录子实体的数量。每个实验重复3次。
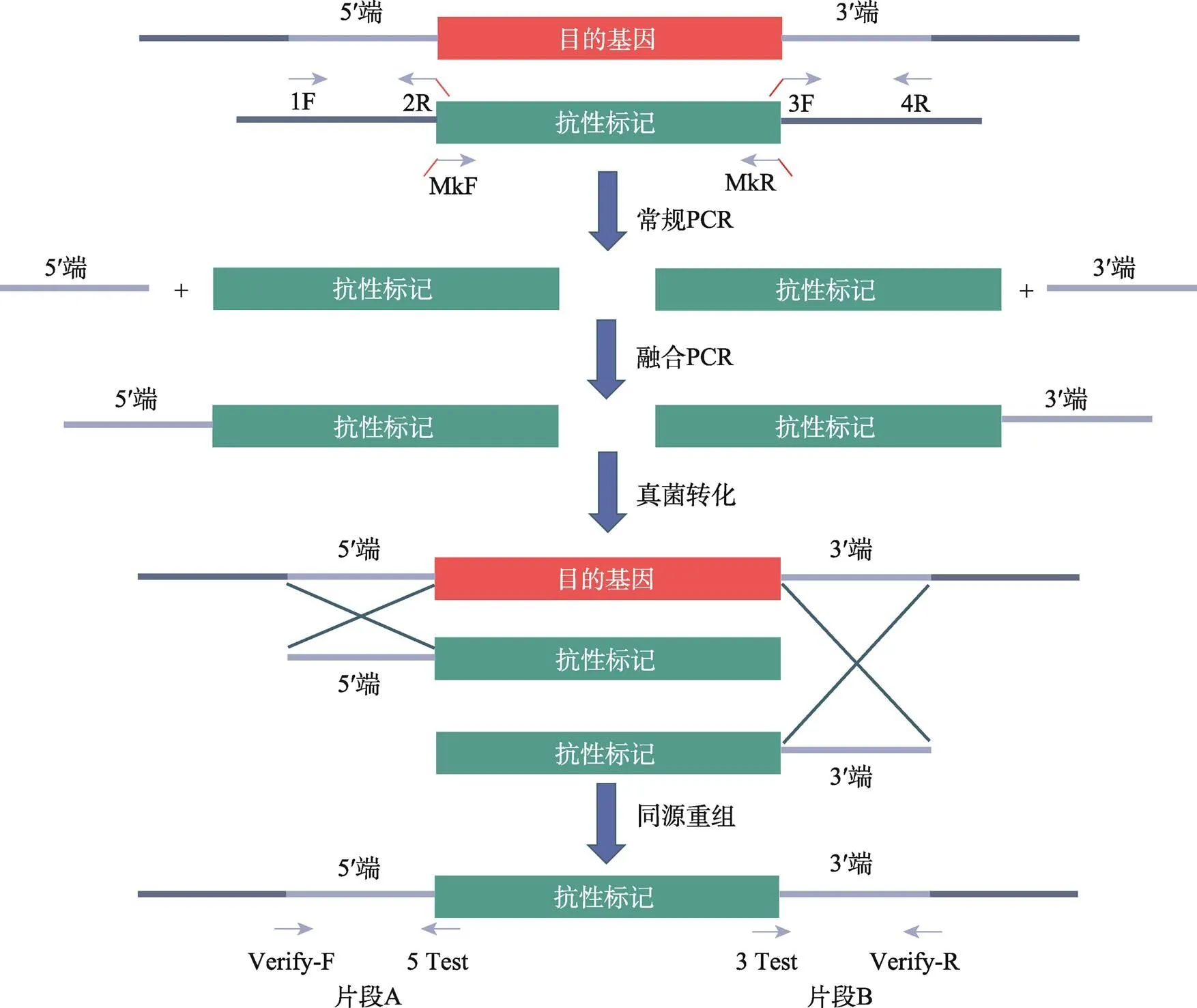
图1 构建PaLPMO11家族突变体的流程图
(2) 木质纤维素培养基培养:将不同交配型的WT和突变型菌株混匀破碎,分别接种于添加了木质素、木屑和微晶纤维素的M0培养基上,于27℃光照条件下培养两周。在培养过程中观察并记录各个菌株的生长速率、菌落大小、菌落形态、色素沉着、子实体的形成以及子囊孢子的产生、喷发和萌发情况。每个实验重复3次。
1.5 菌丝ROS水平分析
将WT、ΔE和ΔAΔBΔCΔDΔE不同交配型菌株混匀破碎后分别接种在M2培养基上,于27℃黑暗培养3天,分别使用DAB染色试剂盒和NBT染色试剂对菌丝进行染色,检测菌丝组织中过氧化物和超氧化物的积累[19]。在过氧化物酶的存在下,过氧化氢与DAB反应会生成棕褐色沉淀;NBT在超氧化物酶催化下,与超氧离子反应生成蓝紫色沉淀。菌丝染色强度与过氧化物和超氧化物的积累成正比关系[20]。置于水平摇床避光孵育3 h,菌丝体出现颜色变化时除去染色液,使用PBS洗涤3次,终止显色反应。观察比对各菌株菌丝染色程度。每个实验重复3次。
1.6 纤维素酶活测定
将不同交配型的WT、ΔE和ΔAΔBΔCΔDΔE菌株分别接种到纤维素诱导培养基上生长,以促进纤维素作用酶的分泌。27℃、150 r/min、光照条件下培养3天、5天、7天、9天,取上清液离心后即得粗酶液。参照国标QB2583-2003及Dashtban[21]和Thankappan[22]的方法对滤纸酶活(filter paper activity,FPA)、内切葡萄糖苷酶活(endoglucosidase activity,EG activity)、外切葡聚糖酶活(cellobiohydrolase activity,CBH activity)和β-葡萄糖苷酶活(β-D-glucosidase activity,BG activity)进行测定,具体操作方法如下。每组样品均设置3组重复。
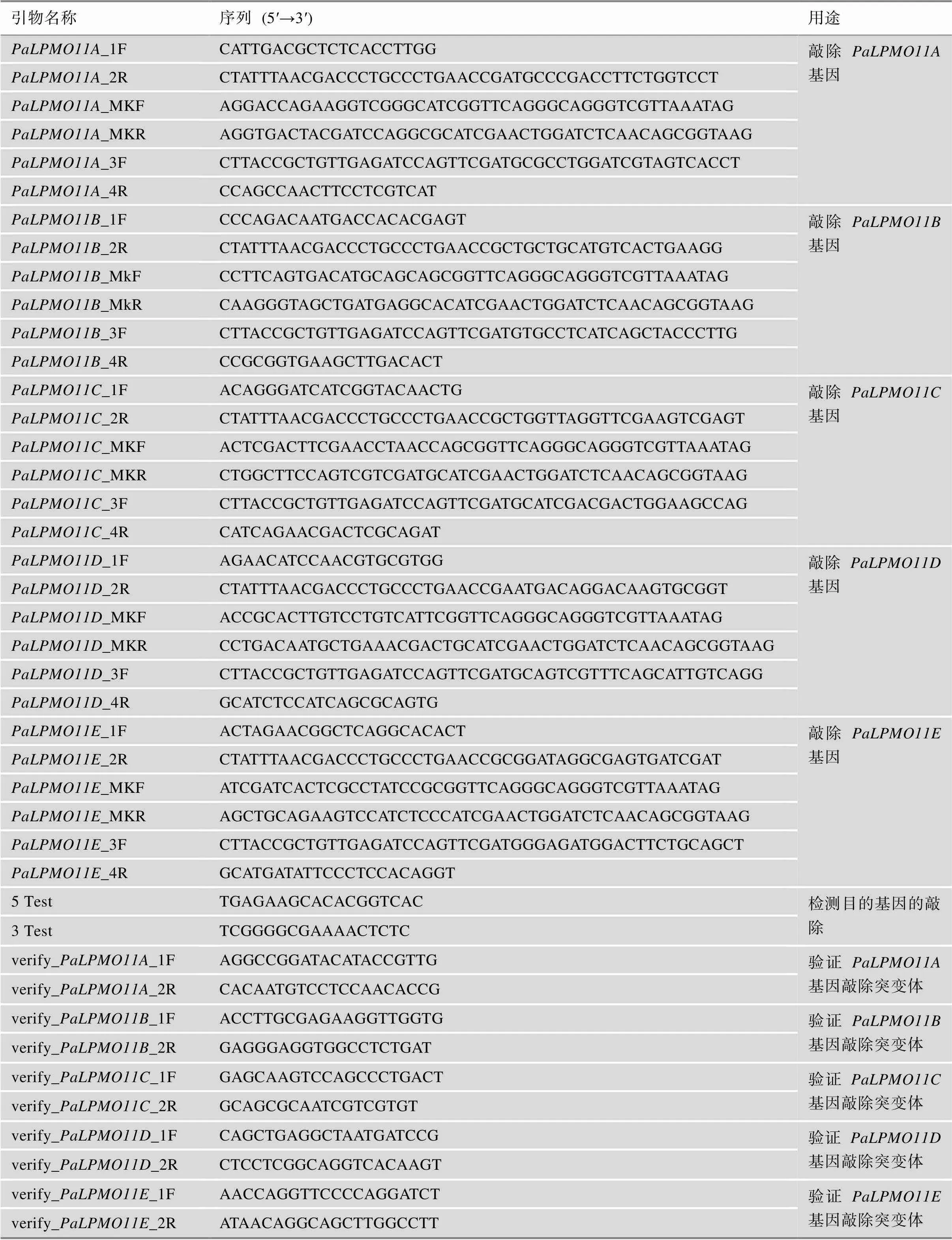
表1 本研究使用的引物
1.6.1 FPA和EG activity测定
FPA和EG酶活单位(U)定义为:在实验反应条件下,每分钟释放1 µmol葡萄糖所需的酶量。纤维素酶可以降解纤维素底物,生成还原性单糖。还原性单糖与3,5-二硝基水杨酸(DNS)沸水浴后发生显色反应,生成棕红色氨基化合物。在一定范围内,反应颜色深浅与酶解反应单位时间内释放的还原性单糖浓度呈正比,在540 nm波长处可以检测到其吸光度。根据葡萄糖标准曲线计算酶解反应释放的还原性单糖含量,从而计算酶活。FPA实验以滤纸为反应底物,EG activity实验以CMC-Na为反应底物。将粗酶液与底物混合,50℃水浴1 h,进行反应。加入DNS后轻轻混合,沸水浴5 min后立即置于冰上冷却,取200 μL反应液于96孔板中,540 nm处测定吸光度。每个样品均设置空白对照组。
1.6.2 CBH activity和BG activity测定
CBH和BG酶活单位(U)定义为:在实验反应条件下,每分钟释放1 µmol硝基苯酚(pNP)所需的酶量。无色底物pNPG 和pNPC在β-葡萄糖苷酶和外切葡聚糖酶的催化下,水解成有色物质pNP,在405 nm波长处有最大吸收峰。根据pNP标准曲线计算酶解反应释放的pNP含量,从而计算酶活。将粗酶液与底物混合,50℃水浴30 min,进行反应。水浴后立即置于冰上冷却,向各离心管中加入2% Na2CO3终止反应。取200 μL反应液于96孔板中,405 nm处测定吸光度。每个样品均设置空白对照组。
2 结果与分析
2.1 PaLPMO11家族蛋白的生物信息学分析
2.1.1 PaLPMO11的系统发育分析
真菌米曲霉(4MAI_A)和烟曲霉(7P3U_A)编码的LPMO11蛋白相似度为39.63%,由此推测AA11家族LPMO氨基酸序列差异虽然较大,但构成组氨酸支架的两个组氨酸和AA11家族独有的轴位点酪氨酸侧链均严格保守,该酪氨酸侧链不直接参与金属铜离子的配位,而是与其附近的丙氨酸相互作用[11]。5个基因编码的蛋白PaLPMO11A、PaLPMO11B、PaLPMO11C、PaLPMO11D和PaLPMO11E与AoAA11蛋白以及AfAA11蛋白均有较高的一致性(均在27.9%以上)。与AoAA11蛋白相似度分别为37.04%、27.93%、35.94%、45.07%和40.64%;与AfAA11蛋白相似度分别为37.43%、49.02%、48.6%、38.39%和49.05%。因此,推测5个编码的蛋白可能是与AoAA11和AfAA11蛋白类似的裂解多糖单加氧酶。使用MEGA 11.0对12个LPMO11蛋白构建系统发育树(图2A)。系统发育分析显示,PaLPMO11D和AoAA11、AfAA11B和FfAA11亲缘关系近;AfAA11A和PaLPMO11C处于同一分支上,表明亲缘关系最近;其次是与PaLPMO11E关系较近;而PaLPMO11A和PaLPMO11B位于该进化树的最外侧。由图2B可知,5个PaLPMO11蛋白在催化位点核心保守区域显示出较高相似度,预测两个高度保守的组氨酸会与金属铜离子螯合成T形的组氨酸支架,从而构成催化活性位点[23]。而PaLPMO11B在多序列比对结果中显示缺少轴位点酪氨酸侧链,因此在预测蛋白三级结构中,无法预测出其催化活性位点的保守氨基酸所处位置(图3)。根据系统发育分析结果和多序列比对结果显示,推测5个PaLPMO11蛋白可能分属于AA11家族的不同亚家族。
2.1.2 PaLPMO11蛋白的三级结构模型预测
和基因编码蛋白的三级结构与活性位点保守氨基酸预测如图3所示。5个PaLPMO11蛋白预测具有与AoAA11蛋白相似的整体拓扑结构,均具有典型的反向平行β-三明治核心结构。将除PaLPMO11B外的4个PaLPMO11预测蛋白的催化活性位点的保守氨基酸与AoAA11蛋白的活性位点的保守氨基酸进行比较,所有原子的均方根偏差(root mean square deviation,RMSD)在0.77~0.97 Å范围内显示出较高水平的结构重叠。根据系统进化分析、多序列比对和蛋白三级结构模型预测结果推测,的5个LPMO11蛋白可能是与AoAA11类似的蛋白,可以通过氧化还原反应裂解糖苷键,从而降解某种顽固多糖[11]。
A: PaLPMO11蛋白的系统发育树。AA11蛋白氨基酸序列来源于CAZy数据库和NCBI网站,基因库序列号显示在系统发育树中。B: LPMO11s和PaLPMO11s的部分氨基酸序列比对。三角形表示参与铜离子配位的两个绝对保守的组氨酸残基,以及AA11家族特有的轴向酪氨酸残基。
2.2 ΔPaLPMO11突变体的构建
2.2.1五个基因敲除表达盒的构建
按1.3构建PaLPMO11家族突变体所述方法构建突变体。利用4种不同的抗性标记geneticin、nourseothricin、hygromycin和phleomycin代替5个基因序列,敲除这5个基因,并对5个单突变体进行抗性验证和PCR验证。以WT基因组DNA为模板,PCR扩增得到5个基因上游片段PaLPMO11-5′和下游片段PaLPMO11-3′约1000 bp片段。以质粒pBC- Hygromycin、pBC-Nourseothricin、pBC-Geneticin或pBC-Phleomycin DNA为模板,扩增获得HygroR基因片段(3657 bp)、NourR基因片段(1773 bp)、GeneR基因片段(2342 bp)或PhleR基因片段(3100 bp)。将各个基因的上下游片段和抗性标记基因片段进行融合PCR扩增,分别构建的敲除表达盒-5′-HygroR和HygroR--3′;的敲除表达盒-5′-NourR和NourR--3′;的敲除表达盒-5′-GeneRand GeneR--3′;的敲除表达盒-5′-PhleR和PhleR--3′以及的敲除表达盒-5′-GeneR和GeneR--3′。5个基因敲除表达盒的构建结果见附图1和附图2。通过原生质体转化方法,将敲除表达盒转化到ΔPaMus51菌株的原生质体中,筛选具有相应抗性的转化子,利用两对验证引物(verify__1F和5T、verify__2F和3T)对筛选出来的转化子进行PCR验证(验证引物序列见表1)并纯化,扩增得到的同源重组片段与目的序列大小一致,序列比对结果与目标序列一致。以上结果均表明,目的基因片段与抗性基因片段在目的敲除基因位点发生同源重组置换,证实抗性标记基因成功整合在目的基因座中,成功构建PaLPMO11家族5个基因缺失菌株。
A: 目标蛋白三级结构预测。B:PaLPMO11蛋白与AoAA11蛋白的整体叠加。AoAA11蛋白用黄色标注,PaLPMO11蛋白用蓝色标注。C:PaLPMO11蛋白(蓝色)与AoAA11蛋白(黄色)的活性位点的重叠。
2.2.2 ΔPaLPMO11突变体的PCR验证
单突变体构建成功后,通过遗传杂交构建10个双突变型菌株ΔAΔB(ΔPaLPMO11AΔΔPaLPMO11B)、ΔAΔC、ΔAΔD、ΔAΔE、ΔBΔC、ΔBΔD、ΔBΔE、ΔCΔD、ΔCΔE和ΔDΔE,10个三重突变型菌株ΔAΔBΔC、ΔAΔBΔD、ΔAΔBΔE、ΔAΔCΔD、ΔAΔCΔE、ΔAΔDΔE、ΔBΔCΔD、ΔBΔCΔE、ΔBΔDΔE和ΔCΔDΔE,5个四重突变型菌株ΔAΔBΔCΔD、ΔAΔBΔCΔE、ΔAΔBΔDΔE、ΔAΔCΔDΔE和ΔBΔCΔDΔE以及缺失所有PaLPMO11家族基因的五重突变体菌株ΔAΔBΔCΔDΔE。具有相同抗性双重突变型菌株ΔAΔD和五重突变体菌株ΔAΔBΔCΔDΔE均通过PCR验证。ΔPaLPMO11突变体的PCR验证结果见附图3。
2.3 PaLPMO11家族基因的缺失影响P. anserina利用碳源的能力
2.3.1 菌株在M2培养基上的生长发育
M2培养基是以糊精作为唯一碳源、能满足生长和发育的基础培养基。将WT和突变型菌株接种到M2培养基上,27℃培养1周。除ΔAΔBΔCΔDΔE外的突变型菌株均不受影响,没有发现WT和其他突变型菌株在菌落大小、形态和色素沉着、子实体形成以及子囊孢子产生、喷发和萌发方面存在明显差异(数据未显示)。而ΔAΔBΔCΔDΔE出现色素沉着的时间和子实体生成时间与其他菌株出现差异,且菌落大小、菌丝密度、子实体形成数量及子囊孢子喷发数量都远低于WT (图4)。与WT相比,ΔAΔBΔCΔDΔE的生长有最显著的延迟,且提前进入衰老状态。培养第7天起,ΔAΔBΔCΔDΔE开始停止生长并伴随明显的色素沉着。结果表明,PaLPMO11家族单个基因在的生长发育过程中似乎没有发挥关键作用,但5个基因同时敲除,会引起菌株生长发育和有性生殖明显延缓并导致菌株提前衰老。
2.3.2 菌株在木质素培养基上的生长发育
能够高效利用木质纤维素作为碳源,通过分泌胞外酶使其能够有效地渗透和降解植物生物质[24]。为了确定PaLPMO11家族基因在木质纤维素利用中的作用,将WT与31个突变型菌株进行表型分析比较。用木质素、木屑和微晶纤维素等量代替糊精作为唯一的碳源,评估突变体利用木质纤维素的能力。
在以纯木质素作为唯一碳源的培养基上培养时,WT菌丝呈纺丝状生长,在培养基上只形成菌丝体,而不生成子实体[25]。除ΔAΔBΔCΔDΔE外,其余菌株生长速度很快,菌丝体在培养6天后几乎覆盖了整个培养基。在木质素平板上,ΔAΔBΔCΔDΔE的生长直径与菌丝密度明显减少(图5),在培养5天时,生长直径比WT减少了19%;在培养7天时,直径减少了近31%。结果表明,ΔAΔBΔCΔDΔE利用木质素的能力降低。除ΔAΔBΔCΔDΔE外,其他突变型菌株的生长与WT几乎没有差异。这表明,5个基因之间可能在降解木质素以获取营养物质的过程中具有补偿效应,在PaLPMO11家族基因一个或多个基因缺失的情况下,菌株的生长状态并没有显著改变,而PaLPMO11家族的基因全部缺失时,菌株的生长和菌丝量都会相应地下降减少。
2.3.3 菌株在木屑培养基上的生长发育
在以木屑为唯一碳源的培养基上培养时,除ΔAΔBΔCΔDΔE外,WT和其他突变型菌株的子实体在培养6天后开始发育,数量众多,并在2天后喷发具有活性的子囊孢子。且除ΔAΔBΔCΔDΔE以外,其他突变型菌株的子实体萌发时间、生长速率和菌丝密度与WT并无明显差异。而5个基因的同时缺失会导致菌株生长速率延缓,子囊孢子的形成时间延迟,子实体的产生显著减少,这与M2基础培养基上的观察结果一致。
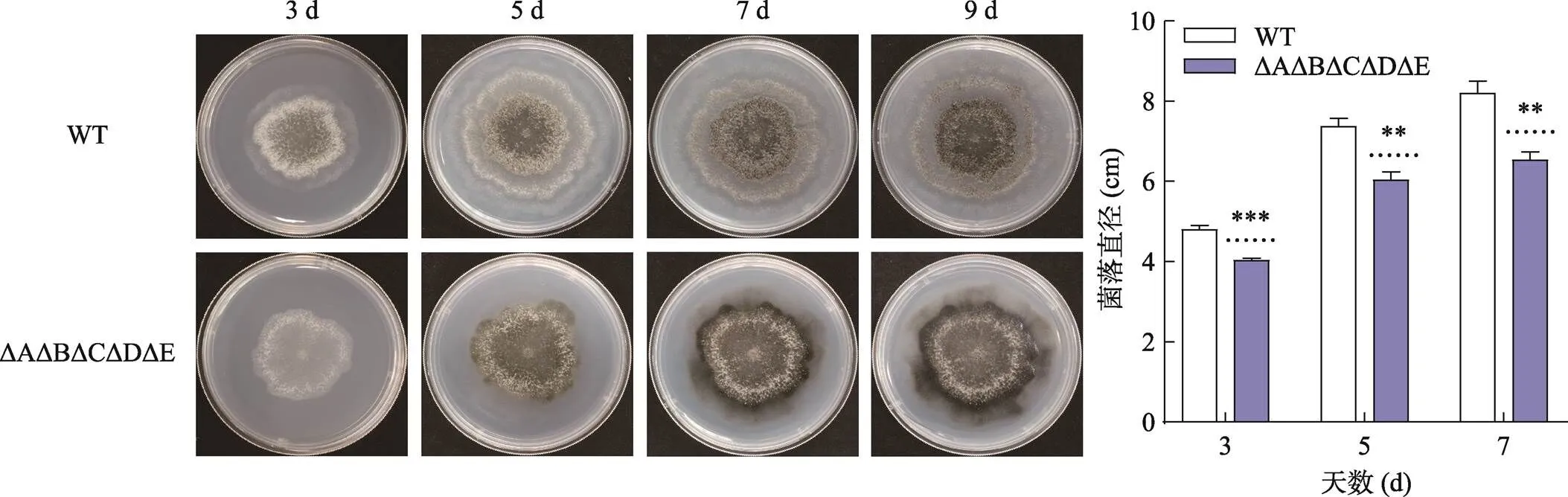
图4 WT与ΔAΔBΔCΔDΔE菌株在M2培养基上的生长发育情况
将不同交配型菌株混合破碎,取5 µL菌悬液滴在M2培养基中央,27℃光照培养3天(d)后,拍照并记录菌株生长直径。野生型与其他突变株间生长直径并无显著差异。检验:*<0.05,**<0.01,***<0.001。
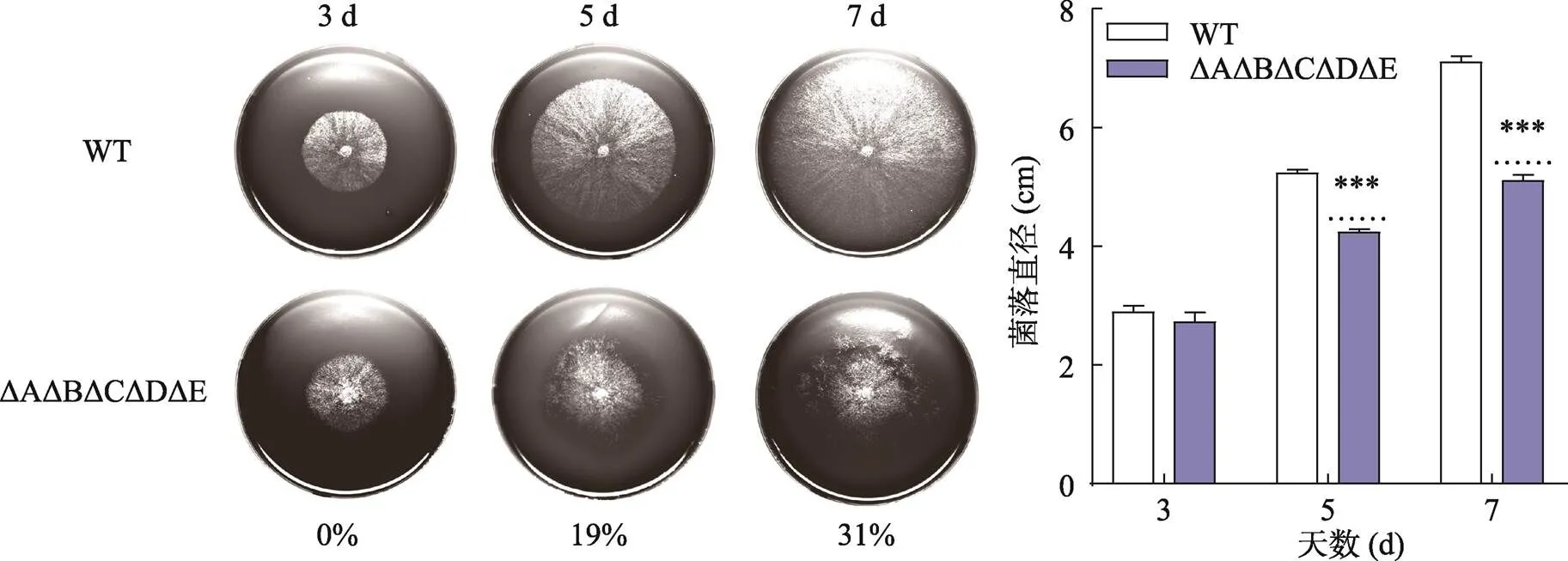
图5 WT与ΔAΔBΔCΔDΔE菌株在木质素培养基上的生长发育情况
将不同交配型菌株混合破碎,取5 µL菌悬液滴在木质素培养基中央,27℃光照培养3天(d)后,拍照并记录菌株生长直径。图中数字表示突变型菌株与野生型菌株相比,生长直径减少的百分比。野生型与其他突变株间生长直径并无显著差异。检验:*<0.05,**<0.01,***<0.001。

图6 WT与ΔAΔBΔCΔDΔE菌株在木屑培养基上的生长发育情况
将不同交配型菌株混合破碎,取5 µL菌悬液滴在木屑培养基中央,27℃光照培养9天(d)后,拍照并观察培养基上及培养皿盖上的子实体密度。子实体为可见的黑色小点。野生型与其他突变株间子实体数量并无明显差异。
丝状真菌的子实体是区分营养生殖和有性生殖的重要生殖器官[26];在真菌生命周期中,子实体的形成是最为复杂的发育过程之一,需要消耗大量的能量和营养物质[27,28]。观察喷发到培养基盖子上的子囊孢子密度(图7A),发现敲除基因的突变型菌株喷发的孢子数明显少于WT和其他突变体,ΔAΔBΔCΔDΔE的产孢数是最少的(图片未完全展示)。为了量化菌株产孢数,对WT和敲除基因的16个突变型菌株进行子实体数量统计与显微镜观察(图7B)。每个菌株均设置了3组重复。由图7D可知,ΔBΔCΔE子实体数量为5739,ΔAΔBΔCΔE子实体数量为5296,ΔAΔCΔDΔE子实体数量为4812,ΔAΔBΔCΔDΔE子实体数量为379,与WT的子实体数量7216相比,分别减少了20%、27%、33%和95%,均存在显著差异。统计上述菌株子囊孢子的萌发率,WT菌株子囊孢子萌发率为95.5%,ΔBΔCΔE子囊孢子萌发率为92%,ΔAΔBΔCΔE子囊孢子萌发率为81.5%,ΔAΔCΔDΔE子囊孢子萌发率为70%,ΔAΔBΔCΔDΔE子囊孢子萌发率为51.5% (图7E)。子实体是真菌体内的产孢结构,孢子在子实体内形成并于成熟后喷发,是真菌进行繁殖的主要途径[29]。ΔAΔBΔCΔDΔE菌株进入有性生殖阶段后,会生成部分异常的子实体,部分子实体无内容物,不含有活性的子囊孢子(图7C),因而无法正常排出子囊孢子,导致喷发到培养基盖子上的子囊孢子密度远小于其他菌株。实验结果显示,基因缺失的突变体的子囊孢子萌发率和子实体数量均呈现下降趋势,说明基因可能与子囊孢子萌发和子实体形成有关;除ΔAΔBΔCΔDΔE外,WT和其余突变体的生长速率和菌丝密度无明显差异。
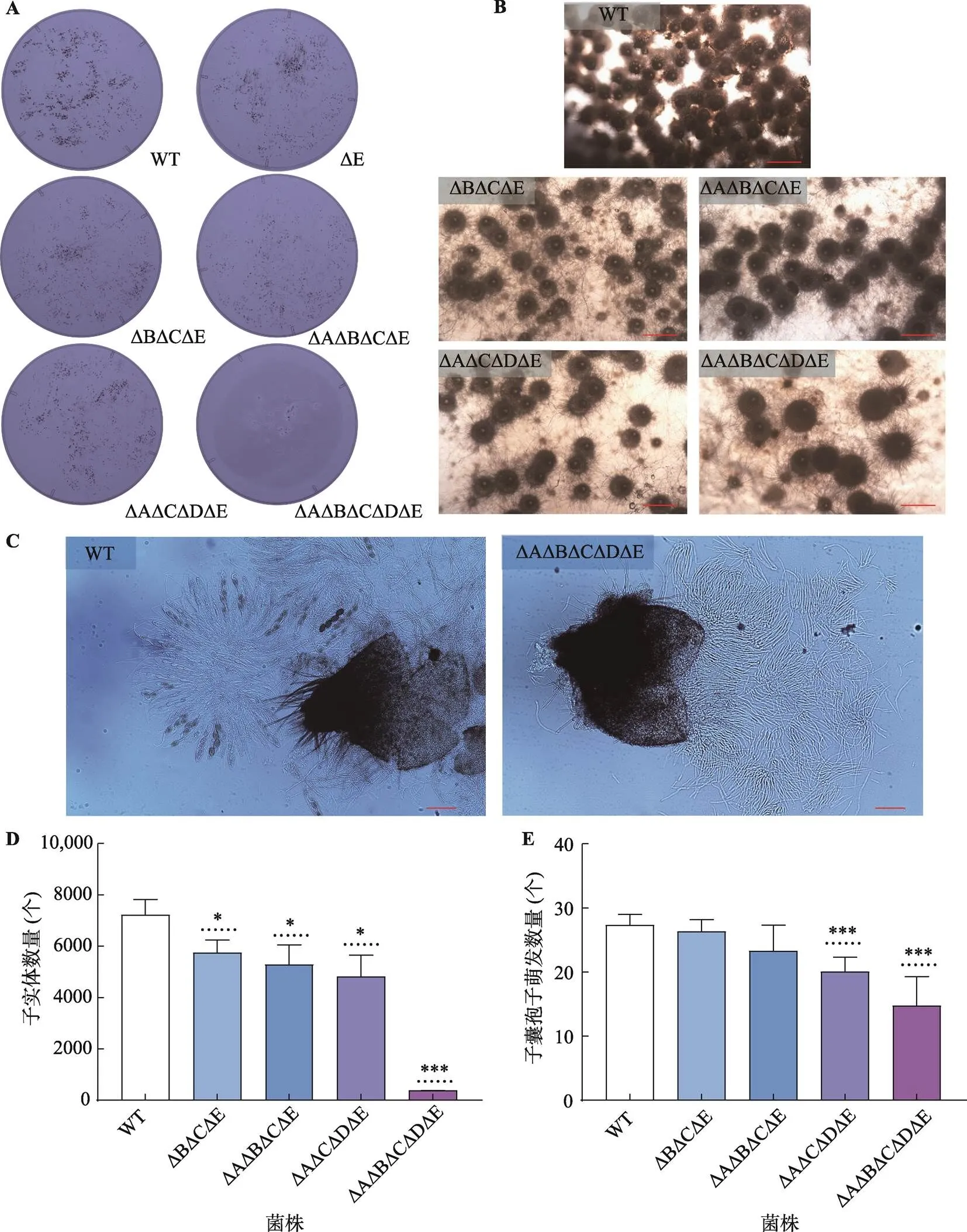
图7 WT和部分PaLPMO11E基因缺失突变型菌株的子囊孢子密度图、显微观察、子实体数量统计与子囊孢子萌发率统计
A:野生型和部分基因缺失突变型菌株的子囊孢子密度图。菌株于木屑培养基上培养8天后会将孢子喷发至培养皿盖子上,对接有子囊孢子的培养皿盖拍照。B:野生型和突变型菌株于倒置显微镜下的观察结果。不同交配型菌株于M2培养基27℃光照培养5天后,置于倒置显微镜下观察子实体形态与数量。标尺:500 µm。C:WT及ΔΔBΔCΔDΔE子实体放大图。在M2培养基27℃光照培养5天后,挑取子实体置于载玻片上,用盖玻片盖上碾压,置于倒置显微镜下观察并拍照。标尺:100 µm。D:子实体数量统计。不同交配型菌株在M2培养基上27℃光照7天后,在显微镜下对野生型和突变型菌株的子实体数量进行计数统计。E:子囊孢子萌发率统计。将不同交配型菌株在M2培养基上进行杂交,培养7天后开始喷发子囊孢子,用接种孢子培养基接住喷发出来的子囊孢子。每个菌株挑取200个子囊孢子于萌发孢子培养基上黑暗培养1天后,统计子囊孢子的萌发率。检验: *<0.05,**<0.01,***<0.001。
这表明基因可能参与了有性生殖过程,而PaLPMO11家族5个基因的同时缺失,会导致菌株营养生长和有性生殖过程进一步受损,不仅表现为菌落的生长直径明显降低和子实体数量大幅减少,还表现为生成异常子实体,导致菌株的低育性。由此推测上述突变型菌株可能无法从木屑中正常获取足够的碳源,利用营养物质的能力受损,导致菌株营养生长期间能量储备不足,无法正常供应有性生殖过程的能量需求,从而造成菌株有性生殖过程部分受损,其中ΔAΔBΔCΔDΔE的表型最为明显。
2.3.4 菌株在微晶纤维素平板上的生长发育
在以微晶纤维素为唯一碳源的培养基培养时,野生型菌株生成的子实体以接种点为中心呈环状分布[20],通过计算培养皿上的成熟子实体数量来衡量菌株的生育力。由图8A可知,敲除基因的突变型菌株形成更大更弥散的子实体环,子实体分布呈现发散状,且子实体数量少于WT,这一点与菌株在木屑平板上生长得到的结果一致。这一表型在ΔAΔBΔCΔDΔE菌株上尤为明显,ΔAΔBΔCΔDΔE的子实体数量与WT相比下降了19倍。在其他突变型菌株中没有观察到这种表型,它们所生成的子实体环与WT无太大差异,均在培养皿中央生成成熟的子实体环。实验结果表明,基因可能在的有性生殖过程中发挥了作用,基因缺失菌株会形成一个更大更弥散的子实体环,子实体密度减少(图8B)。由实验结果推测,基因缺失菌株可能无法充分利用微晶纤维素碳源以维持自身生长发育所需,导致有性生殖过程在一定程度上受到影响,因而生成的子实体数量减少。PaLPMO11家族基因的同时敲除,会对的生长发育和有性繁殖造成更大程度的影响,导致菌株生长发育明显延缓,生成空心子实体,子实体数量与子囊孢子萌发率显著下降。而PaLPMO11家族基因间可能发挥相互代偿的作用,一个基因或多个基因缺失时,PaLPMO11家族的其他基因可能发挥类似的功能,以代偿其他基因的缺失,从而保证菌株可以维持正常的生理过程。
2.4 过量生成的过氧化氢可能加速了菌株的衰老
活性氧(ROS)是细胞有氧代谢过程中产生的副产物,主要由超氧阴离子自由基和过氧化氢组成[30]。生物体内存在一系列抗氧化系统使ROS的产生和消除处于平衡状态,当ROS在生物体内过量产生时,会开始攻击细胞内脂质、蛋白质和DNA,导致细胞损伤和凋亡[30,31]。对WT、ΔE和ΔAΔBΔCΔDΔE菌株菌丝的ROS水平进行分析,染色结果如图9所示。NBT染色观察发现,WT和ΔE菌丝的蓝紫色显色最深,且WT和ΔE之间菌丝染色并无明显差异,而ΔAΔBΔCΔDΔE蓝紫色染色最浅,说明ΔAΔBΔCΔDΔE超氧化物的分泌量明显减少。DAB染色观察发现,除培养第1天外,DAB染色结果与NBT染色结果一致,WT和ΔE染色程度无差异,均呈现明显的棕褐色,ΔAΔBΔCΔDΔE菌丝染色程度最浅。培养1天时,ΔAΔBΔCΔDΔE菌丝密度低于WT,但其DAB染色程度却深于WT,结果表明ΔAΔBΔCΔDΔE菌株可能在培养第1天时分泌了大量的过氧化氢。由上述结果推测,单突变型菌株体内可能存在其他相似功能的基因能弥补其缺失,因而其染色结果与WT无明显差异。再结合在M2和木质素平板上的表型分析结果,ΔAΔBΔCΔDΔE菌株均出现提前衰老的现象。由此推测,ΔAΔBΔCΔDΔE衰老的潜在机制可能是由于菌株生长前期体内积累了过量过氧化氢,而过量的过氧化氢可能会导致菌株进入过度氧化应激状态,导致分子氧化损伤增加,从而促进了突变菌株的衰老过程,导致突变型菌株提前进入衰老状态。
2.5 PaLPMO11基因参与纤维素降解过程
纤维素可以用于乙醇生产等工业生物炼制应用,而纤维素的降解利用却受到如物化特性、结构特点以及组成成分等因素的阻碍[32,33]。因此,提高木质纤维素的降解效率、减少工业生产成本对于木质纤维素的工业应用具有重要意义。滤纸酶活是包括内切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶三种纤维素降解酶在内的总纤维素酶活[34]。单突变体ΔE的四种纤维素酶活力与WT差异不大,而ΔAΔBΔCΔDΔE的酶活降低最为显著。培养7天时,ΔAΔBΔCΔDΔE的FPA酶活是WT酶活的53%,EG酶活是WT酶活的61%,CBH酶活是WT酶活的61%,BG酶活是WT酶活的32%;菌株培养9天时,ΔAΔBΔCΔDΔE的FPA活性、EG活性、CBH活性和BG活性分别是WT酶活的45%、58%、36%和35% (图10)。实验结果表明,单个基因的缺失,对总纤维素酶活影响不明显,而5个基因的同时缺失,总纤维素酶活显著下降,但却仍有野生型45%以上的活力,说明基因之间可能存在功能冗余,且5个基因功能可能与纤维素降解有关。
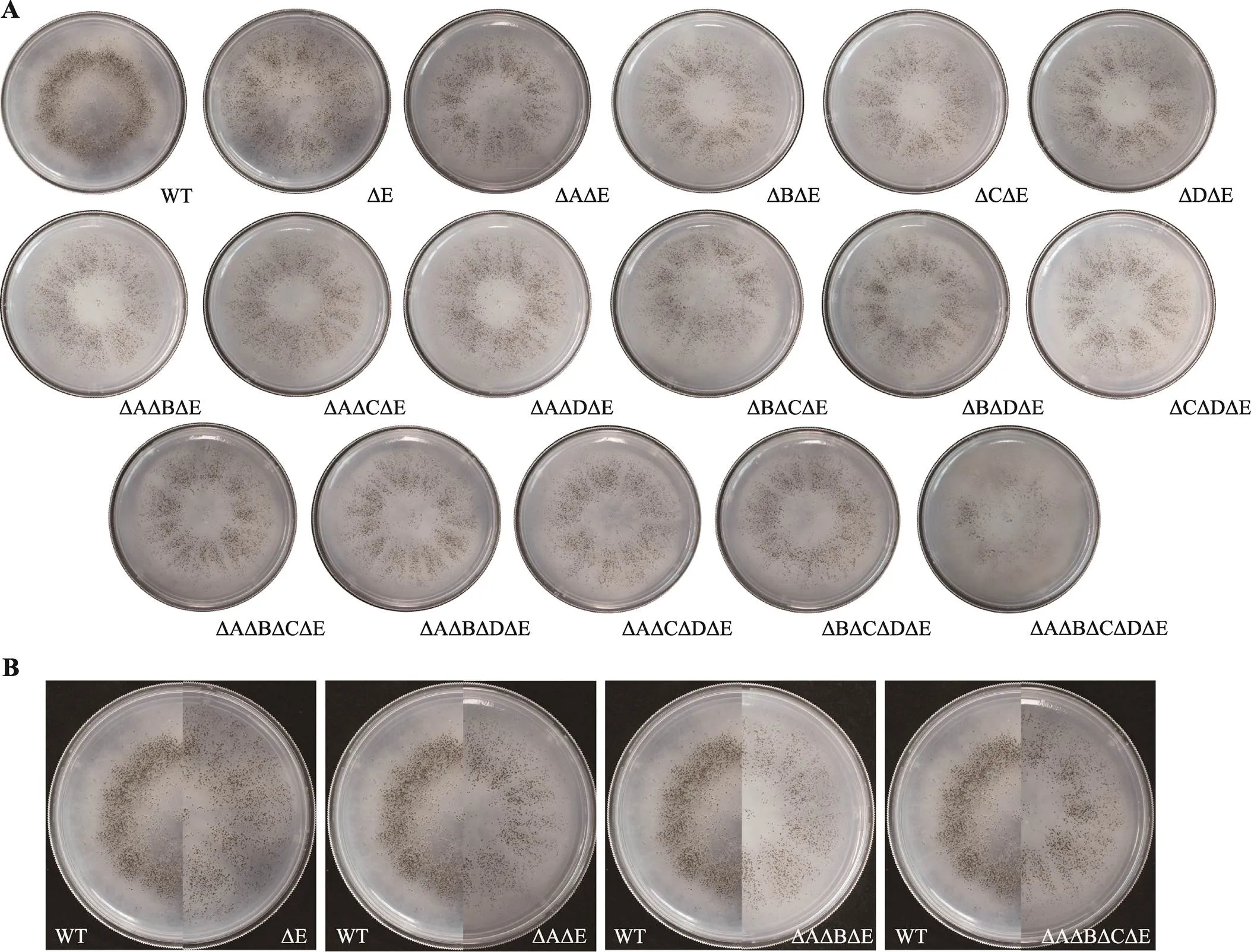
图8 WT与突变型菌株在微晶纤维素培养基上的生长发育情况与对比图
子实体为可见的黑色小点。将不同交配型菌株混合破碎,取5 µL菌悬液滴在微晶纤维素培养基中央,培养8天后拍照。除基因缺失突变型菌株外,未发现野生型与其他突变株间子实体数量与分布存在显著差异。A:野生型和基因缺失突变型菌株于微晶纤维素培养基培养的结果图。B:野生型和突变型菌株放大对比图。
3 讨论
目前仅有来自米曲霉、富士镰刀菌和烟曲霉的LPMO11蛋白被纯化和表征[11~13]。所有已知真菌的LPMO均具有保守的中央反向平行β-折叠核心结构,而暴露在平坦核心结构表面的金属铜离子会与两个高度保守的组氨酸提供的3个氮原子配位,形成T形的组氨酸支架,从而构成催化活性位点[10,35]。全基因组序列中存在5个可能编码LPMO11蛋白序列的基因,分别为和。进行生物信息学分析发现,5个PaLPMO11蛋白均含有构成催化活性位点的两个高度保守的组氨酸残基和β-三明治折叠核心结构,而PaLPMO11B缺乏AA11家族独有的轴位点酪氨酸侧链。因此推测5个PaLPMO11蛋白可能属于AA11家族的不同亚家族,可以通过氧化还原反应裂解糖苷键,从而降解某种难降解多糖。
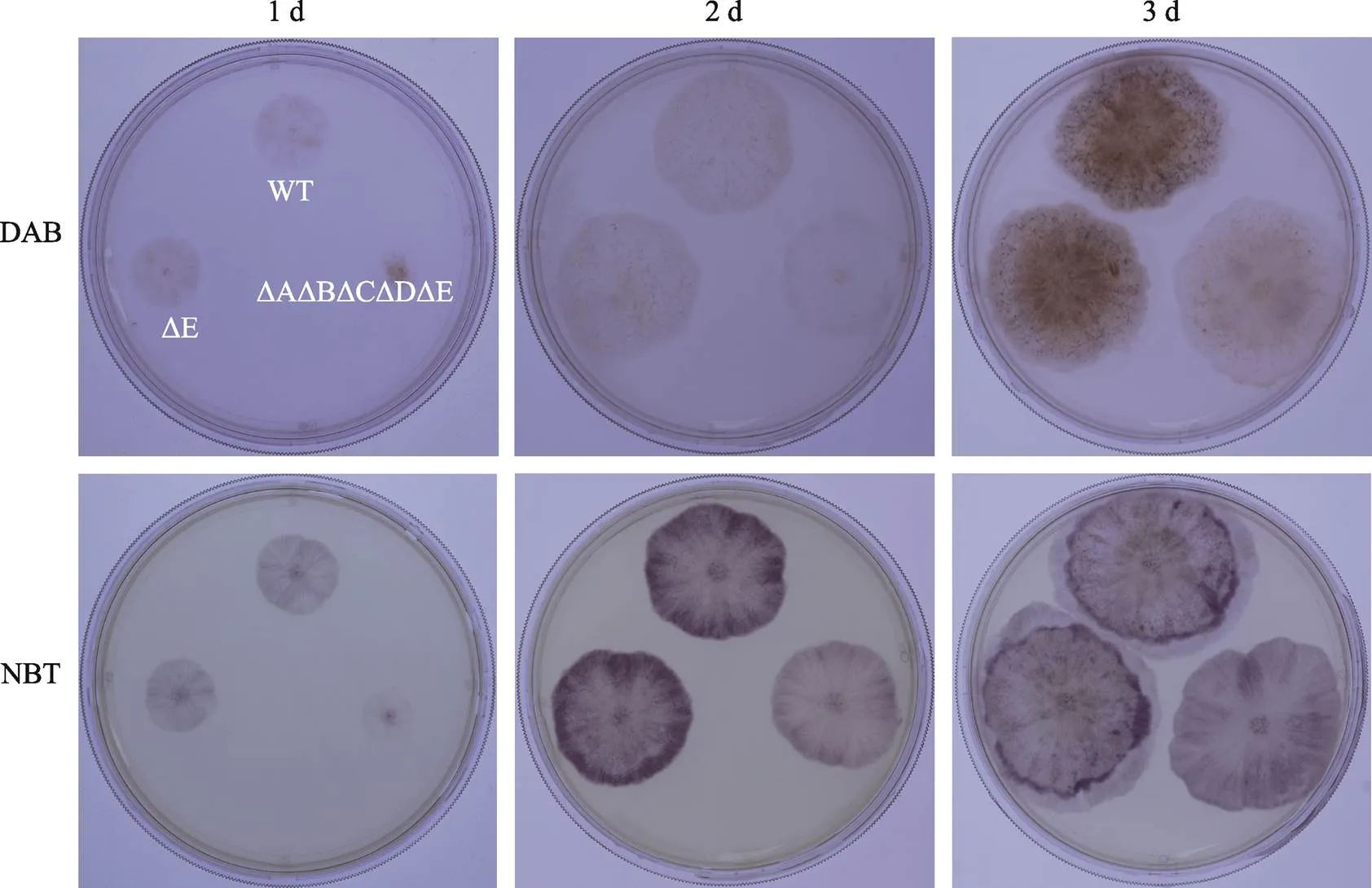
图9 DAB和NBT染色分析野生型菌株与突变型菌株菌丝ROS水平
将不同交配型菌株混合破碎,取5 µL菌悬液滴在M2培养基中央。黑暗培养1、2、3天(d)后,用DAB和NBT对菌株菌丝进行染色。
将100 µL菌悬液接种于50 mL 微晶纤维素液体培养基中,150 r/min恒温摇床培养3、5、7、9天(d),提取粗酶液,测定不同菌株的纤维素酶活。检验:*<0.05,**<0.01,***<0.001。
多项研究表明,是一种寿命有限的丝状真菌,其衰老的表型为:菌株生长速率和生殖能力的下降、气生菌丝形成的减少以及生长停滞会伴随着明显的色素沉着[36,37]。本研究也发现ΔAΔBΔCΔDΔE在M2和木质素培养基上均表现出异常的菌落形态和显著的生长减少,与WT的蓬松菌落表面不同,ΔAΔBΔCΔDΔE菌株的环状菌落仅形成稀疏的气生菌丝;生殖能力显著下降,仅在内圈生成少数子实体;培养5天后,ΔAΔBΔCΔDΔE加速了外圈色素的过度积累(图4,图5)。子实体是真菌体内的产孢结构,孢子在子实体内形成并于成熟后喷发,是真菌繁殖的主要途径,该过程需要消耗大量的能量和营养物质[26,29]。如图6、图7和图8所示,基因可能与子实体分布、形成和子囊孢子萌发有关。同时敲除PaLPMO11家族的5个基因,会导致菌株子实体数量明显减少和生成部分异常子实体,进而造成有性生殖部分受损。ΔAΔBΔCΔDΔE菌株子实体数相较于WT减少95%,子囊孢子萌发率仅为51.5%,说明PaLPMO11家族基因可能是控制菌株有性生殖的关键基因。适量的过氧化氢可以作为一种信号分子,发挥一定的生理作用,但高浓度的过氧化氢可能会引发氧化应激,导致程序性细胞死亡[38]。根据前人的研究报道,用400 μmol/L的过氧化氢会诱导H9C2细胞发生氧化应激,导致细胞活力下降[39];在中,WT菌株体内过氧化氢释放量会随着培养时间的延长而不断增加,菌株培养后期释放的过氧化氢量约是培养前期的3倍,证实了衰老与氧化应激的增加有关[40];在水稻中,基因的突变会导致过氧化氢的积累,从而导致叶片过早衰老[41]。在M2培养基上培养的第1天,ΔAΔBΔCΔDΔE菌株产生了大量过氧化氢(图9),再结合前面表型分析所得结果推测,过量的过氧化氢可能引发了ΔAΔBΔCΔDΔE菌株过度氧化应激状态,增强了自由基对细胞的氧化损伤,从而促进了突变菌株的衰老过程。综上,中5个基因的完全缺失会导致菌株出现严重的生长缺陷、形态变化和寿命缩短等情况。因而,PaLPMO11家族基因在丝状真菌的生长发育和有性生殖过程中可能是必不可少的。根据实验结果推测PaLPMO11家族基因功能冗余,5个基因间可能相互协调调控碳源利用与菌株的生长发育、有性生殖和衰老,一个或多个基因缺失时,其他基因可能在一定程度上能替代或补充其功能,使能够应对AA11家族单个或多个基因的缺失。在41种常见的真菌基因组中,是含有最多降解木质纤维素蛋白酶类基因的丝状真菌[15],具有强大的木质纤维降解能力,能够将木质纤维素转化成如生物乙醇等不同的高附加值产品[42]。将作为真菌降解木质纤维素的模式生物进行研究,发现一些新的木质纤维素降解相关基因和酶,阐明这些酶和基因在真菌中的作用机制,有望为深入了解真菌降解复杂多糖、提高生物能源生产效率等领域的研究提供参考[16,43]。如图10所示,单突变体的滤纸酶活、内切葡聚糖酶活、外切葡聚糖酶活和β-葡萄糖苷酶活与WT没有显著差异,而5个基因的同时缺失,会导致菌株总纤维素酶活显著下降,但却仍有野生型45%以上的酶活力。在中,基因缺失突变体降解纤维素的效率比WT提高了近两倍[44];和漆酶基因亚家族1的突变体降解纤维素的能力明显下降[25,45];证实了它们均参与了纤维素降解过程。在本研究中,出乎意料的是,PaLPMO11家族基因家族贡献了接近一半的纤维素酶活力。根据结果推测,基因之间可能存在功能冗余,且PaLPMO11家族的5个基因功能可能与纤维素降解有关,PaLPMO11家族的裂解多糖单加氧酶的完全失活可能会改变的纤维素降解机制。
综上所述,中AA11家族的裂解多糖单加氧酶基因参与菌株的生长发育、有性生殖、衰老和纤维素降解过程。本研究结果为系统阐述丝状真菌中木质纤维素降解的调控机制提供参考。
附加材料见文章电子版www.chinagene.cn。
[1] Tran MH, Yu JH, Lee EY. Microwave-assisted two-step liquefaction of acetone-soluble lignin of silvergrass saccharification residue for production of biopolyol and biopolyurethane., 2021, 13(9): 1491.
[2] Francois JM, Alkim C, Morin N. Engineering microbial pathways for production of bio-based chemicals from lignocellulosic sugars: current status and perspectives., 2020, 13: 118.
[3] Zhang ZR, Song JL, Han BX. Catalytic transformation of lignocellulose into chemicals and fuel products in ionic liquids., 2017, 117(10): 6834–80.
[4] Upton BM, Kasko AM. Strategies for the conversion of lignin to high-value polymeric materials: review and perspective., 2016, 116(4): 2275–306.
[5] Lyu LT, Chu YD, Zhang SF, Zhang Y, Huang QT, Wang S, Zhao ZK. Engineering the oleaginous yeastfor improved resistance against inhibitors in biomass hydrolysates., 2021, 9: 768934.
[6] Couturier M, Navarro D, Chevret D, Henrissat B, Piumi F, Ruiz-Dueñas FJ, Martinez AT, Grigoriev IV, Riley R, Lipzen A, Berrin JG, Master ER, Rosso MN. Enhanced degradation of softwood versus hardwood by the white-rot fungus., 2015, 8: 216.
[7] Xia W, Xu XX, Qian LC, Shi PJ, Bai YG, Luo HY, Ma R, Yao B. Engineering a highly active thermophilic β-glucosidase to enhance its pH stability and saccharification performance., 2016, 9: 147.
[8] Calderaro F, Keser M, Akeroyd M, Bevers LE, Eijsink VGH, Várnai A, van den Berg MA. Characterization of an AA9 LPMO from,LPMO9B, under industrially relevant lignocellulose saccharification conditions., 2020, 13(1): 195.
[9] Theibich YA, Sauer SPA, Leggio LL, Hedegård ED. Estimating the accuracy of calculated electron paramagnetic resonance hyperfine couplings for a lytic polysaccharide monooxygenase., 2021, 19: 555–67.
[10] Span EA, Marletta MA. The framework of polysaccharide monooxygenase structure and chemistry., 2015, 35: 93–99.
[11] Hemsworth GR, Henrissat B, Davies GJ, Walton PH. Discovery and characterization of a new family of lytic polysaccharide monooxygenases., 2014, 10(2): 122–126.
[12] Wang DM, Li J, Salazar-Alvarez G, McKee LS, Srivastava V, Sellberg JA, Bulone V, Hsieh YSY. Production of functionalised chitins assisted by fungal lytic polysaccharide monooxygenase., 2018, 20(9): 2091–2100.
[13] Rieder L, Petrović D, Väljamäe P, Eijsink VGH, Sørlie M. Kinetic characterization of a putatively chitin-active LPMO reveals a preference for soluble substrates and absence of monooxygenase activity., 2021, 11(18): 11685–11695.
[14] Støpamo FG, Røhr ÅK, Mekasha S, Petrović DM, Várnai A, Eijsink VGH. Characterization of a lytic polysaccharide monooxygenase fromshows functional variation among family AA11 fungal LPMOs., 2021, 297(6): 101421.
[15] Levasseur A, Drula E, Lombard V, Coutinho PM, Henrissat B. Expansion of the enzymatic repertoire of the CAZy database to integrate auxiliary redox enzymes., 2013, 6(1): 41.
[16] Dicko M, Ferrari R, Tangthirasunun N, Gautier V, Lalanne C, Lamari F, Silar P. Lignin degradation and its use in signaling development by the coprophilous ascomycete, 2020, 6(4): 278.
[17] El-Khoury R, Sellem CH, Coppin E, Boivin A, Maas MFPM, Debuchy R, Sainsard-Chanet A. Gene deletion and allelic replacement in the filamentous fungus., 2008, 53(4): 249–258.
[18] Goswami RS. Targeted gene replacement in fungi using a split-marker approach., 2012, 835: 255–269.
[19] Yan LY, Zhang HJ, Zheng YQ, Cong YQ, Liu CT, Fan F, Zheng C, Yuan GL, Pan G, Yuan DY, Duan MJ. Transcription factor OsMADS25 improves rice tolerance to cold stress., 2021, 43(11): 1078– 1087.闫凌月,张豪健,郑雨晴,丛韫起,刘次桃,樊帆,郑铖,袁贵龙,潘根,袁定阳,段美娟. 转录因子OsMADS25提高水稻对低温的耐受性. 遗传,2021, 43(11): 1078–1087.
[20] Tangthirasunun N, Navarro D, Garajova S, Chevret D, Tong LCH, Gautier V, Hyde KD, Silar P, Berrin JG. Inactivation of cellobiose dehydrogenases modifies the cellulose degradation mechanism of, 2017, 83(2): e02716–16.
[21] Dashtban M, Maki M, Leung KT, Mao CQ, Qin WS. Cellulase activities in biomass conversion: measurement methods and comparison., 2010, 30(4): 302–309.
[22] Thankappan S, Kandasamy S, Joshi B, Sorokina KN, Taran OP, Uthandi S. Bioprospecting thermophilic glycosyl hydrolases, from hot springs of Himachal Pradesh, for biomass valorization., 2018, 8(1): 168.
[23] Bennati-Granier C, Garajova S, Champion C, Grisel S, Haon M, Zhou SM, Fanuel M, Ropartz D, Rogniaux H, Gimbert I, Record E, Berrin JG. Substrate specificity and regioselectivity of fungal AA9 lytic polysaccharide monooxygenases secreted by., 2015, 8: 90.
[24] Demoor A, Silar P, Brun S. Appressorium: the breakthrough in., 2019, 5(3): 72.
[25] Xie N, Ruprich-Robert G, Silar P, Chapeland-Leclerc F. Bilirubin oxidase-like proteins from: promising thermostable enzymes for application in transformation of plant biomass., 2015, 17(3): 866–875.
[26] Fu C, Thielhelm TP, Heitman J. Unisexual reproduction promotes competition for mating partners in the global human fungal pathogen., 2019, 15(9): e1008394.
[27] Wang M, Gu BL, Huang J, Jiang S, Chen YJ, Yin YL, Pan YF, Yu GJ, Li YM, Wong BHC, Liang Y, Sun H. Transcriptome and proteome exploration to provide a resource for the study of., 2013, 8(2): e56686.
[28] Orban A, Weber A, Herzog R, Hennicke F, Rühl M. Transcriptome of different fruiting stages in the cultivated mushroomsuggests a complex regulation of fruiting and reveals enzymes putatively involved in fungal oxylipin biosynthesis., 2021, 22(1): 324.
[29] Schumacher DI, Lütkenhaus R, Altegoer F, Teichert I, Kück U, Nowrousian M. The transcription factor PRO44 and the histone chaperone ASF1 regulate distinct aspects of multicellular development in the filamentous fungus., 2018, 19(1): 112.
[30] Ismail HF, Hashim Z, Soon WT, Rahman NSA, Zainudin AN, Majid FAA. Comparative study of herbal plants on the phenolic and flavonoid content, antioxidant activities and toxicity on cells and zebrafish embryo., 2017, 7(4): 452–465.
[31] Zhang HR, Li BX, Sun ZM, Zhou H, Zhang SS. Integration of intracellular telomerase monitoring by electrochemiluminescence technology and targeted cancer therapy by reactive oxygen species., 2017, 8(12): 8025–8029.
[32] Kalyani D, Tiwari MK, Li JL, Kim SC, Kalia VC, Kang YC, Lee JK. A highly efficient recombinant laccase from the yeastand its application in the hydrolysis of biomass., 2015, 10(3): e0120156.
[33] Abraham RE, Wong CS, Puri M. Enrichment of cellulosic waste hemp () hurd into non-toxic microfibres., 2016, 9(7): 562.
[34] Zhang XJ, Qu YB, Qin YQ. Expression and chromatin structures of cellulolytic enzyme gene regulated by heterochromatin protein 1., 2016, 9: 206.
[35] Laurent CVFP, Sun PC, Scheiblbrandner S, Csarman F, Cannazza P, Frommhagen M, van Berkel WJH, Oostenbrink C, Kabel MA, Ludwig R. Influence of lytic polysaccharide monooxygenase active site segments on activity and affinity., 2019, 20(24): 6219.
[36] Silar P.: from laboratory to biotechnology. Genomics of soil- and plant-associated fungi. 2013, 283–309.
[37] Bernhardt D, Hamann A, Osiewacz HD. The role of mitochondria in fungal aging., 2014, 22: 1–7.
[38] Pellavio G, Sommi P, Anselmi-Tamburini U, DeMichelis MP, Coniglio S, Laforenza U. Cerium oxide nanoparticles regulate oxidative stress in hela cells by increasing the aquaporin-mediated hydrogen peroxide permeability., 2022, 23(18): 10837.
[39] Gao H, Liu XP, Tian KM, Meng YC, Yu CC, Peng YF. Insight into the protective effect of salidroside against H2O2-induced injury in H9C2 cells., 2021, 2021: 1060271.
[40] Wiemer M, Osiewacz HD. Effect of paraquat-induced oxidative stress on gene expression and aging of the filamentous ascomycete., 2014, 1(7): 225–240.
[41] Wu LW, Ren DY, Hu SK, Li GM, Dong GJ, Jiang L, Hu XM, Ye WJ, Cui YT, Zhu L, Hu J, Zhang GH, Gao ZY, Zeng DL, Qian Q, Guo LB. Down-regulation of a nicotinate phosphoribosyltransferase gene,, leads to withered leaf tips., 2016, 171(2): 1085–1098.
[42] Bian HY, Wu XX, Luo J, Qiao YZ, Fang GG, Dai HQ. Valorization of alkaline peroxide mechanical pulp by metal chloride-assisted hydrotropic pretreatment for enzymatic saccharification and cellulose nanofibrillation., 2019, 11(2): 331.
[43] Couturier M, Tangthirasunun N, Ning X, Brun S, Gautier V, Bennati-Granier C, Silar P, Berrin JG. Plant biomass degrading ability of the coprophilic ascomycete fungus., 2016, 34(5): 976–983.
[44] Brun S, Malagnac F, Bidard F, Lalucque H, Silar P. Functions and regulation of the Nox family in the filamentous fungus: a new role in cellulose degradation., 2009, 74(2): 480–496.
[45] Xie N, Chapeland-Leclerc F, Silar P, Ruprich-Robert G. Systematic gene deletions evidences that laccases are involved in several stages of wood degradation in the filamentous fungus., 2014, 16(1): 141–161.
Identification and functional study of AA11 family polysaccharide monooxygenase genes in filamentous fungus
Wenzhen Du1, Yuanjing Li2, Jialing Wu1, Siyu Chen1, Liang Jiang1, Gang Liu1, Ning Xie1
The lytic polysaccharide monooxygenase (LPMO) in the auxiliary active protein family (AA family) catalyzes the oxidative depolymerization of various refractory carbohydrates including cellulose, chitin and starch. While accumulating studies investigate the enzymology of LPMO, the research on the inactivation ofgenes has been rarely explored. In this study, fivegenes()()()()()of the AA11 family in the filamentous funguswere knocked out by homologous recombination. Single mutants ΔPaLPMO11A (ΔA), ΔPaLPMO11B (ΔB), ΔPaLPMO11C (ΔC), ΔPaLPMO11D (ΔD) and ΔPaLPMO11E (ΔE) were constructed, and then all polygenic mutants were constructed via genetic crosses. The differences in the growth rate and sexual reproduction between wild type and mutant strains were observed on different carbon source media. The alteration of oxidative stress and cellulose degradation ability were found on DAB and NBT staining and cellulase activity determination. These results implicated thatgenes play a key role in the growth, development, and lignocellulose degradation of. The results showed that the spore germination efficiency, growth rate and reproductive capacity of mutant strains including ΔBΔCΔE, ΔAΔBΔCΔE, ΔAΔCΔDΔE and ΔAΔBΔCΔDΔE was significantly decreased on different cellulose carbon sources and the remaining strains have no difference. The reduced utilization of various carbon sources, the growth rate, the spore germination rate, the number of fruiting bodies, the normal fruiting bodies, the shortened life span and the ability to degrade cellulose were found in strains which all five genes in the PaLPMO11 family were deleted. However, the strain still had 45% cellulase activity compared to wild type. These results suggest thatgenes may be involved in the growth and development, sexual reproduction, senescence and cellulose degradation of. This study provides information for systematically elucidating the regulatory mechanism of lignocellulose degradation in filamentous fungus.
filamentous fungi;;; gene knockout; lignocellulose degradation
2023-08-21;
2023-10-28;
2023-11-10
国家重点研发计划项目(编号:2021YFA0910800),广东省自然科学基金项目(编号:2021A1515012166,2021A1515012118)和国家自然科学基金项目(编号:31601014,22078199)资助[Supported by the National Key R&D Program of China (No. 2021YFA0910800), the Basic and Applied Basic Research Fund of Guangdong Province (Nos. 2021A1515012166, 2021A1515012118), and the National Natural Science Foundation of China (Nos. 31601014, 22078199)]
杜文珍,硕士研究生,专业方向:微生物遗传学。E-mail: 2100251038@email.szu.edu.cn
谢宁,博士,副教授,博士生导师,研究方向:微生物遗传学。E-mail: ning.xie@szu.edu.cn
10.16288/j.yczz.23-223
(责任编委: 刘钢)