基于距离编码-图神经网络的药物靶标作用关系预测
2023-12-13翁兴娜李建华
翁兴娜,高 创,李建华
(华东理工大学 信息科学与工程学院,上海 200237)
1 引 言
众所周知,准确预测药物-靶标相互作用(Drug-Target Interactions,DTI)是药物发现和药物重定位的基础之一.药物发现是指从大量的分子中为靶标寻找潜在的候选药物,药物重定位是指为现有药物寻找新应用.相比于代价高昂的药物发现,药物重定位是现代化学药物研发过程中较常采用的方法.例如,早期作为镇静剂与止吐剂的药物“沙利度胺”于1998年被FDA批准应用于治疗麻风结节性红斑[1].预测药物靶标相互作用旨在为现有靶标确定新药物或为现有药物发现新靶标.
分子对接和机器学习方法长期以来一直是药物发现的重要工具[2].分子对接方法依赖药物与靶标蛋白质的三维结构进行模拟,但在实验中部分生物大分子的三维结构数据难以获得[3],因此无法处理大批量的药物组学数据.传统的机器学习方法广泛应用于定量构效活性关系(QSAR)、蛋白质化学计量学(PCM)等领域来进行DTI建模[4],如支持向量机、随机森林、朴素贝叶斯、K近邻和人工神经网络等方法通过计算蛋白质与配体化合物之间的特征向量或相似性来预测相互作用,但此类方法很难从公开数据集中找到具有较高质量的负样本数据集[5],因此存在正负样本高度不平衡,预测结果不准确的风险.研究人员尝试使用卷积神经网络提高DTI的预测能力,但卷积神经网络的平移不变性使其无法处理药物靶标信息这样的非欧式空间数据,因此预测精确度远不如传统机器学习方法[6].
近年来,图神经网络在图数据[7]学习方面有出色的表现,因此在节点分类和链路预测任务中得到了广泛的应用[5,8,9,10].而本文的图数据描述的是药物靶标相互作用图,本质上也是一个图结构,其中药物和靶标节点可以看作图中的节点(node),药物靶标相互作用看作边(edge),TriModel方法[11]中也将药物靶标相互作用预测视为链路预测问题的一种.针对机器学习方法中在高度不平衡数据上预测精度较低的问题,Gao等人[12]提出了基于异质信息的图神经网络方法GCNDTI来对其进行解决,验证了图神经网络方法在药物靶标作用预测任务上的可行性.然而,图神经网络仍存在无法识别图网络中具有相似拓扑结构节点的缺点[13,14,15],许多研究者在使用图神经网络来表达图网络的结构特征时,大多直接对大图特征进行提取,这类算法的计算效率不高且泛化能力较差[14,16,17].
针对图神经网络方法在药物靶标预测任务中无法识别具有相似拓扑结构节点的缺陷,本文提出一种基于距离编码-图神经网络的方法(Distance Encoding Drug-Target Interaction,DEDTI)来对其进行解决,主要贡献有以下两个方面:1)对图中每个节点进行距离编码,使得具有相似拓扑结构的不同节点可以生成不同的低维嵌入表征,提高了药物靶标数据在图神经网络中的表达能力;2)使得药物靶标节点在图神经网络中训练时具有距离属性而非简单的one-hot特征,提升了使用图神经网络方法预测药物相关靶标的性能.
2 相关工作
2.1 药物靶标预测研究现状
基于机器学习的计算预测方法可以更快的预测可能存在的DTI相互作用,是现代生物信息学研究的重要组成部分.例如,Yamanishi等人[18]应用经典的支持向量机(Support Vector Machine,SVM)框架,第一个提出了使用来自药物和靶标的二分图信息的统计模型来预测DTI.Mei等人[19]为了进一步优化模型效果,提出了从邻居节点中提取相互作用特征的模型来预测药物和靶标作用.Xia等人[20]应用了拉普拉斯正则化最小二乘法,并将相似性和交互性内核结合到预测框架中.
随着研究的深入,深度学习算法在图网络数据上的应用越发广泛,在网络数据上应用深度学习模型可以结合图异构信息,捕获高维的复杂生物网络拓扑特征.Olayan等人[21]将药物靶标关系信息表示在异质图网络中,通过非线性融合的方法结合相似性特征和异质图嵌入特征训练随机森林模型预测DTI.类似地,Sameh K等人[11]提出了TriModel模型来同时学习药物和靶标的矢量嵌入特征,从而计算DTI得分.Tang等人[22]将DTI预测看作异质网络上的降噪问题,利用异质节点之间的关联关系降低网络稀疏性并提高预测精度.Gao等人[12]提出了GCNDTI,利用药物靶标节点、节点的邻域信息和局部拓扑结构的低维向量信息得到节点间的概率.但现有的DTI预测方法在异质图中还存在无法区分相似拓扑结构的不足,且提取药物靶标图网络数据中节点特征的能力较弱,需要提出新的方法来解决上述问题.
2.2 图神经网络的发展
图神经网络(Graph Neural Network,GNN)的概念在2009年首次被提出[7],早期的图神经网络通过反复交换邻域信息来更新节点状态,直到达到稳定的平衡状态,被归为循环图神经网络的一种.后来又发展了一些处理图结构数据的GNN方法,与循环图神经网络相比,卷积图神经网络[23](GCNs)解决了循环层相互依赖的问题,极大地提高了图神经网络的效率、灵活性和通用性.图自编码器[24](GAEs)可用于学习网络嵌入和生成图,Cao等人[25]提出的DNGRs和Wang等人[26]提出的SDNE是典型的使用多层感知器来构建用于网络嵌入学习的GAE.
图神经网络的研究与图嵌入密切相关.在网络任务中,虽然矩阵分解方法可以高效的生成特征嵌入,但其无法聚合节点属性特征的问题一直未能被解决.图神经网络可以高效率的更新节点,且属于启发式学习,因此相关算法被越来越多的应用到图网络领域任务中.图神经网络算法利用丰富的节点间关系来抽取网络层特征,在基于图的应用中日渐流行,包括半监督节点分类[23]、链路预测[9]和节点分类[27]等.药物靶标数据库如DrugBank、KEGG等,很大程度上是由药物、靶标的节点和边组成的网络数据,预测新药物靶标之间的相互作用的任务可以被看作基于图嵌入的链路预测问题.
为了将网络结构数据从原始网络空间转化到嵌入空间,可以采用不同的模型来合并不同类型的信息.常用的图嵌入模型[28]包括基于矩阵分解模型(SVD等)的图嵌入、基于随机游走模型(DeepWalk[29]、node2vec[30]等)的图嵌入、基于图神经网络(SDNE、GCN[23]、GraphSage[31]等)的图嵌入及其变体[28].此处将基于随机游走的图嵌入看作矩阵分解模型的变体,因为DeepWalk、node2vec等算法可以统一到闭型(closed form)的矩阵分解框架中[32].针对图数据中的关系,研究者们受到DeepWalk和node2vec中提取图表示方法的启发,使用随机游走(random walk)和最短路径方法(the shortest path)来对图网络数据进行编码.DeepWalk通过随机游走的方法对不同跳的节点进行采样,获得子网节点关系的节点表征.node2vec在DeepWalk的基础上,引入宽度优先算法和深度优先算法,对下一跳的随机游走算法进行概率分配,而概率由中心节点与邻域节点之间的最短路径决定.广度优先注重聚合数据的局部特征,深度优先能更好的遍历整个子网,扫描节点间的同质性.因此将二者结合起来,可以更好地得到图网络数据的嵌入特征.
在区分蛋白质、基因网络等任务中通常认为具有相似结构的分子可能具备相似的功能特性,因此度量图的相似度即图同构问题是图学习的一个核心问题.而目前解决图同构问题最有效的算法是Weisfeiler-Lehman测试算法[13],WL测试判断两个拓扑图结构是否同构的步骤与图卷积神经网络的逐层迭代十分相似,区别主要在于单射函数[13],即算法是否将两个非同构图映射到相同的嵌入表征向量中.在图神经网络算法中,两个节点映射到同一位置的条件是两个节点必须具有相同的子图拓扑结构,而WL算法判断同质图的能力强于图卷积神经网络.
图神经网络解决了卷积神经网络无法处理非欧式结构数据的问题[33],但在实际应用中还存在缺陷.在图网络中,如果两个节点位置不同,但节点的邻域节点结构相似或相同,图神经网络迭代聚合并更新节点信息后,将得到相同的节点嵌入表征.对于部分图网络数据来说,增加图神经网络的层数可以区分相似或相同拓扑结构的子图.但是对于高对称性的正则图,无论图神经网络进行多少次的迭代也无法区分节点,且网络层数过多时图神经网络会出现过平滑问题,有研究[23]尝试使用one-hot编码方法计算节点之间的距离,模型使用one-hot编码依赖于全图结构信息,因此训练时无法在具有新节点的图网络数据上进行迁移学习,使得模型变为了直推式任务.本文的研究方法将距离编码加入图神经网络算法中,利用邻域拓扑信息标记中心节点,使得图神经网络可以为具有相同子图结构的中心节点生成不同的特征向量,保证了图神经网络的单射性,使得基于距离编码的图神经网络算法在判断图同构任务时超过一阶WL测试的效果.
3 DEDTI方法
在药物靶标预测任务中有效表示图结构是使用图数据进行学习的前提.图神经网络利用节点属性和图结构来学习图特征向量.受从网络拓扑结构中提取数据特征的方法启发[34],本文针对药物靶标之间存在的相互作用边关系编码,将其作为网络结构特征,由此提出一种基于距离编码-图神经网络的方法来预测药物靶标相互作用.
DEDTI方法由特征生成、模型预测、训练优化3部分组成.特征生成以节点和图网络作用信息等拓扑结构特征作为输入,输出包含图距离编码信息的特征矩阵.模型预测根据药物靶标节点的低维特征信息,构建图神经网络模型预测节点间的相互作用概率得分.训练优化通过迭代计算与更新得到多个模型参数的最优值.图1给出了DEDTI的架构图,其中图1(a)所示为表示药物靶标相互作用的拓扑图网络示例,图1(b)、图1(c)、图1(d)分别为距离编码、模型预测和训练优化过程示意图.其中距离编码的流程由选取子图、距离编码和矩阵构造3部分组成.
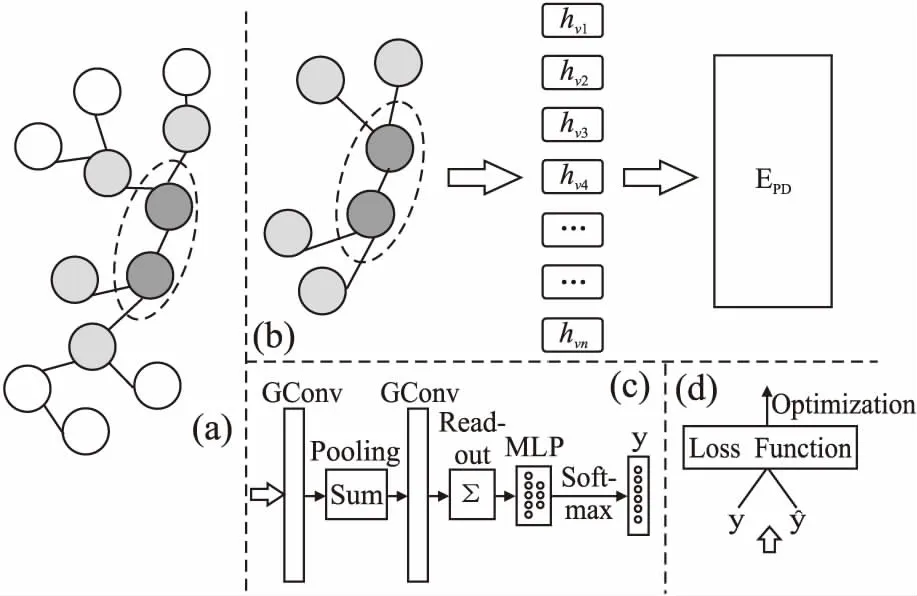
图1 DEDTI架构图Fig.1 DEDTI architecture
3.1 选取子图
图网络随着药物靶标节点的增多而逐渐变大,因此子图的大小决定图神经网络聚合邻域信息的数量.使用G=(E,V)表示图,G′表示子图,E表示存在相互关系的边的集合,V表示药物和靶标的节点集合,使用节点度作为图的初始特征.
在药物靶标标准数据集Yamanishi_08中,选用相同跳数从药物靶标图网络中聚合邻域信息时,存在部分数据集的整图与其他数据集的子图大小相仿的情况,即G′=G.为避免在子图选取时出现上述问题,本文先统计数据集中每个节点与不同跳数邻域节点之间的距离,统计结果如表1所示.随机选取一个中心节点vi,使用最短路径方法(The shortest path)统计图中其它节点与该节点的最短路径长度,称为步长,表1中步长-1表示该节点为不可达节点,0表示自身节点,如表1中统计数据所示,当步长大于7时,各数据集中的节点距离与其他步长的节点距离之比逐渐降低为0,因此统计过远的邻域节点信息对于节点的特征表示没有明显的区别,还会造成邻接矩阵的信息冗余,所以统计时将步长大于等于9的节点视为一类节点.从表1中的统计信息可以发现,不同数据集的最大子图大小不同,可达节点的步长也有差异.
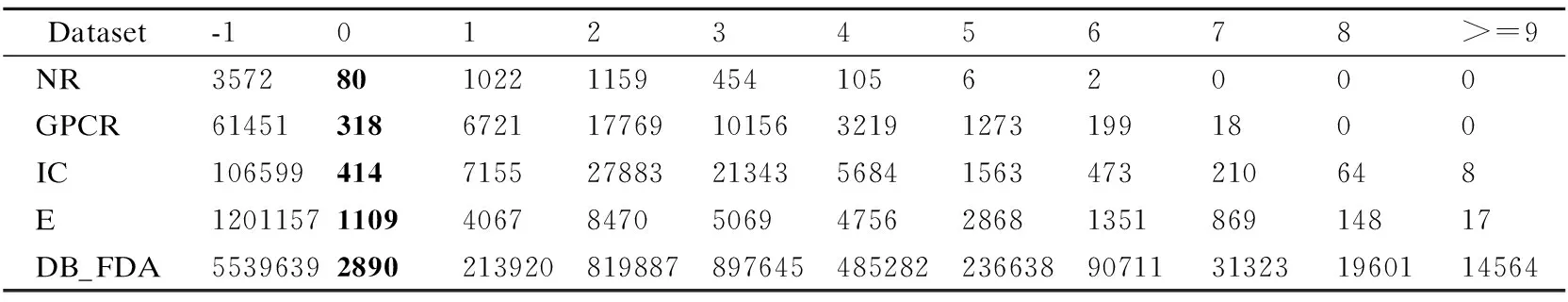
表1 各数据集中节点步长数据统计Table 1 Statistics of node step in five datasets
在选取子图时,可以使用固定步长与不固定步长两种方式确定步长.固定步长是指针对5种数据集采用同一步长计算子图;非固定步长指针对各数据集设置不同步长,即不同数据集选取的子图大小不同.从表1中可以看出,当步长相同时,从不同数据集中提取的子图节点数量所占的比例是不同的.例如当步长小于等于3时,NR数据集中提取的子图节点的比例为全图节点的95.8%,而在DrugBank_FDA数据库中仅为68.7%.子图过大不仅会影响图网络性能,还会造成信息冗余的问题.子图过小会导致聚合到的节点信息不足.当步长为非固定步长时,可以灵活控制步长,使得不同数据集中的子图所聚合的节点数量始终占比在80%左右,因此在进行实验时设置步长为非固定步长.步长大小的设置与GNN的层数及传播深度密切相关.GNN的层数L决定了图神经网络中心节点聚合邻域信息的跳数.如果子图大小小于GNN层数,则存在相同节点在传播过程中被重复聚合多次的可能,从而无法起到扩散信息的作用.
3.2 距离编码
DEDTI通过距离编码来保证图神经网络的单射性,因此距离编码是DEDTI中最重要的步骤.在距离编码过程中,使用sn表示子图的跳步数量,然后任选一中心节点vi,计算中心节点周围的关系边eij∈E的sn跳子图G′.在子图G′中,根据其他节点相对边eij的位置进行编码.距离编码是计算子图内中心节点vi与其他所有节点之间的边eij之间的相对距离,而不是直接对节点在全图中的绝对距离进行计算,因此具有很好的泛化能力和扩展能力.
GCNDTI[12]通过提取节点嵌入特征预测DTI,更关注节点本身.而DEDTI关注的是网络中的边和节点之间的距离等网络结构信息.DEDTI使用随机游走(Random Walk)和最短路径算法(The Shortest Path)两种方法来生成节点到边eij的距离,两种方法的计算过程不同.图2以最短路径方法为例,利用最短距离算法来计算中心节点vi和vj与子图内所有邻居节点之间的最短距离特征矩阵,分别生成距离编码后,将两个节点的最短距离拼接成二元数组hij∈n×2,其中hij表示子图中所有节点相对于边eij的距离特征,n表示子图内包含的节点数目.随后对特征数组设置步长阈值,方便后续使用one-hot向量表示该特征向量,大于阈值的步长以阈值为准.经过距离编码后,保证了GNN的单射性,使得GNN可以根据每个节点独特的距离属性特征区分具有相同拓扑结构的中心节点.保证GNN的单射性是确保GNN能达到WL测试性能的基础.
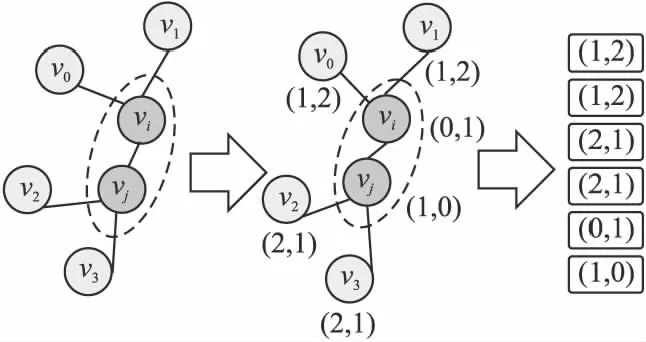
图2 距离编码过程Fig.2 Process of positional encoding
使用距离编码进行聚合解决了图神经网络中池化方法无法保证单射性的问题.距离编码在对不同中心节点进行特征提取时,虽然聚合的节点数量和拓扑结构是相同的,但是由于邻域节点的结构特征不同,所以得到的节点属性也是不同的.如图3所示,在未加入距离编码之前,再多层的图神经网络也无法辨别v1和v8两个中心节点.加入距离信息后,图神经网络迭代至第2层时就可以精准地辨别两个正则节点.图3中使用不同灰度标记不同步长的节点v1和v8表示中心节点,(v2、v3、v8)和(v1、v7、v6)表示一跳节点,(v4、v5、v6、v7)和(v2、v3、v4、v5)表示二跳节点,如图3中椭圆阴影部分,两个中心节点在进行第2次迭代时,聚合信息就开始出现差异.距离编码通过不同跳步来提取一定邻域范围内的子图节点特征,不需要再通过增加图神经网络的层数来聚合远距离邻域节点的有效信息,即舍弃使用深层网络来分辨正则图结构,避免了图神经网络层数过多引发过平滑的问题.
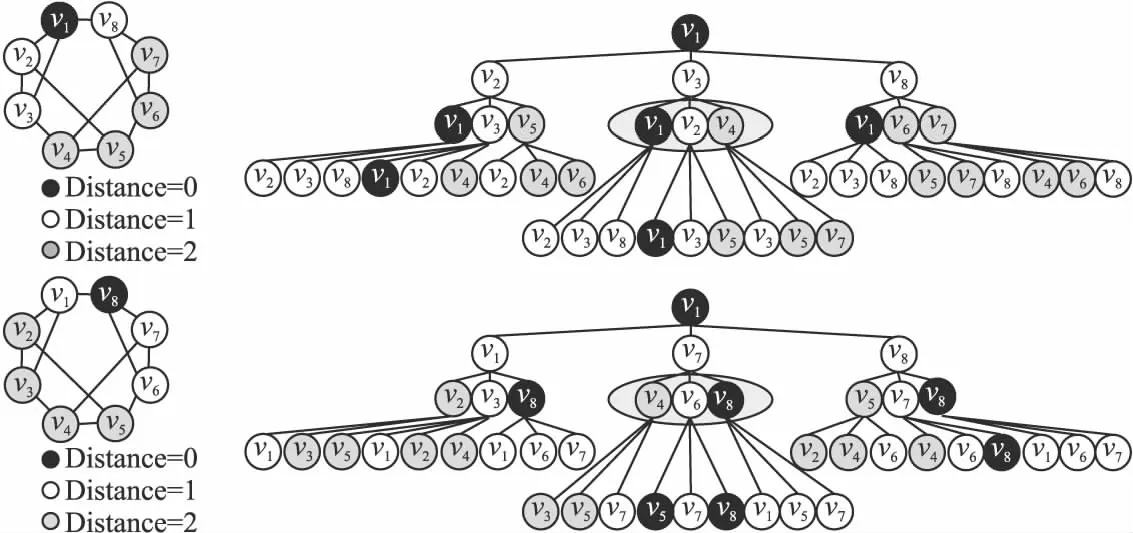
图3 距离编码辨别相似节点过程Fig.3 Process of positional encoding to identify similar node
3.3 特征生成
特征生成即使用新的子图索引更新子图特征矩阵,与经过距离编码后的特征矩阵拼接后生成包含子图节点距离信息的特征矩阵.特征矩阵包含所有对已编码边特征的处理以及聚合.
在距离编码后,每两个中心节点之间的边关系都由二元组组成的特征矩阵表示.但考虑到需要使用该特征矩阵进行训练,两列的特征矩阵表达的网络特征较少,训练模型时容易出现欠拟合现象.因此在构成下游训练任务所需的特征矩阵之前,根据二元组自身步长,使用one-hot编码表示该二元组,从而增强特征表示.one-hot向量的维度与步长的最大值或者阈值是一致的,例如二元组(1,2)的最大步长为5,则生成特征的形式为(0,1,0,0,0)与(0,0,1,0,0).为保证矩阵数据的均衡分布,再用0补充0维数据变为(0,0,1,0,0,0)与(0,0,0,1,0,0),计算生成了fij=n×2×6的三维矩阵.最后使用连接方法将二元组特征纵向拼接为(0,0,1,1,0,0),生成最终的编码特征Fij=n×6.为了提高网络结构数据的特征表达能力,在对关系边进行距离编码后加上节点度属性来表示节点的重要性.为方便药物和靶标节点之间的横向比较,采用对数方法对节点度进行归一化.矩阵特征生成后,可以作为输入数据送入下游模型进行预测任务.
3.4 模型预测
模型预测即采用图神经网络方法预测药物靶标节点之间原来不存在的作用边,预测过程如图1(c)所示.模型采用距离编码后的特征向量作为输入,预测后输出节点vi和vn之间的概率分数pin,与图神经模型的预测步骤基本一致,其中需要注意模型聚合器的选用.
GNN的池化有两种类型的操作,一种是针对图层面(Graph-level),比如分子图分类.一种是针对节点层面(Node-level),比如节点的分类或回归.预测靶标节点是否与药物节点有相互作用,属于节点分类任务.因此需要聚合节点的邻域节点特征,得到表示节点属性的向量,后续对该向量进行分类操作.在节点分类任务中引入池化操作,其中READOUT是最简单的池化操作.公式如式(1)所示[35]:
y=
(1)
不同种类的聚合器适用于不同的图网络任务.平均池化聚合器适用于节点分类任务,尤其当节点重复少、具有特征信息,任务与节点分布的特征有关、与局部结构无关时更为适用.最大池化聚合器仅整合子图结构中的最重要节点,丢失了节点的邻域信息,也无法聚合到节点分布信息.求和池化聚合器可以学习子图结构信息,也可以学习到边的相互作用标签和子图节点数量等特征.DEDTI需要聚合图网络中节点的属性信息,因此选用求和池化聚合器.
3.5 训练优化
训练优化是为了优化图神经网络每层的参数,使得最终的预测值接近真值.为了使模型的训练数据分布与真实数据分布相近,DEDTI引入与正样本数量相同的负样本来增强训练数据集,负样本是在数据集中选取不存在作用关系的节点和之间的边来替换真实边,然后使用交叉熵损失函数[12]来优化迭代模型参数.参数优化过程如图1(d)所示,公式如式(2)所式:
(2)
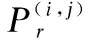
(3)
在训练的过程中,使用随机梯度下降(Stochastic Gradient Descent,SGD)算法来加快模型的收敛,取得最优值.模型使用分块训练的方法,并加入了DropOut层随机关闭神经元来缓解模型训练和预测过程中出现的过拟合现象.
4 实验结果与分析
本文采用Yamanishi_08和DrugBank_FDA数据集,使用AUC和AUPR的评价标准衡量DEDTI方法与其他方法在预测药物靶标相互作用方面的优劣,并对新发现的药物靶标关系进行验证.
4.1 数据来源
训练数据主要使用药物靶标之间的相互作用数据.使用到的网络图数据来自于Yamanishi_08[18],Yamanishi_08的药物靶标相互作用数据由KEGG、BRENDA、SuperTarget和DrugBank这4个数据库中的已被验证的DTI相互作用数据组成,并根据受体蛋白质的种类分为酶(Enzymes,E)、核受体(Nuclear Receptors,NR)、离子通道(Ion Channels,IC)及G蛋白偶联受体(G Protein-Couple Receptors,GPCR)4类子数据集.DrugBank_FDA(DB_FDA)是DrugBank数据集的数据子集,该数据集包含了一部分美国食品药品药物监督管理局(Food and Drug Administration,FDA)批准通过的药物靶标相互作用数据.Yamanishi_08相较于DrugBank_FDA数据量较少,因此DrugBank_FDA数据集成为了另一个重要的数据来源.如表2所示,实验数据中包含2414种药物、2397种靶标和15008种药物靶标相互作用(DTI)关系数据.DEDTI的核心是从药物靶标数据中提取图网络的结构特征,并将距离编码后的信息作为节点的属性特征,因此不涉及各类相似性数据,只需将药物靶标之间的相互作用关系进行特征生成即可.也正因此,距离编码的方法可以方便地扩展到各种图神经网络中,具有较强的泛化能力.
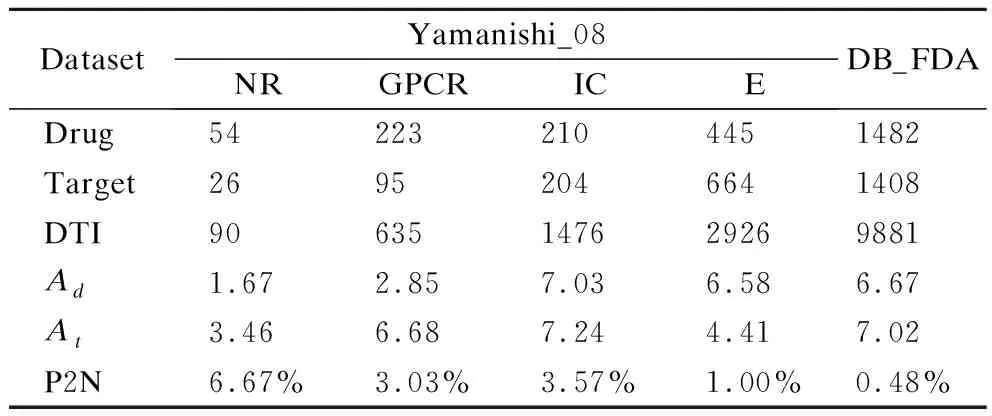
表2 药物靶标关系数据集Table 2 Statistic of the DTI databases
4.2 评价类型与标准
本文使用的评估指标为曲线下面积(Area Under the Curve,AUC)、精确召回曲线下面积(Area Under the Precision-Recall Curve,AUPR).这两个指标常用于药物靶标相互作用预测的相关工作[36].AUC指标用于衡量正负样本相对平衡的数据的精确度.AUPR在正负样本不平衡的数据中,能更灵敏地感知样本结果相关性和获得正相关结果的数量[37].如表2中P2N(正负样本比)指标所示,本文使用的Yamanishi08和DrugBank_FDA数据集中Yamanishi_08的正负样本比全部低于1∶9,DrugBank_FDA的正负样本比低于1∶100.因此AUPR指标更能衡量DEDTI方法的效果.
随着深度学习在DTI领域的应用,研究人员更希望能预测出新药物和新靶标之间的作用关系.由于距离编码在表示数据时使用的是节点之间的关系结构数据,未涉及到药物和靶标的相似性数据,因此DEDTI使用预测类型来对比DEDTI与其他方法的预测能力.指未知部分DTI,即遮盖部分药物靶标作用关系,将其标签统一设置为1,预测所遮盖的作用关系信息.数据集中按照8∶1∶1的比例对训练集、测试集和验证集进行划分.
本文将DEDTI与以下5种方法进行对比:BLM-NII[19]、NRLMF[38]、DDR[21]、TriModel、GCNDTI,所有方法均按照AUC和AUPR指标进行衡量.
4.3 实验设置
实验中首先确定使用不同种类的编码函数(最短路径方法和随机游走方法)提取的节点特征对下游任务性能的影响,然后通过实验确定子图大小.再使用消融实验确定GNN模型中的隐藏层维度、学习率和丢弃率等最优参数.
参数不同时,模型结果自然不同.在子网提取、距离编码和图神经网络预测步骤中,相对核心的是进行距离编码.距离编码方式决定了节点的表征方式.为了使图网络数据在GNN上的表达效果达到一阶WL测试的效果,节点编码后必须保持单射性,即不同的节点编码后属性不同.因此实验中采用随机游走(rw)、最短路径(sp)和随机游走+最短路径(sp+rw)3种参数类型.实验数据集选择IC数据集,实验结果如图4(a)所示.在子网提取中,步长大小决定了子网络的大小.步长为1时,仅聚合了11%的图网络数据.步长为5时,聚合了98%的图网络数据.步长实验结果如图4(b)所示.
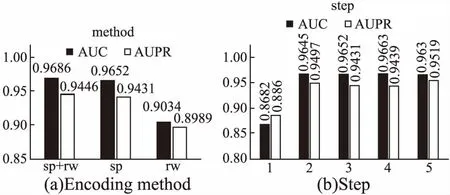
图4 IC数据集上的模型参数实验Fig.4 Experiment of model parameters on IC dataset
由图4(a)可知,编码方法对最终结果产生的影响较大,而最短路径和随机游走方法的组合在消除此影响的同时获得了最佳的表现.在其他领域的预测任务中,还可以使用PageRank等算法来实现距离编码的目的.由图4(b)可知,不同数据集适合不同的步长阈值,当小于阈值时,模型性能随阈值变化而变化.当步长超出一定阈值时,性能表现趋于稳定.经过实验发现,在较大的数据集Drugbank_FDA中,步长阈值为3时最佳;在较小的NR数据集中,步长阈值小于3时效果均不错.因此,确定3为所有数据集的步长.
在确定子图提取、距离编码的参数后,需要进一步确定下游GNN中的超参数:学习率α、丢弃率β和隐藏层维度数d.在GCNDTI的实验中发现,加深图神经网络的层数来捕捉远处节点的特征会引发过平滑现象,因此GNN舍弃使用此方法来聚合节点特征,只将GNN层数设置为两层.超参数的对比实验参数设置包括:学习率α∈[0.001,0.0001,0.00001]、丢弃率β∈[0.1,0.01,0.001]以及隐藏层数维d∈度[32,64,128].3种类型参数的实验结果展示在图5(a)、图5(b)、图5(c)中.可以看出不同参数对最终的AUC、AUPR均有一定影响,选取其中最好的结果参数α=0.0001、β=0.001、d=64作为最终参数.为两层图神经网络设置相同的隐藏层维度.尽管模型采用了随机丢弃的策略,但当学习率α=0.01时,还是出现了过拟合问题,因此在训练时设置α=0.001,并采用分块训练、权重衰减的方法来缓解该现象.
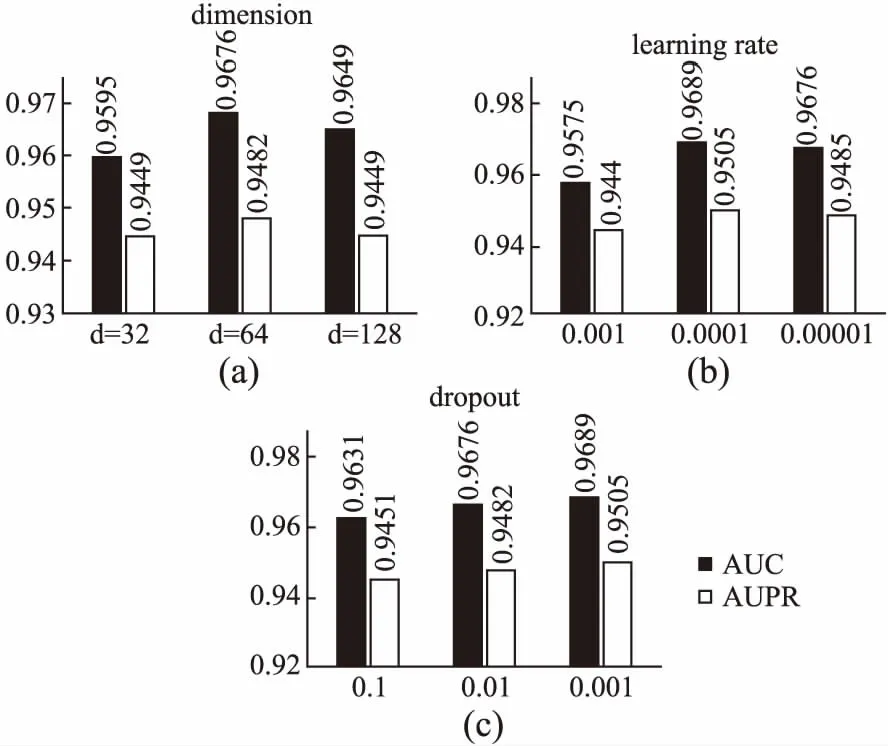
图5 维度、学习率和丢弃率对 DEDTI 模型的影响Fig.5 Influence of dropout、learning rate and dimension
4.4 对比实验以及实验分析
GCNDTI利用异质图中的特征信息有效地预测了DTI任务,但节点矩阵的特征编码使用的是one-hot编码.对比试验不仅证明了DEDTI在药物靶标预测领域的有效性,更证明了距离编码相较于one-hot编码的优越性,且图神经网络的预测性能在引入距离编码后得到了进一步提升.
表3所示为DEDTI与5种优秀的药物靶标方法及GCNDTI的对比结果.DEDTI在AUC指标上相比于TriModel方法稍显不足,但超过了其他4种基准模型;在AUPR指标上取得了最好的效果.从AUC指标的结果看,除了TriModel以外,DEDTI比其他4个模型平均提高了3.1%,并在IC数据集上取得了最优结果,其他4个数据集上的结果与最优的TriModel差值不超过0.03.从AUPR指标看,DEDTI在5个数据集上的结果均达到最优结果,平均提高了19.08%,效果优于其他5个基准模型.综上可得,DEDTI在预测药物靶标相互作用方面具有出色的表现.
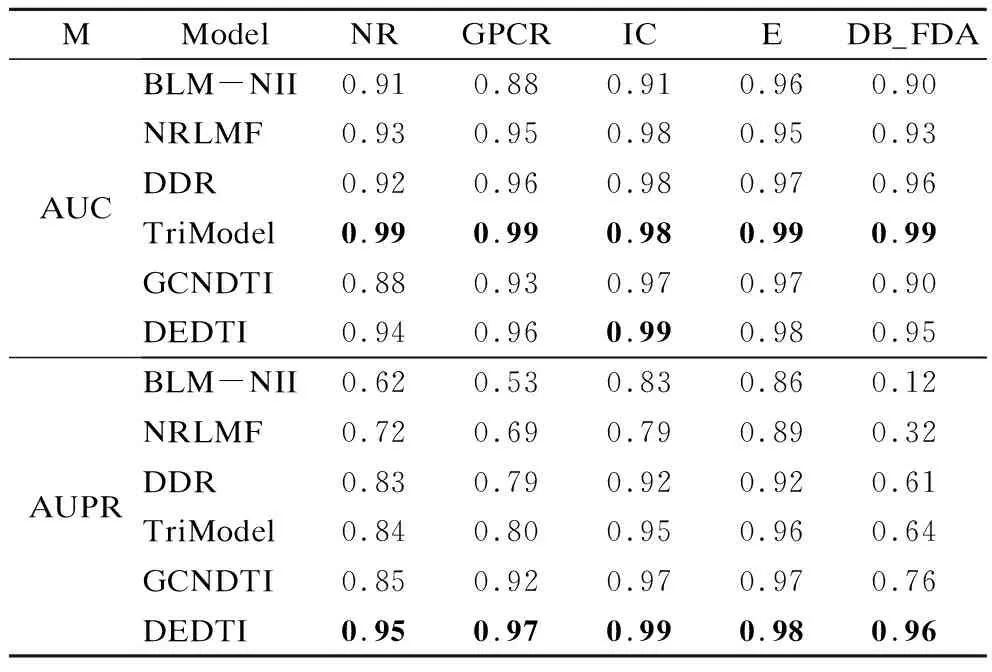
表3 5种数据集中,各种药物靶标模型指标结果Table 3 A comparision with atate-of-art models on standard datasets
DEDTI具有较强的鲁棒性,不易受数据集规模的影响.5种基准模型在各类数据集上的AUC指标均取得了较好的效果.但在不平衡样本更关注的AUPR指标下,使用矩阵分解和二分局部方法已无法实现有效地预测,而DDR和TriModel方法在小数据集上虽然表现不佳,但其预测有效性仍保持在60%以上.但在大型数据集DrugBank_FDA中,以上两种方法的预测有效性普遍低于60%,4种方法的AUPR值平均降低了59%、45.25%、25.5%和 24.75%.GCNDTI的平均降幅为16.75%,而DEDTI降幅仅8.25%.由此可见DEDTI在不同数据集上具有更强的稳定性,受数据分布和数据量的影响较小.
综合AUC和AUPR指标来看,相较于GCNDTI,DEDTI在稀疏数据中具有更强的表达能力.GCNDTI由于受到图神经网络层数的限制,存在模型参数优化不充分、训练轮数不足的问题.而DEDTI使用距离编码的方式,聚合了各中心节点与子图中其他节点的距离信息,因此所聚合的信息量数倍于GCNDTI所聚合到的信息量,所以DEDTI在稀疏数据集上更具优势.
GNN模型可以聚合邻域节点特征,因此其性能优于传统的矩阵分解和二分局部模型.距离编码增强了图结构数据在图神经网络中的表达能力,提高了GNN预测的性能.BLM_NII借助药物和靶标节点之间的相似性数据预测药物靶标相互作用.NRLMF使用矩阵分解的方法,通过药物靶标相似性信息提取特征矩阵.DDR借助不同跳数的子网中的路径,通过相似信息融合得到特征矩阵.TriModel使用三元组表示随机初始化的节点和边的邻接矩阵,生成向量表征表达数据特征,从而预测节点之间的评分.GCNDTI使用图卷积网络聚合网络信息,使得图卷积网络可以充分利用图上的异质信息预测DTI.以上5种基准模型均使用节点和相似性信息来预测DTI,但忽略了可以使用网络数据的结构信息对节点进行深层表达,同时增强自身的泛化能力.为了填充边信息,DEDTI使用边特征对节点进行距离编码,将边特征作为图神经网络的输入恰好符合图神经网络聚集邻域节点信息的特性,从而提高了图神经网络的预测能力.
DEDTI弥补了使用one-hot编码的模型缺陷,向模型中灵活地加入未经训练的新节点,且无需进行重新训练.相较于使用one-hot编码的直推式模型,DEDTI属于归纳式模型,此类模型使用预训练参数来预测新节点的相互作用关系,节省了模型大量的调参时间.
4.5 药物靶标发现与验证
验证图神经网络预测的未知药物靶标关系是否准确十分重要.在实验中,本文将10次预测结果排序,取top-k得分的结果进行验证.由于在数据处理部分,DEDTI通过掩盖一部分已知的相互作用,将其作为负样本,因此预测结果中会出现一部分已知的相互关系.如表4中的D01161和hsa2100,在训练集中为已知的药物靶标相互作用关系,在生成负样本数据时掩盖了这对数据,经过预测得到它们相互作用的标签,且预测得分较高.因此验证了DEDTI模型对于未知药物靶标关系预测的能力.
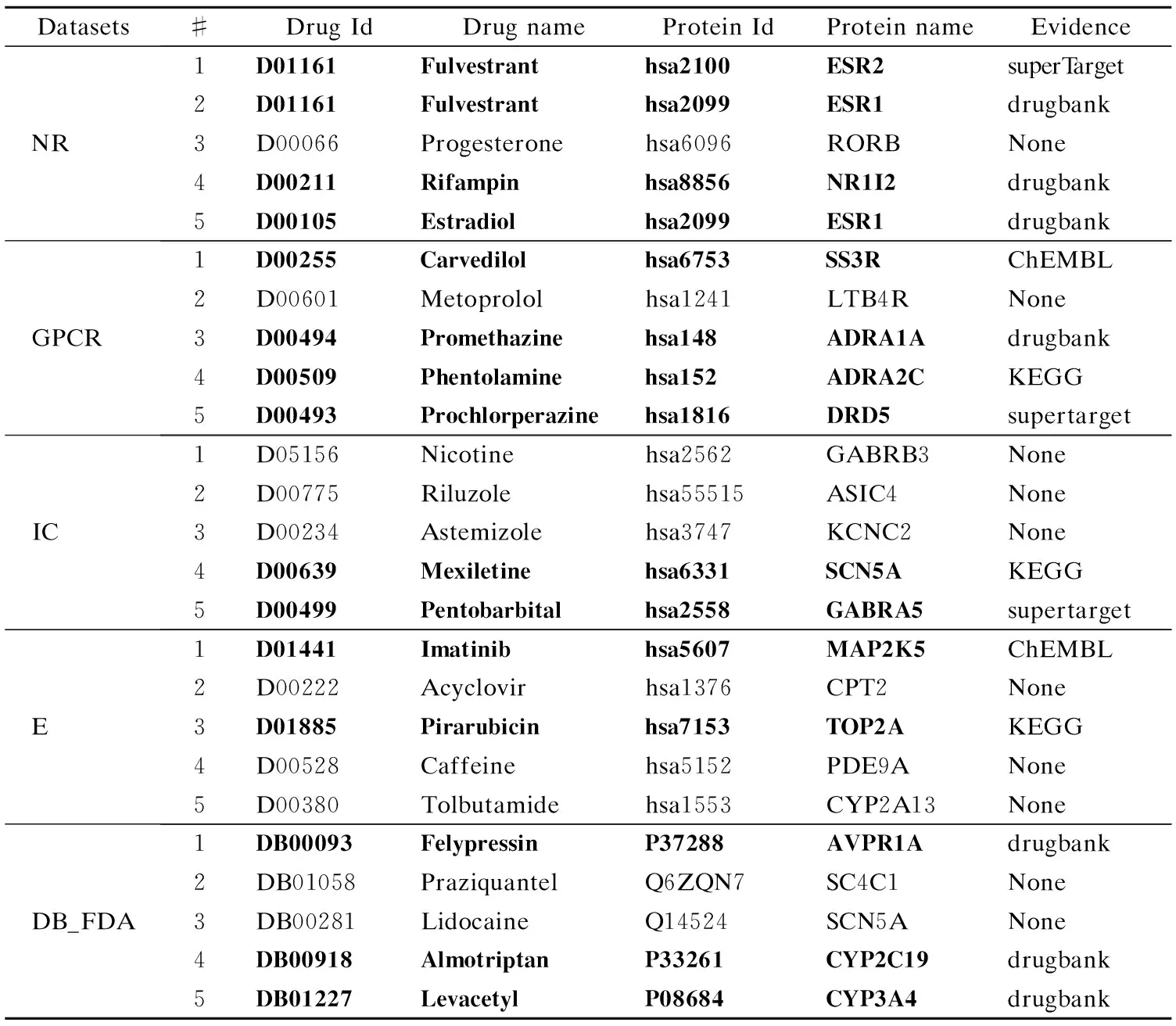
表4 验证各数据集中top-5 的预测结果Table 4 Validate the prediction results of top-5 in each dataset
去除已知的相互作用后,本文将top-K的数据对在常见的6个药物靶标数据库中进行未知边验证.在每个数据集中取排名前5的药物靶标对进行验证,最终的验证结果如表4所示.由此可见DEDTI对未知药物靶标作用关系的有效性.
统计数据集中top-10的预测结果在权威药物靶标数据库上验证DTI结果的数量,从表5中可知,与其他基准模型相比,DEDTI中得到验证的结果总数与TriModel和GCNDTI相近.在NR和GPCR数据集中,DEDTI验证为真的数量略低于IC和E数据集的结果,表明DEDTI在较大型数据集上的预测能力还有一定的提升空间.
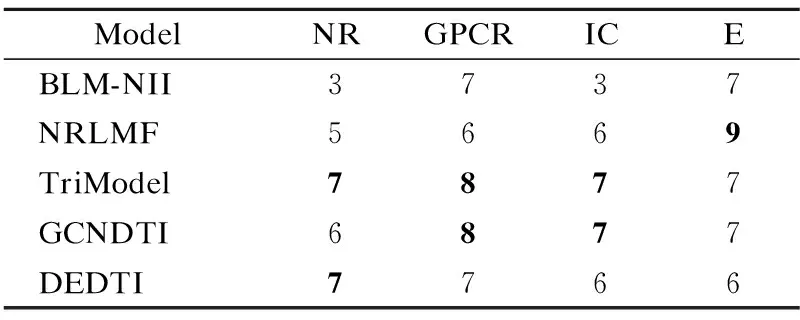
表5 top-10中得到验证的数目Table 5 Number of proven DTIs among the predicted top-10 unknown interaction
5 结束语
本文基于距离编码为图网络中的节点添加属性,解决了one-hot编码带来的直推式模型的问题.并提高了图网络数据在GNN模型上的表达能力来提升图神经网络的性能.本文提出了DEDTI 方法,详细介绍了子图选取、距离编码和特征生成的具体实现步骤,经过大量对比实验,确定了步长、编码方法以及用于预测结果的GNN网络超参数.将AUC和AUPR作为评价指标,对比了主流基准模型(包括GCNDTI)的性能,最后在经典的药物靶标数据库中验证了DEDTI方法预测未知药物靶标对的能力,并取得了明显的预测效果.后续将探索该模型在更大的交互数据集与实际业务场景药物靶标数据集中的表现,并将该方法泛化到中药方剂等图网络数据的实际应用中,以期发挥该模型更大的作用.
