CRISPR/Cas9系统介导的棉花GhNAC3基因编辑
2023-12-05王志军何宗铃张国丽焦灰敏桑玉伟马盼盼
王志军,何宗铃,张国丽,焦灰敏,桑玉伟,马盼盼*
(1 新疆农垦科学院 生物技术研究所/作物种质创新与基因资源利用兵团重点实验室,新疆石河子 832000;2 新疆生产建设兵团第一师农业科学研究所,新疆阿拉尔 843300)
棉花是中国最重要的纤维作物和重要的油料作物,新疆是中国最大的产棉区,然而,新疆棉花生产频繁受到干旱胁迫,对棉花安全生产造成较大不利影响。筛选棉花抗旱品种或者利用转基因技术提高棉花的抗旱能力成为有效的应对措施。目前,已有一些棉花抗旱相关基因的功能被报道,其中NAC转录因子家族是植物所特有,其不仅参与植物在生长发育方面的调节,在调控植物响应不同逆境胁迫方面也发挥着关键性作用。在植物中过表达NAC转录因子,可以增强植株对逆境的抗性,并且有些可以增强对多种胁迫的抗性[1]。目前,NAC转录因子在几十种植物中被发现,并对该家族基因的时空及诱导表达特性、结构及功能等方面的研究取得了较快的进展。在不同物种中均有报道,说明NAC转录因子参与植物抗旱性的调节。烟草NAC转录因子NtNAC053能增强烟草在干旱胁迫下的存活率[2]。番茄中过表达NAC家族转录因子SlNAP1显著提升番茄的抗旱性[3]。小麦抗旱基因TaNAC071-A可提高小麦在干旱胁迫下水分利用效率、存活率[4]。水稻中转录因子OsNAC2d正调控水稻响应干旱胁迫[5-6]。胡美玲[7]研究表明,棉花中GhNAC3受PEG和NaCl诱导显著上调表达,另外,对GhNAC3启动子分析发现,该基因的启动子区域具有响应干旱、盐和ABA的一些顺式作用元件。无论在拟南芥还是在棉花中该基因的超量表达都会提高转基因植物对干旱胁迫的抗性[7]。探究GhNAC3对于棉花响应干旱胁迫的作用,对棉花抗旱分子育种具有重要的意义。
基因组定向编辑技术对特定靶基因进行精确修饰,在作物基因功能研究和品种改良过程中具有广阔的应用潜力。CRISPR/Cas9基因组编辑技术,具有操作简便、成本低、效率高等优点。该技术只需表达Cas9核酸酶和1个经过设计的单导向RNA(sgRNA),就可以完成对基因组特定位点的切割,其序列特异性通过改变sgRNA中20个核苷酸的引导序列而实现[8],目前,CRISPR/Cas9系统已经用来在多种植物中定向突变目标基因,比如有研究利用该系统敲除了水稻的OsPDS基因,获得了水稻白化体突变植株[9]。
目前,虽然对NAC转录因子功能进行了广泛研究,但是NAC转录因子耐旱性相关的分子机制还需要进一步深入研究,在棉花上取得的研究进展还较为有限。棉花中NAC转录因子如何参与棉花抵御非生物逆境的调控机制还有待进一步研究,因此,本研究利用CRISPR/Cas9系统定向突变棉花的GhNAC3基因,获得了目标基因序列碱基发生缺失的转基因植株,并获得了植株同时黄化和矮化的突变体,这些研究结果可以充分证明利用CRISPR/Cas9系统定向突变棉花的基因,是研究基因功能的有效手段;另一方面,筛选鉴定抗旱相关的基因资源,对培育干旱耐受型棉花品种具有十分重要的意义。
1 材料和方法
1.1 试验材料
CRISPR/Cas9植物基因敲除用到的载体有pRGEB32-7和pGTR4(华中农业大学金双侠课题组馈赠),大肠杆菌菌株为T1,农杆菌菌株为LBA4404,均为本实验室保存。主要酶及试剂:Ex-Taq酶(Takara公司),琼脂糖凝胶DNA回收试剂盒、质粒提取试剂盒(南京诺唯赞公司)、植物基因组DNA提取试剂盒(北京天根)、Bsa Ⅰ(New England Biolabs公司)、Exnase Ⅱ(南京诺唯赞公司)。
1.2 试验方法
根据棉花GhNAC3(Gh_D02G0790)序列特征,利用CRISPR-GE在线软件http://skl.scau.edu.cn/home/在第一外显子区域设计编辑靶点(图1),并将2个靶点序列在棉花基因组数据库中进行序列比对,以消除非特异性靶点干扰。
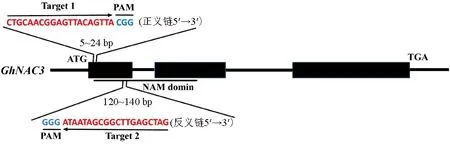
图1 GhNAC3的2个gRNA靶点位置Fig.1 Position of two gRNA targets in the GhNAC3 gene locus
1.2.1 小片段扩增和连接
设计好的引物由公司合成(上海生物工程股份有限公司),纯化级别为PAGE。gRNA+tRNA组合序列已经插入pGTR4载体,第1次PCR:1、2两片段分别从含有pGTR4载体的质粒中扩增。PCR扩增体系为20 μL体系,反应条件:95 ℃,4 min;95 ℃,30 s;55 ℃,30 s;72 ℃,20 s,3个循环;95 ℃,30 s;60 ℃,30 s;72 ℃,20 s,27个循环,最后72 ℃延伸5 min。第2次PCR使用重叠延伸PCR,使2个独立的PCR产物借助接头经 Taq 酶延伸成为融合片段,将2个小片段进行拼接,电泳检测,并用试剂盒进行回收、纯化,所用引物见表1。
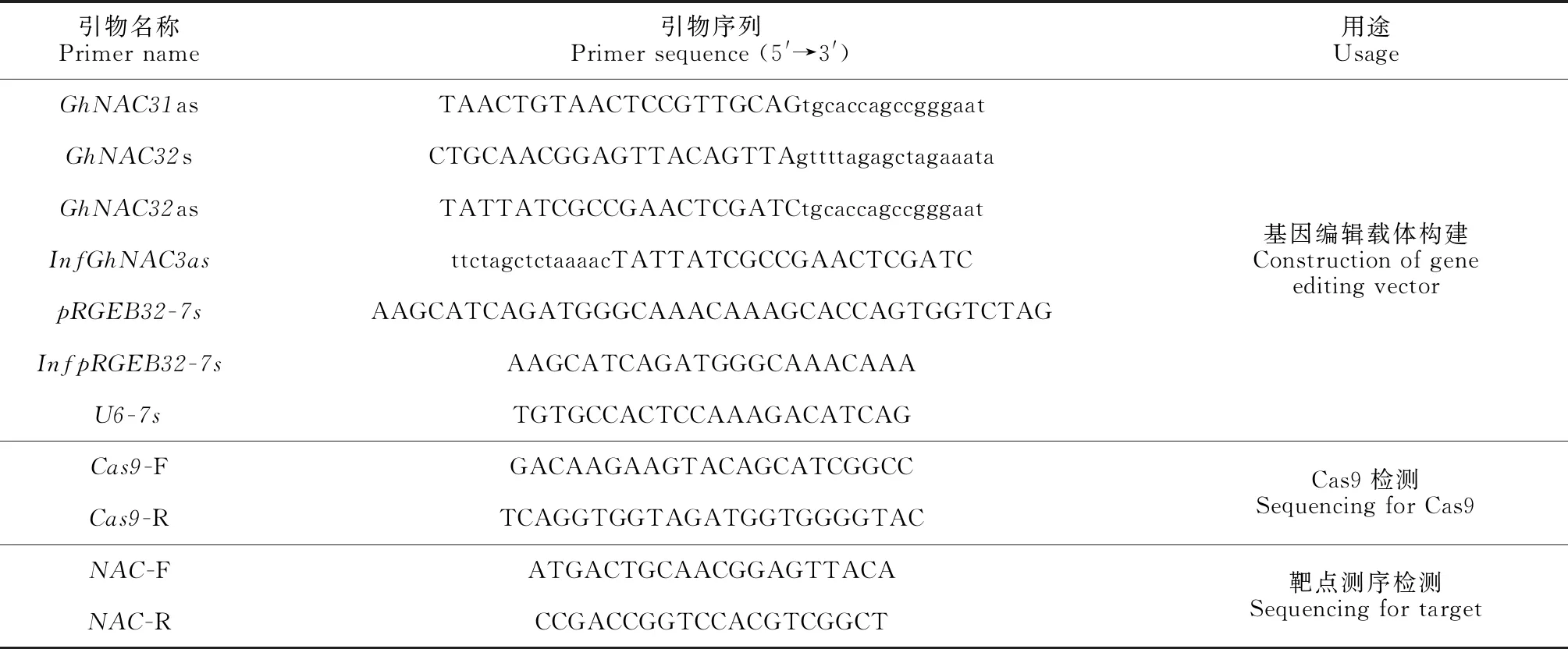
表1 所用的引物序列
1.2.2 载体酶切与连接
提取含pRGEB32-7载体的质粒,测定浓度,使用限制性核酸内切酶BsaⅠ进行单酶切。100 μL酶切体系中,其中pRGEB32-7载体10 μg(根据浓度计算体积),10× cut smart Buffer 10 μL,BsaⅠ酶4 μL,ddH2O补足至100 μL。37 ℃酶切6 h,可以适当延长酶切时间,使反应更充分。连接载体使用南京诺唯赞的非连接酶依赖型单片段快速克隆试剂盒(ClonExpress Ⅱ One Step Cloning Kit),连接方法参照试剂盒说明书,其中目的片段100 ng,线性化表达载体100 ng,Exnase Ⅱ酶1 μL,CEBuffer 2 μL。37 ℃水浴30 min,冰浴5 min。连接产物可以-20 ℃长期保存。
1.2.3 转化农杆菌
(1)取2 μL构建成功的pRGEB32-GhNAC3重组质粒加入100 μL冰浴的农杆菌感受态细胞(LBA4404),轻轻混匀,放入电击杯中,电压设置为1 800 V,电击转化农杆菌;(2)将电转值大于5的转化质粒迅速加入500 μL LB液体培养基(不含抗生素),28 ℃培养3 h,5 000 r/min离心5 min,去除上清500 μL,剩余菌体悬浮,涂布于含1/1000抗生素(Kan和Rif)的LB固体培养基中,28 ℃倒置培养48 h;(3)挑取单克隆菌株,含1/1000抗生素(Kan和Rif)的LB液体培养基,220 r/min培养16~24 h,PCR鉴定为阳性菌株。
1.2.4 pRGEB32-GhNAC3棉花遗传转化
含pRGEB32-GhNAC3的基因编辑载体的LBA4404农杆菌浸染陆地棉YZ-1的下胚轴,具体操作步骤参照朱守鸿[10]方法并稍作改良。
1.2.5 基因编辑株系的靶点分析与鉴定
为检测GhNAC3的靶点突变情况,在2个靶点序列两侧分别设计引物GhNAC3-F/GhNAC3-R,扩增产物长度为507 bp,通过植物DNA提取试剂盒提取转基因植株叶片的DNA,进行PCR扩增检测,扩增片段回收纯化连接T载体,转化大肠杆菌感受态T1,在含1/1000抗生素(kan)的LB培养基摇菌,提取质粒,寄送生工生物工程(上海)股份有限公司进行测序,并将测序结果与野生型DNA的扩增测序结果进行比对分析,鉴定突变植株的基因变异类型。
2 结果与分析
2.1 GhNAC3靶点设计和CRISPR-GhNAC3载体构建
利用在线数据库cotton FGD(https://cottonfgd.net/)下载GhNAC3(Gh_D02G0790)的全长序列,CDS序列和氨基酸序列,GhNAC3位于棉花D02染色体,DNA序列编码区全长为1 241 bp,由3个外显子和2个内含子组成,将上游靠近启动子区域第一外显子作为突变区域设计靶点,利用靶位点设计软件CRISPR-GE(http://skl.scau.edu.cn/home/)[11]在线设计,并将2个靶点序列在棉花基因组数据库中进行序列比对,以消除非特异性靶点干扰。选取第一外显子的正义链+25 bp到+27 bp处的CGG为PAM(protospacer-adjacent motif)序列,+5 bp到+24 bp之间的20 bp碱基作为靶点1序列,第一外显子的反义链+117 bp到+119 bp处的GGG碱基为PAM序列,+120 bp到+140 bp区间的20 bp碱基作为靶点2的编辑序列(图1)。
GhNAC3的氨基酸序列结构分析表明,GhNAC3氨基酸序列具备典型的NAM结构域,在氨基酸序列11~134之间,具有典型的NAC类蛋白的结构特征,共包含1个结构域,编辑靶点2位于该结构域。
2.2 GhNAC3基因遗传转化
为使重组CRISPR/Cas9载体中的2个靶点序列由AtU6启动子驱动,通过酶切、连接将双链靶点序列与AtU6-sgRNA载体连接,将回收纯化的Atu6-sgRNA片段与BsaⅠ酶切线性化的CRISPR-Cas9(pRGEB32-7)载体连接(图2),通过T4 DNA连接酶进行连接并转化大肠杆菌感受态细胞T1,在含Kan(50 mg/mL)抗性的LB平板上挑取阳性克隆提取质粒并进行酶切验证,经测序分析表明,重组载体中的2个靶点序列(图3)与设计序列一致(表1),说明靶点准确连接到CRISPR/Cas9载体上,鉴定正确的重组质粒电击转化农杆菌感受态(LB4404)。参照朱守鸿[10]的棉花遗传转化方法,转化受体材料YZ-1。

LB.左边界;RB.右边界。图2 2个靶位点sgRNA表达盒与CRISPR/Cas9组装成的表达载体LB. Left border; RB. Right border.Fig.2 Cloning of two sgRNA cassettes into the CRISPR/Cas9 vector
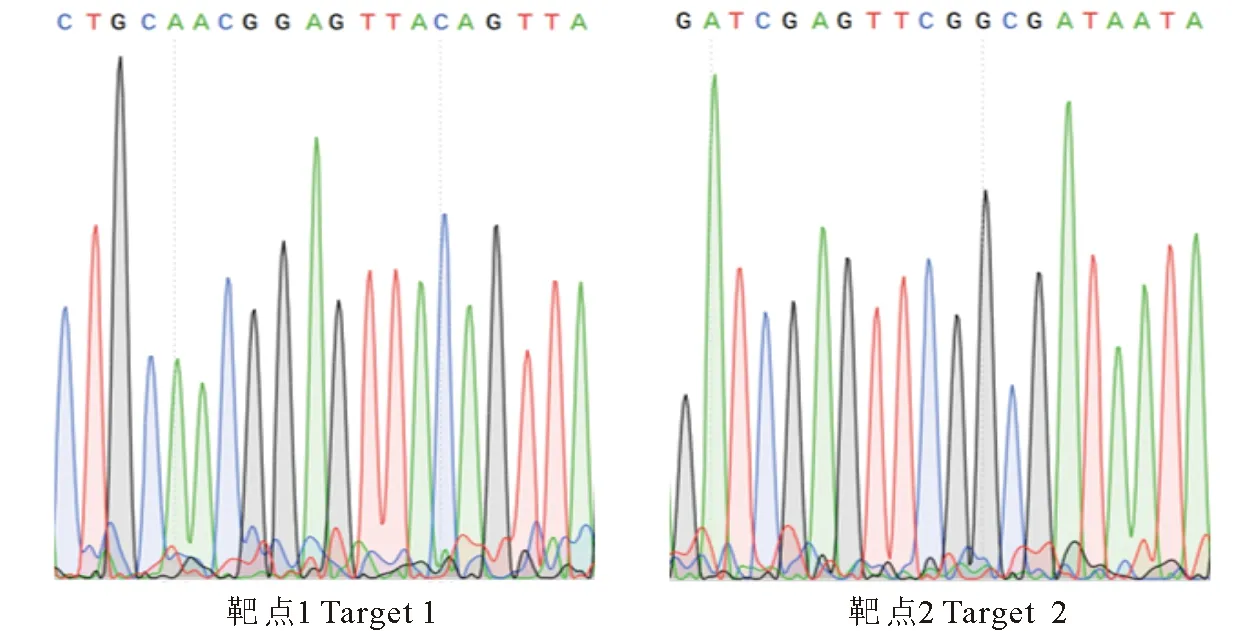
图3 CRISPR-GhNAC3 重组载体中2个靶点序列测序结果Fig.3 Sequencing results for the two target sequences inserted in CRISPR-GhNAC3 recombinant vector
无菌苗的获得,种子去壳后,用0.1%的升汞消毒15 min,用无菌水清洗6次;将消毒后的种子接种于无菌苗培养基M0(1/2 MS 培养基+15 g/L葡萄糖+6.5 g/L琼脂粉),28 ℃暗培养5~7 d备用;愈伤组织的诱导,无菌条件下,将暗培养5~7 d的YZ-1幼苗,使用无菌手术刀切除根部和子叶,将下胚轴切成0.5~0.8 cm的小段备用;用OD值为0.8左右的pRGEB32-GhNAC3农杆菌菌液(含50 mg/mL乙酰丁香酮)侵染下胚轴15 min;去除多余菌液,迅速将下胚轴倒入无菌滤纸上,铺平,用滤纸将下胚轴表面的菌液吸干,20 ℃,共培养基暗培养48 h,28 ℃选择培养基含1/1000抗生素(Cef和kan)光照培养,25~30 d继代1次;选择培养70 d左右,将下胚轴(褐化严重的去除)移至含1/1000抗生素(Cef和kan),2,4-D减半愈伤诱导培养基中,25 d左右继代1次,直至出现颗粒状的胚性愈伤;选择胚性愈伤继代于分化培养基上,20 d 左右继代1次,及时淘汰未分化愈伤组织,直至出现黄绿色颗粒的球形胚;挑取球形胚继续在分化培养基诱导,直至子叶胚的出现;将萌发出幼根和幼叶的子叶胚移至生根培养基中,继续培养,直至成苗。
获得组培再生棉苗,再生苗水培炼苗后直接移栽到营养土中,获得生长发育良好的转基因再生植株(T0),组织培养再生苗过程见图4,第2年将编号为5的T0代种子播种于大田,获得了同时黄化和矮化的突变体3株(图4)。根据Cas9 DNA序列,设计特异性引物,扩增目的片段大小为512 bp,提取组培获得的正常再生植株的DNA,对Cas9蛋白检测为阳性株的进行靶位点区域测序。结果表明,CRISPR-GhNAC3的再生植株中,有16株检测到Cas9蛋白基因,表明CRISPR-GhNAC3载体被成功转入YZ-1受体,靶位点可能被成功编辑(图5)。

图4 CRISPR-GhNAC3遗传转化过程Fig.4 Process of genetic transformation of CRISPR-GhNAC3

图5 GhNAC3棉花株系PCR验证Fig.5 The PCR validation of CRISPR-GhNAC3 cotton lines
2.3 基因编辑类型检测
为了进一步确定GhNAC3基因的靶标位点是否产生突变,对PCR检测到Cas9蛋白基因的5、6、7、8、9、10的T0代植株的目标基因靶点及其上、下游序列进行PCR扩增和测序分析(图5和图6)。以T0代GhNAC3突变体棉花DNA为模板的PCR反应中,扩增片段大小为507 bp,对PCR扩增产物进行测序分析表明,共有6株阳性株靶点序列发生缺失突变,其中7,8,9株在2个靶点区域均发生碱基片段缺失,靶点1缺失片段大小为23~28 bp不等,靶点2缺失片段大小为3 bp,以上结果表明靶点的缺失会导致氨基酸序列移码变异,从而产生表型变异突变体。编号为5的T1代植株,其在靶点1区域出现大小为26 bp的片段缺失,田间出现同时黄化和矮化的表型突变,共3株,也可以验证上述结论。

红色字母为靶点序列,蓝色大写字母表示PAM序列,删除线表示缺失碱基,-.缺失,WT.野生型。图6 转化植株编辑靶点的PCR产物测序序列与野生型(WT)序列的比对结果The red letters are the target genome sequence; The blue capital letters denote PAM;Dashes strike through indicate the deleted bases; -. Deletion; WT. Wild type.Fig.6 Sequence alignment of the edited targets for the GhNAC3 mutants and WT line
3 讨 论
新疆是中国主要植棉区,2022年新疆棉花产量539.1万t,较上年增加26.2万t,占全国棉花总产量的比重持续提升,达到90.2%,创历史新高,新疆棉花总产、单产、种植面积、商品调拨量连续28年位居全国第一(国家统计局)。但是,近年来新疆棉花种植业的发展越来越受到资源环境约束,最重要的一点就是水资源不足[12],干旱已经成为新疆棉花产业安全的最大威胁。干旱可以严重制约农作物的生长,导致产量降低,同时在干旱胁迫下,植物的生长、发育乃至光合与呼吸作用都会受到不良影响,甚至会改变植物叶片大小,影响植物茎杆延伸和根的增殖,扰乱植物体内水分平衡[13]。因此,提高作物抗旱能力可以直接影响产量。传统育种有着操作复杂,育种周期长,利用生物技术手段研究农作物在干旱胁迫条件下的分子机制,创造新的耐旱品种成为了农作物抗逆品种改良的研究热点[14]。
目前,克隆的抗旱基因已有较多报道,其中NAC转录因子在调控植物响应逆境胁迫方面也发挥着至关重要的作用。在植物中过表达NAC基因发现,部分该家族基因可以增强植株对逆境的抗性,并且有些可以增强对多种胁迫的抗性[1]。超量表达拟南芥NAC家族基因ANAC019、ANAC055和ANAC072,超表达植株耐旱性得到改善,同时发现多个与逆境胁迫响应相关的基因被诱导表达[15]。在水稻中超表达SNACl能够显著增强水稻对干旱胁迫的忍耐能力,正常生长环境下,转基因水稻表型无明显差异。但是,在生殖生长期进行严重的干旱处理,发现,超量表达SNACl的水稻材料坐果率高于对照22%~34%,转基因水稻也可以显著增强在营养生长期的干旱胁迫[16]。超表达大麦中SNACl同源基因HvSIVACl可以增强植株抵御干旱胁迫能力,表现为改善叶片光合活性,减少叶片的水分蒸腾作用,并增加生物产量[17]。康振生院士团队通过全基因组关联分析方法,克隆了1个小麦抗旱基因TaNAC071-A,过表达该基因则能够显著增强小麦抗旱性[18]。水稻中过表达OsNAC14基因可提高水稻耐旱性[19]。水稻中的OsNAC2通过ABA介导的通路在水稻干旱和耐盐性中起正向调控作用,OsNAC05则负责根径增大和抗旱性;同时,OsNAC6也可增强水稻抗旱性[20]。一些NAC转录因子则通过调控胁迫相关基因应对胁迫,过表达OsNAC1的转基因植株,通过调控许多胁迫相关基因,如OsSRO1c和OsPP18,显著提高了水稻对干旱和盐的耐受性[21]。
番薯(Ipomoeabatatas)中ItbNAC62启动子中发现了ABA和茉莉酮酸甲酯(methyl jasmonate,MeJA)响应的顺式作用元件(cis-acting elements,CAEs),表明该基因通过ABA或MeJA信号通路参与了抗旱性[22]。在辣椒(Capsicumannuum)中,CaNAC46可通过上调LBD18和IAA4的表达来促进侧根的形成和根的伸长,从而增强其对干旱和盐胁迫的耐受性,是正调控因子[23]。在云杉(Piceawilsonii)中,过表达PwNAC11显著提高了ROS的清除能力,并有助于保护干旱胁迫下的过氧化损伤,同时PwNAC11作为一种ABA信号因子,通过促进气孔的关闭和ABA的合成,直接或间接地提高了植物对干旱胁迫的耐受性[24]。一些NAC转录因子通过调控ROS含量和ABA合成来应对胁迫。在小麦中,TaNAC69的过表达增强了小麦的脱水耐受性[25]。此外,其他作物中也有关于NAC基因响应干旱胁迫的报道,如鹰嘴豆、棉花、玫瑰等[26]。
CRISPR/Cas9系统具有方便、高效、周期短以及载体结构简单等优点。高效定向的CRISPR/Cas9系统能使受体材料的目的基因产生突变,达到目的基因的编辑效果,被广泛应用于各种作物中,比如水稻[27]、玉米[28]和烟草[29]等。王加峰等[30]利用CRISPR/Cas9技术对调控水稻产量千粒重的基因TGW6定点编辑,T0代该基因突变频率约为90%,其中纯合缺失突变率高达51%,T1代纯合缺失突变体千粒重显著增加。中国水稻研究所[31]采用CRISPR/Cas9技术对控制水稻香味的BADH2基因进行敲除,结果表明突变体材料中香味物质显著增加。Li等[32]利用CRISPR/Cas9技术以非同源重组的末端(NHEJ)修复方式对水稻5-烯醇丙酮酸莽草酸-3-磷酸合成酶基因EPSPS进行编辑,实现了EPSPS基因保守区域2个重要氨基酸的定点置换,在T0代获得具有草甘膦抗性的突变体材料。Wang等[33]利用CRISPR/Cas9技术修饰稻瘟病侵染效应因子基因(OSERF922),以提高水稻对稻瘟病的抗性。Tang等与袁隆平团队[34]利用CRISPR/Cas9技术敲除水稻中与镉离子吸收和积累相关的基因,获得了抗镉毒害的水稻品种。杜邦先锋公司利用CRISPR/Cas9技术,将糯玉米性状直接引入具有优良性状的杂交种中,解决了糯玉米产量低的问题,同年,该公司利用CRISPR/Cas9技术将1个玉米内源启动子GOS2精确插入到玉米中乙烯应答调控因子ARGOS8的5′端,获得耐旱材料[35]。本研究以棉花抗旱基因GhNAC3基因为对象进行研究,构建了2个单靶点CRISPR/Cas9重组载体,采用U6启动子启动sgRNA,35S启动子高效表达Cas9蛋白,最终达到成功编辑GhNAC3基因的目的。
