自动化合成生物技术在DNA组装与微生物底盘操作中的应用
2023-11-21陈永灿司同张建志
陈永灿,司同,张建志
(中国科学院深圳先进技术研究院,深圳合成生物学创新研究院,中国科学院定量工程生物学重点实验室,广东 深圳 518055)

微生物细胞工厂在现代工业生物技术中起着核心作用。然而,天然微生物的产物种类有限且产量较低,无法满足各个领域对化学品的生产需求。传统的微生物育种方法主要依赖于天然微生物的筛选和非理性诱变育种技术,随机性强且耗时长[1-3]。为了提高生产水平,研究者利用基因工程技术对细胞代谢途径进行了改造和设计,构建异源分子的生物合成途径。然而,由于细胞网络的复杂性和非线性特征,人工引入的外源通路和底盘基因组突变往往难以实现预期功能,需要对大量基因元件、线路和系统进行反复合成和调试,缺乏对细胞工厂的理性设计能力[4],这导致了构建人工微生物细胞工厂时间和成本的增加[5]。
为了解决这些困难,依托自动化设施平台的自动化合成生物技术应运而生。自动化合成生物设施平台又称自动化合成生物铸造厂(biofoundry),以合成生物学基础研究为理论基础,把自动化工业的智能制造理念引入到合成生物学研究中。基于智能化、自动化及高通量设备和机器人系统,结合设计软件与机器学习,开发自动化合成生物技术,快速、低成本、多循环地完成“设计-构建-测试-学习”的闭环,服务于包括细胞工厂构建在内的合成生物学研究与产业应用[6]。目前,在全球范围内相继投资建设了数十个自动化合成生物铸造厂,并成立了“全球合成生物设施联盟”(Global Biofoundry Alliance,GBA),进行基础设施、数据资源、合成生物标准的开放共享,共同应对自动化合成生物研究的技术难题[7]。自动化合成生物技术依托合成生物设施软硬件平台,将生物体系设计、构建、表征与分析等合成生物技术工艺进行自动化开发与应用,以减少人员干预并降低错误率,提高通量和可重复性[8]。
自动化合成生物技术的一个重要应用领域就是开发标准化生物元件库、模块化的DNA组装技术和精准的基因编辑方法,以实现对细胞工厂的规模化设计、组装和改造[9-12]。在构建人工细胞工厂的过程中涉及 “设计-构建-测试-学习”(designbuild-test-learn,DBTL)的循环步骤,其中“构建”是最耗时、耗力的过程之一[13]。细胞工厂“构建”步骤主要需要完成两类实验任务:①工程DNA的构建;②底盘细胞的操作[8,12]。为了自动化完成这些实验任务,需要开发对应的硬件平台、软件系统和工艺流程。在工程DNA的构建过程中,涉及基因合成、PCR扩增、酶切、组装、提取纯化、测序等多个步骤。而微生物细胞底盘的操作包括工程DNA转化、菌落涂布、菌落挑取、细胞裂解和荧光分选等验证流程。开发相应的硬件和软件系统是实现生物学实验操作自动化的关键。在硬件系统方面,标准化的实验容器及其配套设备,包括机械臂、液体工作站、分液器、封膜仪和撕膜仪等,均是必需的。而在软件系统方面,集成软件可以控制自动化实验仪器并管理物料与数据,实现实验过程的自动化控制和数据分析。针对Thermo、TECAN等主流合成生物自动化品牌商的设备特点以及在自动化设施平台搭建过程中的设备选型方案,Thurow K.和Junginger S.进行了专门的比较和论证[14]。更进一步,自动化合成生物研究通过引入人工智能算法,大幅提升研究过程中实验对象、方法、技术的标准化和模块化水平,不但可以快速积累大批优质生物元件,还可以产生高质量、大规模的实验数据,实现数据驱动的DBTL自动化闭环,不断提升细胞工厂的构建速度、研发效率和理性设计水平[8,12,15-17]。本文将重点针对自动化细胞工厂构建过程中的DNA组装及微生物细胞操作两个自动化合成生物技术比较关注的方向的进展进行介绍。对于自动化基因编辑,特别是基于CRISPR(clustered regularly interspaced palindromic repeats)家族及其衍生技术的自动化基因组工程[18-21],以及DBTL循环中的其他步骤,例如“设计”[22]、“测试”[12,23]和“学习”[24-25],读者可参考其他相关的专业文献报道。
1 自动化DNA组装
为了获得符合下游实验要求的DNA组装产物,并提高DNA组装自动化的成功率和效率,需要对自动化DNA组装进行预先顶层设计。合成生物学设计是除集成控制软件外合成生物自动化设施在软件方面的另一个重要组成,主要包括零件与系统设计、DNA组装图谱设计[26]。其中,DNA组装图谱设计以工程DNA序列为输入,基于所计划采用的DNA组装方法,输出产生目标DNA序列所需的元件序列和组装路径。DNA组装图谱设计工具是自动化DNA合成和组装的关键共性技术。Vector NTI[27]是早期开发的一款商业化序列分析和设计工具集成套件,但无法满足利用标准化元件进行大量DNA组装的设计需求。美国能源部Agile Biofoundry研发了网页版DNA组装设计软件j5[28],可根据用户选择或软件推荐的组装方法设计组装图谱和组装过程,并编译产生可用于移液工作站、微流控等的操作指令,大大提高了设计效率。波士顿大学Douglas Densmore团队开发了RAVEN工具[29],不仅实现了DNA设计自动化,还可通过自主学习算法基于实验结果以交互方式优化组装设计。此外,伊利诺伊大学iBioFAB团队研发了DNA组装设计软件iBioCAD[30],可基于酵母体内组装、Gibson组装、连接酶循环反应(ligase cycling reaction, LCR)和Golden Gate等不同方法进行工程DNA的构建设计;该团队还开发了PlasmidMaker[31],允许用户从网站前端界面提交质粒构建需求,由系统自动化完成引物设计后根据任务清单触发机器人系统完成自动化DNA组装。爱丁堡大学EGF团队针对同源臂设计、序列优化、组装规划、计算机辅助酶切、测序验证等DNA组装流程中的不同环节,开发了包含一系列DNA组装工具的CUBA软件平台(https://cuba.genomefoundry.org/)。然而,针对合成生物自动化发展的新需求,如大规模DNA序列的组合式并行设计、大片段DNA可视化分析和操作、基于机器学习的DNA组装反馈优化等,还需要开发更多的DNA组装设计工具。
基于上述生物设计自动化(bio-design automation,BDA)软件完成DNA组装方案设计后,需要采用一系列高通量DNA组装方法快速、高效地完成从单个转录单元到整个合成生物系统的拼装和构建[32]。这些方法包括如下几类:①基于限制性内切酶的组装技术,如Golden Gate、BioBrick、Flexi Cloning、MASTER等方法,此类方法基于限制性内切酶使基因片段和载体产生互补的黏性末端序列,并通过DNA连接酶进行无痕组装;②基于同源重组的组装技术,包括Gibson、Gateway、Echo Cloning、Creator等体外酶法组装,以及DNA assembler等酵母胞内组装方法,此类方法简便高效,既可用于单片段的克隆,也可用于多片段与载体的组装,且不受到酶切位点的限制;③基于寡核苷酸的架桥法组装技术,该方法通过设计与相邻DNA的两端序列互补的单链桥接寡核苷酸(bridging oligo),在较低温度下进行退火,从而使上游片段的3′端与下游片段的5′端连接,将2个DNA片段组装成单个的线性片段。然后以组装好的线性片段为模板来组装互补链,通过多次热循环,将线性DNA片段组装成环形质粒;④基于可编程核酸酶的组装技术,如基于CRISPR系统Cpf1蛋白的大DNA片段体外编辑CCTL方法(Cpf1-assisted Cutting and Taq DNA ligase-assisted Ligation)、基于Ago蛋白的PlasmidMaker方法等。
其中,Golden Gate组装方法[图1(a)]和Gibson组装方法[图1(b)]都可以一步实现转录单元的高效率、规模化拼装,有利于节省后续对构成转录单元的生物元件进行优化和替换的时间,是自动化合成生物研究中最常用的DNA组装方法[32]。Gibson方法依赖T5核酸外切酶、DNA聚合酶和TaqDNA连接酶的组合作用。首先T5核酸外切酶作用于待组装的双链DNA片段后生成可互补配对的黏性末端并自发配对成双链DNA结构,利用DNA聚合酶将空隙补齐,在TaqDNA连接酶作用下,缺口被连接形成完整的DNA分子。Golden Gate组装方法基于ⅡS型限制性内切酶在同一反应体系中进行酶切和质粒组装。ⅡS型限制性内切酶,如BsaⅠ、BsmBⅠ等,在其识别序列的外侧进行切割产生4 bp的黏性末端。因此,只需在相邻片段上合理地设计4 bp的互补序列接口(linker),就可进行无痕组装。理论上,4 bp互补区域有256种不同组合,可实现多片段一次性组装,且不受重复序列的影响。同时,通过多种ⅡS型限制性内切酶的替换使用,可实现模块化的多轮逐级组装(hierarchical assembly)。但在实际应用中,不同接口序列的组装效率、特异性不同,需要进行优化[30]。此外,基于Golden Gate方法的DNA组装需要移除目的基因和质粒载体序列中的BsaI、BsmBI等酶切位点,会增加DNA组装工艺的复杂性和成本。
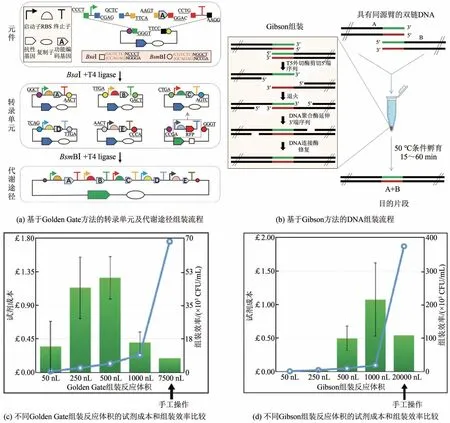
图1 Golden Gate和Gibson组装自动化[33]Fig. 1 Automated DNA assembly methods[33]
研究者分别采用Golden Gate[图1(c)]和Gibson[图1(d)]组装方法以红色荧光蛋白为报告基因完成了相应转录单元的半自动化构建[33]。研究者主要基于超声纳升移液仪Echo 550、自动化PCR仪和384孔板,实施了2片段DNA的快速组装和工程DNA转化等生物学操作,并通过Sanger测序和凝胶电泳的方法对质粒进行了验证。基于Echo,单次移液体积可减少至2.5 nL,连接反应体系缩小至50 nL,是常规人工操作所需体积的1/20~1/100,不仅实现了较高的组装成功率,亦可有效地降低试剂和连接酶的成本。基于Golden Gate组装方法,伊利诺伊大学赵惠民团队[11]开发了类转录激活因子感受器核酸酶(transcription activatorlike effector nucleases, TALEN)表达质粒的自动化组装方法。研究者将192个人基因组位点信息输入自主开发的DNA组装设计软件Script Generator对TALEN序列进行设计,并生成自动化组装流程脚本,通过连接酶反应、大肠杆菌转化和培养、质粒提取、酶切验证等过程的自动化运行,iBioFAB平台在17 h内对444种DNA元件或试剂进行了3648步移液工作,实现了15个DNA片段的一步法组装;每天内可以构建400对编码TALEN蛋白的DNA序列,正确组装效率达96%以上,且每对TALEN表达载体的构建成本仅为2.1美元(商业售价的0.3%)。
此外,TAR(transformation associated recombination)、BASIC(Biopart Assembly Standard for Idempotent Cloning)、USER(uracil-specific excision reagent)等方法也因其可实现多片段、调控元件、催化元件、代谢途径等的一步无痕组装,被应用于自动化设施平台。例如,美国Amyris公司采用TAR组装方法,可在酿酒酵母体内实现最多12个DNA片段的一次性组装,正确率>25%;London Biofoundry基于BASIC方法和自动化设施平台,搭建了全自动化的多片段DNA构建平台DNA-BOT(图2)[34]。DNA-BOT读取元件和接头存储信息以及构建设计信息并生成自动化脚本文件,分配给4个功能执行模块:①剪接反应模块,将所需生物元件从其保存质粒上剪切成DNA片段,并在其两端分别连接相应的Prefix和Suffix接头序列;②核酸纯化模块,采用磁珠法纯化带有21 bp黏性末端的生物元件;③组装模块,将生物元件与相应的载体骨架进行组装;④转化模块,将构建的组装体系转化至大肠杆菌感受态细胞,并涂布于筛选平板,用于后续的分析验证。基于该平台,通过1578步移液操作,38个磁珠法核酸提取、96个热激转化(96孔板)的自动化操作,一步实现了88个各包含3个DNA片段的质粒组装。与传统人工所需的5 h操作时间相比,其操作时间(非运行时间)缩短至1.5 h,每个质粒的构建成本仅为1.5~5.5美元,同时具有较高的质粒构建成功率。
2 自动化细胞底盘操作
在人工细胞工厂的构建阶段,微生物细胞底盘的操作是自动化合成生物技术主要关注的一个方面。底盘细胞是细胞工厂的“硬件”基础,理想的细胞底盘需要具有较好的稳定性和鲁棒性,并且在不被外源工程DNA元件影响原始代谢和生长的情况下与其进行适配[35-36]。然而,自动化设施平台的终端产品,即人工细胞工厂,仅仅是目标化合物工业化生产的起点。而且,自动化设施平台改造细胞工厂不是一蹴而就的,在生物过程放大过程中仍然需要对细胞工厂进行重新设计、迭代,甚至需要更换更加合适的细胞底盘。自动化合成生物技术有助于揭示微生物复杂表型的分子机制,拓展对传统微生物的理解,进一步利用这些信息定制目标细胞工厂。
目前,在绿色生物制造领域应用比较成熟的细胞底盘包括大肠杆菌、酿酒酵母、谷氨酸棒状杆菌等模式微生物。在手工操作方面,模式微生物的生物学操作工艺成熟,大肠杆菌和酿酒酵母作为应用最广泛的模式微生物,具有培养条件简单、生长繁殖快、遗传工具成熟、遗传背景清晰等特点,也是目前研究者最为关注、容易实现自动化操作的细胞底盘[10,37]。随着生物技术从早期的形态学观察发展到分子水平的分子生物学、系统生物学及合成生物学,非模式生物与模式生物之间的技术障碍正在逐渐缩小。模式生物建立的部分方法和工具可以直接或改造后应用于非模式生物,指导非模式生物的元件挖掘、细胞工厂的建立和改造优化等。然而,由于跨物种的不兼容性,某些基于模式微生物开发的生物技术很难平移至非模式微生物,如RecA蛋白介导的同源重组功能。尽管一些前沿技术为在非模式微生物细胞工厂中的应用进行了相应的方法改造,但仍有很大的探索空间。采用非模式微生物作为细胞工厂改造的宿主面临的主要挑战不仅仅是缺乏基因工具,更重要的是对其代谢网络和调控信息的理解有限[38]。因此,需要对不同生长环境下的非模式微生物的代谢情况进行详细的分析,例如遗传或化学扰动,以深入了解代谢及调控机制。自动化合成生物技术自动化、标准化、高通量、信息化和智能化的特性可加速重要非模式菌株的系统开发和应用推广[39-40]。
针对微生物细胞底盘的操作而言,主要涉及细胞培养、工程DNA转化、菌落涂布、菌落挑取、细胞裂解、荧光分选等验证流程。在设备方面,除了使用标准化的实验容器(例如符合SBS标准的96孔、384孔微孔板等)以外,还需整合与之适配的多通道自动化移液机械臂的液体工作站、微孔板离心机、分液器、封膜仪、撕膜仪、自动化震动培养箱/摇床、自动化PCR仪、高通量毛细管电泳仪、自动化菌落涂布挑选仪等以完成包括DNA构建在内的细胞底盘操作。此外,还需要通过硬件接口将这些仪器设备与机械手、传送带、AGV智能小车等自动化转运设备对接,从而按照实验流程进行样品、试剂、耗材等的传输。最终,实现噬菌体、细菌及真菌等不同尺度生命体的自动化构建与测试[8,12,17,41]。但是,针对特殊环境需求的非模式细胞底盘则需要对自动化设施进行定制化设计、装备或升级。例如,定制化搭建厌氧功能岛以维持全程无氧环境;搭建自动化光照培养反应器以适配光照细胞的培养和基因操作等。下面将对微生物底盘自动化操作过程中最为关注的细胞培养和DNA转化环节进行介绍。
2.1 自动化细胞底盘培养
标准化的自动化DNA组装构建、细胞底盘操作等生物学流程,基本上都是基于微孔板(96孔板等)完成移液、微生物培养或储存甘油菌等操作。与非自动化方法一致,自动化微生物细胞培养也包括振荡培养和静置培养两种方式。振荡培养有利于改善氧气供应和营养分布,促进微生物生长,对大多数微生物的常规培养来说通常是首选。商业化设备方面,Thermo Fisher公司开发了Cytomat系列,LiCONiC公司开发了StoreX系列,WakenBtech公司开发了NeXCell R系列,HighRes Biosolutions公司开发了SteriStore系列等自动化培养箱。以Thermo Fisher 公司Cytomat系列为例,这些培养箱内置即用型的条形码读码器对微孔板进行标记和位置追踪,通过与现有自动化硬件或平台进行无缝集成。根据型号的不同,最短取板时间低于10 s,最大可同时存储240块深孔板、504块96或384孔板或672块1536孔板,并可智能化控制温度和湿度,各平板分别设置振荡速度。此外,一些研究团队自研开发了基于微流控的高通量液滴微生物培养方法[42-44],也可拓展至自动化设施平台。
然而,对于一些有特殊培养环境需求的微生物,如绝对厌氧、光照、嗜高压等微生物,目前还没有成熟的自动化设施平台可以将其所需的厌氧操作箱、光照反应器等进行自动化集成。因此,针对这些非模式微生物,需要进行定制化的设备开发,并且需要将它们与自动化平台的其他硬件设备进行端口串联。虽然目前在这方面仍存在一些挑战,但随着技术的不断发展,可以期待未来会有更多的解决方案出现,以满足特殊微生物的自动化需求。
2.2 自动化细胞底盘DNA转化
理想的微生物细胞工厂需要具有较好的稳定性和鲁棒性,自动化合成生物技术及自动化设施平台可以从多个方向加速或拓展细胞底盘的开发。工程DNA的转化是自动化生物过程最为烦琐的步骤之一,且十分耗时。目前,伊利诺伊大学iBioFAB[11]、爱丁堡大学EGF[33,45]、帝国理工学院London Biofoundry[46]等平台已经开发了基于常规液体工作站、微孔板或微流控的化学转化(热激法)或电转化,用于TALEN蛋白表达质粒制备、番茄红素代谢途径组装等。
酿酒酵母具有培养条件简单、生长繁殖快、GRAS(generally regarded as safe,通常认为是安全的)、遗传操作工具成熟等优势。但是由于酿酒酵母代谢网络十分复杂,单基因或多基因的编辑可能限制了目标产物的生产,传统的改造方法通量较低限制了细胞工厂的构建[36]。自动化的高通量基因编辑技术在基因组水平上改造细胞生理功能构建细胞工厂,可提高细胞工厂的生产性能。London Biofoundry基于自主开发的软件平台AMOS将实验设计软件JMP和Echo纳升移液工作站进行集成,开发了高通量的酿酒酵母转化方法(图3)[46]。首先,在大肠杆菌中实现了88个包含荧光蛋白编码基因的质粒构建、验证和提取的自动化操作。构建好的质粒通过多通道移液器转移至预先装有酿酒酵母S288c感受态的96孔深孔板中,采用聚乙二醇(polyethylene glycol,PEG)介导的化学转化法将质粒与感受态细胞进行混合,将工程DNA高通量的转化至细胞底盘中。然后,采用多通道移液器将3 μL稀释后的细胞涂布于Omnitray平板进行克隆筛选。类似地,基于微孔板的自动化底盘细胞转化也可应用在大肠杆菌[47]、谷氨酸棒状杆菌[48]、枯草芽孢杆菌[49]及丝状真菌[50]等细胞底盘。

图3 London Biofoundry开发的高通量酿酒酵母转化流程[46]Fig. 3 High-throughput transformation process for Saccharomyces cerevisiae[46]
此外,George Church团队[51]基于微流控芯片开发了可用于大肠杆菌自动化、规模化、微量化DNA电转化的平台EP/EWD(EP: Electroporation,电穿孔;EWD:Electrowetting-On-Dielectric,介电润湿电极),并对其电转化过程参数进行了优化(图4)。该装置的芯片底盘集成了22个700 μm长的介电润湿EWD电极,研究者采用6个EP/EWD芯片进行了44次独立的报告质粒转化实验。在平均电场强度2.25 kV/mm条件下,质粒的转化效率高达8.6 ×108CFU/μg,大肠杆菌的存活率为1.5%~2.3%;因为采用微流控操纵微流体(微升或纳升)的系统技术,单个转化体系的体积仅为200 nL,并且在进行平行转化实验的过程中未观察到交叉污染现象。同样地,高通量的微流控电转化方法也在微藻细胞工厂的构建中得到了应用[52]。

图4 基于微流控芯片的高通量大肠杆菌转化系统[51]Fig. 4 High-throughput transformation of Escherichia coli[51]
一些研究团队还专注针对一些具有挑战性的非模式微生物细胞底盘开发了自动化工艺流程。例如,Joosu Kuivanen等[53]开发了一套基于原生质体的高通量工程DNA转化和筛选工艺流程,用于黑曲霉全基因组编辑。Tecan公司则利用Freedom EVO 150移液工作站、8通道移液机械臂(LiHa)、纳升移液器和自动化成像识别系统(Pickolo)开发了一套针对丝状真菌克隆挑取的自动化工艺流程(https://www.tecan.com/hubfs/Tecan_Journal/201401/16_17_Customizing_automated_colony_picking_012014.pdf)。这些自动化设施平台的引入和应用,极大地简化了微生物细胞底盘DNA转化等关键步骤的操作,显著减少了时间又降低了劳动成本,使得细胞工厂的构建过程更加高效、精确和可重复。
3 自动化合成生物技术在细胞底盘构建中的应用
自动化技术在微生物细胞工厂研究中的应用为加速DBTL工程循环和标准化实验提供了解决方案。传统的手工实验操作限制了循环效率和设计通量,人为错误和偏差也导致实验操作的不一致性和主观性。自动化技术的应用改变了生物实验的范式,实现了实验操作的多通道和实验过程的标准化。细胞工厂的构建方法正朝着全自动化方向发展,推动以石化原料为基础的工业向可循环、可持续的生物基绿色制造转变。在此过程中,全自动的DBTL循环将是下一波创新,能够引导自动化的代谢途径组装和底盘细胞优化,并提高循环效率。本节将从生物合成基因簇挖掘、代谢通路工程改造和底盘细胞优化三方面介绍国内外研究者在微生物细胞工厂自动化快速构建领域取得的科研进展。
3.1 天然产物生物合成基因簇的快速挖掘与表征
武汉大学刘天罡课题组[54]基于Auto-HTP自动化高通量平台及“基因簇功能元件理性可控重组”策略,实现了丝状真菌未知萜类基因簇的高效挖掘(图5)。该研究以米曲霉为细胞底盘,利用近缘宿主解决同源基因异源表达的适配性和产量低的问题。通过模块化组合重构了39个丝状真菌来源的萜类生物合成基因簇,构建了173个质粒,并在细胞底盘内重构208株突变体,形成了包含185个萜类产物的化合物库,同时解决了研究通量、产物集中度低的问题,提高了新化合物发现的效率。最终,筛选到具备高抗炎活性的二倍半萜类产物mangicol J,并对生物合成机理进行了解析,通过对细胞底盘代谢工程改造,解决了目标产物产量低的问题。
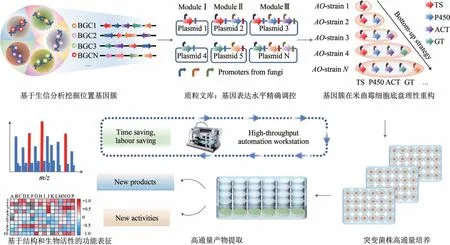
图5 自动化工作站(auto-HTP)工作流程[54]Fig. 5 Workflow of auto-HTP[54]
赵惠民课题组与Wilfred A. van der Donk课题组及Douglas A. Mitchell 课题组合作开发了一个基于自动化设施可快速、高通量、自动、可扩展的发现核糖体合成和翻译后修饰肽(ribosomally synthesized and post-translationally modified peptides,RiPPs)的平台FAST-RiPPs(图6)[55]。研究人员通过生信分析发现了96个疑似RiPPs合成基因簇序列,基于iBioFAB平台自动化的重构这些基因簇,对基因序列进行密码子优化、保留前导肽序列、融合His标签序列,在大肠杆菌细胞底盘中进行异源表达和肽结构表征,最终,RiPPs合成基因簇的构建成功率达到86%。对获得的基因簇进行异源表达筛选,发现27个基因簇成功实现了异源表达,合成了30种RiPPs,有7种表现出了对超级细菌的抑制作用,并且首次被发现对肺炎克雷伯菌有抑制作用。

图6 RiPPs结构式、合成机制及FAST-RiPPs流程[55]Fig. 6 Overview of RiPPs and FAST-RiPPs pipelines[55]
本课题组基于深圳设施平台对环二肽[56]、羊毛硫肽[37]、萜类[57]等天然产物的生物合成基因簇开展了一系列自动化、高通量的基因组挖掘和工程改造研究。其中,本课题组采用模块化DNA组装方法,规模化构建了古菌细胞膜极性脂的生物合成通路,在酵母底盘中“即插即用”式引入产甲烷古菌(Methanosarcina acetivorans)、硫化古菌(Sulfolobus islandicusas)、Asgard古菌等不同来源的关键合成基因,成功合成了具备古菌醚键特征的杂合极性脂DGGGO-FA,首次在真核细胞中实现了古菌来源极性细胞膜脂的异源合成(图7)。基于基因敲除、回补等方法,对古菌细胞膜极性脂类合成关键酶/通路功能表征,探究了DGGGOFAs的生物合成机制,发现酿酒酵母中二酰甘油酰基转移酶DGAT是关键合成酶,催化DGGGOH古菌醇和Acyl-CoA缩合产生DGGGO-FA。同时,证实了古菌中也广泛存在DGAT酶,并具备合成DGGGO-FA的能力,对目前古菌能量存储方式的认知提出了挑战[57]。

图7 重组酿酒酵母中古菌特征脂类合成途径(蓝色背景)及酵母内源脂类合成途径(黄色背景)[57]Fig. 7 Biosynthetic pathways of archaeal phospholipids, hybrid neutral lipids (DGGGO-FAs)and triacylglycerol (TAG) in engineered S. cerevisiae[57]
3.2 代谢通路组合优化
赵惠民团队利用iBioFAB自动化设施平台,集成贝叶斯机器学习算法搭建了一个完全由机器学习算法驱动的生物系统设计优化平台——BioAutomata,实现了“设计-构建-测试-学习”的全自动化运行,通过大肠杆菌细胞底盘完成了番茄红素代谢途径的组合优化构建、产品合成和筛选分析(图8)[10]。在完成初始的设计和设置后,机器学习算法驱动自动化设备执行实验操作,并将数据反馈给算法模型,由算法评估并确定下一轮实验的步骤。研究者基于该平台以大肠杆菌BL21(DE3)为细胞底盘,对包含3个基因表达框的番茄红素合成途径进行了组合优化。利用iBioFAB平台,完成了代谢途径的组装(Gibson和Golden Gate方法)、大肠杆菌的化学转化(热激法)、细胞培养和番茄红素提取等生物学过程。以各个基因的启动子强度为变量,以番茄红素的产量为目标函数生成实验设计。通过对所有13 824种可能性的算法评估,只需测试其中不到1%的组合,就可以得到最优的代谢途径组合方案,使番茄红素的产量提高了1.77倍。结合了机器学习算法的全自动设施平台,可以执行实验操作并即时分析数据,以迭代的方式优化指定的生物过程,降低实验成本,促使细胞工厂构建向更加自动化和智能化方向发展。与此类似,Carbonell等[10]依托Hamilton Microlab STAR平台对启动子强度、代谢途径基因顺序和基因拷贝数等因素进行组合优化,在大肠杆菌细胞底盘成功将(2S)-乔松素的产量提高了500倍。

图8 基于iBioFAB平台开发的机器学习算法驱动的生物系统设计优化平台BioAutomata在大肠杆菌细胞底盘进行番茄红素代谢途径组合优化构建、番茄红素合成和筛选[10]Fig. 8 The application of an integrated robotic system coupled with machine learning algorithms to fully automate the DBTL process for biosystems design[10]
美国劳伦斯-伯克利国家实验室的研究人员基于贝叶斯机器学习算法及高通量、标准化的DNA组装方法,开发了一套自动推荐工具(automated recommendation tool,ART)。使用基于采样的优化,ART对下一轮工程周期的构建菌株进行推荐,并提供其生产水平的概率预测(图9)[58-59]。研究人员选择了5个基因,每个基因受细胞内不同启动子或其他机制的调控,总共包含将近8000种潜在的生物合成途径。对其中250种组合进行自动化实验验证,用于训练算法模型,解析输入参数(氨基酸合成)与输出参数(基因表达)之间的关联,从而推测剩余7000多种组合中的每一种对应的色氨酸合成的情况。最终,色氨酸的产量相较当时报道最高值增加了106%左右,比用于训练模型的最优组合高17%左右。

图9 ART可基于较小训练数据集为下一轮实验提供预测及建议[59]Fig. 9 ART provides predictions and recommendations for the next experimental cycle[59]
3.3 细胞底盘优化
模式微生物方面,George Church团队[60]在大肠杆菌中开发的多重自动化基因组工程(multiplex automated genome engineering,MAGE)进行大规模的细胞编程和进化等。赵惠民团队开发了以酿酒酵母为细胞底盘的RNAi(RNA interference)辅助的自动化基因组进化方法RAGE(RNAi-Assisted Genome Evolution)(图10)[61]。基于引入异源RNA干扰途径的酵母底盘及iBioFAB自动化平台,利用标准化的全长互补DNA文库构建编码过表达和敲低突变的基因调控元件库,并在其中引入δ位点序列作为基因组整合的供体,通过CRISPR/Cas9引入DNA双链断裂(DNA doublestrand break, DSB)实现了高效、无标记的文库基因组整合。通过自动化移液工作站和96孔板,利用PEG介导的化学转化法将质粒文库转化至细胞进行表型筛选,转化效率达到104细胞/μg质粒;通过自动化平台可每次实施192个平行转化实验,得到106以上的克隆突变体。通过高通量测序(next generation sequencing,NGS)方法对得到的突变体进行测序分析,基因突变位点覆盖了酿酒酵母基因组全部编码基因的90%以上。经过6周共计3轮的工程菌株自动化构建和筛选,快速获得了当时已报道乙酸耐受性能最高的酵母菌株。

图10 酿酒酵母自动化多重基因组进化技术[61]Fig. 10 Scheme of automated RNAi-assisted genome evolution in S. cerevisiae[61]
在非模式底盘方面,中国科学院天津工业生物技术研究所王猛团队[48]依托高通量基因编辑与筛选自动化平台,在谷氨酸棒状杆菌中开发了多元自动化基因组编辑方法MACBETH(multiplex automatedCorynebacterium glutamicumbase editing method),实现了质粒构建、基因组编辑、克隆筛选和表型验证的全流程自动化操作,可实现每月数千突变株的编辑。其中,基于CRISPR/nCas9(D10A)-AID(activation-induced cytidine deaminase)方法对94个转录因子进行自动化、高通量的基因编辑,86%的目的基因编辑效率达50%以上。此外,该团队还开发了基于液滴微流控的链霉菌高通量培养和筛选技术,检测分选速度达到每小时1万菌株,人工混库的单轮分选富集率超过了330倍(图11)[43]。通过优化启动子强度、分泌蛋白表达等方面的优化和测试,成功从随机突变文库中筛选获得纤维素酶产量提高211.4%的菌株。同时,由于链霉菌孢子的稳定性,该平台也可适用于生长中期胞内、胞外目标蛋白的筛选,在次级代谢产物如抗生素等筛选方面展示出较好的应用前景。

图11 基于液滴微流的链霉菌高通量筛选技术[43]Fig. 11 Flow chart of high throughput screening of Streptomyces using fluorescence-activated droplet sorting[43]
4 展 望
自动化合成生物技术基于其自动化、标准化、高通量的特性以及信息化、智能化的发展,有望突破依赖手工实验的传统生物学研究范式,在多个方面为微生物细胞工厂的快速设计和构建提供重要的技术和平台支撑。在微生物细胞工厂快速构建方面,自动化合成生物技术已在模式菌株领域有了较为广泛的应用,并取得了一些令人瞩目的研究成果。但是,如何将这些使能技术及经验扩展至其他重要的非模式微生物,实现从天然驯化到理性设计的转变,克服非模式微生物复杂代谢网络和有限理化知识造成的阻碍是人工微生物细胞工厂构建面临的主要问题之一。目前暂未有针对非模式微生物的全自动设施平台,如何面对非模式微生物多样性的生理特性,如厌氧、光照、高温或者高压等,实现非模式微生物高通量、自动化操作也是自动化设施平台面临的主要问题之一[38,40]。另一方面,即使是对常用的细胞底盘如大肠杆菌、酿酒酵母而言,目前大多数自动化设施平台仍未能实现微生物细胞工厂构建的全自动化。如前所述,微生物细胞工厂的构建是指构建工程DNA并将其导入底盘细胞,其中工程DNA的构建主要包括元件准备、多元件组装和基因组工程。自动化DNA组装和底盘细胞操作是共性的合成生物自动化工艺,涉及基因合成、PCR扩增、酶切、DNA组装、核酸提取纯化、测序、工程DNA转化、菌落涂布、菌落挑取、细胞裂解和荧光分选等流程。我们对主要自动化设施平台在DNA组装和底盘细胞转化方面的能力和进展进行了总结和比较[12]。除了非模式微生物给自动化构建提出的广泛性挑战外,对构建的工程DNA和底盘细胞的验证性测试也为全流程自动化提出了新工艺开发、新设备与软件研发的需求。
自动化设施平台是一种综合性平台,集成了移液工作站、高通量检测与分析设备、处理分析软件、人员/数据管理和运行系统。目前合成生物自动化设备、机器人集成等核心技术由Thermo、Beckman、Tecan、HiRes、Hamilton、Roche等国外公司掌控。iBioFAB、EGF两个设施平台均由Thermo提供集成方案,实现了全自动整合。然而,建立不同类型的自动化设施平台所需的设备成本,尤其是构建和测试环节,可能至少需要数千万元,对任何研究机构或者团队都可能是最大的一个挑战。往往搭建一个完全自动化的平台都是从小规模开始,以缓慢的速度向自动化和集成化发展。例如,美国能源部Agile Biofoundry、澳大利亚AusFAB、Australian Genome Biofoundry、天津大学Biofoundry等则主要由可以进行高通量操作的单台仪器组成。英、美等国现有的自动化设施中大部分已建成的平台更专注于某种细胞底盘或者DBTL循环中的特定任务,仍然存在一定的局限性,包括复杂线路设计能力不强、底盘细胞单一、大片段DNA的制造成本高、高通量测试手段少、与下游应用衔接不紧密等问题。此外,采用Thermo等国外厂商的方案及设备搭建的全自动化平台存在一些局限。其中之一是,生物学实验的需求往往是个性化的,平台的灵活性往往难以适应不同实验需求,且平台维护成本高、难以升级改造。另一个挑战是不同自动化系统和软件平台的集成和互操作性不高。
国产化自主研发可在一定程度上解决上述问题,同时避免核心设备仅依靠进口,途径单一。但是,任何设备的开发或者更新,都需要进行大量试错,以确保所有组件都经过验证并在自动化平台中稳健运行。在实现国产化路径方面,“引进-消化-吸收-创新”的模式可极大地缩短国产自主研发的周期,为国内研发和制造提供基础;加强合成生物学及自动化领域的人才培养和团队建设,提高人才储备;高校和科研院所可通过引入政府或企业资金,促进科研机构在自动化合成生物技术领域的创新研发和产业化推进;加强合作与开放创新,各团队可基于各自特点建设半自动化的设施模块,可降低投入成本;不同自动化实验室需要实现更高的标准化(硬件、计量、流程语义、数据等标准内容)、数据来源追踪、可溯性(步骤、操作方法、材料、设备等信息,数据采集记录,样本、实验材料信息,数据处理和分析方法,软件、算法版本等)和再现性,从而可与具有不同专业知识和设备的实验室进行合作及交互,实现生物学实验的分布式操作。自动化设施平台的国产化替代,也面临一些难点:需要生物学、自动化、人工智能等不同领域专家合作克服技术壁垒;赢得与欧美等自动化厂商的市场竞争;短期内仍处于国产设备和试剂耗材性能不佳的窘境;建立可持续的生态系统,将产学研进行深度联合等。
自动化合成生物技术改变了生物学的研究范式,然而在建设及使用中也需要考虑技术变革带来的一些科学问题,如:自动化设施平台如何成为工业4.0的重要参与者而非辅助技术?自主研发或低成本商用设备与更成熟、整合度更高的设备相比如何选择?选择时需要权衡哪些因素(时间、效率、资源可用性等)?自动化设施平台是否会引起任何伦理问题?自动化设施平台是否可以引起代谢工程领域的一场革命?国内的自动化设施平台仍处于起步阶段,如何搭建、迭代,适用于哪些科学问题,建设及使用过程中会引起哪些问题,都值得探讨和跟进。
