开花期番茄circRNA的测序分析及功能分析
2023-11-18殷子贺杨成成赵钰慧赵丽吕秀荣杨振超武永军
殷子贺,杨成成,赵钰慧,赵丽,吕秀荣,杨振超,武永军
开花期番茄circRNA的测序分析及功能分析
殷子贺,杨成成,赵钰慧,赵丽,吕秀荣,杨振超,武永军
西北农林科技大学生命科学学院/园艺学院,陕西杨凌 712100
【背景】开花期作为植物生长发育过程中至关重要的时期之一,直接影响植物果实成熟和种子发育。circRNA是一类普遍存在于真核细胞的共价闭环RNA分子,在番茄的发育过程和应激反应中起重要调节作用。但目前番茄的circRNA研究主要集中在果实和叶片中,缺少对番茄花期circRNA较为系统的研究。【目的】鉴定和分析开花期番茄circRNA,对番茄中miRNA、circRNA的功能研究有重要意义,也为番茄生长、发育和胁迫响应机制研究奠定基础。【方法】选择开花期番茄植株的花、根、叶3个组织样品进行circRNA测序,每个样品3个重复,鉴定circRNA并对其基本特性进行分析;筛选组织特异性的circRNA检测成环能力,并对鉴定的circRNA宿主基因进行GO分析和KEGG分析,通过生物信息学方法预测分析circRNA的作用方式和作用位点,构建番茄响应生长发育的潜在circRNA-miRNA-mRNA互作调控网络。【结果】利用高通量测序方法,共获得532个circRNA,其中83%为外显子类型,且circRNA在花期番茄各染色体中的分布不均匀,其中1号染色体产生的circRNA最多,5号染色体最少。对开花期番茄花、叶、根3个组织的circRNA差异表达分析表明,花与叶中差异表达的circRNA有79个、花与根中差异表达的circRNA 133个、叶与根中差异表达的circRNA 132个。其中花、叶、根3个组织中均有显著差异表达的circRNA 14个。从14个差异表达circRNA中随机选择8个circRNA进行成环能力检测,结果表明这8个circRNA均具有成环能力。GO分析和KEGG分析表明开花期番茄中的circRNA主要与核酸、蛋白及其他小分子物质结合以及各类生物大分子的合成和代谢相关。最后构建了14个circRNA、10个miRNA和136个mRNA组成的番茄circRNA-miRNA-mRNA互作调控网络。【结论】开花期番茄circRNA具有明显的组织特异性,共鉴定到342个组织特异性的circRNA,其中均显著特异性表达的14个,成功鉴定成环8个,构建了开花期番茄特异性的circRNA-miRNA-mRNA互作调控网络,为后续开花期circRNA的研究奠定了基础。
circRNA;番茄;开花期;miRNA;ceRNA
0 引言
【研究意义】番茄()属于茄科(),由南美洲驯化而来,在世界各地广泛种植,是人类饮食中重要的营养来源,也是研究果实的典型模式植物。开花期作为番茄生长发育最关键的阶段之一,开花的时期、数目、质量直接决定着番茄的产量和经济效益[1]。环状RNA(circular RNA,circRNA)作为非编码RNA的一种,广泛参与植物的生长发育和非生物胁迫响应,在转录和转录后水平上对基因表达起着重要的调控作用。因此,研究开花期番茄的circRNA,对探索开花期番茄的分子机制有重要的理论意义。【前人研究进展】circRNA是一种共价闭合的环状RNA分子,相比普通RNA,circRNA没有5′末端和3′ ploy(A)尾巴,它以共价键连接首尾,从而形成封闭的环状结构,这一结构使它不易被核酸外切酶R (RNase R)降解[2]。一般来说,根据circRNA与其宿主基因的位置关系,可将其分为外显子circRNA、内含子circRNA和基因间circRNA三种类型[3]。前人研究表明,特定的植物circRNA在一定条件下,可以通过竞争性内源RNA(competing endogenous RNA,ceRNA)方式调节基因表达并对植物的生长发育(尤其是花发育)起至关重要的作用[4-6]。近些年来,一些关于番茄circRNA的报道也逐渐被关注。Zuo等[7]在番茄果实中预测到一些circRNA参与了冷胁迫响应过程。Tan等[8]在番茄果实中鉴定了一些来源于果实色素生物合成基因的circRNA,并表明其中有些circRNA的丰度受到果实成熟的调控。Wang等[9]证明了circRNA在番茄果实乙烯途径中的复杂调控作用,并表明番茄果实中乙烯途径可能受各种circRNA调控。此外,非编码RNA,如miRNA和长链非编码RNA(lncRNA)可能通过调控花组织内膜的形成,与多种分子结合,影响代谢途径,诱导细胞凋亡,从而在开花番茄植株的生长发育和细胞凋亡过程中发挥重要作用[10]。Zuo等[11]研究了番茄果实成熟过程中可能与潜在靶向的乙烯或类胡萝卜素途径相关的转录因子和关键酶通过差异表达的circRNA。Yang等[12]揭示了番茄叶片中多个被分离出来的circRNA在低温胁迫下受到不同的调控,并绘制了低温胁迫下的circRNA-miRNA-mRNA潜在网络。【本研究切入点】目前,有关番茄circRNA的研究大多是在果实和叶片中进行,研究方向也主要是胁迫响应和果实发育方面居多,对开花期番茄circRNA的研究较少。【拟解决的关键问题】本研究从开花期番茄的ssRNA-seq(链特异性RNA测序)文库中鉴定和分析circRNA,获得高质量的circRNA序列信息,又从中选取部分circRNA进行成环验证;并对其中少数靶标miRNA进行推测,建立开花期番茄的circRNA-miRNA-mRNA互作网络。
1 材料与方法
1.1 植物材料与生长条件
本研究使用的番茄品种为Ailsa Craig。番茄植株的生长环境为光照/黑暗16 h/8 h,光照强度250 μmol∙m-2∙s-1,昼/夜温度26 ℃/18 ℃,湿度80%。番茄第一次开花时,收集植株顶端的第3片叶、去除萼片的花和根作为试验材料(每个样品包含3个生物学重复)。取样后,立即用液氮速冻,并置于-80 ℃储存。
1.2 circRNA文库的构建及测序
使用TRIzol(Invitrogen, Carlsbad, CA, USA)提取花期番茄植物各组织(叶、花和根)的总RNA,使用Ribo-Zero Gold kit(Epicentre, San Diego, CA, USA)分别处理开花期番茄植株的花、叶和根3个组织总RNA样品。总RNA样品经DNase I(NEB,MA,USA)处理后,使用NEBNext®Ultra™ Directional RNA(NEB,MA,USA)进行rRNA消除。对得到的RNA进行RNase R(Epicentre, San Diego, CA, USA)处理去除线性RNA分子,参照Yang等[13]的方法构建circRNA-seq文库,并按照供应商推荐的方案在Illumina Hiseq 4000(LC Bio,China)上进行了配对末端测序。
1.3 circRNA鉴定
使用StringTie对基因组比对后的Reads进行转录本重构。使用cuffcompare对重构的转录本进行相互比较,并删除冗余的转录本。基于Prensner等[14]的方法,对重构的转录物进行过滤和筛选。并将重构后的转录本分别使用软件CIRI和find_circ进行circRNA检测,将CIRI和find_circ鉴定的所有circRNA进行交集分析,能够在CIRI和find_circ两个软件中同时预测到的circRNA作为circRNA鉴定的最终结果。
1.4 circRNA 表达水平及差异表达分析方法
circRNA表达量采用RPM(back-spliced reads per million mapped reads)进行标准化计算。参照Yang等[13]的方法,circRNA的差异表达分析采用差异倍数(Fold Change)和value进行差异circRNA 筛选,筛选条件为:log2Fold Change的绝对值≥1且value≤0.05。
1.5 circRNA的验证和qRT-PCR鉴定
从所有已鉴定的circRNA中,基于碱基位点数目和高度差异表达选择8个circRNA用于试验验证。使用十六烷基三甲基溴化铵(CTAB)方法提取基因组DNA作为circRNA验证过程中不同引物的阴性对照[15]。将用于RNA-seq的总RNA样品用DNase I去除DNA污染后,根据Epicenter Ribo-Zero Gold Kit程序去除rRNA,并且通过在37 ℃下与3 μg RNase R孵育15 min去除线性RNA。使用Primer 5设计每种circRNA的两套引物:发散引物,预期仅可扩增跨反向剪接连接处的circRNA,以及用于扩增线性mRNA形式的收敛引物(引物信息见表1)。
通过反转录试剂盒(Promega, Madison, WI,将2 μg总RNA逆转录为cDNA。使用随机引物进行cDNA合成。以反转录后的cDNA作为模板进行实时定量PCR检测。使用SYBR Green PCR Master Mix(Promega, Madison, WI,)和Bio-Rad CFX manager 3.1(Bio-Rad, Hercules, CA USA)程序进行qRT-PCR。内参基因使用,通过2-ΔΔCT方法进行定量分析数据。所有结果使用3个生物学重复的SD值(平均值±标准差)表示。RT-qPCR引物使用发散引物R和F,引物信息见表1。
1.6 共表达网络的建立
基于针对miRBase 21.0版的比对,采用Targetscans(v7.0)和miRanda分析预测开花期番茄circRNA的miRNA结合位点,该方法利用了34种植物物种中的miRNA(http://www.mirbase.org/)。利用生物信息学方法预测circRNA与miRNA的互作可能性及对应的互作位点,使用PlantCircNet(http://bis.zju. edu.cn/plantcircnet/)可视化circRNA-miRNA-mRNA相互作用网络。所有检测到的circRNA和miRNA均用于相互作用分析。
2 结果
2.1 番茄circRNA的鉴定
从番茄开花植株的叶片、花和根中共获得731 707 984个原始读数,在去除衔接子和引物序列以及短的低质量序列后,其中99.98%是清洁读数。结果表明,RNA-seq读数和番茄参考基因组均具有较高的质量。发现平均82.76%的读取可以映射到唯一的基因组位置(表2)。
在比较基因组读数的基础上,利用StringTie对转录本进行重组和组装。经线性RNA去除后,使用CIRI和find_circ两款circRNA预测软件分别鉴定得到 1 137和2 144个circRNA,通过对两款软件鉴定到的circRNA进行交集汇总,总共得到2 749个circRNA,选取两款软件共同鉴定到的532个circRNA(附表)用于后续分析(图1-A)。这些circRNA有较强的组织特异性,在所有的532个circRNA中只有74个(14%)circRNA可以在番茄花、叶、根3个组织中同时鉴定得到,其中根组织特异性circRNA在番茄花期中鉴定的最多,总共为219个,占所有番茄circRNA总数的41.2%(图1-B)。
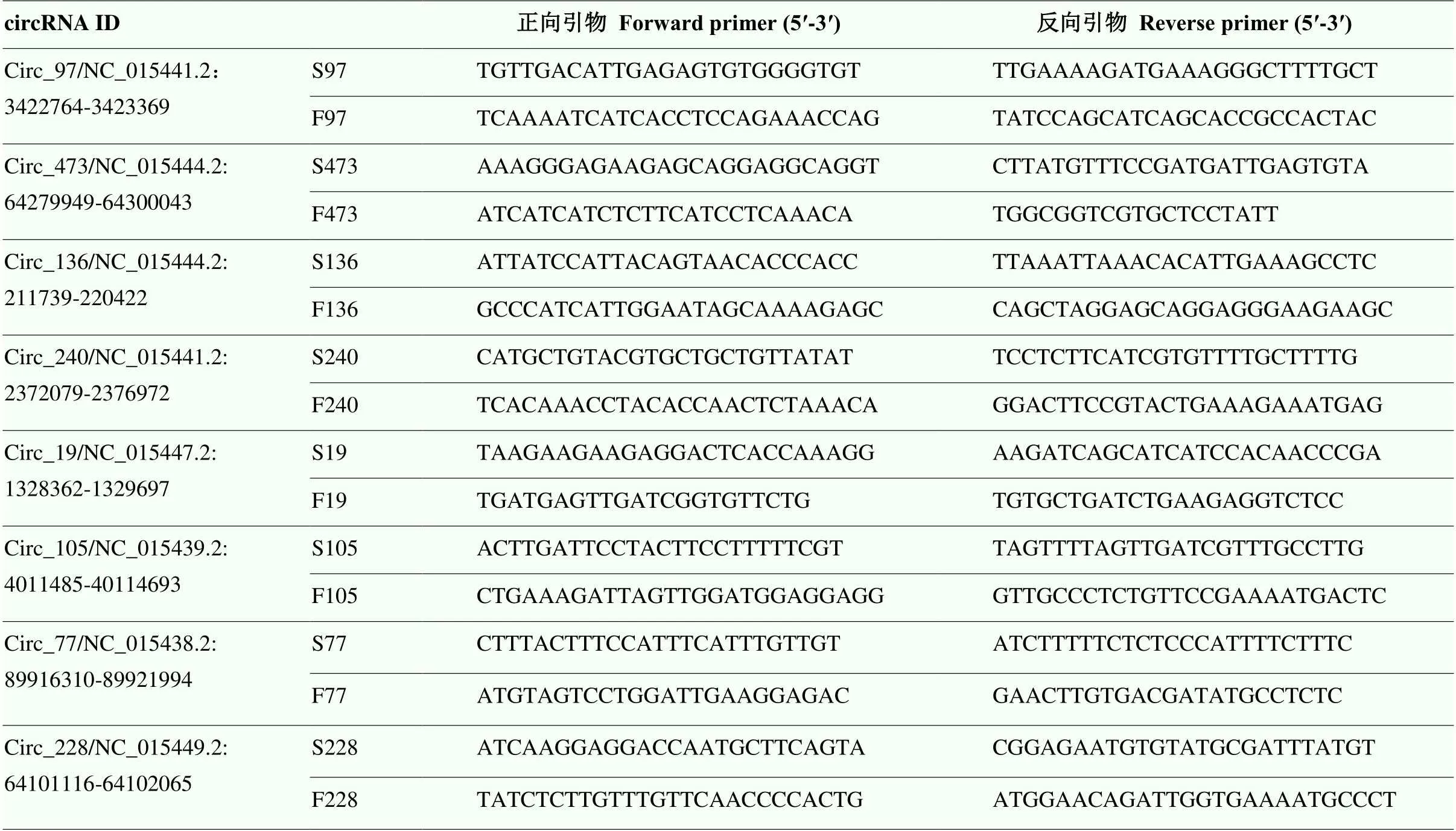
表1 引物序列
F:发散引物;S:收敛引物 F: Divergent primer; S: Convergent primer
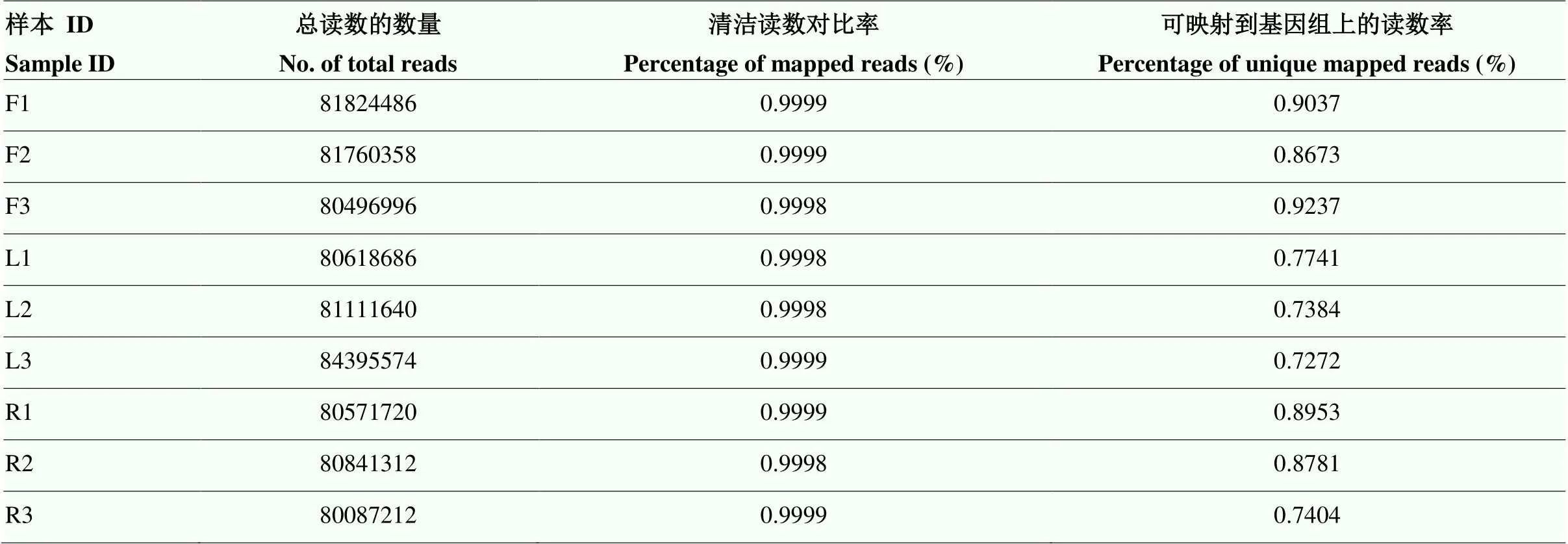
表2 circRNA测序数据结果统计
F:花;L:叶;R:根。下同 F: Flower; L: Leaf; R: Root. The same as below
2.2 番茄circRNA的分类及特征
对上述鉴定所得的532个circRNA类型分析显示,开花期番茄circRNA中,占比最高的为外显子circRNA(83%),其次为基因间circRNA(16%),内含子circRNA在花期番茄circRNA中鉴定最少,仅占1%(图2-A)。circRNA在花期番茄各染色体中的分布不均匀(图2-B),但相较于其他染色体,1号染色体对circRNA的产生明显具有更大的贡献,而5号染色体形成circRNA的能力明显要低于其他染色体,且本研究发现来源于线粒体的circRNA中基因间circRNA占有较大比例。
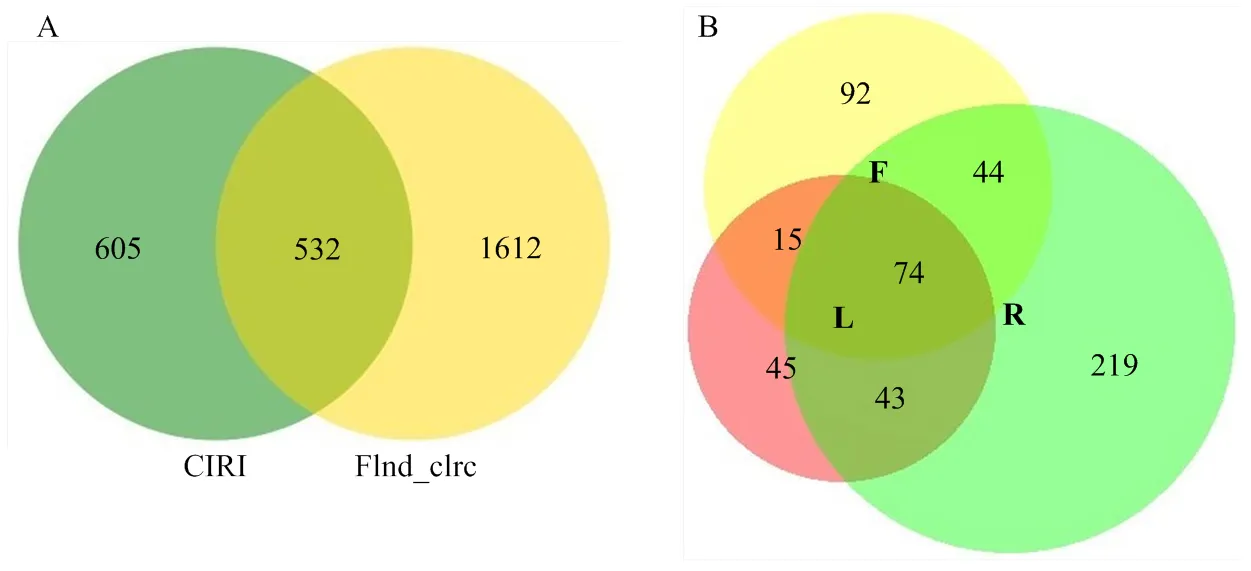
A:利用CIRI和find_circ鉴定的番茄circRNA;B:番茄各组织总circRNA维恩图分析
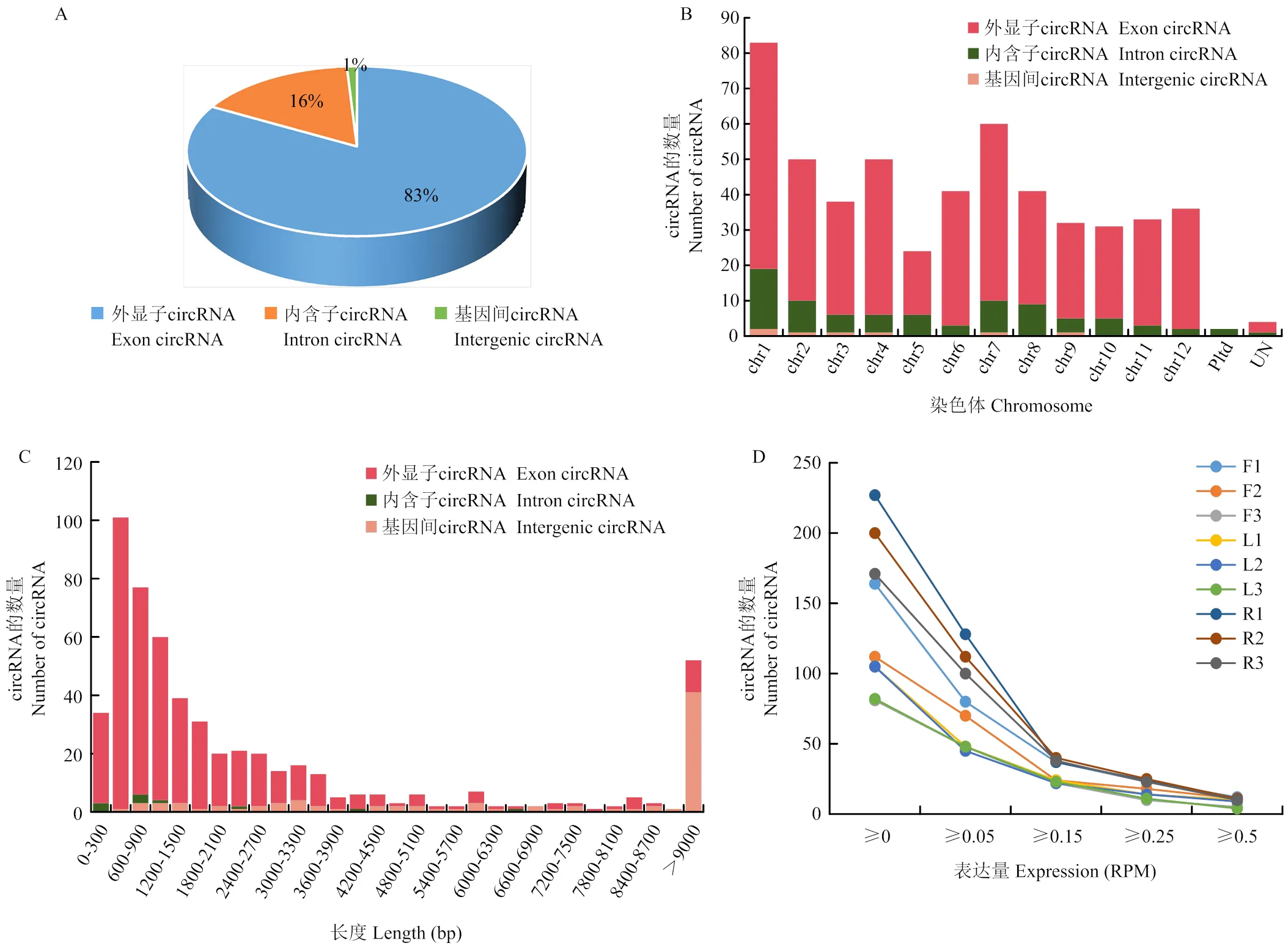
A:鉴定的番茄 circRNA分类图;B:番茄各染色体circRNA数量及类型比例分析;C:番茄中不同长度circRNA的分布及类型;D:番茄花、叶、根中不同表达量(RPM)的circRNA数量
此外,随着circRNA长度的增加,circRNA的数目不断减少(图2-C)。值得关注的是,超过80.6%的番茄circRNA长度较短(≤3 600 bp),外显子circRNA一般是较短的circRNA(<3 600 bp)。而在较长的circRNA(>3 600 bp)中,基因间circRNA占有更大的比例,尤其是在9 000 bp以上的circRNA中,基因间circRNA所占比例超过了77.5%,约为基因间circRNA整体占比的5倍。根据circRNA RPM表达量值,分别统计了RPM表达量≥0.0、0.05、0.15、0.25、0.5的circRNA数目(图2-D)。结果显示,随着circRNA表达量的升高,circRNA的数目逐渐减少。但是,虽然花组织和叶组织circRNA的数目随表达水平的变化趋势类似,但是相比于花组织和叶组织,根组织中RPM<0.15时,circRNA的数目明显更多;在RPM>0.15后,根组织的circRNA数目才与叶组织和花组织中的数目类似,呈现出基本稳定的减少趋势(图2-D)。
2.3 番茄circRNA差异表达分析
由于表达值分析中开花期番茄花、叶、根3个组织中circRNA显示出一定的差异性,因此,本研究对上述开花期番茄花、叶、根,3个组织的circRNA进一步进行差异表达分析。聚类图(图3-A)显示了532个番茄circRNA在花、叶、根3个组织中的总体表达水平分布。根据聚类结果,将番茄3个组织的circRNA进行差异表达统计,总共得到花与叶中差异表达的circRNA 79个,花与根中差异表达的circRNA 133个,叶与根中差异表达的circRNA 132个。分别对这些circRNA进行表达水平统计,结果显示,与根组织相比,叶与花中的circRNA主要呈现出上调趋势(图3-B)。番茄circRNA在花、叶、根3个组织中的具体表达如火山图所示(图3-C—E)。
对上述鉴定的circRNA进一步进行交集分析(图3-F),共得到花期番茄花、叶、根3个组织均有显著差异表达的circRNA 14个,对这14个显著差异表达的circRNA分层聚类的结果(图3-G)显示,这14种新型circRNA在花、叶、根3个组织中表达的重复性较高,同时聚类树图将这14种circRNA分为3大类型。其中,circ_301、circ_714具有类似的表达模式,均在叶中的表达量较小,而在花与根中表达水平较高;circ_113、circ_239、circ_250、circ_116、circ_7、circ_433在叶中表达水平更高;circ_228、circ_504在根中有高表达;circ_37、circ_325、circ_609、circ_1118在花中表达量较高。研究结果表明,circRNA在花期番茄的叶、花和根中有组织特异性表达。
2.4 番茄circRNA的成环能力及差异表达验证
为了确认对番茄circRNA的鉴定,基于BS位点数目和高度差异表达筛选了8个circRNA用于试验验证。扩增结果显示,所有候选circRNA均可被收敛引物在cDNA样品和基因组DNA样品中扩增,而利用发散引物时只可以在cDNA样品中顺利扩增,不能在基因组DNA样品中扩增得到(图4-A、B),证明8个候选circRNA可以在番茄体内成环。
进一步对这8个候选circRNA进行qRT-PCR验证,结果显示circRNA在番茄不同组织中表现不同的调控模式(图4-C)。circRNA19、circRNA240在番茄花组织中具有高水平表达,而circRNA105、circRNA473在番茄叶组织中具有极显著的高水平表达,但在番茄根组织中表达量较低;与circRNA105和circRNA473正好相反,circRNA 136在番茄根组织中具有高水平表达,但在番茄叶组织中表达量较低。此外,circRNA97与circRNA228在花、根、叶组织中并没有显著的表达差异。
2.5 GO/KEGG通路分析差异表达的circRNA-宿主基因
由于circRNA与其亲本蛋白编码基因具有相似或相反的表达模式,为了探索番茄差异表达circRNA的推定功能,对circRNA-宿主基因进行GO富集和KEGG途径分析。GO富集将532个circRNA(342个显著性差异的基因数目)的宿主基因分为89个功能项,包括3个主要的GO分类类别(“生物过程”“细胞组分”和“分子功能”),分别包含28、23和38个功能项。KEGG途径分析将circRNA的宿主基因分为78个功能项。在细胞组分类别中,circRNA主要集中在细胞质(GO:0005737)、胞质部分(GO:0044444)和质膜(GO:0005886)中。表明开花期番茄的circRNA很可能通过ceRNA方式与胞质中的miRNA结合,从而调控基因表达;也可能是为后续果实发育积累一些物质和能量条件。对于分子功能,富集的GO类别主要包括核苷酸结合(GO:0000166)、转移酶活性(GO:0016740)、离子结合(GO:0043167)、碳水化及衍生物结合(GO:0097367)和激酶活性(GO:0016301),推测circRNA可能通过与核酸、蛋白以及其他小分子物质的结合,来调节植物开花期的分子功能。对于生物过程,大多数circRNA-宿主基因主要参与生物调节(GO:0065007)、细胞代谢过程调节(GO:0031323)、基因表达调控(GO:0010468)、细胞分解代谢过程(GO:0044248)和RNA代谢过程调节(GO:0051252)。推测开花期番茄circRNA主要通过在胞质中结合核酸、蛋白以及一些小分子物质,来参与番茄开花过程中生物调控、细胞代谢调控以及基因表达调控等过程(图5-A、表3)。

A:番茄circRNA在花、叶、根3个组织中的总体表达水平分布聚类图;B:番茄花、叶、根3个组织的circRNA进行差异表达统计和上/下调水平统计;C、D和E分别显示番茄叶片、花和根中DE circRNA基因(Degs)的火山图,C是F-vs-L的DEGS_Volcano,D是L-vs-R的DEGS_Volcano,E是F-vs-R的DEGS_Volcano,红色、绿色和灰色分别表示上调、下调和非DEG;F:番茄花、叶、根3个组织的差异表达的circRNA交集分析;G:对番茄花、叶、根3个组织均显著差异表达的14个circRNA分层聚类的结果
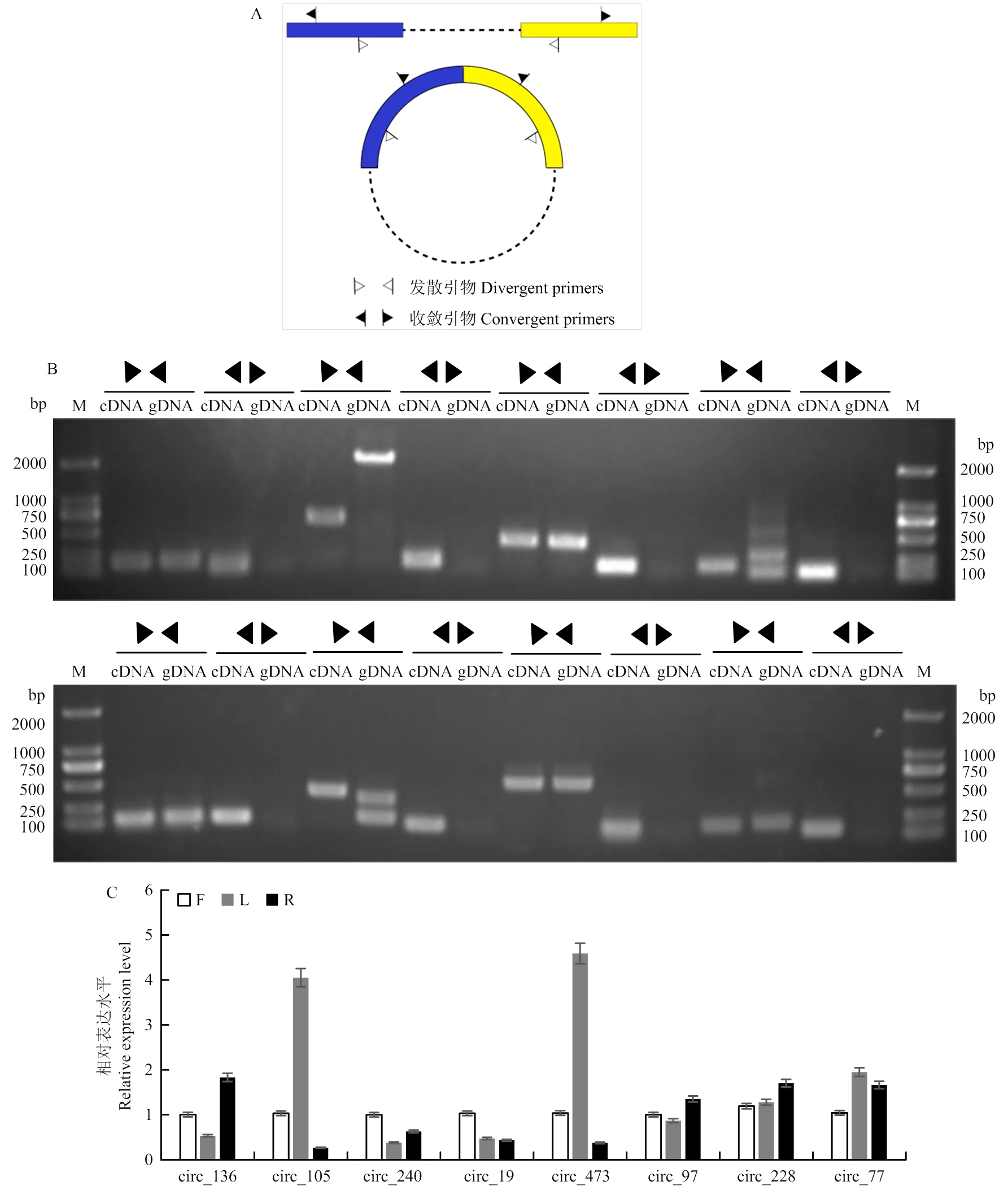
A:circRNA成环验证原理;B:番茄circRNA的成环能力检测;C:番茄差异表达circRNA的验证
为了更进一步了解基因的生物学功能,将532个(232个显著性差异的基因数目)差异表达的circRNA宿主基因分配到78个通路。KEGG途径分析显示,circRNA的亲本基因主要与某些生物大分子的合成、代谢有关,例如丙氨酸、天冬氨酸和谷氨酸的代谢,磷酸戊糖途径,花生四烯酸代谢,脂肪酸的降解,精氨酸的生物合成等。且circRNA-宿主基因在丙氨酸、天冬氨酸和谷氨酸的代谢通路显著富集,戊糖磷酸途径富集程度较高,丙氨酸,天冬氨酸和谷氨酸的代谢具有显著性差异的基因数目最多,戊糖磷酸途径和脂肪酸降解具有显著性差异基因较多(图5-B、表4)。表明开花期番茄circRNA的许多亲本基因参与氨基酸、糖、脂肪酸等生物大分子的合成和代谢途径,推测这些可能是为后期番茄果实发育和成熟积累的养分和能量。也进一步证明,circRNA确实在开花期番茄中对番茄的生长发育起到积极的调控作用。

表3 差异表达circRNA的宿主基因GO注释
仅列出基因数量最多的3种GO类别 Only the three GO term with the largest number of genes are listed
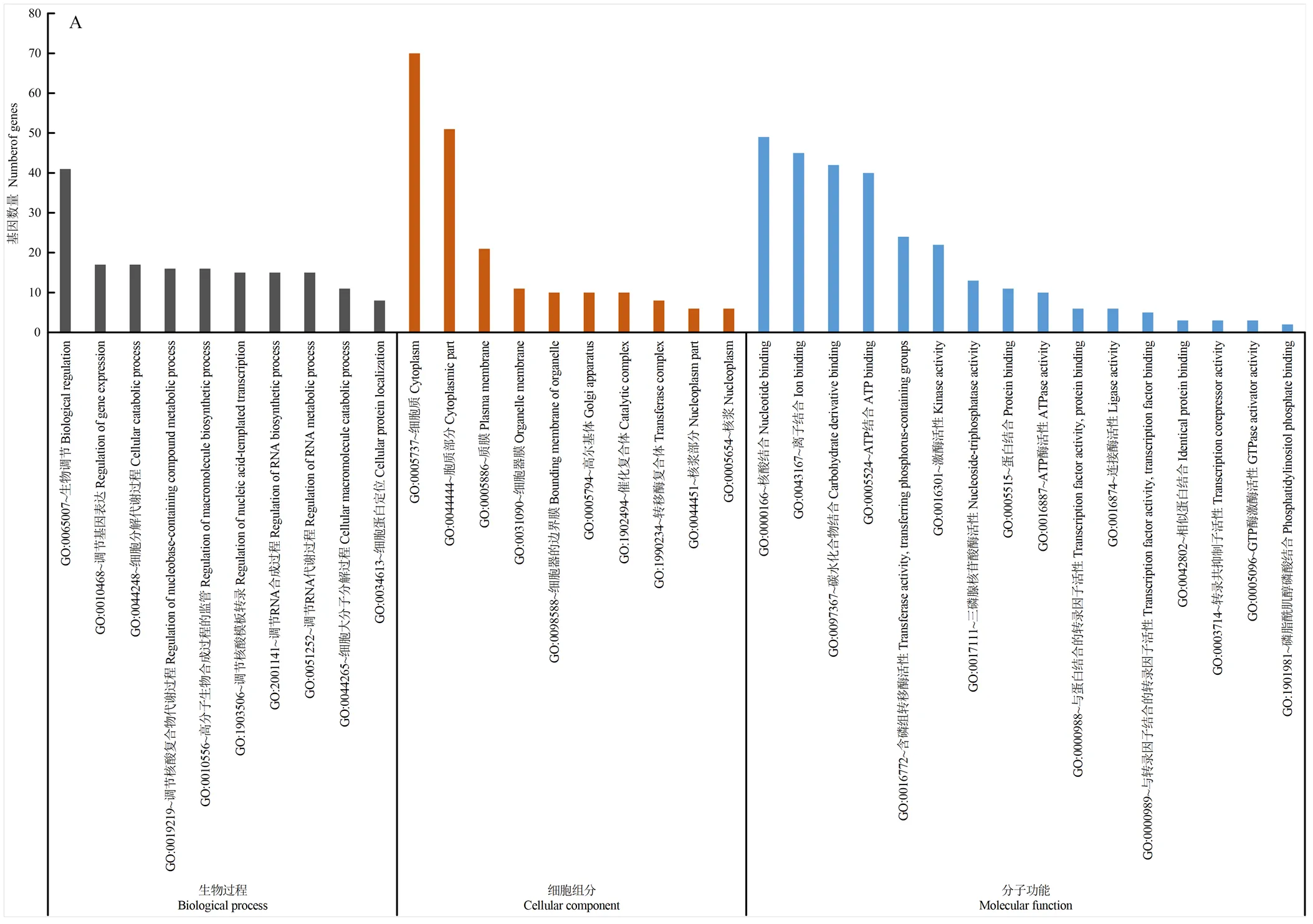
A:番茄中circRNA顺式靶基因的基因本体富集分析(GO),垂直轴代表基因数目,水平轴代表富集项目列出了Biological process(BP)前10条,Cellular component(CC)前10条,Molecular function(MF)前16条;B:番茄中circRNA顺式靶基因的KEGG途径富集(前24条),垂直轴上的项目/路径按照路径的分类绘制(不同的颜色代表不同的类别),水平轴代表富集系数
A: The vertical axis represents the number of genes in tomato, the horizontal axis represents the enrichment items listing the top 10 biological process (BP), cellular component (CC) Top 10 bars, Top 16 of molecular function (MF); B: KEGG pathway enrichment of circRNA cis-target genes in tomato (top 24), items / paths on the vertical axis are drawn by the classification of paths (different colors represent different categories), the horizontal axis represents the enrichment coefficient
图5 GO/KEGG通路分析差异表达的circRNA-hosting gene
Fig. 5 GO/KEGG pathway analysis Gene ontology enrichment analysis (GO) of circRNA cis-target genes in circRNA-hosting gene
2.6 开花期番茄circRNA-miRNA-mRNA共表达网络
为了揭示circRNA是否可以靶向miRNA并进一步影响基因的转录后调控,研究circRNA、miRNA和mRNA之间的潜在相互作用。首先,采用Targetscans(v7.0)和miRanda软件对开花期番茄的circRNA和miRNA结合位点进行预测,发现14个circRNA是两个软件共同预测到的circRNA(表5)。结果显示有14个circRNA、10个miRNA和136个mRNA可能存在潜在的相互作用。其中多个circRNA(或miRNA)被预测与一个以上的miRNA(或circRNA)相互作用。其中circ_1403可以分别与sly-miR167b-3p、sly-miR9479-3p结合;而sly-miR5303可与circ_1420和circ_547进行结合;sly-miR396b也可与circ_1112和circ_473进行结合。sly-miR482d-5p也有circ_703和circ_733两个circRNA富集。其中circRNA结合最多的miRNA是sly-miR9478-3p,它可以与circ_10、circ_176和circ_1079这3个circRNA结合。此外,还发现circ_473可直接作用于靶基因Solyc01g110700.2。随后对这14个circRNA亲本基因的功能注释进行研究,结果表明,在生物过程中,主要与蛋白结合(GO:0005515)、ATP结合(GO:0005524)、辅酶结合(GO:0006306)以及在含磷酸酐中作用于酸酐的水解酶活性(GO:0016818)有关;在分子功能中,主要与谷氨酸代谢(GO:0006536)、胞外多糖的生物合成(GO:0045226)、DNA模板化转录调控(GO:0006355)有关;在细胞组分中,主要存在于细胞核(GO:0005634)和线粒体基质(GO:0005759)中(表6)。
表4 差异表达circRNA的宿主基因KEGG注释 Table 4 KEGG annotations of the host genes with differentially expressed circRNA KEGG通路名称Name of the KEGG pathwayKEGG编号KEGG number宿主基因数量No. of host genes 丙氨酸,天冬氨酸和谷氨酸的代谢Alanine, aspartate and glutamate metabolismsly0025030 精氨酸的生物合成Arginine biosynthesissly0022012 磷酸戊糖途径Pentose phosphate pathwaysly0003010 脂肪酸的降解Fatty acid degradationsly00071 6 脂肪酸的生物合成Fatty acid biosynthesissly00061 5 脂肪酸代谢Fatty acid metabolismsly01212 3 花生四烯酸代谢Arachidonic acid metabolismsly00590 2 仅列出基因数量最多的7种KEGG通路Only the seven KEGG pathways with the largest number of genes are listed
表5 开花期番茄circRNA的miRNA靶基因及结合位点 Table 5 miRNA target genes and binding sites of tomato circRNAs during flowering Trans IDmiR ID结合位点Binding sites靶向基因数量No. of target genes circ_1420/NC_015447.2: 2008132-2031198sly-miR530338 circ_547/NC_015449.2: 53700671-5371156238 circ_459/NC_015444.2: 21380917-21381396sly-miR9476-5p5 circ_1112/NC_015444.2: 64279962-64300043sly-miR396b30 circ_473/NC_015444.2: 64279949-64300043 circ_10/NC_015444.2: 208457-238616sly-miR9478-3p 4 circ_1079/NC_015444.2: 230400-238546 circ_176/NC_015444.2: 210427-211962 4 circ_437/NC_015438.2: 97435162-97451603sly-miR156e-3p 4 circ_704/NC_015438.2: 95819397-95828905sly-miR482d-5p 1 circ_733/NC_015438.2: 95819397-95825691 circ_739/NC_015438.2: 90765535-90770648sly-miR1916 6 circ_309/NC_015445.2: 2493994-2498871sly-miR1918 3 circ_1403/NC_015439.2: 47259582-47353207sly-miR9479-3p 1 sly-miR167b-3p 2
随后用PlantCircNet(http://bis.zju.edu.cn/plantcircnet/)对circRNA、miRNA和mRNA的共表达网络进行可视化分析(图6)。表5是开花期番茄circRNAs与靶向miRNA的结合位点。其中circ_1420、circ_1112、circ_547和circ_473对应miRNA富集的mRNA相对较多。而这些miRNA的主要功能也有其他研究者提出,例如sly-miR167b-3p、sly-miR156e-3p、sly- miR396b、sly-miR9479-3p这4个miRNA主要与非生物胁迫、干旱响应和不同光质处理有关[16-19]。sly-miR5303、sly-miR482d-5p、sly-miR9478-3p、sly-miR1916、sly- miR1918、sly-miR9476-5p这6个miRNA主要与免疫反应、病害感染、植物激素有关[20-23]。推测可能是番茄开花期的生长发育环境刺激了这些circRNA- miRNA-mRNA的表达,导致响应开花的下游基因表达以及各种发育信号应答反应。
图6 开花期番茄circRNA-miRNA-mRNA共表达网络的可视化图 Fig. 6 Visualization plot of the circRNA-miRNA-mRNA co-expression network in flowering tomato
表6 circRNA-miRNA-mRNA模块中靶基因GO注释 Table 6 GO annotation of target genes in the circRNA-miRNA-mRNA module GO类别GO categoryGO名称GO nameGO ID宿主基因数量No. of target genes 生物过程Biological process谷氨酸代谢Glutamate metabolic processGO: 0006536 4 胞外多糖的生物合成Extracellular polysaccharide biosynthetic processGO: 0045226 4 DNA模板化转录调控Regulation of transcription, DNA-templatedGO: 0006355 3 细胞组分Cellular component细胞核NucleusGO: 000563410 线粒体基质Mitochondrial matrixGO: 0005759 2 分子功能Molecular functionATP结合ATP bindingGO: 000552412 蛋白结合Protein bindingGO: 000551512 辅酶结合Coenzyme bindingGO: 0050662 4
P-value<0.05:均具有显著性富集 P-value<0.05: All were significant enriched
3 讨论
3.1 开花期番茄中circRNA的鉴定与特征分析
研究表明,circRNA作为关键的调控分子,以ceRNA的形式参与植物的生长发育,在转录和转录后水平上对基因表达起着重要的调控作用[5,24-25]。番茄作为果实研究的模式作物,一些与番茄果实成熟[8]和果实着色[9]有关的circRNA已被鉴定。但对番茄开花相关的研究还主要集中在编码基因上,对circRNA研究相对较少。因此,揭示开花相关的circRNA对进一步了解番茄的生长发育和生理代谢通路具有重要意义。本研究探索了circRNA在番茄开花过程中的表达和潜在功能,发现342个circRNA在花、叶和根中有显著差异,根组织特异性circRNA在花期番茄中鉴定的最多,这与之前在大豆中的报道类似[26]。表明花期番茄的circRNA在不同组织中具有一定的特异性,其中根组织中剪接生成的circRNA最多。推测可能是由于花期番茄处于生长发育与生殖发育的交接期,需要从土壤中汲取更多的营养和水分所导致。
此外,在分析circRNA的特性时,发现番茄开花期过程中,circRNA来源于不同的基因组区域,但本研究鉴定的绝大多数属于外显子circRNA(83%),这与之前对拟南芥(85.1%)[27]、番茄(86.40%)[28]、白菜(70.49%)[29]和芸苔(87.63%)[30]等许多植物物种的研究一致。此外,与番茄果实着色[8]和番茄乙烯途径[9]中染色体上的circRNA分布一致,相较于其他染色体,1号染色体的circRNA密度最高,说明circRNA在花期番茄各染色体中的分布不均匀。对circRNA进行分类后发现,随着番茄circRNA长度的增加,circRNA的数目不断减少,外显子circRNA作为最常见的类型,在长度较短的circRNA(≤3 600 bp)中所占比例明显较高,这与番茄果实得到的结果类似[8]。表明番茄在生长过程中更有可能形成较短的外显子circRNA来调控番茄的发育。结合前人研究,在番茄生长发育的重要时期(开花期、着色期和完熟期)中,1号染色体剪接的circRNA均最丰富,且容易形成长度较短的circRNA参与开花、果实着色以及果实成熟过程。证明番茄在不同发育阶段的circRNA具有高度保守性,且1号染色体剪接的circRNA可能与番茄的生长发育密切相关。
3.2 番茄响应开花期相关的circRNA成环验证及生物学作用分析
前期研究表明,番茄的circRNA具有高度保守性以及组织特异性。因此,本研究参考Yang等[13]的方法,利用发散引物和收敛引物挑选了开花期差异表达的8个circRNA进行成环验证,验证成功后对其在花、根、叶不同组织中的表达量进行了检测,从分子生物学角度进一步证实了circRNA在番茄开花期中具有组织特异性。
由于circRNA的功能可能与其亲本基因的功能有关,本研究使用GO和KEGG分析注释了circRNA亲本基因的生物学作用,以便于更好地理解circRNA在番茄开花期中的潜在功能。发现circRNA亲本基因可能参与番茄花期发育相关的各种重要过程和功能,如核酸结合、转移酶、激酶活性、生物调节、基因表达调节、细胞的分解代谢途径等。KEGG分析表明,circRNA的亲本基因涉及78条通路。另外,在所列出的前24条KEGG通路中发现,在丙氨酸、天冬氨酸和谷氨酸代谢、磷酸戊糖途径、花生四烯酸的代谢以及脂肪酸代谢合成中均有较高的富集。这些途径可以为植物的生长发育提供大量的能量。此外,这些circRNA被发现在细胞质和质膜上大量表达。前人研究表明细胞核中的circRNA主要起调控亲本基因的作用,而存在于细胞质中的circRNA则起ceRNA的作用[3]。综上,在开花期番茄中,circRNA可以在根、花、叶组织中特异性表达,预测参与多种核酸结合、基因表达调控和分解代谢途径,以及各类生物大分子的合成与代谢。进一步验证了花期番茄的circRNA以ceRNA的方式,调控番茄开花期的基因表达与调控,从而在番茄花期的生长发育和能量代谢过程中发挥重要作用。
3.3 番茄响应开花期相关的潜在circRNA-miRNA- mRNA调控模块
研究表明,circRNA可以作为ceRNA,通过与mRNA、miRNA或RNA结合蛋白(RBP)相互作用来直接调节转录[31-32]。然而对植物中circRNA-miRNA- mRNA网络的预测,与动物和人类相比相对较少,目前只在拟南芥[24]、油菜[25]、番茄[12]和大豆[26]等少数作物中有研究。本研究基于ceRNA作用原理构建了circRNA-miRNA-mRNA相关网络,发现在番茄开花期中有14个circRNA、10个miRNA和165个mRNA可以参与相关共表达网络的构建。其中,大多数circRNA和miRNA是一一对应的关系;也有多个circRNA靶向一个miRNA的情况出现;但是circRNA靶向多个miRNA的只有一个,即circ_1403可以分别靶向sly-miR167b-3p、sly-miR9479-3p。这表明circRNA可以与miRNA相互作用,通过miRNA响应元件(microRNA response elements,MREs)与miRNA结合,从而缓解miRNA导致的基因沉默或降解,促使miRNA靶基因的基因表达量升高,参与调节番茄花期的生长发育。在预测网络中还注意到一些已报道的miRNA,例如miR156、miR167、miR396、miR482、miR1918和sly-miR5303均参与了番茄生长发育、乙烯的生物合成以及果实成熟等过程[9,33-34]。miR1916、miR9476、miR9478和miR9479在番茄生长发育研究中较少有人报道,但在响应非生物胁迫和病害感染免疫应答中有作用[16,35-37]。番茄的花期生长发育并不是一个独立的过程,不仅需要为幼苗期的番茄发育到生殖期提供生理生化保障,也需要为下一步的果实成熟积蓄能量。因此,推测花期番茄的circRNA-miRNA- mRNA可能在番茄生长发育、果实成熟以及胁迫应答过程中起到协同调控的作用。
学生是学校构成的基本主体,是学校管理的主要对象,不管是哪种规章制度,都需要学生和教师双方的共同发展,按照科学的管理制度,提高学生的管理工作,调动师生的积极性。管理制度的规划是不可打破的,这样会让更多的学生懂得约束自己的行为,若想要真正地做到人性化和制度化的融合,这就需要管理人员转变思想,在处理相关问题上坚持以“学生为本”的管理理念,使学生的管理工作充分抓住重点,这都符合人性化和制度化管理的基本要求。
4 结论
通过在番茄花期生长发育过程中分析circRNA表达谱特征,确定了532个circRNA,其中342个circRNA具有组织特异性表达特征。此外,在染色体分布上,circRNA表现出高度的保守性。circRNA宿主基因的功能表征表明,circRNA主要与番茄花期生长发育中的生物大分子合成及代谢有关,如氨基酸合成代谢、磷酸戊糖途径、花生四烯酸的代谢和脂肪酸代谢等过程。此外,发现14个circRNA含有miRNA结合位点,表明circRNA在基因表达中作为转录后调控元件发挥作用。本研究揭示了circRNA在番茄花期的生长发育过程中的潜在功能。这些结果极大地提高了对番茄开花植株发育过程中circRNA的理解,研究结果也为进一步研究circRNA在番茄开花期的功能奠定了基础。
参考文献References
[1] FORNARA F, DE MONTAIGU A, COUPLAND G. SnapShot: Control of flowering in Arabidopsis. Cell, 2010, 141(3): 550-550.e2.
[2] JECK W R, SHARPLESS N E. Detecting and characterizing circular RNAs. Nature Biotechnology, 2014, 32(5): 453-461.
[3] JECK W R, SORRENTINO J A, WANG K, SLEVIN M K, BURD C E, LIU J Z, MARZLUFF W F, SHARPLESS N E. Circular RNAsare abundant, conserved, and associated with ALU repeats. RNA, 2013, 19(2): 141-157.
[4] CONN V M, HUGOUVIEUX V, NAYAK A, CONOS S A, CAPOVILLA G, CILDIR G, JOURDAIN A, TERGAONKAR V, SCHMID M, ZUBIETA C, CONN S J. A circRNA from SEPALLATA3 regulates splicing of its cognate mRNA through R-loop formation. Nature Plants, 2017, 3(5): 17053.
[5] CHENG J P, ZHANG Y, LI Z W, WANG T Y, ZHANG X T, ZHENG B L. A lariat-derived circular RNA is required for plant development in Arabidopsis.Science China Life Sciences, 2018, 61(2): 204-213.
[6] ZENG R F, ZHOU J J, HU C G, ZHANG J Z. Transcriptome-wide identification and functional prediction of novel and flowering-related circular RNAs from trifoliate orange (Poncirus trifoliata L. Raf.). Planta, 2018, 247(5): 1191-1202.
[7] ZUO J H, WANG Q, ZHU B Z, LUO Y B, GAO L P. Deciphering the roles of circRNAs on chilling injury in tomato. Biochemical and Biophysical Research Communications, 2016, 479(2): 132-138.
[8] TAN J J, ZHOU Z J, NIU Y J, SUN X Y, DENG Z P. Identification and functional characterization of tomato circRNAs derived from genes involved in fruit pigment accumulation. Scientific Reports, 2017, 7(1): 8594.
[9] WANG Y X, WANG Q, GAO L P, ZHU B Z, LUO Y B, DENG Z P, ZUO J H. Integrativeanalysis of circrNAs acting as ceRNAs involved in ethylene pathway in tomato. Physiologia Plantarum, 2017, 161(3): 311-321.
[10] YANG Z C, YANG C C, WANG Z Y, YANG Z, CHEN D Y, WU Y J. LncRNA expression profile and ceRNA analysis in tomato during flowering. PLoS ONE, 2019, 14(1): e0210650.
[11] ZUO J H, GRIERSON D, COURTNEY L T, WANG Y X, GAO L P, ZHAO X Y, ZHU B Z, LUO Y B, WANG Q, GIOVANNONI J J. Relationships between genome methylation, levels of non-coding RNAs, mRNAs and metabolites in ripening tomato fruit. The Plant Journal, 2020, 103(3): 980-994.
[12] YANG X D, LIU Y H, ZHANG H, WANG J Y, ZINTA G, XIE S B, ZHU W M, NIE W F. Genome-wide identification of circular RNAs in response to low-temperature stress in tomato leaves. Frontiers in Genetics, 2020, 11: 591806.
[13] YANG Z C, YANG Z, XIE Y G, LIU Q, MEI Y H, WU Y J. Systematic identification and analysis of light-responsive circular RNA and co-expression networks in lettuce (Lactuca sativa). G3 (Bethesda, Md), 2020, 10(7): 2397-2410.
[14] PRENSNER J R, IYER M K, BALBIN O A, DHANASEKARAN S M, CAO Q, BRENNER J C, LAXMAN B, ASANGANI I A, GRASSO C S, KOMINSKY H D, CAO X H, JING X J, WANG X J, SIDDIQUI J, WEI J T, ROBINSON D, IYER H K, PALANISAMY N, MAHER C A, CHINNAIYAN A M. Transcriptome sequencing across a prostate cancer cohort identifies PCAT-1, an unannotated lincRNA implicated in disease progression. Nature Biotechnology, 2011, 29(8): 742-749.
[15] MURRAY M G, THOMPSON W F. Rapid isolation of high molecular weight plant DNA. Nucleic Acids Research, 1980, 8(19): 4321-4326.
[16] TRIPATHI A, GOSWAMI K, TIWARI M, MUKHERJEE S K, SANAN-MISHRA N. Identification and comparative analysis of microRNAs from tomato varieties showing contrasting response to ToLCV infections. Physiology and Molecular Biology of Plants, 2018, 24(2): 185-202.
[17] LIU M M, YU H Y, ZHAO G J, HUANG Q F, LU Y E, OUYANG B. Profiling of drought-responsive microRNA and mRNA in tomato using high-throughput sequencing.BMC Genomics, 2017, 18(1): 481.
[18] ZHOU R, YU X Q, OTTOSEN C O, ZHANG T L, WU Z, ZHAO T M. Unique miRNAs and their targets in tomato leaf responding to combined drought and heat stress. BMC Plant Biology, 2020, 20(1): 107.
[19] LIU Y, SU W J, WANG L J, LEI J, CHAI S S, ZHANG W Y, YANG X S. Integrated transcriptome, small RNA and degradome sequencing approaches proffer insights into chlorogenic acid biosynthesis in leafy sweet potato. PLoS ONE, 2021, 16(1): e0245266.
[20] MIOZZI L, NAPOLI C, SARDO L, ACCOTTO G P. Transcriptomics of the interaction between the monopartite phloem-limited geminivirus tomato yellow leaf curl Sardinia virus and Solanum lycopersicum highlights a role for plant hormones, autophagy and plant immune system fine tuning during infection. PLoS One, 2014, 9(2): e89951.
[21] ZHAO M, JI H M, GAO Y, CAO X X, MAO H Y, OUYANG S Q, LIU P. An integrated analysis of mRNA and sRNA transcriptional profiles in tomato root: Insights on tomato wilt disease. PLoS ONE, 2018, 13(11): e0206765.
[22] HONG Y H, MENG J, HE X L, ZHANG Y Y, LUAN Y S. Overexpression of MiR482c in tomato induces enhanced susceptibility to late blight. Cells, 2019, 8(8): 822.
[23] LÓPEZ-GALIANO M J, SENTANDREU V, MARTÍNEZ-RAMÍREZ A D, RAUSELL C, REAL M D, CAMAÑES G, RUIZ-RIVERO O, CRESPO-SALVADOR O, GARCÍA-ROBLES I. Identification of stress associated microRNAs in Solanum lycopersicum by high-throughput sequencing. Genes, 2019, 10(6): 475.
[24] MENG X W, ZHANG P J, CHEN Q, WANG J J, CHEN M. Identification and characterization of ncRNA-associated ceRNA networks in Arabidopsis leaf development. BMC Genomics, 2018, 19(1): 607.
[25] LIANG Y W, ZHANG Y Z, XU L A, ZHOU D, JIN Z M, ZHOU H Y, LIN S E, CAO J S, HUANG L. CircRNA expression pattern and ceRNA and miRNA-mRNA networks involved in anther development in the CMS line of Brassica campestris. International Journal of Molecular Sciences, 2019, 20(19): 4808.
[26] ZHAO W, CHENG Y H, ZHANG C, YOU Q B, SHEN X J, GUO W, JIAO Y Q. Genome-wide identification and characterization of circular RNAs by high throughput sequencing in soybean. Scientific Reports, 2017, 7(1): 5636.
[27] CHEN G, CUI J W, WANG L, ZHU Y F, LU Z G, JIN B. Genome-wide identification of circular RNAs in Arabidopsis thaliana. Frontiers in Plant Science, 2017, 8: 1678.
[28] WANG W H, WANG J L, WEI Q Z, LI B Y, ZHONG X M, HU T H, HU H J, BAO C L. Transcriptome-wide identification and characterization of circular RNAs in leaves of Chinese cabbage (Brassica rapa L. ssp. Pekinensis) in response to calcium deficiency- induced tip-burn. Scientific Reports, 2019, 9: 14544.
[29] BABAEI S, SINGH M B, BHALLA P L. Circular RNAs repertoire and expression profile during Brassica rapa pollen development. International Journal of Molecular Sciences, 2021, 22(19): 10297.
[30] ZHOU R, XU L P, ZHAO L P, WANG Y L, ZHAO T M. Genome-wide identification of circRNAs involved in tomato fruit coloration. Biochemical and Biophysical Research Communications, 2018, 499(3): 466-469.
[31] HANSEN T B, JENSEN T I, CLAUSEN B H, BRAMSEN J B, FINSEN B, DAMGAARD C K, KJEMS J. Natural RNA circles function as efficient microRNA sponges. Nature, 2013, 495(7441): 384-388.
[32] MEMCZAK S, JENS M, ELEFSINIOTI A, TORTI F, KRUEGER J, RYBAK A, MAIER L, MACKOWIAK S D, GREGERSEN L H, MUNSCHAUER M, LOEWER A, ZIEBOLD U, LANDTHALER M, KOCKS C, LE NOBLE F, RAJEWSKY N. Circular RNAs are a large class of animal RNAs with regulatory potency. Nature, 2013, 495(7441): 333-338.
[33] ARORA S, PANDEY D K, CHAUDHARY B. Target-mimicry based diminution of miRNA167 reinforced flowering-time phenotypes in tobacco via spatial-transcriptional biases of flowering-associated miRNAs. Gene, 2019, 682: 67-80.
[34] MA L L, MU J L, GRIERSON D, WANG Y X, GAO L P, ZHAO X Y, ZHU B Z, LUO Y B, SHI K, WANG Q, ZUO J H. Noncoding RNAs: functional regulatory factors in tomato fruit ripening. Theoretical and Applied Genetics, 2020, 133(5): 1753-1762.
[35] CANDAR-CAKIR B, ARICAN E, ZHANG B H. Small RNA and degradome deep sequencing reveals drought-and tissue-specific micrornas and their important roles in drought-sensitive and drought- tolerant tomato genotypes. Plant Biotechnology Journal, 2016, 14(8): 1727-1746.
[36] KAUR P, SHUKLA N, JOSHI G, VIJAYAKUMAR C, JAGANNATH A, AGARWAL M, GOEL S, KUMAR A. Genome-wide identification and characterization of miRNAome from tomato (Solanum lycopersicum)roots and root-knot nematode (Meloidogyne incognita) during susceptible interaction. PLoS ONE, 2017, 12(4): e0175178.
[37] CHEN L, MENG J, HE X L, ZHANG M, LUAN Y S. Solanum lycopersicum microRNA1916 targets multiple target genes and negatively regulates the immune response in tomato. Plant, Cell & Environment, 2019, 42(4): 1393-1407.
Sequencing and Functional Analysis of Tomato circRNA During Flowering Stage
YIN ZiHe, YANG ChengCheng, ZHAO YuHui, ZHAO Li, LÜ XiuRong, YANG ZhenChao, WU YongJun
College of Life Sciences/College of Horticulture, Northwest A&F University, Yangling 712100, Shaanxi
Abstract:【Background】As one of the most important periods in plant growth and development, the flowering period directly affects fruit ripening and seed development. circRNAs are a class of covalent closed-loop RNA molecules that are ubiquitous in eukaryotic cells and play an important role in the regulation of tomato development and stress response. However, the current circRNA studies on tomato mainly focus on fruit and leaves, and there is a lack of systematic studies on tomato circRNA at flowering stage.【Objective】Identification and analysis of circRNs in flowering tomato could be of great significance for the functional study of miRNA and circRNA in tomato, and also layed a foundation for the study of tomato growth, development and stress response mechanism.【Method】 circRNA sequencing was performed on 3 tissue samples of flowers, roots and leaves of flowering tomato plants, with 3 replicates for each sample. circRNAs were identified and their basic characteristics were analyzed. The cycle-forming ability of tissue-specific circRNAs was screened, and the host genes of identified circRNAs were analyzed by GO analysis and KEGG analysis. The mode and site of action of circRNAs were predicted and analyzed by bioinformatics methods to construct a potential circRNA-miRNA-mRNA interaction regulatory network in response to tomato growth and development.【Result】A total of 532 circRNAs were obtained by high-throughput sequencing, 83% of which were exon types. The distribution of circRNA in each chromosome of flowering tomato was uneven, among which chromosome 1 produced the most circRNAs and chromosome 5 produced the least. circRNAs differentially expressed in flower, leaf and root tissues of flowering tomato showed that 79 circRNAs were differentially expressed in flower and leaf, 133 circRNAs were differentially expressed in flower and root, and 132 circRNAs were differentially expressed in leaf and root tissues. Among them, 14 circRNAs were differentially expressed in flower, leaf and root tissues. The cyclization ability of 8 circRNAs randomly selected from 14 differentially expressed circRNAs was tested, and the results showed that all 8 circRNAs had cyclization ability. GO analysis and KEGG analysis showed that circRNAs in flowering tomato were mainly related to the binding of nucleic acids, proteins and other small molecules, as well as the synthesis and metabolism of various biological macromolecules. Finally, the tomato circRNA-miRNA-mRNA interaction regulatory network composed of 14 circRNAs, 10 miRNAs and 136 mRNAs was constructed.【Conclusion】A total of 342 tissue-specific circRNAs were identified, among which 14 were significantly expressed, and 8 were successfully identified. A circRNA-miRNA-mRNA interaction regulatory network was constructed for tomato at flowering stage. This study laid a foundation for the subsequent research of circRNA in flowering period.
Key words: circRNA; tomato; flowering stages; miRNA; ceRNA
开放科学(资源服务)标识码(OSID):
收稿日期:2023-04-03;
接受日期:2023-06-09
基金项目:陕西省农业部技术创新引导专项(2021QFY08-02)
联系方式:殷子贺,E-mail:ziheyin@nwafu.edu.cn。通信作者武永军,E-mail:wuyongjun@nwafu.edu.cn
(责任编辑 赵伶俐)
