水稻Pi9位点6个稻瘟病抗性基因特异分子标记的开发及应用
2023-11-18杨好黄衍焱易春霖石军谭楮湉任文芮王文明
杨好,黄衍焱,易春霖,石军,谭楮湉,任文芮,王文明
水稻位点6个稻瘟病抗性基因特异分子标记的开发及应用
杨好1,黄衍焱1,易春霖1,石军2,谭楮湉1,任文芮1,王文明1
1四川农业大学西南作物基因资源发掘与利用国家重点实验室,成都 611130;2绵阳市农业科学研究院水稻研究所/厅市共建作物特色资源创制及应用四川省重点实验室,四川绵阳 621023
【目的】水稻位点由多个串联的同源基因组成,从中已克隆了超过10个优良的稻瘟病抗性基因。论文旨在鉴别水稻亲本位点抗性基因的组成,促进该位点基因快速、精准地应用于水稻抗性育种。【方法】对比位点已克隆抗性基因的序列,从中发掘各基因特异的核苷酸位点;然后将各目标基因分别与数据库(Rice Resource Center)中的155个水稻基因组进行比对分析,进一步从中筛选出特异最强的核苷酸位点,用于、、、、和6个抗性基因特异分子标记的开发;以24个抗稻瘟病单基因系、阳性对照和110个四川盆地水稻亲本为鉴定对象,通过优化PCR扩增条件、测序或基因组数据分析检验分子标记鉴定结果的准确性。由于该位点基因的同源性较高、基因间常常拥有一些相同的特异位点,导致许多抗性基因很难用一对分子标记进行精准鉴定,因此采用多对分子标记共同鉴定的方式;另外许多特异位点为单碱基差异,因此需要对差异位点旁的碱基进行特异突变,使引物的3′端具有两个碱基的错配,以提高PCR扩增的特异性。【结果】最终为6个位点的抗性基因开发了有效的分子标记,发现32.09%的参试水稻材料含有位点抗性基因。、、、、和分别存在于1、7、8、14、23和33个水稻材料中,其中仅存在于单基因系中。这些抗性基因通常两个或多个组合在一起,同时存在于一个水稻材料中。其中往往与或成对出现,仅在成恢993、HR2168和绵恢365 3个亲本中单独存在。雨恢38含有的位点抗性基因最多,分别有、、和。川谷B、川农4B、内香6B和双1B中均同时含有、和3个抗性基因。千乡654B中含有和两个抗性基因。【结论】为位点的6个同源稻瘟病抗性基因开发了特异的分子标记,明确了四川盆地110个水稻亲本位点的抗性基因组成,揭示了位点抗性基因丰富的组合形式,为水稻育种的抗原选择提供了明确参考。
水稻;稻瘟病;位点;同源基因;分子标记
0 引言
【研究意义】稻瘟病是威胁水稻安全生产的重要病害之一,通过抗性育种导入或聚合抗性基因是提高水稻抗性最经济有效的途径。在已知的抗性基因中,位点串联的同源基因最多,一般在9—15个[1-4],从中克隆了[5-6]、[7]、[8]、[7-8]、[3]、[1]、[2]和[9]等10余个广谱抗性基因,其中、和等基因在全国各稻作区均具有良好的稻瘟病抗性,并得到了广泛应用[10-13]。为了在育种中更加高效而精准地利用位点的稻瘟病抗性基因,需要有特异的分子标记从众多同源基因中精准地鉴别各个抗性基因,从而明确水稻亲本中位点的抗性基因组成。进一步利用这些分子标记进行辅助选育,促进位点抗性基因在抗病育种中的应用,提高水稻对稻瘟病的抗性,为水稻的安全生产提供保障。【前人研究进展】水稻位点中串联多个同源基因,导致该位点功能基因的克隆、区分及鉴别相对困难。在水稻抗病材料二八占中,位点包含12个同源基因,其中和-的DNA序列完全相同,介导了的广谱抗性。和也有一样的DNA序列,与仅有8个SNP的差异,但不具有抗稻瘟病的功能[2]。与基因序列相同的(),赋予了谷梅4号广谱的稻瘟病抗性,其编码的蛋白与无抗性功能的同源蛋白R4相比仅有4个氨基酸的差异[1]。不仅串联的同源(旁系同源基因)同源性高,不同水稻材料中克隆的直系同源(orthologous gene)基因间也非常保守,例如中花11中克隆的与Toride 1中克隆的编码相同的氨基酸序列,和C101A51中克隆的编码的抗性蛋白间仅有8个氨基酸的差异[7-8]。而从地谷B中克隆的虽然与同位点的其他抗性基因的同源性较低,但与其他位点的抗性基因相比,仍然与同位点的其他抗性基因在同一进化分支中[9]。【本研究切入点】由于位点抗性基因的同源性高,有的串联在一个基因位点中,因此使用连锁标记等方法很难准确地区分和鉴别该位点的抗性基因。另外,位点抗性基因具有广谱的稻瘟病抗性,在水稻抗性育种中具有很高的应用价值,然而绝大部分水稻亲本没有明确的抗性基因组成,因此本研究通过比对位点已知同源基因的DNA序列,分别筛选、、、、和特异的核苷酸多态性位点,开发特异的分子标记引物对。以24个抗稻瘟病单基因系、阳性对照和110个四川盆地水稻亲本为鉴定对象,通过优化PCR扩增条件、测序或基因组数据分析,检验分子标记鉴定结果的准确性。【拟解决的关键问题】为位点的6个抗性基因、、、、和开发特异的分子标记,明确110个参试水稻亲本位点的抗性基因组成。
1 材料与方法
试验于2022年9月至2023年7月在四川农业大学西南作物基因资源发掘与利用国家重点实验室完成。
1.1 水稻材料
24份抗稻瘟病单基因系水稻材料,110份四川盆地常用水稻育种亲本材料,以及对照材料地谷B、谷梅4号、日本晴(Nipponbare,NPB)、中花11(Zhonghua 11,ZH11)和丽江新团黑谷(Lijiangxintuanheigu,LTH)(表1)。
1.2 核苷酸多态性序列的筛选及引物设计
通过在线多序列比对网站(https://www.novopro. cn/tools/muscle.html)分析位点8个同源基因,、、、、、、和,在编码区的核苷酸多态性位点,然后在水稻资源中心(http://ricerc.sicau.edu.cn/)的155个水稻资源(包括籼稻、粳稻、非洲栽培稻和野生稻等)基因组中分析这些多态性位点的特异性,在特异性最强位置开发分子标记。当单核苷酸差异位点作为分子标记引物的3′末端时,则将其相邻的第二位碱基进行特异突变,形成新的错配位点,使引物与非目标结合位点的3′末端始终保持两个碱基的错配,减少PCR过程中非特异扩增的产生。
1.3 水稻DNA提取
水稻DNA采用CTAB法提取:取100 mg水稻叶片装入1.5 mL离心管中,液氮中磨碎后,加入500 μl CTAB溶液。然后放置在65 ℃烘箱中萃取,每隔10 min混匀一次。30 min后将样品转移到室温,冷却后加500 μL氯仿/异戊醇(24﹕1)溶液,混匀并在室温静置5 min。然后12 000 r/min离心15 min,将上清转移到新的1 mL离心管中,加入等体积无水乙醇,混匀后放置在-20 ℃冰箱中10 min。然后12 000 r/min离心10 min,弃上清,加入1 L预冷的75%无水乙醇洗涤沉淀两次,室温下干燥沉淀,最后加入100 μL ddH2O溶解DNA,放置于-20 ℃冰箱备用。
1.4 PCR扩增条件及分子标记引物对的准确性检验
PCR扩增所用的DNA聚合酶来源于北京庄盟国际生物基因科技有限公司(货号:ZT101),PCR扩增体系:2.0 μL模板DNA(400 ng·μL-1),2.0 μL Buffer,0.5 μl dNTP(2.5 mmol·L-1each),0.5 μL正向引物(10 μmol·L-1),0.5 μL反向引物(10 μmol·L-1),0.5 μL Taq酶(2.5 U·μL-1),14.0 μL ddH2O,共20 μL。
为了提高分子标记的准确性,以单基因系、NPB()、ZH11(/)、谷梅4号(和)为阳性对照,感病材料LTH等为阴性对照,调整和优化PCR扩增条件(表2)。为了检验标记结果,如果被标记材料在水稻资源中心数据库中有全基因组序列(蜀恢498、蜀恢527、蜀恢548、地谷B、雅恢2115、宜香1B、华占、五山丝苗、中花11、日本晴和LTH),则通过序列分析检验标记结果的准确性。如果标记结果中没有这些材料,将随机进行测序分析检验,最终为、、、、和6个同源抗性基因开发有效而准确的分子标记引物对(表2)。
2 结果
2.1 Pi2和Piz-t分子标记的开发及应用
特异分子标记引物对中,正向引物Pi2/z-t-F的特异性不强,而反向引物Pi2-R位于多态性丰富的区域,具有很强的特异性(图1-A)。使用这对分子标记,24个单基因系中仅含有的单基因系材料IRBLz5-CA被特异地标记,在110个水稻亲本中,有22个材料被标记(图1-B、表3),其中包括雅恢2115、华占、蜀恢548和五山丝苗4个有全基因组数据的材料。经比对分析,这4个材料均含有,另外也有研究报道雅恢2115和蜀恢548含有[14-15],蜀恢586经过PCR扩增和测序,确认含有。说明的分子标记具有很强的特异性。
由于拥有的核苷酸特异位点较少,经反复试验,设计了3个特异分子标记共同鉴别。如图1-C所示,分子标记引物对Piz-t-F4和Piz-t-R4不能区分、与3个基因;引物对Pi2/z-t-F和Piz-t-R2不能区分、与3个基因;引物对Pi2/z-t-F和Piz-t-R3不能区分与两个基因。同时使用这3个分子标记,24个单基因系中仅含有的材料IRBLzt-T被同时标记,水稻亲本材料中有13个被同时标记(表4)。其中包括蜀恢498和中花11,分析其全基因组序列,证明含有,泸恢8258、明恢86和蜀恢137经过PCR扩增和测序,确认含有。说明使用这3对分子标记可以准确地标记含有的水稻材料。
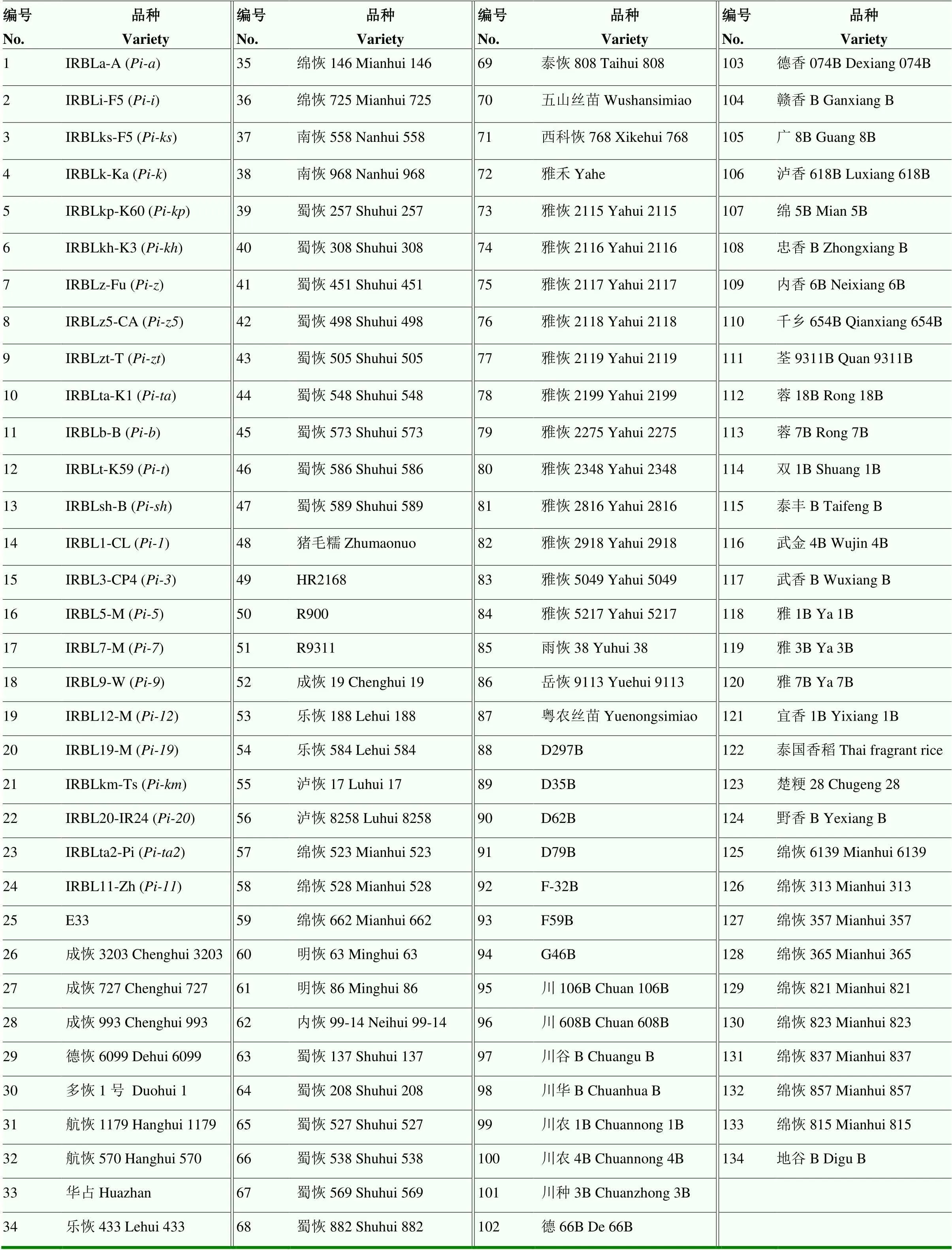
表1 供试水稻品种
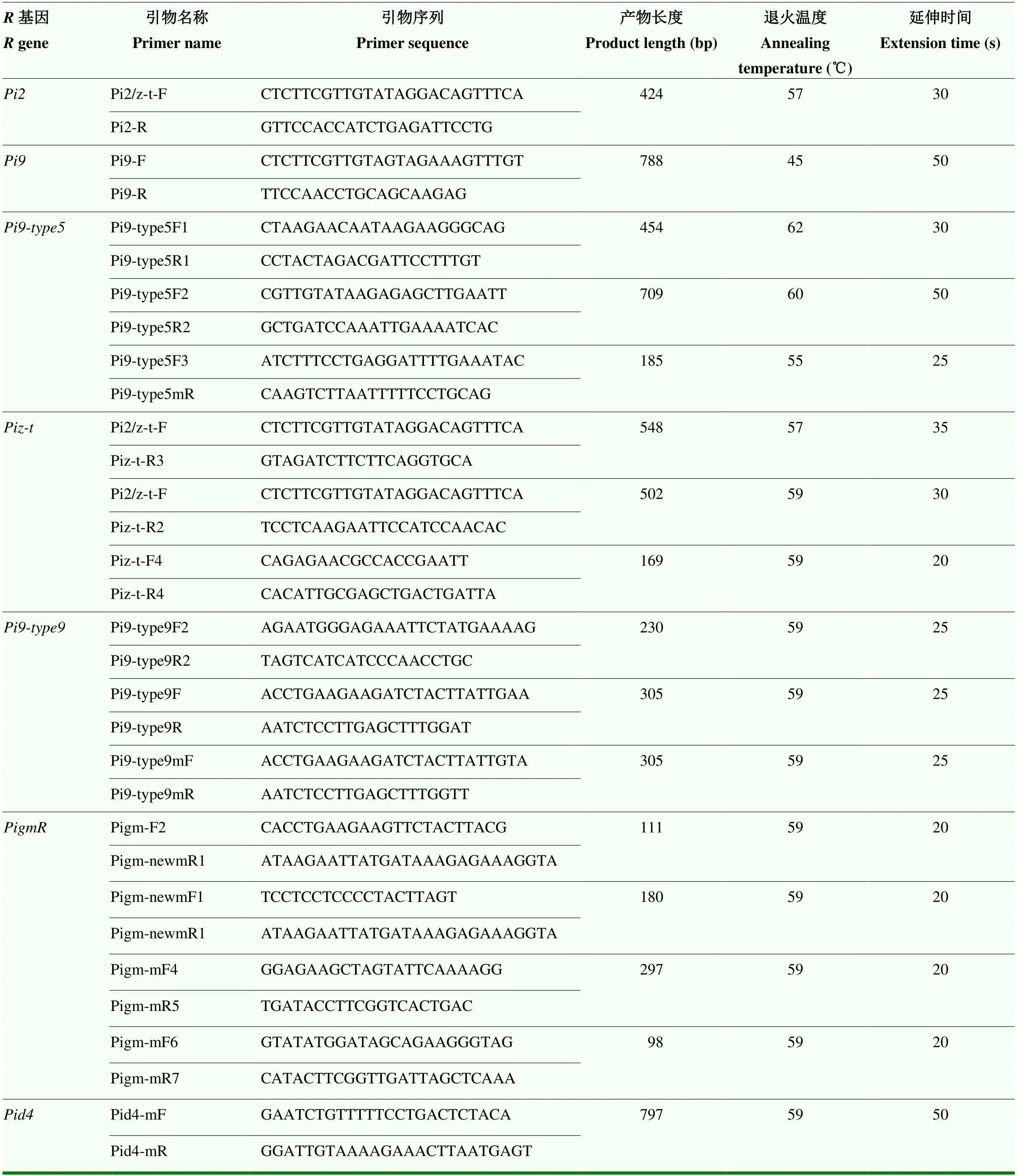
表2 分子标记引物对及PCR扩增条件
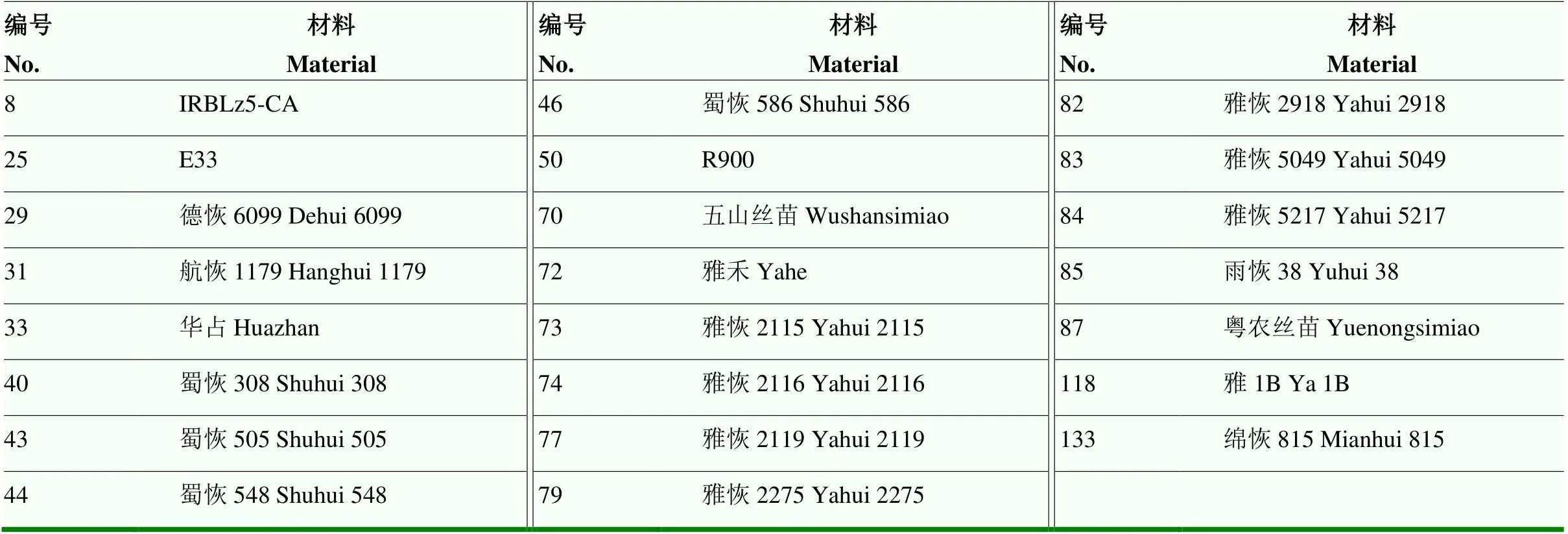
表3 含有Pi2的水稻材料
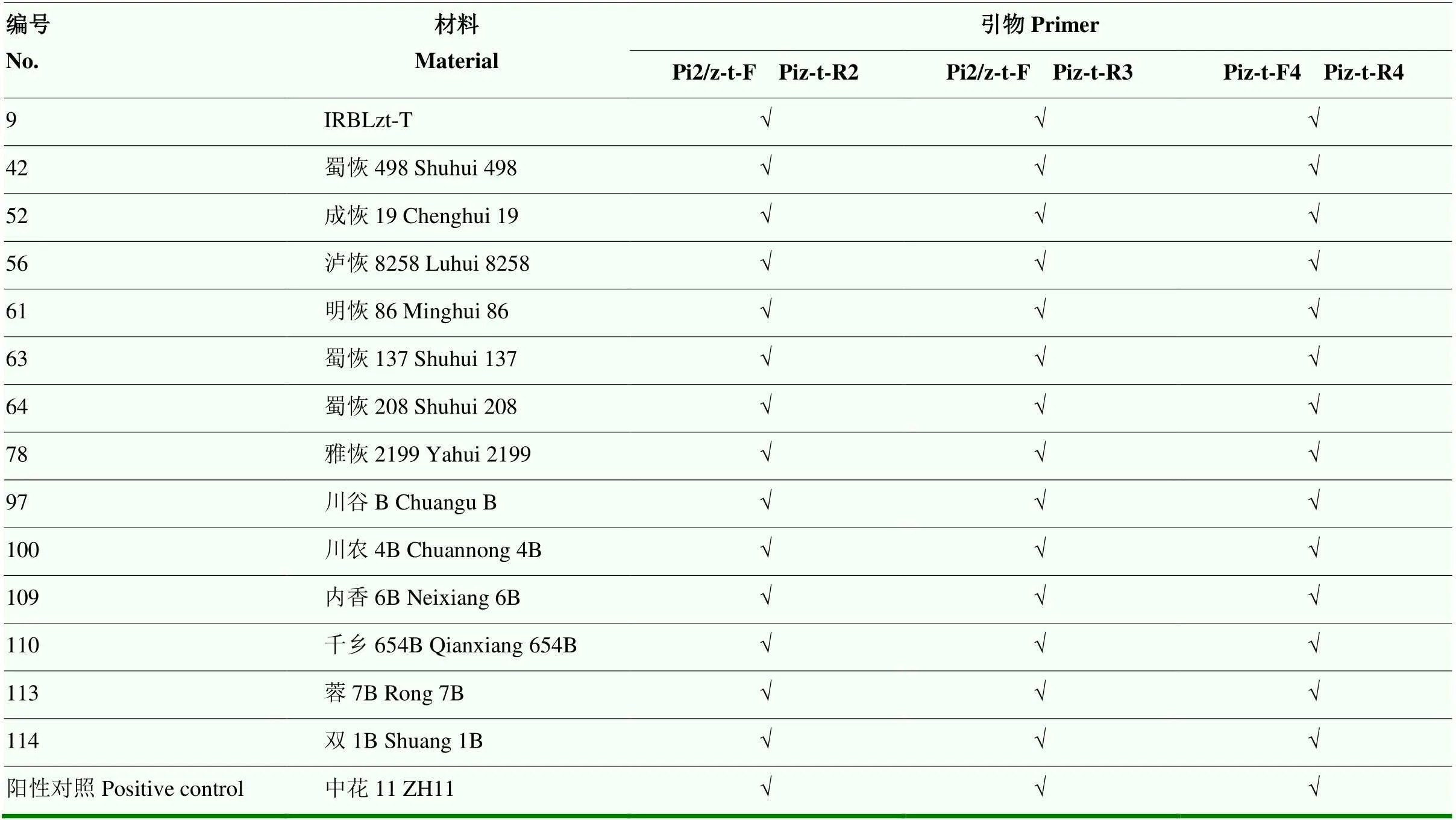
表4 含有Piz-t的水稻材料
2.2 Pi9和Pi9-type5分子标记的开发及应用
在分子标记的引物对中,正向引物在其他同源基因中均有5个以上的错配位点,具有很高的特异性。反向引物在中无错配位点,但在其他同源基因中均有6个以上的错配位点(图2-A)。使用这对引物鉴定,发现所有参试材料中,仅含有的单基因系IRBL9-W能够被标记。说明该分子标记具有很高的特异性,不存在于110个水稻亲本中。
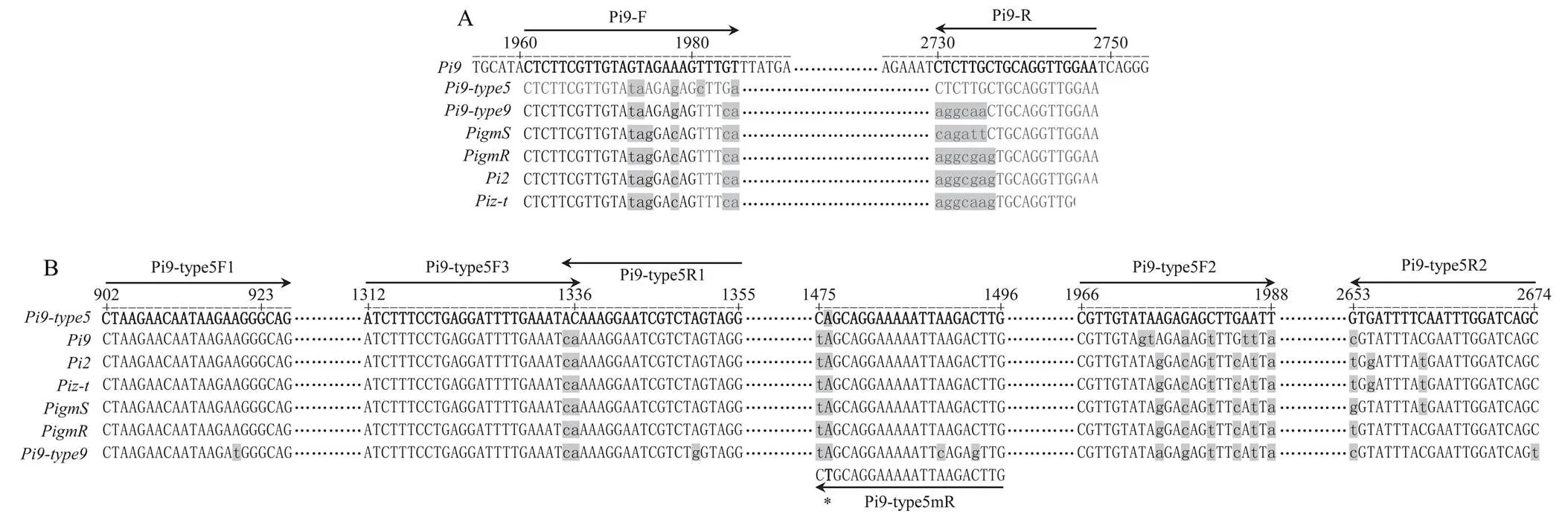
A:Pi9分子标记引物对Pi9-F和Pi9-R所在位置核苷酸的多态性分析The nucleotide polymorphism analysis of primer pair, Pi9-F and Pi9-R, for the molecular markers of Pi9。B:Pi9-type5分子标记引物对Pi9-type5F1和Pi9-type5R1,Pi9-type5F2和Pi9-type5R2,Pi9-type5F3和Pi9-type5mR所在位置核苷酸的多态性分析。箭头标记引物位置及方向,*表示引物中引入的特异错配碱基,以提高PCR扩增的特异性。灰色阴影标记的小写字母为核苷酸多态性位点,灰色阴影标记的大写字母为引物特异性突变创造的错配位点The nucleotide polymorphism analysis of primer pairs, Pi9-type5F1 and Pi9-type5R1, Pi9-type5F2 and Pi9-type5R2, Pi9-type5F3 and Pi9-type5mR, for the molecular markers of Pi9-type5. The arrows indicate the site and direction of primers. * indicates artificial mutated base in primer for improving specificity of PCR amplification. The shaded lowercase letters indicate the polymorphic nucleotide sites in homologous genes. The shaded uppercase letters indicate the primer mismatched site generated by artificial mutated base in homologous genes
经反复试验,发现需要用3个分子标记引物对共同鉴定。引物Pi9-type5F1尽管在已知的同源基因中几乎无特异性(图2-B),然而其3′末端的两个碱基在155个水稻种质资源中的某些未知同源基因序列中有较高的特异性。经Pi9-type5F1与Pi9-type5R1、Pi9-type5F2与Pi9-type5R2、Pi9-type5F3与Pi9-type5mR共同鉴定,发现它们标记的材料完全相同(表5),包括两个单基因系IRBLz5-CA()和IRBLzt-T(),以及31个水稻亲本材料。这3个分子标记利用不同的核苷酸多态性位点得到了相同的结果,说明3个分子标记对均有很好的特异性。抗病单基因系IRBLz5-CA()和IRBLzt-T()携带的抗性基因分别来源于克隆这两个抗性基因的品种C101A51和Toride 1[16],在这两个材料中,与的启动子及编码区序列一致[8]。序列比对发现与的序列一致,因此单基因系IRBLz5-CA和IRBLzt-T分别携带的抗性基因与即为,另外,经PCR扩增和测序,再次确认两个单基因系含有。分析雅恢2115、华占、蜀恢548和五山丝苗4个亲本材料的基因组序列,发现它们均含有。雅恢2116和粤农丝苗经过PCR扩增和测序,确认含有。表明这3个分子标记均有很好的特异性,能够有效地鉴别。

表5 含有Pi9-type5的水稻材料
2.3 PigmR分子标记的开发及应用
在开发分子标记的过程中,诸多基于特异性核苷酸位点的分子标记引物对依然能从中花11、NPB或者LTH等明确不含有的水稻材料中扩增出标记片段。经过不断筛选、试验和检验,最终发现Pigm-F2和Pigm-newmR1、Pigm-newmF1和Pigm-newmR1、Pigm-mF4和Pigm-mR5、Pigm-mF6和Pigm-mR7 4个分子标记引物对可以准确地将从参试水稻材料中鉴别出来(图3)。在110个水稻亲本中共发现8个含有的材料(表6)。其中包含地谷B,使用序列在地谷B的基因组中比对,明确地谷B中含有。另外随机挑选水稻亲本材料蓉18B和川谷B,通过PCR对进行克隆及测序分析,明确蓉18B和川谷B中含有。说明使用这些分子标记共同鉴定具有良好的特异性。

箭头标记引物位置及方向,*表示引物中引入的特异错配碱基,以提高扩增的特异性。灰色阴影标记的小写字母为核苷酸多态性位点,灰色阴影标记的大写字母为引物特异性突变创造的错配位点
2.4 Pid4分子标记的开发及应用
由于在DNA序列上与其他同源基因、、和等有明显的差异(图4-A),特异的核苷酸多态性位点很难通过与这些基因的同源比对中获得。而水稻资源中心的155个水稻资源(包括籼稻、粳稻、非洲栽培稻和野生稻等)基因组数据为分析的特异核苷酸多态性位点提供了充足的同源基因资源。通过比对分析,最终将分子标记设计在特异性最强位点(图4-A)。以克隆的地谷B为阳性对照,最终从参试材料中鉴定出了7个水稻亲本含有,而所有的单基因系不含该基因(图4-B、表7),川农4B、内香6B和双1B经过PCR扩增和测序,确认含有。

表6 含有PigmR的水稻材料
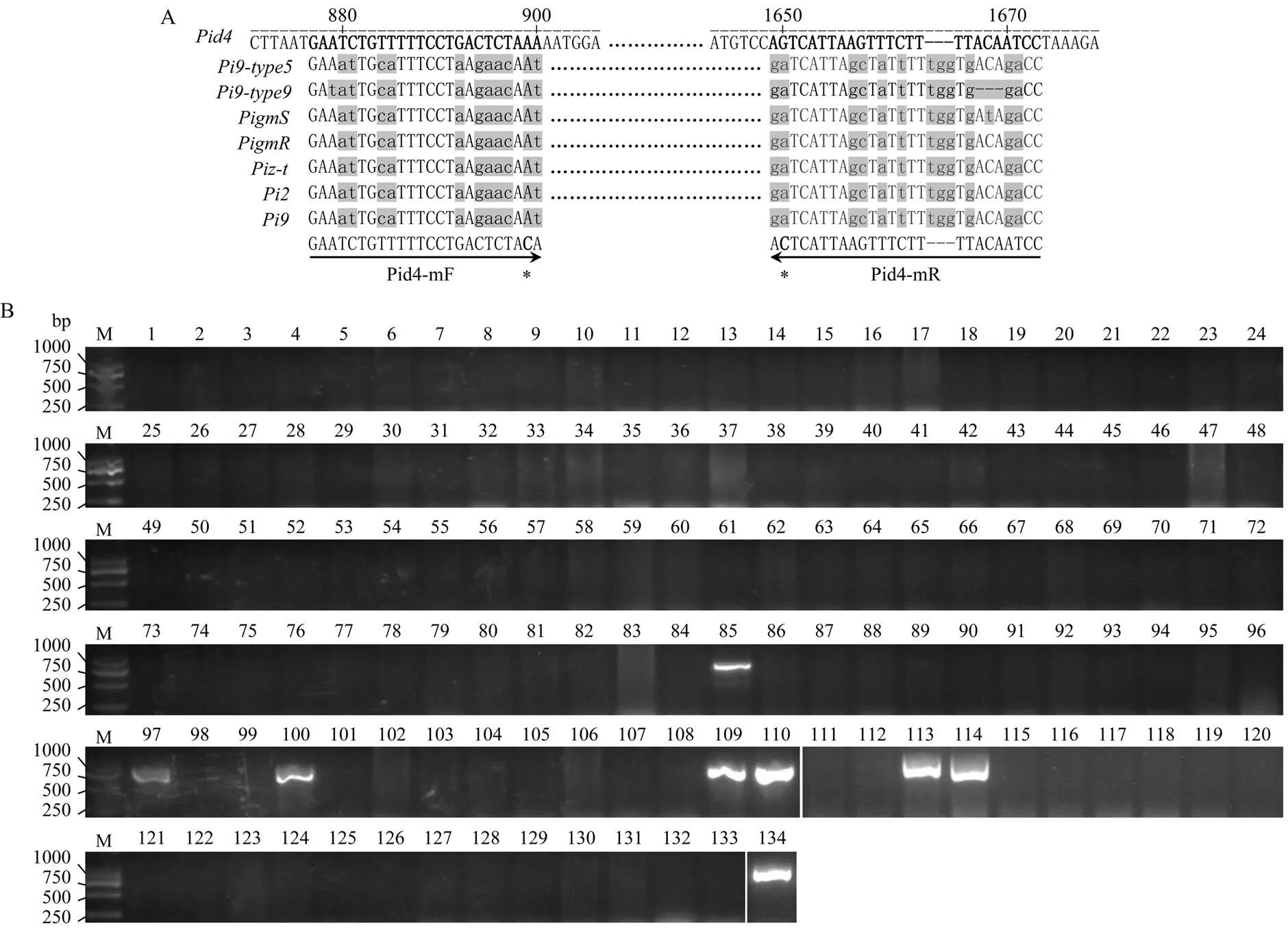
A:Pid4分子标记引物对的核苷酸多态性分析。箭头标记引物位置及方向,*表示引物中引入的特异错配碱基,以提高扩增的特异性。灰色阴影标记的小写字母为核苷酸多态性位点,灰色阴影标记的大写字母为引物特异性突变创造的错配位点The nucleotide polymorphism analysis of primer pairs for the molecular marker of Pid4. The arrows indicate the site and direction of primers. * indicates artificial mutated base in primer for improving specificity of amplification. The shaded lowercase letters indicate the polymorphic nucleotide sites in homologous genes. The shaded uppercase letters indicate the primer mismatched sites generated by artificial mutated base in homologous genes。B:分子标记鉴定参试水稻品种中的Pid4 The molecular marker was used to characterize the Pid4 in tested rice varieties。M:DNA marker

表7 含有Pid4的水稻材料
2.5 Pi9位点同源稻瘟病抗性基因在四川盆地水稻亲本中的应用现状
根据鉴定结果,32.09%的参试水稻材料含有位点抗性基因,、、、、和分别存在于1、7、8、14、23和33个材料中(表3—表7)。其中仅在阳性单基因系IRBL9-W中存在,因此在参试的水稻亲本中均不存在。根据表8所展示的水稻亲本在位点的抗性基因组成,发现绝大多数材料同时含有多个同源抗性基因。和同时存在于23个水稻材料的位点中,其中包括单基因系IRBLz5-CA()、华占、蜀恢548和五山丝苗。分析华占、蜀恢548和五山丝苗的基因组数据,证实同时含有和。另外还有8个水稻材料中同时含有和,其中包括单基因系材料IRBLzt-T()和蜀恢498,分析蜀恢498的基因组数据,证实蜀恢498的位点同时含有和。
分子标记鉴定结果显示,地谷B中含有和,分析其基因组序列证实了这一鉴定结果。另外,千乡654B中含有和;川谷B、川农4B、内香6B和双1B中均同时含有、和3个抗性基因;雨恢38中含有、、、4个抗性基因。
位点中,仅含有一个抗性基因的水稻材料较少,如含有的单基因系IRBL9-W,含有的成恢993、HR2168和绵恢365,含有的蓉18B。这些结果表明,四川盆地主要水稻亲本材料中,位点的抗性基因组合形式丰富,为水稻抗性育种贡献了重要的基因资源。
3 讨论
3.1 基因编码区开发分子标记的优势
分子标记是PCR扩增出的特异片段,一般通过其大小、多少、有无或是否能被酶切等方式标记和鉴别特定的基因,分子标记的引物对一般位于基因的连锁区或基因内。连锁区的分子标记,如SSR等利用基因连锁区的差异进行特异标记。这样的分子标记往往受连锁累赘或遗传背景等问题的限制,有可能因染色体发生交换而出现错选和漏选目标基因的情况[17],在遗传背景相近或者标记位点无差异的两个材料间很难进行有效的鉴定与筛选。更不能鉴别出不同遗传背景材料中所含的相同抗性基因,比如中花11中克隆的和Toride 1中克隆的,它们编码氨基酸序列相同的抗性蛋白[7-8],二八占中克隆的与谷梅4号中克隆的拥有完全一样的DNA序列[1-2],本研究通过分子标记及基因组序列分析发现,地谷B中也含有。说明相同的抗性基因可能来源于血缘遗传,但也可能源于相同或相似选择压力下趋同的进化。
在目标基因的编码区开发分子标记,可以有效地摆脱分子标记辅助筛选过程中面临的连锁累赘和遗传背景等问题的制约,更精准地鉴别目标基因。由于位点是由多个串联的同源基因组成,有些基因间的同源性非常高,导致分子标记的准确性面临着很大的挑战。例如与附近串联的无抗性功能的编码的蛋白仅有4个氨基酸差异[1]。在实验过程中,有些的分子标记引物对能在LTH或不含有目标基因的阴性对照材料中扩增出标记片段。为了解决这一问题,首先将目标基因的特异核苷酸多态性位点排列出来,分析这些位点在不同同源基因中的分布特点,然后从中筛选多个特异位点开发分子标记,通过多重PCR筛选,逐步将高度同源的基因排除,从而提高鉴定结果的准确性。尽管目前位点的多个抗性基因均开发了特异的分子标记[17-21],有些标记成功地应用到了分子辅助育种中[22-25],但这些分子标记局限于材料的遗传背景,只能对特定材料进行分子辅助选育。本研究开发的分子标记,不仅能用于分子标记辅助育种,还能突破遗传背景的限制,用于鉴别水稻亲本及资源材料中的抗性基因组成,为水稻抗性育种明确更多种质资源的抗性基因组成,促进抗性育种的发展。

表8 水稻亲本基因组中Pi9位点稻瘟病抗性基因的组成
3.2 Pi9位点的抗性可能由多个抗性基因协同产生
位点串联着多个同源基因,大多数情况下由其中一个抗性基因赋予了水稻广谱的稻瘟病抗性,如、[8][5]和[2]等。本研究通过分子标记鉴别,揭示了位点抗性基因组成的复杂性。在参试水稻材料中,仅有5个材料的位点含有单个抗性基因。其他位点含有抗性基因的材料中,除了和不能同时存在外,该位点的抗性基因均能以不同的组合方式聚合在一起,其中主要以与或基因组合的方式存在(表8)。在以往的研究中也有相似的发现,如中花11中串联同源基因()对()的抗性功能有重要贡献[3]。谷梅4号中,和两个基因在抗性中起着相反的作用,相互协作,平衡抗性与产量[1]。位点之所以只能克隆出其中某一个抗性基因,原因可能在于该位点不同抗性基因所编码的蛋白识别不同的效应因子。因此串联多个同源基因的位点可能是一个聚合了多个抗性基因的位点,赋予了水稻更广谱的抗性。在全国各个稻作区,位点的抗性基因一般比其他位点的抗性基因具有更强的稻瘟病抗性[26-28],而且、、和等广谱抗病基因在全国均有广泛的应用[29-32]。由于不同材料中位点串联着不同数量的基因,因此杂交时该位点的重组可能使子代形成不同于双亲的基因组合方式,这可能是该位点快速进化和一直保持较高稻瘟病抗性的原因之一。另外,尚不清楚位点不同抗性基因如何共同发挥抗病功能,这为深入解析位点的抗病机制提供了新的思路。
4 结论
成功地为位点的6个同源稻瘟病抗性基因、、、、和开发了特异的分子标记,明确了四川盆地110个水稻亲本位点的抗性基因组成。参试水稻亲本中不存在,而、、、和5个基因通常以两个或多个基因组合的方式存在于36.36%的水稻亲本中。
致谢:中国农业大学赵文生教授提供了24份抗稻瘟病单基因系水稻材料,四川农业大学黄富教授和王玉平教授、四川省绵阳市农业科学研究院石军研究员和四川省内江市农业科学研究院李小艳博士提供了110份四川盆地常用水稻育种亲本材料,四川省农业科学研究院植物保护研究所彭云良教授提供了地谷B和谷梅4号。在此一并表示感谢!
[1] DENG Y, ZHAI K, XIE Z, YANG D, ZHU X, LIU J, WANG X, QIN P, YANG Y, ZHANG G,. Epigenetic regulation of antagonistic receptors confers rice blast resistance with yield balance. Science, 2017, 355(6328): 962-965.
[2] SU J, WANG W, HAN J, CHEN S, WANG C, ZENG L, FENG A, YANG J, ZHOU B, ZHU X. Functional divergence of duplicated genes results in a novel blast resistance geneat thelocus. Theoretical and applied genetics, 2015, 128(11): 2213-2225.
[3] XIE Z, YAN B, SHOU J, TANG J, WANG X, ZHAI K, LIU J, LI Q, LUO M, DENG Y, HE Z. A nucleotide-binding site-leucine-rich repeat receptor pair confers broad-spectrum disease resistance through physical association in rice. Philosophical transactions of the Royal Society of London. Series B, Biological Sciences, 2019, 374(1767): 20180308.
[4] 黄衍焱, 李燕, 王贺, 王文明.水稻小种特异性抗稻瘟病基因的等位性变异研究进展.植物病理学报, 2023, doi: 10.13926/j.cnki. apps.000866.
HUANG Y Y, LI Y, WANG H, WANG W M. Allelic variation in the race-specific blast resistance genes in rice. Acta Phytopathologica Sinica, 2023, doi: 10.13926/j.cnki.apps.000866. (in Chinese)
[5] QU S, LIU G, ZHOU B, BELLIZZI M, ZENG L, DAI L, HAN B, WANG G L. The broad-spectrum blast resistance geneencodes a nucleotide-binding site-leucine-rich repeat protein and is a member of a multigene family in rice. Genetics, 2006, 172(3): 1901-1914.
[6] ZHOU B, DOLAN M, SAKAI H, WANG G L. The genomic dynamics and evolutionary mechanism of thelocus in rice. Molecular Plant-Microbe Interactions, 2007, 20(1): 63-71.
[7] ZHOU Y, LEI F, WANG Q, HE W, YUAN B, YUAN W. Identification of novel alleles of the rice blast-resistance genethrough sequence-based allele mining. Rice, 2020, 13(1): 80.
[8] ZHOU B, QU S, LIU G, DOLAN M, SAKAI H, LU G, BELLIZZI M, WANG G L. The eight amino-acid differences within three leucine-rich repeats betweenandresistance proteins determine the resistance specificity to. Molecular Plant-Microbe Interactions, 2006, 19(11): 1216-1228.
[9] CHEN Z, ZHAO W, ZHU X, ZOU C, YIN J, CHERN M, ZHOU X, YING H, JIANG X, LI Y,. Identification and characterization of rice blast resistance geneby a combination of transcriptomic profiling and genome analysis. Journal of Genetics and Genomics, 2018, 45(12): 663-672.
[10] 马军韬, 李文华, 张国民, 王永力, 张丽艳, 孟文凯. 水直播条件下黑龙江省不同稻区稻瘟病菌致病性分析. 植物保护学报, 2020, 47(5): 987-996.
MA J T, LI W H, ZHANG G M, WANG Y L, ZHANG L Y, MENG W K. Pathogenicity offrom different rice growing regions of Heilongjiang Province under water direct seeding. Journal of Plant Protection, 2020, 47(5): 987-996. (in Chinese)
[11] 田珂, 杨武, 李泌, 徐鑫, 刘新琼, 刘学群, 王春台. 鄂西南地区2012-2014年稻瘟病菌致病性变化分析. 华中农业大学学报, 2017, 36(5): 10-14.
Tian K, YANG W, LI M, XU X, LIU X Q, LIU X Q, WANG C T. Pathogenicity changes ofin south-western Hubei Province during 2012-2014. Journal of Huazhong Agricultural University, 2017, 36(5): 10-14.(in Chinese)
[12] 马继琼, 孙一丁, 杨奕, 李进斌, 许明辉. 适用于云南粳稻种植区的稻瘟病抗性基因分析. 分子植物育种, 2021, 19(5): 1556-1568.
MA J Q, SUN Y D, YANG Y, LI J B, XU M H. Analysis of rice blast resistance genes suitable forrice planting areas in Yunnan. Molecular Plant Breeding, 2021, 19(5): 1556-1568. (in Chinese)
[13] 张姝, 钟雪莲, 乔贵艳, 沈丽, 周天云, 彭云良. 川渝黔稻瘟病菌毒性的区域分化研究. 西南农业学报, 2017, 30(2): 359-365.
ZHANG S, ZHONG X L, QIAO G Y, SHEN L, ZHOU T Y, PENG Y L. Difference in virulence offrom Sichuan, Chongqing and Guizhou. Southwest China Journal of Agricultural Sciences, 2017, 30(2): 359-365. (in Chinese)
[14] SHI J, LI D, LI Y, LI X, GUO X, LUO Y, LU Y, ZHANG Q, XU Y, FAN J, HUANG F, WANG W. Identification of rice blast resistance genes in the elite hybrid rice restorer line Yahui 2115. Genome, 2015, 58(3): 91-97.
[15] HASSAN B, PENG Y T, LI S, YIN X X, CHEN C, GULZAR F, ZHOU S X, PU M, JI Y P, WANG Y P, ZHAO W, HUANG F, PENG Y L, ZHAO Z X, WANG W M. Identification of the blast resistance genes in three elite restorer lines of hybrid rice. Phytopathology Research, 2022, 4(1): 15.
[16] Tsunematsu H, Yanoria M J T, Ebron L A, Hayashi N, Ando I, Kato H, Imbe T, Khush G S. Development of monogenic lines of rice for blast resistance. Breeding Science, 2000, 50(3): 229-234.
[17] 陈嘉林, 余敏祥, 何旎清, 黄凤凰, 杨德卫. 稻瘟病抗病基因分子标记的开发与育种利用. 分子植物育种, http://kns.cnki.net/kcms/ detail/46.1068.S.20220112.1946.012.html.
Chen J L, Yu M X, He N Q, Huang F H, Yang D W. Development and breeding application of molecular markers for rice blast resistance genes. Molecular Plant Breeding, http://kns.cnki.net/ kcms/detail/46.1068.S.20220112.1946.012.html. (in Chinese)
[18] HAYASHI K, YOSHIDA H, ASHIKAWA I. Development of PCR- based allele-specific and InDel marker sets for nine rice blast resistance genes. Theoretical and Applied Genetics, 2006, 113(2): 251-260.
[19] 华丽霞, 汪文娟, 陈深, 汪聪颖, 曾烈先, 杨健源, 朱小源, 苏菁. 抗稻瘟病基因特异性分子标记的开发. 中国水稻科学, 2015, 29(3): 305-310.
HUA L X, WANG W J, CHEN S, WANG C Y, ZENG L X, YANG J Y, ZHU X Y, SU J. Development of specific DNA markers for detecting the rice blast resistance gene alleles. Chinese Journal of Rice Science, 2015, 29(3): 305-310. (in Chinese)
[20] 杨立明, 纪剑辉, 周颖君, 方继朝, 刘永锋, 罗玉明. 水稻稻瘟病抗性基因-InDel标记的开发与评价. 分子植物育种, 2017, 15(2): 594-598.
YANG L M, JI J H, ZHOU Y J, FANG J C, LIU Y F, LUO Y M. The development of the rice blast resistance genes-InDel markers and its evaluation in application. Molecular Plant Breeding, 2017, 15(2): 594-598. (in Chinese)
[21] 王芳权, 陈智慧, 许扬, 王军, 李文奇, 范方军, 陈丽琴, 陶亚军, 仲维功, 杨杰. 水稻广谱抗稻瘟病基因功能标记的开发及应用. 中国农业科学, 2019, 52(6): 955-967.doi: 10.3864/j.issn.0571- 1752.2019.06.001.
WANG F Q, CHEN Z H, XU Y, WANG J, LI W Q, FAN F J, CHEN L Q, TAO Y J, ZHONG W G, YANG J. Development and application of the functional marker for the broad-spectrum blast resistance genein rice. Scientia Agricultura Sinica, 2019, 52(6): 955-967. doi: 10.3864/j.issn.0571-1752.2019.06.001. (in Chinese)
[22] 谢旺有, 陈锦文, 谢少和, 陈惠清, 王天生, 余文昌, 侯凯强. 抗稻瘟病基因导入籼稻恢复系泉恢039的应用研究. 福建农业学报, 2022, 37(10): 1266-1274.
XIE W Y, CHEN J W, XIE S H, CHEN H Q, WANG T S, YU W C, HOU K Q. Introduction of blast resistantrice restorer line Quanhui 039. Fujian Journal of Agricultural Sciences, 2022, 37(10): 1266-1274. (in Chinese)
[23] 柳絮, 张华, 宣宁, 李平波, 张梦琦, 姚方印. 水稻抗虫()和抗稻瘟病(,)材料的创制. 山东农业科学, 2021, 53(2): 89-93.
LIU X, ZHANG H, XUAN N, LI P B, ZHANG M Q, YAO F Y. Breeding of new rice varieties with rice blast and insect resistances. Shandong Agricultural Sciences, 2021, 53(2): 89-93. (in Chinese)
[24] 王哉, 蒋英健, 谢留杰, 王敏天, 黄丛林, 陈嘉乐, 阮文晓. 分子标记辅助选育含抗稻瘟病基因的水稻新品系. 浙江农业科学, 2023, 64(8): 1945-1948.
WANG Z, JIANG Y J, XIE L J, WANG M T, HUANG C L, CHEN J L, RUAN W X. The new rice lines containing rice blast resistance genewere selected by molecular marker assisted breeding. Journal of Zhejiang Agricultural Sciences, 2023, 64(8): 1945-1948. (in Chinese)
[25] 于连鹏. 黑龙江省主栽水稻品种、和抗瘟基因检测和抗性评价[D]. 大庆: 黑龙江八一农垦大学, 2017.
YU L P.,andgenes detection and blast resistance evaluation of main rice varieties in Heilongjiang Province[D]. Daqing: Heilongjiang Bayi Agricultural University, 2017. (in Chinese)
[26] 张亚玲, 高清, 赵羽涵, 刘瑞, 付忠举, 李雪, 孙宇佳, 靳学慧. 黑龙江省水稻种质稻瘟病抗性评价及抗瘟基因结构分析. 中国农业科学, 2022, 55(4): 625-645. doi: 10.3864/j.issn.0578-1752.2022.04.001.
ZHANG Y L, GAO Q, ZHAO Y H, LIU R, FU Z J, LI X, SUN Y J, JIN X H. Evaluation of rice blast resistance and genetic structure analysis of rice germplasm in Heilongjiang Province. Scientia Agricultura Sinica, 2022, 55(4): 625-645. doi: 10.3864/j.issn.0578-1752.2022.04.001. (in Chinese)
[27] 陈晴晴, 杨雪, 张爱芳. 长江中下游区试水稻品种稻瘟病抗性评价及抗性基因检测. 南方农业学报, 2022, 53(1): 21-28.
CHEN Q Q, YANG X, ZHANG A F. Evaluation of rice blast resistance and detection of resistance genes of rice varieties in regional trials in the middle and lower reaches of Yangtze River. Journal of Southern Agriculture, 2022, 53(1): 21-28. (in Chinese)
[28] 董丽英, 刘树芳, 田维逵, 周伍民, 张先闻, 李迅东, 杨勤忠. 云南省稻瘟病菌群体的致病性分析及交配型分布. 植物保护学报, 2023, 50(2): 316-324.
DONG L Y, LIU S F, TIAN W K, ZHOU W M, ZHANG X W, LI X D, YANG Q Z. Pathogenicity and mating type of rice blast fungusisolates in Yunnan Province. Journal of Plant Protection, 2023, 50(2): 316-324. (in Chinese)
[29] 桑世飞, 王亚男, 王君怡, 曹梦雨, 孙晓涵, 姬生栋. 9个抗稻瘟病基因在291份水稻种质资源中的分布及组合特征. 河南农业科学, 2022, 51(12): 19-27.
SANG S F, WANG Y N, WANG J Y, CAO M Y, SUN X H, JI S D. Distribution and combination features of nine blast resistant genes in 291 rice germplasm resources. Journal of Henan Agricultural Sciences, 2022, 51(12): 19-27. (in Chinese)
[30] 何弯弯, 王健康, 丁成伟, 郭荣良, 吴玉玲, 王友霜, 赵轶鹏, 胡婷婷.、、、和在粳稻品种(系)中的分布及对穗颈瘟的抗性. 西南农业学报, 2022, 35(3): 497-502.
HE W W, WANG J K, DING C W, GUO R L, WU Y L, WANG Y S, ZHAO Y P, HU T T. Distribution of,,,andgenes inrice varieties (lines) and resistance to panicle blast. Southwest China Journal of Agricultural Sciences, 2022, 35(3): 497-502. (in Chinese)
[31] 沈浙南, 时焕斌, 邱结华, 王聪聪, 孟帅, 寇艳君. 江苏省212份水稻材料中抗稻瘟病基因的情况分析. 分子植物育种, 2023, 21(13): 4344-4351.
SHEN Z N, SHI H B, QIU J H, WANG C C, MENG S, KOU Y J. Analysis of rice blast resistance genein 212 rice materials in Jiangsu Province. Molecular Plant Breeding, 2023, 21(13): 4344-4351. (in Chinese)
[32] 刘军化, 黄成志, 蒋静玥, 吕直文, 刘忠贤, 蔡钟亚, 雷树凡. 87份水稻材料中抗稻瘟病基因的分子检测. 西南农业学报, 2022, 35(9): 2030-2037.
LIU J H, HUANG C Z, JIANG J Y, LÜ Z W, LIU Z X, CAI Z Y, LEI S F. Molecular detection of blast resistance genes in 87 rice materials. Southwest China Journal of Agricultural Sciences, 2022, 35(9): 2030-2037. (in Chinese)
Development and application of specific molecular markers for six homologous rice blast resistance genes inLocus of rice
YANG Hao1, HUANG YanYan1, YI ChunLin1, SHI Jun2, TAN ChuTian1, REN WenRui1, WANG WenMing1
1State Key Laboratory of Crop Gene Exploration and Utilization in Southwest China, Sichuan Agricultural University, Chengdu 611130;2Institute of Rice Research, Mianyang Academy of Agricultural Sciences/Crop Characteristic Resources Creation and Utilization Key Laboratory of Sichuan Province, Mianyang 621023, Sichuan
【Objective】Theresistance gene locus, conferring a broad-spectrum resistance against, is consist by several tandem homologous genes. over 10 resistance genes have been cloned from this gene locus. This study aims to clarify thegene composition atlocus in rice resource materials and promote the application of those genes in rice resistance breeding.【Method】Comparing the DNA sequence of clonedgenes atlocus, the specific nucleotide polymorphism sites were screened as the candidate sites. Subsequently, eachgene was blasted with 155 rice genomes in the database of Rice Resource Center. The most specific nucleotide polymorphism sites were picked out from the candidate site in each gene to develop primer pair of molecular markers. The PCR product of primer pairs was used to mark indicatedgene in tested rice materials via parameter optimization. To verify the results, thegenes were cloned from indicated rice variety randomly and examined by Sanger sequencing, or analyzed thegenes from the genome database if the genome sequence of indicated rice variety exists in Rice Resource Center. Thegenes inlocus have high homology, which cause same specific nucleotide polymorphism sites existing in differentgenes. Therefore, somegenes are hardly identified by one molecular marker. For this case, several molecular markers were employed to identify the indicatedgene simultaneously. Moreover, some specific nucleotide polymorphism sites are single nucleotide polymorphism (SNP), in where the primers of molecular markers have a mismatched base. In order to improve the specificity of PCR amplification, the adjacent base of SNP was mutated to generate two mismatched bases at 3′ site of primer.【Result】Finally, the valid molecular markers were developed for eachgene and identified 32.09% tested materials containinggenes atlocus.,,,,andare present in 1, 7, 8, 14, 23 and 33 tested materials, respectively. Theonly presents in monogenic line but not in rice parent lines. The other genes are usually present in two or more gene combinations in rice parent lines. Theoften presents in pair withand, and presents alone in three rice parents, Chenghui 993, HR2168 and Mianhui 365. Yuhui 38 contains the mostgenes atlocus, including,,and. Chuangu B, Chuannong 4B, Neixiang 6B and Shuang 1B contain,and. Qianxiang 654B containsand.【Conclusion】This study successfully developed specific molecular markers for six homologous rice blast resistance genes inlocus and identified thegene composition inlocus for 110 rice parent lines that used in rice breeding in Sichuan basin. It also discovered different types ofgenes combination atlocus and provided a clear reference for choosing the resistance source in rice breeding.
rice; rice blast;locus; homologous gene; molecular marker

2023-07-23;
2023-09-06
国家自然科学基金(U19A2033,31901839)、四川省青年科技创新研究团队(2022JDTD0023)
杨好,E-mail:2365657091@qq.com。黄衍焱,E-mail:h1985yy@163.com。杨好和黄衍焱为同等贡献作者。通信作者王文明,E-mail:j316wenmingwang@163.com
(责任编辑 岳梅)
