桃生长势相关基因PpSAUR73功能鉴定
2023-11-18杨丽曹洪波张学英翟含含李辛淼彭佳伟田义陈海江
杨丽,曹洪波,张学英,翟含含,李辛淼,彭佳伟,田义,陈海江
桃生长势相关基因功能鉴定
1河北农业大学园艺学院,河北保定 071000;2喀什职业技术学院,新疆喀什 844000;3河北农业大学山区研究所/河北省山区农业技术创新中心/国家北方山区农业工程技术研究中心,河北保定 071001
【目的】克隆桃树势相关基因,分析其对多种激素的响应,鉴定其在调控拟南芥生长势中的作用,为科学合理调控树势提供分子依据。【方法】以‘中油蟠9号’为材料进行激素处理,利用实时荧光定量分析在24 h内的动态响应;以桃品种‘久艳’为材料克隆;构建过量表达载体,将其转化拟南芥;对转基因拟南芥进行表型观察,对同时播种的转基因与野生型拟南芥进行萌芽率统计,对萌发一致的生长7 d拟南芥进行根长及下胚轴的测量,对萌发一致的拟南芥进行不同浓度的激素处理;取7 d大小的两个转基因株系及野生型拟南芥材料进行转录组测序,对差异表达基因进行功能分析、KEGG通路富集分析,并分析调控基因。【结果】能够对激素处理做出快速响应。过表达能够影响拟南芥种子萌芽,转基因拟南芥幼苗下胚轴及根长较野生型长,莲座大,整体长势好于野生型,且降低了对生长素的敏感性。过表达的转录组分析结果显示,在两个对比组中均表达的差异基因有128个,其中有84个上调基因,44个下调基因,并对20个表达量较高的差异基因进行了描述。对过表达产生的差异表达基因进行GO功能显著性富集分析,结果表明差异表达基因在细胞组分方面富集的基因最多,定位在细胞质、细胞膜、细胞器和细胞外区域。对差异表达基因进行KEGG通路富集分析,结果表明差异表达基因主要富集到苯丙氨酸生物合成通路、植物激素信号转导通路、淀粉蔗糖代谢通路等代谢通路。在苯丙氨酸生物合成通路中,能够调控编码过氧化物酶基因AT1G05260、AT3G01190、AT3G32980、AT5G15180表达上调,过氧化物酶与木质素合成相关,木质素含量与植株长势呈显著相关,暗示过表达可能参与调控拟南芥木质素合成从而调控长势。在植物激素信号转导通路中,生长素信号转导中的一些生长素响应基因等表达下调,脱落酸信号转导途径中磷酸酶蛋白表达上调,而脱落酸信号通路基因表达下调。能够调控拟南芥长势,参与多种激素信号转导。【结论】能够快速对激素做出响应,且能够调控转基因拟南芥长势;过量表达导致的差异基因主要富集到苯丙氨酸生物合成通路、植物激素信号转导通路、淀粉蔗糖代谢通路等代谢通路;调控IAA、ABA信号转导,推测其在桃树的生长发育过程中起到了重要作用。
桃;;基因表达;激素处理;信号转导
0 引言
【研究意义】桃(L.)属于蔷薇科(Rosaceae)植物,落叶小乔木。桃树营养生长旺盛,新梢生长量大,一年可形成多次副梢,生产中经常通过修剪以及喷施生长调节剂对树势进行控制,否则便会造成树冠郁闭,影响树体的通风透光,从而影响果实品质。因此,挖掘桃树势生长调控基因,明确树势调控机制十分必要。【前人研究进展】生长素调节植物生长发育,早期生长素反应基因介导其对植物生长发育的基因组效应。()于1987年被发现,是最大的早期生长素响应基因家族[1]。在前人提出的生长素介导细胞扩增的酸生长机制中发挥核心作用[2]。尽管早期发现了它们的生长素反应性,但其功能和作用方式长期以来仍有待解析[3]。近年来,人们对在动态调控生长和适应性调控生长中的重要性以及SAUR蛋白作用的分子机制日益了解。目前,该基因家族已在拟南芥、棉花、茄子及葡萄等多个物种中得到鉴定[4-5]。一些主要在下胚轴或其他伸长组织中表达,在调节细胞伸长中发挥重要作用[6-7]。能够通过负调节PP2C蛋白磷酸酶来调节质膜H+-ATP酶活性,从而促进细胞扩增[7]。在拟南芥内胚层中表达,参与侧根发育,过表达的拟南芥会出现下胚轴伸长,影响侧根发育,使花瓣膨胀和花序茎发生扭曲[8]。此外,一些还可以对生长素的合成运输产生负调控,过表达的拟南芥幼苗下胚轴变短且直,并且可以通过施加外源生长素缓解这种症状[9]。在水稻中,过表达对生长素的生物合成和运输产生负调控,降低生长素的转运活性[10],这表明不同的SAUR蛋白可能具有相反的功能。还可以受到大量其他上游因素的调控,从而根据内部和环境信号动态调节生长[11-15]。此外,一些在叶片衰老、细胞分裂及响应干旱、低温、病虫害等过程中发挥作用[8-10]。【本研究切入点】桃是原产我国的一种极为重要的大宗水果,但其树体偏大,普遍存在树冠郁闭不便管理的问题,大量使用人工导致生产成本增加[16-17]。笔者课题组前期在对不同树势的桃树进行转录组数据分析时获得了与调控树势相关的18个关键基因,并对其中的进行了功能鉴定,发现该基因过量表达促进器官伸长[18]。为进一步探究其余的差异基因是否也存在相似或者相反的表型,对转录组中筛选得到的与树势相关的(XM_007201374.1)进行功能鉴定,探究过表达拟南芥是否具有调控长势的功能,并观察过表达拟南芥对激素的响应情况。【拟解决的关键问题】克隆桃并稳定遗传转化拟南芥,进行表型观察及转录组测序分析,结合表达对激素的响应,鉴定的基因功能,为调控桃树体长势提供分子依据。
1 材料与方法
1.1 试验材料
试验于2022年在河北农业大学园艺学院、保定市满城区国家良种基地进行。
本试验用于克隆基因的RNA提取自桃品种‘久艳’,砧木为‘毛桃’,采用常规田间管理。用于田间激素处理的RNA提取自桃品种‘中油蟠9号’,由保定市满城区国家桃良种基地提供。
本研究使用的拟南芥为哥伦比亚型(Col-0)。
1.2 方法
1.2.1 构建桃与拟南芥长势相关蛋白系统发育树 通过文献获得拟南芥()SAUR家族与长势相关的蛋白序列信息,将桃PpSAUR5、PpSAUR73和拟南芥SAUR蛋白序列置于MEGA7软件中,使用ClustalW程序进行多重序列比对,将得到的结构采用邻接法(Neighbor-Joining,NJ),参数默认,Bootstrap邻接值设置为1 000,构建系统发育树。
1.2.2 桃对激素的瞬时响应 于2022年9月20日,选取长至20 cm的‘中油蟠9号’嫩梢对其进行生长调节剂处理。试验以喷施0.7%无水乙醇溶液为对照,分别向叶面喷施100 mg∙L-1吲哚-3-乙酸IAA、100 mg∙L-1赤霉素(GA)、50 mg∙L-1邻氨甲酰苯甲酸(NPA)、50 mg∙L-1的乙烯利(ETH)和100 mg∙L-1的脱落酸(ABA)。取样时间为处理后0 min、3 min、5 min、1 h、3 h和24 h。样品采集部位为叶片,直接在液氮中冷冻后储存于-80 ℃。
利用OminiPlant RNA Kit(康为世纪,北京,CW2598S)提取样品RNA,采用HiFiScript gDNA Removal RT MasterMix(康为世纪,北京,CW2020M)试剂盒进行反转录,合成cDNA第一链,用于quantitative real-time PCR(qRT-PCR)分析,仪器为LightCycler®96(Roche,德国)。选取桃树(Prupe.6G163400)作为内参基因[19],每个样品3次重复,用2-ΔΔCT方法计算基因相对表达量[20]。反应条件设定:预变性95 ℃、300 s;95 ℃、10 s变性,58 ℃、10 s退火,72 ℃、10 s后延伸,循环45次。本研究所需引物见表1。
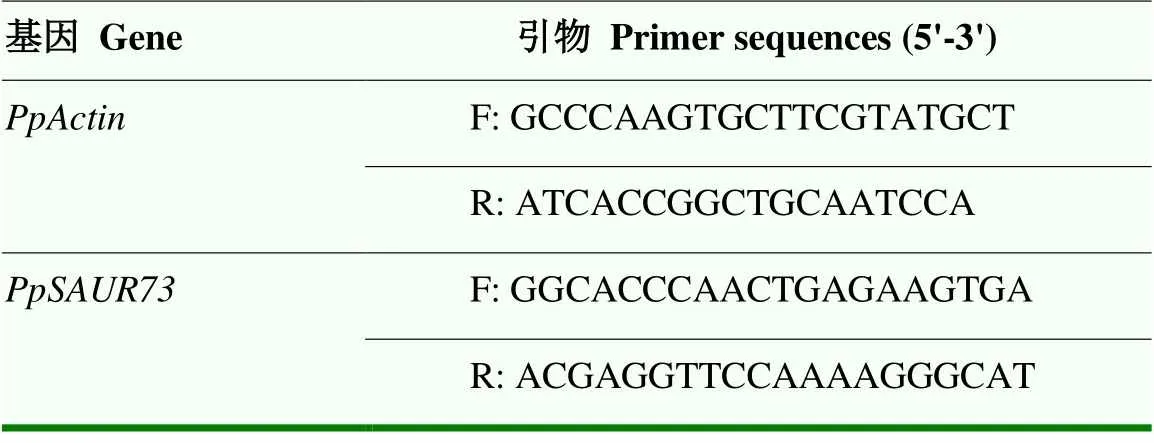
表1 qRT-PCR验证引物序列
1.2.3 桃转基因拟南芥表型观察 构建PRI101-FLAG-过表达载体,利用花序浸染法转化拟南芥[18],获得转基因株系。在同一四分板MS培养基播种野生型与3个转基因株系拟南芥种子,每个株系播种20粒种子,4 ℃春化2 d,之后放置在组培室培养,每12 h进行一次萌发率统计(种子露出白色胚根即为成功萌发),试验重复3次。萌发率(%)=萌发种子数/培养皿中全部种子数×100%。游标卡尺统计根长、下胚轴长度。
1.2.4转基因拟南芥转录组测序分析 选取MS培养基中生长7 d的野生型与转基因拟南芥幼苗用液氮速冻,干冰运输至百迈客公司,文库质检合格后,使用Illumina NovaSeq6000测序平台进行测序,利用百迈客云平台进行数据分析。
2 结果
2.1 桃PpSAUR73与拟南芥长势相关蛋白的系统发育树
为进一步研究PpSAUR73与拟南芥的系统进化关系,采用MEGA7软件构建了桃、拟南芥长势相关的SAUR蛋白系统进化树。PpSAUR73与AtSAUR14、15、19蛋白的关系较近(图1)。
2.2 桃PpSAUR73对激素的瞬时响应
为了探究桃对各种激素的响应情况,对‘中油蟠9号’嫩梢进行激素喷施并取样,定量分析的表达量。结果显示,桃能在喷施IAA后3 min内出现快速响应,表达量是对照(0 min)的3.1倍,在喷施后3 h时表达量达到对照的14.9倍,在24 h时为对照的4.6倍;在喷施NPA后,的表达量呈先下降再上升趋势,在1 h时的表达量仅为对照的0.54倍,24 h时上升到与对照持平水平;在喷施ABA后,表达量整体呈上升趋势,最高在3 h时,为对照的3.4倍;在喷施乙烯后,的表达量呈上升下降再上升下降的趋势,1 h时表达量最高,为对照的2.4倍(图2)。
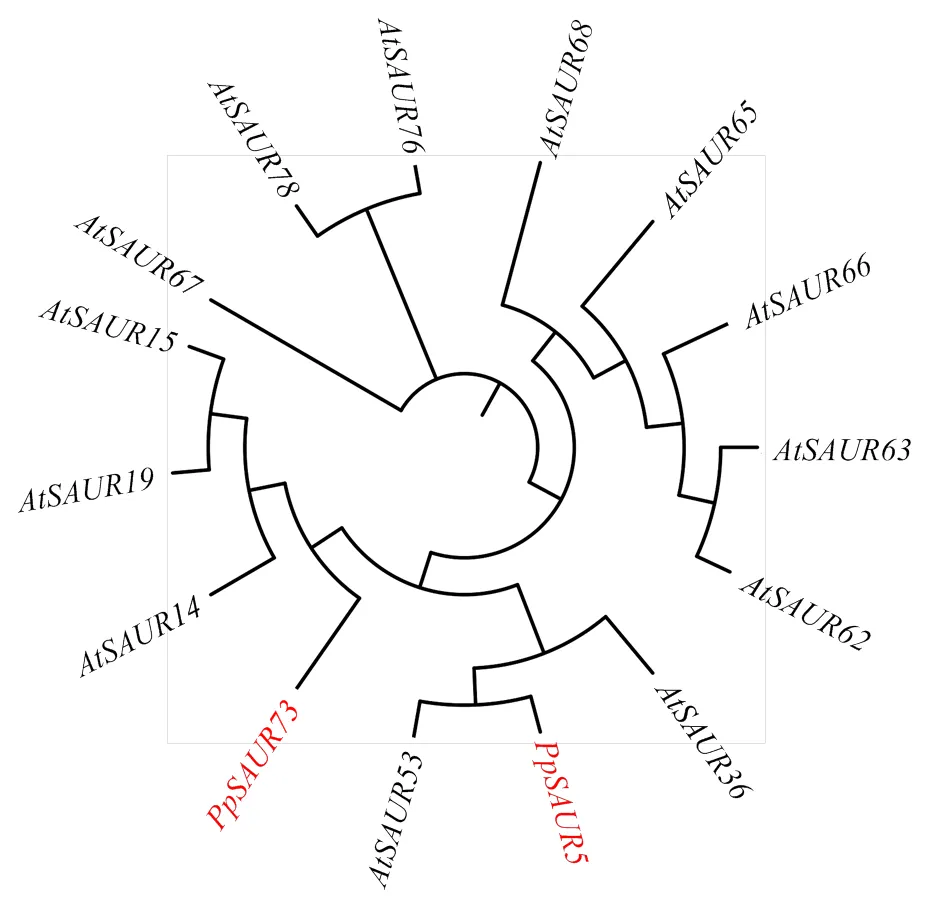
图1 PpSAUR73与拟南芥长势相关蛋白的系统发育树
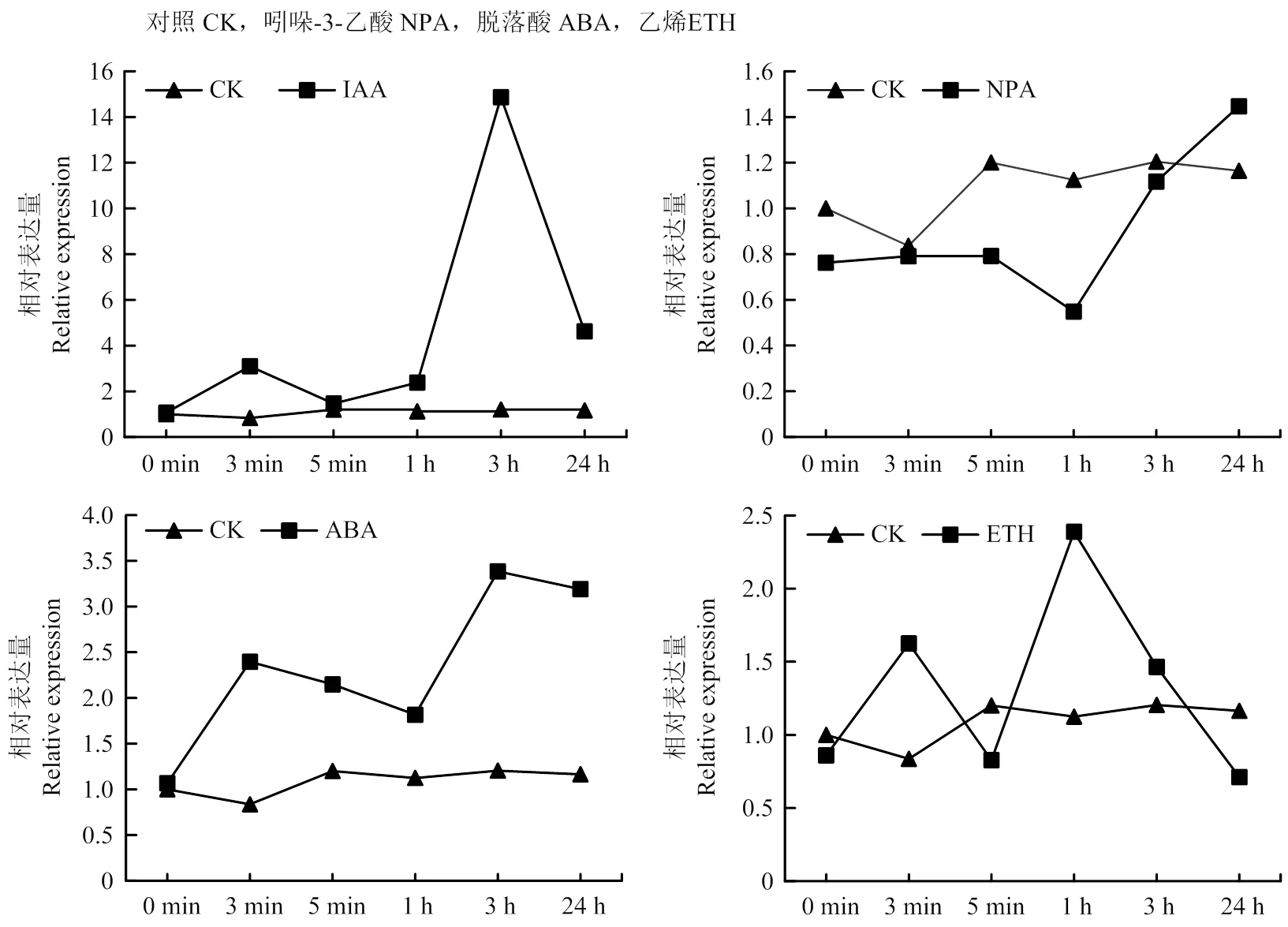
图2 PpSAUR73表达对激素的响应分析
2.3 PpSAUR73转基因拟南芥表型观察
为探究的基因功能,构建了过表达载体,遗传转化拟南芥,获得转基因拟南芥。结果显示,种子萌芽率在24 h时出现显著性差异,野生型拟南芥的萌芽率仅45%,但转基因拟南芥能够达到70%,甚至96%(图3)。表明过量表达能够影响种子萌芽。
对萌发7 d的转基因拟南芥的根长、下胚轴进行观察统计。结果显示,相较于野生型,转基因株系的根长显著增加,分别增加了32%、43%和43%;下胚轴长度分别显著增加60%、59%和74%(图4),表明也具有促进器官伸长的功能。此外,过表达的拟南芥较野生型的莲座大,一直到结果期,过表达的拟南芥都表现出长势显著好于野生型。

Col-0:野生型;SAUR73-1、SAUR73-14、SAUR73-2:转基因拟南芥株系。不同小写字母表示差异显著(P<0.05)。下同

图4 野生型及转基因拟南芥表型观察
2.4 PpSAUR73过表达影响拟南芥根系对IAA和GA的敏感性
为了探究对激素的响应,对野生型与过表达拟南芥进行了不同浓度的IAA、GA及NPA处理(图5)。结果显示,与野生型相比,转基因株系对IAA的敏感性大幅度降低。与Col-0相比,50 nmol∙L-1IAA对3个转基因拟南芥根长生长的抑制率分别降低了11%、4%和6%;75 nmol∙L-1IAA对转基因拟南芥根长生长的抑制率分别降低约8%、6%和4%;100 nmol∙L-1IAA对转基因拟南芥根长生长的抑制率分别降低约7%、7%和6%,随着处理浓度增大,IAA对各拟南芥根系的抑制作用逐渐增大。与野生型相比,转基因株系对GA与NPA的敏感性明显增加。50 μmol∙L-1的GA对各拟南芥根长生长的抑制差异并不显著,均约为23%;100 μmol∙L-1GA对不同拟南芥根长生长的抑制作用存在显著性差异,分别约为28%、36%、38%和47%;150 μmol∙L-1GA对拟南芥根长生长的抑制作用更加明显,抑制率分别约为46%、53%、55%和57%。10和20 μmol∙L-1NPA对转基因拟南芥根长生长的抑制率均显著高于野生型。由此可知,过量表达降低了植株对IAA的敏感性,提高了对GA与NPA敏感性。
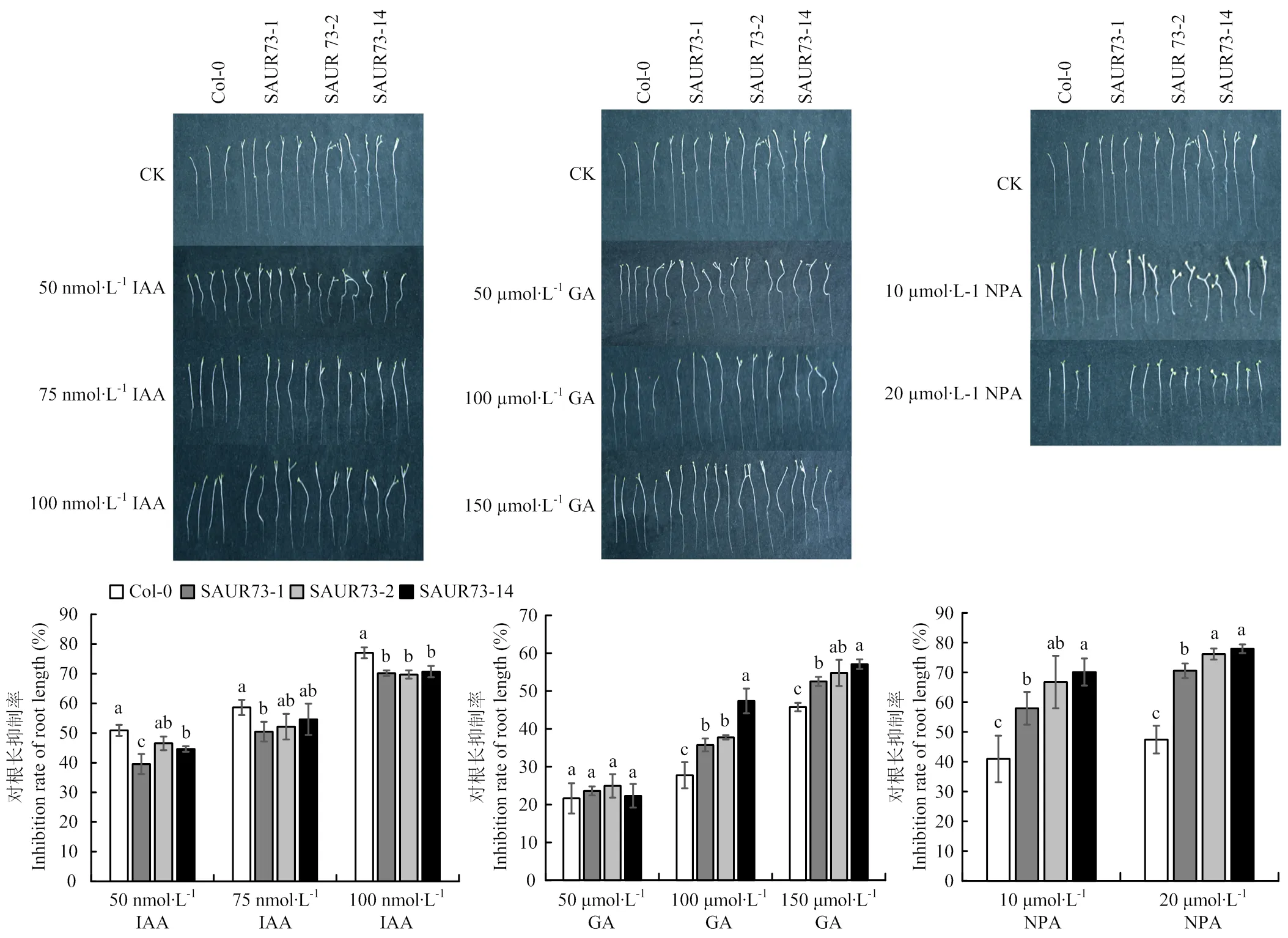
图5 IAA、GA及NPA对拟南芥根长生长的影响
2.5 转录组数据的实时荧光定量PCR验证
对野生型(CK)及SAUR73-1、SAUR73-14两个转基因拟南芥株系进行转录组测定,从转录组中筛选出9个基因进行qRT-PCR验证(图6)。转录组测得的FPKM值与这9个基因相对表达量的变化趋势一致,从而验证了RNA-seq数据的可靠性。
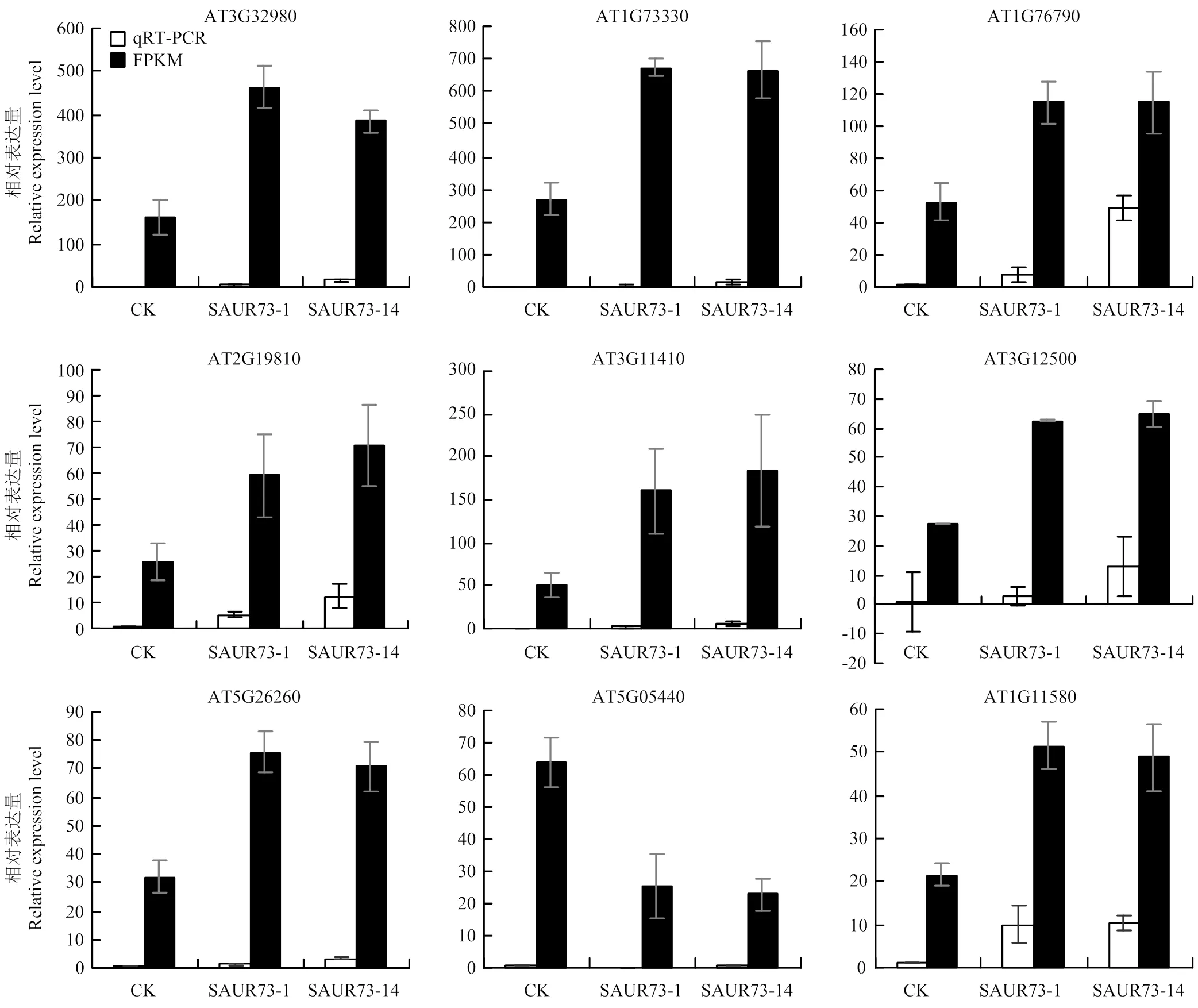
图6 差异表达基因的qRT-PCR验证
2.6 差异基因表达分析
将SAUR73-1、SAUR73-14分别与CK进行差异比较分析(图7),显示在两个对比组中均表达的差异基因有128个,其中有84个上调基因,44个下调基因,对这些差异基因的表达绘制热图,筛选出20个表达量相对高的差异基因(表2)。
2.7 差异基因功能分析
将CK与SAUR73-1、SAUR73-14的共同差异基因进行GO功能显著性富集分析。结果表明,差异表达基因在细胞组分方面富集的基因最多,定位在细胞质、细胞膜、细胞器和细胞外区域等;其次是生物学过程方面富集的基因,与细胞过程、代谢过程、生物调节和对刺激的反应等有关;在分子功能方面富集的基因较少,与其他物质结合、催化活性、核酸结合转录因子活性、结构分子活性、抗氧化活性以及分子功能调节剂等有关(图8-A)。对差异表达基因进行KEGG通路富集分析(图8-B),结果显示,差异基因主要富集到苯丙氨酸生物合成通路、植物激素信号转导通路、淀粉蔗糖代谢通路、MAPK信号通路。其中,苯丙氨酸生物合成通路与植物激素信号转导通路可能与生长势密切相关。
2.8 苯丙氨酸代谢合成途径
对苯丙氨酸代谢合成通路进行分析(图9),挖掘差异表达基因(表3),发现差异基因中AT1G05260、AT3G01190、AT3G32980、AT5G15180 4个编码过氧化物酶基因表达上调,而过氧化物酶在木质素的合成过程中发挥关键作用[21],推测过表达可能参与调控木质素合成。

A:样品间差异表达基因维恩图;B:共同差异表达基因热图
2.9 植物激素信号转导通路
KEGG通路富集显示(图10),过表达拟南芥的差异基因在生长素信号转导、脱落酸信号转导通路富集。对上述通路中的差异表达基因进行分析,发现过量表达下调生长素的响应基因AT1G16510()、AT1G56150()、AT1G75580()、AT3G12830()、AT4G34770()表达,其中基因AT1G16510()与AT1G75580()还可对植物的衰老进行调控,AT1G56150()表达对ABA信号传导和叶绿体的功能状态进行调控[22]。在脱落酸信号转导通路,过量表达上调磷酸酶蛋白AT3G11410()表达,下调脱落酸信号通路基因AT5G05440(),编码ABA信号传导负调节器,而能够调节丝氨酸、苏氨酸磷酸酶活性。尽管是生长素响应基因,但对脱落酸通路有调控作用,并且田间激素喷施试验也发现对脱落酸有响应。

表2 SAUR73部分共同差异基因的信息描述
3 讨论
3.1 PpSAUR73对激素的响应
广泛参与细胞生理发育过程,涉及植物生长发育的激素和环境调控过程,而植物激素之间也存在相互作用,互相影响[23]。作为生长素响应基因,拟南芥能够对IAA作出快速响应[8],在IAA处理30 min后,—都发生两到三倍的上调[24],过表达—能够对ETH作出响应,突变体对ETH的敏感性降低[25]。本研究通过对田间桃树进行激素喷施,发现对IAA能够做出快速响应,并且发现其也能对ABA、ETH作出响应。转拟南芥影响种子萌芽,而种子萌芽受到赤霉素的调控[26],但转录组数据中并没有富集到赤霉素信号转导,推测是在种子萌芽期间影响赤霉素水平,但在幼苗及成苗以后不影响。以上均说明可能参与多种激素信号转导过程。拟南芥种子的萌发、根长、下胚轴长度等与激素水平有关,且激素在调控树势生长中也具有重要的作用,有研究表明苹果倒贴皮和矮化中间砧可以降低韧皮部IAA水平从而使树体矮化[27];ABA水平与核桃实生苗生长势呈负相关[28];GA处理24 h后,栽培种和野生种曼陀罗种子的发芽率和幼苗的鲜质量、干质量、根长以及根表面积均显著提高[29]。本研究发现拟南芥萌发、根长、下胚轴长度都发生了变化,能够促进器官伸长,猜测该基因可能具有影响激素水平的功能,而激素是调控树体生长发育的关键因素之一,由此推测,该基因也可能通过影响激素水平而调控桃树树势。
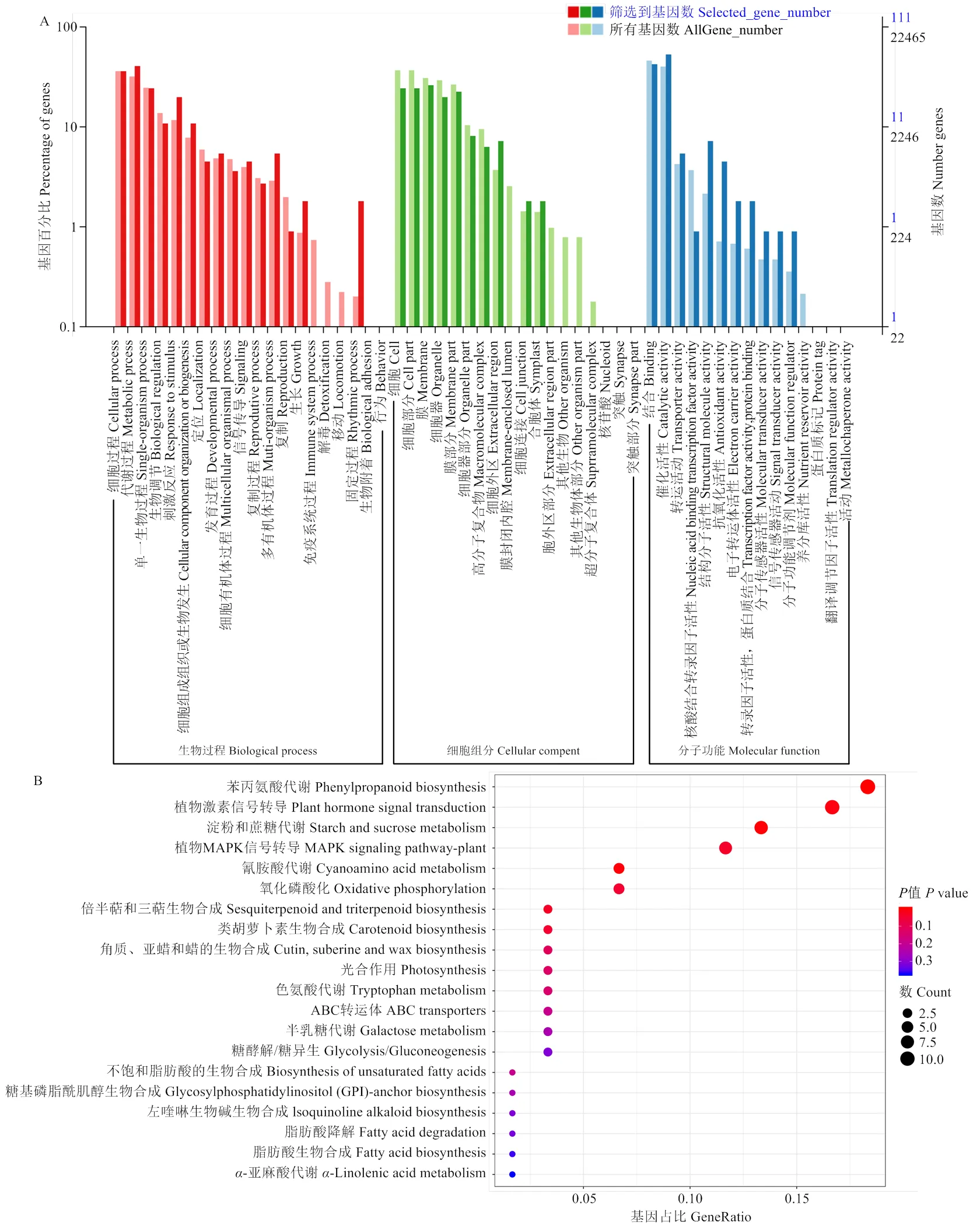
A:SAUR73共同差异表达基因的GO功能类别;B:SAUR73共同差异表达基因的KEGG代谢通路富集

表3 苯丙氨酸合成代谢通路中涉及的差异基因
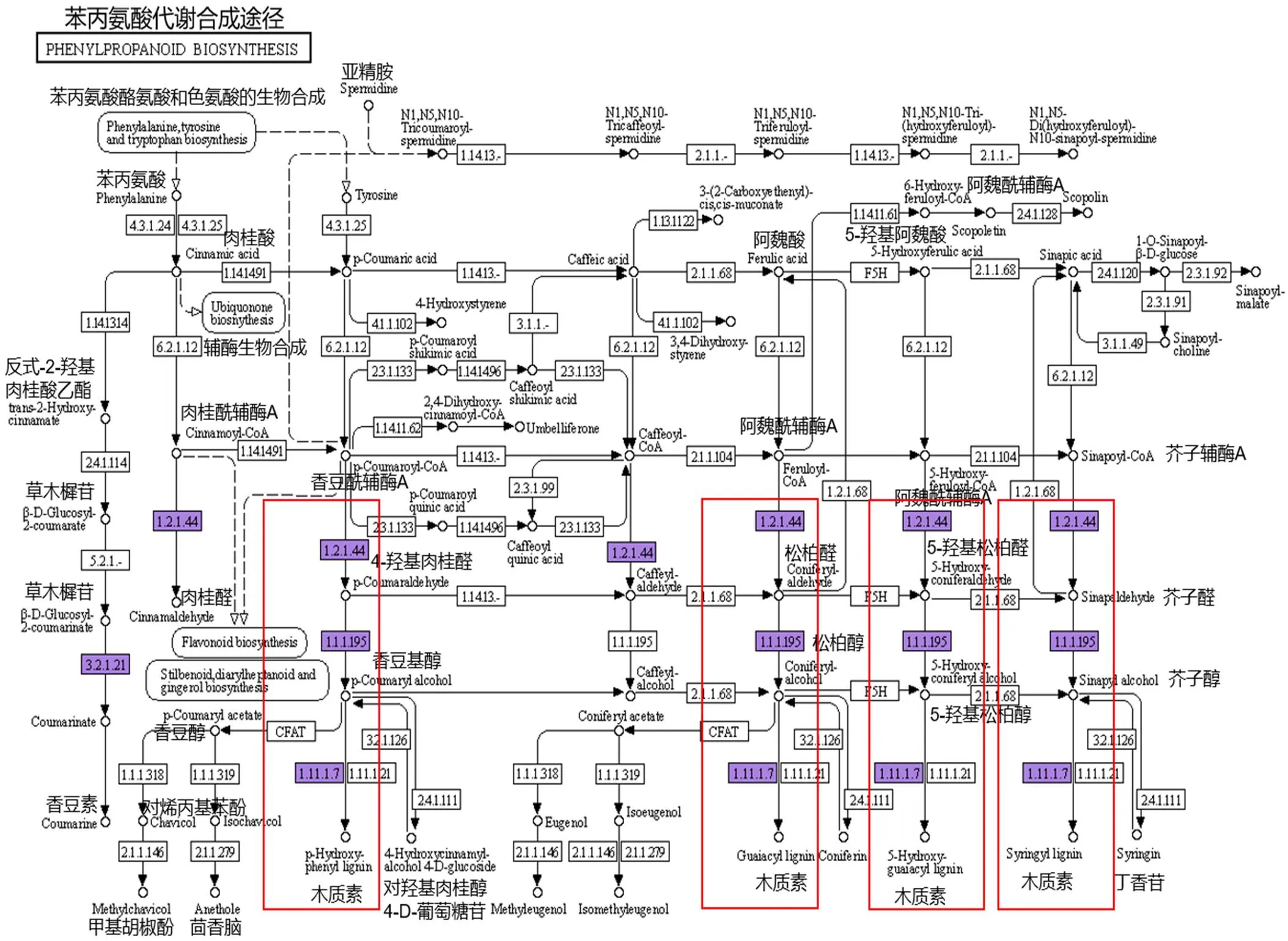
图 9 苯丙氨酸合成代谢通路图

图10 植物激素信号转导中差异表达基因聚类热图
3.2 过表达PpSAUR73使器官伸长
拟南芥中发现多个基因能够调控细胞伸长,定位在细胞质膜上,在下胚轴和根中高表达,正向调控拟南芥的生长和生长素转运,过表达使细胞扩张导致下胚轴长度增加[6,24-25,30];过表达会导致细胞与器官的伸长并且影响顶钩的发育[11];在拟南芥中发现过表达融合蛋白能够使下胚轴、雄蕊丝变长,并且发现下胚轴中积累的IAA较多[6];—通过影响拟南芥的乙烯受体信号传导并促进植物生长,使子叶、莲座叶变大,叶片表皮细胞面积变大[25];过表达—也均被发现能够促进细胞伸长[6,24,30-35]。笔者实验室前期研究也发现,过表达能够使拟南芥器官伸长[18]。与前人研究一致,本研究中的表型试验表明过表达能够使转基因拟南芥的根及下胚轴伸长、莲座变大。
3.3 转录组分析
过表达影响生长素水平、生长素极性转运和生长素通路基因的表达[6,8,24,36]。同样在本研究转录组测序中发现,过表达对IAA信号转导发生调控,下调生长素的响应基因AT1G16510()、AT1G56150()、AT1G75580()、AT3G12830()、AT4G34770()表达,其中AT1G16510()与AT1G75580()基因还可对植物的衰老进行调控。过表达上调拟南芥表达,下调的表达。由此猜测过表达也对拟南芥的IAA水平产生影响。前人研究表明,已有许多对脱落酸有反应[11],如与脱落酸信号转导相关[10,37]。同样,本研究发现能够调控IAA信号转导,且能够调控ABA信号转导,这与相似。转录组数据显示在野生型与SAUR73-1、SAUR73-14株系中均表达的差异基因有128个,在差异基因的KEGG代谢途径分析中显示,有4个编码过氧化物酶基因表达上调,而过氧化物酶在木质素的合成过程中发挥关键作用,而已有研究表明木质素水平与长势呈显著相关[38-43],这暗示很可能是通过促进木质素的合成来调控植株的生长势。前人研究发现可通过与PP2C.D磷酸酶相互作用并抑制其磷酸酶活性,使质外体酸化,细胞壁松弛,促进细胞的伸长生长[2,7,24,44-45]。本研究转录组测序结果显示,对PP2CA表达量产生了上调,但是否存在与相似的功能还需要进一步鉴定。
4 结论
本研究发现能够快速对激素做出响应,且能够调控转基因拟南芥长势;过量表达导致的差异基因主要富集到苯丙氨酸生物合成通路、植物激素信号转导通路、淀粉蔗糖代谢通路等代谢通路;调控IAA、ABA信号转导,推测其在桃树的生长发育过程中发挥重要作用,研究结果为采用分子手段控制树势提供了参考。
[1] STORTENBEKER N, BEMER M. The SAUR gene family: The plant’s toolbox for adaptation of growth and development. Journal of Experimental Botany, 2019, 70(1): 17-27.
[2] SPARTZ A K, LOR V S, REN H, OLSZEWSKI N E, MILLER N D, WU G S, SPALDING E P, GRAY W M. Constitutive expression of() in tomato confers auxin-independent hypocotyl elongation. Plant Physiology, 2017, 173(2): 1453-1462.
[3] BEMER M, VAN MOURIK H, MUIÑO J M, FERRÁNDIZ C, KAUFMANN K, ANGENENT G C. FRUITFULL controls SAUR10 expression and regulatesgrowth and architecture. Journal of Experimental Botany, 2017, 68(13): 3391-3403.
[4] LI X H, LIU G Y, GENG Y H, WU M, PEI W F, ZHAI H H, ZANG X S, LI X L, ZHANG J F, YU S X, YU J W. A genome-wide analysis of the small auxin-up RNA (SAUR) gene family in cotton. BMC Genomics, 2017, 18(1): 815.
[5] WU J, LIU S Y, HE Y J, GUAN X Y, ZHU X F, CHENG L, WANG J, LU G. Genome-wide analysis of SAUR gene family in Solanaceae species. Gene, 2012, 509(1): 38-50.
[6] CHAE K, ISAACS C G, REEVES P H, MALONEY G S, MUDAY G K, NAGPAL P, REED J W.SMALL AUXIN UP RNA63 promotes hypocotyl and stamen filament elongation. The Plant Journal, 2012, 71(4): 684-697.
[7] SPARTZ A K, REN H, PARK M Y, GRANDT K N, LEE S H, MURPHY A S, SUSSMAN M R, OVERVOORDE P J, GRAY W M. SAUR inhibition of PP2C-D phosphatases activates plasma membrane H+-ATPases to promote cell expansion in. The Plant Cell, 2014, 26(5): 2129-2142.
[8] KONG Y Y, ZHU Y B, GAO C, SHE W J, LIN W Q, CHEN Y, HAN N, BIAN H W, ZHU M Y, WANG J H. Tissue-specific expression of SMALL AUXIN UP RNA41 differentially regulates cell expansion and root meristem patterning in. Plant and Cell Physiology, 2013, 54(4): 609-621.
[9] PARK J E, KIM Y S, YOON H K, PARK C M. Functional characterization of agene in apical hook development in. Plant Science, 2007, 172(1): 150-157.
[10] KANT S, BI Y M, ZHU T, ROTHSTEIN S J. SAUR39, a small auxin-up RNA gene, acts as a negative regulator of auxin synthesis and transport in rice. Plant Physiology, 2009, 151(2): 691-701.
[11] KODAIRA K S, QIN F, TRAN L S P, MARUYAMA K, KIDOKORO S, FUJITA Y, SHINOZAKI K, YAMAGUCHI-SHINOZAKI K.Cys2/His2 zinc-finger proteins AZF1and AZF2negatively regulate abscisic acid-repressive and auxin-inducible genes under abiotic stress conditions. Plant Physiology, 2011, 157(2): 742-756.
[12] FAVERO D S, LE K N, NEFF M M. Brassinosteroid signaling converges with suppressor of phytochrome b4-#3 to influence the expression of small auxin up rna genes and hypocotyl growth. The Plant Journal, 2017, 89(6): 1133-1145.
[13] HU W F, YAN H W, LUO S S, PAN F, WANG Y, XIANG Y. Genome-wide analysis of poplar SAUR gene family and expression profiles under cold, polyethylene glycol and indole-3-acetic acid treatments. Plant Physiology and Biochemistry, 2018, 128: 50-65.
[14] OH E, ZHU J Y, BAI M Y, ARENHART R A, SUN Y, WANG Z Y. Cell elongation is regulated through a central circuit of interacting transcription factors in thehypocotyl. eLife, 2014, 3: e03031.
[15] VAN MOURIK H, VAN DIJK A D J, STORTENBEKER N, ANGENENT G C, BEMER M. Divergent regulation ofSAUR genes: a focus on the SAUR10-clade.BMC Plant Biology, 2017, 17(1): 1-14.
[16] 翟宇杰. 不同品种桃树生长势的差异及相关基因的表达与调控[D]. 保定: 河北农业大学, 2020.
ZHAI Y J. Difference of growth potential of different peach cultivars and expression and regulation of related genes [D]. Baoding: Hebei Agricultural University, 2020. (in Chinese)
[17] 周晓雅. 多效唑抑制桃新梢生长的效应及相关分子机理研究[D]. 保定: 河北农业大学, 2020.
ZHOU X Y. Study on the effect of paclobutrazol on inhibiting the growth of peach shoots and its related molecular mechanism [D]. Baoding: Hebei Agricultural University, 2020. (in Chinese)
[18] 翟含含, 翟宇杰, 田义, 张叶, 杨丽, 温陟良, 陈海江. 桃SAUR家族基因分析及功能鉴定. 园艺学报, 2023, 50(1): 1-14.
ZHAI H H, ZHAI Y J, TIAN Y, ZHANG Y, YANG L, WEN Z L, CHEN H J. Genome-wide identification of peach SAUR gene family and characterization ofgene. Acta Horticulturae Sinica, 2023, 50(1): 1-14. (in Chinese)
[19] 王文然, 樊秀彩, 张文颖, 刘崇怀, 房经贵, 王晨. 果树赤霉素代谢与信号途径研究进展. 生物技术通报, 2017, 33(11): 1-7.
WANG W R, FAN X C, ZHANG W Y, LIU C H, FANG J G, WANG C. Study progress on gibberellin metabolism and signaling transduction pathway in fruits trees. Biotech Bulletin, 2017, 33(11): 1-7. (in Chinese)
[20] 徐献斌, 耿晓月, 李慧, 孙丽娟, 郑焕, 陶建敏. 基于转录组分析ABA促进葡萄花青苷积累相关基因. 中国农业科学, 2022, 55(1): 134-151. doi: 10.3864/j.issn.0578-1752.2022.01.012.
XU X B, GENG X Y, LI H, SUN L J, ZHENG H, TAO J M. Transcriptome analysis of genes involved in ABA-induced anthocyanin accumulation in grape. Scientia Agricultura Sinica, 2022, 55(1): 134-151. doi: 10.3864/j.issn.0578-1752.2022.01.012. (in Chinese)
[21] WEI J H, SONG Y R. Recent advances in study of lignin biosynthesis and manipulation. Journal of Integrative Plant Biology, 2001, 43(8): 771-779.
[22] ZENG Y, ZHAO T H, KERMODE A R. A conifer ABI3-interacting protein plays important roles during key transitions of the plant life cycle. Plant Physiology, 2013, 161(1): 179-195.
[23] SWARUP R, PERRY P, HAGENBEEK D, VAN DER STRAETEN D, BEEMSTER G T S, SANDBERG G, BHALERAO R, LJUNG K, BENNETT M J. Ethylene upregulates auxin biosynthesis inseedlings to enhance inhibition of root cell elongation. The Plant Cell, 2007, 19(7): 2186-2196.
[24] SPARTZ A K, LEE S H, WENGER J P, GONZALEZ N, ITOH H, INZÉ D, PEER W A, MURPHY A S, OVERVOORDE P J, GRAY W M. The saur19 subfamily of small auxin up RNA genes promote cell expansion. The Plant Journal, 2012, 70(6): 978-990.
[25] LI Z G, CHEN H W, LI Q T, TAO J J, BIAN X H, MA B, ZHANG W K, CHEN S Y, ZHANG J S. Retraction Note: Three SAUR proteins SAUR76, SAUR77 and SAUR78 promote plant growth in. Scientific Reports, 2022, 12: 1994.
[26] 吴慧娟, 刘艳, 王锡锋. 小麦矮缩病毒引起的植株矮化与赤霉素代谢的相关性分析. 中国农业科学, 2017, 50(17): 3337-3343. doi: 10.3864/j.issn.0578-1752.2017.17.008.
WU H J, LIU Y, WANG X F. Correlational analyses between dwarfing of plant height induced by wheat dwarf virus (WDV) infection and gibberellin metabolism. Scientia Agricultura Sinica, 2017, 50(17): 3337-3343. doi: 10.3864/j.issn.0578-1752.2017.17.008. (in Chinese)
[27] LOCKARD R G, SCHNEIDER G W. Stock and scion growth relationships and the dwarfing mechanism in apple//Horticultural Reviews. Wiley Online Books, 1981: 315-375.
[28] 张志华, 刘新彩, 刘彦红, 高仪, 王红霞. 核桃幼树内源激素与生长势的关系. 林业科学, 2006, 42(9): 131-133.
ZHANG Z H, LIU X C, LIU Y H, GAO Y, WANG H X. Study on relationship between hormone and growth vigor of young. Scientia Silvae Sinicae, 2006, 42(9): 131-133. (in Chinese)
[29] 闫艳华. 不同外源激素处理对曼陀罗种子萌发及幼苗生长的影响. 北方园艺, 2020(23): 115-122.
YAN Y H. Effects of different exogenous hormones on seed germination and seedling growth of. Northern Horticulture, 2020(23): 115-122. (in Chinese)
[30] STAMM P, KUMAR P P. Auxin and gibberellin responsiveSMALL AUXIN UP RNA36 regulates hypocotyl elongation in the light. Plant Cell Reports, 2013, 32(6): 759-769.
[31] HARMER S L, BROOKS C J. Growth-mediated plant movements: hidden in plain sight. Current Opinion in Plant Biology, 2018, 41: 89-94.
[32] SHIN J H, MILA I, LIU M C, RODRIGUES M A, VERNOUX T, PIRRELLO J, BOUZAYEN M. The RIN-regulated Small Auxin-Up RNA SAUR69 is involved in the unripe-to-ripe phase transition of tomato fruit via enhancement of the sensitivity to ethylene. The New Phytologist, 2019, 222(2): 820-836.
[33] WANG J J, SUN N, ZHANG F F, YU R B, CHEN H D, DENG X W, WEI N. SAUR17 and SAUR50 differentially regulate PP2C-D1 during apical hook development andopening in. The Plant Cell, 2020, 32(12): 3792-3811.
[34] HOU K, WU W, GAN S S. SAUR36, a small auxin up RNA gene, is involved in the promotion of leaf senescence in. Plant Physiology, 2013, 161(2): 1002-1009.
[35] 余洪, 易倩, 张曼曼, 朱世平, 王福生, 赵晓春. CclSAUR49基因的表达特征及对类柠檬苦素生物合成的影响. 果树学报, 2021, 38(8): 1240-1251.
YU H, YI Q, ZHANG M M, ZHU S P, WANG F S, ZHAO X C. Characteristics of CclSAUR49 expression and its effect on limonoids bio-synthesis in citrus. Journal of Fruit Science, 2021, 38(8): 1240-1251. (in Chinese)
[36] REN H, GRAY W. SAUR proteins as effectors of hormonal and environmental signals in plant growth. Molecular Plant, 2015, 8(8): 1153-1164.
[37] KANT S, PENG M S, ROTHSTEIN S J. Genetic regulation by NLA and microRNA827 for maintaining nitrate-dependent phosphate homeostasis in. PLoS Genetics, 2011, 7(3): e1002021.
[38] 李伟. 桤木苗期不同无性系PAL和木质素及生长关系的研究[D]. 北京: 中国林业科学研究院, 2001.
LI W. Study on the relationship between PAL and lignin and growth of different clones ofat seedling stage [D]. Beijing: Chinese Academy of Forestry, 2001. (in Chinese)
[39] 佘恒志, 聂姣, 李英双, 刘星贝, 胡丹, 马珊, 次仁卓嘎, 汪灿, 吴东倩, 阮仁武, 易泽林. 不同抗倒伏能力甜荞品种茎秆木质素及其单体合成特征. 中国农业科学, 2017, 50(7): 1202-1209. doi: 10.3864/j.issn.0578-1752.2017.07.003.
SHE H Z, NIE J, LI Y S, LIU X B, HU D, MA S, CIRENZHUOGA, WANG C, WU D Q, RUAN R W, YI Z L. Lignin and lignin monomer synthetic characteristics of culm in common buckwheat with different lodging resistance capabilities. Scientia Agricultura Sinica, 2017, 50(7): 1202-1209. doi: 10.3864/j.issn.0578-1752.2017.07.003. (in Chinese)
[40] 郭光艳, 柏峰, 刘伟, 秘彩莉. 转录因子对木质素生物合成调控的研究进展. 中国农业科学, 2015, 48(7): 1277-1287. doi: 10.3864/ j.issn.0578-1752.2015.07.03.
GUO G Y, BAI F, LIU W, BI C L. Advances in research of the regulation of transcription factors of lignin biosynthesis. Scientia Agricultura Sinica, 2015, 48(7): 1277-1287. doi: 10.3864/j.issn.0578- 1752.2015.07.03. (in Chinese)
[41] 胡丹, 刘星贝, 汪灿, 杨浩, 李鹤鑫, 阮仁武, 袁晓辉, 易泽林. 不同抗倒性甜荞茎秆木质素合成关键酶基因的表达分析. 中国农业科学, 2015, 48(9): 1864-1872. doi: 10.3864/j.issn.0578-1752.2015. 09.20.
HU D, LIU X B, WANG C, YANG H, LI H X, RUAN R W, YUAN X H, YI Z L. Expression analysis of key enzyme genes in lignin synthesis of culm among different lodging resistances of common buckwheat (moench). Scientia Agricultura Sinica, 2015, 48(9): 1864-1872. doi: 10.3864/j.issn.0578-1752.2015. 09.20. (in Chinese)
[42] 吕婷雯, 沈汝波, 杨洪强, 范伟国, 张瑞雪, 王利, 徐颖, 曹辉, 宁留芳, 周春然. 施加有机肥下木质素对平邑甜茶根系活力及根际土壤微生态的影响. 山东农业大学学报(自然科学版), 2018, 49(4): 561-565.
LÜ T W, SHEN R B, YANG H Q, FAN W G, ZHANG R X, WANG L, XU Y, CAO H, NING L F, ZHOU C R. Effects of lignin on root activity ofrehd. and microecology in rhizosphere soil under organic fertilizer. Journal of Shandong Agricultural University (Natural Science Edition), 2018, 49(4): 561-565. (in Chinese)
[43] 孙瑞敏, 翟梦华, 李虎, 李孟华, 王凤, 张桦. 木质素合成与梭梭生长发育的关系研究. 现代农业科技, 2019(5): 126-127.
SUN R M, ZHAI M H, LI H, LI M H, WANG F, ZHANG H. Study on the relationship between lignin synthesis andgrowth and development. XianDai NongYe KeJi, 2019(5): 126-127. (in Chinese)
[44] KATHARE P K, DHARMASIRI S, DHARMASIRI N. SAUR53 regulates organ elongation and apical hook development in. Plant Signaling & Behavior, 2018, 13(10): e1514896.
[45] WONG J H, SPARTZ A K, PARK M Y, DU M M, GRAY W M. Mutation of a conserved motif of PP2C.D phosphatases confers SAUR immunity and constitutive activity. Plant Physiology, 2019, 181(1): 353-366.
Functional Identification of Peach Gene
YANG Li1, CAO HongBo1, ZHANG XueYing1, ZHAI HanHan2, LI XinMiao1, PENG JiaWei1, TIAN Yi3, CHEN HaiJiang1
1Horticultural Department, Agricultural University of Hebei, Baoding 071000, Hebei;2Kashgar Vocational and Technical College, Kashgar 844000, Xinjiang;3Mountainous Areas Research Institute, Hebei Agricultural University/Technology Innovation Center for Agriculture in Mountainous Areas of Hebei Province/National Engineering Research Center for Agriculture in Northern Mountainous Areas, Baoding 071001, Hebei
【Objective】 The object of this study was to isolate a peach potential-related gene, to analyze its expression response to hormones, and to identify its role in regulating seedling growth in transgenic Arabidopsis, so as to provide the molecular basis for the regulation of tree potential. 【Method】 Using Zhongyou Pan 9 as the material for hormone treatment, the real-time fluorescence quantitative analysis was used to analyze the dynamic response ofwithin 24 hours.was cloned from the peach variety Jiuyan. PpSAUR73 overexpression vector was constructed and transformed into. Phenotypic observation of genetical modified Arabidopsis was carried out, and the germination rate statistics of both genetically modified and wild-type Arabidopsis sown simultaneously were performed too. The root length and hypocotyl of 7-day growing Arabidopsis with consistent germination were measured, and Arabidopsis with consistent germination was treated with different concentration hormone. Transcriptome sequencing was performed using 7-day-old seedlings, and the differentially expressed genes were analyzed by functional analysis, KEGG pathway enrichment analysis, and regulatory genes analysis, respectively.【Result】could respond quickly to hormone treatments. The overexpression ofcould affect the germination of Arabidopsis seeds. The hypocotyl and root length of seedlings were longer than those of wild type. In addition, the rosette of transgenic Arabidopsis was larger, and the overall growth potential was larger than wild type. The transgenic Arabidopsis showed decreased sensitivity to auxin. The transcriptome analysis of overexpressingshowed that there were 128 differentially expressed genes in both control groups, including 84 up-regulated genes and 44 down-regulated genes, and 20 differentially expressed genes were described. The GO function significant enrichment analysis of the differentially expressed genes generated by overexpression ofshowed that the differentially expressed genes were the most abundant in cell components, located in cytoplasm, cell membrane, organelle and extracellular regions. KEGG pathway enrichment analysis on differentially expressed genes were conducted, and the results showed that the differentially expressed genes in pairwise comparisons CK vsand CK vswere mainly enriched in phenylalanine biosynthesis pathway, plant hormone signal transduction pathway, starch sucrose metabolic pathway and other metabolic pathways. In the phenylalanine biosynthesis pathway,could regulate the upregulation of peroxidase encoding genes,,and. Peroxidases were associated with lignin synthesis, and lignin content was significantly correlated with plant growth, suggesting that overexpression ofmight be involved in regulating lignin synthesis inand thus growth. In plant hormone signal transduction pathway, the expression of some auxin responsive genes of,,,andin abscisic acid signal transduction pathway was up-regulated, and the expression of abscisic acid signal pathway genewas down-regulated.could regulate the growth ofand participate in multiple hormone signal transduction pathways. 【Conclusion】This study found thatcould quickly respond to hormones and regulate the growth in transgenic. The differentially expressed genes caused by overexpressed genes caused by overexpression ofwere mainly enriched in metabolic pathways, such as phenylalanine biosynthesis pathway, plant hormone signaling pathway, and starch sucrose metabolism pathway.also played an important role in IAA and ABA signal transduction pathways, it was speculated that it played an important role in the growth and development of peach trees.
peach;; gene expression; hormone treatment; signal transduction

10.3864/j.issn.0578-1752.2023.20.012
2023-03-24;
2023-06-30
财政部和农业农村部:国家现代农业产业技术体系资助项目(CARS-30-2-03)、河北省重点研发计划(20326804D)、热杂果现代种业科技创新团队(21326310D)
杨丽,E-mail:1097125451@qq.com。通信作者陈海江,chenhaijiang2001@163.com。通信作者田义,tianyi@hebau.edu.cn
(责任编辑 赵伶俐)
