基于生物信息学构建胃癌患者预后比例风险回归模型
2023-11-10孙晨夏云红
孙晨, 夏云红
安徽医科大学第一附属医院北区 安徽省公共卫生临床中心肿瘤科,安徽合肥 230000
胃癌(gastric cancer,GC)是最常见的消化系统恶性肿瘤,其病死率在恶性肿瘤中位居第二位[1]。由于早期胃癌多为轻度或无症状,故以晚期胃癌最为常见,平均5年生存率低于20%[2]。若在胃癌侵袭胃肌层前进行根治性手术化疗,术后5年生存率可达90%[3]。由于用于诊断胃癌的生物标志物数量有限,早期胃癌的检出率较低[4];因此,临床迫切需要能够有效识别和准确预测预后的分子标记物,用以改善胃癌患者的早期检测和治疗,并为开发胃癌靶点治疗提供策略[5]。本文从癌症基因组图谱(the cancer genome atlas,TCGA)数据库下载胃癌测序数量和临床样本,探讨GC发生发展中的关键基因和构建胃癌患者预后风险模型,为胃癌患者靶向治疗提供参考。
1 资料和方法
1.1 数据资料
从TCGA(https://portal.gdc.cancer.gov/)下载胃癌高通量表达数据,去掉临床信息存在缺失项的样本,共获得321例样本的mRNA转录测序信息和临床数据,其中包括胃癌组织293例,癌旁正常组织28例。基因表达数据用于差异基因分析和功能富集分析等,得到的差异表达基因结合患者临床信息用于构建比例风险回归模型(Cox)。
1.2 差异表达基因的筛选和功能富集分析
利用R语言软件limma包对胃癌组织和癌旁正常组织的基因表达数据进行差异分析,以错误发现率<0.05和基因表达差异倍数取对数绝对值>2.0为基因差异显著性阈值,获得差异表达基因。利用pheatmap包绘制热图显示差异分析结果。采用R语言软件clusterProfile包的非中心超几何分布完成GO和KEGG富集分析,并用ggplot2包和GOplot包显示分析结果。
1.3 Cox风险回归模型构建
提取TCGA数据库患者临床信息,根据患者年龄、性别、临床T分期、N分期、M分期及美国癌症联合委员会胃癌临床病理分期以及组织病理分级等临床指标,利用R语言软件Survival、Survminer包,对差异表达基因进行单因素Cox比例风险回归初步分析筛选(P<0.001),进一步利用双向逐步回归法进行多因素Cox比例风险回归分析,获得胃癌患者预后基因(P<0.05),构建总体生存期(overall survival,OS)预测模型,计算预后风险评分(risk score,RS),根据RS中位值将患者分为高、低风险组。采用Kaplan-Meier法进行Log-rank检验多基因预后模型的预后预测价值;计算ROC曲线下面积(area under the curve,AUC)评价多基因预后模型的有效性和敏感性。绘制列线图预测患者预后情况。P<0.05为差异具有统计学意义。
2 结 果
2.1 差异表达基因的筛选
从TCGA胃癌高通量测序数据中最终获得930个差异表达基因。相较于癌旁正常组织,胃癌组织中表达上调基因436个,表达下调基因494个(P<0.05;图1)。
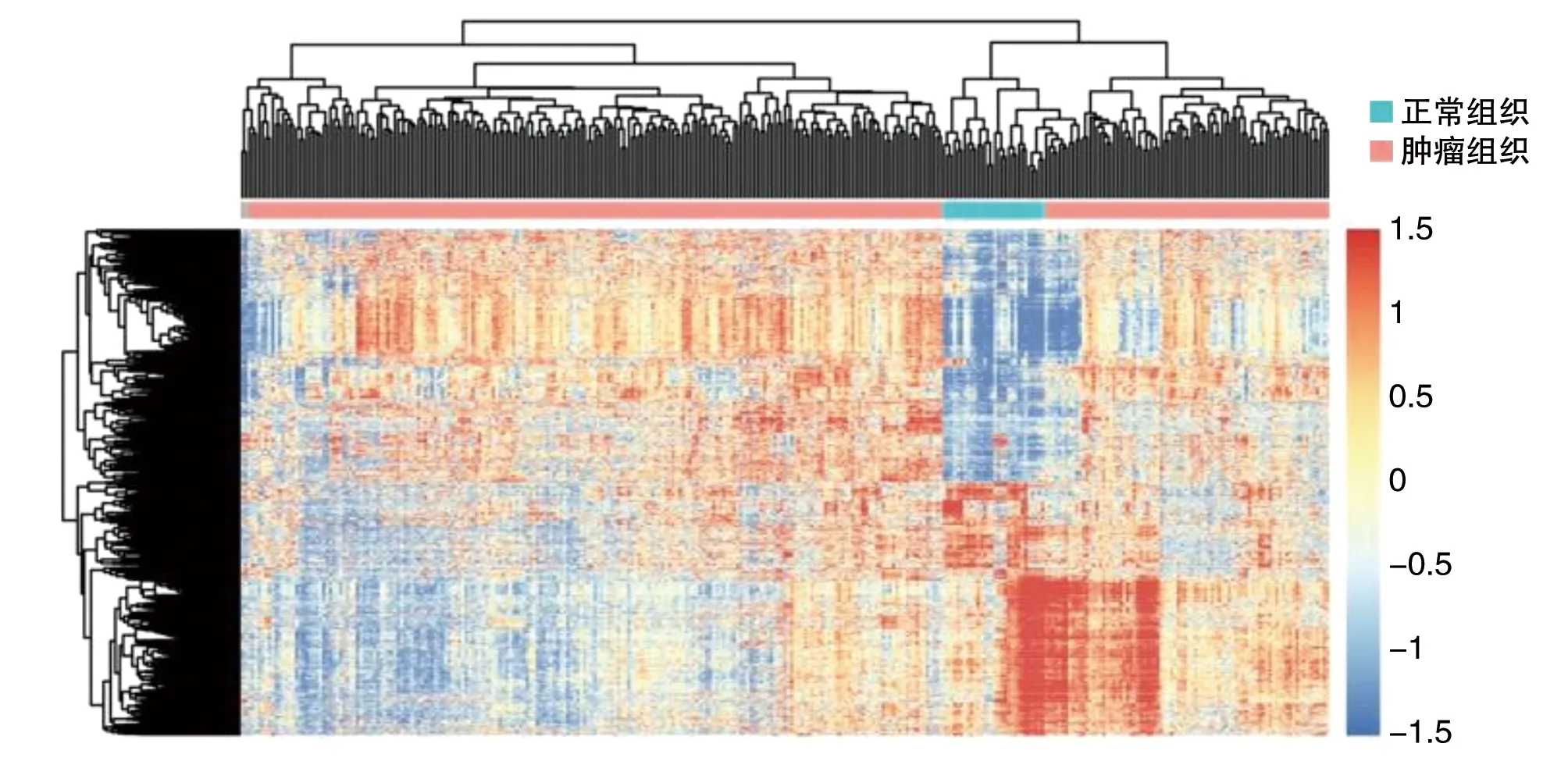
图1 TCGA胃癌数据集差异表达基因分层聚类热图
2.2 差异表达基因的功能富集
KEGG功能富集分析显示,上调表达基因主要富集在细胞周期、白细胞介素(interleukin,IL)-17信号通路、细胞因子-细胞因子受体相互作用等通路;下调表达基因主要富集在神经活性配体-受体相互作用、细胞色素P450对外源性物质的代谢、cAMP信号通路(图2)。
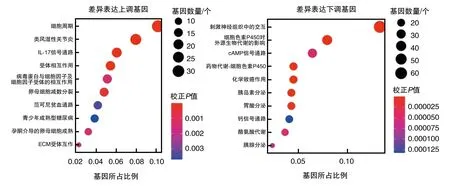
图2 KEGG功能富集分析
GO功能富集分析显示,上调表达基因主要富集在核分裂、着丝粒区染色体、细胞外基质结构成分等注释项中;下调表达基因主要富集在肌肉系统过程、突触膜、离子门控通道活性等注释项中(图3)。

图3 差异表达基因的GO功能富集分析
2.3 多基因Cox预后模型的建立
利用单因素Cox比例风险回归分析共筛选到73个可能影响患者总体生存期的基因;通过多因素Cox比例风险回归分析得到10个基因,并建立多基因预后预测模型。其中,SRMS、CTLA4、MFAP2、ADAMTS18和PPEF1在胃癌组织中显著高表达,ACADL、AR、MFAP5、RORB和DCHS2则显著低表达(P<0.001)。
依据预后模型(PI=-0.119×SRMS-0.299×CTLA4+0.114×ACADL+0.082×MFAP2+0.065×ADAMTS18-0.104×AR-0.028×MFAP5+0.162×RORB+0.099×DCHS2+0.172×PPEF1)中位RS值(1.028)将293例患者分别纳入高、低风险组,低风险组生存率显著高于高风险组(P<0.001;图4A)。ROC曲线AUC为0.628;患者1年、3年和5年ROC曲线AUC分别为0.627、0.713和0.847(图4B)。预后评分越高(图4C),死亡风险越大(图4D)。多基因预后模型各基因在高、低风险组的表达量见图3E。通过列线图中差异基因表达评分可以定量预测胃癌患者1、3、5年总体生存率(图5)。
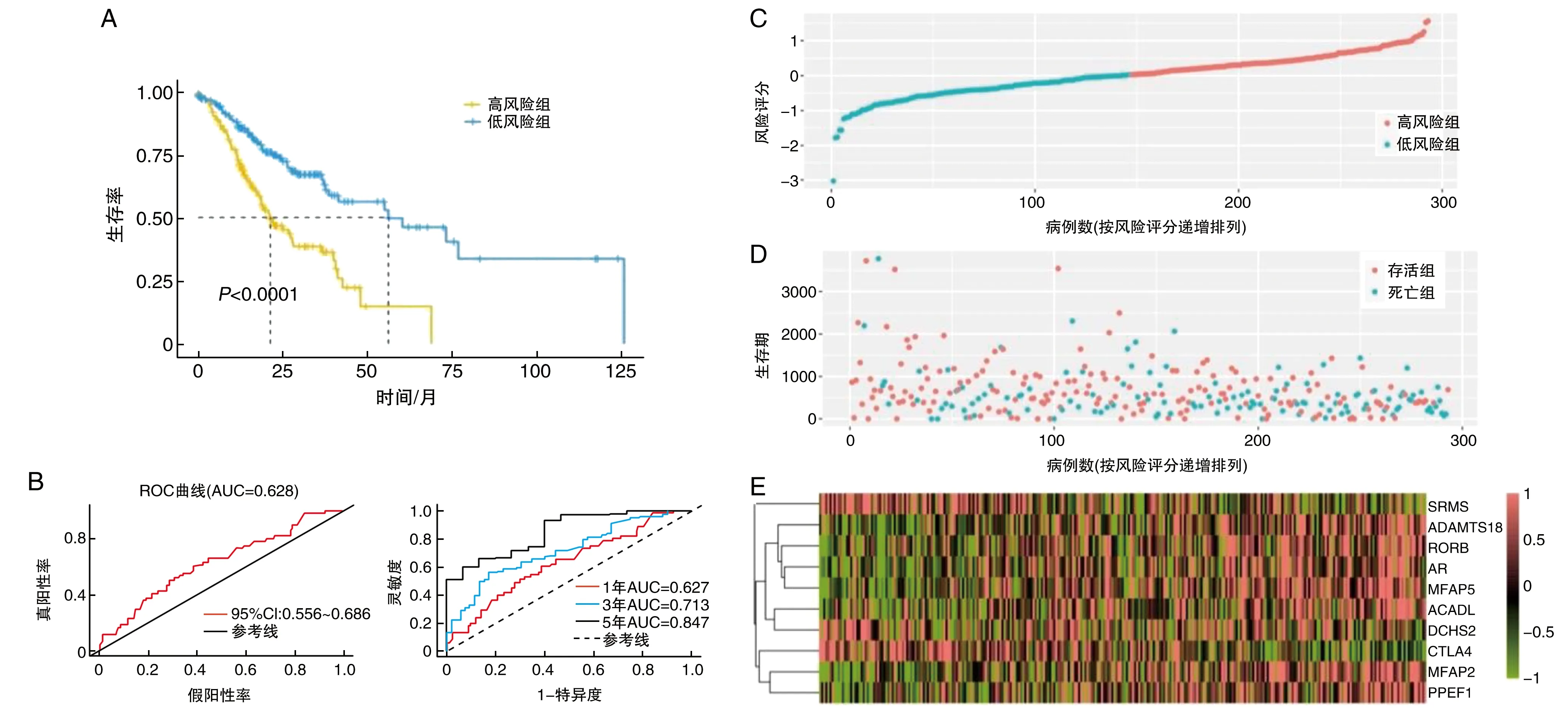
图4 胃癌预后模型在TCGA整体数据集中高风险和低风险患者分布情况A为生存率;B为ROC曲线;C为风险评分;D为生存期;E为各基因的表达。
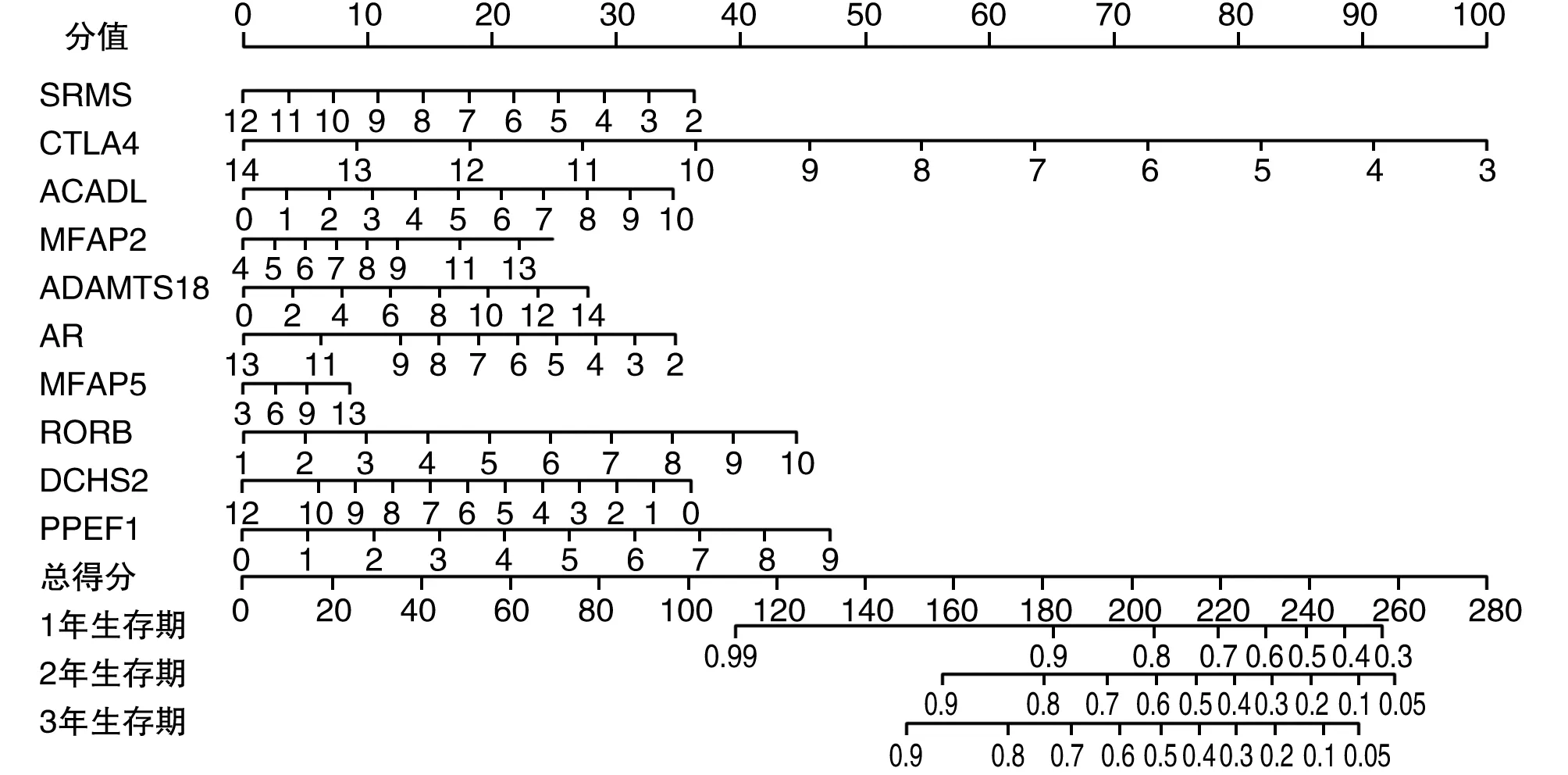
图5 胃癌患者预后模型列线图
2.4 预后模型不同风险胃癌患者临床特征比较
高风险组和低风险组病理分期、肿瘤大小、远处转移、肿瘤分级比较,差异均有显著性(P<0.05;表1)。
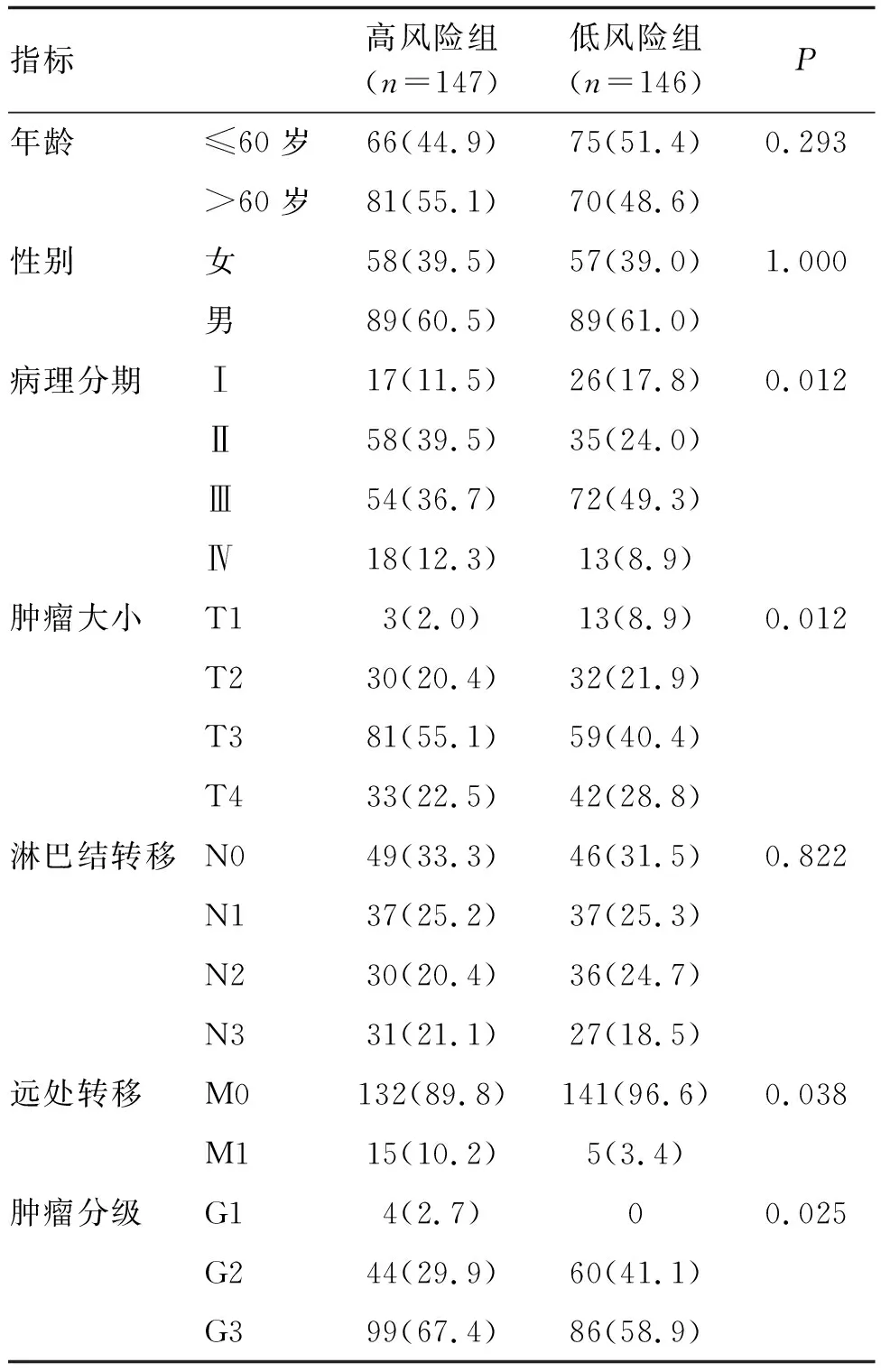
表1 预后模型不同风险胃癌患者临床特征比较 例(%)
2.5 胃癌患者预后的Cox回归分析
将多基因预后模型和TCGA胃癌数据集患者临床指标纳入Cox回归分析,其中多基因预后模型风险评分为连续变量,整理后的患者临床指标为分类变量。单因素Cox回归分析显示病理分期、淋巴结转移、肿瘤大小、年龄和预后模型风险评分是影响胃癌患者总体生存期的危险因素(P<0.001;表2)。多因素Cox回归分析显示年龄和预后模型风险评分是影响胃癌患者预后的独立因素(P<0.001;表3)。
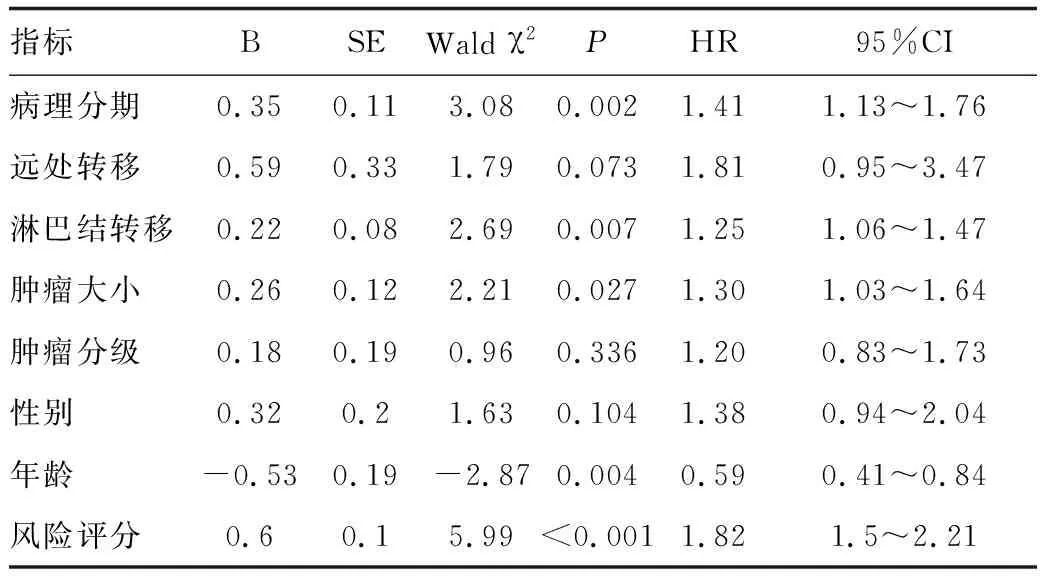
表2 单因素Cox回归分析TCGA胃癌患者总体生存期的影响因素

表3 多因素Cox回归分析结果
3 讨 论
胃癌的治疗效果和策略取决于诊断的癌症阶段。早期胃癌多为轻度或无症状,故早期诊断非常具有挑战性。本研究对胃癌数据集进行生物信息学分析,利用双向逐步回归法对930个差异表达基因进行Cox风险回归分析,构建了一个由10个基因构成的预后风险评分模型。模型对胃癌患者的预后有良好的预测价值和预测效能,且与胃癌患者的肿瘤分期、大小、分级和远处转移有关。
模型基因与肿瘤的进展和预后相关,SRMS、CTLA4、MFAP2、ADAMTS18和PPEF1在胃癌组织中显著高表达,ACADL、AR、MFAP5、RORB和DCHS2则显著低表达。SRMS是一种非受体酪氨酸激酶,调节信号通路,可作为结直肠癌患者潜在治疗靶点[6]。研究胃癌蛋白质组发现SRMS是唯一的差异表达激酶[7]。胃癌组织CTLA4相对于正常黏膜显著上调,CTLA4的激活导致胃癌患者的总生存期缩短[8],与本研究分析结果一致。MFAP2促进胃癌和甲状腺乳头状癌的增殖、迁移、侵袭和上皮-间质转化[9-10],shRNA沉默能够抑制裸鼠致瘤性和肿瘤转移[11]。PPEF1参与乳腺癌发生的分子机制,可作为预后和诊断标志物[12]。本研究发现,ADAMTS18在胃癌组织中高表达,然而在其他多肿瘤中表达显著下调,且与启动子高度甲基化有关,被认定为肿瘤抑制基因[13-15]。ADAMTS18在胃癌中的功能还有待进一步研究。ACADL在肝细胞癌与临床预后不良相关,抑制Hippo/YAP通路,恢复ACADL表达导致癌细胞周期阻滞和生长抑制[16]。AR诱导和促进G1周期素依赖性激酶的活性,是前列腺癌的关键因子[17]。MFAP5是一种细胞外基质糖蛋白,显著降低膀胱癌细胞增殖、迁移和侵袭能力[18]。在胃癌和结直肠癌中,DCHS2非常规移码突变可导致癌细胞黏附和极性功能失活[19]。
本研究构建的多基因预后风险模型尚未见报道,其功能值得进一步研究。该模型在临床预测胃癌的预后方面具有较好的效果,可以作为预测胃癌预后潜在标志物组合,并为寻找靶向治疗药物提供数据支持。本研究尚需进一步收集大样本临床数据验证模型的有效性和可靠性。
