大豆BZR1基因家族进化与油菜素内酯响应分析
2023-11-08严新悦左春柳郭新月李艳玲丁洪玲
张 晴, 严新悦, 左春柳, 郭新月, 李艳玲, 张 岚, 丁洪玲, 袁 敏
(1.华北理工大学 生命科学学院,河北 唐山 063210; 2.华北理工大学 理学院,河北 唐山 063210)
油菜素内酯(brassinosteroid,BRs)是植物体内的甾醇类激素,在种子萌发、光形态发生、细胞伸长、逆境胁迫响应等多个生物学过程中发挥重要的作用[1].BRs信号由细胞质膜上的受体BRI1和共受体BAK1的胞外结构域共同感知,通过信号传导过程激活转录因子BZR1和BES1,进而调控下游基因的表达[2].BZR1和BES1作为BZR(brassinazole resistant transcription factor)家族的成员,二者在蛋白水平上具有88 %的序列同源性.虽然都是BRs信号通路中的转录因子,二者的作用有所区别.BZR1结合CGTGT/CG序列抑制BRs调控基因的表达,而BES1结合E-box序列激活BRs调控基因的表达[3-7].
随着测序技术的发展和基因组数据的不断更新,现已有多个物种的BZR1家族被鉴定.高粱SbBZR1-3,SbBZR1-4及SbBZR1-9基因的相对表达量在不同的外源激素处理下因处理时间延长而发生明显的变化,推测高粱BZR1基因家族在激素应答过程中发挥重要的作用[8];玉米ZmBES1/BZR1基因家族大部分成员的表达受到脱落酸(ABA)和不同波长的光处理诱导,表明ZmBES1/BZR1参与了玉米ABA和光介导的信号通路,且在不同器官和发育阶段对ABA信号的响应表现出很大的差异[9-10];苹果BZR1基因通过调控内源BRs的含量以及参与BRs信号转导来调控果树的株高[11-13].
大豆是一种重要的豆科植物,是中国重要的粮食作物之一,其种子含有丰富的植物蛋白质,具有极高的营养价值,常被制成各种豆制品,有利于人体的健康和生长发育.目前已有研究鉴定了部分豆科植物的BZR1基因家族成员[14],但仍缺乏系统的进化和功能分析.本研究将利用生物信息学分析方法结合相关实验,对大豆BZR1基因家族进行鉴定,从蛋白理化性质、基因系统进化、基因结构、基因复制方式以及表达和亚细胞定位等方面进行系统分析,为揭示该家族在BRs信号通路中的功能和调控机制奠定基础.
1 材料和方法
1.1 基因家族成员的鉴定及理化性质分析
拟南芥BZR1家族成员的蛋白序列从TAIR网站(http://www.arabidopsis.org/)下载.大豆、蒺藜苜蓿和葡萄的全基因组数据均从JGI数据库(https://phytozome.jgi.doe.gov/pz/portal.html)下载,版本分别为Glycine max Wm82.a4.v1,Mtruncatula_285_Mt4.0和Vvinifera_457_v2.1.水稻基因组文件从水稻数据库(http://rice.plantbiology.msu.edu/index.shtml)获取.
为了鉴定大豆BZR1基因家族的成员,利用本地BLAST+[15]软件将拟南芥BZR1蛋白序列分别与大豆、蒺藜苜蓿、葡萄和水稻的基因组数据进行序列比对筛选,设置筛选条件为一致性>40 %,e值<1e-5.将筛选得到的蛋白质序列提取出来,提交至Pfam(http://pfam.xfam.org/)和SMART(http://smart.embl-heidelberg.de/)网站预测结构域信息,保留具有BES1_N结构域的蛋白序列,确定其为BZR1基因家族成员;使用ExPASy中的ProtParam(https://web.expasy.org/protparam/)预测蛋白质的氨基酸个数、分子量、等电点、疏水性和不稳定系数;使用Cell-PLoc 2.0中的Plant-mPLoc(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)预测亚细胞定位.
1.2 系统发育树的构建
使用MUSCLE[16]对筛选获得的BZR1家族成员进行蛋白序列比对,并用MEGA11[17]中的最大似然法(maximum likelihood,ML)构建系统发育树,设置bootstrap值为1 000,模型为JTT.
1.3 基因结构和结构域
将所研究物种的BZR1家族蛋白序列提交至MEME(http://meme-suite.org/)预测保守基序,设置预测保守基序个数为10,长度为6~50 aa,模式为zoop;提交至Pfam(https://pfam.xfam.org/)网站,预测结构域信息,设置e值为1e-5.根据保守基序和结构域的起始位置在图中标注包含关系;在基因组gff3文件中提取BZR1基因家族成员的位置信息,使用TBtools[18]的Gene Structure View(Advanced)程序绘制基因结构组合图.
1.4 共线性分析
利用MCScan X[19]软件分析大豆BZR1基因的复制类型,并对其基因组内的共线性进行分析.使用BLAST对5个物种的全基因组蛋白序列进行两两比对,设置e值为1e-5,将比对结果提交至MCScanX进行物种间和物种内的共线性分析,从结果中提取BZR1基因所在的共线性区块,并统计共线性基因对的数量.
1.5 蛋白质互作
利用STRING(https://string-db.org/)数据库分别预测15个GmBZR1蛋白的相互作用蛋白,限制最低互作得分为0.700(高可信度),设置显示的互作蛋白最多不超过5个,构建蛋白质互作网络[20].
1.6 GmBZR1s的表达及亚细胞定位分析
从GEO公共数据库下载BRs处理大豆幼苗的转录组数据,提取GmBZR1s的表达FPKM值,绘制柱状图.
GmBZR1.11蛋白磷酸化状态和亚细胞定位的检测在烟草叶片中进行.构建p35S:AgBZR1s-YFP表达载体,转化农杆菌GV3101.利用农杆菌介导的转染技术注射烟草下表皮细胞,48 h后,激光聚焦显微镜下观察基因表达定位情况.同时,1 μM的BRs处理烟草叶片1 h,提取全蛋白,Western-blot 检测GmBZR1.11的磷酸化水平.
2 结果与分析
2.1 GmBZR1基因家族成员的鉴定及理化性质分析
拟南芥共包含8个具有BES1-N特征结构域的BZR1家族成员,AtBZR1,AtBES1,AtBEH1~4,AtBMY2和AtBMY4.AtBMY2和AtBMY4具有额外的β-淀粉酶结构域,因此被定义为淀粉酶样蛋白.本研究以拟南芥BZR1蛋白作为靶序列,从大豆、蒺藜苜蓿、葡萄和水稻中分别鉴定出15,7,7和6个BZR1家族成员.根据染色体定位和基因位置,15个大豆BZR1基因家族成员依次被命名为GmBZR1.1-GmBZR1.15.GmBZR1s蛋白序列长度在263~705 aa之间,分子量在29.63~78.84 kDa之间.除GmBZR1.1,GmBZR1.5,GmBZR1.6和GmBZR1.15这4个蛋白质是酸性蛋白质外,其余12个成员均为碱性蛋白质;除GmBZR1.1和GmBZR1.5的不稳定系数小于40外,其余蛋白质不稳定系数均大于40,属不稳定蛋白质(表1).

表1 大豆BZR1家族成员理化性质及定位分析Tab.1 Physicochemical Properties of BZR1 Family Members in Soybean
2.2 系统发育分析
Ⅰ拟南芥BZR1s通常被分为3组(Ⅰ,Ⅱ,Ⅲ),BRs信号通路的关键转录因子成员AtBZR1/BES1在Ⅰ组;AtBEH3和AtBEH4在Ⅱ组;淀粉酶样蛋白AtBMY2和AtBMY4在Ⅲ组.根据拟南芥BZR1s的分组和进化树的拓扑结构,大豆BZR1s同样被分为3组,Ⅰ组包含7个GmBZR1成员,Ⅱ和Ⅲ组分别包含4个GmBZR1成员.进化树显示,与大豆亲缘关系最近的是蒺藜苜蓿,其次为双子叶物种拟南芥和葡萄,最后是单子叶物种水稻.基于GmBZR1s构建的系统发育树的拓扑结构与各物种亲缘关系相符(图1).

图1 BZR1基因家族系统发育树Fig.1 Phylogenetic Tree of BZR1 Gene Family
2.3 GmBZR1s蛋白保守基序和基因结构分析
为了探究GmBZR1s蛋白的功能,首先分析预测了GmBZR1s蛋白的特征结构域和保守基序.所有成员均具有BES1-N特征结构域,Ⅲ组成员具有额外的β-淀粉酶结构域.Ⅰ和Ⅱ组成员具有相似的基序组成,而Ⅲ组成员的基序组成具有明显差异.首先,Ⅲ组成员特有的基序2,3,4,6和7组成其β-淀粉酶结构域(Glyco_hydro_14,又称BAM-like结构域).其次,Ⅲ组成员都缺乏基序9和10.基序9是一个富含丝氨酸的基序,被认为是GSK3激酶的磷酸化位点.基序10包含14-3-3蛋白结合位点,据报道,拟南芥BZR1和BES1在该位点被14-3-3蛋白结合后会被滞留在细胞质中.Ⅲ组成员特异的基序组成暗示其发生了明显的功能分化,具备额外的淀粉酶功能(图2b).
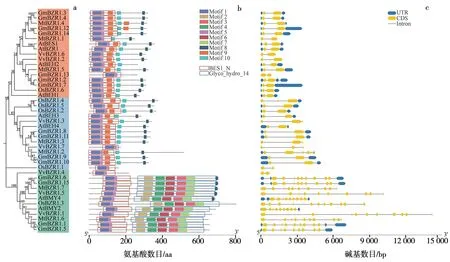
a.BZR1基因家族系统发育树; b.保守基序与结构域分析; c.基因结构分析.图2 BZR1家族成员保守基序和基因结构分析Fig.2 Conserved Motif and Gene Structure Analysis of BZR1 Gene Members
基因结构分析显示,聚在同一进化分支上的BZR1基因在外显子和内含子的数目和位置方面都非常接近.Ⅰ和Ⅱ组成员,除MtBZR1.2外,均含有2~3个编码框(coding sequence,CDS);而Ⅲ组成员的CDS数量较多,除VvBZR1.4外,每个成员均含有10个CDS(图2c).
2.4 GmBZR1s基因复制类型分析
植物基因的复制有多种不同方式,并且基因组复制是基因家族扩张的一个重要来源.本研究发现,80 %的大豆BZR1基因来自全基因组加倍或片段复制(WGD或segmental duplication);6.67 %和13.33 %的大豆BZR1基因分别来自分散基因复制(dispersed duplication)和串联基因复制(tandem duplication);单基因复制(singleton duplication)和近端基因复制(proximal duplication)并没有对大豆BZR1基因产生影响(图3).结果表明,不同复制方式对大豆BZR1成员的影响不同.与大豆全基因组相比,全基因组加倍或片段复制对该家族的影响更大.
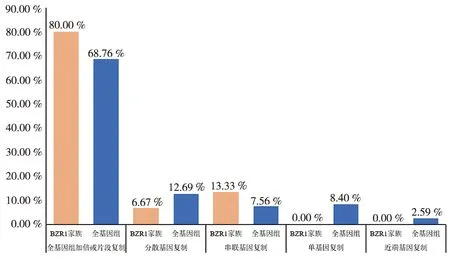
图3 不同复制类型对大豆BZR1同源基因对的贡献率Fig.3 Contribution of Different Duplication Types to the BZR1 Duplicated Gene Pairs
2.5 GmBZR1s基因共线性分析
为了更进一步探索研究大豆BZR1基因的进化过程,本研究进行了共线性分析,在大豆基因组内找到18对BZR1旁系同源基因对.同义置换率Ks值可以反映物种分歧的时间和基因组加倍事件发生的时间.已有研究表明,豆科与其它双子叶植物共同祖先在大约(1.3~1.5) 亿a前发生了一次全基因组三倍化事件(Ks≈1.310),豆科植物在大约(5 900) 万a前发生了一次全基因组二倍化事件(Ks≈0.627),大豆在大约1 300 万a前发生了一次物种内独有的全基因组二倍化事件(Ks≈0.164).本研究计算了大豆BZR1旁系同源基因对的Ks值,并与基因组加倍事件的Ks值相比较,推算大豆BZR1旁系同源基因对的产生时间.8对大豆BZR1旁系同源基因对的Ks大于1.310,6对大豆BZR1旁系同源基因对的Ks为0.627~1.310,各有2对大豆BZR1旁系同源基因对的Ks分别为0.164~0.627和0~0.164.由此可见,大部分大豆BZR1旁系同源基因对(8+6)与双子叶物种共同祖先经历的三倍化事件以及豆科植物经历的二倍化事件相关,均产生于大豆物种形成之前,说明该家族基因在整个进化进程中均比较保守(图4).

不同颜色线条代表不同Ks区间.红色线条为Ks>1.310; 橙色线条为0.627 为了研究GmBZR1s参与调控的信号网络,本研究利用STRING数据库预测了GmBZR1s的相互作用蛋白.Ⅰ组中的6个成员(GmBZR1.2,1.3,1.4,1.7,1.12和1.14)均与BIN2激酶家族成员相互作用,这与拟南芥中AtBZR1和AtBIN2在BRs信号转导通路中相互作用相一致;Ⅱ组中的4个成员(GmBZR1.8,1.9,1.10和1.11)与含有HSP20结构域的热休克蛋白相互作用;Ⅲ组中的4个成员(GmBZR1.1,1.5,1.6和1.15)与含有α-淀粉酶和糖苷水解酶相关结构域的蛋白相互作用,这与其含有β-淀粉酶结构域相吻合;特殊的是,GmBZR1.13不与上述任何蛋白相互作用,可能是由于其缺少某些保守基序(图5). 黑色字体标注GmBZR1蛋白; 深蓝色字体标注BIN2家族成员蛋白; 紫色字体标注含有HSP20结构域的蛋白; 绿色字体标注与GmBZR1.13相互作用的蛋白; 浅蓝字体标注含有Alpha-amylase和Carbohydrate-binding module_48两个结构域的蛋白; 红色字体标注含有Glyco_hydro_77结构域的蛋白; 黄色字体标注含有Alpha-amyl_C2和Alpha-amylase两个结构域的蛋白.图5 GmBZR1相互作用蛋白网络Fig.5 Protein Interaction Network of GmBZR1 Proteins 为了探索GmBZR1是否也在BRs信号通路中发挥作用,分别在转录和蛋白磷酸化修饰方面分析了GmBZR1是否响应BRs.结果表明,绝大多数GmBZR1基因的表达在BRs处理前后没有明显变化,只有GmBZR1.2在BRs处理后表达降低,GmBZR1.8和GmBZR1.11在BRs处理后表达升高.文献报道,拟南芥AtBZR1的蛋白磷酸化水平受到BRs的调控,BRs处理诱导AtBZR1蛋白的去磷酸化.本研究检测了BRs处理前后GmBZR1.11蛋白的磷酸化状态,结果表明,BRs处理后GmBZR1.11蛋白的磷酸化水平明显降低,推测GmBZR1同样作为转录因子在BRs信号通路中发挥重要作用.亚细胞定位分析显示,GmBZR1蛋白在细胞核和细胞膜均有十分明显的表达(图6). a.BRs处理前后GmBZR1s转录水平的变化,误差线代表3次生物学重复间的标准误差,*代表显著性差异p<0.05; b.BRs处理前后GmBZR1.11蛋白的磷酸化水平变化; c.GmBZR1.11的亚细胞定位.图6 BRs诱导GmBZR1.11蛋白去磷酸化Fig.6 BRs Induced the Dephosphorylation of GmBZR1.11 作为植物特有的一类转录因子基因家族,BZR1基因不仅在BRs信号通路中发挥关键作用,还参与调控BRs与其他激素信号和光信号等途径的交互作用[21-23].目前,在多种植物中都鉴定出了BZR1家族成员,拟南芥、高粱和水稻中分别鉴定出8,9和6个BZR1成员[8,24].本研究利用同源比对和保守结构域分析相结合的方式,在大豆中鉴定出15个BZR1基因家族成员,划分为3组,与拟南芥中BZR1的分类相似. 特征结构域与基因结构分析显示,在所选5个物种的BZR1蛋白中,N端都存在一个高度保守的BES1_N结构域,表明BZR1可能存在功能上的高度保守.此外,Ⅲ组成员,除含有BES1_N特征结构域外,还含有β-淀粉酶结构域,其所含CDS的数量多于其他2组,暗示其可能具有额外的功能.转录组数据显示,在RNA水平上,BRs处理前后绝大多数GmBZR1基因表达并没有明显的变化,但苹果MdBZR1基因在外源BRs处理之后的表达量会显著升高,说明BRs对不同物种BZR1基因的影响是有区别的.拟南芥BZR1的蛋白磷酸化水平受到BRs的调控,BRs处理诱导AtBZR1蛋白的去磷酸化[4].本研究证实,在蛋白水平上,BRs处理后大豆BZR1蛋白磷酸化水平明显降低,推测BRs也可以通过改变大豆BZR1蛋白的磷酸化状态调控下游基因的表达. 全基因组加倍(或称多倍化)是植物基因组进化过程中一个非常普遍的现象,这种遗传效应使植物基因组中出现大量的重复基因,为植物基因组功能进化提供了原始遗传材料,也为基因家族的扩张和功能分化奠定了基础[25-26].本研究所选物种中,除单子叶物种水稻外,其余物种均经历了一次真双子叶共同祖先物种的全基因组三倍化事件(eudicot-common hexaploidy,ECH,又称γ事件);葡萄基因组在经历了此次三倍化事件后没有发生过其他多倍化事件,因此保留了比较完整的双子叶物种祖先的染色体结构;拟南芥基因组在经历了真双子叶共同祖先物种的三倍化事件后,又经历了2次全基因组二倍化事件;大豆和蒺藜苜蓿经历了一次豆科共有的全基因组二倍化事件;大豆物种在大约1 300 万a前又经历了一次特异性的全基因组二倍化事件[27-28].本研究鉴定到15个大豆BZR1家族成员,其家族成员数量明显多于拟南芥等物种,暗示大豆的特异性加倍事件可能在该家族的扩张中发挥重要的作用.共线性分析表明,18对大豆BZR1旁系同源基因对中的14对,与双子叶物种共同祖先经历的三倍化事件以及豆科植物经历的二倍化事件相关,均产生于大豆物种形成之前,说明该家族基因在整个进化进程中均比较保守. 综上所述,本研究从大豆中鉴定出15个BZR1基因家族成员,并从蛋白理化性质、基因结构、基因系统进化、复制方式以及表达与定位等方面进行了系统分析.研究结果不仅为其他物种BZR1家族的研究提供了思路,也为揭示该家族在大豆BRs信号通路中的功能和调控机制奠定了基础.2.6 GmBZR1s互作蛋白分析
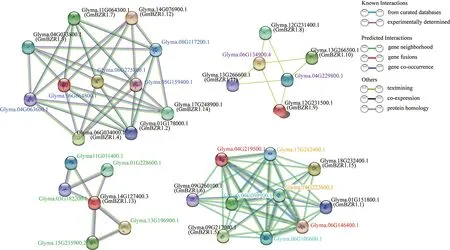
2.7 GmBZR1s的表达及亚细胞定位分析

3 讨 论
