密花豆叶绿体基因组序列特征及密码子偏好性分析
2023-11-02吴民华叶晓霞谭靖怡王燊沈诗焕黄琼林
吴民华 叶晓霞 谭靖怡 王燊 沈诗焕 黄琼林





DOI:10.3969/j.issn.2095-1191.2023.06.005
摘要:【目的】分析密花豆葉绿体基因组序列特征及密码子偏好性,为密花豆种质鉴定、分子育种及资源保护利用提供理论参考。【方法】利用高通量测序技术对密花豆的叶绿体基因组进行测序,结合生物信息学软件和工具对序列进行拼接、注释及序列特征和密码子偏好性分析,并通过构建系统发育进化树解析密花豆的进化地位。【结果】密花豆叶绿体基因组全长为152275 bp,是由83924 bp的大单拷贝(LSC)区、25113 bp的反向重复A(IRA)区、18125 bp的小单拷贝(SSC)区和25113 bp的反向重复B(IRB)依次排列而成的环状双链四分体分子。密花豆叶绿体基因组共注释到129个基因,包括84个蛋白编码基因(PCGs),8个rRNA和37个tRNA。密花豆叶绿体基因组在自然选择为主的多因素作用下,偏好使用以A或T结尾的密码子,最优密码子是GCT、AGA、CGA、AAT、TGT、CAA、GAA、GGT、CAT、ATA、TTA、AAA、TTT、CCT、TCA、ACT、TAT和GTT。从密花豆叶绿体基因组检测到117个SSR位点,由单、二、三、四核苷酸重复基元组成,其中以单、二核苷酸复基元数量较多,分别占SSR位点总数的50.4%和36.8%,且单、二、三、四核苷酸重复基元均以A或T及其组合为主。叶绿体基因组在密花豆属内和属间均存在较明显的碱基突变,密花豆与同属的美丽密花豆的叶绿体基因组序列相似性最高,说明二者亲缘关系最近。【结论】密花豆叶绿体基因组具有植物叶绿体基因组典型的结构特点,在密花豆属内及属间均有较好的鉴别效果,且其密码子偏好性是多因素共同作用的结果,其中自然选择是主要决定因素,但碱基突变以及其他因素对密码子使用偏好也有一定影响。
关键词:密花豆;叶绿体基因组;序列特征;密码子偏好性;系统发育分析
中图分类号:S567.19 文献标志码:A 文章编号:2095-1191(2023)06-1633-13
Sequence characteristics and codon bias analysis of chloroplast genome from Spatholobus suberectus Dunn
WU Min-hua1,YE Xiao-xia1,TAN Jing-yi2,WANG Shen2,SHEN Shi-huan3,
HUANG Qiong-lin1*
(1School of Basic Medicine,Guangdong Medical University,Zhanjiang,Guangdong 524023,China;2School of Pharmacy,Guangdong Medical University,Zhanjiang,Guangdong 524023,China;3The First Clinic Medicine College,Guangdong Medical University,Zhanjiang,Guangdong 524023,China)
Abstract:【Objective】This work was aimed to clarify the sequence features and codon bias of chloroplast genome from Spatholobus suberectus Dunn,and provide theoretical reference for germplasm identification,molecular breeding and resource conservation and utilization of S. suberectus. 【Method】S. suberectus chloroplast genome was sequenced using high-throughput sequencing technology and then was assembled,annotated and analyzed by means of bioinformatics softwares and tools. A phylogenetic tree was also constructed to understanding the evolution status of S. suberectus. 【Result】The chloroplast genome of S. suberectus was an annular double-chain molecule with a size of 152275 bp,which was successively composed of a large single copy(LSC) with 83924 bp,an inverted repeat A(IRA)with 25113 bp,a small single copy(SSC) with 18125 bp and an inverted repeat B(IRB)with 25113 bp. The chloroplast genome annotated 129 genes,including 84 protein-coding genes(PCGs),8 rRNA and 37 tRNA. The chloroplast genome of S. suberectus tended to use the codon ended with A or T under the action of multiple factors dominated by natural selection,and the preference codons were identified as GCT,AGA,CGA,AAT,TGT,CAA,GAA,GGT,CAT,ATA,TTA,AAA,TTT,CCT,TCA,ACT,TAT,GTT. A total of 117 SSR loci were detected from the chloroplast genome of S. suberectus,which were composed of mononucleotide,dinucleotide,trinucleotide and tetranucleotide repeat motifs. The number of mononucleotide and dinucleotide repeat motifs was higher,accounting for 50.4% and 36.8% of the total number of SSR loci respectively,and the mononucleotide,dinucleotide,trinucleotide and tetranucleotide repeat motifs were mainly A or T and their combinations. There were obvious base mutations in the chloroplast genome both within and among the genera Spatholobus. The chloroplast genome sequences of S. suberectus and S. pulcher were the most similar,and the two were most closely related.【Conclusion】The chloroplast genome of S. suberectus possesses typical structure of plant chloroplast genome and preferable identification capability among the Spatholobus and other genera. The codon bias of S. suberectus chloroplast genome is a synergistical result of multiple factors. Among them,natural selection contributes most,and the base mutation and others also have a certain impact.
Key words: Spatholobus suberectus Dunn; chloroplast genome; sequence characteristics; codon bias; phylogenetic analysis
Foundation items: Guangdong Basic and Applied Basic Research Foundation(2018A030310116); Zhanjiang Science and Technology Plan Project (2019A01008)
0 引言
【研究意义】密花豆(Spatholobus suberectus Dunn)为豆科(Leguminosae)密花豆属(Spatholobus)攀援植物,为我国特有的物种,主要分布在广东、广西、福建等岭南省(区)(中国植物志编辑委员会,1995)。密花豆的藤茎可供药用,称鸡血藤,因可分泌红棕色树脂而得名。鸡血藤苦泄温通、微甘能补,入肝、肾经,既能活血通络而止痛,又补血舒筋而止痛,可用于月经不调、痛经、经闭、风湿痹痛、麻木瘫痪、血虚萎黄等病症的治疗(国家药典委员会,2020),为我国传统常用大宗中药材,也是鸡血藤胶囊、花红片、妇科千金片等十余种知名中成药的主要组方原料。由于鸡血藤的显著疗效和广泛应用,导致密花豆种质资源被大量开采和挖掘,野生资源的种群遭到严重破坏,生存状态变差,更新迟缓。密花豆在广东和广西有一定规模的仿野生栽培,但规范化程度低,无法满足市场供应和临床需求(荣广天,2016;萧晓吉等,2022)。加强密花豆种质鉴定、系统发育分析、栽培育种等基础研究已成为鸡血藤资源保护和可持续利用的重要课题。叶绿体基因组是存在于核基因组之外的另一种遗传系统(孙孟涛等,2022),具有序列短而易获取和解析(樊守金等,2022)、基因组成较恒定但有适中的碱基突变和核苷酸置换率(倪梁红等,2015)等特点。另外,叶绿体基因组中存在类型较丰富、多态性较高的简单重复序列(Simple sequence repeat,SSR),可用于植物分子标记的开发(王化坤等,2006)。开展叶绿体基因组的高通量测序及其序列解析,对推动密花豆种质鉴别、系统发育分析、分子标记辅助育种等研究具有重要的意义。【前人研究进展】被子植物的叶绿体基因组呈保守的环状双链四分体结构,长度为115~165 kb,由大单拷贝(Large single copy,LSC)区、反向重复A(Inverted repeat A,IRA)区、小单拷贝(Small single copy,SSC)区和反向重复B(Inverted repeat B,IRB)4个部分依次排列而成(Daniell et al.,2016;李裕华等,2020)。叶绿体基因组一般含有110~130个功能基因,除了少数功能未明确外,绝大多数与植物光合作用和叶绿体自身复制相关(张同武,2012;Zhang et al.,2012;赵振宁等,2023)。叶绿体基因组序列的编码区和非编码区具有不同的进化速率,可提供丰富的分类信息,其中编码区序列的变异水平较低,适用于属、科或更高阶物种之间的分类研究,而非编码区的突变程度较高,可用于属内甚至种内物种的多样性分析(倪梁红等,2015)。目前,叶绿体基因组序列在物种鉴别、多样性分析和系统发育分别等方面已有广泛应用。Choi等(2018)利用中药番杏叶绿体基因组序列探究中药番杏在石竹科内的系统发育关系和进化地位,结果显示番杏属与松叶菊属的亲缘关系更近。Zhou等(2018a)比较中药大黄3种同属基源植物[掌叶大黄(Rheum palmatum)、唐古特大黄(R. tanguticum)和药用大黄(R. officinale)]的叶绿体基因组序列,结果发现三者间存在8个高变异区域,表明叶绿体基因组可用作大黄药材鉴别的超级DNA条形码。王崇等(2021)基于甘薯叶绿体基因组序列开发出11个叶绿体SSR分子标记及其扩增引物,将其应用于104个甘薯品种的遗传多样性分析,并构建其叶绿体SSR指纹图谱。由于选择压力和碱基突变等因素的作用,植物在进化过程中会出现密码子偏好性的现象(Athey et al.,2017)。这种现象普遍存在于植物叶绿体基因组,且具有物种特异性(Ma et al.,2014;原晓龙等,2021)。密码子偏好性与mRNA的转录调控、外源基因的表达及蛋白的翻译速度和准确率密切相关(Chen et al.,2017;Zhou et al.,2018b),从而影响基因及其编码蛋白的结构和功能(Zelasko et al.,2013)。此外,密码子偏好性还可揭示物种之间进化关系,近缘物种的密码子使用模式通常较相似。目前已有大量植物叶绿体基因组密码子偏好性被研究报道。Wang等(2022)比较了40个山茶科物种的密码子偏好性,结果发现其密码子偏好性主要取决于自然选择,且间接影响这些物种特有基因的表达,并基于密码子偏好性可进一步解析物种间的系统发育关系。Li等(2023)分析了3份咖啡种质的密码子偏好性,基于此发现烟草和酒酿酵母是咖啡基因表达的良好宿主,为后续咖啡基因转化和分子育种提供参考。【本研究切入点】密花豆的叶绿体基因组信息匮乏,目前鲜见有关密花豆叶绿体基因组序列特征及密码子偏好性分析的研究报道,限制了密花豆种质资源的保护和开发利用。【拟解决的关键问题】利用高通量测序技术对密花豆的叶绿体基因组全序列进行测序,结合生物信息学方法分析其序列特征,探究密花豆叶绿体基因组的密码子偏好性,并通过构建系统发育进化树解析密花豆的进化地位,为密花豆种质鉴定、分子育种及资源保护利用提供数据支持。
1 材料与方法
1. 1 样本采集及处理
密花豆采集于广西钦州市灵山县,清洗干净后置于硅胶袋中保存。参照吴民华等(2023)的方法,采用磁珠法植物基因组DNA提取试剂盒(北京康为生物科技有限公司)提取密花豆的基因组DNA,并采用NexteraXT DNA文庫制备试剂盒(Illumina,美国)构建的DNA文库,后续用于高通量测序。
1. 2 序列获取、拼接和注释
采用Illumina NovaSeq测序平台进行双末端测序,测得的原始序列经NGS QC Toolkit进行过滤处理,获得高质量读序。运用SPAdes软件以De novo模式进行序列组装,并使用PLANN工具对叶绿体基因进行注释,并将拼接序列和注释信息上传至GenBank数据库(登录号为OQ809071)。最后,使用OGDRAW工具制作叶绿体基因组图谱,使其结构可视化。
1. 3 叶绿体基因组密码子偏好性分析
1. 3. 1 基因筛选 以密花豆叶绿体基因组所有的蛋白编码基因(Protein coding genes,PCGs)为对象,剔除重复序列、长度小于300 bp的序列、非ATG作为起始密码子的序列、终止密码子异常及内部存在终止密码子的序列后,余下的序列用于密码子偏好性分析。
1. 3. 2 密码子組成分析 采用CodonW和CUSP等软件计算PCGs的相对同义密码子使用度(Relative synonymous codon usage,RSCU)、有效密码子数(Effective number of codons,ENC)、总GC含量(GCall)及各编码密码子的首位、次位和末位碱基的GC含量(依次以GC1、GC2和GC3表示)等参数,并利用SPSS 22.0进行各参数间的相关分析。但RSCU>1.00时,表示该密码子使用频率高于其他同义密码子;当RSCU≤1.00时,表示该密码子使用频率等于或高于其他同义密码子。ENC取值范围为20.00~61.00,其值越大,密码子偏好性越弱,反之密码子偏好性越强。
1. 3. 3 绘图分析 采用Origin进行中性绘图分析、ENC-plot分析和PR2-plot分析,以确定影响密花豆叶绿体基因组密码子偏好性的因素。(1)以GC1和GC2的平均值GC12为纵坐标,GC3为横坐标,绘制中性分析散点图,并进行直线拟合,获得回归方程和相关系数(R2),再根据回归系数(即回归方程的斜率)和R2判断密码子偏好性的影响因素。若回归系数趋向0,说明密码子偏好性由自然选择主导;若回归系数趋向1.000,则碱基突变是影响密码子偏好性的主要因素。(2)以ENC作为纵坐标,GC3为横坐标绘制散点图,并参考杨祥燕等(2022)的公式计算各基因的预期ENC(ENCexp),再以ENCexp为横坐标,GC3为横坐标绘制标准曲线,获得ENC-plot分析散点图。若绝大部分基因分布在标准曲线的远处,则说明自然选择是影响密码子偏好性的主要因素,反之,碱基突变是影响密码子偏好性的主要因素。然后计算ENC比值,即ENC比值=(ENCexp-ENC实际)/ENCexp,并统计各组段的基因个数和频率,直观展示ENC-plot的分析结果。(3)以G3/(G3+C3)为横坐标,A3/(A3+T3)为纵坐标,绘制PR2-plot分析散点图,对各密码子第3位碱基的使用差异进行分析。根据图中各点所处的位置判断密码子偏好的方向和程度,若位于中心位置,表示其密码子偏好性不受自然选择的影响。
1. 3. 4 最优密码子确定 将所有用于密码子偏好性分析的基因按ENC从小到大进行排序,在ENC最大和最小两侧各取5个基因分别建成高表达基因组和低表达基因组,计算各密码子在2个表达基因组的RSCU差值(ΔRSCU),参考Wright(1990)的方法,以RSCU>1.00且ΔRSCU>0.08为标准,确定密花豆叶绿体基因组的最优密码子。
1. 4 叶绿体基因组SSR分析
参考吴民华等(2023)设定的参数,分别采用MISA在线工具分析密花豆叶绿体基因组的SSR序列特征。
1. 5 叶绿体基因组比较分析
从GenBank下载的美丽密花豆(Spatholobus pulcher)、紫矿(Butea monosperma)、鸡冠刺桐(Erythrina crista-galli)和常春油麻藤(Mucuna sempervirens)叶绿体基因组序列。分别采用IRcope和mVISTA比较密花豆叶绿体基因组与美丽密花豆、紫矿、鸡冠刺桐和常春油麻藤的叶绿体基因组在全序列和SC/IR边界的差异。
1. 6 系统发育分析
从GenBank下载豆科的美丽密花豆(S. pulcher)、紫矿(B. monosperma)、鸡冠刺桐(E. crista-galli)、常春油麻藤(M. sempervirens)、大果油麻藤(Mucuna macrocarpa)、木豆(Cajanus cajan)、虫豆(Cajanus crassus)、蔓草虫豆(Cajanus scarabaeoides)、苦参(Sophora flavescens)、苦豆子(Sophora alopecuroides)和越南槐(Sophora tonkinensis),蔷薇科的皱皮木瓜(Chaenomeles speciosa)和木瓜(Chaenomeles sinensis),景天科的四叶红景天(Rhodiola quadrifida)和唐古红景天(Rhodiola tangutica)等3个科共15种植物的叶绿体基因序列,并以百合(Lilium brownii)作为外群,采用MAFFT进行多重比对和校正,随后使用RAxML软件基于GTRGAMMA模式构建系统发育进化树。
2 结果与分析
2. 1 叶绿体基因组结构分析结果
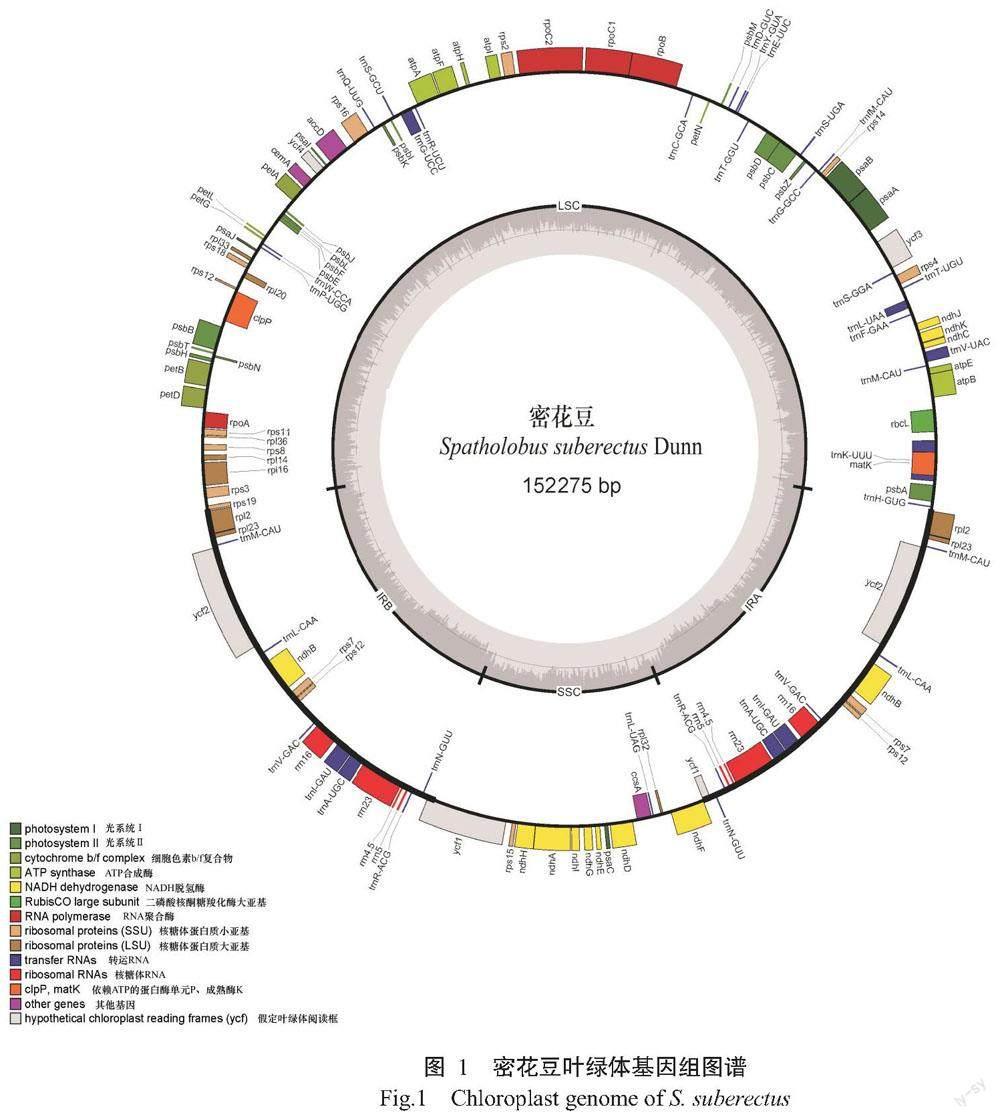
如图1所示,密花豆叶绿体基因组全长为152275 bp,由83924 bp的LSC、25113 bp的IRA、18125 bp的SSC和25113 bp的IRB依次排列而成的环状双链四分体结构。密花豆叶绿体基因组的GC含量为35.2%。
如表1所示,密花豆叶绿体基因组共注释到129个基因,包括84个PCGs、37个tRNA和8个rRNA。从拷贝数上看,除4个PCGs(ndhB、rps7、rpl2和rpl23)、7个tRNA(trnM-CAU、trnL-CAA、trnV-GAC、trnI-GAU、trnA-UGC、trnR-ACG和trnN-GUU)及全部rRNA为双拷贝外,其余基因均为单拷贝。从内含子数量来看,共有16个基因存在1个内含子,包括9个PCGs(atpF、petB、petD、ndhA、ndhB、rps16、rpl2、rpl16和rpoC1)和7个tRNA(trnK-UUU、trnG-UCC、trnL-UAA、trnV-UAC、trnI-GAU和trnA-UGC),有2个PCGs(clpP和ycf 3)各含有2个内含子,其他基因则无内含子。
2. 2 密码子偏好性分析
2. 2. 1 密码子碱基组成分析 从密花豆叶绿体基因组中筛选出42个用于密码子偏好性分析的PCGs,共含有17483个密码子。由表2可知,42个PCGs的GC1为29.75%~57.56%,平均为45.26%,GC2为25.88%~54.68%,平均为38.40%,GC3为20.59%~35.83%,平均为26.27%,GCall为25.95%~43.15%,平均为36.64%,密码子3位碱基的GC含量依次降低,说明密码子以A/T结尾的频率远高于以G/C结尾的频率。ENC为35.25~54.32,平均为44.38,ENC在45.00以上的PCGs数量占50%,说明密花豆叶绿体基因组的密码子使用偏好性不明显。
GC含量、ENC和密码子数量进行相关分析,如表3所示。GC1、GC2和GC3均与GCall呈极显著相关(P<0.01,下同);GC1与GC3呈显著正相关(P<0.05,下同),说明碱基组成有一定的相似性,而GC2与GC3无显著相关性(P>0.05),说明碱基组成存在明显差异。ENC与GC1和GCall均呈显著正相关,与GC3呈极显著正相关,说明ENC主要受GC3影响,与GC1和GCall存在一定的关联。此外,密码子数量与GC2呈显著负相关,且两者对ENC均无明显影响。
2. 2. 2 中性绘图分析 密花豆叶绿体基因组中性绘图分析结果如图2所示。GC12为0.29~0.43,GC3为0.21~0.26,全部基因均处于坐标轴对角线的左上侧,GC12与GC3的相关系数为0.045,回归系数为0.307,说明二者无明显相关性,暗示GC12和GC3的进化方式并无关联。自然选择和碱基突变均影响密花豆叶绿体基因组的密码子偏好性,但自然选择是主要决定因素。
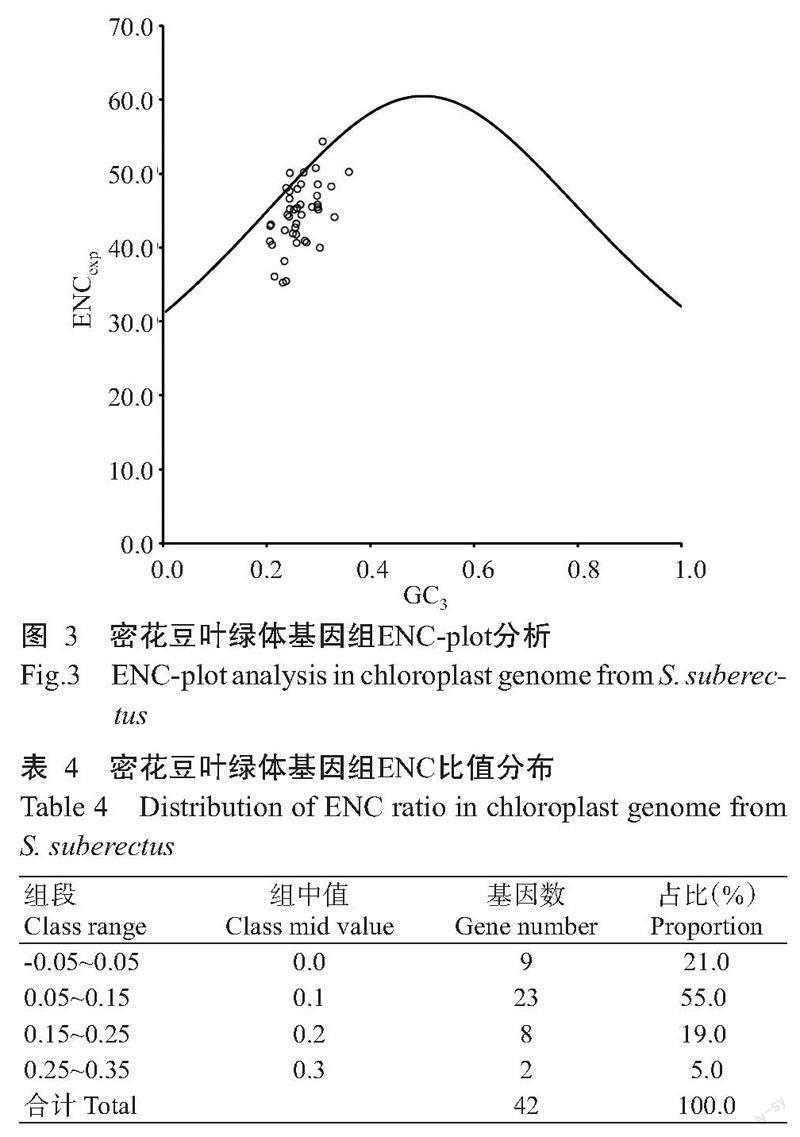
2. 2. 3 ENC-plot分析 密花豆叶绿体基因组ENC-plot分析如图3所示,大多数基因处于标准ENC曲线的下方,ENC实际距离ENCexp较远,仅有少数基因分布在曲线附近,说明密花豆叶绿体基因组的密码子偏好性主要是由自然选择作用的结果,碱基突变的作用较小。由表4可知,ENC比值位于-0.05~0.05区间的基因有9个,这些基因的ENC实际趋近于ENCexp,而位于其他区间的基因有33个,这些基因的ENC实际与ENCexp相差甚远,进一步证实密花豆叶绿体基因组的密码子偏好性主要由自然选择起决定作用。
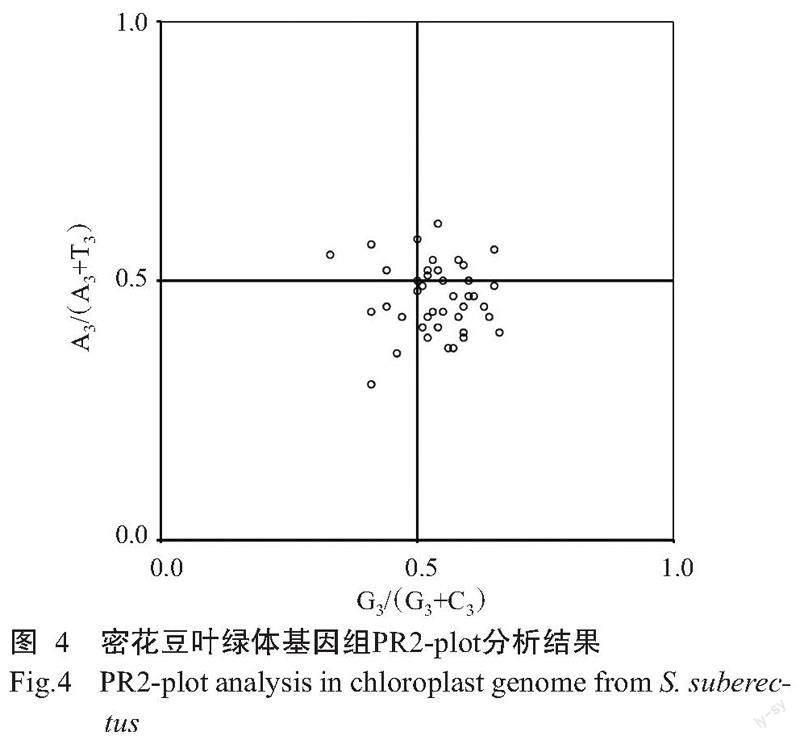
2. 2. 4 PR2-plot分析 由图4可知,图中大多数基因位于右下象限内,说明在密码子末位碱基的使用频率上,T高于A,且G高于C。在其他3个象限内,也有少量基因零散分布,提示密花豆叶绿体基因组的密码子偏好性是多因素共同作用的结果。
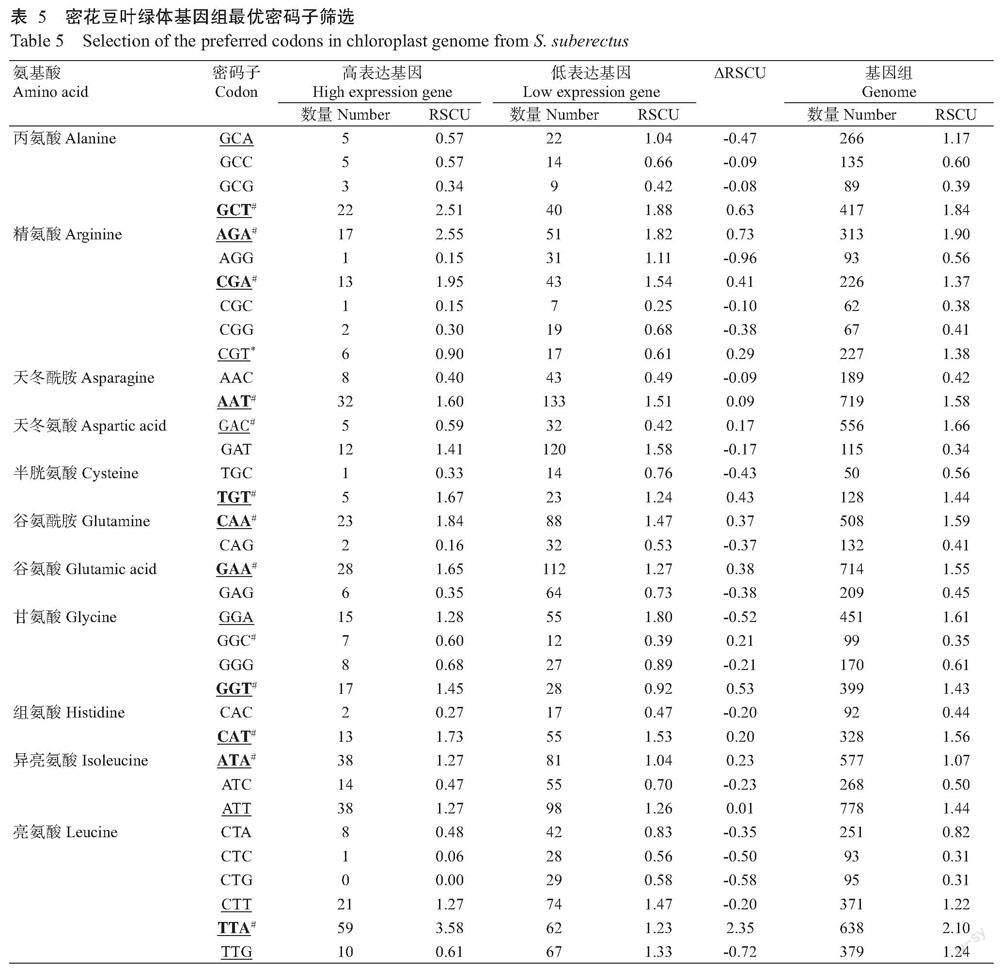
2. 2. 5 最优密码子的确定 密花豆叶绿体基因组编码各种氨基酸的密码子及其RSCU如表5所示。编码亮氨基酸的TTA具有最大RSCU,达2.10,而编码亮氨酸的CTC、CTG和编码丝氨酸的AGC具有最小的RSCU,为0.31。共有30种密码子的RSCU>1.00,其中29种密码子是以A或T结尾,仅有1个以G结尾,即编码亮氨酸的TTG。
由表5还可知,根据高表达基因和低表达基因的RSCU计算所得ΔRSCU,该值大于0.08的密码子有24个。按照预设的最优密码子标准,即ΔRSCU>0.08且RSCU>1.00,最终确定密花豆叶绿体基因组编码各种氨基酸的最优密码子共有18个,分别是GCT、AGA、CGA、AAT、TGT、CAA、GAA、GGT、CAT、ATA、TTA、AAA、TTT、CCT、TCA、ACT、TAT和GTT。
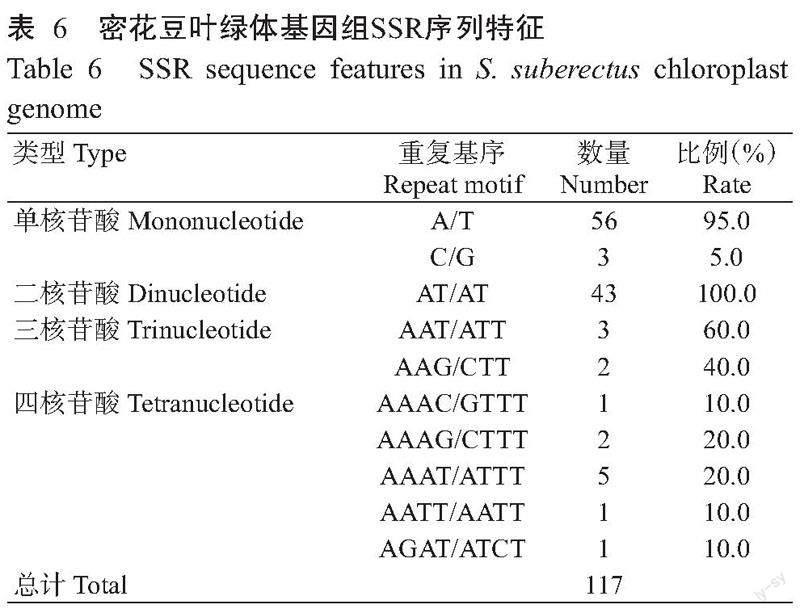
2. 3 SSR序列特征分析结果
由表6可知,密花豆叶绿体基因组共有117个SSR位点,分别由单、二、三、四核苷酸重复基元组成,无五、六核苷酸重复基元。其中单核苷酸重复基元数量最多,占SSR位点总数的50.4%;其次是二核苷酸重复基元,占SSR位点总数的36.8%。在单、二、三、四核苷酸重复基元中,分别以A/T、AT/AT、AAT/ATT和AAAT/ATTT为主要重复基元。可见,密花豆叶绿体基因组SSR主要是由A或T及其组合构成。
2. 4 SC/IR边界比较分析结果
如图5所示,5种植物叶绿体基因组相邻的SC和IR之间均分别形成4个边界,即JLB、JSB、JSA和JLA。除了常春油麻藤的JLB位于rps3和间隔区外,其余植物的JLB均位于rps19基因内,分别在5'端的51、51、49和68 bp处。密花豆和美丽密花豆的JSB均位于ycf1基因中,紫矿的JSB位于ycf1和ndhF基因重叠区,鸡冠刺桐的JSB則位于ycf1和ndhF基因间隔区,而常春油麻藤的JSB位于ndhF基因中。5种植物的JSA均位于ycf1基因的另一个拷贝中,分别在5'端的500、476、475、1249和471 bp处。5种植物的JLA均位于基因间隔区,其中密花豆、美丽密花豆、紫矿和常春油麻藤的JLA位于rpl2基因的另一个拷贝和trnH基因之间,而鸡冠刺桐的JLA则位于rps19和trnH基因之间。
2. 5 序列差异分析结果
豆科蝶形亚科菜豆族刺桐亚族5种植物密花豆、美丽密药豆、紫矿、鸡冠刺桐和常春油麻藤的叶绿体基因组序列差异如图6所示,这些植物的叶绿体基因组之间存在较明显的序列差异,非编码区的碱基突变程度高于基因的编码区,且SC的变异程度较IR大。密花豆与同属的美丽密花豆的核苷酸序列相似性最高,但仍有多个区域存在碱基变异。可见,叶绿体基因组在密花豆属内及属间均有较好的鉴别效果。
2. 6 系统发育分析结果
由图7可知,密花豆与同属的美丽密花豆最先聚在一起,形成密花豆属分支,随后再与紫矿属、木豆属、刺桐属、黧豆属和苦参属共同聚成豆科分支;景天科和蔷薇科植物分别单独聚成一分支。可见,系统发育进化树的聚类效果良好,可直观反映密花豆的进化地位。
3 讨论
随着新一代高通量测序技术的快速发展和序列解析方法的更新迭代,细胞器基因组的序列测定和解读程序更趋容易、成本更加低廉(Heather and Chain,2016)。本研究发现,密花豆叶绿体基因组为长度为152275 bp的环状双链四分体分子,编码129个基因,从功能上看主要是与光合作用和叶绿体自身复制相关的基因,其结构和基因组成与大多数植物如高良姜(黄琼林,2021)等单子叶植物、铁冬青(黄红宝等,2022)等双子叶植物相似,具有植物叶绿体基因组的典型特征。本研究系统地探究了密花豆叶绿体基因组的密码子偏好性,结果发现密码子3个位置的GC含量存在明显差异,且GC1>GC2>GC3,说明密码子首位、次位和末位的碱基组成不同,且末位碱基以A/T居多,与千果榄仁(余潇等,2023)、金银花大毛花(张加强等,2023)等植物的研究结果一致。通过绘图分析,发现密花豆叶绿体基因组的密码子偏好性主要是自然选择的结果,而碱基突变以及其他因素也参与该进化过程,也是造成楸树(李凤等,2021)、菠萝(杨祥燕等,2022)等多种植物密码子偏好性的原因。密花豆叶绿体基因组RSCU>1.00的密码子中有96.7%是以A或T结尾,而且确定的最优密码子均以A或T结尾,表明密花豆在叶绿体进化过程中偏好使用以A或T结尾的密码子。相同情况也出现在露兜树(吴民华等,2023)、峨眉凤仙花(赵秋燕等,2023)等植物中。上述结果暗示,陆生植物叶绿体基因组具有较保守的GC含量和密码子使用偏好性。
SSR是植物基因组存在一种短的高频重复DNA序列,是植物分子育种、遗传进化分析等研究中常用的分子标记(黄琼林,2022)。本研究在密花豆的叶绿体基因组中共找到117个SSR位点,主要分布在LSC和基因间隔区,且以A/T单核苷酸重复为主要类型,推测与密花豆叶绿体基因组的低GC含量相关。密花豆的SSR分布和类型与闭鞘姜(吴永飞,2023)、祁连圆柏叶(赵明等,2023)等大多数植物相似。
在前人研究中,黄琼林等(2015,2016)利用叶绿体基因matK和rbcL成功鉴别密花豆(鸡血藤)与大血藤、常春油麻藤等常见易混淆品,证实了叶绿体基组序列应用于密花豆品种鉴定的可行性。本研究序列比较分析也发现,密花豆不仅与常春油麻藤等属外植物的叶绿体基因组存在较明显的序列差异,与同属的美丽密花豆也有多处碱基突变,表明叶绿体基因组是探究密花豆真伪鉴别和系统关系的理想分子工具。
由于叶绿体基因组是研究被子植物系统进化关系和分类地位的理想工具(Song et al.,2019)。本研究基于叶绿体基因组分析密花豆在豆科内的进化地位,结果发现密花豆与美丽密花豆最先形成独立分支,形成单系类群,说明二者的亲缘关系最近,与传统植物分类相符(中国植物志编辑委员会,1995)。在科内,密花豆属则与紫矿属的分类位置更近。目前GenBank数据库中可获取的密花豆属植物叶绿体基因组信息尚少,后续仍需利用更多的密花豆属植物叶绿体基因组序列来构建密花豆的系统发育进化树,以期全面地理解密花豆及近缘物种的进化关系,为密花豆的分子辅助育种提供参考。
叶绿体基因组不仅是研究植物物种鉴定和系统进化的理想工具,也是基因遗传转化的良好介质。自1988年首次在衣藻中成功实现叶绿体遗传转化后(Boynton et al.,1988),拟南芥、烟草和番茄等多种高等植物也先后实现了基因遗传转化(崔柳青等,2012)。随着药用植物的叶绿体基因组序列相继研究和解析,基于叶绿体基因工程培养高产、优质、强抗逆性的药用植物优势植株有望实现。本研究开展的密花豆叶绿体基因组测序及序列特征分析,为密花豆的叶绿体遗传转化提供丰富的基因信息,叶绿体基因工程将是今后密花豆品种选育、性状改良和增产丰产的重要研究方向之一,进而缓解密花豆的资源匮乏难题。
4 结论
密花豆叶绿体基因组具有植物叶绿体基因组典型的结构特点,在密花豆属内及属间均有较好的鉴别效果,且其密码子偏好性是多因素共同作用的结果,其中自然选择是主要决定因素,但碱基突变以及其他因素对密码子使用偏好也有一定影响。
参考文献:
崔柳青,李一帆,潘卫东. 2012. 叶绿体基因工程研究进展[J]. 生物技术通报,(6):1-6. [Cui L Q,Li Y F,Pan W D. 2012. Progress of studies on chloroplast genetic engineering[J]. Biotechnology Bulletin,(6):1-6.] doi:10. 13560/j.cnki.biotech.bull.1985.2012.06.025.
樊守金,郭秀秀. 2022. 植物叶绿体基因组研究及应用进展[J]. 山东师范大学学报(自然科学版),37(1):22-31. [Fan S J,Guo X X. 2022. Advances in research and application of plant chloroplast genome[J]. Journal of Shandong Normal University (Natural Science),37(1):22-31.] doi:10.3969/j.issn.1001-4748.2022.01.003.
國家药典委员会. 2020. 中华人民共和国药典(一部)[S]. 北京:中国医药科技出版社. [Chinese Pharmacopoeia Commission. 2020. Pharmacopoeia of the People’s Republic of China (Volume 1)[S]. Beijing:China Medical Science Press.]
黄红宝,何应会,黄欣,孙开道,秦波,黄耀恒. 2022. 铁冬青叶绿体全基因组及系统进化分析[J]. 农业研究与应用,35(5):7-14. [Huang H B,He Y H,Huang X,Sun K D,Qin B,Huang Y H. 2022. Chloroplast whole genome of Ilex rotunda and its phylogenetic analysis[J]. Agricultural Research and Application,35(5):7-14.] doi:10.3969/j.issn. 2095-0764.2022.05.002.
黄琼林,马新业,詹若挺,陈蔚文. 2015. 鸡血藤及其混伪品matK基因分析和分子鉴定[J]. 北方园艺,(17):94-98. [Huang Q L,Ma X Y,Zhan R T,Chen W W. 2015. Comparative analysis and molecular identification of matK gene from Spatholobus suberectus and its adulterants[J]. Northern Horticulture,(17):94-98.] doi:10.11937/bfyy.201517024.
黄琼林,马新业,詹若挺,陈蔚文. 2016. 基于rbcL条形码的鸡血藤真伪鉴别[J]. 江苏农业科学,44(6):57-60. [Huang Q L,Ma X Y,Zhan R T,Chen W W. 2016. Authentication of Spatholobus suberectus and its adulterants based on rbcL barcode[J]. Jiangsu Agricultural Science,44(6):57-60.] doi:10.15889/j.issn.1002-1302.20012.016.06.01.
黄琼林. 2021. 高良姜叶绿体基因组测序与特征分析[J]. 热带作物学报,42(1):1-6. [Huang Q L. 2021. Complete sequencing and analysis of chloroplast genome from Alpinia officinarum Hance[J]. Chinese Journal of Tropical Crop,42(1):1-6.] doi:10.3969/j.issn.1000-2561.2021. 01.001.
黄琼林. 2022. 高良姜转录组简单重复序列位点分析[J]. 时珍国医国药,33(2):386-389. [Huang Q L. 2022. Analysis on SSR loci from Alpinia officinarum transcriptome[J]. Lishizhen Medicine and Materia Medica Research,33(2):386-389.] doi:10.3969/j.issn.1008-0805.2022.02.33.
李凤,辛静,辛雅萱,肖遥,屈亚亚,王军辉,麻文俊,辛培尧. 2021. 楸树叶绿体基因组密码子偏性分析[J]. 南方农业学报,52(10):2735-2743. [Li F,Xin J,Xin Y X,Xiao Y,Qu Y Y,Wang J H,Ma W J,Xin P Y. 2021. Codon usage bias of chloroplast genome in Catalpa bungei C. A. Mey.[J]. Journal of Southern Agriculture,52(10):2735-2743.] doi:10.3969/j.issn.2095-1191.2021.10.013.
李裕華,任永康,赵兴华,刘江,韩斌,王长彪,唐朝晖. 2020. 禾本科主要农作物叶绿体基因组研究进展[J]. 生物技术通报,36(11):112-121. [Li Y H,Ren Y K,Zhao X H,Liu J,Han B,Wang C B,Tang Z H. 2020. Research progress on chloroplast genome of major gramineous crops[J]. Biotechnology Bulletin,36(11):112-121.] doi:10. 13560/j.cnki.biotech.bull.1985.2020- 0285.
倪梁红,赵志礼,米玛. 2015. 药用植物叶绿体基因组研究进展[J]. 中药材,38(9):1990-1994. [Ni L H,Zhao Z L,Mi M. 2015. Research progress of chloroplast genome in medicinal plants[J]. Journal of Chinese Medicinal Materials,38(9):1990-1994.]doi:10.13863/j.issn1001-4454. 2015.09.046.
荣广天. 2016. 密花豆种质资源ISSR分析[D]. 长沙:中南林业科技大学. [Rong G T. 2016. ISSR analysis on the diversity of Spatholobus suberectus germplasms[D]. Chang-sha:Central South University of Forestry & Technology.]
孙孟涛,张峻鑫,黄体冉,杨明峰,马兰青,段留生. 2022. 虎杖叶绿体基因组结构与变异分析[J]. 生物工程学报,38(5):1953-1964. [Sun M T,Zhang J X,Huang T R,Yang M F,Ma L Q,Duan L S. 2022. Genome structure and variation of Reynoutria japonica Houtt. chloroplast genome[J]. Chinese Journal of Biotechnology,38(5):1953-1964.] doi:10.13345/j.cjb.210843.
王崇,王连军,杨新笋,雷剑,柴沙沙,张文英,焦春海,田小海. 2021. 104个甘薯品种的cpSSR指纹图谱构建及遗传多样性分析[J].热带作物学报,42(6):1549-1556. [Wang C,Wang L J,Yang X S,Lei J,Chai S S,Zhang W Y,Jiao C H,Tian X H. 2021. Construction of cpSSR fingerprints and genetic diversity analysis of 104 sweet potato varieties[J]. Chinese Journal of Tropical Crops,42(6):1549-1556.] doi:10.3969/j.issn.1000-2561.2021. 06.006.
王化坤,娄晓鸣,章镇. 2006. 叶绿体微卫星在植物种质资源研究中的应用[J]. 分子植物育种, 4(S1):92-98. [Wang H K,Lou X M,Zhang Z. 2006. Application in germplasm resource research using chloroplast simple sequence repeat[J]. Molecular Plant Breeding,4(S1):92-98.]
吴民华,邹振宁,叶晓霞,梁艳清,陈妹,黄琼林. 2023. 露兜树叶绿体基因组结构与序列特征分析[J]. 中药新药与临床药理,34(1):115-122. [Wu M H,Zou Z N,Ye X X,Liang Y Q,Chen M,Huang Q L. 2023. Structure and sequence characteristics of chloroplast genome from Pandanus tectorius[J]. Traditional Chinese Drug Research and Clinical Pharmacology,34(1):115-122.] doi:10.19378/j.issn.1003-9783.2023.01.016.
吴永飞,杨雪莲,颜丽,王霞,胡小京. 2023. 闭鞘姜叶绿体基因组特征及其系统发育分析[J]. 种子, 42(4):10-17. [Wu Y F,Yang X L,Yan L,Wang X,Hu X J. 2023. Analysis on chloroplast genome characteristics and phylogeny of Helenia speciosa[J]. Seed,42(4):10-17.] doi:10.16590/j.cnki.1001-4705.2023.04.010.
萧晓吉,李苗苗,詹若挺,韩正洲,刘军民. 2022. 密花豆野生及栽培资源调查与分析[J]. 中药材,45(7):1592-1297. [Xiao X J,Li M M,Zhan R T,Han Z Z,Liu J M. 2022. Resource investigation and analysis of wild and cultivated Spatholobus suberectus[J]. Journal of Chinese Medicinal Materials,45(7):1592-1597.] doi:10.13863/j.issn 1001-4454.2022.07.010.
楊祥燕,蔡元保,谭秦亮,覃旭,黄显雅,吴密. 2022. 菠萝叶绿体基因组密码子偏好性分析[J]. 热带作物学报,43(3):439-446. [Yang X Y,Cai Y B,Tan Q L,Qin X,Huang X Y,Wu M. 2022. Analysis of codon usage bias in the chloroplast genome of ananas comosus[J]. Chinese Journal of Tropical Crops,43(3):439-446.] doi:10.3969/j.issn.1000-2561. 2022.03.001.
余潇,赵振宁,杜春. 2022. 千果榄仁叶绿体基因组特征及密码子偏好性分析[J]. 山西农业大学学报(自然科学版),42(6):81-92. [Yu X,Zhao Z N,Du C. 2022. Analysis of the complete chloroplast genome sequence characteristics and its code usage bias of Terminalia myriocarpa[J]. Journal of Shanxi Agricultural University(Natural Science Edition),42(6):81-92.]doi: 10.13842/j.cnki.issn 1671-8151.202207004.
原晓龙,刘音,康洪梅,陈中华,李云琴,王毅. 2021. 蒜头果叶绿体基因组密码子偏好性分析[J]. 西南林业大学学报
(自然科学),41(3):15-22. [Yuan X L,Liu Y,Kang H M,Chen Z H,Li Y Q,Wang Y. 2021. Analysis of codon usage bias in chloroplast genome of Malania oleifera[J]. Journal of Southwest Forestry University,41(3):15-22.] doi:10.11929/j.swfu.202001047.
张加强,刘慧春,王杰,许雯婷,周江华,周开元. 2023. 金银花大毛花叶绿体基因组密码子的偏好性分析[J]. 浙江农业学报,35(4):821-830. [Zhang J Q,Liu H C,Wang J,Xu W T,Zhou J H,Zhou K Y. 2023. Analysis on codon usage patterns of chloroplast genome of Lonicera japonica cv. Damaohua[J]. Acta Agriculturae Zhejiangensis,35(4):821-830.] doi:10.3969/j.issn.1004-1524.2023.04.09.
张同武. 2012. 植物细胞器基因组测序、组装及比较基因组学研究[D]. 杭州:浙江大学. [Zhang T W. 2012. The whole genome sequencing,assembly and comparative genomic research of plant organelle[D]. Hangzhou:Zhe jiang University.]
趙明,张宏斌,李伟,赵祜,刘建海,赵国生,赵兴鹏,吕东. 2023. 祁连圆柏叶绿体基因组序列特征分析[J]. 植物资源与环境学报,32(3):1-11. [Zhao M,Zhang H B,Li W,Zhao H,Liu J H,Zhao G S,Zhao X P,Lü D. 2023. Ana-lysis on chloroplast genome sequence characteristics of Juniperus przewalskii[J]. Journal of Plant Resource and Environment,32(3):1-11.] doi:10.3969/j.issn.1674-7895.2023.03.01.
赵秋燕,曹孟会,李新艺,周敏,魏春梅,张茜,瞿素萍,黄海泉,黄美娟. 2023. 濒危植物峨眉凤仙花叶绿体基因组分析[J]. 福建农业学报,38(2):174-182. [Zhao Q Y,Cao M H,Li X Y,Zhou M,Wei C M,Zhang X,Zhai S P,Huang H Q,Huang M J. 2023. Complete chloroplast genome of endangered Impatiens omeiana[J]. Fujian Journal of Agricultural Sciences,38(2):174-182.] doi:10. 19303/j.issn.1008-0384.2023.02.007.
赵振宁,孙浩田,宋雨茹,余潇. 2023. 山楂属植物叶绿体基因组特征与密码子偏好性分析[J]. 江苏农业学报,39(2):504-517. [Zhao Z N,Sun H T,Song Y R,Yu X. 2023. Chloroplast genome characteristics and codon usage bias analysis of Crataegus L.[J]. Jiangsu Journal of Agricultural Sciences,39(2):504-517.] doi:10.3969/j.issn.1000-4440. 2023. 02.024.
中国植物志编辑委员会. 1995. 中国植物志(41卷)[M]. 北京:科学出版社:190-192. [Committee of Flora of China. 1995. Flora of China (Volume 41)[M]. Beijing:Science Press:190-192.]
Athey J,Alexaki A,Osipova E,Rostovtsev A,Santana-Quintero L V,Katneni U,Simonyan V,Kimchi-Sarfaty C. 2017. A new and updated resource for codon usage tables[J]. BMC Bioinformatics,18:391. doi:10.1186/s12859-017-1793-7.
Boynton J E,Gillham N W,Harris E H,Hosler J P,Johnson A M,Jones A R,Randolph-Anderson B L,Robertson D,Klein T M,Shark K B. 1988. Chloroplast transformation in Chlamydomonas with high velocity microprojectiles[J]. Science,240(4858):1534-1538. doi:10.1126/science. 2897716.
Chen S Y,Li K,Cao W Q,Wang J,Zhao T,Huan Q,Yang Y F,Wu S H,Qian W F. 2017. Codon-resolution analysis reveals a direct and context-dependent impact of indivi-dual synonymous mutations on mRNA level[J]. Molecular Biology and Evolution,34(11):2944-2958. doi:10.1093/molbev/msx229.
Choi K S,Kwak M,Lee B,Park S. 2018. Complete chloroplast genome of Tetragonia tetragonioides:Molecular phylogenetic relationships and evolution in Caryophyllales[J]. PLoS One,13(6):e0199626. doi:10.1371/journal.pone. 0199626.
Daniell H,Lin C S,Yu M,Yu M,Chang W. 2016. Chloroplast genomes:Diversity,evolution,and applications in genetic engineering[J]. Genome Biology,17(1):134. doi:10.1186/s13059-016-1004-2.
Heather J M,Chain B. 2016. The sequence of sequencers:The history of sequencing DNA[J]. Genomics,107(1):1-8. doi:10.1016/j.ygeno.2015.11.003.
Li Y Q,Hu X,Xiao M K,Huang J X,Lou Y Q,Hu F G,Fu X F,Li Y N,He H Y,Cheng J H. 2023. An analysis of codon utilization patterns in the chloroplast genomes of three species of Coffea[J]. BMC Genomics Data,24(1):42. doi:10.1186/s12863-023-01143-4.
Ma L N,Cui P,Zhu J,Zhang Z H,Zhang Z. 2014. Translational selection in human:More pronounced in housekeeping genes[J]. Biology Direct,9:17. doi:10.1186/1745-6150-9-17.
Song Y,Zhang Y J,Xu J,Li W M,Li M F. 2019. Characteri-zation of the complete chloroplast genome sequence of Dalbergia species and its phylogenetic implications[J]. Scientific Report,9(1):20401. doi:10.1038/s41598-019-56727-x.
Wang Z J,Cai Q W,Wang Y,Li M H,Wang C C,Wang Z X,Jiao C Y,Xu C C,Wang H Y,Zhang Z L. 2022. Compara-tive analysis of codon bias in the chloroplast genomes of Theaceae species[J]. Frontiers in Genetics,13:824610. doi:10.3389/fgene.2022.824610.
Wright F. 1990. The ‘effective number of codons’ used in a gene[J]. Gene,87(1):23-29. doi:10.1016/0378-1119(90)90491-9.
Zelasko S,Palaria A,Das A. 2013. Optimizations to achieve high-level expression of cytochrome P450 proteins using Escherichia coli expression systems[J]. Protein Expression and Purification,92(1):77-87. doi:10.1016/j.pep. 2013.07.017.
Zhang T W,Fang Y J,Wang X M,Deng X,Zhang X W,Hu S N,Yu J. 2012. The complete chloroplast and mitochondrial genome sequences of Boea hygrometrica:insights into the evolution of plant organellar genomes[J]. PLoS One,7(1):e30531. doi:10.1371/journal.pone.0030531.
Zhou Y X,Nie J,Xiao L,Hu Z G,Wang B. 2018a. Comparative chloroplast genome analysis of rhubarb botanical origins and the development of specific identification mar-kers[J]. Molecules,23(11):2811. doi:10.3390/molecules 23112811.
Zhou Z,Dang Y,Zhou M,Yuan H,Liu Y. 2018b. Codon usage biases co-evolve with transcription termination machinery to suppress premature cleavage and polyadenylation[J]. Elife,7:e33569. doi:10.7554/eLife.33569.
(責任编辑 陈 燕)
收稿日期:2023-04-28
基金项目:广东省基础与应用基础研究基金项目(2018A030310116);湛江市科技计划项目(2019A01008)
通讯作者:黄琼林(1986-),https://orcid.org/0000-0001-5248-8253,博士,副教授,主要从事医学生物化学研究工作,E-mail:perfecthql @163.com
第一作者:吴民华(1981-),httpss://orcid.org/0000-0003-4454-5507,博士,副教授,主要从事中药分子药理研究工作,E-mail:wugdmczp @gdmu.edu.cn
