大黄鱼Cspg4基因的表达调控
2023-10-31罗阿蓉李完波王志勇
罗阿蓉,李完波,王志勇
(集美大学水产学院、农业农村部东海海水健康养殖重点实验室,福建厦门 361021)
0 引言
大黄鱼是我国养殖量最大的海水鱼类,但各种病害频发,而且病原复杂多样、防控困难,使养殖业遭受巨大损失。从大黄鱼自身的免疫机制入手,阐明其免疫抗病机制,可为制定防治措施提供理论基础[1-3]。本课题组前期对81尾1.5龄健康大黄鱼脾脏进行了转录组测序,结合全基因组测序数据进行表达数量性状位点分析(expression quantitative trait locus,eQTL),发掘出诸多调控免疫相关靶基因的顺式作用eQTL(未发表数据),这些靶基因有的在免疫系统中充当关键枢纽,如参与免疫调节的硫酸软骨素蛋白聚糖4(chondroitin sulfate proteoglycan 4,Cspg4)基因。调控Cspg4基因的eQTL中最显著的单核苷酸多态性(single nucleotide polymorphism,SNP)位点位于脂质磷酸磷酸酶相关蛋白1型(lipid phosphate phosphatase-related protein type 1,lppr1)的内含子中。
Cspg4是一种由硫酸软骨素和核心蛋白组成的具有多个亚结构的跨膜蛋白,在细胞生长、连接、迁移和细胞外基质中起作用。近几年的研究发现,Cspg4基因在人类多种恶性肿瘤细胞及组织中高表达,如恶性黑色素瘤[4]、成胶质细胞瘤[5]、乳腺癌[6]、头颈肿瘤[7]、胰腺肿瘤[8]和难治性混合性白血病[9]等。它能促进肿瘤细胞的多种生物学行为,如增殖、运动、侵袭和抗凋亡,这些行为在肿瘤的发生发展过程中具有重要作用[10-11]。人类和犬类的Cspg4氨基酸序列具有82%的同源性,Riccardo等[12]研究发现人的Cspg4 DNA能使犬口腔恶性黑色素瘤中Cspg4特异性免疫和生存期延长,针对Cspg4的异种疫苗能够克服宿主对“自我”抗原的无反应性,对治疗犬类恶性黑色素瘤初步有效。另外研究者们利用模式生物也对Cspg4基因进行了多方面的研究。Lee等[13]发现Cspg4在斑马鱼胚胎发育中通过调控Wnt/平面细胞极性信号通路调控体轴组织,而Cspg4的过表达还会导致睫状视网膜动脉阻塞。受Cspg4基因座的转录调节,在没有心功能不全或纤维脂肪症、掌跖角化病和脱发的情况下,桥粒斑蛋白(desmoplakin,Dsp)的缺失可导致小鼠致死性心律失常,类似人类的心脏皮肤综合症[14]。鼠体内还存在Cspg4同源蛋白的神经元胶原抗原(neuron-glia antigen2,Ng2)。Zhu等[15]利用细菌人工染色体修饰技术,制备了在Ng2阳性(Ng2+)细胞中特异性表达红色荧光蛋白DsRed或噬菌体Cre重组酶的转基因小鼠,结果发现Ng2+细胞在体内外均可形成原生质星形胶质细胞和少突胶质细胞。Wilms等[16]证明脊椎动物特异性氧化还原酶谷氧还蛋白2 (Grx2c)的胞浆异构体调节转录因子Sp-1的氧化还原状态,使其与Cspg4基因的启动子和增强子区域结合,导致编码蛋白水平的增加,从而增强了胶质细胞和胶质瘤细胞的迁移能力。
本研究就上述Cspg4基因的表达调控机制进行初步探索。通过PCR产物的Sanger测序验证该eQTL区域中最显著的SNP位点的真实性,随后构建Cspg4基因候选启动子区不同长度缺失片段的重组质粒,利用双荧光素酶报告系统检测重组质粒的活性并确定Cspg4基因的核心转录活性区域,在此基础上插入此基因的不同单倍型调控序列进行验证,为揭示该eQTL与靶基因之间的表达调控方式打下基础。
1 材料与方法
1.1 实验材料
1.1.1 实验动物
本实验室前期对81尾1.5龄的健康大黄鱼进行了全基因组重测序(从鳍条提取DNA)和转录组测序(从脾脏提取RNA),所用大黄鱼来自宁德市三都澳养殖渔排。DNA和RNA的提取按照常规方法进行,动物实验操作遵循集美大学水产学院动物伦理委员会的要求进行。
1.1.2 菌株和细胞
大肠杆菌菌株DH5α CompetentE.coliStrain购自南京诺唯赞生物科技有限公司;萤火虫pGL3-basic载体菌株、内参海参pRL-TK载体菌株为本实验室保存,HEK 293T细胞购自上海富衡生物科技有限公司,经传代、冻存保存于本实验室。
1.1.3 引物
本研究所用引物由厦门铂瑞生物科技有限公司和生工生物工程(上海)有限公司合成,其名称及序列如表1所示。

表1 本研究所用引物序列及名称Tab.1 Sequence and name of primers used in this experiment
1.2 实验方法
1.2.1 SNP位点验证
前期研究获得了调控Cspg4基因的顺式eQTL,其中最显著的eQTL位于大黄鱼基因组17号染色体13952653 bp的一个SNP(Chr17_13952653)上。为了检验该SNP是否真实存在而非测序错误,搜寻用于eQTL分析的81个个体的全基因组重测序数据,获取该SNP位点的基因型信息,并进行汇总统计。同时,从参考基因组上获取该SNP两侧各250 bp的序列,使用NCBI网站“Primer-BLAST”在线工具分别设计PCR引物(chr17_13952653-F/R,见表1)。然后,根据基因组重测序获得该SNP位点的基因型,每个位点、每种基因型分别挑选若干个体的鳍条DNA 为模板,使用高保真 Taq 酶进行 PCR 扩增,扩增产物送至厦门铂瑞生物科技有限公司进行Sanger测序,并将测序结果与基因组重测序结果进行比较。PCR反应程序如下:95 ℃ 3 min;95 ℃ 15 s,55 ℃ 20 s,72 ℃ 30 s,35个循环;72 ℃ 5 min;4 ℃保存。取 2 μL PCR反应后产物,用 2%琼脂糖凝胶电泳检测扩增产物是否条带单一。
1.2.2Cspg4启动子活性分析
从大黄鱼参考基因组中获得Cspg4基因启动子区序列信息,以大黄鱼鳍条DNA为模板,扩增Cspg4候选启动子序列。为了鉴定大黄鱼Cspg4启动子序列中的核心区域,对其进行不同长度片段的截短。使用2× Phanta©Max Master Mix,带有SmaⅠ和HindⅢ两个酶切位点的相应引物(pGL3-Cspg4-P-R,pGL3-Cspg4-P1~P7-F,见表1)分别进行扩增,以获得全长启动子和各个截短的片段。PCR反应程序如下:95 ℃ 3 min;95 ℃ 15 s,58 ℃ 20 s,72 ℃ 4 min,35个循环;72 ℃ 5min;10 ℃保存。根据各试剂盒说明书进行PCR产物纯化和pGL3-Cspg4-P1~P7重组质粒的构建及鉴定。构建成功的重组质粒转染至HEK 293T细胞并进行双荧光素酶活性测定。
为预测大黄鱼Cspg4基因的5' UTR上游候选启动子序列中潜在核心区域的分布,本研究使用在线程序神经网络启动子Neural Network Promoter Prediction预测BDGP,阈值设为0.8。
1.2.3 Chr17_13952653位点不同单倍型序列对Cspg4基因转录的调控作用验证
通过上述Cspg4启动子活性分析实验确定启动子的核心区域后,在核心区前插入含Chr17_13952653位点的序列,以此来研究包含该SNP位点的序列对靶基因Cspg4启动子的调控情况。
根据大黄鱼参考基因组设计引物(pGL3-chr17-1kb-F/R,见表1)扩增包含Chr17_13952653位点的序列,其片段大小为1157 bp,2条引物分别带有MluI与SmaI酶切位点。从81个重测序样品中选取Chr17_13952653位点基因型为AA和CC的个体,以其鳍条DNA为模板,使用2× Phanta©Max Master Mix进行PCR扩增,PCR反应程序如下:95 ℃ 3 min;95 ℃ 15 s,55 ℃ 20 s,72 ℃ 70 s,35个循环;72 ℃ 5min;4℃保存。根据各试剂盒说明书进行PCR产物纯化和pGL3-Cspg4-A/C重组质粒的构建及鉴定。构建成功的重组质粒转染至HEK 293T细胞并进行双荧光素酶活性测定。
1.2.4Cspg4基因不同单倍型调控序列生物信息学分析
为了计算包含Chr17_13952653位点的调控序列内的连锁不平衡,本研究提取区域内所有的SNP及InDel并进行可视化展示,具体步骤如下:
1)用GATK V3.8[17]的HaplotypeCaller模块,提取含Chr17_13952653位点的调控序列1157 bp内所有的SNP及InDel,用CombineVariants模块合并VCF文件。
2)通过PopLDdecay V3.41[18]计算该区域内的连锁不平衡,即每个SNP或InDel之间的r2值,统计与Chr17_13952653该SNP强连锁的SNP或InDel。
3)通过R 4.0.3中的TrackViewer包,对强连锁的SNP或InDel在基因组上的位置,进行可视化展示。
2 结果与分析
2.1 最显著eQTL突变位点的验证
2.1.1 最显著SNP位点所在短片段序列扩增与测序结果
含Chr17_13952653 SNP位点的短片段扩增产物电泳检测结果如图1所示,在266 bp左右有单一条带,符合预期结果。将上述扩增产物进行Sanger测序,测序结果如图2所示,发现各测序样品该SNP位点的基因型与基因组重测序获得的结果完全一致,纯合子测序峰图为单峰,杂合子测序峰图为双峰。以上结果表明该显著SNP位点均真实存在于实验群体中。


2.1.2 最显著eQTL位点的基因型统计
从81个全基因组重测序数据中,获取显著影响Cspg4基因表达的Chr17_13952653位点的基因型,结果显示Chr17_13952653位点为A/C突变,A的频率为0.34,C的频率为0.66。其中AA基因型个体4个,AC基因型个体47个,CC基因型个体30个。
2.2 Cspg4启动子活性分析
2.2.1Cspg4候选启动子序列分析及潜在核心区域预测
本研究成功扩增到Cspg4基因2076 bp的启动子序列,另包含20 bp的外显子1的部分编码序列,具体序列信息见图3。使用在线程序BDGP启动子预测大黄鱼Cspg4候选启动子序列中潜在的核心区域,结果表明有1个潜在的核心区域存在于该基因的候选启动子序列中,位置及得分见表2,序列具体位置如图3所示。

表2 大黄鱼Cspg4候选启动子序列中预测的核心区域的分布Tab.2 The distribution of predicted core regions within the candidate promoter sequence of Cspg4 in large yellow croaker

2.2.2Cspg4候选启动子区活性分析
双荧光素酶报告系统分析结果显示,Cspg4基因候选启动子区不同长度缺失片段的相对荧光活性由强到弱的顺序为:P4、P3、P7、P2、P1、P6、P5(见图4),其中pGL3-Cspg4-P4重组质粒的启动子活性最高。pGL3-Cspg4-P1~P3重组质粒相较于pGL3-Cspg4-P4的启动子活性逐渐降低,表明在-2076~-1202区域可能存在负调控元件(negative regulatory element,NRE);在-1202~-762和-190~+20区域可能为该启动子的核心区域,存在基本调控元件;而pGL3-Cspg4-P5~P7重组质粒的启动子活性逐渐增强,表明在-762~-190区域也可能存在负调控元件。

2.3 Chr17_13952653位点不同单倍型序列对Cspg4基因转录的调控作用
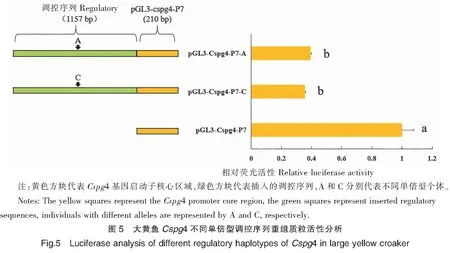
据上述大黄鱼Cspg4候选启动子区活性分析,推测可知在-1202~-762和-190~+20区域可能为该启动子的核心区域。本研究选择pGL3-Cspg4-P7(-190~+20)作为Cspg4基因的核心区域,在此核心区域前插入含Chr17_13952653位点共1157 bp的调控序列,以此来研究该序列对靶基因Cspg4启动子的表达调控情况。
双荧光素酶报告系统分析结果显示,pGL3-Cspg4-P7-A和pGL3-Cspg4-P7-C重组质粒的相对荧光活性均显著低于对照组pGL3-Cspg4-P7,而pGL3-Cspg4-P7-A和pGL3-Cspg4-P7-C重组质粒之间相对荧光活性无显著差别,pGL3-Cspg4-P7-C重组质粒活性略低(如图5,P<0.05),表明含Chr17_13952653 位点的1157 bp序列中可能含有减弱启动子活性的调控序列,即含有沉默子。
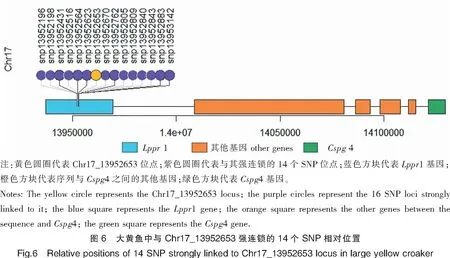
连锁不平衡分析发现,包含Chr17_13952653 位点的1157 bp 序列中有14个r2=1的与Chr17_13952653位点强连锁的SNP位点(见表3、图6)。这提示该序列对Cspg4基因转录的弱化作用,可能也涉及诸多强连锁碱基的协同作用。
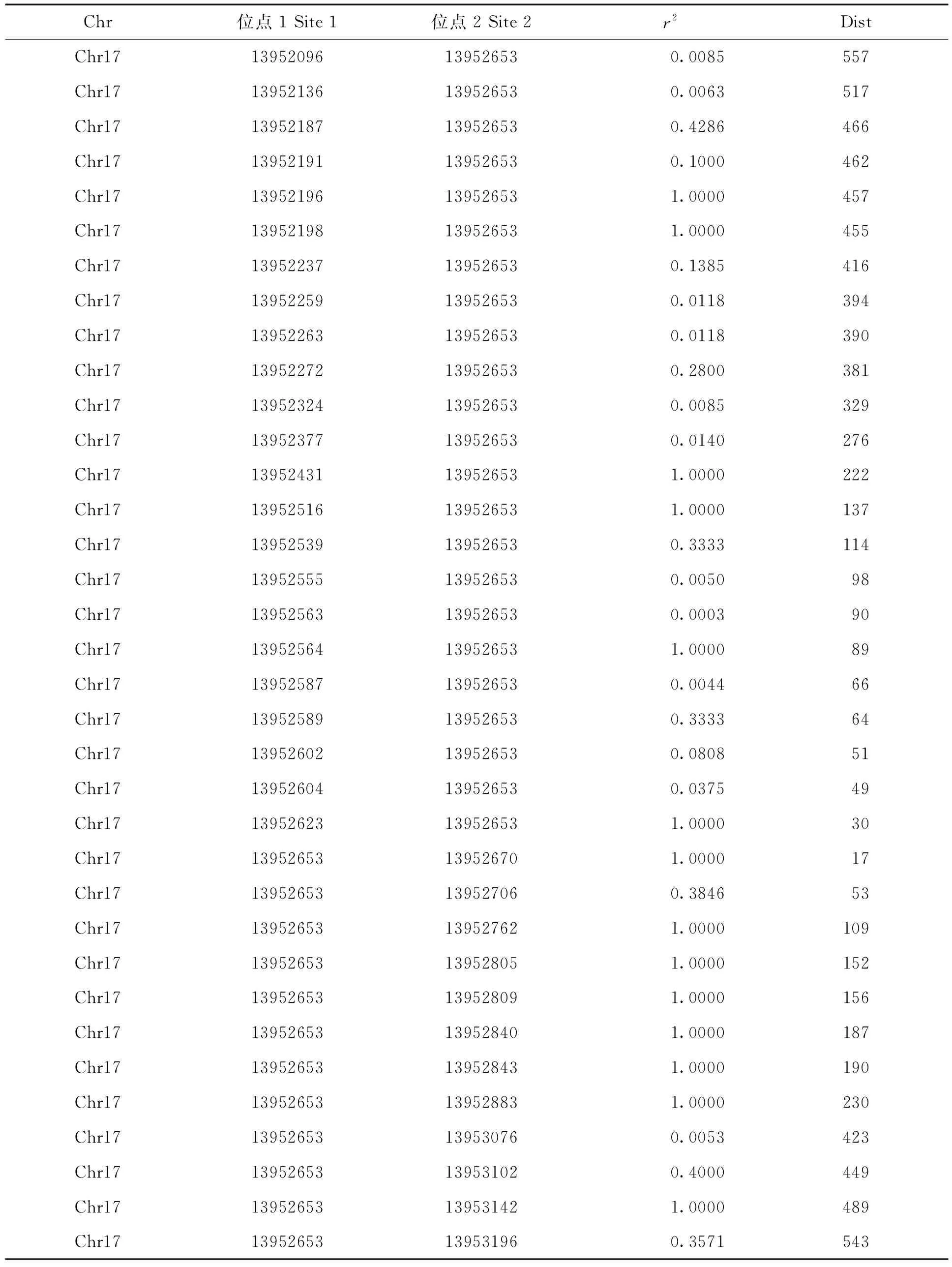
表3 Cspg4不同单倍型调控序列区域内连锁不平衡分析Tab.3 Results of linkage disequilibrium in the region of different allele sequences of Cspg4
3 讨论
表达QTL分析被认为是定位基因表达调控位点的一种有效方法。为了解大黄鱼脾脏eQTL中挖掘到的显著SNP位点对生物信息学分析预测到的靶基因是否存在调控作用,本研究首先通过Sanger测序验证了81尾实验鱼中该SNP位点的基因型,进而通过构建候选启动子与调控序列片段的重组质粒测定调控序列的活性。
RNA聚合酶及转录因子通常结合作用在基因的启动子区,欲了解基因表达调控模式必须研究启动子的分子结构和功能[19]。本研究成功扩增到大黄鱼Cspg4候选启动子2096 bp序列并预测该序列中潜在的核心区域,结果表明有1个潜在的核心区域存在于该基因的候选启动子序列中。随后构建Cspg4基因候选启动子区不同长度缺失片段的重组质粒。分析结果显示:pGL3-Cspg4-P4重组质粒的启动子活性最高,而pGL3-Cspg4-P1~P3重组质粒相较于pGL3-Cspg4-P4的启动子活性逐渐降低。表明:在-2076~-1202区域可能存在负调控元件;在-1202~-762和-190~+20区域可能为该启动子的核心区域,存在基本调控元件。而pGL3-Cspg4-P5~P7重组质粒的启动子活性逐渐增强,说明在-762~-190区域可能存在负调控元件。
在转录激活过程中发生的一系列级联反应最终会作用到核心启动子上,直接或间接影响核心启动子上发生的基础转录作用。核心启动子是所有能转录调控RNA聚合酶的转录因子的终极作用靶标[20]。因此,为了研究含Chr17_13952653位点的序列对Cspg4的调控作用,据上述启动子活性分析结果,将存在Cspg4基因的核心区域的pGL3-Cspg4-P7(-190~+20)重组质粒作为对照组,构建含Chr17_13952653位点的不同单倍型调控序列的重组质粒。双荧光素酶报告系统结果显示,pGL3-Cspg4-P7-A和pGL3-Cspg4-P7-C重组质粒的相对荧光活性均显著低于对照组pGL3-Cspg4-P7,而pGL3-Cspg4-P7-A和pGL3-Cspg4-P7-C重组质粒之间相对荧光活性无显著差别。pGL3-Cspg4-P7-C重组质粒活性略低,表明含Chr17_13952653 位点的1157 bp序列可能为减弱启动子活性的调控序列,但该位点的A/C变异可能不直接影响该调控序列的功能。连锁不平衡分析发现,该序列中有14个与Chr17_13952653位点强连锁的SNP位点(见图6),且r2值均等于1(见表3)。
生物的基因组存在大量调控基因表达的序列,包括启动子、增强子、沉默子和绝缘子等。其中的沉默子是一类主动行使抑制启动子活性的调控元件,这些序列能招募转录因子到启动子上,进而执行特定的功能。已有研究表明许多不同类型的沉默子可影响基因调控的多个方面,如转录因子活性、内含子剪接、染色质结构、3' 上游非翻译信号识别及转录起始复合物的装配等,最终下调基因表达[21]。目前已报道的研究显示:沉默子无特定结构或保守序列,它们可能是一段包含有特异性阻遏蛋白结合位点的短核酸序列;也可能作为一个包含多个调控元件的复杂区域存在,如酵母HMR-E(hidden mating region-E)沉默子[22]。大部分沉默子具有非启动子特异性的特点。Zhao等[23]报道在多巴胺转运蛋白基因(human dopamine transporter gene,hDAT)内含子中发现一个121 bp的沉默子,它不仅可在体外下调hDAT启动子活性,还可以使异源性SV40(simian virus 40)启动子活性下降80%。典型沉默子对基因表达的阻遏作用表现出位置和方向的非依赖性,主要通过干扰转录起始复合物的装配这一主动抑制过程调控转录,但也有些沉默子对此表现出不同程度的依赖[24]。B淋巴细胞瘤-2基因(bcl-2)是细胞凋亡的抑制因子。Young等[25]在此基因的5' UTR中鉴定出一个约1.3 kb的负调控元件,将其插入P1启动子下游约2.4 kb处,启动子的活性未受影响,表明该NRE以特定位置的方式降低bcl-2 P1启动子的表达。一些沉默子元件具有远距离作用顺式连接启动子的特性,本研究中含Chr17_13952653位点的调控序列相对荧光活性显著低于对照组,该结果表明在大黄鱼脾脏基因中也可能存在类似沉默子抑制转录过程的顺式作用元件。目前研究者们对大黄鱼的顺式调控元件及其功能的研究,主要体现在启动子与转录因子结合层面。如Sun等[26]发现大黄鱼Toll样受体家族中tlr5m和tlr5s的基因组结构和启动子结构可能存在差异,其中oct1和nf-κb结合位点可能是tlr5s启动子的顺式调控元件,nf-κb/p65通过与nf-κb结合位点结合,在tlr5s启动子激活中发挥重要作用。Zhang等[27]发现大黄鱼中存在对哺乳动物巨噬细胞的发育至关重要的转录因子pu.1的两种转录本pu.1a和pu.1b,它们都可以通过结合其启动子的不同E26转化特异性基序,上调组织蛋白酶(Cathepsin S)的表达,并且它们的三个结构域都是启动Ctss表达的必要条件。Xu等[28]发现磷酸化的转录因子stat1通过结合其启动子区域,显著上调了干扰素调节因子1(irf1)的启动子活性。但是本研究在启动子基础上进一步探究eQTL分析获得的不同单倍型调控序列对靶基因的作用,为表型与变异之间建立起连接。因此,利用启动子、增强子、沉默子等元件构建人工表达载体,精确调控目的基因的表达,深入研究有关基因表达调控机制可作为未来研究的基础。
综上所述,通过对大黄鱼17号染色体13952653 bp前后长度为1157 bp的调控序列对靶基因Cspg4启动子的表达调控研究,为Cspg4基因在大黄鱼脾脏中的转录调控及功能研究奠定了基础。但迄今为止,对该基因参与免疫系统的调节且在大黄鱼脾脏组织表达的分子机制还知之甚少,其具体表达调控方式与作用机制有待进一步深入研究。
