噬菌体Z基因组生物合成通路的研究进展
2023-10-30陈荟玉赵素文
陈荟玉,赵素文
综 述
噬菌体Z基因组生物合成通路的研究进展
陈荟玉,赵素文
上海科技大学iHuman研究所与生命科学与技术学院,上海 201210
噬菌体基因组中存在丰富的碱基修饰,主要用于逃避宿主内切酶的切割。40多年前,在蓝藻噬菌体S-2L的DNA中,研究者发现2-氨基腺嘌呤(Z)完全取代腺嘌呤(A),与胸腺嘧啶(T)形成具有三根氢键的互补配对,形成了特殊的Z基因组。近年来,研究者在多个噬菌体中发现并证实了噬菌体Z基因组生物合成通路。该通路是一个多酶系统,其中包含由噬菌体DNA编码的2-氨基脱氧腺苷琥珀酸合成酶(PurZ)、脱氧腺苷三磷酸水解酶(dATPase/DatZ)、脱氧腺苷/脱氧鸟苷三磷酸的焦磷酸水解酶(DUF550/MazZ)和DNA聚合酶(DpoZ)。本文在简述噬菌体中各种修饰核苷发现历史的基础上,详细介绍了Z基因组生物合成通路中多种酶的研究进展,最后对Z基因组及其合成通路中多种酶的应用进行了展望,以期为该领域的研究提供借鉴和参考。
Z基因组;噬菌体;DNA修饰;2-氨基腺嘌呤;生物合成通路
众所周知,DNA由四种标准脱氧核苷构成,每种脱氧核苷都拥有一个独特的碱基,分别是腺嘌呤(adenine,A)、胸腺嘧啶(thymine,T)、胞嘧啶(cytosine,C)、鸟嘌呤(guanine,G),这四种标准碱基构成了生命的遗传密码表。除了四种标准碱基,在病毒、原核及真核的基因组中,还广泛存在着多种天然的碱基修饰,即各种化学基团连接到标准碱基上的现象。这些修饰对基因组的稳定性、基因的复制和表达调控有着重要的影响[1]。在真核生物中,修饰通常比较简单,如甲基修饰、羟甲基修饰等;而在细菌及噬菌体中,存在氨基酸、胺类、糖类等更加复杂的修饰[2]。对于噬菌体来说,这些复杂修饰能够帮助其逃避宿主的防御系统,如限制-修饰系统[2,3]。可以说,这些修饰就是噬菌体和宿主进行漫长军备竞赛的一类利器,也是人们研究两者从古至今如何互相影响和进化的一类有效证据。
从20世纪50年代开始,噬菌体DNA中的碱基修饰就陆续地被人们发现。在已明确生物合成通路的噬菌体碱基修饰中,有部分修饰是在四种标准脱氧核苷聚合成DNA链后才发生的,这类通常被称作表观遗传修饰;而另一部分碱基修饰发生在DNA聚合之前,基因组中的某种碱基全部被另一种修饰碱基替换,这打破了人们对生命遗传物质的传统认知。本文将介绍其中一种更为特殊的修饰碱基——2-氨基腺嘌呤(2-aminoadenine,简称Z)。因该碱基的修饰发生在碱基配对边,所以其改变了正常DNA碱基“A与T形成两根氢键、G与C形成三根氢键”的配对原则。伴随着噬菌体中各种修饰核苷的发现历史,本文将对Z基因组的相关研究进行系统地阐述:Z基因组及其合成通路如何一步步被发现、目前的研究进展及其应用的展望。
1 噬菌体基因组中存在丰富的碱基修饰
1.1 发生在碱基非配对边上的修饰
早在1953年,Wyatt等[4]就在大肠杆菌噬菌体T2、T4和T65(phage T2, T4, T65)的DNA中发现5-羟甲基胞嘧啶(5-hydroxymethylcytosine,5-hmC)修饰,该修饰能够保护DNA免受CRISPR-Cas9降解[5]。随后,多种胞嘧啶修饰被发现。例如,Teng等[6]发现了5-甲基胞嘧啶(5-methylcytosine,5mC);Hattman 等[7]发现了5-羟基胞嘧啶(5-hydroxycytosine,5hC)。
1962年,Kallen等[8]发现,多种噬菌体DNA中含有完全取代胸腺嘧啶的5-羟甲基尿嘧啶(5-(hydroxymethyl)uracil,5-hmU),包括芽孢杆菌噬菌体SPO1、Φe、SP8、H1、2C和SP82(phage SPO1, Φe, SP8, H1, 2C, SP82)。多种噬菌体还对DNA上的5-hmU进行进一步修饰,生成如α-腐胺胸腺嘧啶(α-putrescine thymine,α-putT)、α-谷氨酰胺胸腺嘧啶(α-glutamine thymine,α-gluT)、5-(2-氨基乙基)尿嘧啶(5-(2-aminoethyl)uracil,5-NeU)、5-(2-氨基乙氧基)甲基尿嘧啶(5-(2-aminoethoxy)methyluracil,5-NeOmU)[9~11]等更加复杂的修饰。
1983年,Swinton等[12]发现大肠杆菌噬菌体Mu(phage Mu)中约15%的腺嘌呤被N6 -氨甲酰甲基腺嘌呤(N6-carbamoylmethyladenine,ncm6A)所取代,其基因组编码的一种乙酰转移酶家族蛋白可能与该碱基的合成有关。
2016年,Thiaville 等[13]在大肠杆菌噬菌体9g(phage 9g)中发现了7-甲脒基-7-脱氮鸟嘌呤(7-formamidino-7-deazaguanine,G+),其合成通路涉及到等一系列噬菌体基因组编码的基因。在测试的214种Ⅱ型限制性内切酶中,有 152种都不能顺利切割含有G+的DNA[14]。除了G+,还存在几种7-脱氮鸟嘌呤类似物:7-酰胺基-7-脱氮鸟嘌呤(7-amido-7-deazaguanine,ADG)、7-氰基-7-脱氮鸟嘌呤(7-cyano-7-deazaguanine,PreQ0)、7-氨甲基-7-脱氮鸟嘌呤(7-aminomethyl-7- deazaguanine,PreQ1)[15]。
上述绝大多数碱基修饰都发生在嘧啶环的5位或者嘌呤环的7位[2],这两个位点并不在碱基的配对边,因此对碱基互补配对的直接影响不大。
1.2 Z碱基——发生在碱基配对边上的修饰
1977年,苏联莫斯科国立大学的Kirnos等[16]首次报道,在蓝藻噬菌体S-2L(Cyanophage S-2L)基因组中,2-氨基腺嘌呤(Z)完全取代了腺嘌呤(A)。由于多出的2-氨基处于配对边上,Z跟T配对会形成三根氢键[16,17](图1A)。在正常DNA中,A跟T配对只能形成两根氢键。这一微小的变化却可以较大地改变DNA的结构和理化性质。和正常的DNA相比,dZ-DNA(dZ-DNA指Z完全取代A的DNA)具有更高的解链温度(melting temperature,Tm)、更强的弯曲刚度(flexural rigidity)、更长的持久长度(persistence length)、更短的静态轮廓长度(static contour length)[17~20]。Z碱基也让蓝藻噬菌体S-2L能够抵抗绝大部分识别序列中含有A的限制性内切酶的切割[21,22]。Tan等[23]在研究DNA的转录效率时发现,含Z的DNA还对T7 RNA聚合酶和人RNA聚合酶II介导的转录具有显著的抑制作用。
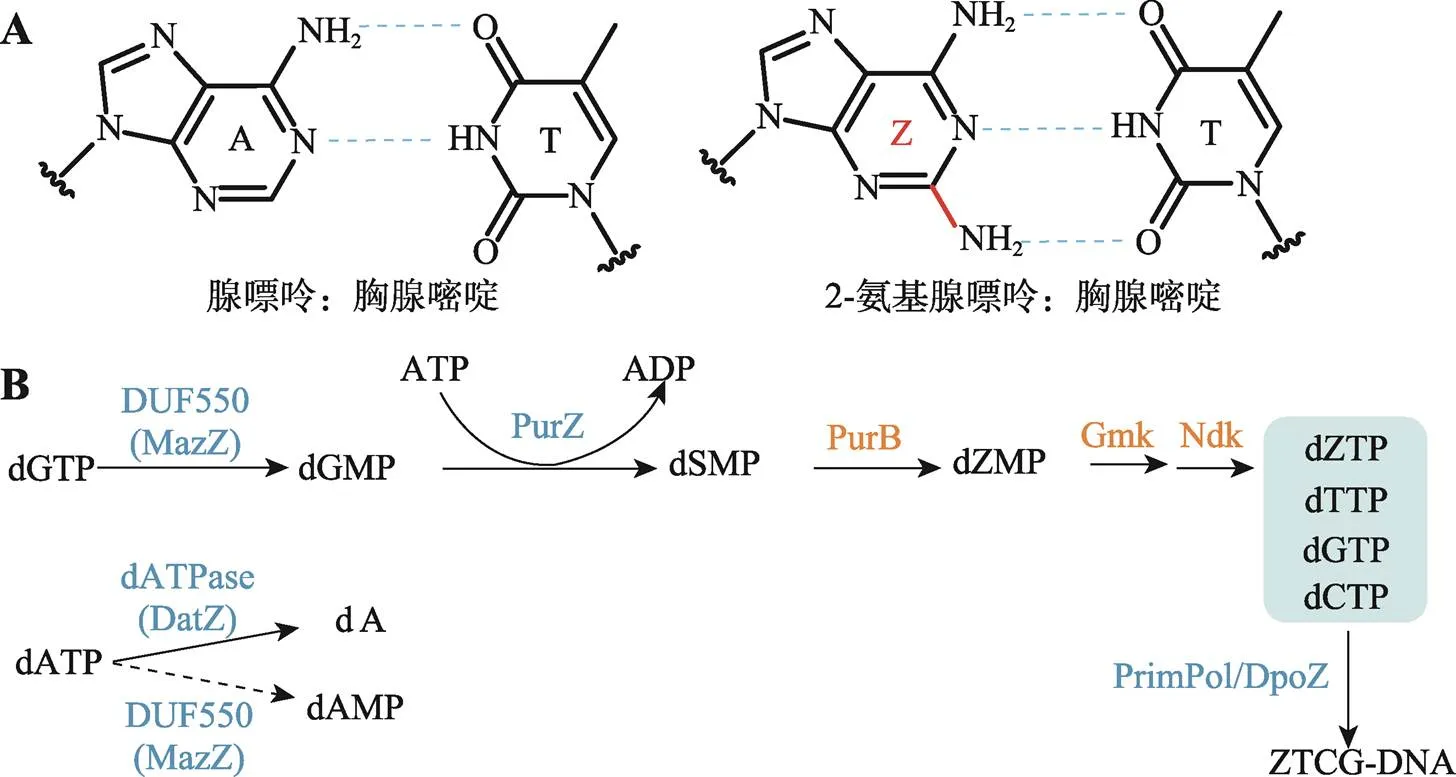
图1 A:T配对和Z:T配对的比较及Z基因组的生物合成通路
A:A:T配对和Z:T配对示意图。图中蓝色虚线表示氢键;B:Z基因组的生物合成通路。蓝色标注酶为噬菌体编码,橙色标注酶为宿主编码(也有少量噬菌体中编码PurB,见图2A)。
发现蓝藻噬菌体S-2L的科学家在当时没有条件对这个噬菌体进行测序,他们将S-2L及其宿主菌赠与了多个感兴趣的课题组,其中包括法国巴斯德研究所的Philippe Marliere和Pierre A. Kaminski等。到20世纪90年代末,巴斯德研究所的这些科学家使用Sanger测序法完成了对噬菌体S-2L的测序[24]。但可能由于S-2L的高GC含量以及Z对A的取代,这次测序结果中存在着不少错误,这些错误使得通路关键酶经过合成纯化后并不具备活性,从而将Z基因组合成通路的发现往后又推迟了十几年。
2 噬菌体DNA中的2-氨基腺嘌呤(Z)及其合成通路
从1977年到2021年以前,蓝藻噬菌体S-2L都是唯一已知的Z基因组噬菌体,这不禁让人怀疑Z基因噬菌体的存在是非常罕见的。然而,随着测序价格的降低,测序变得越来越普遍,公共数据库也开始不断出现Z基因组噬菌体的测序数据——当然,测序者并不知道它们的基因组中含有Z,因为通常使用的二代测序方法受到测序原理的限制,是无法检测出DNA上碱基修饰的。只有通过仔细的序列分析、结构建模、分子对接,以及关键酶的克隆表达纯化和活性实验,Z基因组的生物合成通路才有可能被揭示出来。相应地,数据库中的Z基因组噬菌体才能根据含有Z基因组生物合成通路的相关基因而被鉴定出来。
2021年,噬菌体Z基因组的生物合成通路被赵素文/张雁/赵惠民合作团队以及法国巴斯德研究所Pierre A. Kaminski/Marc Delarue/Philippe Marliere合作团队各自独立阐明(图1B)[25~29]。这些课题组主要的研究对象,并不是测序结果存在问题的噬菌体S-2L,而是其他测序结果准确的Z基因组噬菌体,如鲍曼不动杆菌噬菌体SH-Ab 15497(phage SH-Ab 15497)和霍乱弧菌噬菌体PhiVC8 (phage PhiVC8)。
这几个课题组对Z基因组生物合成通路的研究结果是高度一致的。首先,噬菌体会编码dATP/dGTP焦磷酸水解酶(deoxyadenosine/deoxyguanosine triphosphate pyrophosphatase,DUF550,或称MazZ)将脱氧鸟苷三磷酸(dGTP)去磷酸化成脱氧鸟苷单磷酸(dGMP),然后,噬菌体编码的2-氨基脱氧腺苷琥珀酸合成酶(deoxy-2′-aminoadenylosuccinate synthetase,PurZ)在腺苷三磷酸(ATP)、L-天冬氨酸(L-Asp)和Mg2+存在时,将dGMP转化为2-氨基脱氧腺苷琥珀酸(2-aminodeoxyadenosylsuccinate,dSMP,或称ADAS)。接下来,宿主体内的腺苷琥珀酸裂解酶(aminoadenylosuccinate lyase,PurB)将dSMP转为2-氨基脱氧腺苷单磷酸(2-aminodeoxyadenosine monophosphate,dZMP),再经宿主的鸟苷激酶(guanosine kinase,GmK)和核苷二磷酸激酶(nucleoside diphosphate kinase,Ndk)磷酸化为2-氨基脱氧腺苷三磷酸(2-aminodeoxyadenosine triphosphate,dZTP)。最终,噬菌体通过自身编码的DNA聚合酶(DNA polymerase,DpoZ或PrimPol)完成dZ-DNA的聚合。为保证基因组中Z对A的完全取代,噬菌体编码了两个酶,脱氧腺苷三磷酸水解酶(deoxyadenosine triphosphate hydrolase,dATPase,或称DatZ)和脱氧腺苷/脱氧鸟苷三磷酸的焦磷酸水解酶(DUF550,或称MazZ)来水解dATP,降低它的浓度,杜绝噬菌体的DNA聚合酶对dATP的使用。其中,dATPase可以专一地把dATP转化为dA,而DUF550因为可以将dATP去磷酸化为dAMP,所以也可以协助降低dATP的浓度。还有一部分Z基因组噬菌体中缺少编码DUF550和dATPase的基因,被注释为“dUTPase”(deoxyuridine triphosphate hydrolase)的功能未知基因可能替代它们行使类似的功能(图2A)。
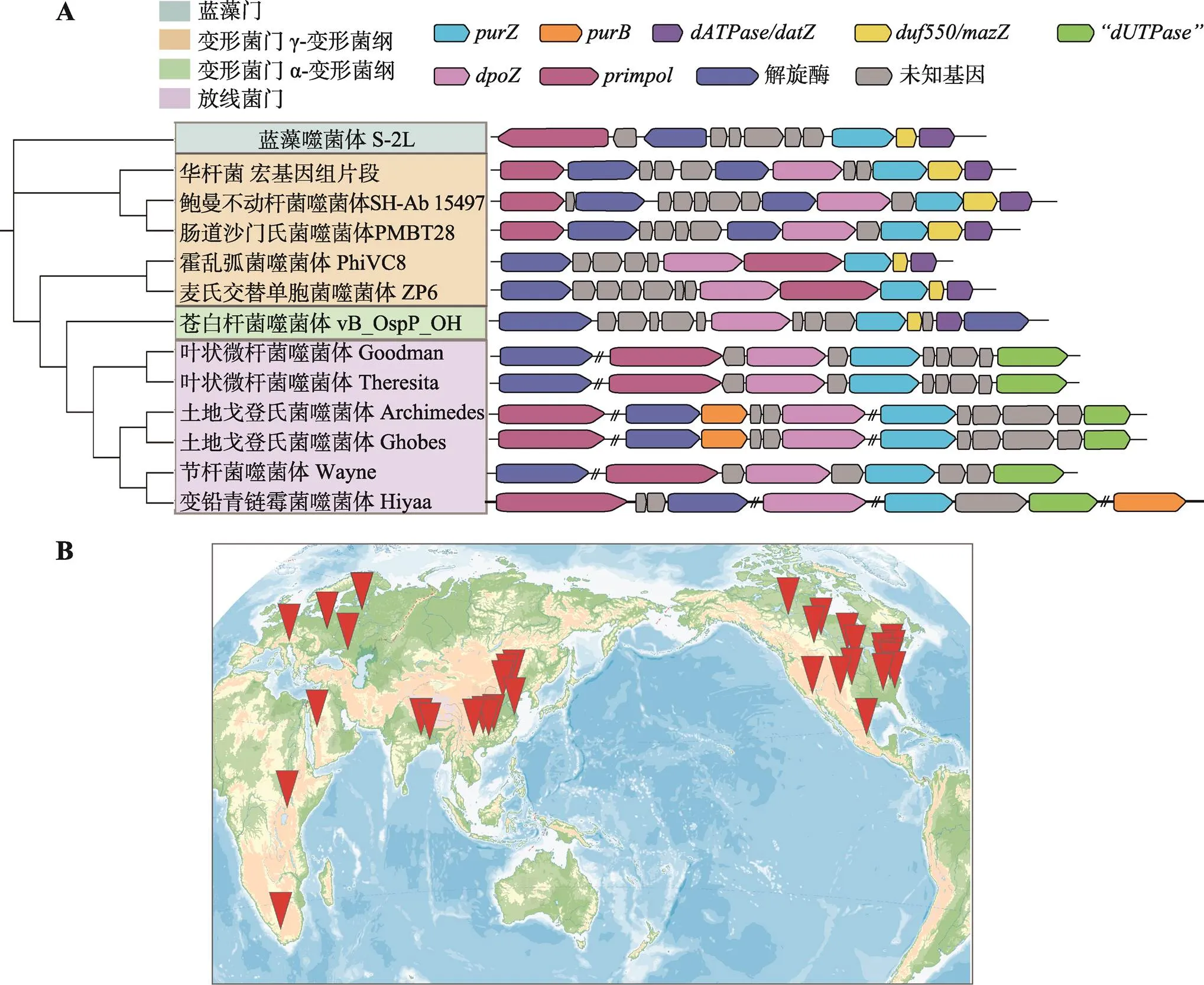
图2 噬菌体Z基因组合成通路的基因簇及在全球的地理分布
A:噬菌体中负责合成Z基因组的基因簇。最左边是各噬菌体的PurZ的系统发育树(使用IQ-TREE软件构建最大似然树,替代模型为“LG+I+G4”,步长为1000);用不同颜色标注出了噬菌体对应宿主的不同细菌门(纲);B:Z基因组噬菌体在全球的分布图。地图下载自中国地图出版社与国家基础地理信息中心联合编制的“世界地形图”,详见地图浏览(mnr.gov.cn)。
Philippe Marliere和Pierre A. Kaminski团队还指出,Z基因组噬菌体是相当古老的生物。他们的推理如下:目前已知的Z基因组噬菌体的宿主细菌主要分布于变形菌门、蓝藻门和放线菌门,而这些噬菌体Z基因组生物合成通路中的几个关键酶,PurZ,dATPase和DNA聚合酶的进化树拓扑结构是一致的,这表明Z基因组在三个门的细菌分开(约35亿年前)之前就已经存在。有意思的是,Z碱基在年代早于地球的碳质陨石中被鉴定出来[30],表明了该碱基可能是地外生命的遗传物质,且可能参与了地球生命的起源。
赵素文/张雁/赵惠民团队重新对公共数据库中含有PurZ的基因组进行了盘点,他们发现,除了噬菌体S-2L,还有122个分离噬菌体和13个宏基因组测序片段中也具有Z基因组合成通路中的关键酶PurZ,它们构成了目前已知的Z基因组噬菌体名单(表1)。这些噬菌体广泛分布在世界各地[25,26](图2B),是已知分离噬菌体总量的约1%,对应的宿主能归属到细菌的4个门,分别为变形菌门、放线菌门、厚壁菌门和蓝藻门(表1)。变形菌门的这些宿主分布于α、β和γ变形菌纲,涉及到7个细菌目,其中以γ变形菌纲-弧菌目-弧菌科-弧菌属为宿主的噬菌体较为常见;放线菌门的宿主主要分布于放线菌纲的放线菌目、链霉菌目和微球菌目中,其中以微球菌目-微球菌科-节杆菌属为宿主的噬菌体数量众多;目前,人们在厚壁菌门和蓝藻门的宿主中只分别观察到一种Z基因组噬菌体的存在。虽然目前发现的Z基因组噬菌体数量并不算多,但其存在于多个细菌门的现象就可以表明,Z基因组噬菌体已经在地球上存在了几十亿年而未灭绝,并能够以多种类型的细菌作为宿主,显示出了顽强的生命力。相信随着采样和测序工作的稳步推进,更多的Z基因组噬菌体将会被人们源源不断地发现。
在所有Z基因组噬菌体中,鲍曼不动杆菌噬菌体15497、霍乱弧菌噬菌体phiVC8、土地戈登氏菌噬菌体Ghobes(phage Ghobes)和节杆菌噬菌体Wayne(phage Wayne)的DNA经酶解后的脱氧核苷产物,经过高效液相色谱-紫外法(HPLC-UV)鉴定,确实都存在Z取代A的现象[25,27]。实验还表明,前述几种噬菌体的DNA都不能被识别序列中含A的限制性内切酶所消化,唯一的例外是一个底物范围很宽的I[25],该酶以能够耐受各种DNA修饰而著称[21]。
2.1 Z基因组噬菌体中的PurZ是PurA的一种同源蛋白
关于Z基因组噬菌体生物合成通路的发现,可追溯到20多年前Pierre A. Kaminski课题组对噬菌体S-2L的测序[24]。他们发现S-2L的基因组中包含了一个腺苷琥珀酸合成酶(adenylosuccinate synthetase,PurA)同源物的开放阅读框(后称为PurZ),并认为PurZ会催化一个类似的反应来合成Z核苷酸[2,24]。PurA是所有细胞生物共有的,是腺嘌呤核苷酸从头合成通路中的一个酶,其在GTP、L-天冬氨酸(L-Asp)和Mg2+的存在下催化次黄嘌呤核苷酸(inosine monophosphate,IMP)生成腺苷琥珀酸(adenosylsuccinate)、GDP和磷酸根,后续再通过腺苷琥珀酸裂解酶(PurB)催化,脱去富马酸生成AMP[32](图3A)。而在S-2L中发现的PurZ,则催化了一个类似的反应(图3B)。和PurA相比,PurZ中IMP结合口袋的底物在两个小地方发生了变化,一是碱基由I变为G,二是核苷变为脱氧核苷;而GTP结合口袋的底物,则从GTP变成了ATP。系统发育分析显示,这些噬菌体PurZ与某些古菌的PurA的进化关系最近,而与原核和真核生物的PurA关系较远[33]。其实,除了噬菌体,少数细菌中也存在着PurZ类似物[26],目前尚不清楚其在细菌中的功能。
2021年,PurZ的体外功能分别得到了赵素文/张雁/赵惠民团队和Pierre A. Kaminski课题组充分的实验验证。体外实验显示,噬菌体S-2L、SH-Ab 15497、phiVC8肠道沙门氏菌噬菌体PMBT28 (phagePMBT28)和华杆菌(bacterium)宏基因组组装片段中的PurZ(后续分别称为CpPurZ、ApPurZ、VpPurZ、SpPurZ、SbPurZ)在与底物dGMP、ATP、L-Asp、Mg2+共同孵育后具有很强的酶活性(dGMP、ATP的kcat/KM范围分别为7.5~171.9、1.8~67.1 [mM–1s–1])[25,26]。将dGMP换成GMP/IMP,或者把ATP换成其他NTPs都没有活性。对于SbPurZ,dGMP换成dIMP还有38%的活性,但dIMP的kcat/KM约只有在体内的真正底物dGMP的1/15[25]。SpPurZ和ApPurZ反应的产物通过高效液相色谱-二级质谱联用法(HPLC-MS/MS)确证为ADP和dSMP[25](图3B)。虽然另一组研究者发现,将CpPurZ分别与dGMP、ATP/dATP、L-Asp、Mg2+孵育,有相当的活性,且HPLC-MS/MS显示底物为dSMP和ADP/dADP[28],但考虑到Z基因组噬菌体存在不止一条降低dATP浓度的途径,生理情况下的dATP浓度应该很低,不太可能与ATP竞争这个口袋。
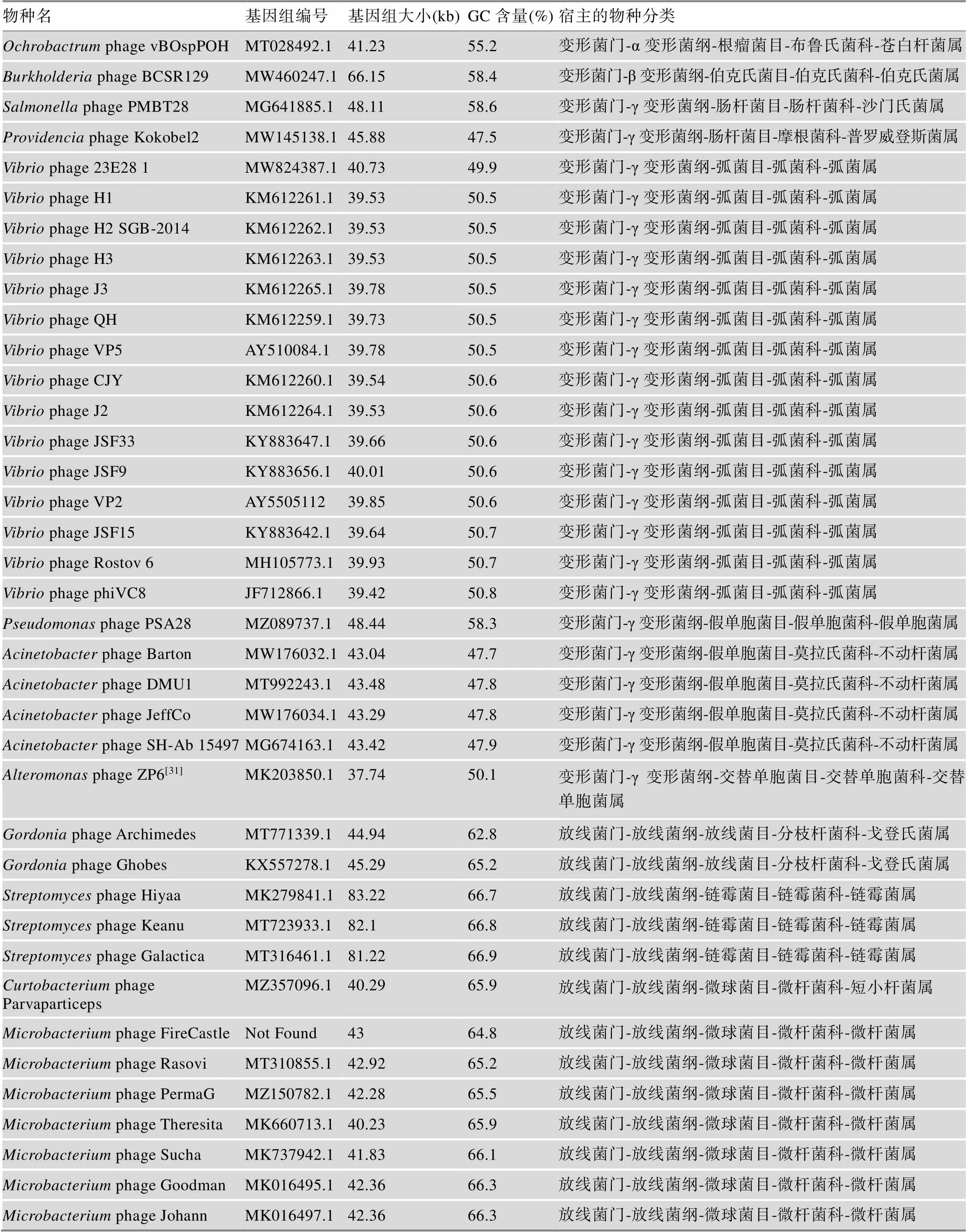
表1 已分离测序的Z基因组噬菌体名单
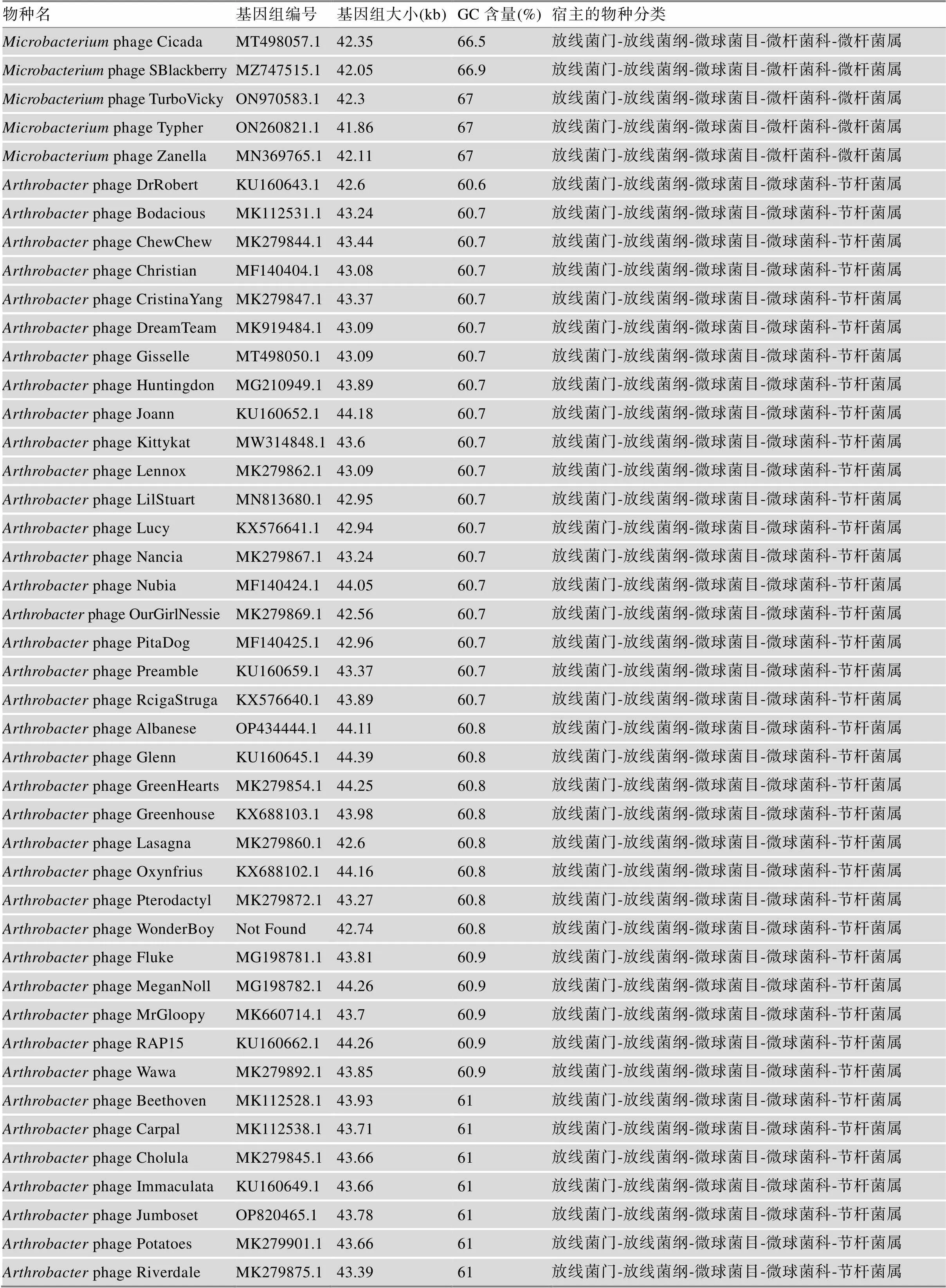
续表
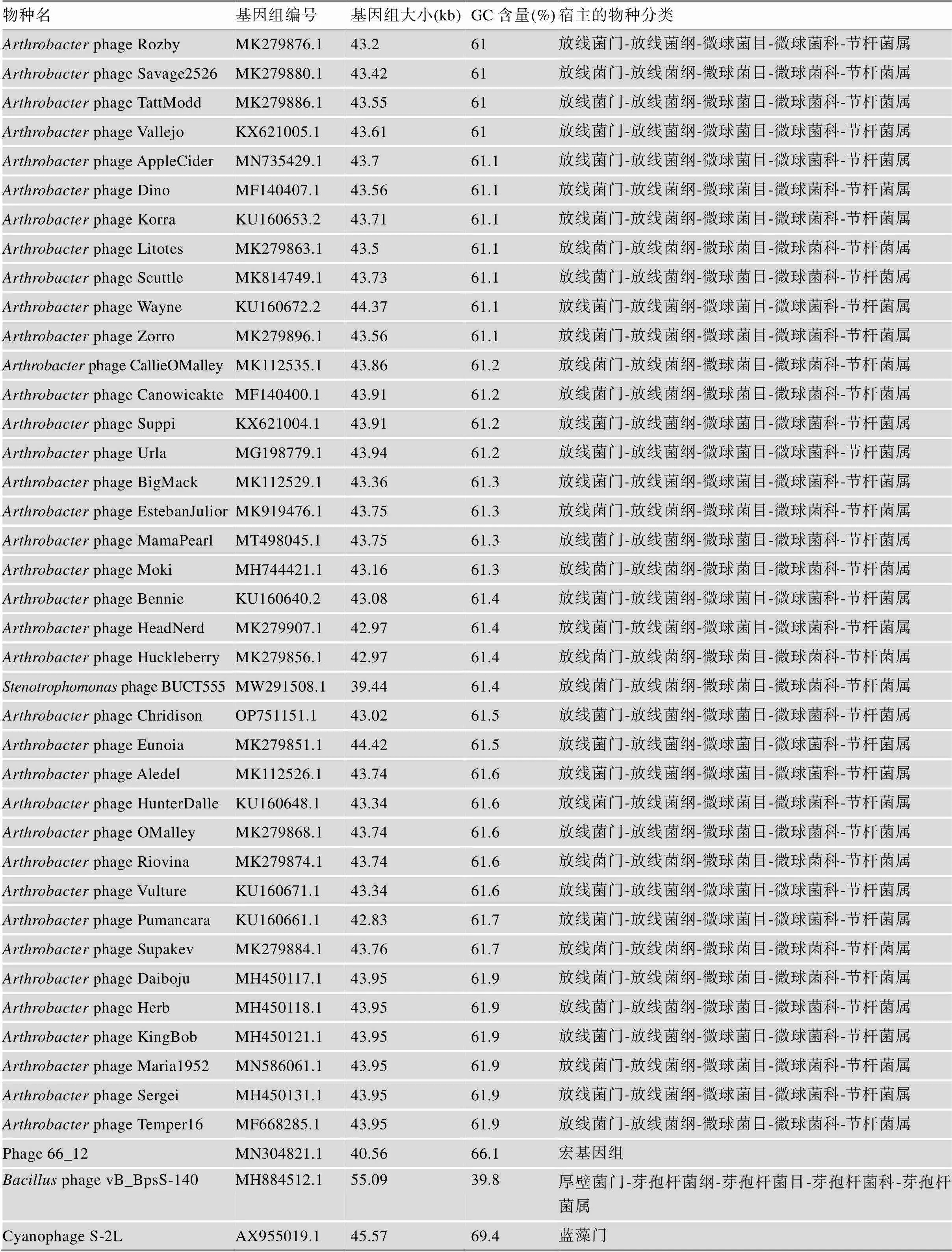
续表
名单先按照宿主的物种分类排序,再按照基因组GC含量排序。以上信息来源于NCBI网站对应噬菌体基因组页面,例如phage DMU1的信息来源:eotide-NCBI (nih.gov)。
A:AMP从头合成通路中的两个步骤;B:Z基因组噬菌体产生dZMP的通路与细胞生物从头合成AMP的通路类似。
值得一提的是,根据2004年测序结果注释出的噬菌体S-2L CpPurZ,在经过蛋白表达后并没有活性,而根据2021年第二次测序结果注释出的CpPurZ表现出了应有的活性。把两次测序结果进行对比可知,第一次测序时基因上有两个测序错误[24],正是这两个测序错误导致了Z基因组生物合成通路的发现被推迟了十几年。
法国巴斯德研究所Pierre A. Kaminski和Marc Delarue 课题组分别解析了VpPurZ和CpPurZ的晶体结构[26,28],这使人们能够更深入地理解这类蛋白功能变化的原因。其中,VpPurZ有apo-PurZ(PDB:6FLF),结合ATP的(PDB:6FM0),结合dGMP、ATP、Mg2+的(PDB:6FM1)以及结合AMPPcP(β,γ -亚甲基腺苷5′-三磷酸,β,γ-methyleneadenosine 5′-triphosphate)、dGMP、L-Asp的(PDB:6TNH)的四种晶体结构。CpPurZ结构中结合了dGMP和dATP(PDB:7ODX)[28]。和PurA一样,PurZ也是二聚体[34,35]。对于dGMP和ATP/dATP这两个主要的配体,VpPurZ(PDB:6FM1)和CpPurZ具有类似的结合方式。与经典的大肠杆菌K-12(K-12) 的PurA(EcPurA,PDB:1CG0)相比,VpPurZ(PDB:6FM1)的(d)NMP结合口袋中的Ser14取代了Asp13,与dGMP的嘧啶部分形成氢键,正好可以容纳底物上额外的2-氨基。同时,Ile234对Thr271的取代,则改变了临近残基Val236的构象,而Val236的不同构象,可以决定底物是脱氧核糖核苷还是核糖核苷。在PurZ中,Val236的侧链靠近底物的糖环,空间位阻和非极性环境,决定了该处无法再容纳核糖上的2′-OH (图4A)。对于NTP结合口袋,VpPurZ中Asn297取代了EcPurA中的Asp333,与腺嘌呤形成氢键,有利于配体从GTP转变为ATP,与K331类似位置的Phe295仍与ATP嘌呤部分形成堆叠作用(图4B)。
对于NTP口袋的297位结合位点,天津大学张雁/上海科技大学赵素文/深圳大学李猛和刘杨团队通过对Z基因组噬菌体关键位点的保守性分析发现,目前已知的所有感染放线菌门细菌的Z基因组噬菌体,其PurZ的297位仍是Asp (图4B右),意味着这些噬菌体的NTP结合口袋很可能仍然结合GTP而非ATP[33]。巧合的是,上述经过活性验证的PurZ均不来自感染放线菌门的噬菌体,所以它们的底物组合均为dGMP和ATP。通过对土地戈登氏菌噬菌体Archimedes(phage Archimedes,宿主为放线菌门)的PurZ(后称为GpPurZ)进行活性实验,他们发现,GpPurZ只有在以dGMP和GTP组合作为底物时才展现出很强的酶活性(GTP的kcat/KM为2.8~3 [mM–1s–1],反应也需要L-Asp和Mg2+)[33]。与SbPurZ类似,虽然将GpPurZ的底物dGMP换成dIMP也有约50%的活性,但dIMP的kcat/KM约只有真正底物dGMP的1/13。另外,相应底物在经过GpPurZ孵育后的HPLC-MS/MS实验结果显示,产物仍然是dSMP[33]。也就是说,这两类PurZ都能够将dGMP生成dSMP,但用来反应的磷酸盐供体并不相同:感染放线菌门的Z基因组噬菌体使用GTP,而其他Z噬菌体则使用ATP。
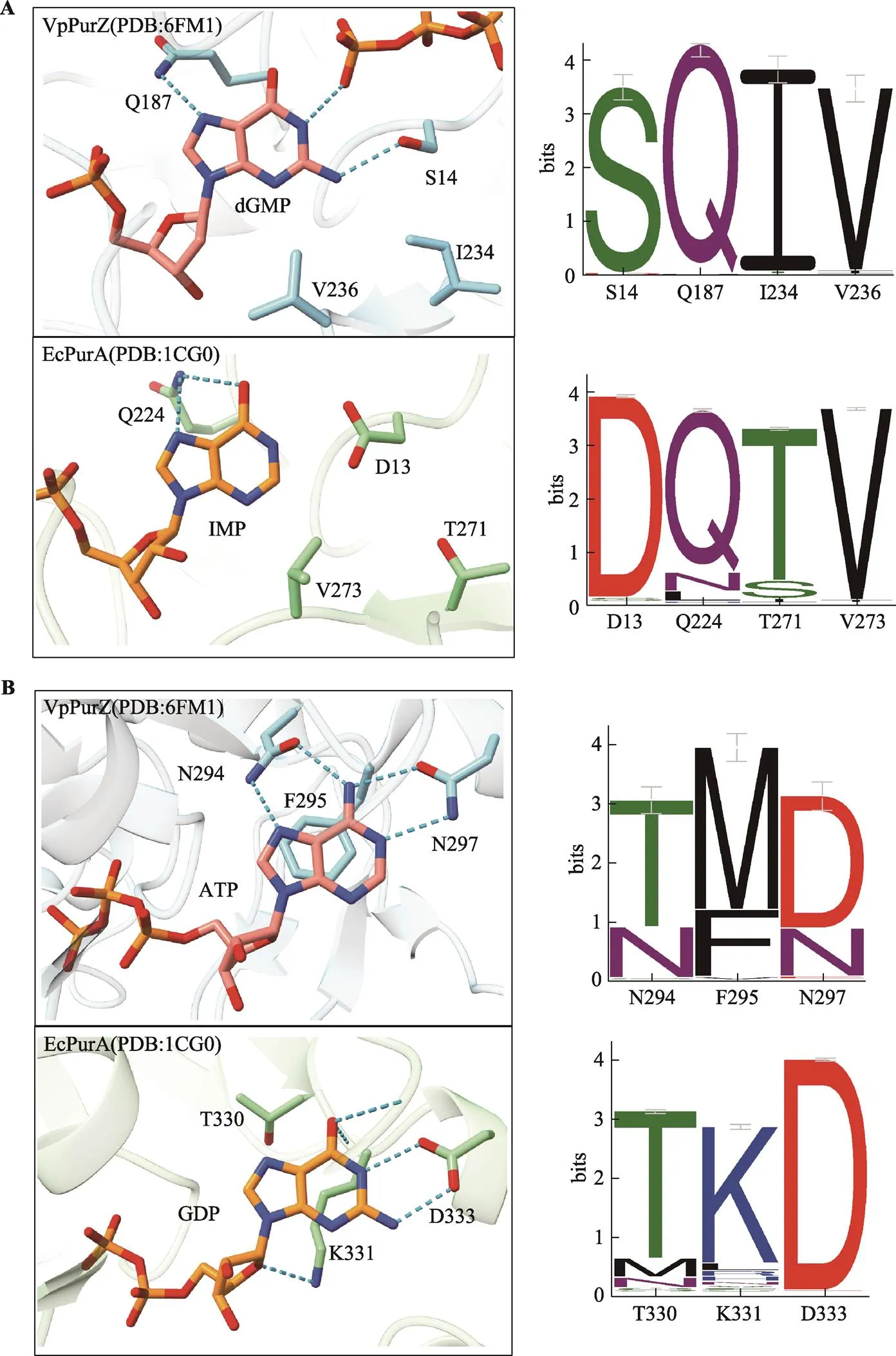
图4 VpPurZ和EcPurA中口袋与底物结合示意图
A:(d)NMP口袋与底物结合示意图;B:NTP口袋与底物结合示意图。蓝色虚线表示氢键,右侧均为相应位点在所有Z基因组噬菌体PurZ/经典PurA中的保守性。
对于上述提到的PurZ的关键残基位点,有不少突变实验证明了它们的重要性。例如对于SbPurZ的实验,所有突变都不同程度地降低了其活性:Ser15突变成Asp失活(6FM1:Ser14),说明了Ser15结合dGMP的重要性;Thr274突变成Gly(6FM1:Thr262)使Asp的KM升高271倍,说明Thr274对结合Asp至关重要;突变ATP周围三个残基的任意一个(Asn306突变为Thr,Phe307突变为Lys,Asn309突变为Asp) (6FM1:Asn294, Phe295, Asn297)都会导致ATP的KM上升几十倍,但并没有一个突变体可以使用GTP[25]。而对于GpPurZ,Asp306突变为Asn (6FM1:Asn297)使GTP的kcat/KM约只有原来的1%,但并没有检测到这种突变体对ATP的活性[33]。值得注意的是,上述任意一种突变体都不能成功改造PurZ,使其底物从原来的dGMP转变成IMP[25],这说明PurZ和PurA底物的变化需要多点突变的协同,是长期演化的结果,以至于单突变体根本无法做到底物的切换。
2.2 Z基因组噬菌体通过编码dATPase基因防止dATP进入DNA
为了探究是否还有其他酶参与Z基因组生物合成,赵素文/张雁/赵惠民团队了通过分析在基因组上的邻近区域,发现多个噬菌体基因组中都存在一个HD结构域蛋白。HD结构域通常是一种结合DNA的结构域,而这些噬菌体中的HD结构域蛋白和普里斯特氏菌(megaterium)中发现的脱氧核苷三磷酸水解酶存在远缘关系,说明该蛋白的功能可能也是脱氧核苷三磷酸水解酶。经过HPLC-MS实验验证,S-2L和SH-Ab 15497中该酶的底物是dATP,在水解dATP时,可以经过一步反应就切掉三磷酸,将dATP变为dA,所以该酶是脱氧腺苷三磷酸水解酶,并因此被命名为dATPase (或称为DatZ[36]) (图2A)。实验还表明,S-2L、SH-Ab 15497和PMBT28的dATPase的活性,都是在使用Co2+作为金属辅因子时最高。另外,该酶是dATP专一性的,对包括dZTP在内的其他NTPs和dNTPs的活性非常低[25]。dATP的表观KM值在先前报道的细菌胞内dATP浓度范围内,为6.5~74.8 mM[25]。当然,除了直接催化dATP,dATPase还可催化dADP和dAMP分别脱去焦磷酸和磷酸,生成dA[25]。
Marc Delarue 课题组解析了S-2L dATPase的晶体结构[36]。目前有三种含不同结合配体的结构:结合dA、没加金属离子但结合了一个Zn2+的(PDB:6ZPA),结合dA、加入且结合了两个Co2+的(PDB:6ZPB),结合dATP、加入EDTA金属螯合剂但仍结合了一个Zn2+的(PDB:6ZPC)。从结构中可以观察到,与碱性磷酸酶和3′-5′外切酶[37,38]类似,S-2L dATPase使用典型的双金属离子机制使dATP去磷酸化(图5)。这些噬菌体dATPase的多序列比对显示[36],所有稳定两种催化金属离子的残基都是严格保守的;与α-,β-和γ-磷酸具有相互作用的Arg19、Lys81和Lys116(使用S-2L dATPase编号,下同)是保守的;与碱基具有相互作用的Trp20、Ile22、Pro79是保守的或发生保守取代的。
总之,dATPase可以降低dATP在胞浆里的浓度,使dATP和dZTP相比,在被噬菌体的DNA聚合酶DpoZ整合进入Z基因组时失去竞争力,从而解释了Z噬菌体基因组中A完全缺失的原因。
2.3 Z基因组噬菌体通过编码duf550/mazZ基因提供PurZ的原料dGMP
赵素文/张雁/赵惠民团队还在多个噬菌体的基因附近,发现了另外一种编码核苷三磷酸水解酶的基因:/,相应的蛋白是脱氧腺苷/脱氧鸟苷三磷酸的焦磷酸水解酶(简称DUF550[25]或MazZ[28])。S-2L DUF550的活性实验表明,活性最高的底物是dGTP/GTP,产物是 dGMP/GMP,包括dZTP在内的其他dNTP都不是该酶的底物[28]。SH-Ab 15497 DUF550活性最高的两个底物是dATP和dGTP,相应的产物是dAMP和dGMP[25]。这两种DUF550都是结合Co2+时活性最高[25](图2A)。
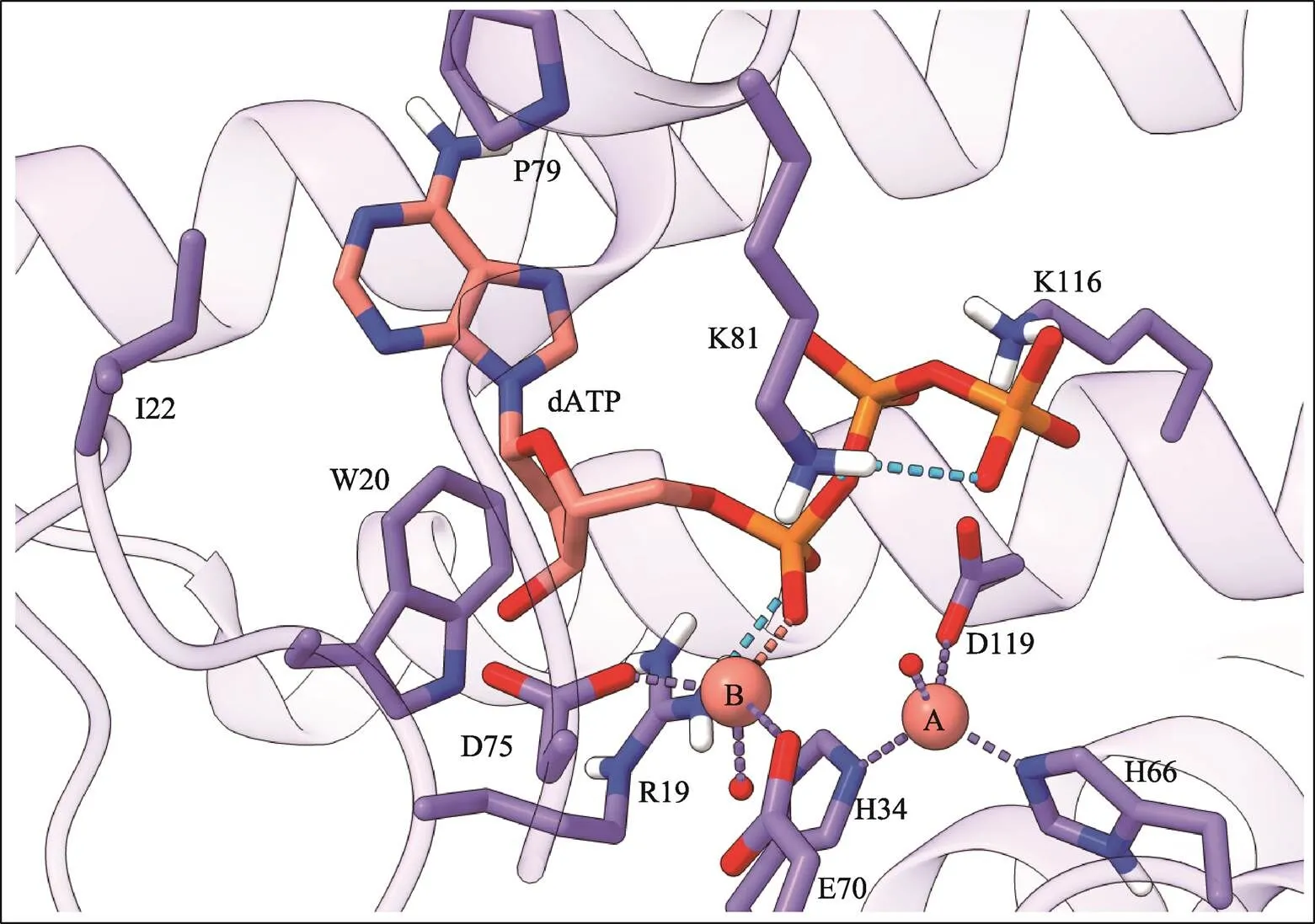
图5 Cyanophage S-2L dATPase (6ZPB) 的催化口袋示意图
S-2L dATPase使用经典的双金属离子(A和B点,结合Co2+有最高活性)机制去磷酸化。蓝色虚线为氢键,紫色和粉色虚线为金属离子键。
根据序列特征,Marc Delarue 课题组将DUF550分成两类:DUF550-1,存在于SH-Ab 15497、PMBT28等噬菌体中;DUF550-2,存在于PhiVC8、交替单胞菌噬菌体ZP6(phage ZP6)等噬菌体中。
Marc Delarue 课题组解析了S-2L DUF550、dGDP和三个Mn2+的复合物结构(图6A,PDB:7ODY)[28]。在该结构中,三个Mn2+的位置与弯曲杆菌中的一种脱氧尿苷焦磷酸水解酶[39](PDB:1W2Y)的三个Mg2+位置是基本相同的(图6A,A、B、C位置)。离子A与Glu34、Glu35、Glu38配位,离子B与Glu50和Asp53配位,而离子C与Glu38和Glu50配位(图6B)。除Glu34外,S-2L DUF550中的Ile56、Asp60和与催化离子配位的残基,在所有DUF550-1中都严格保守。DUF550-2中,Glu35、Glu38、Glu50和Glu53(S-2L MazZ编号)同样保守,但碱基结合残基与DUF550-1不一致,可能意味着底物的另一种结合模式[28]。因为上述实验观察到DUF550也具有水解dATP的活性,所以根据目前结构猜测,其结合dATP时,可能需要Asn20发生构象翻转,使用侧链的羰基结合腺嘌呤的6-氨基。
综上,噬菌体中的DUF550不仅可以给PurZ的反应提供原料dGMP,可能也协助降低了dATP在胞浆中的浓度。
2.4 Z基因组噬菌体利用宿主的腺苷琥珀酸裂解酶和鸟苷激酶合成dZTP
噬菌体在利用PurZ合成出dSMP后,与腺嘌呤核苷酸的合成通路类似地,接下来又利用宿主体内的腺苷琥珀酸裂解酶(PurB)、鸟苷激酶(Gmk)和核苷二磷酸激酶(Ndk)完成自身dZMP和dZTP的合成。目前除了链霉菌噬菌体Hiyaa(phage Hiyaa)、土地戈登氏菌噬菌体Ghobes和Archimedes的基因组编码了PurB类似物[27](图2A),其他含PurZ的噬菌体基因组中并未发现PurB类似物;并且研究者在这些噬菌体基因组中也并未发现编码可能催化dZMP磷酸化的相关激酶基因。
实验证实,SbPurZ反应后的产物和重组大肠杆菌PurB共同孵育后能够生成dZMP[25],同样地,VpPurZ和PhiVC8宿主霍乱弧菌()的PurB在ATP、dGMP、L-Asp和Mg2+条件下的产物也是dZMP,说明宿主的PurB能够被噬菌体利用来生成dZMP。最后,dZMP经过Gmk和Ndk的处理(或Gmk单一处理[25]),即可转化为dZTP[26]。为了进一步说明噬菌体这两步反应对宿主的依赖性,Pierre A. Kaminski课题组构建了霍乱弧菌 O1的突变体(使PurB表达减少),和霍乱弧菌O1的敲除株。他们发现含噬菌体PhiVC8对含突变体宿主的感染率仅是原来的10–5倍,而对敲除株宿主则是直接失去了感染性[26]。这些结果说明,宿主的和基因对Z基因组噬菌体的遗传物质复制起到关键的作用。
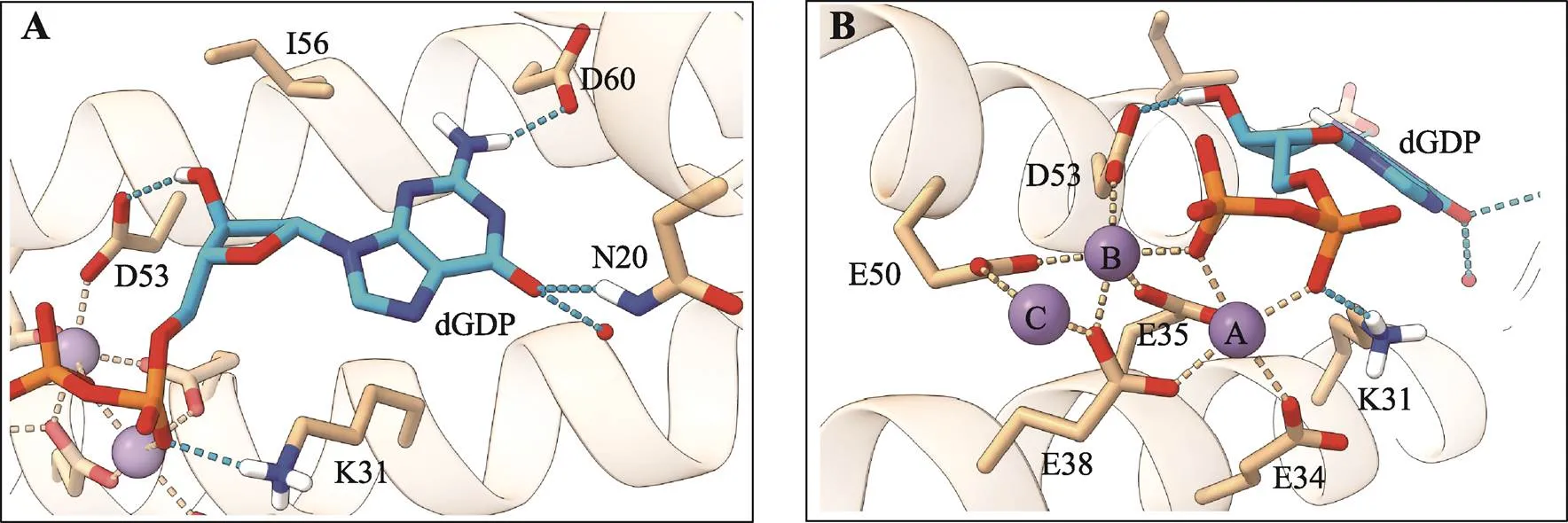
图6 S-2L MazZ (7ODY) 的催化口袋示意图
A:底物dGDP与残基结合示意图;B:三个催化金属离子(结合Co2+时有最高活性)与残基结合示意图。蓝色虚线为氢键,黄色虚线为金属离子键。
2.5 Z基因组噬菌体中的DNA聚合酶DpoZ
所有细胞生物和病毒都通过DNA或RNA聚合酶来复制它们的基因组。因此,噬菌体利用宿主体内的PurB、Gmk和Ndk成功合成dZTP后,还需要DNA聚合酶来发挥功能,使dZTP最终整合到其基因组中。已知的DNA聚合酶可以分成几个家族:A, B, C, D, X, Y, RT和PrimPol[40,41]。除了S-2L,其他一些含有PurZ的噬菌体基因组中都编码了DNA聚合酶A家族(DNA polA)的基因(图2A),与大肠杆菌DNA polA的Klenow片段相对应,含有具有校对功能的3′-5′外切酶结构域和聚合功能的5′-3′聚合酶结构域[27]。对于PhiVC8、SH-Ab 15497、Wayne等宿主涉及3个细菌门的10个噬菌体,Philippe Marliere课题组以相同方法分别构建其PurZ和DpoZ的系统发育树,发现两者的分支关系几乎一致(除放线菌门-放线菌纲-链霉菌目的Hiyaa,其基因组是其他噬菌体的两倍大)[27],说明这两种酶很可能共存于一个以Z为信息载体的共同祖先中,随着35亿年前的放线菌、蓝藻和变形菌一直演化至今。
Philippe Marliere课题组还发现,DpoZ相比A会更偏好Z的聚合[27]。在分别将dZTP和dATP生成到24 bp长度的实验中,四种噬菌体(PhiVC8、SH-Ab 15497、Ghobes、Wayne)的DpoZ在经过统一突变外切酶结构域使其失活后,都是聚合dZ比聚合dA有更高的效率。PhiVC8和SH-Ab 15497的DpoZ以dZTP作为底物的效率分别是以dATP为底物的90和29倍。另外,SH-Ab 15497的引物延伸实验还显示,无论模板中是否有Z,DpoZ都更偏好dZTP底物,大肠杆菌Klenow酶则展示出对dATP底物和含A模板的偏好性[27]。
Marc Delarue课题组希望能够找到这些噬菌体DpoZ结构上的特殊之处,来解释其更偏好Z的原因。于是,他们解析了PhiVC8 apo-DpoZ的晶体结构(PDB:7PBK)(图7A),其由3′-5′外切酶和5′-3′聚合酶结构域组成。在这个二聚体结构中,两个单体具有不同的构象,主要涉及拇指(thumb)区和3′-5′外切酶(exonuclease)区的构象变化,因此分别被称为“thumb-exo”关闭和开启状态构象[42]。而典型的polA结构的开放和闭合形式主要涉及的是手指(finger)区[43,44]。Marc Delarue等[42]认为,可能并不是酶本身的活性位点导致对Z的偏好,而是新生碱基对的双键/三键造成了酶不同的动力学方向。即,较强的新生Z:T对(三键)使聚合酶主要停留在延伸模式(“thumb-exo”关闭状态),而较弱的A:T对(双键)更容易导致聚合酶的校对模式(“thumb-exo”开启状态),造成新合并碱基的切除[42](图7B)。然而,仍缺少有力的实验来证明这一观点。
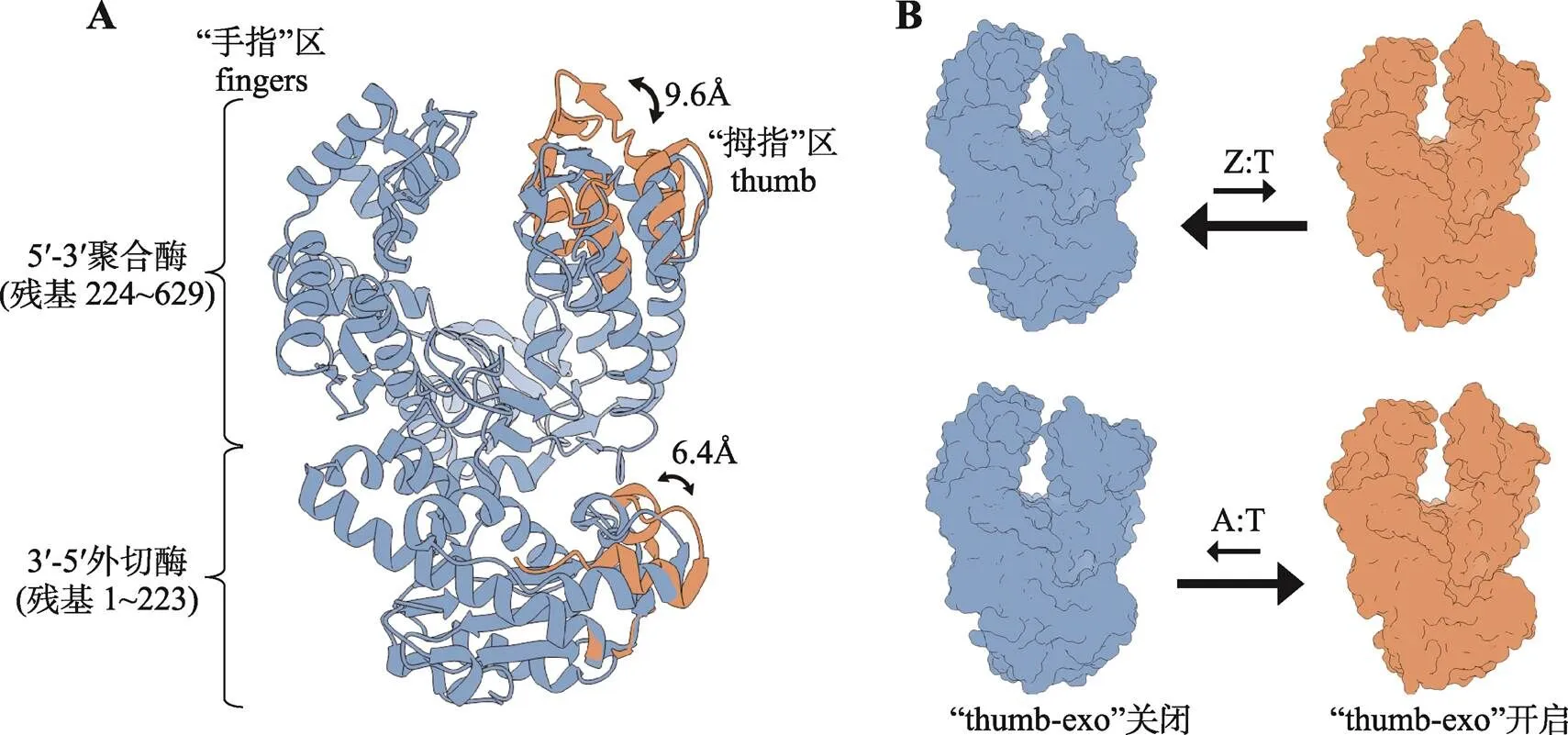
图7 PhiVC8 DpoZ (7PBK) 的结构域组成及两种构象
A:PhiVC8 DpoZ由聚合酶结构域和外切酶结构域组成。蓝色为“thumb-exo”关闭构象,橙色为“thumb-exo”开启构象。B:新生碱基对的双键/三键造成了酶的不同动力学方向。
不同于其他Z基因组噬菌体,S-2L的DNA聚合酶属于另外一个家族,PrimPol。S-2L PrimPol从N到C端包含三个结构域:AEP结构域、PriCT-2结构域、VirE家族结构域。随着从C到N端结构域的逐步缺失,聚合酶活性逐渐降低,但只要存在AEP结构域,就基本保持活性。虽然Z基因组噬菌体中PrimPol酶并不对dZTP或者dATP的使用具有选择性,聚合酶活性实验显示,相比于dZTP,S-2L PrimPol更倾向于使用dATP;但如果在加入PrimPol的同时也加入S-2L dATPase,则聚合酶产生的DNA中便无法观察到A的存在[36]。
考虑到上述Marc Delarue等[42]的观点缺乏有力的实验证明,以及S-2L的PrimPol聚合酶并不支持这一观点,因此,对于噬菌体能够避免A进入基因组而完全使用Z的原因,赵素文/张雁/赵惠民团队持有不同的看法。他们认为,噬菌体编码的DNA聚合酶并没有那么特殊,它也可以使用dATP,关键是要在细胞内提高dZTP的浓度和降低dATP的浓度。他们的实验表明,只要dZTP的浓度和dATP的浓度相当,那么利用普通聚合酶(如和Q5)生成的DNA产物中,Z就已经可以100%取代A(尚未发表),因为Z可以轻松地通过配对能量上的优势胜出。
3 结语和展望
对噬菌体中Z碱基及其生物合成通路的研究不仅刷新了人们对生物学基本观念的认知,而且由此发现的Z核苷及Z基因组合成通路中的各种酶都有着广泛的应用前景。
噬菌体中Z基因组生物合成通路的发现,使在细胞生物的基因组中引入Z成为可能。法国巴斯德所的Marc Delarue课题组发现,在大肠杆菌相容表达质粒中导入、和基因并表达培养后,能够观察到质粒和染色体上分别平均有12.1%和3.9%的A被Z替代,且缺少其中任意一个基因,替代率就会降低[28]。所以,通过引入Z基因组噬菌体合成通路中噬菌体编码的上述三个基因,细菌DNA上就能出现Z碱基。这也说明细菌的DNA聚合酶在在体的情况下是可以使用dZTP进行DNA聚合的。
Z碱基合成通路的发现,也使批量生产Z核苷成为可能,为合成生物学提供了便利,借天然酶来实现多种类型核苷合成。例如,张雁/赵惠民/刘胜男课题组成功地使用华杆菌宏基因组噬菌体片段的PurZ(SbPurZ)和大肠杆菌的PurB将黄嘌呤脱氧核苷酸(xanthine deoxynucleotide,dXMP)转化为异鸟嘌呤脱氧核苷酸(isoguanine deoxynucleotide,dBMP)[45](图8)。若对PurZ家族进行改造,还有可能实现更加多样的嘌呤脱氧核苷的酶法合成。
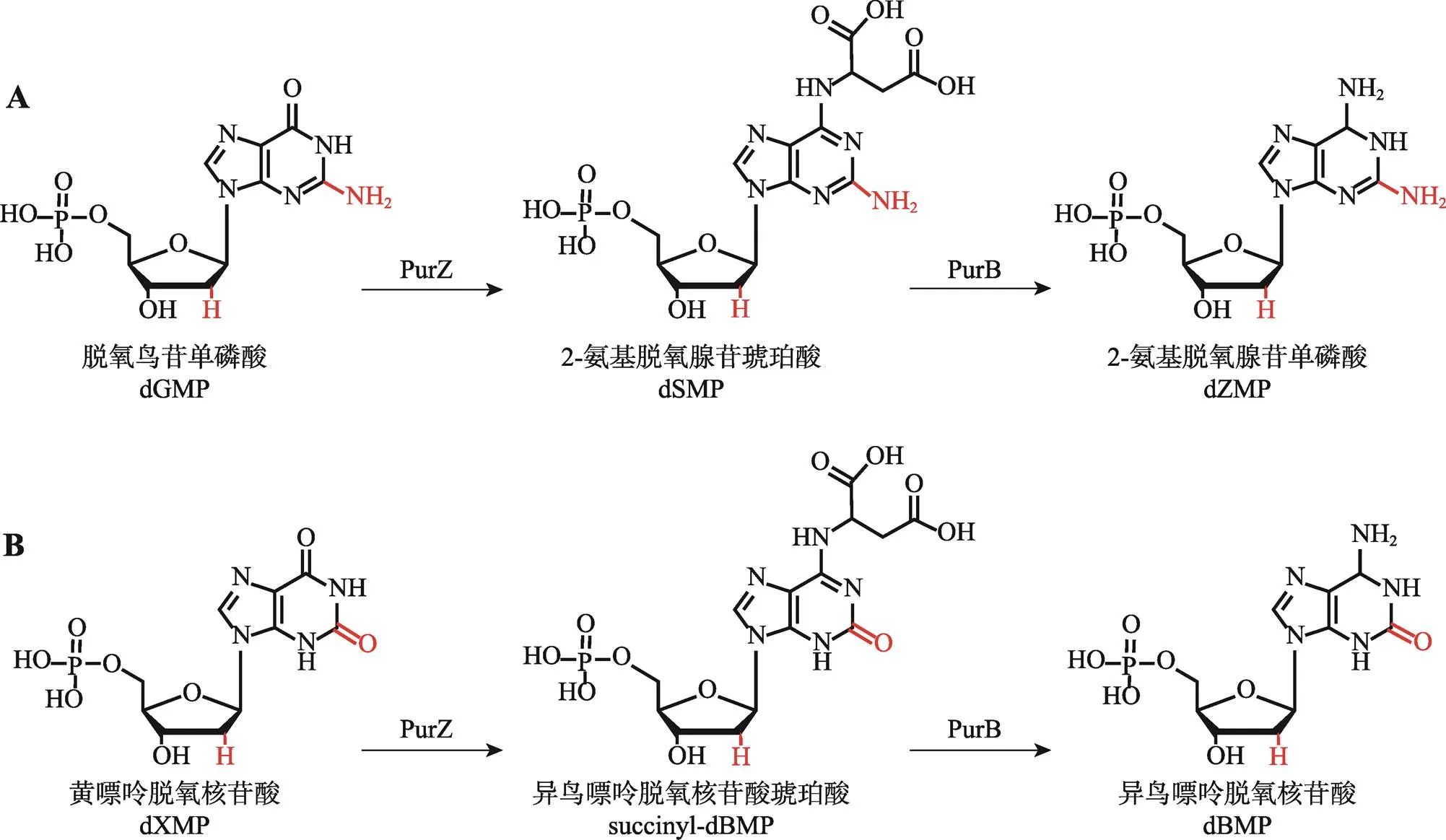
图8 PurZ和PurB可以催化dXMP成为dBMP
A:Z-噬菌体中利用PurZ和PurB合成dZMP;B:不经过酶改造,利用SbPurZ和大肠杆菌PurB就可以催化dXMP成为dBMP。
非经典核苷、DNA的生物合成,还可以应用于DNA存储、DNA折纸等新兴技术[46,47]。DNA本质就是储存遗传信息的载体,在如今数据量爆炸的时代,DNA存储凭借着存储密度高、稳定性强、维护成本低等特点,已成为国家重点研究的新兴技术[48]。上述报道的DNA修饰酶可以作为工具酶,参与信息存储系统的重新编写等过程,有利于DNA存储走向更广阔的应用场景。
在医疗领域,dZ-DNA还可能应用于医疗诊断和治疗。张等[49]的研究表明,与仅含有天然四碱基的DNA相比,包含合成碱基Z(非上述Z,6-氨基-5-硝基-3-(1′-β-D-2′-脱氧核糖)-2(1H)-吡啶酮)和P(2-氨基-8-(1′-β-D-2′-脱氧核糖)-咪唑[1,2-a]-1,3,5-三嗪- 4(8H)-丙酮)的DNA链能够更好地与癌细胞结合。因此,dZ-DNA也可能被设计成探针,用于疾病的诊断。
另外,目前噬菌体疗法面临的一大挑战,就是细菌利用限制修饰系统或CRISPR-Cas系统进行的噬菌体抵御,导致疗法效果下降[50]。由于Z基因组噬菌体对多种识别A的限制性内切酶的高抵抗性[21,22],可能使得天然或改造的Z基因组噬菌体具有治疗耐药细菌感染的潜力。对于CRISPR-Cas系统,目前只有含糖基化5-hmdC修饰的大肠杆菌噬菌体T4抵抗CRISPR-Cas9系统的报道[5],对于含其他修饰噬菌体的抵抗情况,仍有待进一步探索。
综上所述,本文重点介绍的Z基因组生物合成通路不仅对噬菌体自身的生存有着重要意义,还有着广阔的应用前景:该通路中各种酶的发现,能够成为合成生物学的砖瓦,用来合成非常规嘌呤或在细胞生物的基因组中引入Z;也可以利用此通路批量合成dZ-DNA,作为材料或诊断试剂使用;而Z基因组噬菌体,也具有潜在的抵抗耐药菌的能力,将来可能在噬菌体疗法中发挥重要作用。
[1] Liyanage VRB, Jarmasz JS, Murugeshan N, Del Bigio MR, Rastegar M, Davie JR. DNA modifications: function and applications in normal and disease States., 2014, 3(4): 670–723.
[2] Weigele P, Raleigh EA. Biosynthesis and function of modified bases in bacteria and their Viruses., 2016, 116(20): 12655–12687.
[3] Gommers-Ampt JH, Borst P. Hypermodified bases in DNA., 1995, 9(11): 1034–1042.
[4] Wyatt GR, Cohen SS. The bases of the nucleic acids of some bacterial and animal viruses: the occurrence of 5-hydroxymethylcytosine., 1953, 55(5): 774–782.
[5] Bryson AL, Hwang Y, Sherrill-Mix S, Wu GD, Lewis JD, Black L, Clark TA, Bushman FD. Covalent modification of bacteriophage T4 DNA inhibits CRISPR-Cas9., 2015, 6(3): e00648.
[6] Kuo TT, Huang TC, Teng MH. 5-Methylcytosine replacing cytosine in the deoxyribonucleic acid of a bacteriophage for Xanthomonas oryzae., 1968, 34(2): 373–375.
[7] Swinton D, Hattman S, Benzinger R, Buchanan-Wollaston V, Beringer J. Replacement of the deoxycytidine residues in Rhizobium bacteriophage RL38JI DNA., 1985, 184(2): 294–298.
[8] Kallen RG, Simon M, Marmur J. The new occurrence of a new pyrimidine base replacing thymine in a bacteriophage DNA:5-hydroxymethyl uracil., 1962, 5: 248–250.
[9] Lee YJ, Dai N, Walsh SE, Müller S, Fraser ME, Kauffman KM, Guan CD, Corrêa IR, Weigele PR. Identification and biosynthesis of thymidine hypermodifications in the genomic DNA of widespread bacterial viruses., 2018, 115(14): E3116–E3125.
[10] Warren RA. Modified bases in bacteriophage DNAs., 1980, 34: 137–158.
[11] Kropinski AM, Bose RJ, Warren RA. 5-(4- Aminobutylaminomethyl)uracil, an unusual pyrimidine from the deoxyribonucleic acid of bacteriophage phiW-14., 1973, 12(1): 151–157.
[12] Swinton D, Hattman S, Crain PF, Cheng CS, Smith DL, McCloskey JA. Purification and characterization of the unusual deoxynucleoside, alpha-N-(9-beta-D-2′- deoxyribofuranosylpurin-6-yl)glycinamide, specified by the phage Mu modification function., 1983, 80(24): 7400–7404.
[13] Thiaville JJ, Kellner SM, Yuan YF, Hutinet G, Thiaville PC, Jumpathong W, Mohapatra S, Brochier-Armanet C, Letarov AV, Hillebrand R, Malik CK, Rizzo CJ, Dedon PC, de Crécy-Lagard V. Novel genomic island modifies DNA with 7-deazaguanine derivatives., 2016, 113(11): E1452–E1459.
[14] Tsai R, Corrêa IR, Xu MY, Xu SY. Restriction and modification of deoxyarchaeosine (dG+)-containing phage 9 g DNA., 2017, 7(1): 8348.
[15] Hutinet G, Kot W, Cui L, Hillebrand R, Balamkundu S, Gnanakalai S, Neelakandan R, Carstens AB, Fa Lui C, Tremblay D, Jacobs-Sera D, Sassanfar M, Lee YJ, Weigele P, Moineau S, Hatfull GF, Dedon PC, Hansen LH, de Crécy-Lagard V. 7-Deazaguanine modifications protect phage DNA from host restriction systems., 2019, 10(1): 5442.
[16] Kirnos MD, Khudyakov IY, Alexandrushkina NI, Vanyushin BF. 2-Aminoadenine is an adenine substituting for a base in S-2L cyanophage DNA.,1977, 270(5635): 369–370.
[17] Khudyakov IY, Kirnos MD, Alexandrushkina NI, Vanyushin BF. Cyanophage S-2L contains DNA with 2,6-diaminopurine substituted for adenine., 1978, 88(1): 8–18.
[18] Cheong C, Tinoco I, Chollet A. Thermodynamic studies of base pairing involving 2,6-diaminopurine., 1988, 16(11): 5115–5122.
[19] Sági J, Szakonyi E, Vorlícková M, Kypr J. Unusual contribution of 2-aminoadenine to the thermostability of DNA., 1996, 13(6): 1035–1041.
[20] Cristofalo M, Kovari D, Corti R, Salerno D, Cassina V, Dunlap D, Mantegazza F. Nanomechanics of diaminopurine- substituted DNA., 2019, 116(5): 760–771.
[21] Szekeres M, Matveyev AV. Cleavage and sequence recognition of 2,6-diaminopurine-containing DNA by site- specific endonucleases., 1987, 222(1): 89–94.
[22] Chollet A, Kawashima E. DNA containing the base analogue 2-aminoadenine: preparation, use as hybridization probes and cleavage by restriction endonucleases., 1988, 16(1): 305–317.
[23] Tan Y, You CJ, Park J, Kim HS, Guo S, Schärer OD, Wang YS. Transcriptional perturbations of 2,6-diaminopurine and 2-aminopurine., 2022, 17(7): 1672–1676.
[24] Philippe Marliere, Pierre-Alexandre Kaminski, Frédérique GALISSON, Madeleine BouzonSylvie, PochetJean Weissenbach, William Saurin, Catherine Robert, Virginie Vico. Banque genomique du cyanophage s-2l et analyse fonctionnelle. WO2003093461A2. 2003-11-13.
[25] Zhou Y, Xu XX, Wei YF, Cheng Y, Guo Y, Khudyakov I, Liu FL, He P, Song ZY, Li Z, Gao Y, Ang EL, Zhao HM, Zhang Y, Zhao SW. A widespread pathway for substitution of adenine by diaminopurine in phage genomes., 2021, 372(6541): 512–516.
[26] Sleiman D, Garcia PS, Lagune M, Loc'h J, Haouz A, Taib N, Röthlisberger P, Gribaldo S, Marlière P, Kaminski PA. A third purine biosynthetic pathway encoded by aminoadenine-based viral DNA genomes., 2021, 372(6541): 516–520.
[27] Pezo V, Jaziri F, Bourguignon PY, Louis D, Jacobs-Sera D, Rozenski J, Pochet S, Herdewijn P, Hatfull GF, Kaminski PA, Marliere P. Noncanonical DNA polymerization by aminoadenine-based siphoviruses., 2021, 372(6541): 520–524
[28] Czernecki D, Bonhomme F, Kaminski PA, Delarue M. Characterization of a triad of genes in cyanophage S-2L sufficient to replace adenine by 2-aminoadenine in bacterial DNA., 2021, 12(1): 4710.
[29] Jin JY, Zhou JH. The mystery of Z-genome biosynthesis has been elucidated., 2022, 3(1): 1–5.金交羽, 周佳海. Z-基因组的生物合成奥秘被揭示. 合成生物学, 2022, 3(1): 1–5.
[30] Callahan MP, Smith KE, Cleaves HJ, Ruzicka J, Stern JC, Glavin DP, House CH, Dworkin JP. Carbonaceous meteorites contain a wide range of extraterrestrial nucleobases., 2011, 108(34): 13995–13998.
[31] Wang ZY, Zhang F, Liang YT, Zheng KY, Gu CX, Zhang WJ, Liu YD, Zhang XR, Shao HB, Jiang Y, Guo C, He H, Wang HL, Sung YY, Mok WJ, Wong LL, He JF, McMinn A, Wang M. Genome and ecology of a novel alteromonas podovirus, ZP6, representing a new viral genus,., 2021, 9(2): e0046321.
[32] Honzatko RB, Fromm HJ. Structure-function studies of adenylosuccinate synthetase from Escherichia coli., 1999, 370(1): 1–8.
[33] Tong Y, Wu XY, Liu Y, Chen HY, Zhou Y, Jiang L, Li M, Zhao SW, Zhang Y. Alternative Z-genome biosynthesis pathway shows evolutionary progression from Archaea to phage., 2023, 8(7): 1330-1338.
[34] Wang W, Gorrell A, Honzatko RB, Fromm HJ. A study of Escherichia coli adenylosuccinate synthetase association states and the interface residues of the homodimer., 1997, 272(11): 7078–7084.
[35] Mehrotra S, Balaram H. Kinetic characterization of adenylosuccinate synthetase from the thermophilic archaea Methanocaldococcus jannaschii., 2007, 46(44): 12821–12832.
[36] Czernecki D, Legrand P, Tekpinar M, Rosario S, Kaminski PA, Delarue M. How cyanophage S-2L rejects adenine and incorporates 2-aminoadenine to saturate hydrogen bonding in its DNA., 2021, 12(1): 2420.
[37] Bochner BR, Ames BN. Complete analysis of cellular nucleotides by two-dimensional thin layer chromatography., 1982, 257(16): 9759–9769.
[38] Kim EE, Wyckoff HW. Reaction mechanism of alkaline phosphatase based on crystal structures: Two-metal ion catalysis., 1991, 218(2): 449–464.
[39] Moroz OV, Harkiolaki M, Galperin MY, Vagin AA, González-Pacanowska D, Wilson KS. The crystal structure of a complex of Campylobacter jejuni dUTPase with substrate analogue sheds light on the mechanism and suggests the "basic module" for dimeric d(C/U)TPases., 2004, 342(5): 1583–1597.
[40] Guilliam TA, Keen BA, Brissett NC, Doherty AJ. Primase-polymerases are a functionally diverse superfamily of replication and repair enzymes., 2015, 43(14): 6651–6664.
[41] Braithwaite DK, Ito J. Compilation, alignment, and phylogenetic relationships of DNA polymerases., 1993, 21(4): 787–802.
[42] Czernecki D, Hu HD, Romoli F, Delarue M. Structural dynamics and determinants of 2-aminoadenine specificity in DNA polymerase DpoZ ofphage ϕVC8., 2021, 49(20): 11974–11985.
[43] Li Y, Korolev S, Waksman G. Crystal structures of open and closed forms of binary and ternary complexes of the large fragment of Thermus aquaticus DNA polymerase I: structural basis for nucleotide incorporation., 1998, 17(24): 7514–7525.
[44] Wu EY, Beese LS. The structure of a high fidelity DNA polymerase bound to a mismatched nucleotide reveals an “Ajar” intermediate conformation in the nucleotide selection mechanism., 2011, 286(22): 19758–19767.
[45] Ceze L, Nivala J, Strauss K. Molecular digital data storage using DNA., 2019, 20(8): 456–466.
[46] Zhao FP, Wei YF, Wang XY, Zhou Y, Tong Y, Ang EL, Liu SN, Zhao HM, Zhang Y. Enzymatic synthesis of the unnatural nucleotide 2′-deoxyisoguanosine 5′-monophosphate., 2022, 23(21): e202200295.
[47] Rothemund PWK. Folding DNA to create nanoscale shapes and patterns., 2006, 440(7082): 297–302.
[48] Zhang DL, Ge Q, Feng YB, Chen WG. Comparison and analysis on scientific research programs on DNA data storage., 2022, 42(6): 116–129.张大璐, 葛奇, 冯一博, 陈为刚. DNA数据存储的科研概况国际对比与分析. 中国生物工程杂志, 2022, 42(6): 116–129.
[49] Zhang LQ, Yang ZY, Sefah K, Bradley KM, Hoshika S, Kim MJ, Kim HJ, Zhu GZ, Jiménez E, Cansiz S, Teng IT, Champanhac C, McLendon C, Liu C, Zhang W, Gerloff DL, Huang Z, Tan WH, Benner SA. Evolution of functional six-nucleotide DNA., 2015, 137(21): 6734–6737.
[50] Tian EK, Wang Y, Wu ZX, Wan ZQH, Cheng W. Bacteriophage therapy: retrospective review and future prospects., 2021, 52(2): 170–175.田而慷, 王玥, 吴卓轩, 万紫千红, 程伟. 噬菌体疗法:回顾与展望. 四川大学学报(医学版), 2021, 52(2): 170–175.
Progress on Z genome biosynthetic pathway of bacteriophage
Huiyu Chen, Suwen Zhao
There are abundant base modifications in bacteriophages’ genomes, mainly for avoiding the digestion of host endonucleases. More than 40 years ago, researchers discovered that 2-amino-adenine (Z) completely replaced adenine (A) and forms a complementary pairing with three hydrogen bonds with thymine (T) in the DNA of cyanophage S-2L, forming a distinct “Z-genome”. In recent years, researchers have discovered and validated the biosynthetic pathway of Z-genome in various bacteriophages, constituting a multi-enzyme system. This system includes the phage-encoded enzymes deoxy-2′-aminoadenylosuccinate synthetase (PurZ), deoxyadenosine triphosphate hydrolase (dATPase/DatZ), deoxyadenosine/deoxyguanosine triphosphate pyrophosphatase (DUF550/MazZ) and DNA polymerase (DpoZ). In this review, we provide a concise overview of the historical discovery on diversely modified nucleosides in bacteriophages, then we comprehensively summarize the research progress on multiple enzymes involved in the Z-genome biosynthetic pathway. Finally, the potential applications of the Z-genome and the enzymes in its biosynthetic pathway are discussed in order to provide reference for research in this field.
Z-genome; bacteriophage; DNA modification; 2-aminoadenine; biosynthetic pathway
2023-05-26;
2023-08-17;
2023-08-18
国家自然科学基金项目(编号:32122024),上海市生物大分子与精准医药前沿科学研究基地资助[Supported by the National Natural Science Foundation of China (No. 32122024), and Shanghai Frontiers Science Center for Bomacromolecules and Precision Medicine]
陈荟玉,在读硕士研究生,专业方向:计算化学与生物学。E-mail: chenhy6@shanghaitech.edu.cn
赵素文,博士,研究员,研究方向:计算化学与生物学。E-mail: zhaosw@shanghaitech.edu.cn
10.16288/j.yczz.23-059
(责任编委: 张天宇)