基于机器学习的猪生长性状基因组预测
2023-10-30陈栋王书杰赵真坚姬祥申琦余杨崔晟頔王俊戈陈子旸王金勇郭宗义吴平先唐国庆
陈栋,王书杰,赵真坚,姬祥,申琦,余杨,崔晟頔,王俊戈,陈子旸,王金勇,郭宗义,吴平先,唐国庆
研究报告
基于机器学习的猪生长性状基因组预测
陈栋1,2,3,王书杰1,2,3,赵真坚1,2,3,姬祥1,2,3,申琦1,2,3,余杨1,2,3,崔晟頔1,2,3,王俊戈1,2,3,陈子旸1,2,3,王金勇4,郭宗义4,吴平先4,唐国庆1,2,3
1. 四川农业大学动物科技学院,农业农村部畜禽生物组学重点实验室,成都 611130 2. 四川农业大学,畜禽遗传资源发掘与创新利用四川省重点实验室,成都 611130 3. 四川农业大学动物科技学院,猪禽种业全国重点实验室,成都 611130 4. 国家生猪技术创新中心,重庆 402460
为了比较自动机器学习下不同机器学习模型预测部分猪生长性状与全基因组估计育种值(genomic estimated breeding value,GEBV)的性能,并寻找适合的机器学习模型,以优化生猪育种的全基因组评估方法,本研究利用来自多个公司9968头猪的基因组信息、系谱矩阵、固定效应及表型信息通过自动机器学习方法获取深度学习(deep learning,DL)、随机森林(random forest,RF)、梯度提升机(gradient boosting machine,GBM)和极致梯度提升(extreme gradient boosting,XGB)4种机器学习最佳模型。采用10折交叉验证分别对猪达100 kg校正背膘(correcting backfat to 100 kg,B100)、达115 kg校正背膘(correcting backfat to 115 kg,B115)、达100 kg校正日龄(correcting days to 100 kg,D100)、达115 kg校正日龄(correcting days to 100 kg,D115)的GEBV及其表型进行预测,比较不同机器学习模型应用于猪基因组评估的性能。结果表明:机器学习模型对GEBV的估计准确性高于性状表型;在GEBV预测中,GBM在B100、B115、D100、D115的预测准确性分别为0.683、0.710、0.866、0.871,略高于其他方法;在表型预测中,对猪B100、B115、D100、D115预测性能最好的模型依次为GBM(0.547)、DL(0.547)、XGB(0.672、0.670);在模型训练所需时间上,RF远高于其他3种模型,GBM与DL居中,XGB所需时间最少。综上所述,通过自动机器学习获取的机器学习模型对GEBV预测的准确性高于表型;GBM模型总体上表现出最高的预测准确性与较短训练时间;XGB能够利用最短的时间训练准确性较高的预测模型;RF模型的训练时间远超其他3种模型,且准确性不足,不适用猪生长性状表型与GEBV预测。
基因组估计育种值;生长性状;自动机器学习;性能比较
生长性状是动物生产中最重要的经济性状,受众多基因的调控[1],对养殖业发展有着重要的影响[2,3]。全基因组选择方法通过直接检测基因信息,利用个体全基因组范围内的单核苷酸多态性(single nucleotide polymorphism,SNP)标记数据与表型数据相结合,在候选个体的生命早期估计出可靠性更高的基因组育种值对候选个体进行筛选[4,5]。相比于传统的表型选择方法,全基因组选择方法具有更高的预测准确性,可以更快地获得遗传进展[6~8]。
重测序技术和芯片技术是获取基因组信息的两种方式。测序数据比芯片数据含有更多的基因信息,但成本偏高。基因型填充[9]方法可以准确填充测序数据的SNP芯片缺失信息[10~12],同时也能够对多款不同规格的商业芯片进行SNP位点综合,在节约成本的基础上提高育种准确性。
机器学习是一种高效的数据处理方法,在动物遗传育种领域被广泛应用[13~15]。机器学习可以有效的处理基因组信息中的高维数据并建立非线性模型,从而更准确地预测动物的遗传价值[16]。但模型的选择与参数的优化决定了预测结果的准确性与效率,在机器学习的应用上至关重要。自动机器学习技术可以针对特定问题自动选择超参数,实现模型的优化,克服人工选择和调参的不足,达到高效使用机器学习技术的目的[17]。
本文旨在比较自动机器学习下不同机器学习模型对猪生长性状的全基因组估计育种值(genomic estimated breeding value,GEBV)和表型信息的预测性能,探讨自动机器学习在动物遗传育种中的应用价值和前景,找到合适的机器学习模型优化全基因组评估方法。通过对比不同机器学习模型的预测准确性和模型的训练时长,为猪生长性状的全基因组评估提供更加准确和可靠的机器学习方法。
1 材料与方法
1.1 研究材料
本研究以新希望、铁骑力士、四川巨星农牧、明兴等多家公司饲养的9968头大白猪为研究对象(公7266头,母2702头)。收集所有猪只的芯片数据、系谱及固定效应信息(性别、品系、场、胎次、出生年、测定年);收集达100 kg校正背膘(correcting backfat to 100 kg,B100)、达115 kg校正背膘(correcting backfat to 115 kg,B115)表型数据7871(公5903头,母1968头)条;收集达100 kg校正日龄(correcting days to 100 kg,D100)、达115 kg校正日龄(correcting days to 100 kg,D115)表型数据7113(公5401头,母1712头)条。详细数据组成见表1。
1.2 基因型填充
由于多个公司分别采用不同的商业芯片,无法进行统一评估,于是采用测序数据填充的方法对商业芯片进行合并。将4种康普森基因(不同SNP位点数)芯片和纽勤50K芯片数据进行合并得到位点并集。利用735头(384头杜洛克,277头大白,74头长白猪)猪的基因组重测序数据检测出28,763,360个高质量的SNPs作为芯片填充参考群。筛选商业芯片并集和填充参考群之间共有的SNP位点。利用Beagle(v5.1)软件通过填充参考群对每种商业芯片进行填充,根据共有SNP位点和质量控制,获得9968张含有85,542个SNP位点的高质量SNP数据集。
1.3 GEBV估计
通过一步法(single-step genomic best linear unbiased prediction,ssGBLUP)估计猪各生长性状的GEBV,模型如下:
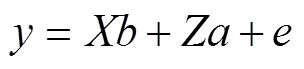
其中,为表型值向量;为固定效应向量;为基因组育种值向量,服从正太分布N(0,σg2),σg2为加性遗传方差,为亲缘关系矩阵;和为和对应的关联矩阵;为残差效应向量,服从正太分布N(0,σe2),σe2为残差方差。
ssGBLUP方法通过结合基于系谱的矩阵和基于基因型的矩阵构建亲缘关系矩阵,从而将无基因型的个体与具备基因型个体包含于同一个亲缘关系矩阵:
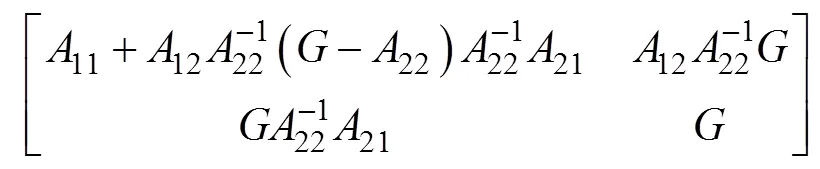
其中,矩阵中子矩阵11、12、21、22的下标1和2分别表示无基因型个体和具基因型个体。矩阵的构建方法为:
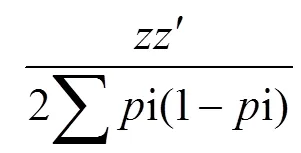
其中,Z矩阵中的元素0-2、1-2、2-2分别代表基因型AA、AB、BB。为位点的第二个等位基因频率。
1.4 机器学习模型
本研究所涉及的机器学习方法均通过H2O.ai平台实现(https://docs.h2o.ai/h2o/-latest-stable/h2o- docs/ index.html)。在模型训练过程中,通过10折交叉验证以确保所有样本数据都被预测且不包括在模型的训练集中。具体方法是将数据集划分为10组,确保每组中的数据量尽可能相同并保证每个公司的数据在不同分组中的分布大致均匀。此外,在4种机器学习方法中都分别指定标签参数为GEBV和表型信息,特征参数为基因型矩阵、系谱矩阵和固定效应,以进行GEBV和表型信息的预测。
1.5 自动机器学习
自动机器学习通过自动搜索和选择模型的最优超参数实现机器学习的自动化,使得机器学习的应用更加高效和便捷[18]。本研究中,指定模型参数include_algos包含“GBM”、“DRF”、“XGBoot”和“DeepLearning”,利用自动机器学习方法对每一个生长性状的GEBV与表型都训练了4种机器学习模型。分别指定标签参数为GEBV和表型信息,特征参数为基因型矩阵、系谱矩阵和固定效应。指定自动机器学习时间为7天,从而在指定时间范围内找到每种方法表现最好的模型参数。利用所获得的模型参数对5种猪生长性状的GEBV与表型进行4种机器学习模型的训练。
1.5.1 GBM模型
梯度提升机(gradient boosting machine,GBM)[19]是一种基于决策树的集成学习算法,通过迭代的方法完成模型的构建。每一次迭代都会训练一个新的分类器,并将其与之前的分类器组合起来[20]。每个新的分类器都会尝试纠正前一个分类器犯的错误[21]。这种迭代过程会一直进行下去,直到分类器的错误率达到一个可接受的水平,完成训练。本研究中,猪生长性状GEBV与表型的GBM预测模型超参数见表2 (未列出的超参数均为默认值,下文相同)。
1.5.2 RF模型
随机森林(random forest,RF)[22]算法是一种集成学习算法,通过将多个决策树组合成一个模型来提高预测性能。RF算法的训练过程中,会对原始数据进行随机采样,以得到多个不同的训练数据集。对于每个训练数据集,都会构建一个独立的决策树,最终将这些决策树组合成一个随机森林。在预测时,随机森林会根据每个决策树的预测结果,投票决定最终的预测结果[23]。该算法能够处理大规模数据集,并且具有较高的准确率和稳定性[24]。本研究中,猪生长性状GEBV与表型的RF预测模型参数见表3。
1.5.3 XGB模型
极致梯度提升(extreme gradient boosting,XGB)[25]模型是一种监督学习算法,它通过梯度提升的方法获取准确的模型,可用于解决分类和回归问题[26]。相比GBM,它能够通过贪心算法找到最优的分裂节点并通过并行处理的方法使得在大数据集能够更快的完成模型训练。本研究中,猪生长性状GEBV与表型的XGB预测模型参数见表4。
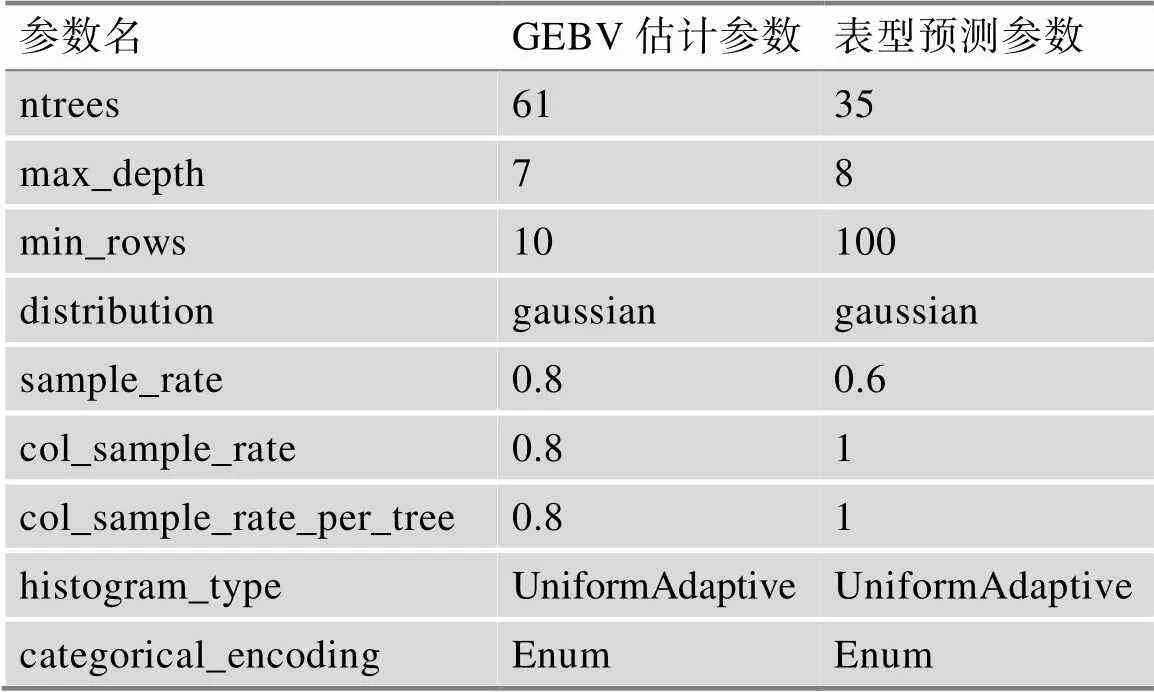
表2 GBM模型超参数表
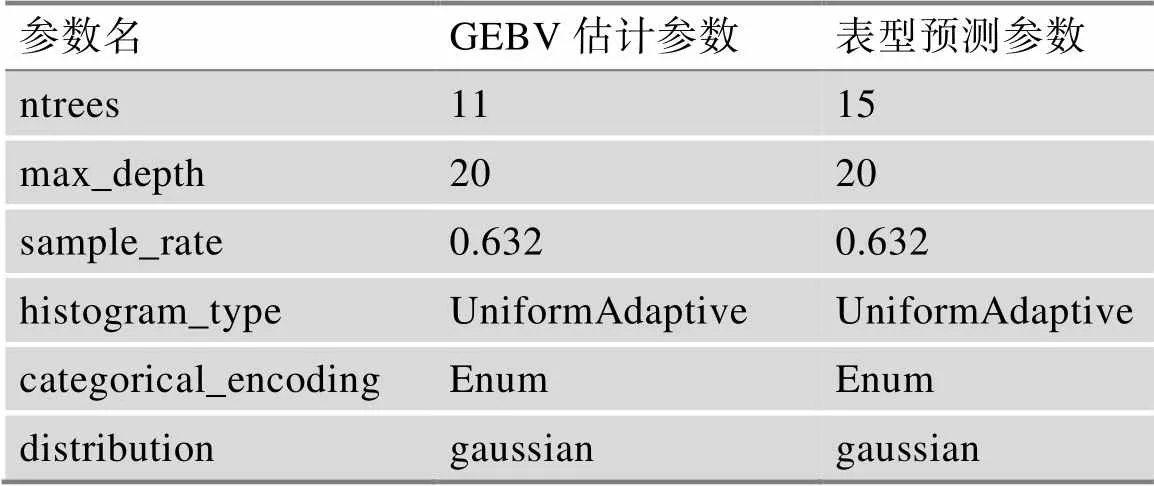
表3 RF模型超参数表
1.5.4 DL模型
H2O平台提供的深度学习(deep learning,DL)方法基于多层前馈人工神经网络,相对于机器学习,DL能够更深入的进行特征提取,模仿神经元的传导构建多层网络框架[27]。人工神经网络中的多层神经元[28]通过逐层对初级信号进行特征转化,不断将原空间样本的特征转化到新的特征空间,自动学习得到新的特征进而更新权重,最终获得表现优秀的DL模型。本研究中,猪生长性状GEBV与表型的DL预测模型参数见表5。
1.6 统计与分析
本研究使用python3.8.13对实验结果进行统计与分析。调用pandas、numpy、scipy、statistics与sklearn.metrics库完成对预测结果指标的计算,利用matplotlib进行结果可视化展示。
1.7 模型性能评判
1.7.1 准确性
本研究中,模型准确性由机器学习方法预测的基因组估计育种值(genome estimated breeding value predicted by the model,MGEBV)与一步法计算的育种值(genome estimated breeding value calculated by single-step method,SGEBV)之间的相关系数()确定,即:
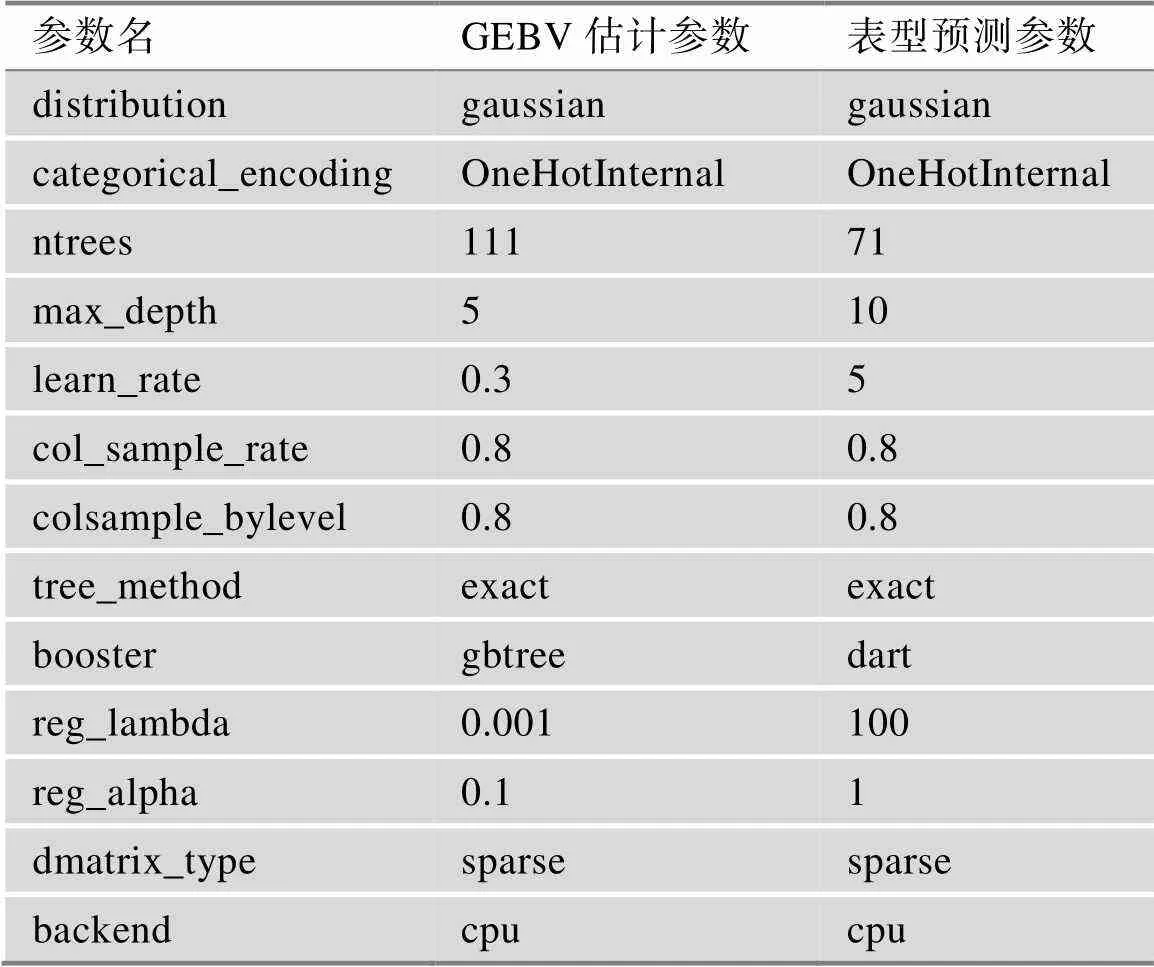
表4 XGB模型超参数表
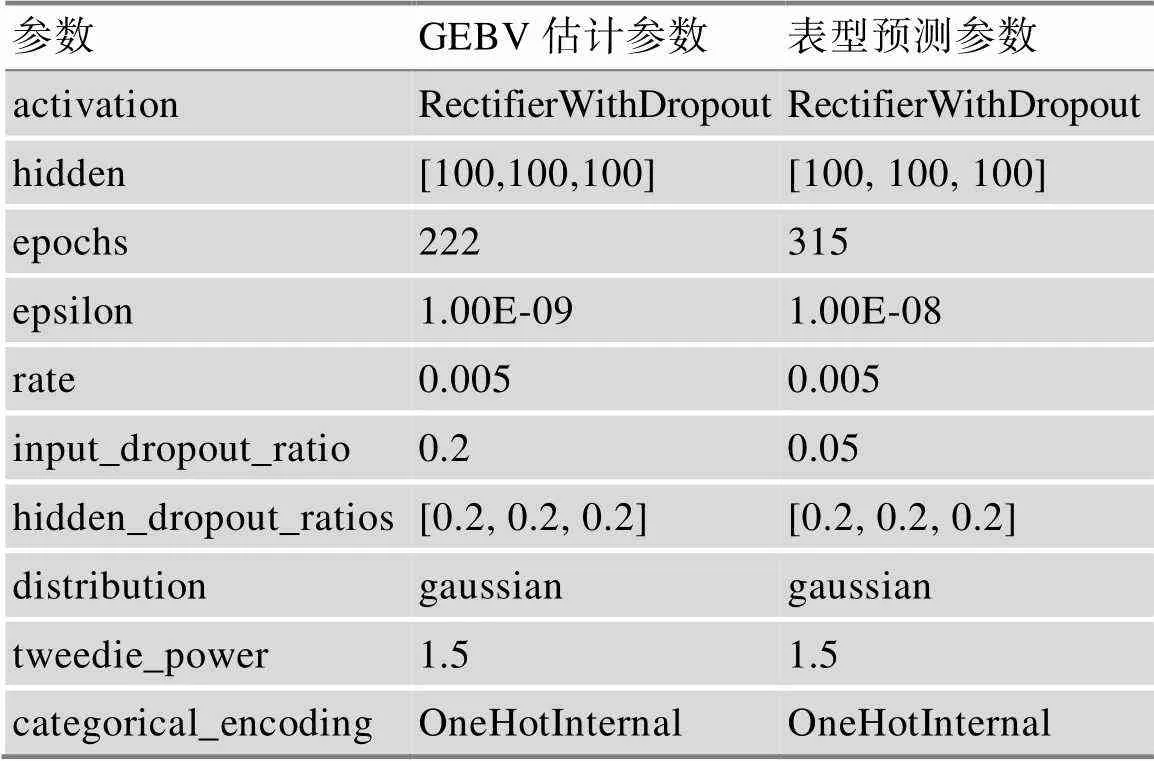
表5 DL模型超参数表
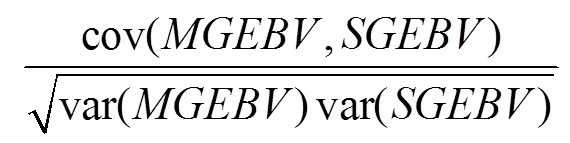
预测值与实际值的相关系数越高表明模型的准确性越高。
1.7.2 模型辅助评估指标
拟合系数(R squared,R2)、偏差(BIAS)、均方误差(mean squared error,MSE)作为模型的辅助评估指标,用于辅助评估机器学习模型的预测性能。它们分别可以表现模型解释变量对于被解释变量的解释程度、模型的预测结果与真实结果的偏离程度以及差异程度。
1.7.3 模型训练时长
全基因组估计育种值与表型往往需要较为庞大的SNP数据,还需要大量迭代运行以实现最佳效果。因此,模型训练时长是GEBV与表型估计应用的关键因素之一。本研究从模型开始训练时计时直至模型完成训练,训练时长越短的方法的实用性就越高。
2 结果与分析
2.1 GEBV预测的机器学习模型整体表现出优秀的预测性能
2.1.1 模型预测性能
经过十倍交叉验证得到了4种机器学习模型,利用模型对猪B100的GEBV预测结果见图1。4种机器学习模型的预测准确性相近,依次为0.683 (GBM)、0.673 (DL)、0.667 (XGB)、0.648 (RF)。4个模型的R2均大于0.4,BIAS在0附近,MSE均在0.3左右,都表明模型具有不错的预测性能。
4种机器学习模型在B115的GEBV预测性能表现上略高于B100,但模型准确性排名与B100GEBV相同(附图1)。4种机器学习模型的准确性结果依次为0.710 (GBM)、0.702 (DL)、0.694 (XGB)、0.671 (RF)。其中GBM模型的R2值达到了0.503,MSE也小于其他3种模型。
4种机器学习模型对于D100与D115的GEBV预测准确性整体上高于B100与B115。机器学习模型D100GEBV的预测准确性依次为0.866(GBM)、0.844 (RF)、0.830 (XGB)、0.824 (DL) (图2)。其中GBM模型R2值达到了0.750。由于校正日龄GEBV的数值远大于校正背膘GEBV,所以MSE在20左右,其中GBM模型最小(16.405)。
各机器学习模型对猪D115的GEBV预测性能与D100GEBV相似,且预测准确性均略高于模型对D100GEBV的预测(附图2)。准确性排名依次为0.871 (GBM)、0.849 (RF)、0.835 (XGB)、0.829 (DL)。其中GBM的R2最高为0.759,且MSE最小(21.583)。
这些结果表明不同模型对不同性状GEBV的估计能力各有不同。但相对于其他3种模型,GBM在猪生长性状的GEBV估计中表现出了更好的性能。
2.1.2 训练时长
生长性状GEBV估计的4种机器学习模型的训练时长对比如图所示(图3),在4种机器学习模型中,RF模型训练时长远超其他模型,这与郭鹏等[6]的结果类似。DL模型与GBM模型居中。XGB模型训练所需时长最少,仅需30 min左右。
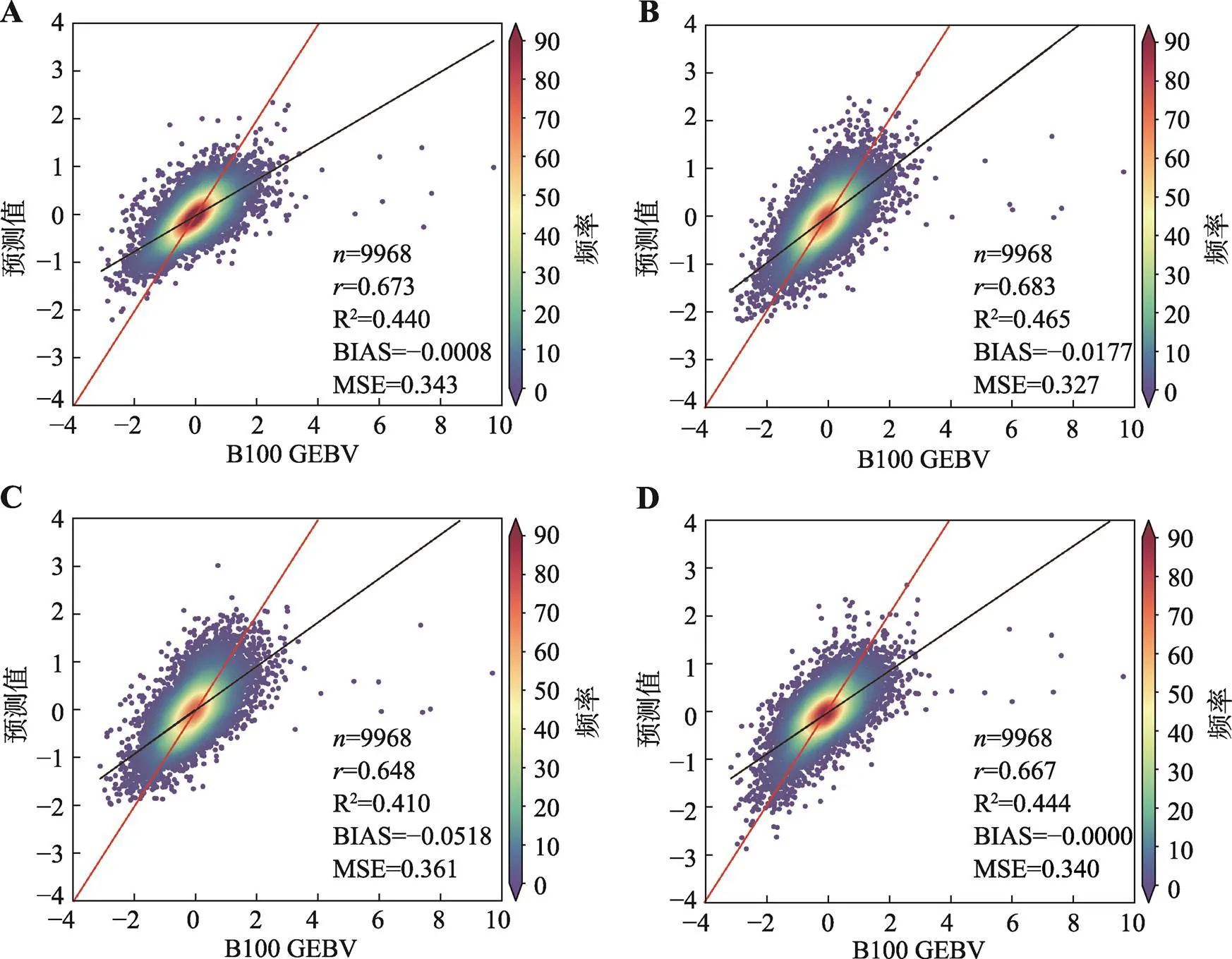
图1 4种机器学习B100 GEBV基因组预测效果
A:DL模型的基因组预测效果;B:GBM模型的基因组预测效果;C:RF模型的基因组预测效果;D:XGB模型的基因组预测效果。黑色直线为预测数据与GEBV的回归线,红色直线为坐标轴的对角线=;R2为拟合系数,BIAS为偏差,MSE为均方误差(下图同)。
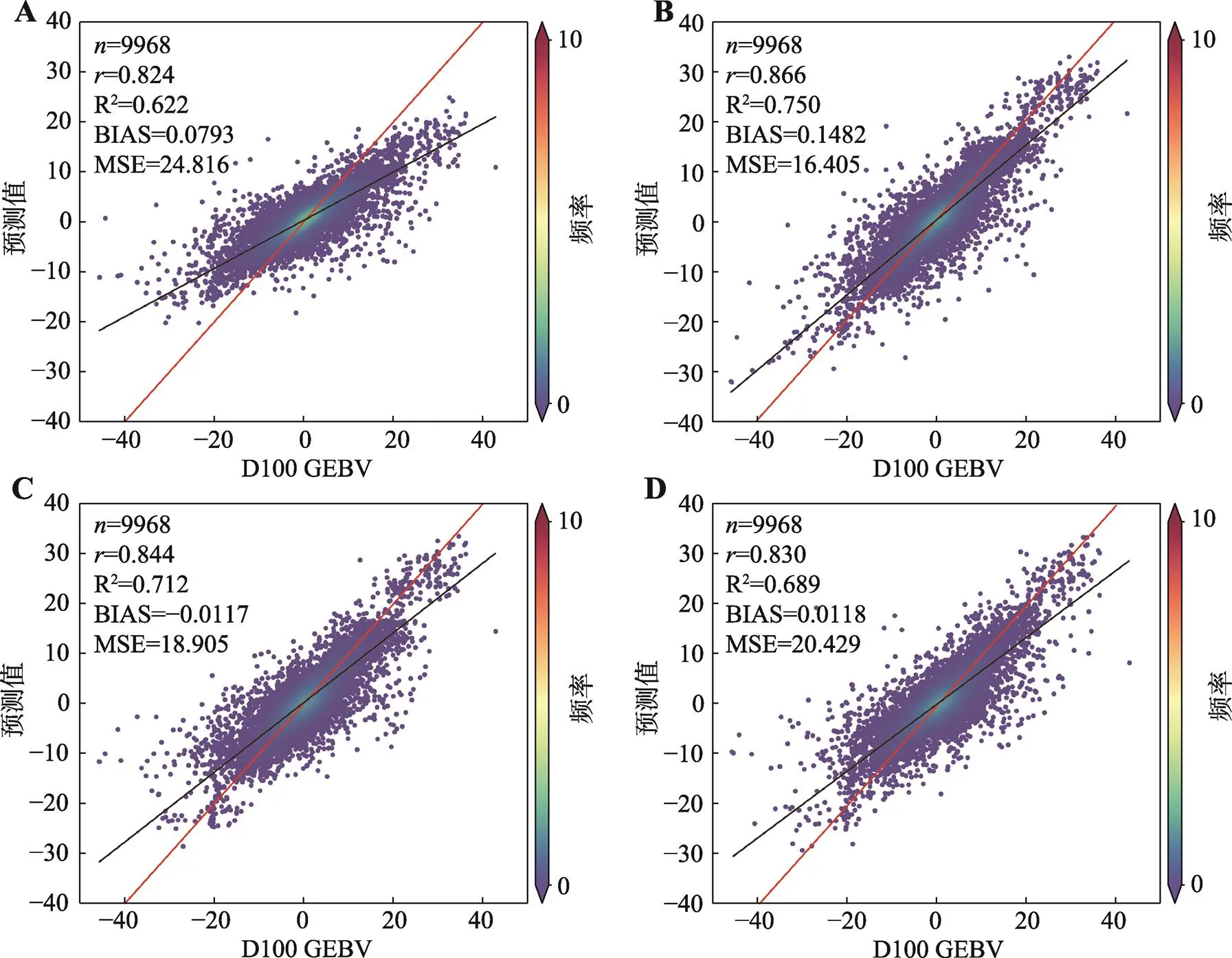
图2 4种机器学习D100 GEBV基因组预测效果
A:DL模型的基因组预测效果;B:GBM模型的基因组预测效果;C:RF模型的基因组预测效果;D:XGB模型的基因组预测效果。
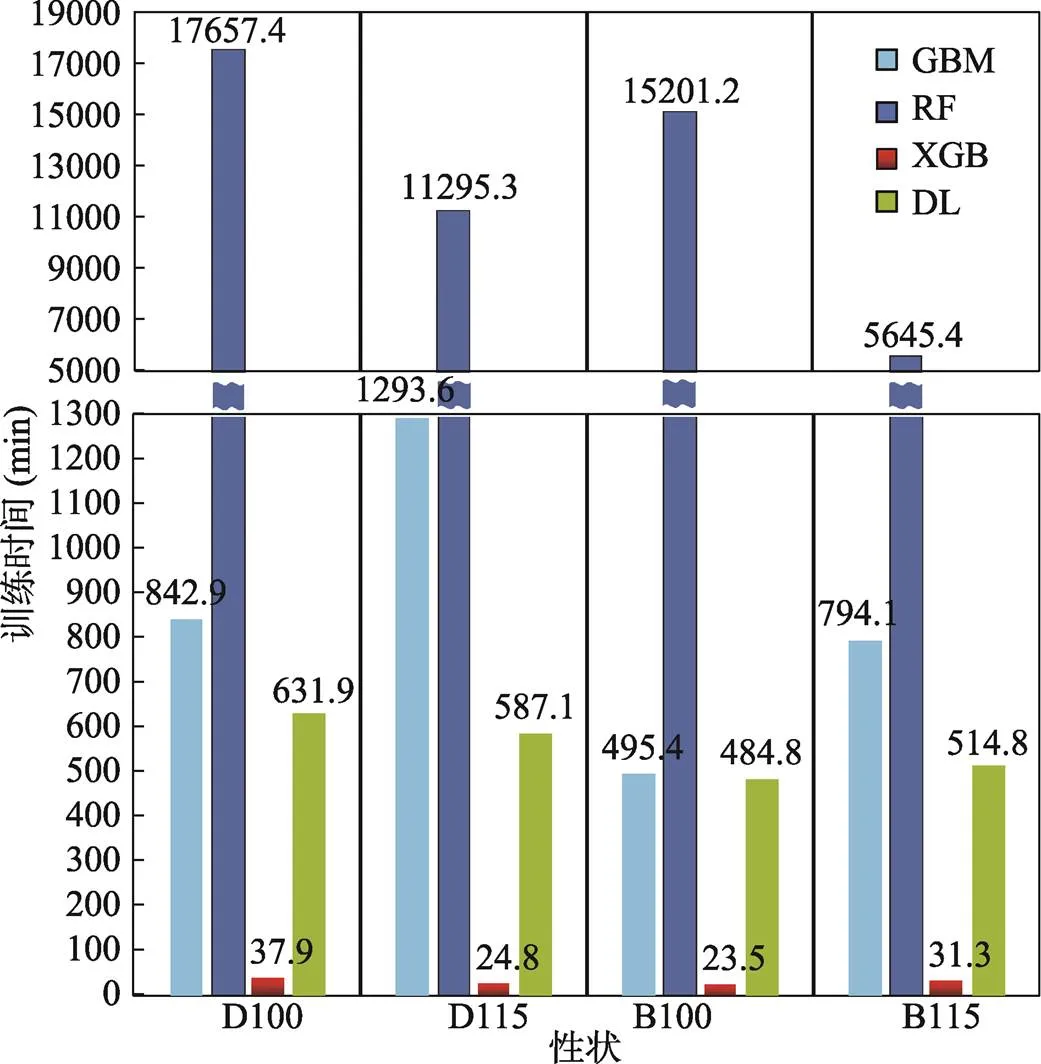
图3 生长性状GEBV估计模型训练时间
2.2 表型预测的机器学习模型整体表现出良好的预测性能
2.2.1 模型预测性能
经过十倍交叉验证得到了4种机器学习模型,利用机器学习模型对猪B100表型预测效果见图4。其准确性依次为0.547 (GBM)、0.539 (DL)、0.509 (XGB)、0.426 (RF)。其中GBM对B100表型预测结果较为准确,RF模型R2为–0.022,预测结果较差。
4种机器学习模型对于B115的预测准确性与B100相似(附图3),依次为0.547(DL)、0.544(GBM)、0.509(XGB)、0.467(RF)。DL表现出了较好的预测性能,其R2为0.296,略高于其他模型。MSE为5.612均低于其他模型。
图5为4种机器学习模型对于D100的预测结果。其准确性依次为0.672 (XGB)、0.599 (GBM)、0.474 (DL)、0.460 (RF)。且XGB模型的R2均远高于其他几种模型,BIAS与MSE也远低于其他模型。
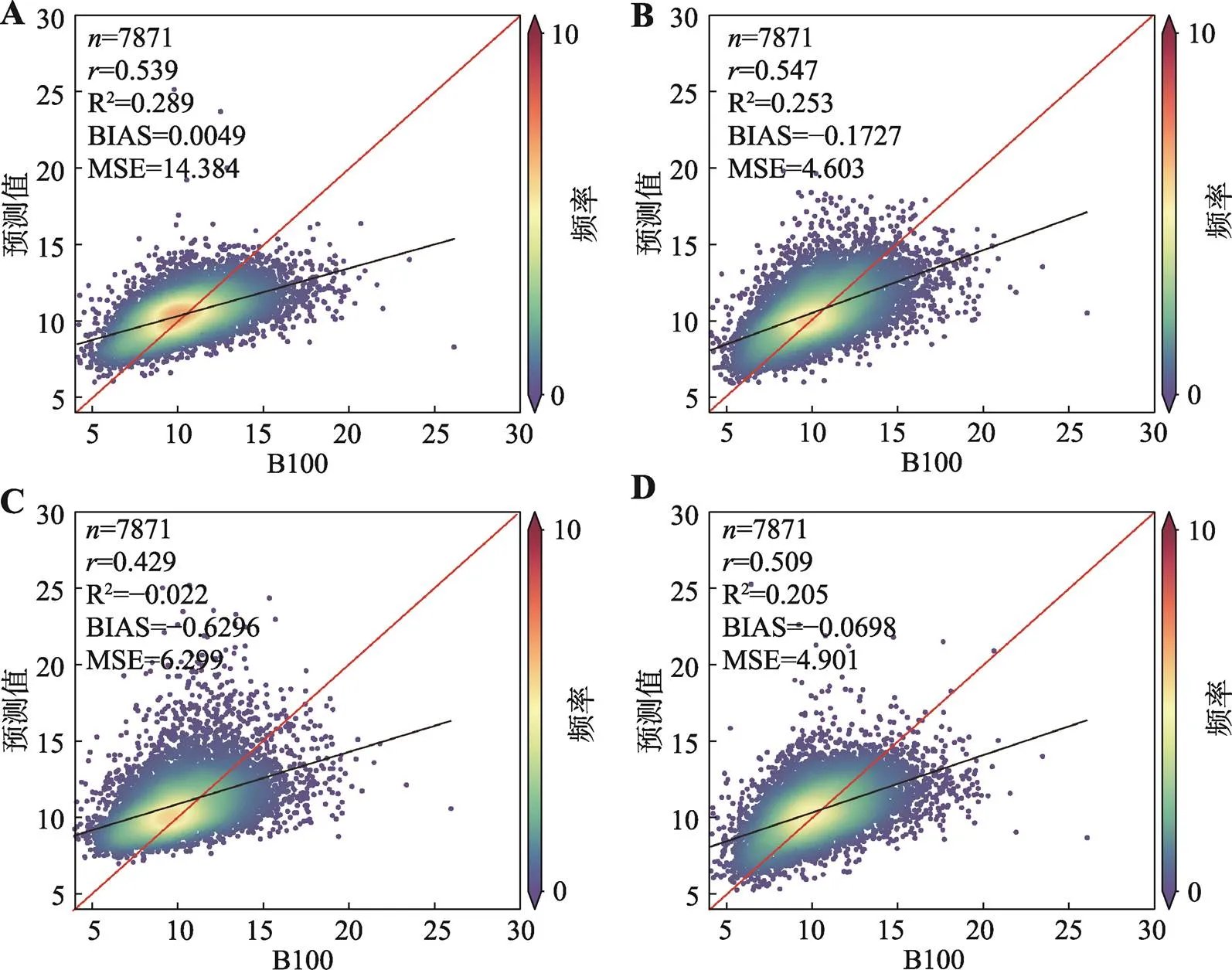
图4 4种机器学习B100表型预测效果
A:DL模型的基因组预测效果;B:GBM模型的基因组预测效果;C:RF模型的基因组预测效果;D:XGB模型的基因组预测效果。
图5 4种机器学习D100表型预测效果
Fig. 5 Prediction effect of D100 phenotype for four types of machine learning
A:DL模型的基因组预测效果;B:GBM模型的基因组预测效果;C:RF模型的基因组预测效果;D:XGB模型的基因组预测效果。
在4种机器学习模型对于D115的预测准确性排名依次为0.670 (XGB)、0.595 (GBM)、0.473 (RF)、0.387 (DL) (附图4)。且XGB模型R2高于其他几种模型,BIAS与MSE也远低于其他模型。表明其对于D100与D115表型信息具有更好的预测性能。
2.2.2 训练时长
生长性状表型预测的4种机器学习模型的训练时长对比如图所示(图6),4种机器学习模型训练时长均小于GEBV的预测。其中,RF模型训练时长远超其他3种模型。DL模型居中。GBM模型与XGB模型训练所需时长最少。
3 讨论
高通量的基因组学的研究为全基因组选择提供了更加丰富的遗传信息,同时也带来了更加冗余的信息影响了传统遗传算法准确性的进一步提升。而机器学习的高速发展为全基因组评估提供了新的计算方法。它让计算机模拟人类的认知过程,从数据中学习并进行求解,而非通过明确的编程方法解决问题[29],能够很好地处理高维数据和非线性关系进而完成动物遗传价值的预测。
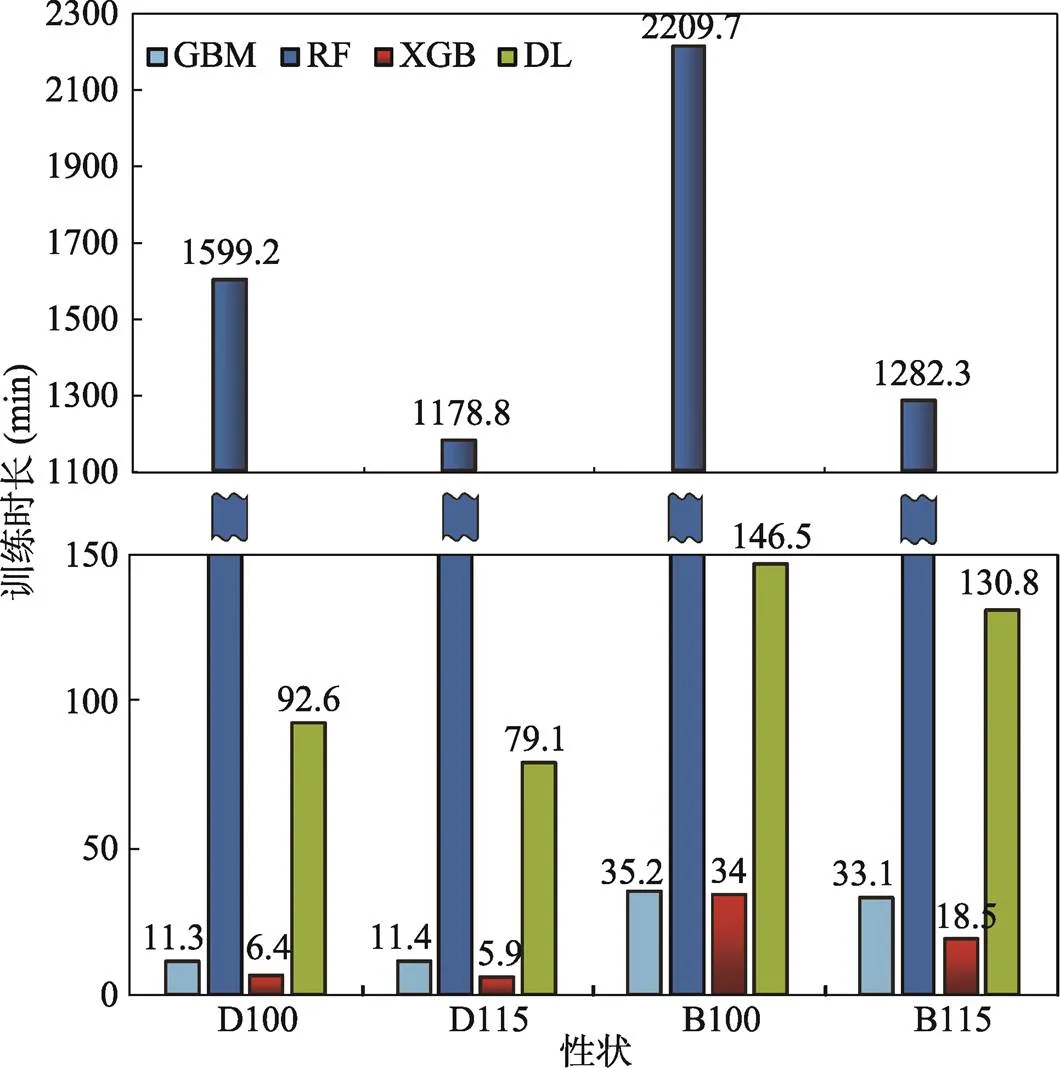
图6 生长性状表型预测模型训练时间
本研究利用自动机器学习方法获得4种猪生长性状与基因组估计育种值的4个机器学习方法的最优模型,比较自动机器学习下不同机器学习模型应用于猪全基因组评估的能力。
在GEBV的预测中,机器学习模型对猪B100、B115、D100和D115的GEBV预测的准确性较高。说明机器学习模型能够根据基因组信息、亲缘系谱信息和固定效应很好的预测猪B100、B115、D100和D115的GEBV。其中,GBM模型在B100、B115、D100和D115的GEBV估计中都表现出了最高的准确性,与Li等[30]对婆罗门牛育种值预测的研究结果表现一致。这表明GBM模型能够较好的捕捉猪D100、D115、B100和B115的GEBV与基因组信息中的特征,并对GEBV做出较好的估计。
相比于GEBV,表型信息与基因组信息之间的关系不够清晰,所以机器学习模型对相同性状GEBV的估计准确性均低于对表型性状的预测。其中机器学习方法对D100和D115的预测准确性较好,对B100和B115的预测准确性一般。虽然GBM模型在B115、D100、D115表型信息的预测中未能表现出最好的预测性能,但其准确性排名始终位于前列。这表明GBM模型能够较好处理猪在生长性状与基因组信息之间的关系,对表型进行较好的预测。而RF模型无论是在GEBV的估计或是在表型信息的预测中,始终表现较差。
此外,在模型训练时长的比较中,RF模型训练时间远超其他3种模型。DL与GBM模型训练时间居中。XGB模型训练时间远远少于其他3种模型。
综上所述,在利用4种机器模型对猪生长性状GEBV与表型的估计中,GEBV的预测准确性均高于表型信息。虽然GBM方法在预测过程中表现出较高的预测准确性与效率,但没有任何一种机器学习模型能在所有性状的GEBV与表型预测中都表现出最好的预测性能。因此,在具体的育种实践中需要育种人员综合考虑准确率与时间因素,选择适当的方法,才能高效的利用机器学习方法优化全基因选择的效率。
附加材料见文章电子版www.chinagene.cn。
[1] Wang KJ, Liu YF, Xu Q, Liu CK, Wang J, Ding C, Fang MY. A post-GWAS confirming GPAT3 gene associated with pig growth and a significant SNP influencing its promoter activity., 2017, 48(4): 478–482.
[2] Guo YM, Huang YX, Hou LJ, Ma JW, Chen CY, Ai HS, Huang LS, Ren J. Genome-wide detection of genetic markers associated with growth and fatness in four pig populations using four approaches., 2017, 49(1): 21.
[3] Ding RR, Yang M, Wang XW, Quan JP, Zhuang ZW, Zhou SP, Li SY, Xu Z, Zheng EQ, Cai GY, Liu DW, Huang W, Yang J, Wu ZF. Genetic architecture of feeding behavior and feed efficiency in a Duroc pig population., 2018, 9: 220.
[4] Vanraden PM. Symposium review: How to implement genomic selection., 2020, 103(6): 5291–5301.
[5] Meuwissen THE, Hayes BJ, Goddard ME. Prediction of total genetic value using genome-wide dense marker maps., 2001, 157(4): 1819–1829.
[6] Guo P, Zhang JB, Cao S. Study on Bayesian method and machine learning genome-wide selection of milk production traits in dairy cows., 2023(5): 56–60+64. 郭鹏, 张建斌, 曹晟. 奶牛产奶性状贝叶斯方法与机器学习全基因组选择研究. 黑龙江畜牧兽医, 2023(5): 56–60+64.
[7] Akanno EC, Schenkel FS, Sargolzaei M, Friendship RM, Robinson JAB. Opportunities for genome-wide selection for pig breeding in developing countries., 2013, 91(10): 4617–4627.
[8] Samorè AB, Fontanesi L. Genomic selection in pigs: State of the art and perspectives., 2016, 15: 211–232.
[9] Marchini J, Howie B. Genotype imputation for genome- wide association studies., 2010, 11(7): 499–511.
[10] Das S, Forer L, Schönherr S, Sidore C, Locke AE, Kwong A, Vrieze SI, Chew EY, Levy S, Mcgue M, Schlessinger D, Stambolian D, Loh PR, Iacono WG, Swaroop A, Scott LJ, Cucca F, Kronenberg F, Boehnke M, Abecasis GR, Fuchsberger C. Next-generation genotype imputation service and methods., 2016, 48(10): 1284– 1287.
[11] Sollero BP, Howard JT, Spangler ML. The impact of reducing the frequency of animals genotyped at higher density on imputation and prediction accuracies using ssGBLUP1., 2019, 97(7): 2780–2792.
[12] Fernandes Júnior GA, Carvalheiro R, de Oliveira HN, Sargolzaei M, Costilla R, Ventura RV, Fonseca LFS, Neves HHR, Hayes BJ, de Albuquerque LG. Imputation accuracy to whole-genome sequence in Nellore cattle., 2021, 53(1): 27.
[13] Alves AAC, Espigolan R, Bresolin T, Costa RM, Fernandes Júnior GA, Ventura RV, Carvalheiro R, Albuquerque LG. Genome-enabled prediction of reproductive traits in Nellore cattle using parametric models and machine learning methods., 2021, 52(1): 32–46.
[14] Zhao W, Lai XS, Liu DY, Zhang ZY, Ma PP, Wang QS, Zhang Z, Pan YC. Applications of support vector machine in genomic prediction in pig and maize populations., 2020, 11: 598318.
[15] Montesinos López OA, Montesinos López A, Crossa J. Random Forest for Genomic Prediction. In: Multivariate Statistical Machine Learning Methods for Genomic Prediction. Cham: Springer International Publishing, 2022, 633–681.
[16] Liang M. Research on genome-wide selection based on machine learning algorithm[Dissertation]. Chinese Academy of Agricultural Sciences, 2021. 梁忙. 基于机器学习算法的全基因组选择研究[学位论文]. 中国农业科学院, 2021.
[17] Barreiro E, Munteanu CR, Cruz-Monteagudo M, Pazos A, González-Díaz H. Net-Net Auto Machine Learning (AutoML) Prediction of Complex Ecosystems. Scientific reports. 2018;8(1): 12340.
[18] Romero RAA, Deypalan MNY, Mehrotra S, Jungao JT, Sheils NE, Manduchi E, Moore JH. Benchmarking AutoML frameworks for disease prediction using medical claims., 2022, 15(1): 15.
[19] Friedman JH. Greedy function approximation: A gradient boosting machine., 2001, 29(5): 1189–1232.
[20] Natekin A, Knoll A. Gradient boosting machines, a tutorial., 2013, 7: 21.
[21] Abdollahi-Arpanahi R, Gianola D, Peñagaricano F. Deep learning versus parametric and ensemble methods for genomic prediction of complex phenotypes., 2020, 52(1): 12.
[22] Breiman L. Random Forests., 2001, 45: 5–32.
[23] González-recio O, Forni S. Genome-wide prediction of discrete traits using Bayesian regressions and machine learning., 2011, 43(1): 7.
[24] Wang FY, Wang YC, Ji XK, Wang ZP. Effective macrosomia prediction using random forest algorithm., 2022, 19(6): 3245.
[25] Chen TQ, Guestrin C. XGBoost: a scalable tree boosting system. In: Proceedings of the 22nd ACM SIGKDD International Conference on Knowledge Discovery and Data Mining, 2016, 785–794.
[26] Hou NZ, Li MZ, He L, Xie B, Wang L, Zhang RM, Yu Y, Sun XD, Pan ZS, Wang K. Predicting 30-days mortality for MIMIC-III patients with sepsis-3: a machine learning approach using XGboost., 2020, 18(1): 462.
[27] Tan XF, Li GS. Overview of deep learning development. In: The 15th National Security Geophysics Symposium. 2019. 谭笑枫, 李广帅. 深度学习发展综述. 见:第十五届国家安全地球物理专题研讨会. 2019.
[28] Nayeri S, Sargolzaei M, Tulpan D. A review of traditional and machine learning methods applied to animal breeding., 2019, 20(1): 31–46.
[29] Zheye Peng, Zijun Tang, Minzhu Xie. Research progress in machine learning methods for gene-gene interaction detection., 2018, 40(3): 218–226. 彭哲也, 唐紫珺, 谢民主. 机器学习方法在基因交互作用探测中的研究进展. 遗传, 2018, 40(3): 218–226.
[30] Li B, Zhang NX, Wang YG, George AW, Reverter A, Li YT. Genomic prediction of breeding values using a subset of snps identified by three machine learning methods., 2018, 9: 237.
Genomic prediction of pig growth traits based on machine learning
Dong Chen1,2,3, Shujie Wang1,2,3, Zhenjian Zhao1,2,3, Xiang Ji1,2,3, Qi Shen1,2,3, Yang Yu1,2,3, Shengdi Cui1,2,3, Junge Wang1,2,3, Ziyang Chen1,2,3, Jinyong Wang4, Zongyi Guo4, Pingxian Wu4, Guoqing Tang1,2,3
This study aimed to assess and compare the performance of different machine learning models in predicting selected pig growth traits and genomic estimated breeding values (GEBV) using automated machine learning, with the goal of optimizing whole-genome evaluation methods in pig breeding. The research employed genomic information, pedigree matrices, fixed effects, and phenotype data from 9968 pigs across multiple companies to derive four optimal machine learning models: deep learning (DL), random forest (RF), gradient boosting machine (GBM), and extreme gradient boosting (XGB). Through 10-fold cross-validation, predictions were made for GEBV and phenotypes of pigs reaching weight milestones (100 kg and 115 kg) with adjustments for backfat and days to weight. The findings indicated that machine learning models exhibited higher accuracy in predicting GEBV compared to phenotypic traits. Notably, GBM demonstrated superior GEBV prediction accuracy, with values of 0.683, 0.710, 0.866, and 0.871 for B100, B115, D100, and D115, respectively, slightly outperforming other methods. In phenotype prediction, GBM emerged as the best-performing model for pigs with B100, B115, D100, and D115 traits, achieving prediction accuracies of 0.547, followed by DL at 0.547, and then XGB with accuracies of 0.672 and 0.670. In terms of model training time, RF required the most time, while GBM and DL fell in between, and XGB demonstrated the shortest training time. In summary, machine learning models obtained through automated techniques exhibited higher GEBV prediction accuracy compared to phenotypic traits. GBM emerged as the overall top performer in terms of prediction accuracy and training time efficiency, while XGB demonstrated the ability to train accurate prediction models within a short timeframe. RF, on the other hand, had longer training times and insufficient accuracy, rendering it unsuitable for predicting pig growth traits and GEBV.
genomic estimated breeding values; growth traits;automated machine learning; performance comparison
2023-04-26;
2023-08-14;
2023-08-16
国家生猪技术创新中心先导科技项目(编号:NCTIP-XD/B01),四川省科技厅项目(编号:2020YFN0024, 2021ZDZX0008, 2021YFYZ0030)和四川省猪创新团队项目(编号:sccxtd-2022-08)资助[Supported by the Strategic Priority Research Program of the National Center of Technology Innovation for Pigs (No. NCTIP-XD/B01), Sichuan Science and Technology Program(Nos.2020YFN0024, 2021ZDZX0008, 2021YFYZ0030), and the Sichuan Innovation Team of Pig(No. sccxtd-2022-08)]
陈栋,硕士,专业方向:畜牧学。E-mail: 1123278154@qq.com
唐国庆,博士,教授,博士生导师,研究方向:猪遗传育种。E-mail: tyq003@163.com
10.16288/j.yczz.23-120
(责任编委: 赵要风)