利用高密度SNP芯片评估中国地方肉牛品种基因组亲缘关系
2023-10-29马浩然张路培金生云宝金山李红艳高会江徐凌洋王泽昭李俊雅
马浩然,张路培,金生云,宝金山,李红艳,高会江,徐凌洋,王泽昭*,李俊雅*
(1.中国农业科学院北京畜牧兽医研究所 牛遗传育种创新团队,北京 100193;2.乌拉盖管理区农牧技术推广中心,乌拉盖 026321;3.乌拉盖管理区供销合作社,乌拉盖 026321;4.内蒙古通辽市畜牧业发展中心,通辽 028000)
基因组选择(genomic selection, GS)技术由Meuwissen等[1]于2001年提出 ,由于该技术能够实现后备牛早期选择,因此可以大幅缩短牛育种世代间隔,提升肉牛育种遗传进展。目前该技术已经成为世界肉牛主要育种技术手段之一[2]。世界范围内牛品种资源十分丰富,2009年,Harris等[3]尝试选择部分代表性牛品种组建参考群进行品种间基因组联合评估。Hayes等[4]使用荷斯坦牛、娟姗牛及其他奶牛品种进行多群体基因组选择研究,其研究证明使用奶牛混合参考群体进行基因组选择会提升目标性状的育种值估计准确性。
上述研究共同发现,多品种GS育种值估计准确性主要受到参考群体规模和验证群体间亲缘关系的影响[5]。全基因组选择准确性依赖于单核苷酸多态性(single nucleotide polymorphism, SNP)位点和数量性状基因座(quantitative trait locus, QTL)之间的连锁不平衡(linkage disequilibrium, LD)程度[6]。而亲缘关系较远的品种间由于受到等位基因频率分布以及基因间或基因与环境间的互作等影响导致QTL及标记LD一致性差异从而使多品种基因组选择的育种值估计准确性降低[7-8]。Wientjes等[9]研究发现,当品种间的遗传关系接近时,种间预测准确性较高。而遗传关系远时利用多品种的混合参考群体进行预测的准确性并不比单一品种准确性高。还有部分研究表明,不仅仅参考群体与候选个体之间的亲缘关系能够影响基因组选择的预测准确性,位于参考群中个体间的亲缘关系同样会影响选择准确性[10-11]。Lund等[12]的研究也同样指出,当远亲品种组合作为基因组选择参考群并使用基因组最佳线性无偏预测(genomic best linear unbiased prediction, GBLUP)模型评估时,育种值估计准确性并未提升,甚至有所降低。但通过使用更复杂的贝叶斯变量选择模型并结合更密集的标记集或标记的功能子集,同时对与QTL强连锁不平衡的基因组标记进行加权,则可能利用远缘品种的遗传信息来提高基因组预测准确性。因此,准确评估不同肉牛品种间的亲缘关系是实现肉牛多品种和跨品种基因组选择的第一步。
相较于系谱数据,高密度SNP芯片分型数据可以更加准确的评估品种间的亲缘关系[13]。目前估计群体间遗传关系的方法主要有以下4种:一是利用预测误差方差差异(prediction error variance of differences, PEVD)评估种间亲缘系数法[14],该方法通过计算品种之间育种值(estimated breeding value, EBV)差异的预测误差方差,来衡量品种间的遗传关联。二是基于广义决定系数 (generalized coefficient of determination, CD)[15]评估品种间亲缘关系。CD 定义为估计育种值比较的可靠性,即预测值差异与真实值差异间相关系数的平方。三是利用预测误差相关系数(prediction error correlation, r) 开展种间亲缘关系评估。该方法是由Lewis等[16-17]提出的基于预测误差方差(prediction error variance, PEV)的简化评估方法,即使用两品种之间的PEV相关系数来衡量亲缘关系。四是基于不同品种间SNP位点与QTL的连锁一致性程度评估品种间亲缘关系。即基因组关系的品种间LD一致性分析[18]。上述4种方法在我国肉牛种间亲缘关系评估工作中的评估性能尚无系统评价。
中国幅员辽阔,是拥有世界上牛品种最多的国家之一,共有130多个牛品种。目前支撑肉牛产业的品种主要有70多种[19]。近年来,随着种业振兴行动相关工作的持续推进,对地方品种优秀种质资源的深度挖掘和新品种培育工作均在持续进行,地方品种对基因选择的需求在不断提升。但大多数品种育种工作仍处于初期阶段,生产性能测定等常规基础育种体系建设仍不健全,育种群规模小、系谱记录不健全等客观因素导致了传统育种无法有效支撑肉牛高效育种工作。对全部品种逐一建设基因组选择参考群体既不经济且实现难度较大,因此探索肉牛多品种基因组选择方法势在必行。
目前,我国尚未开展肉牛多品种基因组选择相关研究工作。本研究基于模拟数据和高密度SNP芯片分型数据,通过使用5种品种间亲缘关系分析方法,对比分析了我国10个地方肉牛品种间的亲缘关系。旨在探索适合我国肉牛多品种间亲缘关系评估的最优策略,为肉牛多品种基因组选择技术研发奠定基础。
1 材料与方法
1.1 实际数据
本研究分析了我国10个地方牛品种基因型数据,分别是:柴达木牛(CDM, n=25)、雷琼牛(LQ, n=26)、凉山牛(LS, n=22)、蒙古牛(MG, n=21)、南丹牛(ND, n=25)、平武牛(PW, n=24)、文山牛(WS, n=24)、西藏牛(XZ, n=26)、延黄牛(YH, n=24)和昭通牛(ZT, n=23)。试验个体均静脉采血20 mL冻存,并用2 mL血液提取DNA,使用Illumina Bovine HD Bead Chip(770K,Illumina, Inc., San Diego, CA)对样本进行基因分型,该芯片由777 962个SNPs位点组成。基因分型和质量控制(quality control, QC)使用 Genome Studio软件进行。除去性染色体及质粒DNA上的42 669个 SNPs位点,常染色体上分布的SNPs位点共计735 293个。
得到基因分型数据后使用PLINK(V1.9)软件对每个品种常染色体位点单独进行质控,其SNPs位点保留标准为:位点检出率(call rates, CR)大于等于90%,个体检出率大于90%,最小等位基因频率(minor allele frequencies, MAF)大于0.01,哈迪-温伯格平衡(Hardy-Weinberg equilibrium, HWE)P值大于1.0×10-6。10个肉牛地方品种基因型质控样本的描述性统计结果见表1。将通过质控后的数据按品种使用BEAGLE(V5.0)对缺失的位点进行填充,填充参数为软件默认参数。
1.2 模拟数据
本研究使用了重抽样模拟方法,在10个地方品种的真实基因型数据基础上,将每个品种基因组数据模拟至1 500头。具体模拟方法及参数设置见Xu等[20]的研究。


表2 表型模拟的遗传参数设置
其中,xij是个体j的第i个QTL的基因型,编码为0、1、2;aj第i个QTL的加性效应;n是QTL的数量。
表型值为:
Pi=TBVi+σei
其中,Pi为模拟表型值,TBVi为第i个个体育种值,σei为第i个个体残差。
1.3 育种值估计模型
在本研究中,使用GBLUP模型进行相应的加性遗传方差以及残差方差估计,其模型如下:
y=Xb+Zg+e
其中,y是个体的表型值;g和b分别是随机加性遗传效应和固定效应,Z和X分别为随机加性遗传效应和固定效应对应的关联矩阵;e是残差向量。
1.4 不同亲缘关系评估方法
本研究首先对模拟数据和实际数据进行主成分分析(principal component analysis,PCA),分别获得模拟数据和真实数据的品种间聚类结果,并将其作为后续分析结果的基准参考。
1.4.1 品种间LD一致性评估法 使用PopLDdecay软件分别计算了模拟数据和实际数据的LD衰减距离,并绘制LD衰减图。然后利用LD的r2值计算品种间的皮尔逊相关系数,并将该值作为不同品种基因组LD一致性衡量指标,评价品种间亲缘关系。r2计算公式如下:
公式中D=f(AB)-f(A)f(B), 其中f(AB)、f(A)、f(a)、f(B)和f(b)分别为单倍型AB的基因型频率,A、a、B和b以及等位基因的频率。
皮尔逊相关的计算公式如下:
公式中rij为计算所得皮尔逊相关系数,COV(i,j)为品种i与品种j的协方差,σi、σj分别为两个品种的标准差。
1.4.2 预测误差方差 本研究中,育种值估计模型的混合模型方程组为:
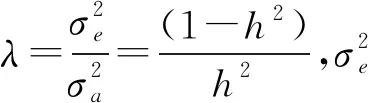
PEVD计算公式如下[14]:
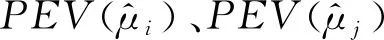
1.4.3 广义决定系数 广义决定系数计算公式如下[17]:
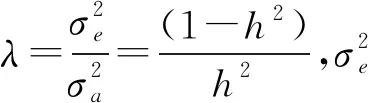
1.4.4 预测误差相关系数 预测误差相关系数计算公式如下:
本研究中群体间PEVD、CD、r、PEV以及PEC的计算使用了不同群体的所有个体两两配对的均值。其中CD值和r值与品种间亲缘关系成正比(数值越大关系越近),而PEVD值则相反(数值越小关系越近)[13]。
2 结 果
2.1 不同地方牛品种PCA分析结果
图1展示的是10个不同地方品种模拟数据和实际数据PCA聚类结果。图1a中,PC1及PC2为第一主成分和第二主成分,分别解释了48.76%以及16.98%的变异。由图可见,10个地方品种模拟数据明显聚集为3大类,其中延黄牛、蒙古牛、西藏牛以及柴达木牛聚为一类,可初步判断上述4个品种间亲缘关系较近。平武牛、昭通牛和凉山牛聚为一类,文山牛、雷琼牛以及南丹牛之间虽然聚类显示一定距离,但相较于其他品种仍然可以被归为一类,品种间亲缘关系较远。图1b展示的是真实基因型数据聚类结果。如图所示,PC1及PC2两个主成分分别解释了56.71%以及4.92%的变异,且在真实群体中10个地方品种整体上也聚类为3大类,每大类包含的品种聚类结果与模拟数据一致。
2.2 不同地方牛品种K-means聚类结果
图2展示的是10个不同地方品种模拟数据的K-Means聚类结果,设定10个初始化聚类中心进行聚类,取最终的聚类中心进行绘图。由图可见,10个地方品种模拟数据明显聚集为3大类,首先,延黄牛、蒙古牛、西藏牛以及柴达木牛聚为一类,其中该类中蒙古牛与延黄牛聚为一类,柴达木牛与西藏牛聚为一类,其次,平武牛、昭通牛和凉山牛聚为一类,其中平武牛与另外两个品种亲缘关系较远,最后,文山牛、雷琼牛以及南丹牛分为一类,但同样,其中文山牛与另外两个品种亲缘关系较远,该结果得到的亲缘关系与PCA聚类分析所展示的结果图一致,在一定程度上证实了PCA分析的可靠性。
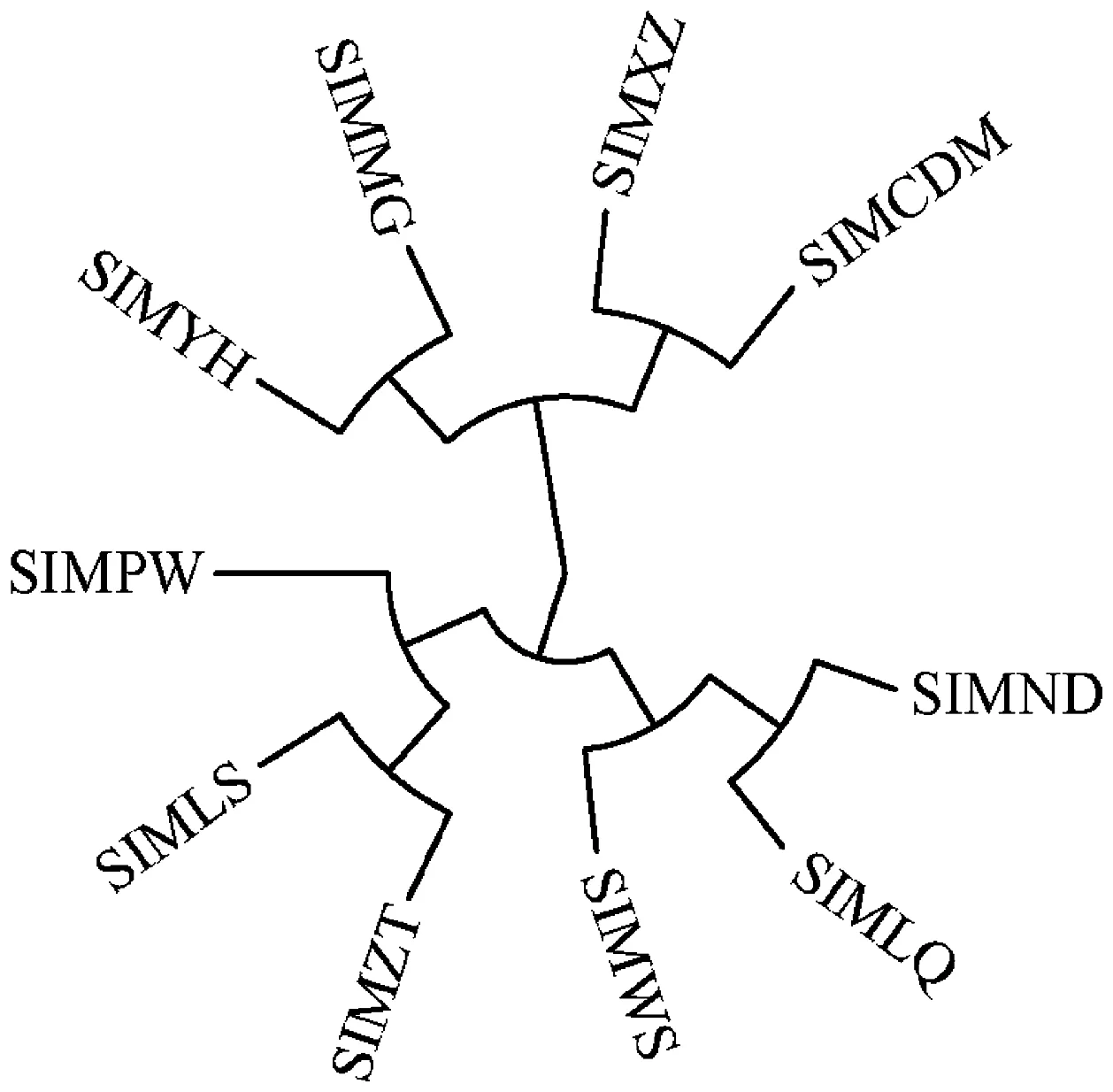
图2 10个不同地方牛品种聚类结果图Fig.2 Clustering results of 10 different indigenous cattle breeds
2.3 种间LD一致性评估法
2.3.1 不同地方牛品种LD衰减结果 图3分别展示了模拟数据和实际数据基因组r2的衰减趋势。模拟数据结果显示,延黄牛、蒙古牛、柴达木牛与西藏牛LD衰减趋势一致,其LD衰减距离分别为82.29、85.41、89.04和87.13 kb,其品种间衰减距离较为相似。南丹牛及雷琼牛2个品种LD衰减距离分别为120.12和123.79 kb。剩余4个品种LD衰减距离分别为105.78、101.26、109.47和102.58 kb,其衰减距离值较为相似。
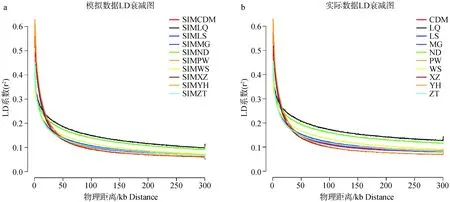
图3 10个不同地方牛品种LD衰减结果图Fig.3 LD decay of 10 different indigenous cattle breeds
2.3.2 LD一致性评估亲缘关系 根据品种间r2计算得到的10个肉牛地方品种间亲缘关系结果如图4所示。如图4a所示,在模拟数据中,蒙古牛与延黄牛亲缘关系最高,品种间r2相关系数为0.64。其次,南丹牛与雷琼牛、延黄牛与西藏牛以及昭通牛与凉山牛3组品种间r2相关系数均为0.63。雷琼牛与延黄牛品种间r2相关系数为0.22,表明两个品种间亲缘关系较远。上述结果与PCA分析展示的聚类结果一致。但其中昭通牛与文山牛的品种间r2相关系数为0.59,且LD衰减趋势较为一致,LD一致性评价结果显示两品种间亲缘关系较为密切,但该发现与PCA结果存在差异。在真实群体的分析结果中(图4b),延黄牛与蒙古牛、昭通牛与凉山牛以及南丹牛与雷琼牛品种间r2相关系数最高(r2=0.74),LD一致性评价结果显示上述品种组合间亲缘关系较为密切。其次,凉山牛与平武牛品种间r2相关系数为0.72,昭通牛与文山牛品种间r2相关系数为0.71,表明上述品种对间存在较高遗传联系。但昭通牛与文山牛LD一致性评价结果与PCA结果存在差异。第三是延黄牛与雷琼牛品种间r2相关系数仅为0.25,表明品种间亲缘关系较远。

图4 10个不同地方牛品种亲缘关系Fig.4 Genomic relationship of 10 different indigenous cattle breeds
2.4 预测误差方差法
表3展示的是基于预测误差方差法计算的群体间亲缘关系结果,表中数字表示预测误差方差,数值越低代表亲缘关系越高。由于实际数据部分表型值缺失,因此本分析方法仅对模拟数据使用。由表可知,10个肉牛地方品种PEVD值范围在0.80~0.87之间。延黄牛与蒙古牛、西藏牛以及柴达木牛PEVD值范围在0.80~0.81之间,初步表明上述4个品种间亲缘关系较近,与PCA聚类分析结果一致。平武牛与昭通牛和凉山牛间聚为一类,其PEVD值范围在0.80~0.82之间。文山牛与雷琼牛以及南丹牛之间PEVD数值为0.83~0.84,说明上述3个品种可聚为一类。但根据表3 展示结果发现,品种间PEVD值较为集中,与上述方法相比种间亲缘关系分层情况不明显。

表3 预测误差方差法计算亲缘关系
2.5 广义决定系数法
表4展示的是基于广义决定系数法计算的群体间亲缘关系结果,数值越高代表亲缘关系越高。同样由于实际数据部分表型值缺失,本分析方法仅对模拟数据使用。由表可知,10个肉牛地方品种CD值范围在0.72~0.79之间。其中,延黄牛与蒙古牛、西藏牛以及柴达木牛间CD值范围在0.78~0.79之间,表明上述4个品种间亲缘关系较近,与PCA聚类分析结果一致。平武牛与昭通牛和凉山牛聚为一类,其CD值范围在0.77~0.78之间。文山牛与雷琼牛以及南丹牛之间CD数值为0.76,说明上述3个品种可聚为一类。但根据表4展示结果发现,品种间CD值相差不大,与上述方法相比种间亲缘关系分层情况不明显。

表4 广义决定系数法计算亲缘关系
2.6 预测误差相关系数法
表5展示的是基于预测误差相关系数法计算的群体间亲缘关系结果,数值越高代表亲缘关系越近。同样由于实际数据部分表型值缺失,本方法仅对模拟数据使用。由表可知,10个地方品种r值范围在0.000 7~0.001 3之间。延黄牛与蒙古牛、西藏牛以及柴达木牛间r值范围为0.001 2~0.001 3,初步表明上述4个品种间亲缘关系较近,与PCA聚类分析结果一致。平武牛与昭通牛和凉山牛可聚为一类,其r值均为0.001 2。文山牛与雷琼牛以及南丹牛之间r数值为0.000 9~0.001 0,说明上述3个品种可聚为一类。

表5 预测误差相关系数法计算亲缘关系
2.7 遗传力大小对亲缘关系评估的影响


表6 不同遗传力对亲缘关系评估的影响
3 讨 论
3.1 种间LD一致性评估法评估结果比较
Ma等[18]的研究指出,种间LD一致性可以反映品种间亲缘关系 。杨祎挺等[29]认为,不同地方猪品种的LD衰减差异大,代表了其种间遗传结构差异性大。因此,若两个品种的LD衰减速度较为一致,一定程度上代表了品种的亲缘关系。本研究中无论在分析模拟数据还是实际数据,种间LD一致性评估法评估结果均与PCA结果保持了高度一致。但该方法的优势在于可以量化品种间亲缘关系,为肉牛多品种基因组选择提供更为准确的参考。因此,基于本研究分析结果,种间LD一致性评估法是一种较为适合评估肉牛地方品种种间亲缘关系的方法。
3.2 品种间遗传关联度量方法比较
Foulley等[30]与Lalo⊇等[31]认为遗传关联性是一个可预测的衡量标准,基于此,Kennedy和Trus[14]、Lalo⊇[15]以及Lewis等[16]分别提出了使用PEVD、CD以及r值衡量群体间的遗传联系,但这3种方法均受到性状的遗传结构、QTL数目、群体大小和结构等因素的影响从而使预测得到的品种间遗传联系产生偏差[32]。在本研究中,由于地方品种的真实表型数据缺失,无法使用实际数据进行评估,但周子文等[13]的研究表明模拟数据与实际数据的结果存在差异,其原因可能是模拟数据中仅考虑了加性效应,并不能很好的反映真实群体性状的遗传结构等,从而使预测产生了偏差。此外,由于3种方法依赖于误差方差的估计,不同的评估模型、性状遗传力也会对结果产生影响[33],同时,评估的准确性也会影响评估种间亲缘关系,研究中使用了多品种基因选择预测模型进行计算,根据Xu等[34]的研究表明,多品种基因组选择的准确性低于传统的基因组选择,进而使误差方差估计出现了偏差,这可能是导致3种方法估计出现偏差的主要原因。同时,Kuehn等[35]研究表明,引入不具有血缘关系的个体会导致预测误差方差水平的降低,从而降低群体的遗传联系。周子文等[13]及Zhang等[36]的研究指出,基于CD值评估品种间亲缘关系时,即使系谱中不存在亲缘关系的个体,也会估计得到较高的遗传关联,从而会过高估计品种间的亲缘程度;基于G矩阵计算的r值较低,无法区分群体间的遗传差异,不能准确反映群体间的实际群体关联。在对品种间的亲缘关系进行评估时,其得到的结果不应与个体的表型产生关联,但本研究中的3种方法均需依赖表型进行相应的计算,因此,如何减少表型对亲缘关系预测的影响仍需进一步探究。
3.2.1 预测误差方差法评估果 PEVD法在评估10个肉牛地方品种亲缘关系时,评估结果与PCA结果较为一致。与周子文等[13]基于高密度SNP芯片估计猪群体间遗传关系的研究结果相似,本研究中10个地方品种间PEVD值范围在0.80~0.87之间,PEVD值较为集中,表明群体间的遗传关联没有显著差异,与LD法相比种间亲缘关系分层情况不明显,很难直观判断品种间的亲缘关系。基于相关结果,本研究同样认为PEVD方法不是理想的度量群体间遗传关系的方法。
3.2.2 广义决定系数法结果 本研究发现,10个地方品种CD值范围在0.72~0.79之间,表明上述品种间均存在较高的群体遗传联系,这与PCA分析结果存在较大差异。因此,该方法同样不是理想的度量不同品种间肉牛遗传关系的方法。
3.2.3 预测误差相关系数法结果 与CD值类似,r值取值范围在0~1之间。本研究发现,10个地方品种r值范围在0.000 7~0.001 3之间。不同品种间,r值聚集程度较为紧密,且均接近于0,表明依据r值结果,10个肉牛地方品种间不存在遗传关联,这与PCA结果和真实情况差距较大,因此,不能真实有效反映品种间的亲缘关系。
3.3 不同遗传力对计算方法的影响
在本研究中模拟了3种遗传力的性状(对应实际应用中的低、中、高遗传力性状),来评估遗传力对不同计算亲缘关系方法的影响。基于LD一致性的亲缘关系计算方法仅依赖于基因组数据,因此,性状的遗传力高低对其没有任何影响。而在PEVD、CD以及r的计算中,需要依赖预测所得的估计育种值计算相关参数,其育种值估计准确性会受到遗传力的显著影响。如使用高遗传力性状评估品种间亲缘关系时,会提升育种值估计的准确性,降低了预测中的误差,进一步提升了预测得到的品种间亲缘关系(表6)。但在选取较低遗传力性状时,育种值估计中的误差较大,导致了种间亲缘关系评估结果与实际不符。所以,使用低、中遗传力的性状计算得到的PEVD、CD以及r值并不能够准确描述品种间的亲缘关系。
4 结 论
本研究对比了5种不同计算品种间亲缘关系的方法,其中以PCA聚类结果为参照,基于LD一致性的亲缘关系评估方法的评估结果与PCA聚类结果一致,且该方法能够使用皮尔逊相关系数量化品种间亲缘关系,具有较好的准确性。PEVD法、CD法与r法3种方法与上述方法相比评估群体间亲缘关系时容易受到性状估计育种值的误差方差影响,从而造成种间亲缘关系评估结果出现误差。因此,基于LD一致性的亲缘关系评估方法是一种较为适合评估肉牛地方品种种间亲缘关系的方法。
