小鼠Usp2基因序列分析、真核表达载体构建和启动子功能鉴定
2023-10-26马天天马白荣靳亚平陈华涛
张 宇,李 超,马天天,马白荣,李 丹,周 栋,靳亚平,陈华涛
(西北农林科技大学动物医学院农业农村部动物生物技术重点实验室,陕西 杨凌 712100)
泛素化作为蛋白质翻译后修饰的一种重要调控机制,是指在泛素激活酶E1、泛素结合酶E2和泛素连接酶E3的共同作用下,蛋白质与泛素分子结合形成底物复合物的过程[1,2]。泛素化在调节蛋白质的稳定性、亚细胞定位和蛋白酶体介导的蛋白质降解过程中发挥重要作用[3,4]。作为一种动态的、可逆的蛋白翻译后修饰过程,泛素化参与调控机体的大量生理功能,如细胞信号转导、DNA损伤修复、细胞分裂分化、炎症和免疫反应等[5-7]。去泛素化是泛素化的可逆过程,通过去泛素化酶(Deubiquitinating enzymes,DUBs)将泛素分子与底物蛋白分离,使底物蛋白免于被泛素化途径降解,对蛋白的稳定和细胞内环境的稳态维持至关重要[8,9]。目前,已知的成员数量最多、结构最具多样性的DUBs家族是泛素特异性蛋白酶(Ubiquitin-specific proteases,USPs)家族[10]。其中,USP2是USPs家族的重要成员,该基因主要编码USP2-45和USP2-69两种蛋白亚型[11,12],参与调控细胞的增殖凋亡和能量代谢等,同时在机体炎症反应和肿瘤发生发展过程中发挥重要作用[13-18]。前期研究发现,Usp2基因丰富表达于哺乳动物的睾丸组织,参与调控精子发生[19]。随着研究的深入,Usp2基因被发现广泛表达于脑、心脏、肝脏、肾脏、骨骼肌和巨噬细胞等器官、组织和细胞中[12,20,21]。
昼夜节律生物钟是一种以近似24 h为周期的内源性自主节律性振荡系统,控制着机体的睡眠-觉醒和进食等生理功能和行为活动,在哺乳动物体温变化、激素分泌和能量代谢过程中发挥重要作用[22]。在细胞层面,哺乳动物生物钟是由生物钟基因(Bmal1、Clock、Pers、Crys和Rev-erbs等)及其编码蛋白(BMAL1、CLOCK、PERs、CRYs和REV-ERBs等)组成的正负反馈转录-翻译环路相互作用而形成的[23]。正调控因子(脑肌类芳香烃受体核转位蛋白1(Brain and muscle Arnt-like protein-1,BMAL1)与昼夜运动输出周期蛋白(Circadian locomotor output cycles kaput, CLOCK)结合形成BMAL1-CLOCK异源二聚体激活负调控因子Pers、Crys以及其他钟控基因的转录,当周期蛋白(Period,PERs)和隐花色素蛋白(Cryptochromes,CRYs)在细胞质中积累到一定数量后转移到细胞核中,抑制BMAL1-CLOCK的转录活性;而细胞质中的酪蛋白激酶CKIδ和CKIε能够引起PERs和CRYs复合物的降解,细胞质中的PERs和CRYs含量下降会再次促发周期起始,如此循环往复[24,25]。已有证据表明,BMAL1-CLOCK异源二聚体可与其下游靶基因启动子特异序列E-box元件结合,激活下游靶基因的转录[26,27]。前期研究发现,小鼠12个组织器官中仅有10个基因共同存在节律性表达,而Usp2基因便是其中之一,节律性表达于哺乳动物绝大多数组织和器官中[13]。进一步研究发现,Usp2基因的主要转录本Usp2-45 mRNA节律性表达于小鼠肝脏组织,而Usp2-69 mRNA的表达却无明显的节律性变化;Usp2-45 mRNA在Bmal1肝脏特异性敲除小鼠的肝脏中几乎完全丧失节律性变化,提示生物钟系统可能选择性调控Usp2-45的节律性表达[13]。然而,目前关于生物钟系统选择性调控Usp2-45节律性表达的分子机制尚不清楚,亟需进一步的深入研究。
本试验对小鼠Usp2基因序列及其编码蛋白的基本理化特性和结构功能进行了预测分析,成功克隆了Usp2-45的编码序列(Coding sequence,CDS),构建了其真核表达载体。同时,本试验成功克隆了小鼠Usp2-45和Usp2-69的启动子区域,构建了不同长度Usp2启动子的荧光素酶报告载体,发现了生物钟系统对Usp2-45和Usp2-69启动子的选择性调控作用,并初步确定了生物钟系统调控Usp2-45转录的重要功能区,为后续深入解析生物钟系统与USP2互作的分子机制提供了前期基础和关键材料。
1 材料与方法
1.1 主要试剂 TRIzol、Quick CutTMXhoⅠ、Quick CutTMKpnI、Quick CutTMBglⅡ、PrimeSTAR®GXL DNA Polymerase和PrimeSTAR®HS DNA Polymerase,均购自日本TaKaRa公司;SYBR®qPCR Mix,购自日本TOYOBO公司;基因组提取试剂盒和无内毒素小提中量试剂盒,均购自北京天根生化科技有限公司;ClonExpress®Ⅱ One Step Cloning Kit,购自南京诺唯赞生物科技股份有限公司;胎牛血清,购自美国HyClone公司;胰蛋白酶、抗生素、DMEM高糖培养基和Opti-MEM培养基,均购自美国Gibco公司;TurboFect Transfection Reagent,购自美国Thermo Fisher科技有限公司;双荧光素酶报告实验试剂盒,购自美国Promega公司;BCA蛋白浓度测定试剂盒,购自江苏凯基生物技术股份有限公司;HRP共轭羊抗兔抗体,购自中国中杉金桥公司;HRP共轭鼠抗β-actin抗体,购自中国三箭公司;兔抗FLAG抗体,购自美国Abcam公司;ECL HRP底物试剂盒,购自北京迪宁生物科技有限公司。
1.2 主要仪器 蛋白电泳仪、核酸电泳仪、PCR扩增仪和荧光定量系统,均购自美国Bio-Rad公司;核酸蛋白测定仪、普通离心机和CO2恒温培养箱,均购自美国Thermo Fisher科技有限公司;低温离心机和手持移液器,均购自德国Eppendorf公司;凝胶成像系统,购自瑞士Roche公司;多功能微孔板检测仪,购自瑞士Tecan公司。
1.3 实验动物、质粒、菌种和细胞 8周龄C57BL/6J小鼠,购自北京维通利华实验动物技术有限公司[生产许可证号:SCXK(京)2016—0011];pcDNA3.1-Flag和pGL4.10[luc2] 载体,均购自美国Promega公司;hBMAL1、mCLOCK、mPER2过表达质粒,由安徽大学秦曦明副教授馈赠;DH5α感受态细胞,购自北京天根生化科技有限公司;小鼠NIH3T3胚胎成纤维细胞和人胚肾HEK293T细胞,均由中国科学院典型培养物保藏委员会细胞库提供。
1.4 生物信息学分析 从NCBI数据库下载小鼠Usp2-45和Usp2-69的CDS区序列信息及其编码蛋白的氨基酸信息,使用ExPASy ProtParam在线工具(https://web.expasy.org/protparam/)分析USP2-45和USP2-69蛋白的基本理化特性,包括氨基酸组成、分子式、理论等电点、脂肪族系数和蛋白亲疏水性;利用SignalP 5.0(https://services.healthtech.dtu.dk/service.php?SignalP-5.0)和TMHMM-2.0(https://services.healthtech.dtu.dk/service.php?TMHMM-2.0)分别分析USP2-45和USP2-69蛋白是否具有信号肽和跨膜结构;使用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)和SWISS-MODEL(https://swissmodel.expasy.org/interactive)分别分析USP2-45和USP2-69蛋白的二级和三级结构。利用DNASTAR软件对不同物种间的Usp2-45和Usp2-69的CDS区进行同源性分析,并利用MEGA 7软件构建系统进化树,NCBI数据库中不同物种的Usp2-45和Usp2-69基因登录号如表1所示。结合Ensembl网站公布的Usp2-45和Usp2-69序列信息,利用Consite网站预测分析Usp2-45和Usp2-69转录起始位点-2 000~+200 bp范围内的启动子区域是否具有生物钟调控的顺式作用元件。

表1 不同物种的Usp2-45和Usp2-69基因登录号Table 1 Usp2-45 and Usp2-69 gene accession numbers in different species
1.5Usp2-45真核表达载体的构建
1.5.1 引物设计和合成 结合pcDNA3.1-Flag真核表达载体序列信息,以XhoⅠ为酶切位点(下划线),使用SnapGene软件设计含pcDNA3.1-Flag同源臂的小鼠Usp2-45 CDS区上、下游引物:F:5′-CGATATCGGATCCGGTACCCTCGAGGCCACCATGC-GTACCTCCTACACGGT-3′,R:5′-GGTCTTTGTAGTCACCGGTCTCGAGCATACGGGAGGGTGGAC-3′,引物交由西安擎科泽西生物科技有限责任公司合成。
1.5.2Usp2-45 CDS区目的片段扩增 称取20 mg小鼠肝脏组织放入1 mL TRIzol中,组织匀浆后提取总RNA,经逆转录获得cDNA。以cDNA为模板进行PCR扩增,反应体系为50 μL:cDNA 2 μL(<200 ng),10 μmol/L上、下游引物各1.5 μL,5×PrimeSTAR Buffer 10 μL,dNTP Mixture 4 μL,PrimeSTAR®HS DNA Polymerase 0.5 μL,补水至50 μL。反应程序:预变性98 ℃ 5 min;变性98 ℃ 10 s,退火55 ℃ 5 s,延伸72 ℃ 90 s,38个循环;终延伸72 ℃ 5 min,16 ℃结束反应。PCR产物进行1.5%琼脂糖凝胶电泳,对符合预期的目的条带进行胶回收纯化,获得Usp2-45的CDS区。
1.5.3 重组质粒的构建和鉴定 对pcDNA3.1-Flag进行单酶切获得线性化载体,将其与已酶切的Usp2-45的CDS区片段进行同源重组反应,反应体系为20 μL:pcDNA3.1线性化载体1 043 ng,Usp2-45 CDS区片段 76 ng,5×CE Ⅱ Buffer 4 μL,Exnase Ⅱ 2 μL,加去离子水补至20 μL;37 ℃孵育30 min。将获得的重组产物进行转化、涂板、挑取单克隆和增菌培养,选取菌落PCR鉴定正确的菌液提取质粒并进行酶切鉴定,将鉴定正确的质粒送至西安擎科泽西生物科技有限责任公司进行测序。将测序信息与NCBI数据库已公布序列进行比对,正确的重组质粒命名为pcDNA3.1-Usp2-45-Flag。
1.5.4 重组质粒转染NIH3T3细胞 将NIH3T3细胞接种于35 mm培养皿中,待细胞密度达70%~80%时进行转染,对照组转染pcDNA3.1-Flag空质粒,试验组转染pcDNA3.1-Usp2-45-Flag。将2 μg质粒与5 μL Turbofect转染试剂加至100 μL Opti-MEM无血清培养基中,混匀并孵育15 min;滴加至培养皿中,混匀后置于37 ℃、5% CO2条件下继续培养。
1.5.5 实时荧光定量PCR(Real-time quantitative PCR,qPCR)检测 转染48 h后,提取细胞样品总RNA并逆转录为cDNA。以cDNA为模板进行qPCR反应,反应体系为20 μL:cDNA 5 μL,SYBR®qPCR Mix 10 μL,上、下游引物各1 μL,ddH2O 3 μL。反应程序:95 ℃ 5 min,95 ℃ 10 s,60 ℃ 30 s,40个循环;95 ℃ 15 s,60 ℃ 60 s,95 ℃ 15 s。引物序列信息如表2所示,选择36b4为内参基因,采用2-ΔΔCt相对定量法计算目的基因的表达变化。
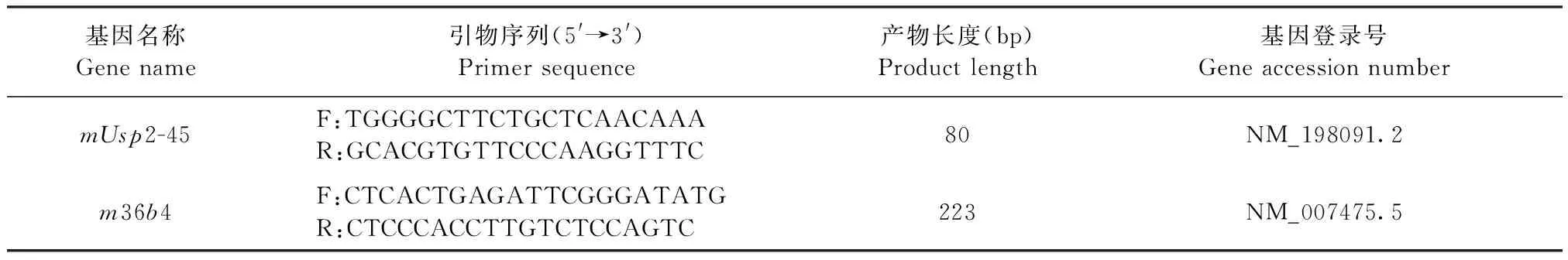
表2 qPCR引物序列Table 2 Primer sequences for qPCR
1.5.6 蛋白质免疫印迹(Western blot,WB)检测 转染48 h后,收集细胞样品并加入RIPA裂解液,提取细胞总蛋白。将蛋白样品稀释至同一浓度后,加入Loading Buffer煮沸使其变性。取20 μg蛋白样品进行SDS-PAGE凝胶电泳,并将蛋白转印至硝酸纤维素(Polyvinylidene fluoride,PVDF)膜,10%脱脂奶粉封闭2 h,TBST洗涤后将PVDF膜分别置于兔抗FLAG抗体和鼠抗β-actin抗体中,4 ℃孵育过夜;TBST洗3次,每次10 min,将PVDF膜分别孵育HRP共扼羊抗兔抗体和HRP共扼羊抗鼠抗体,室温摇床孵育1 h;TBST洗膜后,滴加ECL显色液进行曝光,使用Image J软件对蛋白条带进行灰度分析。
1.6Usp2启动子荧光素酶报告质粒的构建
1.6.1 启动子引物设计和合成 利用Primer Premier 6.0软件设计4对扩增Usp2-69和Usp2-45不同长度启动子的引物,具体序列信息如表3所示。
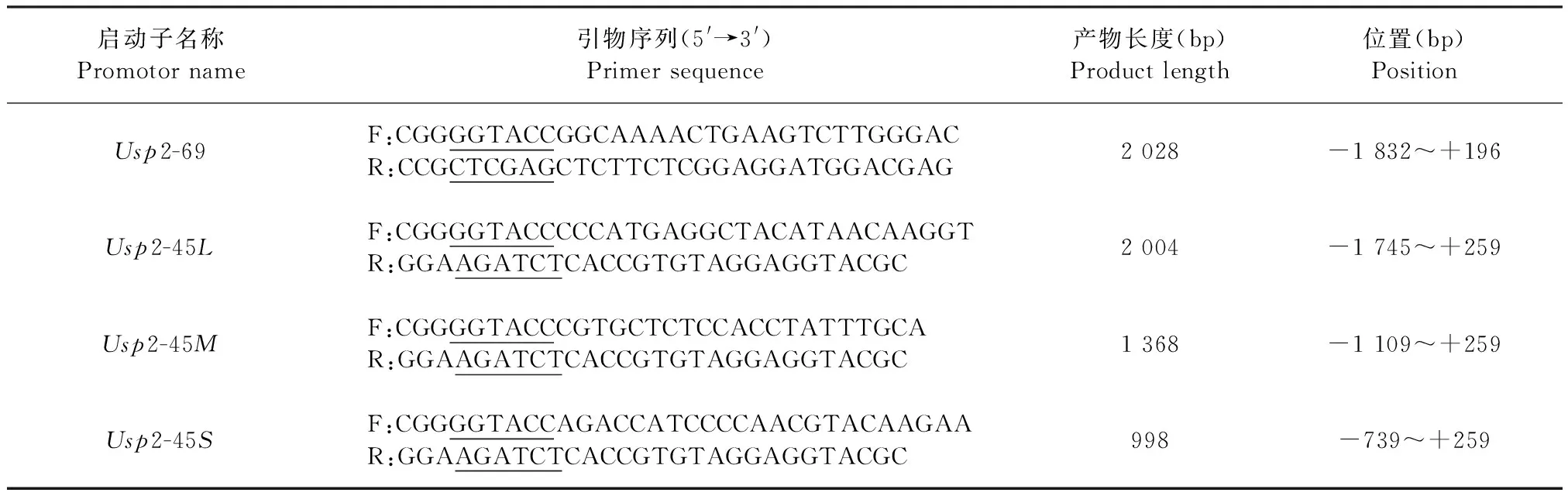
表3 Usp2启动子引物序列Table 3 Primer sequences of Usp2 promoter
1.6.2 启动子片段的扩增 称取100 mg小鼠肝脏提取全基因组,并以此为模板扩增Usp2-69和Usp2-45不同长度的启动子区域,反应体系为50 μL:5×PrimeSTAR GXL Buffer 10 μL,dNTP Mixture 4 μL,上、下游引物各1 μL,模板2 μL(500 ng),PrimeSTAR GXL DNA Polymerase 1 μL,补加ddH2O至50 μL。反应程序:预变性98 ℃ 10 min;变性98 ℃ 10 s,退火55 ℃ 15 s,延伸68 ℃ 150 s,循环35次;终延伸68 ℃ 5 min,16 ℃ 保存。反应结束后进行1.5%琼脂糖凝胶电泳,将目的条带进行胶回收。
1.6.3 启动子重组质粒的构建和鉴定 将获得的Usp2-69、Usp2-45L、Usp2-45M和Usp2-45S启动子片段酶切,酶切产物进行胶回收纯化。将酶切后的启动子片段、pGL4.10[luc2]与T4 DNA连接酶按比例混合,16 ℃连接过夜。后续经过转化、涂板、挑取单克隆、摇菌,提取质粒、酶切鉴定、测序等步骤后,将鉴定正确的质粒分别命名为Usp2-69-Luc、Usp2-45L-Luc、Usp2-45M-Luc和Usp2-45S-Luc。
1.6.4 双荧光素酶报告试验 将HEK293T细胞接种于24孔板中,24 h后将Usp2-45L-Luc或Usp2-69-Luc重组质粒分别与hBMAL1、mCLOCK和mPER2过表达载体共转染至细胞中,具体分组:(1)100 ng Usp2-45L-Luc或Usp2-69-Luc与600 ng pcDNA3.1空载体共转染组;(2)100 ng Usp2-45L-Luc或Usp2-69-Luc与150 ng BMAL1、150 ng CLOCK过表达质粒和300 ng pcDNA3.1共转染组;(3)100 ng Usp2-45L-Luc或Usp2-69-Luc与150 ng hBMAL1、150 ng mCLOCK、300 ng mPER2共转染组。同时,为进一步验证生物钟核心转录因子BMAL1和CLOCK对Usp2-45启动子的调控作用,分别将不同长度的Usp2-45启动子(Usp2-45L-Luc、Usp2-45M-Luc和Usp2-45S-Luc)与BMAL1、CLOCK过表达质粒共转染。以上处理组各转染5 ng pRL-CMV海肾荧光素酶报告质粒(内参质粒),同时每个处理组设置3个复孔。转染36 h后,按照双荧光素酶报告实验试剂盒说明书收集并处理细胞样品,使用多功能酶标仪检测并计算相对荧光值(萤火虫荧光值÷海肾荧光值),数据测量3次取平均值。
1.7 数据统计分析 使用GraphPad Prism 6.0软件对试验数据进行统计学分析,结果以“平均值±标准误”表示,两组间样本均值比较采取t检验,P<0.05表示差异显著,P<0.01表示差异极显著。
2 结果
2.1 小鼠USP2蛋白的基本理化性质 小鼠USP2-45蛋白分子式为C1998H3138N564O590S23,由396个氨基酸组成,其中含量占比前3的氨基酸依次为亮氨酸(11.9%)、丝氨酸(9.3%)和精氨酸(8.1%);不稳定系数52.56,为不稳定蛋白;理论等电点为8.92,脂肪族系数为74.39;平均亲水系数为-0.431,为亲水性蛋白。USP2-69分子式为C3032H4761N881O942S28,由619个氨基酸组成,其中含量占比前3的氨基酸依次为丝氨酸(12.8%)、亮氨酸(10.0%)和精氨酸(8.9%);不稳定系数50.84,为不稳定蛋白;理论等电点为9.34,脂肪族系数为62.89;平均亲水系数为-0.654,为亲水性蛋白。SignalP 5.0和TMHMM-2.0软件分析结果显示,小鼠USP2-45和USP2-69均不存在信号肽,不存在跨膜结构。
2.2 小鼠USP2-45和USP2-69蛋白的二级和三级结构预测 预测结果显示,α-螺旋、延伸链、β-转角和无规则卷曲在USP2-45二级结构中分别占35.86%、15.91%、1.77%和46.46%,在USP2-69二级结构中分别占32.47%、9.69%、2.75%和55.09%。小鼠和人类的USP2-45和USP2-69蛋白三级结构预测结果如图1所示,使用pyMOL软件对小鼠USP2-45(图1A)与人类USP2-45(图1B)进行蛋白结构比对,结果显示,原子位置均方根偏差(Root-mean-square deviation,RMSD)为0.032,两者差异极小;同时,对小鼠USP2-69(图1C)与人类USP2-69(图1D)蛋白进行结构比对,RMSD为0.049,两者差异极小。
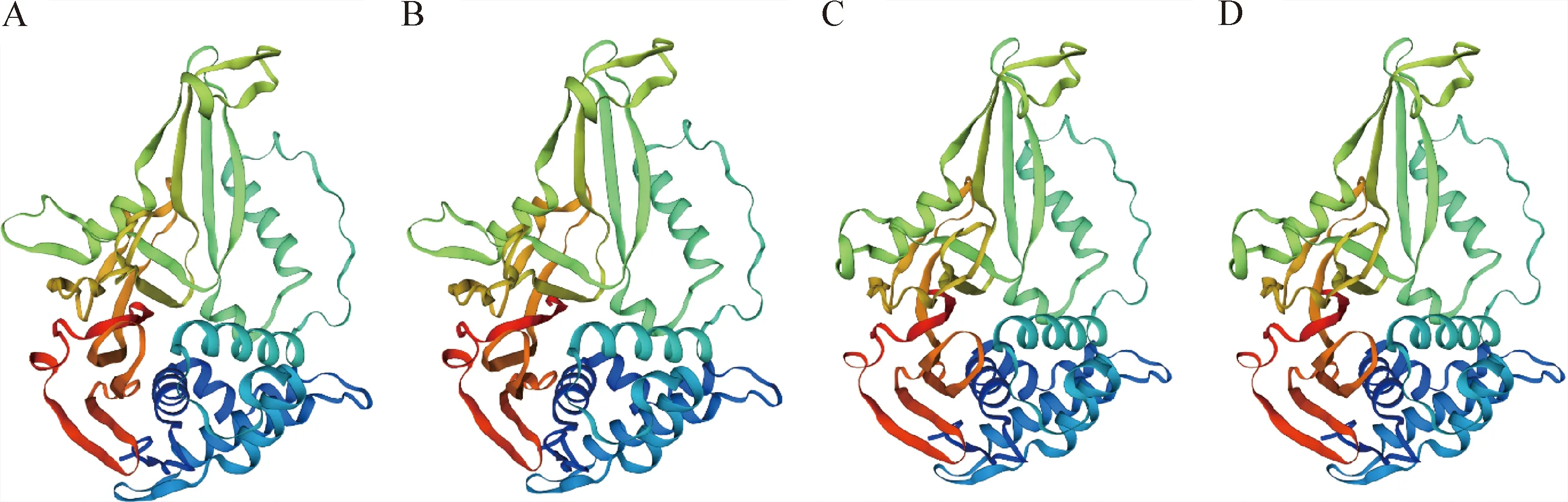
图1 小鼠和人类USP2-45和USP2-69蛋白的三级结构预测Fig.1 Tertiary structure prediction of USP2-45 and USP2-69 proteins in mouse and humanA:小鼠USP2-45蛋白三级结构预测; B:人类USP2-45蛋白三级结构预测; C:小鼠USP2-69蛋白三级结构预测; D:人类USP2-69蛋白三级结构预测A:Predicted tertiary structure of mouse USP2-45 protein; B:Predicted tertiary structure of human USP2-45 protein; C:Predicted tertiary structure of mouse USP2-69 protein; D:Predicted tertiary structure of human USP2-69 protein
2.3 小鼠Usp2-45基因同源性分析和进化树构建 利用生物信息学软件DNASTAR和MEGA 7对不同物种的Usp2-45 CDS区进行同源性分析和比对,结果如图2A所示,小鼠Usp2-45的CDS区与其他9种物种的同源性均保持在89%以上,具有较高的相似性和保守性;系统进化树如图2B所示,小鼠Usp2-45转录本与大鼠的亲缘关系最近,与狗和猫的关系较远。
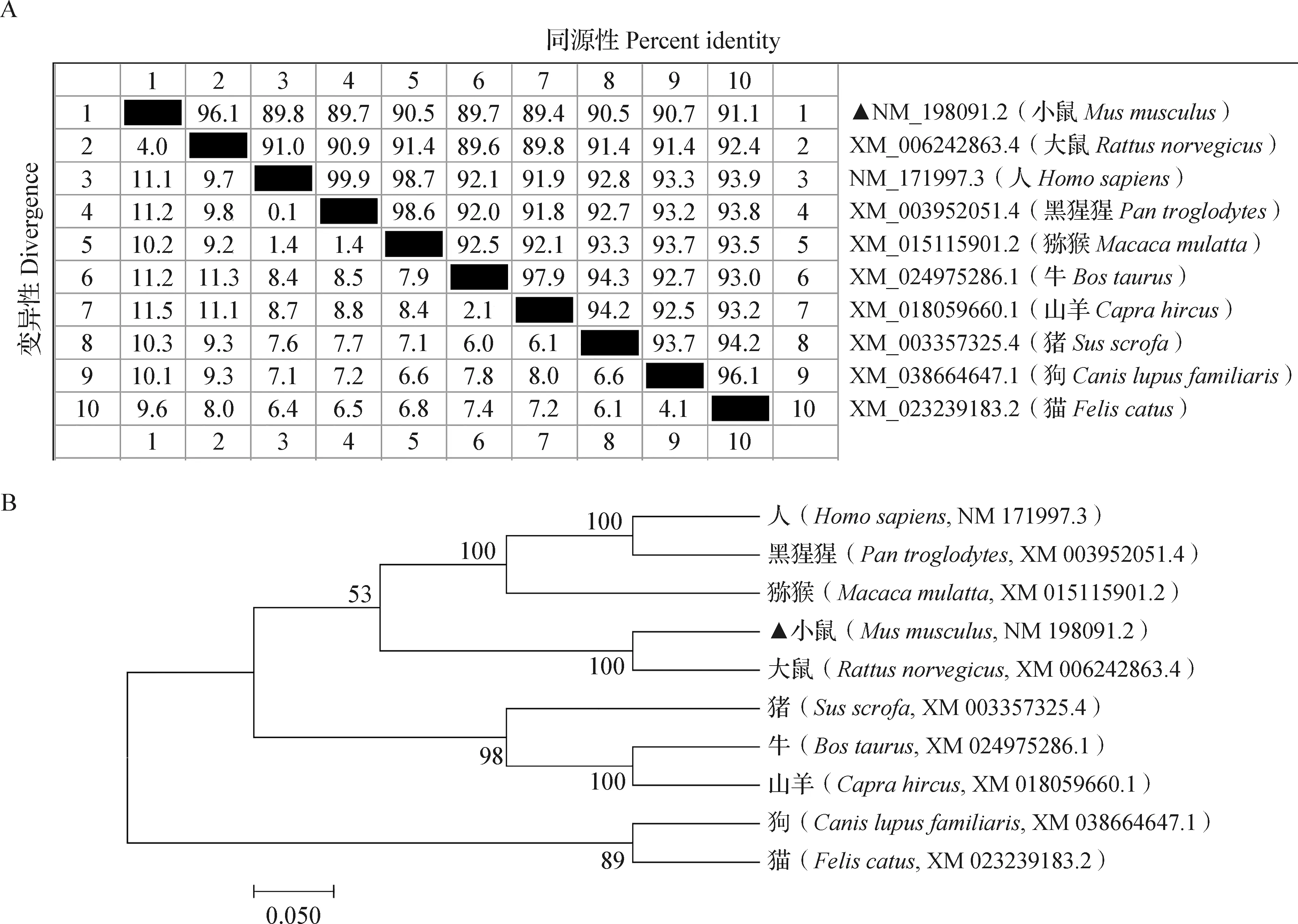
图2 不同物种Usp2-45的CDS区同源性分析(A)和系统进化树分析(B)Fig.2 Homology analysis (A) and phylogenetic tree analysis (B) of Usp2-45 CDS region in different species▲:本试验中的小鼠Usp2-45基因NCBI登录号▲:NCBI accession number of mouse Usp2-45 gene in this study
2.4 小鼠Usp2-69基因同源性分析和进化树构建 同时,对不同物种的Usp2-69 CDS区进行同源性分析和比对,结果如图3A所示,小鼠Usp2-69的CDS区与其他9种物种的同源性均保持在87%以上,具有较高的相似性和保守性;系统进化树如图3B所示,小鼠Usp2-69转录本与大鼠的亲缘关系最近,与狗和猫的关系较远。

图3 不同物种Usp2-69 CDS区的同源性分析(A)和系统进化树分析(B)Fig.3 Homology analysis (A) and phylogenetic tree analysis (B) of Usp2-69 CDS region in different species▲:本试验中的小鼠Usp2-69基因NCBI登录号▲:NCBI accession number of mice Usp2-69 gene in this study
2.5 pcDNA3.1-Usp2-45-Flag的构建 如图4A所示,小鼠Usp2-45 CDS区的PCR扩增产物条带单一,与预期大小相符(1 191 bp)。pcDNA3.1-Usp2-45-Flag的酶切鉴定结果如图4B所示,琼脂糖凝胶电泳后获得的2条条带与预期大小相符(Usp2-45 CDS:1 191 bp;pcDNA3.1-Flag:5 515 bp)。将酶切鉴定正确的质粒测序,测序结果与Usp2-45在NCBI数据库公布的序列信息完全一致,表明pcDNA3.1-Usp2-45-Flag构建成功。
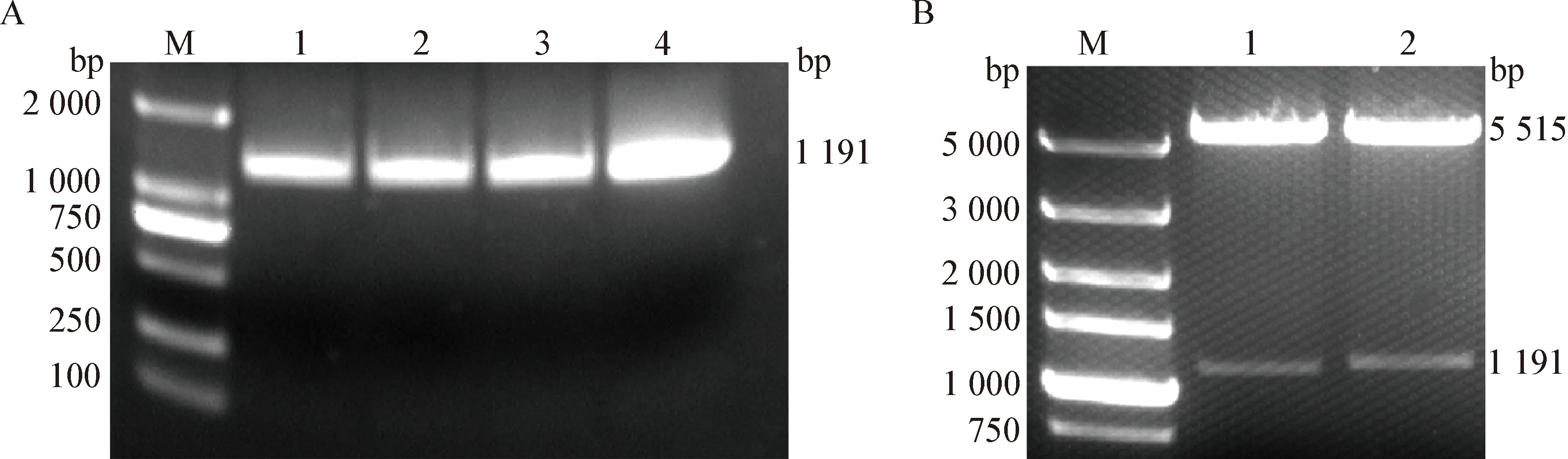
图4 PCR扩增和酶切鉴定Fig.4 PCR amplification and enzyme digestion identification A:Usp2-45 CDS区的PCR扩增(M:DL2 000 DNA分子量标准; 1~4:PCR产物); B:pcDNA3.1-Usp2-45-Flag质粒酶切鉴定(M:DL5 000 DNA分子量标准; 1~2:酶切产物)A:PCR amplification of the Usp2-45 CDS region (M:DL2 000 DNA Marker; 1-4:PCR products); B:Enzyme digestion identification of pcDNA3.1-Usp2-45-Flag plasmid (M:DL5 000 DNA Marker; 1-2:Enzyme digestion products)
2.6Usp2-45在NIH3T3细胞中的过表达 分别将pcDNA3.1-Flag(对照组)和pcDNA3.1-Usp2-45-Flag(试验组)转染至NIH3T3细胞,qPCR结果如图5A所示,试验组的Usp2-45 mRNA表达水平极显著高于对照组(P< 0.01);WB结果如图5B所示,对照组在相应位置无条带,而试验组的USP2-45蛋白成功表达。
2.7 小鼠Usp2-45和Usp2-69的启动子序列分析 利用Consite网站预测分析Usp2-45和Usp2-69转录起始位点-2 000~+200 bp范围内的启动子区域能够与生物钟核心转录因子BMAL1和CLOCK相互作用的关键顺式作用元件。如图6所示,Usp2-45启动子区域存在2个典型的E-box(5′-CACGTG-3′),分别位于其转录起始位点上游-764~-769 bp和-1 389~-1 394 bp区域内;Usp2-69启动子序列仅存在1个典型的E-box,位于其转录起始位点-153~-158 bp区域内。
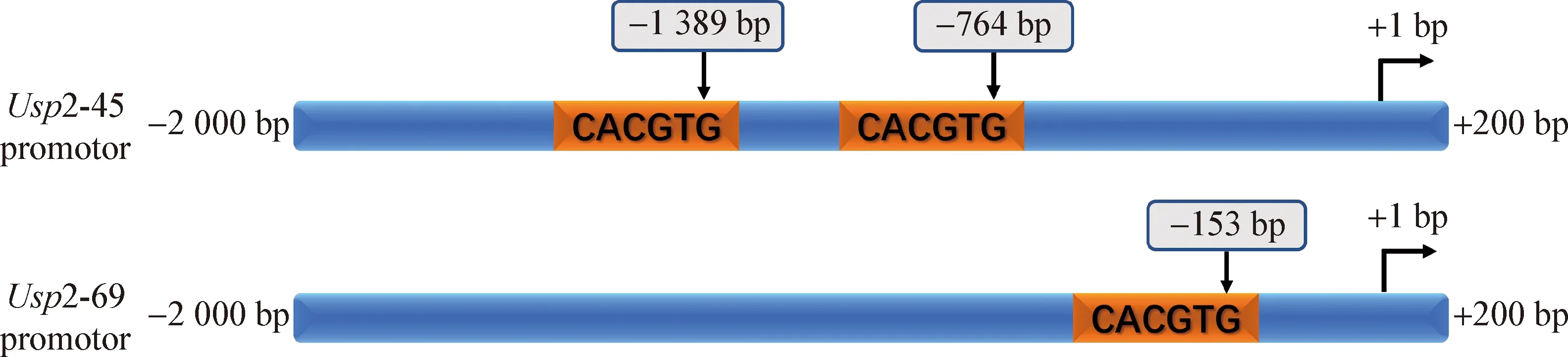
图6 小鼠Usp2-45和Usp2-69启动子结构示意图Fig.6 Schematic diagram of mouse Usp2-45 and Usp2-69 gene promotor structures
2.8 生物钟选择性调控Usp2-45和Usp2-69启动子的转录活性 以小鼠基因组为模板,采用PCR扩增获得Usp2-45L和Usp2-69的启动子区域的DNA片段(Usp2-45L:-1 745~+259 bp,共2 004 bp;Usp2-69:-1 832~+196 bp,共2 028 bp)。将胶回收后的目的片段进行酶切并与pGL4.10[luc2] 载体相连,获得Usp2-45L-Luc和Usp2-69-Luc重组质粒(结果未展示),用于双荧光素酶报告试验。如图7A所示,BMAL1和CLOCK过表达质粒的共转染极显著提高了Usp2-45启动子调控下的荧光素酶活性(P<0.01),生物钟转录抑制因子PER2过表达后显著抑制了Usp2-45启动子调控下的荧光素酶活性(P<0.05);如图7B所示,BMAL1、CLOCK和PER2过表达质粒的共转染对Usp2-69启动子调控下的荧光素酶活性均无显著影响(P>0.05)。
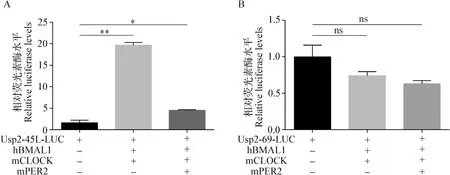
图7 Usp2-45(A)和Usp2-69(B)启动子的双荧光素酶报告试验Fig.7 Dual-luciferase reporter assay of Usp2-45 (A) and Usp2-69 (B) promoters+:添加该质粒; -:不添加该质粒; *:P<0.05,**:P<0.01,ns:无显著性差异; 下图同+:Addition of the plasmid; -:No addition of the plasmid; *:P<0.05,**:P<0.01,ns:No significant difference. The same as below
2.9 生物钟调控Usp2-45转录的启动子功能区鉴定 为了鉴定生物钟调控Usp2-45转录的启动子功能区,在已构建Usp2-45L-Luc(-1 745~+259 bp,2 004 bp)荧光素酶报告质粒的基础上,进一步成功构建了2种不同长度的Usp2-45启动子荧光素酶报告质粒,即Usp2-45M-Luc(-1 109~+259 bp,1 368 bp)和Usp2-45S-Luc(-739~+259 bp,998 bp),启动子序列和E-box元件位置信息如图8 A所示。双荧光素酶报告试验结果显示,BMAL1和CLOCK过表达质粒共转染均极显著提高了不同长度Usp2-45启动子的荧光素酶活性(P<0.01),活性由强到弱依次为1 368、2 004和998 bp。值得注意的是,Usp2-45S-Luc对BMAL1/CLOCK过表达质粒的应答活性极显著低于Usp2-45L-Luc和Usp2-45M-Luc(P<0.01)(图8B)。上述结果提示,生物钟调控Usp2-45转录的重要功能区位于其转录起始位点-740~-1 109 bp的范围内。
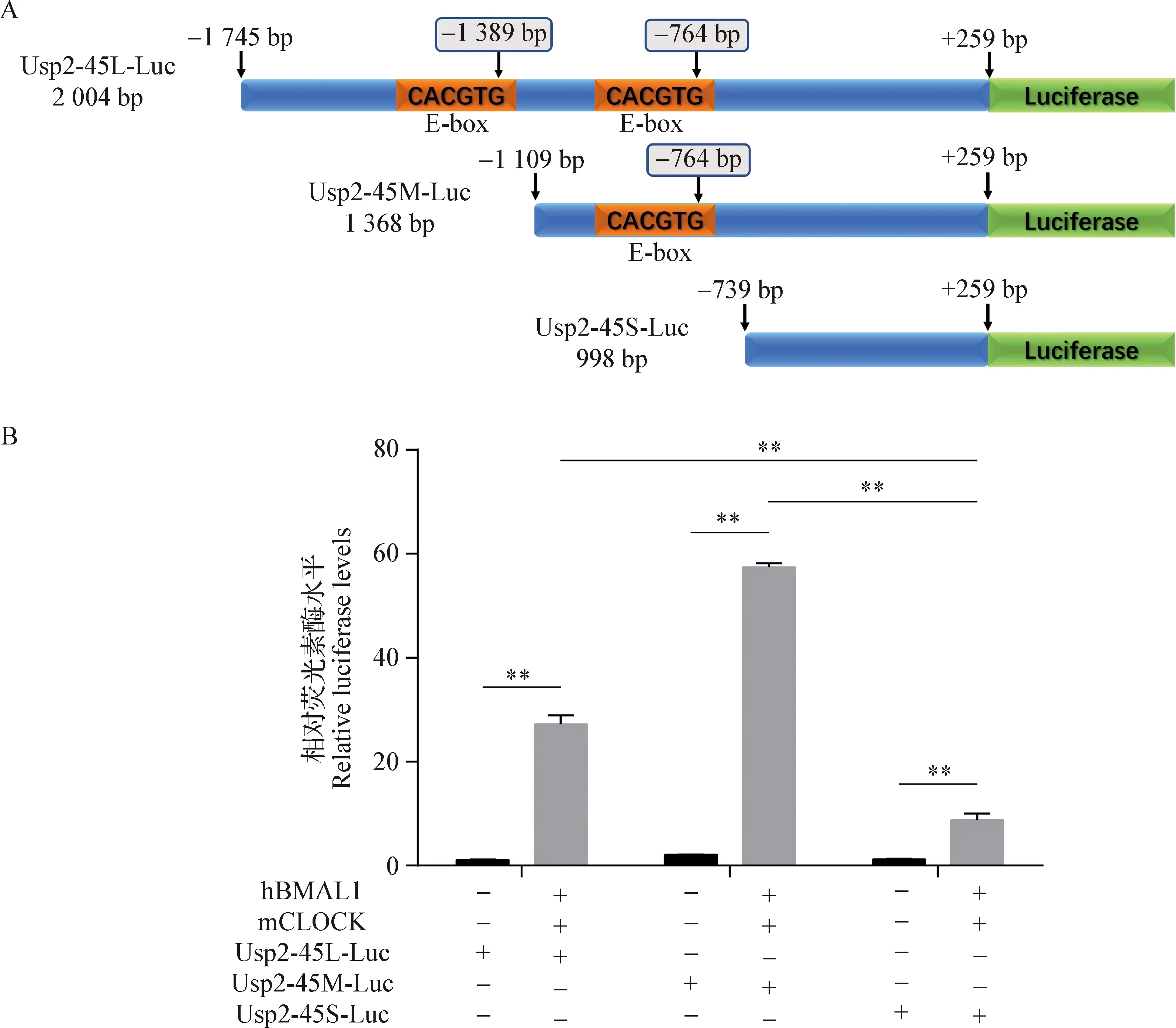
图8 双荧光素酶报告试验检测不同长度的Usp2-45启动子对BMAL1和CLOCK蛋白的应答活性Fig.8 Dual luciferase reporter assay examing the response activities of different lengths of Usp2-45 promoters to BMAL1 and CLOCK proteins A:Usp2-45L-Luc、Usp2-45M-Luc和Usp2-45S-Luc质粒结构示意图; B:双荧光素酶报告试验A:Schematic diagram of Usp2-45L-Luc,Usp2-45M-Luc and Usp2-45S-Luc plasmid structure; B:Dual-luciferase reporter assay
3 讨论
泛素化作为一种重要的蛋白翻译后修饰过程,参与调控细胞周期、细胞增殖和凋亡、细胞分化、转录调控、信号传导、损伤修复以及炎症免疫等几乎一切生命活动,在蛋白的定位和代谢等方面发挥重要作用[20,28]。真核生物体内80%~85%的蛋白降解依赖于泛素化-蛋白酶体途径。同时,泛素化的可逆过程去泛素化在维持蛋白的稳定和细胞稳态方面具有重要意义。去泛素化酶USPs家族在此过程中扮演重要角色[29]。
作为USPs家族的成员,小鼠Usp2基因位于9号染色体(9A5.1),包含21个外显子,经过可变剪接可产生9个转录本,但仅有7个转录本可以编码蛋白[11]。Gousseva和Baker报道了小鼠Usp2基因的结构和组织表达模式,USP2主要存在USP2-45和USP2-69两种蛋白亚型[12],但目前尚缺乏关于小鼠USP2蛋白结构和功能的深入研究。本试验结果显示,USP2-45包含396个氨基酸,USP2-69包含619个氨基酸,二者含量最多的前3种氨基酸均为丝氨酸、亮氨酸和精氨酸;USP2-45和USP2-69蛋白均无信号肽和跨膜结构,且在小鼠和人类两物种之间的三级结构差异较小。不同物种的Usp2-45和Usp2-69同源性分析结果显示,小鼠Usp2-45与其他9种物种的同源性均保持在89%以上,Usp2-69与其他9种物种的同源性均保持在87%以上;进化树结果显示,小鼠Usp2-45和Usp2-69转录本与大鼠的亲缘关系最近。前期研究发现,大鼠中存在小鼠Usp2-45和Usp2-69的直系同源物,并将其命名为睾丸特异性和发育调节蛋白Ubp-t1和Ubp-t2,表明Usp2-45和Usp2-69基因在小鼠与大鼠之间具有较高的相似性和保守性[30]。
尽管USP2-45和USP2-69具有相似的结构和理化特性,二者在同一生理或病理过程中的功能表现却存在明显的差异[14,31]。Tao等通过干扰Usp2-69的表达显著增加了前列腺癌、乳腺癌和结肠癌等细胞系的凋亡率,表明USP2-69具有抗凋亡功能[17];与之相反的是,Gewies等发现,在Hela细胞中过表达UBP41(根据氨基酸序列推断其为USP2-45)可显著诱导其凋亡[32]。前期研究证据表明,Usp2-45节律性表达于小鼠肝脏中,Usp2-69的表达却无明显的节律性变化,Bmal1肝脏特异性敲除小鼠肝脏中Usp2-45的表达几乎完全丧失节律性变化,表明生物钟系统对Usp2-45和Usp2-69具有不同的选择性调控作用[13,33,34]。为深入探究生物钟系统对Usp2-45和Usp2-69的选择性调控机制,本试验通过对Usp2-45和Usp2-69的启动子区域预测分析发现,Usp2-45启动子区域含有2个经典的E-box元件(位于-764~-769 bp和-1 389~-1 394 bp),而Usp2-69仅含有1个E-box元件(位于-153~-158 bp)。双荧光素酶报告试验结果显示,BMAL1/CLOCK对Usp2-69启动子无转录调控活性,但可正向调控Usp2-45的启动子活性,这与Molusky等的研究结果一致[33]。本试验进一步构建了2种不同长度的Usp2-45启动子序列的荧光素酶报告质粒,即Usp2-45M-Luc(-1 109~+259 bp,1 368 bp)和Usp2-45S-Luc(-739~+259 bp,998 bp),结果显示,BMAL1/CLOCK过表达质粒共转染均极显著提高了不同长度Usp2-45启动子的荧光素酶活性,活性由强到弱依次为1 368、2 004和998 bp。上述结果表明,生物钟调控Usp2-45转录的重要功能区位于其转录起始位点-740~-1 109 bp的范围内。结合Koike等在2012年公布的ChIP-seq数据,以及NCBI数据库的Usp2-45碱基序列分析,BMAL1蛋白与Usp2-45基因的结合位点集中在-1 000~-1 400 bp[35],而这与本试验结果高度一致。但生物钟系统调控Usp2-45节律性转录的具体分子机制有待后续更深入的研究。
本试验分析了小鼠Usp2-45和Usp2-69转录本及其编码蛋白的基本理化特性和结构功能,克隆了小鼠Usp2-45的CDS区,并成功构建了pcDNA3.1-Usp2-45-Flag真核表达载体。同时,本试验成功构建了不同长度的Usp2启动子荧光素酶报告载体,通过双荧光素酶报告试验检测到BMAL1/CLOCK对Usp2-69启动子活性无显著影响,但可显著提高Usp2-45的启动子活性,且对启动子区域中-740~-1 109 bp范围内的功能位点调控作用更强,为后续深入研究生物钟系统与USP2互作的分子调控机制提供了前期理论基础和关键材料。
