机器学习在动物基因组选择中的研究进展
2023-10-23李棉燕王立贤赵福平
李棉燕,王立贤,赵福平
机器学习在动物基因组选择中的研究进展
李棉燕,王立贤,赵福平
中国农业科学院北京畜牧兽医研究所/农业部动物遗传育种与繁殖(家禽)重点实验室,北京 100193
基因组选择是指利用覆盖在全基因组范围内的分子标记信息来估计个体育种值。利用基因组信息能够避免因系谱错误带来的诸多问题,提高选择准确性并缩短育种世代间隔。根据统计模型的不同,基因组选择方法可大致分为基于BLUP(best linear unbiased prediction, BLUP)理论的方法、基于贝叶斯理论的方法和其他方法。目前应用较多的是GBLUP及其改进方法ssGBLUP。准确性是基因组选择模型最常用的评价指标,用来衡量真实值和估计值之间的相似程度。影响准确性的因素可以从模型中体现,大致分为可控因素和不可控因素。传统基因组选择方法促进了动物育种的快速发展,但这些方法目前都面临着多群体、多组学和计算等诸多挑战,不能捕获基因组高维数据间的非线性关系。作为人工智能的一个分支,机器学习是最贴近生物掌握自然语言处理能力的一种方式。机器学习从数据中提取特征并自动总结规律,利用该规律与新数据进行预测。对于基因组信息,机器学习无需进行分布假设,且所有的标记信息都能够被考虑进模型当中。相比于传统的基因组选择方法,机器学习更容易捕获基因型之间、表型与环境之间的复杂关系。因此,机器学习在动物基因组选择中具有一定的优势。根据训练期间接受的监督数量和监督类型,机器学习可分为监督学习、无监督学习、半监督学习和强化学习等。它们的主要区别为输入的数据是否带有标签。目前在动物基因组选择中应用的机器学习方法均为监督学习。监督学习可以处理分类和回归问题,需要向算法提供有标签的数据和所需的输出。近年来机器学习在动物基因组选择中的应用不断增多,特别是在奶牛和肉牛中发展较快。本文将机器学习算法划分为单个算法、集成算法和深度学习3类,综述其在动物基因组选择中的研究进展。单个算法中最常用的是KRR和SVR,两者都是通过核技巧来学习非线性函数,在原始空间中将数据映射到更高维的核空间。目前常用的核函数有线性核、余弦核、高斯核和多项式核等。深度学习又称为深度神经网络,由连接神经元的多个层组成。集成学习算法则是指将不同的学习器融合在一起进而得到一个较强的监督模型。近十年来,有关机器学习和深度学习的相关文献呈现了指数型的增长,在基因组选择方面的应用也在逐渐增多。尽管机器学习在某些方面存在明显的优势,但其在估计动物复杂性状基因组育种值时仍面临诸多挑战。部分模型的可解释性低,不利于数据、参数和特征的调整。数据的异质性、稀疏性和异常值也会造成机器学习的数据噪声。还有过拟合、大标记小样本和调参等问题。因此,在训练模型时需要谨慎处理每一个步骤。文章介绍了基因组选择传统方法及其面临的问题、机器学习的概念和分类,探讨了机器学习在动物基因组选择中的研究进展及目前存在的挑战,并给出了一个案例和一些应用的建议,以期为机器学习在动物基因组选择当中的应用提供一定参考。
机器学习;深度学习;基因组选择;动物育种
0 引言
许多重要动物性状都呈现出了复杂的遗传机制,这使得个体基因位点的识别变得困难。基因组选择是指利用覆盖全基因组范围内的分子标记信息估计个体育种值[1]。基因组选择假设每一个数量性状基因座(quantitative trait locus, QTL)都与全基因组中至少一个分子标记处于连锁不平衡状态,因此可以利用标记效应来解释遗传方差[2]。利用基因组信息进行选择不仅能够避免因系谱错误或丢失造成的问题,而且大幅缩短了育种世代间隔[3]。
统计模型是全基因组选择的核心,也是影响预测准确性和效率的主要因素。根据算法的不同,可将其分为BLUP(best linear unbiased prediction, BLUP)系列、贝叶斯系列和机器学习。BLUP系列又称为直接法,把个体作为随机效应,参考群体和候选群体遗传信息构建的亲缘关系矩阵作为方差协方差矩阵,通过迭代法估计方差组分,进而求解混合模型获取候选群体的个体估计育种值。贝叶斯系列又称为间接法,首先在参考群体中估计标记效应,然后结合候选群体的基因型信息将标记效应进行累加,最后获得候选群体的个体估计育种值。不同方法的选择准确性会根据不同群体和不同性状等实际情况而[4-7]异,但后者的GBLUP(genomic best linear unbiased prediction, GBLUP)方法在研究中出现的频率较高。BLUP系列方法假设所有标记都具有相同的效应,但实际基因组范围中只有少量标记具有主效应,大部分标记的效应较小。相比于BLUP方法,贝叶斯方法对数据的假设条件更符合数量性状的遗传结构,但其采用特定算法估计未知参数时则往往耗时更久。
机器学习是一门人工智能的科学,在经验中学习并改善具体算法。机器学习不仅在理论和工程上是实现自然语言处理的首选,也是最贴近生物掌握自然语言处理能力的一种方式。机器学习已经被成功应用于多个领域,其广泛用途已在书籍和文献中有较多概述[8-10]。机器学习是非参数模型,其算法比线性选择模型复杂得多。近年来,利用机器学习算法进行生物学相关领域的热度不断增高,如人类疾病选择[11-13]、多组学数据整合[14]、植物育种[15-17]等。相比起传统的选择方法,机器学习更容易捕捉基因型之间、表型与环境之间的复杂关系,在动物基因组选择中表现出了一定的优势。因此,本文对基因组选择传统方法、选择评价指标、机器学习在动物基因组选择中的研究进展进行综述。
1 基因组选择
1.1 基因组选择方法
统计模型和计算方法是基因组选择的核心。根据所使用统计模型的不同,基因组选择方法大致可分为3类:基于BLUP理论的方法、基于贝叶斯理论的方法和其他方法。没有任何一个方法能在所有情况下都具有绝对优势。目前应用较多的是GBLUP及其改进方法ssGBLUP(single-step genomic best linear unbiased prediction, ssBLUP)。
1.1.1 基于BLUP理论的方法 主要包括RR-BLUP、GBLUP、ssGBLUP等。BLUP把个体作为随机效应,参考群体和候选群体遗传信息构建的亲缘关系矩阵作为方差协方差矩阵,求解方差组分和混合模型获得个体的GEBV。
1.1.2 基于贝叶斯理论的方法 主要包括BayesA、BayesB、BayesCπ、BayesR等。贝叶斯系列方法通过估计标记效应间接获得基因组估计育种值(genomic estimated breeding value, GEBV)。估计过程分为两步:首先利用参考群个体的表型和全基因组标记基因型信息估计每一个标记的效应值,然后根据候选群中每个位点的标记基因型将标记效应累加获得个体的GEBV[18]。
1.1.3 其他方法 主要包括RKHS(reproducing kernel Hilbert space, RKHS)、半参数方法和机器学习等。由于此类方法中考虑互作效应以及其他非线性效应,使得模型非常复杂或者计算难度较大。因此,目前研究和应用最多的还是前两类方法。机器学习方法将在后文做详细介绍。
1.2 基因组育种值估计准确性
1.2.1 准确性评估指标 基因组育种值估计准确性是指基因组估计育种值与真实值之间的相关系数,是估计育种值研究中最常用的评价指标。在模拟研究中已知真实育种值,育种值估计准确性可以直接计算。但是在实际的育种数据研究中真实育种值未知,应用条件复杂,所以出现了不同的准确性评估方法[19],如真实育种值与估计值之间的皮尔逊相关系数、均方根误差、平均绝对误差等。
1.2.2 影响因素 从选择模型中可以体现影响选择准确性的关键因素。这些因素可大致分为可控因素和不可控因素。
(1)可控因素 即估计过程中需要考虑并可以人为调整的因素,如估计方法、群体大小及遗传关系、标记的数量和密度等。研究表明,随着遗传力水平和标记密度的增加,基因组估计育种值的准确性得到提高[20]。有效群体规模不变时,训练世代越多,基因组估计育种值的准确性和稳定性越好[21]。参考群体越大,选择准确性也会有所提升[22]。
(2)不可控因素 染色体的长度、影响性状的基因数和性状的遗传力等也会影响选择准确性。这些因素会因物种而异,所以导致了基因组选择效果在不同物种、不同群体和不同性状中的相差较大。这些因素都是客观存在、不能人为改变的。
2 机器学习
机器学习属于人工智能的一个分支,是一类算法的总称。机器学习的基本思路是将现实问题转化为数学问题,机器解决数学问题从而解决现实问题。机器学习最初被定义为:计算机无需明确编程即可获得学习能力的研究领域[23]。数据、算法和模型是机器学习的三要素。机器学习选取合适的算法,从已有数据中自动归纳总结规律,利用该规律的结果(模型)与新数据进行选择。
根据训练期间接受的监督数量和监督类型,可将机器学习分为监督学习(supervised learning, SL)、无监督学习(unsupervised learning, USL)、半监督学习(semi-supervised learning, SSL)和强化学习(reinforcement learning, RL)。这些不同类型最主要的区别是训练的样本是否带有标签。所以,在无监督学习中的训练数据是未经标记的,算法会在没有指导的情况下自动学习。
2.1 监督学习
监督学习需要向算法提供有标签的数据和所需的输出。即目标变量的数值必须被确定,以便机器学习算法可以发现特征和目标变量之间的关系。监督学习主要处理分类和回归问题。值得注意的是,在动物基因组选择中所使用的机器学习均为监督学习,如支持向量机回归(support vector regression, SVR)、核岭回归(kernel ridge regression, KRR)、随机森林(random forest, RF)和决策树(decision tree, DT)等。
2.2 无监督学习
无监督学习对于没有给定标签的数据进行自动分析,试图找到隐藏的结构。数据没有类别信息,也没有给定的目标值。该方法可以降低数据特征的维度,使用二维或三维图形更加直观地展示数据信息。非监督学习包括聚类和密度估计,如K-means算法及其一些扩展算法。
2.3 半监督学习
半监督学习结合了监督学习和无监督学习的中间类别,输入的数据部分有标签,部分没有。基本思路是利用数据分布上的模型假设,建立学习器对未标签样本进行选择。半监督学习可以处理分类和回归问题。算法包括一些常用监督学习算法的延伸,如支持向量机(support vector machine, SVM)和图论推理算法等。
2.4 强化学习
强化学习是多学科多领域交叉的一个产物,旨在学习自动决策。该方法普适性强,基于决策进行训练,算法根据输出结果(决策)的成功或错误来训练自己,通过大量经验训练优化后的算法来进行选择,常见的算法包括Q-Learning和时间差分学习等。
3 机器学习在动物基因组选择中的应用
近年来,从数学、统计和计算科学引入的多种机器学习算法推动了生物学领域的快速发展。在基因组选择当中常用的机器学习方法包括SVR、KRR、RF和深度学习等。因其算法的特殊性,机器学习可以在不同维度弥补传统方法的缺陷。不同于传统方法,机器学习不需要对选择的变量进行分布假设[24],并且所有的标记信息都能够被考虑进模型当中。也就是说,那些具有弱效应、高度相关和相互作用的标记物都有机会对模型拟合做出贡献[4]。同时,机器学习的默认参数通常表现较好,无需进行大幅度调参[25]。
由于数据结构的不同,有时单个算法就能够起到很好的选择效果,有时则需要采用集成学习和深度学习。如LIANG等[26]在中国西门塔尔肉牛群体中比较了SVR,KRR,RF,Adaboost.RT和GBLUP等5种基因组选择方法的准确性,结果表明4种机器学习方法相对于传统方法GBLUP平均改进了12.8%、14.9%、5.4%和14.4%。在4种机器学习方法中,集成方法Adaboost.RT具有更高的稳定性,准确性与KRR相当。总体而言,机器学习在动物基因组选择中的应用研究不多,但已经成功应用于多个动物品种,特别是肉牛和奶牛。表1列举了部分机器学习算法在动物基因组选择中的应用研究。
图1展示了在Web of Science网站中搜索“机器学习”“深度学习”“机器学习+基因组选择”和“深度学习+基因组选择”等关键词,获得在2011—2022年之间每一年的相关出版物数量。2022年的查找时间截止为9月6日。折线图能很好地呈现数据的特点和趋势。可见,十年内有关机器学习和深度学习的相关文献呈现了指数型的增长,在基因组选择方面的应用也在逐渐增多。
3.1 单个机器学习算法的应用
KRR是岭回归的核版本,在岭回归的基础上引入了核函数。KRR在原始空间中应用核函数将数据映射到更高维的核空间中,以提供基于正则化最小二乘法的泛化性能[40]。这种方式可以使原始数据分离,从而提高回归和分类问题的准确性和稳定性。目前常用的核函数有线性核、余弦核、高斯核和多项式核等。其中,余弦核捕获了不同样本在每个维度向量之间的余弦距离[41]。AN等[24]开发了一个基于余弦核的KCRR算法用于基因组选择,并将其中的余弦相似性矩阵代替传统G矩阵得到了GBLUP_CS方法。他们利用4个群体的12个具有不同遗传力和遗传结构的复杂性状对比了GBLUP、BayesB、SVR、KCRR和GBLUP_CS,发现KCRR在选择准确性和计算效率方面都表现良好,并且GBLUP_CS的运算速度比GBLUP快了20倍。LU等[42]通过整合核函数和线性回归分类提出了KLRC算法,研究结果表明该算法在人脸识别中具有良好的选择效果。HE等[31]提出了一种KRRC算法,在两个合成数据集和一个真实数据集中进行比较发现其选择效果比所选的KNN、LRC和KLRC效果好。
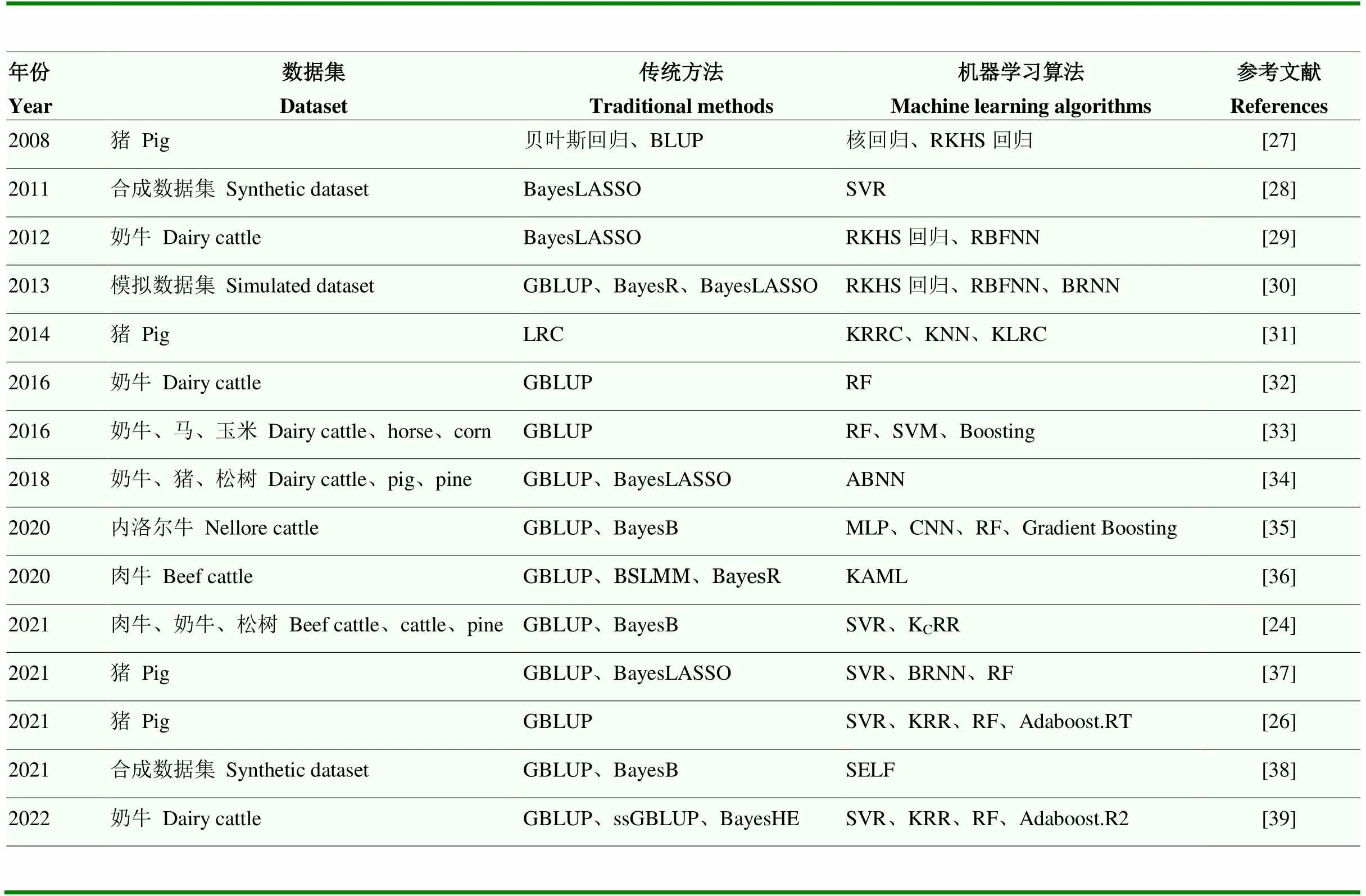
表1 机器学习在动物基因组选择中的应用
RKHS:再生希尔伯特空间;RBFNN:径向基函数神经网络;BRNN:贝叶斯正则化神经网络;LRC:线性回归分类;KNN :K近邻;KLRC:核线性回归分类;ABNN:人工贝叶斯神经网络;MLP:多层感知机;CNN:卷积神经网络;BSLMM:贝叶斯稀疏线性混合模型;KAML:亲缘校正多位点;KCRR:余弦核岭回归;SELF:堆叠集成学习框架
RKHS:Reproducing kernel Hilbert spaces;RBFNN:Radial basis function neural networks;BRNN: Bayesian regularized neural networks;LRC :Linear regression classification;KNN: K nearest neighbor;KLRC:Kernel linear regression classification;ABNN :Artificial Bayesian neural network;MLP :Multilayer perceptron;CNN:Convolutional neural network; BSLMM :Bayesian sparse linear mixed model;KAML Kinship adjusted multi-loci;KCRR:Cosine kernel–based KRR;SELF :Stacking ensemble learning framework
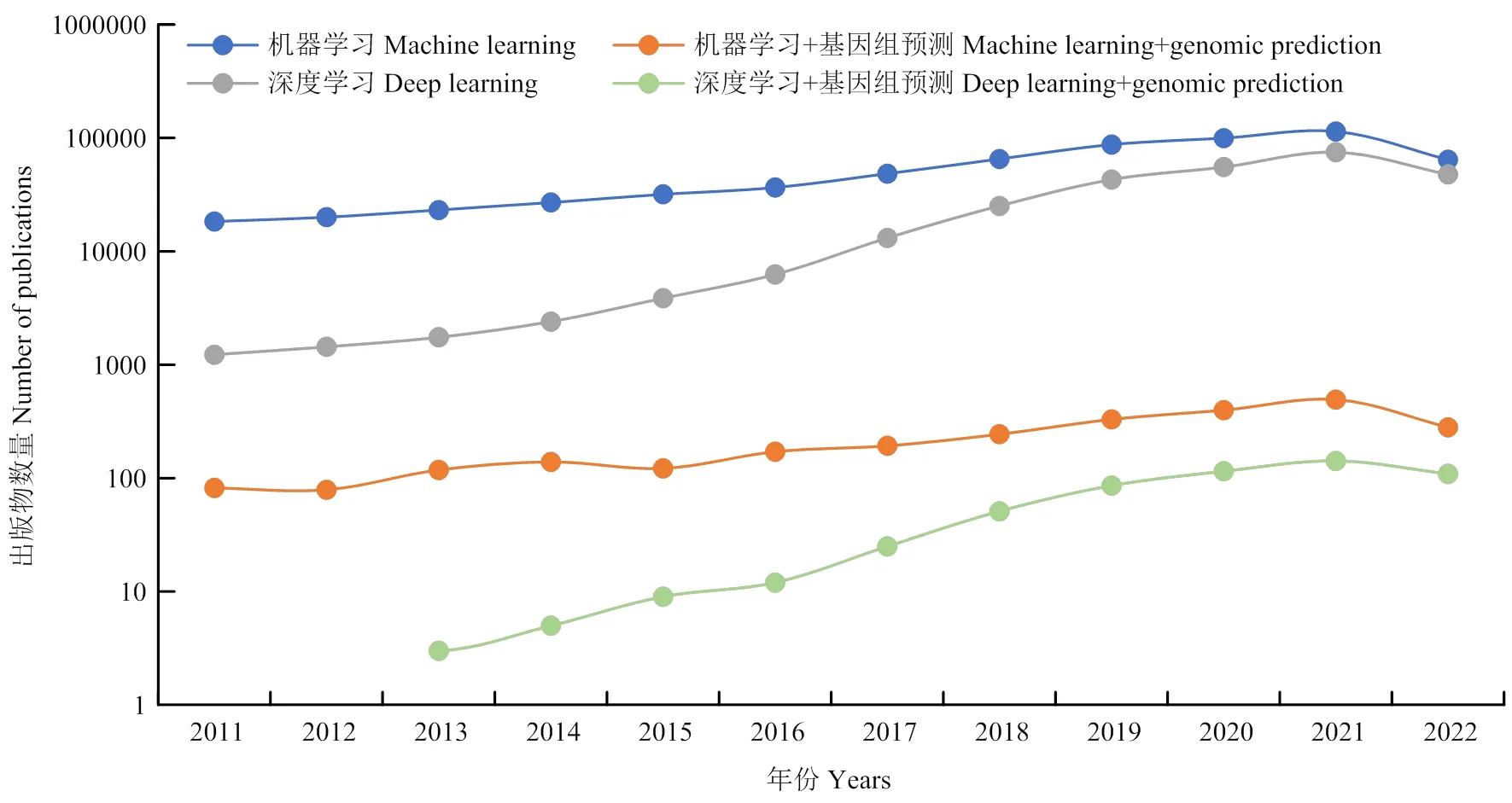
图2 2011-2022年每年发表机器学习相关出版物数量
SVR和KRR都通过核技巧来学习非线性函数,两者的区别在于损失函数不同。MOSER等[43]利用1 945头奶牛对非参数化的SVR和参数化的BayesR、rrBLUP和最小二乘回归对蛋白质百分比和利润指数进行选择,结果表明SVR提供了最高的准确性。LONG等[28]使用ε-SVR、最小二乘SVR与BayesLASSO分别选择了奶牛产奶量和小麦产量,结果表明在奶牛群体中径向基核函数SVR比线性核函数SVR的性能略好,在小麦中ε-SVR相关性则提高了17.5%。径向基核函数比线性核函数具有更好的选择性能,并且在表型可能受到非加性标记效应影响的情况下,其优越性更为明显。与参数化方法一样,机器学习不同算法的效果会因动物和性状的数据特征而异。ALVES等[37]对Nellore牛生殖性状进行基因组选择,发现SVR是Nellore牛生殖性状基因组选择的合适方法,同时说明SVR模型中的最佳核函数参数是特征依赖的。
受到机器学习算法、单核苷酸多态性(single nuclear polymorphism, SNP)加权和混合线性模型的启发,YIN等[36]提出了一种结合交叉验证、多元回归和网格搜索的KAML算法,拟将选择准确性与计算效率相结合。利用模拟和真实数据集进行评估,结果表明KAML算法具有比GBLUP和BayesR更高的选择准确性。
3.2 深度学习的应用
深度学习灵感源自人类大脑,是机器学习的重要分支。因由连接神经元的多个层组成,故又称为深度神经网络[44]。常见的典型深度学习算法有卷积神经网络(convolutional neural networks, CNN)、循环神经网络(Recurrent Neural Network, RNN)和生成对抗网络(generative Adversarial Networks, GANs)等。深度学习覆盖范围广、适应性好、上限高,但是计算量大,便携性差,对硬件要求高。其在基因组选择中的应用在过去十年内逐渐增多,在Web of science网站检索关键词“深度学习+基因组选择”可获得2012—2022年之间深度学习在基因组选择的相关出版物达到554份。而搜索“机器学习+基因组选择”在10年内的结果则是高达1 648份。
基于深度学习的人工智能模型如今代表着基因组学研究中进行功能预测的最新技术水平。NOVAKOVSKY等在综述中回顾了可解释人工智能这一新兴领域的进展,并探讨了关于深度学习方法如何用于调控基因组学的应用[45]。CAMACHO等[29]使用RBFNN方法进行基因组选择发现其选择效果与RKHS回归和BayesLASSO相当,所分析的模拟数据结果表明添加冗余选择变量可能会对非线性回归模型的选择准确性产生不利影响。TUSELL等[30]比较了参数化方法GBLUP、BayesR、BayesLASSO和非参数化方法RKHS回归、RBFNN、BRNN对猪产仔数性状的选择效果,结果表明两者效果相差不大。WALDMANN等[34]提出了一个ABNN模型,利用模拟和真实的猪数据集进行选择,结果表明ABNN显示出比GBLUP和BayesLASSO更好的选择准确性。ABDOLLAHI等[35]比较了两种深度学习方法(MLP和CNN)、两种集成学习方法(RF和GB)和两种参数方法(GBLUP和BayesB)的选择性能,发现MLP和CNN在公牛数据集中的选择效果最低,这可能是因为相对于参数化方法,深度学习的优势取决于控制性状的位点数量和样本大小。目前,深度学习在动植物育种中都取得了一定进展[46-48],更多的应用原理和实例可在MIGUEL等的综述中进行查看[49]。
3.3 集成学习的应用
集成学习算法是指将不同的学习器融合在一起,得到一个较强的监督模型。其基本思想是不同的学习器之间相互纠正错误以达到准确性的提升。目前主流的集成学习有Boosting方法、Bagging方法和Stacking算法。Boosting方法以Adaboosting、提升树和XGBoost为代表,每一次迭代时训练集的选择与前面各轮的学习结果有关,且每次是通过更新各个样本权重的方式来改变数据分布。
Adaboost.RT算法最初由SHRESTHA等[50]开发,在处理回归问题上非常有优势。LIANG等[26]利用集成了SVR、KRR和RF的集成学习算法Adaboost.RT来选择中国西门塔尔肉牛胴体重量、活重和眼肌面积3种经济性状的GEBV,与单个学习器和GBLUP进行比较后发现Adaboost.RT的可靠性和稳定性比其他方法都高。同时,LIANG等[38]构建了一个堆叠集成学习框架(SELF),通过3个数据集的比较分析发现SELF在所有方法中几乎表现得最好。WANG等[39]利用SVR、KRR、RF和Adaboost. R2等4种机器学习回归方法对中国大白猪群体进行了基因组预测,结果表明基于KRR的Adaboost.R2方法一直表现良好,并且最佳超参数对机器学习方法很有用。因此,集成学习在动物基因组选择中具有一定的潜力。
除了使用基因型数据进行预测,还有部分研究利用机器学习整合多组学数据以提高动物复杂性状的选择准确性。FABIO等[51]使用黑腹果蝇群体200自交系中3个数量性状的基因组数据、RNA转录组数据和表型数据,利用机器学习整合不同的信息来源提高了选择准确性。FU等[52]提出了一个集成了多组学信息的CNN模型以优先考虑目标性状的候选基因,并提出了包含已发表的猪多组学数据的ISwine在线知识库。
4 案例分析
选用公共数据库的荷斯坦奶牛数据集进行不同基因组选择方法的比较,进行案例分析。由5 024头公牛组成的德国荷斯坦牛基因组预测群体用于本研究。所有公牛都用Illumina Bovine SNP50珠芯片进行基因分型[53]。质量控制后,剩下42 551个SNP供以进一步分析。每个公牛都有3个性状:产奶量(milk yield, MKG)、乳脂百分比(milk fat percentage, FPRO)和体细胞评分(somatic cell score, SCS)。更多关于该群体的详细信息可在参考文献[54]中查看。采用间接法BayesB、直接法GBLUP和机器学习的KRR和SVR等4种方法,采用5×5倍交叉验证获得GEBV平均值和标准差。评价指标有两个,第一个是准确性,即真实值与预测值之间的皮尔逊相关系数。第二个是误差,即真实值与预测值之间的均方根误差。
得到结果如表2所示,表中评价指标格式为:准确性(均方根误差)。在性状MKG当中,4种方法获得的准确性和误差都没有显著性差异。在性状FPR中,BayesB方法的准确性为0.860,明显优于其他3种方法,AN等[24]的研究中也得到相似的结果。在性状SCS中,机器学习方法KRR和SVR的准确性高于BayesB和GBULP,且均方误也小于它们。本例中仅进行了不同方法性能的比较,对于结果的解读,仍需进一步的分析,如性状的遗传背景和方法的适用情况等。
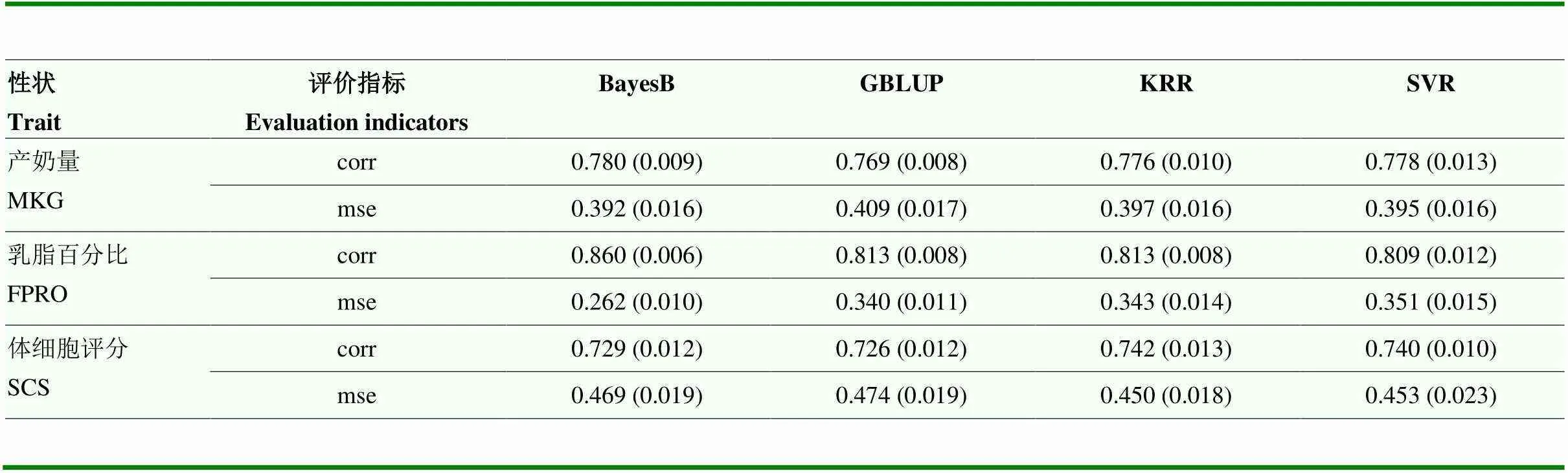
表2 不同方法对奶牛3个性状的基因组估计准确性和均方误比较
corr: Correlation; mse: Mean squared error
5 挑战
尽管机器学习具有很多优点,但其在对动物复杂性状的基因组育种值估计时仍存在一些挑战。
5.1 模型的可解释性
了解模型处理数据的过程能有助于模型和特征的优化。可解释性是指人类能够理解决策原因的程度,模型可解释性则指对模型内部机制的理解以及对模型结果的理解[55]。当机器学习模型的可解释性越高,模型的决策和预测就可以被人理解。一些复杂的模型通常能产生更好的性能,如集成模型和深度学习,但它们的运作原理往往很难被解释清楚[56]。如果不能清晰地解析模型,改进模型和提高估计准确性也会存在困难。性状遗传结构复杂多样,目前还没有一种模型能广泛适用于所有性状[57]。如何选择正确的模型并解读数据分析的过程,是目前研究需要克服的难点之一。
5.2 数据噪声和分布差异
数据噪声是指一个测量变量中的随机错误或偏差,即数据样本中对拟合模型有干扰的信息。机器学习的数据噪声来源于数据的异质性、稀疏性和异常值。常见的噪声数据处理方法包括分箱、聚类和回归等。在基因组选择中,特征受到许多具有微小效应的位点影响时,机器学习往往表现比较糟糕[32]。同时,生物结构、生物环境和批次效应等都会造成基因组学数据的分布差异。
5.3 过拟合
过拟合是指模型在训练集上表现好,但是在测试集上表现不好,模型泛化性能差。模型太复杂、数据噪声、数据量小或数据缺乏代表性等都有可能引起模型过拟合。可以尝试通过以下方法进行改善:(1)增加样本数或样本多样性;(2)剔除噪声数据或改用对噪声不敏感的模型;(3)考虑使用加权或标准化指标来衡量机器学习的表现,如标准化余弦相似度;(4)合并代表性不足的样本[57-58]。在机器学习实际操作中,正则化、提前结束、超参数优化等手段都可用来帮助解决过拟合问题。
5.4 大变量小样本
在基因组选择问题中,大变量小样本是指基因型标记数目远远大于样本观察值。这种现象容易使高维空间包含多余的特征,从而误导模型的训练[59]。机器学习算法通过假设映射函数的数据和结构来解决这个问题,但也因此增大了偏差。所以,研究者在训练模型之前通常采用特征提取和特征选择等降维方法来减少数据的维空间[60]。该问题又会引起计算机计算和储存上的困难,因为大多数的机器学习算法都需要大量的内存来运行和保存结果。这可以使用专用的图形处理单元(graphics processing unit, GPU)和云计算平台进行改善。
5.5 选择模型和超参数
不同模型采用的算法不同,因此擅长处理的数据类型也不同。根据数据结构和数据特征选择合适的模型极为重要。许多文献综述了各种机器学习算法的优势和不足[61-64],以供选择模型时进行参考。训练模型时需要选择合适的超参数,即调参。调参可以权衡模型的偏差和方差,从而提高模型效果及性能。常用的调参方法有手动调参、网格搜索和贝叶斯优化等。目前,一些自动化机器学习平台和工具也可供搜索最佳模型和最佳超参数[65-67]。
5.6 使用建议
由于以上这些挑战,利用机器学习进行选择时需要谨慎处理每一个环节。训练模型前,一方面需要增加数据的样本量和多样性,另一方面则需要尽量剔除造成数据噪声的因素。训练时应根据数据结构选择合适的模型,并不是越复杂越好。默认参数在大多数时候都有较好的表现,但为了获得更好的选择效果,调参工作不建议省略。为避免过拟合,训练结束后应使用交叉验证来评估模型的选择准确性。设置随机种子以便训练过程能够被重复。最后,如果出现了极好或者极差的情况,请认真检查程序,因为这种情况几乎没有在文献中出现过。
6 结语及展望
经典的基因组选择方法在生产实践中发挥了巨大的效力,但在理论和应用方面仍面临一些挑战。由于品种间遗传背景不同,开展基因组选择时跨品种预测准确性难以保证。同时,传统基因组选择方法仅利用到基因组信息,对于多组学信息的利用并不充分。如何将多组学信息进行整合,以提高选择准确度也是需要解决的问题。目前,个体分型主要采用芯片技术。而由于标记密度低,导致该技术依赖于基因组连锁不平衡。测序技术可以解决低密度问题,并且可能实现跨品种预测。所以,测序技术的出现将成为全基因组选择新时代的转折点。但是测序技术成本高、速度慢,对计算资源的配置要求较高。因此,如何快速并有效地储存、处理及分析数据是测序技术应用于全基因组育种的重要挑战[68]。
目前为止,机器学习在动物基因组选择中的应用大多数是处理回归问题,以二元表型的形式完成。研究进展表明,采用机器学习进行动物复杂性状的基因组选择,不仅弥补了传统选择方法的弊端,还能捕获数据之间的非线性关系。众多的优势和功能使机器学习能够很好选择动物个体的遗传价值,这为选择准确性的提升提供了很好的突破契机。但由于数据噪声和分布差异等问题,机器学习算法的表现仍不稳定。机器学习基于算法实现,算法又依赖于超参数的选择。一般情况下,默认超参数都能有不错的表现,所以调参需要谨慎进行。文末给出了一些使用建议,以期有一定的帮助。要将机器学习各类算法的最强作用发挥在基因组选择当中,应从套用运算转为启发学习,思考如何开发在动物品种中具有高准确性和运算速度的选择方法。
除了利用基因组信息外,机器学习还可以在此基础上整合转录组信息、代谢组信息等,以提高育种值估计的准确性。目前这方面的相关研究很少,但是部分已有的研究表明机器学习在多组学数据的整合中很有优势。随着高通量测序和各种分子手段的快速发展,各种组学数据量逐渐庞大起来,如何将这些组学数据进行科学又恰当的整合以提高选择准确性也成为了目前动物育种工作中值得思考的问题之一。
[1] MEUWISSEN T H E, HAYES B J, GODDARD M E. Prediction of total genetic value using genome-wide dense marker maps. Genetics, 2001, 157(4): 1819-1829.
[2] GODDARD M. Genomic selection: prediction of accuracy and maximisation of long term response. Genetica, 2009, 136(2): 245-257.
[3] WELLER J I, EZRA E, RON M.A perspective on the future of genomic selection in dairy cattle. Journal of Dairy Science, 2017, 100(11): 8633-8644.
[4] HABIER D, FERNANDO R L, KIZILKAYA K, GARRICK D J. Extension of the Bayesian alphabet for genomic selection. BMC Bioinformatics, 2011, 12: 186.
[5] MEHER P K, RUSTGI S, KUMAR A. Performance of Bayesian and BLUP alphabets for genomic prediction: analysis, comparison and results. Heredity, 2022, 128(6): 519-530.
[6] LOPES F B, BALDI F, PASSAFARO T L, BRUNES L C, COSTA M F O, EIFERT E C, NARCISO M G, ROSA G J M, LOBO R B, MAGNABOSCO C U. Genome-enabled prediction of meat and carcass traits using Bayesian regression, single-step genomic best linear unbiased prediction and blending methods in Nelore cattle. Animal, 2021, 15(1): 100006.
[7] GUALDRÓN DUARTE J L, GORI A S, HUBIN X, LOURENCO D, CHARLIER C, MISZTAL I, DRUET T. Performances of Adaptive MultiBLUP, Bayesian regressions, and weighted-GBLUP approaches for genomic predictions in Belgian Blue beef cattle. BMC Genomics, 2020, 21(1): 545.
[8] BISHOP C, NASRABADI N. Pattern recognition and machine learning. New York: Springer New York, 2006.
[9] JORDAN M I, MITCHELL T M. Machine learning: trends, perspectives, and prospects. Science, 2015, 349(6245): 255-260.
[10] MURPHY K P. Machine learning: a probabilistic perspective. Cambridge, Mass.: MIT Press, 2012.
[11] ZHANG X H, CHEN S Y, LAI K M, CHEN Z M, WAN J X, XU Y F. Machine learning for the prediction of acute kidney injury in critical care patients with acute cerebrovascular disease. Renal Failure, 2022, 44(1): 43-53.
[12] ARAÚJO D C, VELOSO A A, BORGES K B G, DAS GRAÇAS CARVALHO M. Prognosing the risk of COVID-19 death through a machine learning-based routine blood panel: a retrospective study in Brazil. International Journal of Medical Informatics, 2022, 165: 104835.
[13] BAE S, SAMUELS J A, FLYNN J T, MITSNEFES M M, FURTH S L, WARADY B A, NG D K. Machine learning-based prediction of masked hypertension among children with chronic kidney disease. Hypertension, 2022, 79(9): 2105-2113.
[14] REEL P S, REEL S, PEARSON E, TRUCCO E, JEFFERSON E. Using machine learning approaches for multi-omics data analysis: a review. Biotechnology Advances, 2021, 49: 107739.
[15] YAN J, WANG X F. Unsupervised and semi-supervised learning: the next frontier in machine learning for plant systems biology. The Plant Journal, 2022, 111(6): 1527-1538.
[16] TU K L, WEN S Z, CHENG Y, XU Y N, PAN T, HOU H N, GU R L, WANG J H, WANG F G, SUN Q. A model for genuineness detection in genetically and phenotypically similar maize variety seeds based on hyperspectral imaging and machine learning. Plant Methods, 2022, 18(1): 81.
[17] ESPOSITO S, RUGGIERI V, TRIPODI P. Editorial: machine learning for big data analysis: applications in plant breeding and genomics. Frontiers in Genetics, 2022, 13: 916462.
[18] ZHANG Z, ZHANG Q, DING X D. Advances in genomic selection in domestic animals. Chinese Science Bulletin, 2011, 56(25): 2655-2663.
[19] DAETWYLER H D, PONG-WONG R, VILLANUEVA B, WOOLLIAMS J A. The impact of genetic architecture on genome-wide evaluation methods. Genetics, 2010, 185(3): 1021-1031.
[20] KARIMI K, SARGOLZAEI M, PLASTOW G S, WANG Z Q, MIAR Y. Opportunities for genomic selection in American mink: a simulation study. PLoS One, 2019, 14(3): e0213873.
[21] MUIR W M. Comparison of genomic and traditional BLUP-estimated breeding value accuracy and selection response under alternative trait and genomic parameters. Journal of Animal Breeding and Genetics = Zeitschrift Fur Tierzuchtung Und Zuchtungsbiologie, 2007, 124(6): 342-355.
[22] SONG H L, ZHANG J X, ZHANG Q, DING X D. Using different single-step strategies to improve the efficiency of genomic prediction on body measurement traits in pig. Frontiers in Genetics, 2018, 9: 730.
[23] SAMUEL A L. Some studies in machine learning using the game of checkers. IBM Journal of Research and Development, 1959, 3(3): 210-229.
[24] AN B X, LIANG M, CHANG T P, DUAN X H, DU L L, XU L Y, ZHANG L P, GAO X, LI J Y, GAO H J. KCRR: a nonlinear machine learning with a modified genomic similarity matrix improved the genomic prediction efficiency. Briefings in Bioinformatics, 2021, 22(6): bbab132.
[25] BREIMAN L. Random forests. Machine Language, 2001, 45(1): 5-32.
[26] LIANG M, MIAO J, WANG X Q, CHANG T P, AN B X, DUAN X H, XU L Y, GAO X, ZHANG L P, LI J Y, GAO H J. Application of ensemble learning to genomic selection in Chinese Simmental beef cattle. Journal of Animal Breeding and Genetics = Zeitschrift Fur Tierzuchtung Und Zuchtungsbiologie, 2021, 138(3): 291-299.
[27] GONZÁLEZ-RECIO O, GIANOLA D, LONG N Y, WEIGEL K A, ROSA G J M, AVENDAÑO S. Nonparametric methods for incorporating genomic information into genetic evaluations: an application to mortality in broilers. Genetics, 2008, 178(4): 2305-2313.
[28] LONG N Y, GIANOLA D, ROSA G J M, WEIGEL K A. Application of support vector regression to genome-assisted prediction of quantitative traits. TAG Theoretical and Applied Genetics Theoretische Und Angewandte Genetik, 2011, 123(7): 1065-1074.
[29] GONZÁLEZ-CAMACHO J M, DE LOS CAMPOS G, PÉREZ P, GIANOLA D, CAIRNS J E, MAHUKU G, BABU R, CROSSA J. Genome-enabled prediction of genetic values using radial basis function neural networks. TAG Theoretical and Applied Genetics Theoretische Und Angewandte Genetik, 2012, 125(4): 759-771.
[30] TUSELL L, PÉREZ-RODRÍGUEZ P, FORNI S, WU X L, GIANOLA D. Genome-enabled methods for predicting litter size in pigs: a comparison. Animal, 2013, 7(11): 1739-1749.
[31] HE J R, DING L X, JIANG L, MA L. Kernel ridge regression classification. 2014 International Joint Conference on Neural Networks (IJCNN). July 6-11, 2014, Beijing, China. IEEE, 2014: 2263-2267.
[32] NADERI S, YIN T, KÖNIG S. Random forest estimation of genomic breeding values for disease susceptibility over different disease incidences and genomic architectures in simulated cow calibration groups. Journal of Dairy Science, 2016, 99(9): 7261-7273.
[33] GHAFOURI-KESBI F, RAHIMI-MIANJI G, HONARVAR M, NEJATI- JAVAREMI A. Predictive ability of Random Forests, Boosting, Support Vector Machines and Genomic Best Linear Unbiased Prediction in different scenarios of genomic evaluation. Animal Production Science, 2017, 57(2): 229.
[34] WALDMANN P. Approximate Bayesian neural networks in genomic prediction. Genetics Selection Evolution, 2018, 50(1): 1-9.
[35] ABDOLLAHI-ARPANAHI R, GIANOLA D, PEÑAGARICANO F. Deep learning versus parametric and ensemble methods for genomic prediction of complex phenotypes. Genetics, Selection, Evolution, 2020, 52(1): 12.
[36] YIN L L, ZHANG H H, ZHOU X, YUAN X H, ZHAO S H, LI X Y, LIU X L. KAML: improving genomic prediction accuracy of complex traits using machine learning determined parameters. Genome Biology, 2020, 21(1): 146.
[37] ALVES A A C, ESPIGOLAN R, BRESOLIN T, COSTA R M, FERNANDES JÚNIOR G A, VENTURA R V, CARVALHEIRO R, ALBUQUERQUE L G. Genome-enabled prediction of reproductive traits in Nellore cattle using parametric models and machine learning methods. Animal Genetics, 2021, 52(1): 32-46.
[38] LIANG M, CHANG T P, AN B X, DUAN X H, DU L L, WANG X Q, MIAO J, XU L Y, GAO X, ZHANG L P, LI J Y, GAO H J. A stacking ensemble learning framework for genomic prediction. Frontiers in Genetics, 2021, 12: 600040.
[39] WANG X, SHI S L, WANG G J, LUO W X, WEI X, QIU A, LUO F, DING X D. Using machine learning to improve the accuracy of genomic prediction of reproduction traits in pigs. Journal of Animal Science and Biotechnology, 2022, 13(1): 60.
[40] SAUNDERS C, GAMMERMAN A, VOVK V. Ridge regression learning algorithm in dual variables. Proceedings of the Fifteenth International Conference on Machine Learning. New York: ACM, 1998: 515-521.
[41] KAR A, BHATTACHARJEE D, BASU D K, NASIPURI M, KUNDU M. Human face recognition using Gabor based kernel entropy component analysis. International Journal of Computer Vision and Image Processing, 2012, 2(3): 1-20.
[42] LU Y W, FANG X Z, XIE B L. Kernel linear regression for face recognition. Neural Computing and Applications, 2014, 24(7/8): 1843-1849.
[43] MOSER G, TIER B, CRUMP R E, KHATKAR M S, RAADSMA H W. A comparison of five methods to predict genomic breeding values of dairy bulls from genome-wide SNP markers. Genetics, Selection, Evolution, 2009, 41(1): 56.
[44] BALKENENDE L, TEUWEN J, MANN R M. Application of deep learning in breast cancer imaging. Seminars in Nuclear Medicine, 2022, 52(5): 584-596.
[45] NOVAKOVSKY G, DEXTER N, LIBBRECHT M W, WASSERMAN W W, MOSTAFAVI S. Obtaining genetics insights from deep learning via explainable artificial intelligence. Nature Reviews Genetics, 2023, 24(2): 125-137.
[46] KHAKI S, WANG L Z. Crop yield prediction using deep neural networks. Frontiers in Plant Science, 2019, 10: 621.
[47] MONTESINOS-LÓPEZ O A, MARTÍN-VALLEJO J, CROSSA J, GIANOLA D, HERNÁNDEZ-SUÁREZ C M, MONTESINOS- LÓPEZ A, JULIANA P, SINGH R. A benchmarking between deep learning, support vector machine and Bayesian threshold best linear unbiased prediction for predicting ordinal traits in plant breeding. G3 Genes|Genomes|Genetics, 2019, 9(2): 601-618.
[48] MONTESINOS-LÓPEZ A, MONTESINOS-LÓPEZ O A, GIANOLA D, CROSSA J, HERNÁNDEZ-SUÁREZ C M. Multi-environment genomic prediction of plant traits using deep learners with dense architecture. G3, 2018, 8(12): 3813-3828.
[49] PÉREZ-ENCISO M, ZINGARETTI L M. A guide for using deep learning for complex trait genomic prediction. Genes, 2019, 10(7): 553.
[50] SHRESTHA D L, SOLOMATINE D P. Experiments with AdaBoost. RT, an improved boosting scheme for regression. Neural Computation, 2006, 18(7): 1678-1710.
[51] FABIO M, WEN H, PETER S, CHRISTIAN M, MACKAY TRUDY F C. Leveraging multiple layers of data to predictcomplex traits. G3 (Bethesda, Md), 2020, 10(12): 4599-4613.
[52] FU Y H, XU J Y, TANG Z S, WANG L, YIN D, FAN Y, ZHANG D D, DENG F, ZHANG Y P, ZHANG H H, WANG H Y, XING W H, YIN L L, ZHU S L, ZHU M J, YU M, LI X Y, LIU X L, YUAN X H, ZHAO S H. A gene prioritization method based on a swine multi- omics knowledgebase and a deep learning model. Communications Biology, 2020, 3(1): 502.
[53] MATUKUMALLI L K, LAWLEY C T, SCHNABEL R D, TAYLOR J F, ALLAN M F, HEATON M P, O'CONNELL J, MOORE S S, SMITH T P L, SONSTEGARD T S, VAN TASSELL C P. Development and characterization of a high density SNP genotyping assay for cattle. PLoS One, 2009, 4(4): e5350.
[54] ZHANG Z, ERBE M, HE J L, OBER U, GAO N, ZHANG H, SIMIANER H, LI J Q. Accuracy of whole-genome prediction using a genetic architecture-enhanced variance-covariance matrix. G3, 2015, 5(4): 615-627.
[55] RIBEIRO M T, SINGH S, GUESTRIN C. Model-agnostic interpretability of machine learning. 2016: arXiv: 1606.05386. https://arxiv.org/abs/ 1606.05386.
[56] VELLIDO A, MARTÍN-GUERRERO J, LISBOA P. Making machine learning models interpretable. Proceedings of the ESANN, F, 2012.
[57] ZHANG Q X, ZHANG L N, LIU F, LIU X D, LIU X L, ZHAO S H, ZHU M J. A study of genomic selection on porcine hematological traits using GBLUP and penalized regression methods. Acta Veterinaria et Zootechnica Sinica, 2017, 48(12): 2258-2267.
[58] HE H B, BAI Y, GARCIA E A, LI S T. ADASYN: adaptive synthetic sampling approach for imbalanced learning. 2008 IEEE International Joint Conference on Neural Networks (IEEE World Congress on Computational Intelligence). June 1-8, 2008, Hong Kong, China. IEEE, 2008: 1322-1328.
[59] JAMES G, WITTEN D, HASTIE T, TIBSHIRANI R. An introduction to statistical learning: with applications in R. 2nd ed. Berlin: Springer. 2013.
[60] STANCZYK U, JAIN L C. Feature selection for data and pattern recognition preface. Studies-in-Computational-Intelligence, 2015, 584: 355.
[61] AMANCIO D R, COMIN C H, CASANOVA D, TRAVIESO G, BRUNO O M, RODRIGUES F A, DA FONTOURA COSTA L. A systematic comparison of supervised classifiers. PLoS One, 2014, 9(4): e94137.
[62] LÓPEZ PINEDA A, YE Y, VISWESWARAN S, COOPER G F, WAGNER M M, TSUI F. Comparison of machine learning classifiers for influenza detection from emergency department free-text reports. Journal of Biomedical Informatics, 2015, 58: 60-69.
[63] SAKR S, ELSHAWI R, AHMED A M, QURESHI W T, BRAWNER C A, KETEYIAN S J, BLAHA M J, AL-MALLAH M H. Comparison of machine learning techniques to predict all-cause mortality using fitness data: the Henry ford exercIse testing (FIT) project. BMC Medical Informatics and Decision Making, 2017, 17(1): 174.
[64] UDDIN S, KHAN A, HOSSAIN M E, ALI MONI M. Comparing different supervised machine learning algorithms for disease prediction. BMC Medical Informatics and Decision Making, 2019, 19(1): 281.
[65] FEURER M, KLEIN A, EGGENSPERGER K, Springenberg J T, Blum M, Hutter F. Efficient and robust automated machine learning. Advances in neural information processing systems, 2015, 28: 2755-2763.
[66] OLSON R S, SIPPER M, CAVA W L, Tartarone S, Vitale S, Fu W, Patryk O, Ryan J U, Holmes J H, Moore J H. A system for accessible artificial intelligence. Genetic programming theory and practice XV. Springer. 2018: 121-134.
[67] WARING J, LINDVALL C, UMETON R. Automated machine learning: review of the state-of-the-art and opportunities for healthcare. Artificial Intelligence in Medicine, 2020, 104: 101822.
[68] YIN L L, MA Y L, XIANG T, ZHU M J, YU M, LI X Y, LIU X L, ZHAO S L. The progress and prospect of genomic selection models. Acta Veterinaria et Zootechnica Sinica, 2019, 50(2): 233-242.
Research Progress on Machine Learning for Genomic Selection in Animals
LI MianYan, WANG LiXian, ZHAO FuPing
Key Laboratory of Animal Genetics Breeding andReproduction (Poultry), Ministry of Agriculture, Institute of Animal Sciences, Chinese Academy of Agricultural Sciences, Beijing 100193
Genomic selection is defined as using the molecular marker information that covered the whole genome to estimate individual’s breeding values. Using genome information can avoid many problems caused by pedigree errors so as to improve selection accuracy and shorten breeding generation intervals. According to different statistical models, methods of estimated genomic breeding value (GEBV) can be divided into based on BLUP (best linear unbiased prediction) theory, based on Bayesian theory and others. At present, GBLUP and its improved method ssGBLUP have been widely employed. Accuracy is the most used evaluation metric for genomic selection models, which is to evaluate the similarity between the true value and the estimated value. The factors that affect the accuracy can be reflected from the model, which can be divided into controllable factors and uncontrollable factors. Traditional genomic selection methods have promoted the rapid development of animal breeding, but these methods are currently facing many challenges such as multi-population, multi-omics, and computing. What’s more, they cannot capture the nonlinear relationship between high-dimensional genomic data. As a branch of artificial intelligence, machine learning is very close to biological mastery of natural language processing. Machine learning extracts features from data and automatically summarizes the rules and use to make predictions for new data. For genomic information, machine learning does not require distribution assumptions, and all marker information can be considered in the model. Compared with traditional genomic selection methods, machine learning can more easily capture complex relationships between genotypes, phenotypes, and the environment. Therefore, machine learning has certain advantages in animal genomic selection. According to the amount and type of supervision received during training, machine learning can be classified into supervised learning, unsupervised learning, semi-supervised learning, and reinforcement learning. The main difference is whether the input data is labeled. The machine learning methods currently applied in animal genomic selection are all supervised learning. Supervised learning can handle both classification and regression problems, requiring the algorithm to be provided with labeled data and the desired output. In recent years, the application of machine learning in animal genomic selection has been increasing, especially in dairy and beef cattle. In this review, machine learning algorithms are divided into three categories: single algorithm, ensemble algorithm and deep learning, and their research progress in animal genomic selection were summarized. The most used single algorithms are KRR and SVR, both of which use kernel tricks to learn nonlinear functions and map data to higher-dimensional kernel spaces in the original space. Currently commonly used kernel functions are linear kernel, cosine kernel, Gaussian kernel, and polynomial kernel. Deep learning, also known as a deep neural network, consists of multiple layers of connected neurons. An ensemble learning algorithm refers to fusing different learners together to obtain a stronger supervised model. In the past decade, the related literature on machine learning and deep learning has shown exponential growth. And its application in genomic selection is also gradually increasing. Although machine learning has obvious advantages in some aspects, it still faces many challenges in estimating the genetic breeding value of complex traits in animals. The interpretability of some models is low, which is not conducive to the adjustment of data, parameters, and features. Data heterogeneity, sparsity, and outliers can also cause data noise for machine learning. There are also problems such as overfitting, large marks and small samples, and parameter adjustment. Therefore, each step needs to be handled carefully while training the model. This paper introduced the traditional methods of genomic selection and the problems they face, the concept and classification of machine learning. We discussed the research progress and current challenges of machine learning in animal genomic selection. A Case and some application suggestions were given to provide a certain reference for the application of machine learning in animal genomic selection.
machine learning; deep learning; genomic selection; animal breeding
10.3864/j.issn.0578-1752.2023.18.015
2022-09-17;
2023-06-28
国家自然科学基金面上项目(32172702)、国家重点研发计划(2021YFD130110203)、中国农业科学院科技创新工程(ASTIP-IAS02)、国家生猪产业技术体系(CARS-35)
李棉燕,Tel:15305169095;E-mail:mianyanli@outlook.com。王立贤,E-mail:iaswlx@263.net。李棉燕和王立贤为同等贡献作者。通信作者赵福平,E-mail:zhaofuping@caas.cn
(责任编辑 林鉴非)
