汉滩病毒糖蛋白Gn的T细胞表位预测及其跨种属验证
2023-10-18孙报增秦聪聪张俊琦王永凯张文标刘瑞波白天原张志辉张宇丝杨琨姜东伯
孙报增,秦聪聪,张俊琦,王永凯,张文标,刘瑞波,白天原,张志辉,张宇丝,杨琨,2,3,姜东伯,2,4*
1空军军医大学基础医学院免疫学教研室,陕西 西安 710032;2空军军医大学基础医学院生物武器损伤防治药物重点实验室,陕西 西安 710032;3空军医科大学唐都医院风湿科,陕西 西安 710032;4空军军医大学基础医学院微生物学教研室,陕西 西安 710032
汉坦病毒属(Hantavirus)属于布尼亚病毒科,包括汉滩病毒(Hantaan virus,HTNV)、多布拉-贝尔格莱德病毒、普马拉病毒和首尔病毒等,其在欧亚大陆主要引起肾综合征出血热(hemorrhagic fever with renal syndrome,HFRS)[1-2]。HFRS 以鼠类等啮齿类动物为主要传染源,表现为发热、出血、急性肾功能损伤等。我国是HTNV 所致HFRS 的主要疫源地,占全球感染病例的70%~90%,发病率和病死率均较高[3]。HTNV 为三股单负链RNA 病毒,由S、M 和L片段组成,分别编码RNA 依赖性RNA 聚合酶(RdRp)、糖蛋白(GP)及核衣壳蛋白(NP)[4]。其中GP构成HTNV 的主要抗原,同时可被加工分解成包膜糖蛋白-N 末端(Gn)及包膜糖蛋白-C 末端(Gc),构成病毒吸附蛋白,能够识别并结合细胞表面受体,刺激机体产生中和抗体,在病毒对机体细胞的侵袭及感染过程中发挥重要作用,因此可作为HFRS 疫苗研究的潜在靶点[5]。然而,在现有的表位研究中,筛选鉴定的限制性表位不全面,针对表位的研究比较单一,不能全面揭示表位特性。同时,寻找人类的表位较困难,难以开展相关的实验,而前期研究发现,采用计算机预测的方法具有全面准确、经济效益高的优势[6-8]。因此,本研究对Gn 进行了系统分析,以探索主要组织相容性复合体(MHC)-Ⅰ、Ⅱ类表位免疫反应性的相关规律,指导新型HFRS 表位疫苗的研发,预防汉坦病毒的跨物种传播风险。
1 材料与方法
1.1 Gn 蛋白序列获取 从UniProt 数据库(https://www.uniprot.org/)检索HTNV 76-118 株Gn 蛋白序列(PRO_0000036816)[9],氨基酸序列从aa1至aa649。
1.2 Gn蛋白表位亲和力预测
1.2.1 预测工具的选取 利用5 种预测算法,包括IEDB(http://tools.iedb.org/mhci/)[10]、 SMMPMBEC(https://github.com/ykimbiology/smmpmbec)[11]、NetMHCpan 4.1(https://services.healthtech.dtu.dk/service.php?NetMHCpan-4.1)[12]、SYFPEITHI(http://www.syfpeithi.de/bin/MHCServer.dll/EpitopePrediction.htm)[13]、Rankpep(http://imed.med.ucm.es/Tools/rankpep.html)[14]全长无偏倚地获得高亲和力的表位候选肽,并对HTNV Gc 蛋白序列进行序列寡肽分割,计算MHC-Ⅰ类分子与9 肽表位之间的亲和力;利用4 种预测算法,包括IEDB、NetMHC Ⅱpan3.2(https://services.healthtech.dtu.dk/service.php? NetMHCIIpan-3.2)[12]、SYFPEITHI、Rankpep来预测MHC-Ⅱ类分子与15肽表位之间的亲和力。
1.2.2 MHC-Ⅰ类限制性表位的预测与筛选 对IEDB推荐的人类白细胞抗原(HLA)-Ⅰ类亚型等位基因及Allel Frequency[15]网站(http://www.allelefrequencies.net/)中各地区基因频率>10%的HLA基因型进行亲和力分析,包括HLA-A1(-A*0101、HLA-A*2601、-A*3001、-A*3002)、HLA-A2(HLA-A*0201、-A*0203、-A*0206、-A*0207、-A*0212、-A*6802)、HLA-A3(HLA-A*0301、 -A*1101、 -A*3001、 -A*3101、 -A*3301、 -A*6801)、 HLA-A24(HLA-A*2301、 -A*2402、 -A*2407、-A*3201)、HLA-A*3303、HLA-A*3108、HLA-A*3401、HLA-B7(HLA-B*0702、-B*3501、-B*5101、-B*5301)、HLA-B8(HLA-B*0801)、 HLA-B*1301、 HLA-B*1405、HLA-B15(HLA-B*1501、-B*1502、-B*1506、-B*1512、-B*1521)、 HLA-B*1807、 HLA-B*3508、 HLA-B*3903、HLA-B44(HLA-B*4001、-B*4002、-B*4006、-B*4402、-B*4403、 -B*4404)、 HLA-B46(HLA-B*4601)、 HLAB50(HLA-B*5001)、 HLA-B*5110、 HLA-B*5201、HLA-B*5502、 HLA-B*5601、 HLA-B58(HLA-B*5701、-B*5801)。上述基因型覆盖了超过99.3% 人群的MHC-Ⅰ类亚型。以每种算法结果中前2%的预测表位为候选优势表位,至少在3 种预测算法结果中出现的表位作为优势表位[16]。
1.2.3 MHC-Ⅱ类限制性表位的预测与筛选 MHC-Ⅱ类分子包括4 个HLA-Ⅱ超家族:DRB1(DRB1*0101、DRB1*0301、DRB1*0304、DRB1*0401、DRB1*0405、DRB1*0407、DRB1*0412、DRB1*0701、DRB1*0704、DRB1*0802、DRB1*0803、DRB1*0901、DRB1*1101、DRB1*1104、DRB1*1201、DRB1*1202、DRB1*1301、DRB1*1302、DRB1*1304、DRB1*1401、DRB1*1402、DRB1*1408、DRB1*1409、DRB1*1501、DRB1*1502、DRB1*1503、DRB1*1602),DRB3/4/5(DRB3*0101、 DRB3*0202、 DRB4*0101、 DRB5*0101),DQA1/DQB1(DQA1*0501/DQB1*0201、 DQA1*0501/DQB1*0301、DQA1*0301/DQB1*0302、DQA1*0401/DQB1*0402、DQA1*0101/DQB1*0501、DQA1*0102/DQB1*0602),DPB1(DPB1*0101、DPB1*0201、DPB1*0401、DPB1*0402、DPB1*0501、DPB1*1401)。上述基因型覆盖了超过98%人群的MHC-Ⅱ类亚型,且在Allel Frequency 网站(http://www.allelefrequencies.net/)中各地区的基因型频率>10%。以每种算法结果中前2%的预测表位为候选优势表位,至少在2种预测算法结果中出现的表位作为优势表位。
1.3 HTNV Gn MHC-Ⅰ/Ⅱ类限制性表位的免疫原性分析 高亲和力肽不能充分诱导免疫应答,因此需对优势表位进行免疫原性分析[17]。免疫原性由氨基酸序列所决定。本研究采用VaxiJen v2.0(http://www.ddg-pharmfac.net/vaxijen/VaxiJen/VaxiJen.html)[18]计算9肽、15肽表位的免疫原性,以评分>0.5为阳性标准,即该表位被视为强免疫原性,否则为不具有免疫原性。
为进一步探究高亲和力、强免疫原性的优势表位跨人群、物种的交叉免疫反应性,将各个基因型筛选出来的高亲和力、强免疫原性的优势表位进行对比分析,15 肽表位用其核心9 肽来表示,并采用Excel绘制三维柱状图。
1.4 HTNV Gn MHC-Ⅰ/Ⅱ类限制性表位的保守性预测 为了确定HTNV 76-118毒株Gn序列中优势表位的进化保守性,采用Blastp(https://blast.ncbi.nlm.nih.gov/Blast.cgi)[19]工具预测高亲和力、强免疫原性的9肽和15肽表位的种间种内保守性。其中,种间保守的评价标准为除汉坦病毒(HTNV,taxid:1980471)、人(Human,taxid:9606)、鼠(Mouse,taxid:10088)外的正汉坦病毒(Orthohantavirus,taxid:1980442),而种内保守评价标准为除汉滩病毒76-118 株(Hantaan virus 76-118,taxid:11602)、人(Human,taxid:9606)、鼠(Mouse,taxid:10088)外的汉滩病毒(Hantaan hantavirus,taxid:1980471)。在分析结果中,若E值<10-5,则认为保守。本实验选择种间、种内均保守的表位作为候选靶标。
1.5 双向聚类分析 MHC 分子多态性和表位肽多样性构成了pMHC 相互作用的差异性。为了将其中的关系可视化,利用TBtools[20]对MHC-Ⅰ、Ⅱ基因型与HTNV Gn 表位亲和力的排名进行双向层次聚类。亲和力排序数据通过以2为底的对数和Z得分标准化处理之后,用Complete 方法进行欧氏距离的双向层次聚类。分析包含57 个MHC-Ⅰ类分子与HTNV Gn 9 肽的相互作用及51 个MHC-Ⅱ类分子与HTNV Gn 15肽表位的相互作用。
1.6 模拟pMHC分子对接
1.6.1 HTNV Gn 9肽与MHC-Ⅰ类分子模拟对接HPEPDOCK(http://huanglab.phys.hust.edu.cn/hpepdock/)[21]是一种新型的网络服务器,通过输入9 肽优势表位序列与MHC-Ⅰ类分子PDB 格式文件即可模拟pMHC对接并获得对接模型。使用RCSB PDB数据库(https://www.rcsb.org/)即可获得相关的MHC-Ⅰ类分子3D 结构数据[HLA-A0206(3OXR)、 HLA-A2402(5WWJ)、 HLA-A0101(4NQX)、 HLA-B5301(1A1N)、HLA-B4001(6IEX)、 HLA-B5801(5V5L)、 HLA-B0801(3X13)、H2Kb(6JQ3)、H2-Ld(6L9M)]。对高亲和力、强免疫原性和种间种内均保守的9 肽优势表位与上述MHC-Ⅰ类分子模拟对接,观察对接形式及相应的对接分数。每个9肽与MHC-Ⅰ类分子对接会产生100 个模拟结构,前10 个被选为高置信度对接结果。将高置信度对接结果数据标准化后,绝对值高于总体中位数的数量称为可结合核心数;对接分数绝对值最高者称为最优核心结合力。将上述两种数据利用RStudio制作出气泡图。
1.6.2 HTNV Gn 15肽与MHC-Ⅱ类分子模拟对接使用EpiDOCK(http://www.ddg-pharmfac.net/epidock/EpiDockPage.html)[22]工具对具有高亲和力、强免疫原性和种间种内保守性的人鼠共同的15肽优势表位与MHC-Ⅱ类分子进行模拟对接。15肽表位被作为7个9肽核心进行分子模拟对接,将得到的预测分数减去阈值即为对接分数,对接分数>0则认为是真正的结合物。每种情况的结合分数最大值称为最优核心结合力,绝对值高于总体中位数的数量称为可结合核心数。将上述两种数据利用RStudio制作出气泡图。
1.7 质粒疫苗和灭活疫苗 二价HFRS 灭活疫苗(HANPUWEI®)由中国长春生物制品研究所有限责任公司生产。该疫苗由仓鼠肾细胞中灭活和纯化的HTNV 和SEOV 制成(中国无单价HFRS 疫苗)。质粒pVAX-Gn疫苗为本课题组在先前的研究中合成[23-24]。
1.8 动物分组及免疫 20只无特定病原体的近交系8 周龄雌性BALB/c 小鼠购自空军军医大学实验动物中心。BALB/c小鼠位于MHC单倍型H2-d内。将小鼠随机分为灭活疫苗组(n=10)与pVAX-Gn 疫苗组(n=10)。灭活疫苗组小鼠接种50 μl 1:5 稀释的二价灭活疫苗(长春生物制品研究所有限责任公司,参照说明书确定免疫剂量),pVAX-Gn 疫苗组小鼠注射50 μl 质粒pVAX-Gn 疫苗,均为每3 周注射1 次,共注射2 次。第1 次免疫后5 周通过吸入CO2处死小鼠,采用ELISpot实验评估细胞免疫反应性。所有动物均饲养在无病原体、安静的动物实验室内,用标准饲料和过滤水喂养,每周更换垫料,小鼠均正常进食、饮水,确保10 h以上的连续夜间时间以维持昼夜节律。本动物实验获空军军医大学动物中心伦理委员会审批(2021-1201),实验过程符合国家及单位有关动物管理和使用的规定。
1.9 ELISpot实验验证表位的细胞免疫反应性 由于其生产中固有的困难,仅对HTNV Gn上所预测出的5 个15 肽表位进行合成(ChinaPeptides,中国上海),用20 μg/ml 的PBS 稀释单个肽用于γ 干扰素(INF-γ)ELISpot 分析,4 ℃条件下在包被有IFN-γ 特异性捕获抗体的ELISpot板上过夜。通过吸入CO2处死小鼠并解剖脾脏组织,研磨制成(0.1~2.0)×106个/ml 浓度的单细胞悬液。红细胞裂解后,冲洗脾细胞并重新悬浮。在室温下用含10%胎牛血清的RPMI 1640培养基封闭ELISpot板2 h后,将1×106个脾细胞添加至每个孔中,用终浓度为5 μg/ml 的合成Gn 肽刺激脾细胞,并将平板在37 ℃、5% CO2培养箱中培养24 h,其中,阴性对照为完整的培养基,阳性对照为10 μg/ml 的刀豆蛋白A(Con A)。孵育后,将培养板用去离子水和PBST(含0.05% Tween-20的PBS)分别洗2~3 次,孔内加入100 μl 终浓度为2 μg/ml 生物素化抗IFN-γ抗体,室温孵育2 h。用PBST洗涤后加入链霉亲和素-辣根过氧化物酶(streptavidin-HRP)室温孵育1 h。PBST 洗4 次后加入AEC(DAKEWEI,China)底物显色,并用去离子水洗涤终止反应。待96孔板风干后用CTL ELISpot读取器(美国CTL公司)对板中的斑点进行计数。所有结果均显示为每106个脾细胞的斑点形成单位(SFU)平均值。采用GraphPad Prism 9绘制条形图。
1.10 统计学处理 采用GraphPad Prism 9 进行统计分析。本研究中仅ELISpot 实验涉及到统计学检验,各组数据均以±s 表示,组间差异比较采用t检验。P<0.05为差异有统计学意义。
2 结 果
2.1 HTNV Gn上小鼠H-2和人HLA-Ⅰ、Ⅱ类限制性表位的亲和力、免疫原性及保守性分析 表1 列出了预测工具SMMPMBEC、IEDB、NetMHCⅡpan3.2、NetMHCpan4.1、SYFPEITHI、Rankpep 预测的Gn 优势表位数目。重新计算排除重复9 肽和15 肽后,在H-2亚型中获得了61个优势表位,在HLA-Ⅰ亚型中得到234个优势表位,HLA-Ⅱ亚型中得到212个优势表位。在H-2 亚型中,H-2-Kb 优势表位数目最多,有12 个9 肽。在HLA-Ⅰ类亚型中,HLA-B7 优势表位数目最多,有27 个9 肽。在同等条件下,免疫原性强的表位更有可能充分诱导免疫应答。表1 列出了小鼠和人对Gn的强免疫原性表位数目。排除重复的表位后,在H-2 亚型中获得了23 个强免疫原性优势表位,在HLA-Ⅰ亚型中获得了110 个强免疫原性优势表位,HLA-Ⅱ亚型中获得了42个强免疫原性优势表位。
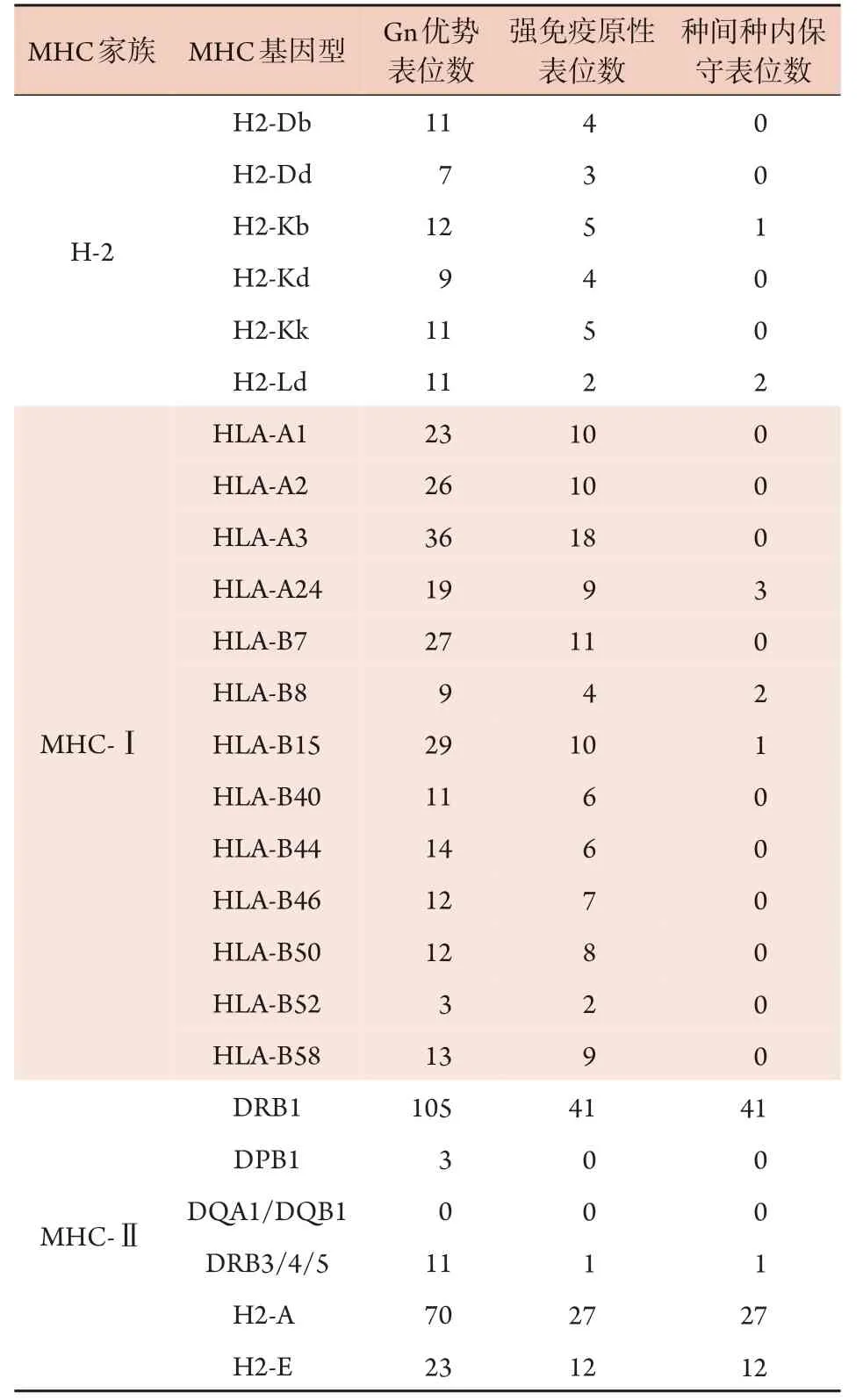
表1 小鼠H-2 和人HLA 分子对Gn 表位的亲和力、免疫原性和保守性分析结果Tab.1 Analysis results of affinity, immunogenicity and conservation of mouse H-2 and human HLA molecules to Gn epitopes
采用Blastp工具预测上述高亲和力、强免疫原性9肽和15肽优势表位的种间种内保守性,结果如表1所示。筛选获得MHC-Ⅰ类限制性表位6 个,MHC-Ⅱ类表位81 个。其中,上述MHC-Ⅰ、Ⅱ类限制性表位中存在3个交叉表位。
各个基因型筛选出来的高亲和力、强免疫原性优势表位比对结果显示,DRB1基因型与各个基因型的高亲和力、强免疫原性表位均有重叠,与H2-A、H2-E基因型的重叠数目最多(图1),而H2-Ld、DPB1等基因型与其他基因型重叠表位较少。

图1 HTNV Gn上各MHC超家族高亲和力、强免疫原性表位的交叉反应性Fig.1 Cross-reactivity of MHC superfamilies with high affinity and strong immunogenicity on HTNV Gn
2.2 HTNV Gn 9 肽、15 肽表位与MHC-Ⅰ、Ⅱ类分子的双向层次聚类揭示跨基因型间交叉免疫反应性
2.2.1 HTNV Gn 9 肽与MHC-Ⅰ类分子的相互作用关系 如图2所示,57个MHC-Ⅰ Gn类分子被分为3组:HLA-Ⅰ亚型组、交叉反应组(HLA 主要)及交叉反应组(H2 主要)。在HLA-Ⅰ亚型组中,HLA-A3201与HLA-B58(-B5701、-B5801)得分相似,表明HLAA3201 基因型在提呈HTNV Gn 抗原过程中表现出HLA-B58 样特征;HLA-A2601 与HLA-A3401 归属于同一类别,两个基因型得分相似;HLA-B4601 与HLA-B15(-1502,-1513,-1521)得分相似,表明HLAB4601 基因型在提呈HTNV Gn 抗原过程中表现出HLA-B15 样特征;HLA-A6801、HLA-A3301、HLAA0301、HLA-A1101 得分相似,归属于同一类别;HLA-B0801、HLA-B1405、HLA-B3903 得分归属于同一类;HLA-B5301 与HLA-B3501、HLA-B3508归属于同一类别。对于交叉反应组(H2 主要),H2-Ld 得分与HLA-B0702相似,归属于同一类;H2-Db、H2-Dd和H2-Kb得分体现出在鼠种内提呈HTNV Gn的相似性。在交叉反应组(HLA 主要)中,H2-Kd 得分与HLA-A24(-A2301、-A2402、-A2407)相似,表明MHC在提呈9肽表位上的跨基因型相似性。
2.2.2 HTNV Gn 15 肽与MHC-Ⅱ类分子的相互作用关系 图3显示了不同MHC-Ⅱ亚型间的差异。51个MHC-Ⅱ类分子被分为3组:HLA-Ⅱ亚型组、交叉反应组(HLA主要)及交叉反应组(H2主要)。在HLA-Ⅱ亚型组中, DRB1(-0407、 -0412、 -0803、 -1402、-1409、-0304、-0301、-0101、-0202、-1202、-1401、-1408、 -1104、 -1301、 -1304) 和 HLA-DQA10101-DQB10501、 HLA-DPA10103(-DPB10101、 -DPB10201、-DPB10401、-DPB10402)归属于同一类别,上述基因型得分相似;DRB1(-1302、-1201、-1501)和HLADQA10501-DQB10201、 HLA-DQA10301-DQB10301、HLA-DQA10401-DQB10402、HLA-DQA10501-DQB10301 基因型相近,表明其在提呈HTNV Gn 抗原过程中表现出相同的特征;DRB1(-0704、-1602、-0701、-0101、-0901、-0401、-0405、-0802)和HLADPA10103-DPB11401 归属于同一类别,基因型得分相似。对于交叉反应组(H2 主要),H2-IAd 得分与HLA-DQA10102-DQB10602相似,归属于同一类,具有相似的抗原呈递结果。在交叉反应组(HLA 主要)中,H2-IEK与DRB0101基因型得分相似。
2.3 MHC分子与表位的模拟分子对接验证亲和力
2.3.1 HTNV Gn 9 肽表位与MHC-Ⅰ类分子对接图4A 所示为每个肽-MHC 对接后的可结合核心数及最优核心结合力的气泡图,3个9肽优势表位均与多个MHC 分子对接较好。表位RYKSRCYIF 与HLAB5801、HLA-A0206、HLA-B5301 等基因型对接的最优核心结合力较为良好,表明该表位与上述基因型分子结合的牢固程度更高,同时,该表位与HLAB5801、HLA-A0206、HLA-B5301、HLA-B0801 等基因型分子对接可结合核心数较多,表明其与基因型分子对接的概率较大; 表位VVYERTYCM、QVVYERTYC与H2-Kb和HLA-B4001基因型对接情况较弱,表明其与上述基因型分子对接的概率及对接牢固程度均较低。此外,HLA-B5801、HLA-A0206、HLA-B5301、HLA-B0801 基因型对3 个优势表位的可结合核心数多,表明上述基因型分子对优势表位的结合概率较大。总体而言,3 个优势表位与人HLA基因型对接时的最优核心结合力和可结合核心数均优于小鼠H-2基因型。如图4B中b、d所示,分别选取表位RYKSRCYIF 与H2-Kb、QVVYERTYC 与H2-Ld作为对接优良的例子,通过相应的对接模拟结构可以看出,在上述两个对接过程的10种对接模拟结构中,9 肽均位于MHC 分子抗原结合槽内。选取表位QVVYERTYC 与H2-Kb、表位VVYERTYCM 与H2-Kb作为对接较差的例子,如图4B中a、c所示,上述两种对接模拟结果中,多数9 肽模拟状态均位于MHC分子抗原结合槽外,其中有一个游离于结合槽;同样的情况见于表位VVYERTYCM 与H2-Kb 对接结果中。通过比较上述分子对接模型,从侧面验证了分子模拟对接存在分子结构上的基础,与分子对接结合可保持一致。
2.3.2 HTNV Gn 15 肽优势表位与MHC-Ⅱ类分子模拟对接 如图5 所示,5 个MHC-Ⅱ类限制性优势表位均与多个MHC基因型分子对接良好,其中,与表位GGIFNITSSMCLVSK 对接良好的基因型最多。此外,DPA1*0201/DPB1*0101 基因型与优势表位对接结果均较差。

图5 HTNV Gn 15肽表位与MHC-Ⅱ类分子对接分数气泡图Fig.5 The bubble map of HTNV Gn 15-mer epitope and MHC-Ⅰ molecule docking
2.4 ELISpot实验评估15肽表位与MHC-Ⅱ类分子对接的细胞免疫反应性 ELISpot实验结果显示,在灭活疫苗组及pVAX-Gn 组中,5 个表位均可引起脾细胞分泌IFN-γ(图6),提示通过表位筛选促进细胞免疫应答确实行之有效。同时,两组的SFU 平均值接近,提示Gn 上的3 个人鼠共优势15 肽表位由Gn 和GP全长所引起的免疫应答效果相近。
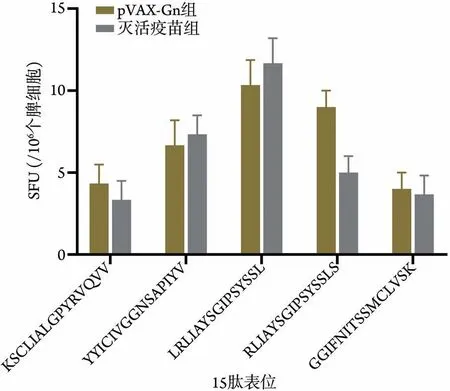
图6 5个15肽表位ELISpot实验结果Fig.6 ELISpot experiment results of 5 15-mer epitopes
3 讨 论
对于HTNV 的预防与控制,现有双价灭活疫苗已被证实可诱导抗病毒应答,但在中和抗体效价、细胞免疫应答和长效免疫记忆方面尚存在不足,同时在T 细胞免疫应答水平方面也有待提高。本研究以比较免疫学和结构生物学原理为基础,选取可特异性激活抗HTNV 免疫应答的Gn 分子为研究对象,对9 肽和15 肽表位免疫反应性进行评估,最终获得高亲和力、强免疫原性、种间种内保守的MHC-Ⅰ类限制性优势表位6个,MHC-Ⅱ类限制性优势表位81个。使用上述方法可精准预测出HTNV Gn上细胞免疫反应性表位,在短时间内找到具有潜在疫苗作用靶点的抗原表位,极大地节省了实验成本。同时,该方法已用于近年流行的新型冠状病毒等病毒的研究[25-26]。
人类遗传因素也可影响对汉坦病毒的易感性及其所致疾病的严重性[27]。有研究发现了HTNV 感染患者中HLA基因型的连锁性,HLA-B*46-DRB1*09和HLA-B*51-DRB1*09 基因型在中国汉族危重HFRS 患者中更常见[28-29]。本研究比较了HLA-Ⅰ类基因型和HLA-Ⅱ类基因型之间HTNV GP 免疫反应性表位的相关性,发现DRB1基因型与HLA-B15、-B46基因型相同的免疫反应性表位较多,提示DRB1-B15、DRB1-B46的化学结构基础相似,具有上述基因的不同地区、不同种族的人群可能对汉滩病毒感染具有相近的免疫应答反应,同一表位引起的效应可能相近。同时,本研究对小鼠基因型与人类基因型的表位进行对比发现,人类DRB1 基因型与小鼠H2-A、H2-E 基因型部分表位更为相近,HLA-A02 基因型与H2-Kd 基因型在提呈HTNV GP 上9 肽表位时亲和力数据呈现相似的趋势,提示在不同物种存在对GP表位相似的反应,具有上述基因型的小鼠与人类在汉滩病毒感染及表位免疫刺激后可能产生相近的免疫应答反应,提示在缺乏人源化转基因小鼠的情况下,使用特定基因型的小鼠可能是实验模型的有用替代物[16,30]。
近年来,随着生物信息学的迅速发展,工具与算法不断更新。不同工具和算法的原理大相径庭,但目的均是对MHC表位的亲和力进行模拟分析。本研究将多个算法进行整合运用,保证了分析的高准确率和泛MHC覆盖率。然而,正因如此,本研究更注重于结果的获取,对其中的算法过程考虑较少,因而衍生出以下问题:(1)不同运算过程将赋予表位一些意料之外的特殊内涵;(2)多种算法多个工具同时使用可造成多重检验问题;(3)由于多重检验造成的假阴性、假阳性和过拟合等;(4)生物信息学算法和数据库的更新及对表位结果的影响;(5)调整工具优先级、参数设置以提高预测准确率的问题。这些问题背后的含义,将是后续深入挖掘探索生物信息学的方向。
综上所述,本研究预测并验证了HTNV Gn上可激活细胞免疫反应性的表位,证实在MHC提呈中病毒抗原存在跨基因、种属、物种的交叉免疫反应性。然而,本研究仅在小鼠脾细胞中进行了表位验证,其在人体内的免疫反应性仍需进一步探讨。与Dawood[25]、Araújo 等[26]通过数据库预测获得SARSCoV-2 结构蛋白相关的强免疫原性、种间种内保守的候选表位相比,本研究创造性地进行种属交叉分析和实验验证,提高了抗原优势表位筛选的系统性和可靠性,为在免疫遗传学背景下开发基于HTNV Gn分子特异性群体免疫保护性表位疫苗提供了理论基础。
