基于全基因组关联分析的青年人群卧推1RM抗阻训练效果预测模型研究
2023-10-10梅涛李燕春李晓霞杨晓琳李亮晏冰何子红
梅涛,李燕春,李晓霞,杨晓琳,李亮,晏冰,何子红
(1.北京体育大学,北京 100084;2.山东体育学院,山东 济南 250102;3.苏丹依德理斯教育大学,马来西亚 丹戎马林 35900;4.国家体育总局体育科学研究所,北京 100061)
上肢肌肉力量在运动中发挥重要的作用,且与健康密切相关。上肢肌肉力量和质量较低被认为与死亡风险的增加有关(Wu et al.,2022)。抗阻训练能够提高上肢卧推1RM重量,改善上肢肌肉力量,并对健康产生积极影响(Polito et al.,2021)。有研究发现,与不进行抗阻训练相比,抗阻训练可将全因死亡风险降低15%,心血管疾病死亡率降低19%(Shailendra et al.,2022)。然而,抗阻训练提高肌肉力量的效果存在较大个体差异(Brigatto et al.,2022;Martins-Costa et al.,2022),且较少被关注(Bonafiglia et al.,2021)。
训练效果的个体差异主要受遗传因素(基因组学)和身体特征因素(表型组学)的影响。遗传学因素中,肌肉力量的遗传度约为52%(Zempo et al.,2017)。候选基因研究已发现4个基因的7个单核苷酸多态性位点(singlenucleotide polymorphisms,SNPs)与抗阻训练效果相关,分别为ACErs4646994/rs1799752/rs4340/rs13447447、ACTN3rs1815739、IL15RArs2296135、PPARArs4253778(Alvarez-Romero et al.,2021),其中ACTN3rs1815739对12周抗阻训练干预产生的肌力提升效果的解释度为2%(Clarkson et al.,2005)。表型组被定义为某一生物体的全部性状特征(Bush et al.,2016;Freimer et al.,2003)。表型因素中,年龄、性别、身体质量指数(body mass index,BMI)、肌肉质量等多个表型指标可影响抗阻训练效果。例如,年龄和性别可以影响特定的肌纤维亚型对抗阻训练的反应,表现为抗阻训练后在年轻女性中,I型纤维的百分比显著增加40%~51%,而年轻男性和老年人未发生此变化(Martel et al.,2006)。抗阻训练后卧推1RM重量的提升同样表现出性别差异(Mayhew et al.,2011)。课题组前期研究也发现,表型指标如卧推初始值、上肢肌肉质量、股直肌和股中间肌厚度等表型因素可解释42.5%的卧推训练效果(梅涛 等,2021)。
随着主动健康领域研究的深入,从个体差异角度制定更加个性化的精准健身方案,对于健康促进具有重要的社会价值。尽管研究已经发现了一些训练效果相关的SNPs,但由于鉴定的SNPs数量有限,已发现的SNPs无法对训练效果进行准确预测。全基因组关联分析(genomewide association studies,GWAS)是近年用于寻找复杂性状遗传标记的重要手段,为训练效果个体差异从基因组层面进行解释提供了可能性。本研究基于中国人群力量训练队列,首次应用GWAS从基因组层面筛选卧推1RM抗阻训练效果的遗传标记,进一步结合表型指标建立卧推1RM抗阻训练效果综合预测模型,以期为制定提高上肢肌肉力量的精准化健身指导方案提供研究依据。
1 研究对象与方法
1.1 研究对象
基于国家重点研发计划课题建立的中国人力量训练队列(梅涛 等,2021)开展研究,样本库共纳入力量训练受试者193名,其中男性95名,女性98名,受试者基本情况见表1。受试者纳入标准:1)体力活动未达到每周150~300 min中等强度有氧运动,或75~150 min高强度有氧运动,或中高强度有氧运动等效组合的成年人(Bull et al.,2020);2)无运动训练损伤风险的健康成年人(Schwartz et al.,2021);3)无不良饮食习惯,并在干预期间保持规律饮食。研究经北京体育大学运动科学实验伦理委员会审查批准。
1.2 抗阻训练方案
受试者进行充分热身后,开始抗阻训练。抗阻训练方案为:以70% 1RM的负荷进行卧推,共5组,每组10次,组间间歇2 min,每周2次,持续12周(梅涛 等,2021)。每4周进行一次1RM测试确定新的训练负荷,以适应力量增长的变化。干预过程中对受试者进行训练负荷的监控,要求受试者按照动作标准完成全部的训练量。若受试者无法独立完成,可在最小辅助情况下完成训练,以保证受试者在完成相同的训练内容后机体受到的训练负荷刺激一致。12周干预前后进行指标测试。
1.3 指标测试
指标测试包含问卷调查、卧推1RM重量测试、身体成分测试和肌肉厚度测试等,其中问卷调查用于干预前受试者的筛选和干预过程中运动量、饮食的监控,卧推1RM重量变化百分比用于判断力量训练后的肌力提升效果,其余表型指标(卧推1RM重量、身体成分和肌肉厚度初始值)用于建立预测模型的预测因子。
1.3.1 问卷调查
调查问卷包括全球体力活动问卷(global physical activity questionnaire,GPAQ)(Bull et al.,2009)、膳食回顾问卷(中国居民营养与健康状况监测食物频率调查问卷)(Liu et al.,2018)和运动风险问卷(physical activity readiness questionnaire,PAR-Q)(Thomas et al.,1992),受试者集中听完讲解和要求后填写,由专人逐一核对完整性和合理性后收集录入。
1.3.2 卧推1RM重量测试
采用力量训练架、杠铃杆和标准化杠铃片测试1RM卧推重量,具体测试过程如下(Pescatello et al.,2014):1)根据受试者的主观感觉预测受试者1RM,以预测值的40%重量进行5~10次卧推作为热身;2)热身后休息1 min;3)在热身重量上增加15~20 kg,要求受试者完成3~5次卧推,作为第1次测试;4)休息2~4 min,在步骤3负荷基础上增加5~10 kg,使受试者最大可完成2~3次卧推;5)休息2~4 min,重复步骤4,尝试最大卧推重量;6)如受试者成功完成,按照步骤5继续增加负荷,直至该负荷下受试者无法成功完成1次卧推;7)休息2~4 min,减小5~10 kg负荷继续测试,此后继续增加或减小负荷,直至受试者能够以标准技术完成1次最大卧推重量,在5次尝试之内确定卧推1RM重量。
1.3.3 身体成分测试
采用双能量X线骨密度仪(Lunar iDXA,美国)测试受试者的身体成分。测试前确保受试者近期未做过钡餐检查、放射性同位素注射及注射或口服造影剂等。提前预热并校准仪器,测试时要求受试者空腹,身着薄衣物,于平卧位开始检查。测试端输入受试者基础参数,选取扫描模式,从头向足侧逐层扫描,获取受试者身体成分数据。
1.3.4 肌肉厚度测试
采用彩色多普勒超声仪(LOGIQ,美国)测试受试者的股直肌、股直肌至股中间肌、胸大肌厚度(Jacob et al.,2021)。股直肌位置标记:受试者平躺位,双腿自然放松,与肩同宽,标记髂前上棘至髌骨上缘连线的中点位置。胸大肌位置标记:受试者平躺位,使用人体测量尺测量,男生标记腋前线至乳头连线的中点位置,女生标记距离腋前线1/3处。股直肌至股中间肌标记:使用肌骨超声12 Mhz线阵列探头,B-Mode在中点处垂直于大腿表面获取股直肌至股中间肌横截面图像。每侧肌肉测试3次取平均值。
1.4 全基因组基因型测试
1.4.1 DNA提取及质量控制
抽取受试者静脉血5 ml,采用磁珠法血液基因组提取试剂盒(天根生化,中国)进行DNA提取,采用Nanodrop 2000(赛默飞,美国)检测DNA浓度和纯度,凝胶电泳检测DNA完整性。要求DNA浓度大于100 ng/μl,样品OD值260/280大于1.8,凝胶电泳主带清晰、无降解条带。
1.4.2 全基因组基因型检测及质量控制
DNA样本质检合格后,采用Infinium芯片(芯片类型:CGA-24v1-0)(Illumina,美国)进行全基因组基因型检测,采用GenomeStudio 2.0(Illumina,美国)数据分析软件分析检测结果,并进行数据格式的转化。以1000 Genomes Phase3 V5(GRCh37/hg19)的中国人全基因组数据为参考模板,采用impute2方法进行基因型填充(填充过滤条件:info≥0.98)。采用PLINK1.9软件对芯片数据进行质量控制,排除标准:1)最小等位基因频率小于5%(maf<0.05);2)不符合哈迪温伯格平衡(Hardy-Weinberg equilibrium,HWE)(P>1×10-5);3)基因型缺失超过10%的SNPs;4)基因型缺失率超过10%的个体。基因型填充后,通过质量控制保留的SNP位点为4 929 604个。采用质控后的SNPs进行后续主成分分析(principal components analysis,PCA)和关联分析等。
1.5 统计学方法
采用SPSS 19.0、GraphPad Prism 8对数据进行统计分析。描述性统计结果以平均数±标准差(M±SD)表示。干预前后卧推1RM变化采用配对样本t检验,卧推1RM训练效果采用干预前后变化百分比表示(卧推1RM%=×100%)。PCA法对人群分层进行质控。应用PLINK 1.9软件,以1RM初始值、性别、年龄以及PCA分析的前5个主成分作为协变量进行线性回归,显著性水平为P<5×10-5,进行全基因组的遗传标记筛选。计算GWAS关联结果的膨胀系数(λ)评价GWAS关联结果的偏倚情况以及是否受到群体分层的影响(Wang et al.,2019),采用R语言包qqman进行GWAS曼哈顿图的绘制。基因组学预测模型建立方法参考Bouchard等(2011)的研究,以1RM变化百分比为因变量,显著性遗传标记为自变量,首先采用向后线性回归筛除冗余SNPs(P>0.1),再采用向前线性回归建立卧推1RM训练效果基因组学模型。采用平均值法计算权重后的基因组多基因得分(genomics polygenic scores,GPGS)得分(GPGS=),以卧推1RM变化百分比为因变量(y),GPGS为自变量(x)进行线性回归。采用K-mean方法对卧推1RM变化百分比进行聚类分析,采用前进法Logistic回归分析,以卧推1RM变化百分比聚类分析结果作为因变量(高反应1,低反应0),以GPGS得分、表型指标[年龄、性别(男性1,女性0)、BMI、身体成分、肌肉厚度]作为自变量(梅涛 等,2021),建立基因组-表型指标训练效果预测综合模型。对Logistic回归模型的拟合度采用似然比检验,并用ROC曲线下面积评价此回归模型的预测能力。显著性水平P<0.05。采用HaploReg v4.1、GTEx、REACTOME PATHWAY数据库(Jassal et al.,2020)对筛选出的遗传标记进行生物信息学分析。
2 研究结果
2.1 卧推1RM抗阻训练效果
结果显示,训练前,受试者的卧推1RM重量为(40.66±20.42)kg,12周抗组训练后,卧推1RM重量为(55.40±22.80)kg,显著提高了36.25%(P<0.01)(图1A),但个体差异明显,卧推1RM变化百分比范围为-31.25%~176.92%(图1B)。

图1 卧推1RM训练效果Figure 1.Training Effect of Bench Press 1RM
2.2 卧推1RM训练效果的GWAS分析
GWAS分析显示,rs79726572、rs6506389、rs7504284等35个SNPs(表2)与12周抗阻训练后卧推1RM变化百分比存在显著关联(P<1×10-5),其中2个SNPs显著性水平为P<1×10-6(图2)。膨胀系数λ=1.007,说明P值的显著性水平不是因为群体分层而致,不存在假阳性。

图2 卧推1RM训练效果GWAS分析的曼哈顿图Figure 2.Manhattan Plot of GWAS Analysis of Bench Press 1RM Training Effect

表2 与卧推1RM训练效果关联的SNPs位点Table 2 SNPs Loci Associated with Bench Press 1RM Training Effect
2.3 卧推1RM训练效果预测的基因组学模型与GPGS得分
以卧推1RM变化百分比为因变量,以GWAS发现的35个SNPs作为自变量,构建卧推1RM训练效果基因组学预测模型,向后线性回归去除冗余SNPs后,8个SNPs位点被保留;向前线性回归7个SNPs纳入训练效果基因组学预测模型(表3),模型总R2=0.396。
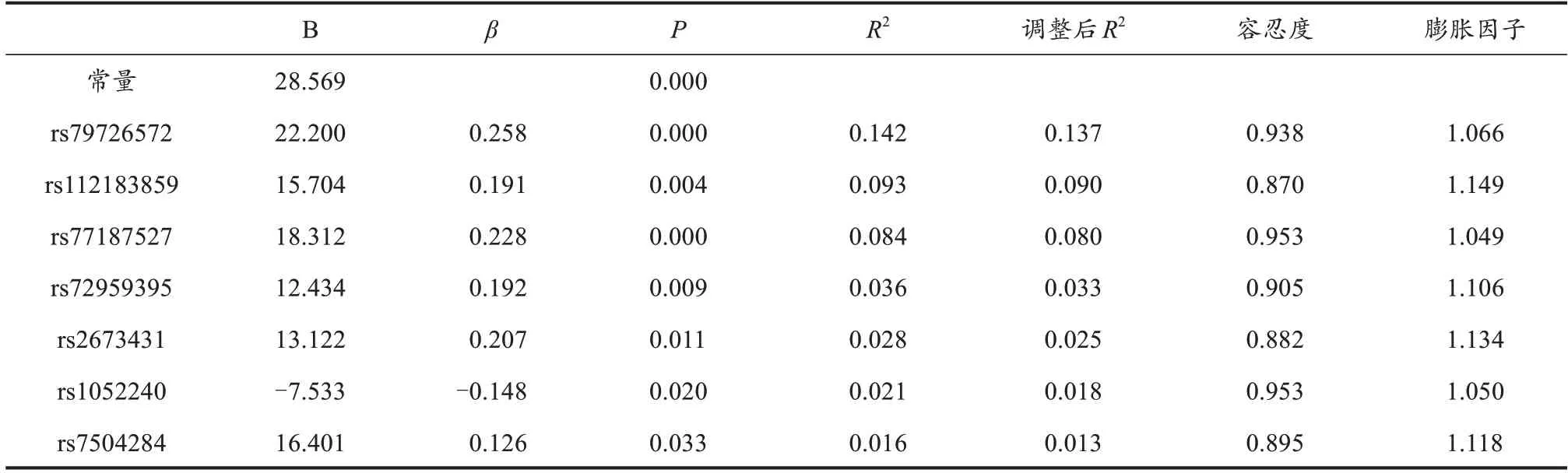
表3 卧推1RM训练效果预测的基因组学模型Table 3 Genomic Model for Predicting the Training Effect of Bench Press 1RM
以卧推1RM变化百分比为因变量,以GPGS得分作为自变量进行线性回归,结果表明,卧推1RM训练效果与GPGS得分呈正相关,y=4.882x+32.51(R2=0.39,P<0.001)(图3)。GPGS平均得分为3.12,分布范围为-3.93~27.21。统计发现,GPGS高于3.12的受试者,其抗阻训练效果高于平均值,占人群总数的43.5%。
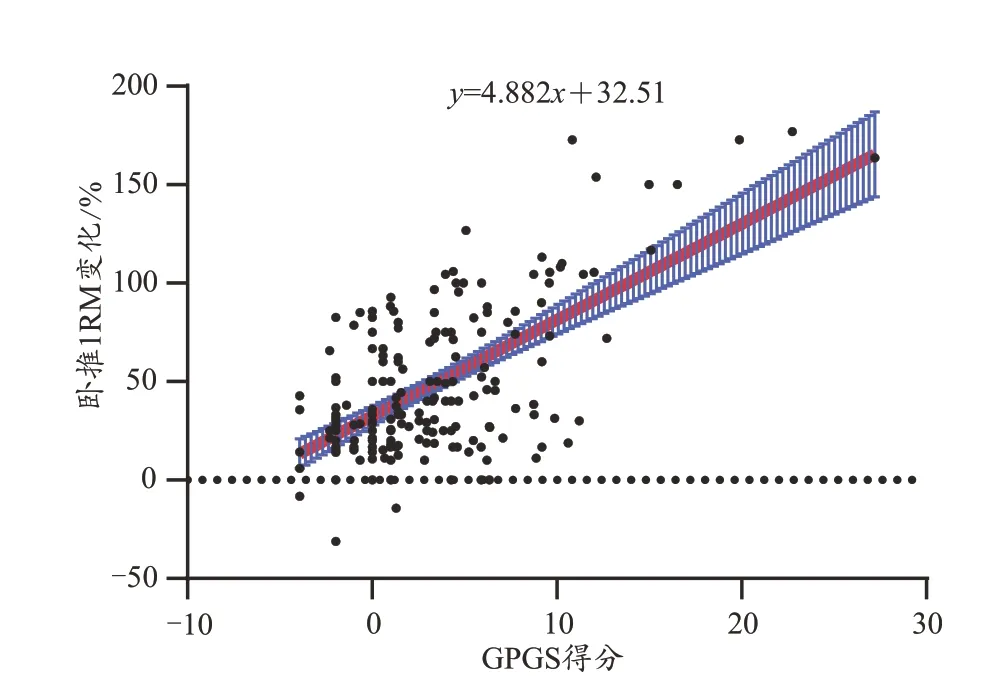
图3 卧推1RM训练效果与GPGS的线性关系Figure 3.Linear Relationship between Bench Press 1RM Training Effect and GPGS
2.4 联合基因组学-表型指标的卧推训练效果预测综合模型
采用K-mean方法对卧推1RM变化百分比进行聚类分析,将受试者分为低反应聚类和高反应聚类,聚类信息见表4所示。

表4 卧推1RM变化百分比聚类分析Table 4 Cluster Analysis of Bench Press 1RM Change Percentages
以卧推1RM变化百分比聚类分析训练效果(低反应0、高反应1)为因变量,GPGS得分、表型指标为自变量进行Logistic回归,构建卧推1RM训练效果的基因组学-表型综合预测模型。结果显示,卧推初始值、BMI、右上肢肌肉质量、左躯干肌肉质量、GPGS得分被纳入预测模型(表5)。采用ROC曲线下面积对上述模型进行检验,结果显示,模型曲线下面积(area under curve,AUC)为0.952(P<0.001),约登指数0.767,阈值(cut off)为0.251(图4)。

图4 联合基因组学-表型指标的卧推训练效果预测模型ROC分析Figure 4.ROC Analysis of the Predictive Model of Bench Press Training Effect with Combined Genomics-Phenotye

表5 联合基因组学-表型指标的卧推训练效果Logistic回归分析Table 5 Logistic Regression Analysis of the Training Effect of Combined Genomics-Phenotye Indicators for Bench Press
2.5 卧推1RM训练效果预测模型中SNPs的生物功能分析
HaploReg v4.1数据库对训练效果相关SNPs生物功能的注释结果见表6。rs79726572位于PARD3B基因内含子,可调控DMRT2、DMRT5、HAD等6个基因模体。rs72959395位于CD109下游,可调控RXRA基因模体。rs2673431位于U6基因上游,可调控CTCF、Mef2、Pou5f1等4个基因模体。rs1052240位于RP11-553K8.5基因内含子,可调控AP-1基因模体,在血液和脑组织中具有组蛋白活性。rs77187527位于TSEN15基因下游,可调控AP-1、Foxa、HNF4等7个基因模体,在心肌、骨骼肌等组织中具有组蛋白活性。rs7504284位于U6基因下游,无可调控的基因模体。
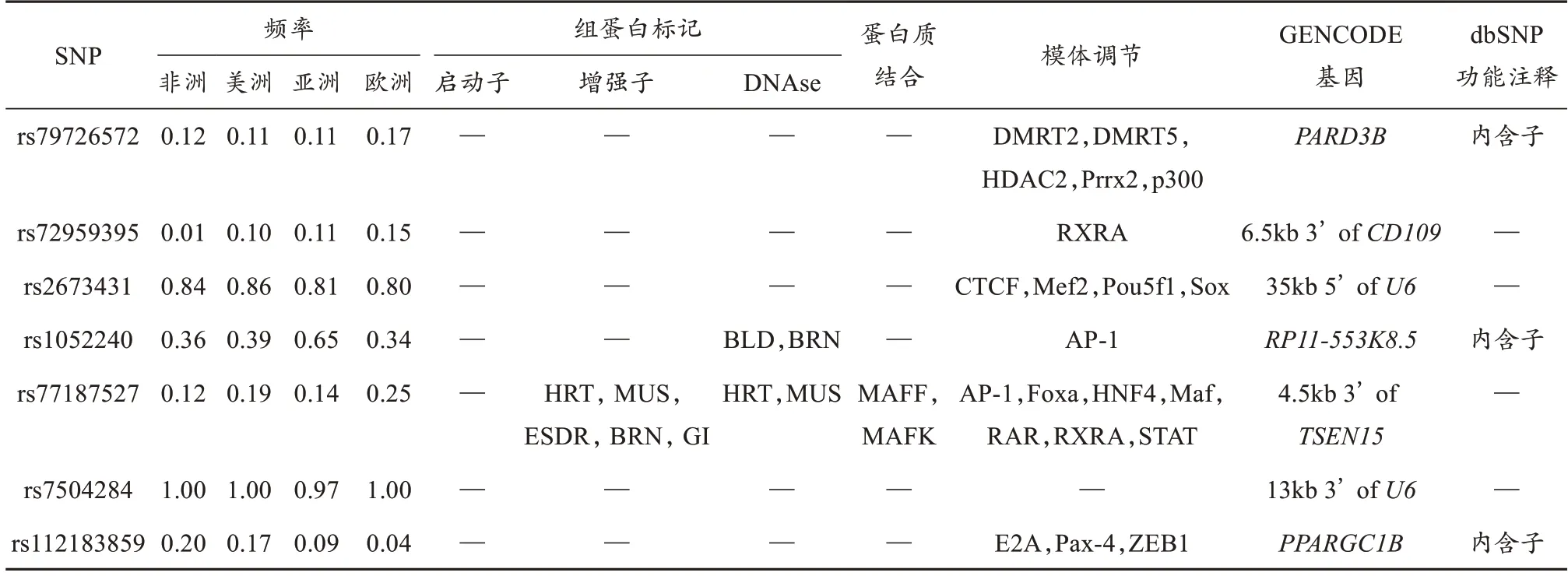
表6 卧推1RM训练效果相关SNPs的生物功能注释Table 6 Biological Function Annotation of SNPs Related to the Training Effect of Bench Press 1RM
对训练效果相关的SNPs调控的基因进行基因本体功能注释(gene ontology,GO),发现共富集到56个生物过程(GOTERM_BP)、9个细胞组分(GOTERM_CC)、38个分子功能(GOTERM_MF)(图5)。值得注意的是,生物过程条目中有5个条目与肌肉生长发育有关:skeletal muscle cell differentiation、muscle organ development、skeletal muscle tissue development、muscle cell fate commitment、positive regulation of skeletal muscle fiber development (表7)。
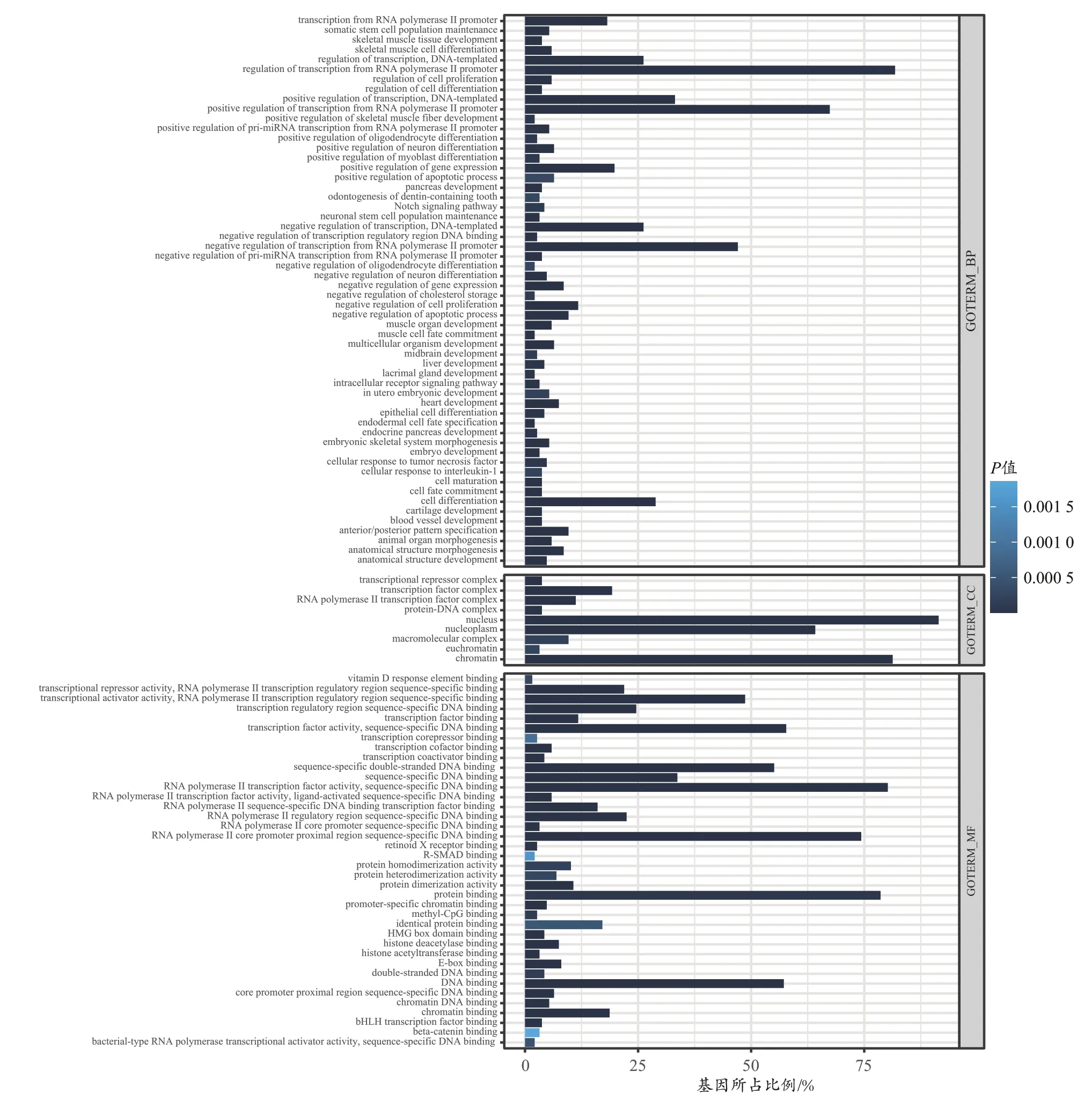
图5 SNPs调控基因GO功能注释Figure 5.Functional Annotation of GO of SNPs Regulatory Genes

表7 骨骼肌生长发育相关的生物过程Table 7 GOTERM_BP Related to Skeletal Muscle Growth and Development
基于REACTOME PATHWAY数据库,对训练效果相关SNPs及其连锁SNPs调控的基因进行通路分析。结果表明,受调控的基因富集于Generic Transcription Pathway、Developmental Biology、RNA Polymerase II Transcription、Gene expression (Transcription)、Myogenesis等37条信号通路(P<0.01,FDR<0.01)(图6)。

图6 训练效果相关SNPs及其连锁SNPs调控基因REACTOME PATHWAY气泡图Figure 6.Bubble Diagram of Training Effect Related SNPs and Their Linked SNPs Regulating the Gene REACTOME Pathway
3 讨论
3.1 卧推1RM训练效果的个体差异性
本研究发现,抗阻训练在提升卧推1RM训练效果上表现出较大的个体差异性。这种个体差异性可能与受试者性别、年龄等因素有关(Alonso-Aubin et al.,2021;Mayhew et al.,2008)。Harmon等(2010)研究发现,12周的上肢渐进式阻力训练后,女性受试者屈肘最大肌力增加了63%,个体差异范围0~9 kg,男性受试者屈肘最大肌力增加了40%,个体差异范围0~12 kg。Miller等(2019)发现,在卧推练习中,卧推功率和卧推达到最大速度的时间存在性别差异,表现为经过训练的男性产生的最大功率在30%~40% 1RM之间,而未经训练的男性则为60%~70% 1RM;经过训练的女性在50% 1RM时产生最大功率,而未经训练的女性在60%~70% 1RM 时产生最大功率。这表明尽管安排了相同的训练方案,但性别不同可能会产生不同的训练效果。有研究发现,抗阻训练的训练效果在不同年龄之间也存在显著差异(Izquierdo et al.,2001)。不同年龄在进行抗阻训练时,上肢力量发展速率有明显的差异,这可能是年龄因素影响抗阻训练效果的原因之一(Barry et al.,2005)。但也有研究报道,两组体力活动背景相似,但年龄不同[分别为(21.2±2.2)岁、(49.7±2.1)岁]的男性,参加8周的中等阻力训练后,卧推1RM增长幅度相似,未表现出差异(Arazi et al.,2013)。这可能跟干预方案、年龄跨度的不同有关。
3.2 基因组学指标对卧推1RM训练效果的预测
遗传因素是影响个体差异的重要因素,从基因组水平上分析个体差异的原因有助于解释训练效果的差异性。本研究发现7个SNPs(rs79726572、rs112183859、rs77187527、rs72959395、rs2673431、rs1052240、rs7504284),可解释卧推1RM训练效果的39.6%。其中rs79726572、rs112183859、rs77187527对卧推1RM训练效果的解释度在5%以上,rs79726572解释度可达13.7%。训练效果的单基因研究中,ACE、ACTN3、CNTF等多个基因被证实对抗阻训练效果有影响。ACEI/D多态性与老年人肌肉体积个体差异相关,但与抗阻训练引起的肌肉肥大反应无关(Charbonneau et al.,2008)。在年轻个体中,ACEII/ID型受试者抗阻训练侧手臂最大自主收缩(maximum voluntary contraction,MVC)增加显著高于DD型受试者,但1RM和肌纤维横截面积(cross-sectional area,CSA)在各基因型间无显著变化;ACEDD/ID受试者非训练侧手臂的1RM和CSA增加大于ACEII受试者,ACEI/D多态性对肌力训练效果的解释度为1%~2%(Pescatello et al.,2006)。基线MVC和训练后的1RM增益约2%可归因于ACTN3R577X基因型(似然比检验P=0.01)(Clarkson et al.,2005)。CCL2基因rs1024610与12周抗阻训练后的MVC变化百分比相关,携带T等位基因的男性受试者可获得更高的训练效果收益;CCR2基因rs3918358、rs1799865与12周抗阻训练后的1RM变化百分比相关,rs3918358 AA基因型和rs1799865 TT基因型可获得更高的训练效果收益,相关的SNPs解释了0.7%~2.5%的力量表型变化(Harmon et al.,2010)。CNTFrs1800169可解释12周上臂单侧抗阻训练后,女性1RM和MVC的训练效果,表现为携带G等位基因的个体能够获得更高的肌肉力量(Walsh et al.,2009)。
相较于单基因,多基因可对训练效果提供更高的解释度。在基于GWAS(2 391 739个SNPs)建立的有氧能力预测模型中,7个SNPs(rs11051548、rs2542729、rs1451462、rs13060995、rs6570913、rs11096663、rs12613181)可解释V˙O2max训练效果的26.0%,预测准确性为86.1%(AUC),建立的力量变化预测模型中,6个SNPs(rs10072841、rs6564267、rs17044554、rs1341439、rs4522375、rs7154161)可解释膝关节峰值力变化的27.5%,预测准确性为77.2%(AUC)(Yoo et al.,2016)。本研究卧推1RM训练效果的基因组学回归模型与GPGS线性模型的拟合度约为0.39,表明单从基因组学角度,无法准确对卧推1RM训练效果进行预判。
3.3 基因组-表型综合预测模型对卧推1RM训练效果的预测
独立的基因组学和表型指标对于卧推1RM训练效果的预测能力有限。本研究建立的基因组-表型综合预测模型AUC为0.952,表明模型具有较好的预测能力。卧推初始值、BMI、右上肢肌肉质量、左躯干肌肉质量、GPGS得分被纳入综合模型,是影响卧推1RM训练效果个体差异的关键表型因素。
对于初始值不同的受试者,获得最佳训练效果的训练方案不同。研究发现,在训练强度上,未经训练的受试者(初始值低)以60% 1RM的训练可获得最佳训练效果,经过训练的受试者(初始值高)则以80% 1RM的训练可获得最佳训练效果;在训练安排上,未经训练的受试者3天/周,而经过训练的受试者2天/周的训练可获得最佳训练效果(Rhea et al.,2003)。BMI也可影响卧推1RM的训练效果,Meta分析显示,抗阻训练可同时改善肥胖青年人群的肌肉力量和BMI(Ribeiro et al.,2022),不同BMI受试者在抗阻训练后肌肉力量均得到提升,但BMI较高的人群在抗阻训练后肌肉力量的增加少于BMI较低的人群(Silva et al.,2021),这使得BMI可以作为预判因子对力量训练效果进行区分。肌肉质量是卧推1RM训练效果的另一个预测因子。初始肌肉质量可影响训练效果,有研究报道,肌肉质量相同的一批受试者在12周抗阻训练后,训练效果个体差异较小;而当肌肉质量不同时,再经12周抗阻训练后,训练效果的个体差异进一步扩大(Nunes et al.,2021)。7周的上肢抗阻训练(强度60%~70% 1RM),无论是快收缩,还是慢收缩形式都能显著增加肱二头肌的厚度(Kojic et al.,2021)。12周的抗阻训练(深蹲+卧推)可以显著增加股直肌、股外侧肌和肱三头肌的肌肉厚度(Gavanda et al.,2020)。
本研究中,基于7个SNPs计算的GPGS被纳入预测模型。我们前期的研究发现,采用表型指标建立的预测模型能够解释卧推1RM训练效果个体差异,但仅能解释卧推1RM训练效果的42.5%,提示表型指标不是影响训练效果的唯一因素(梅涛 等,2021)。但目前多数研究将基因组和表型分别研究,鲜见联合基因组-表型指标对抗阻训练效果进行预测。GPGS的纳入使得模型预测能力提高,对于精准化健身指导方案的制定具有实际应用意义。根据模型计算个体的预测值,如大于阈值,提示该运动方案能够取得较好的训练效果,反之则需更换其他训练方案。
3.4 卧推1RM训练效果相关SNPs的生物信息学分析
卧推1RM训练效果相关SNPs可通过多条信号通路影响骨骼肌生长发育过程,这可能是SNPs影响卧推1RM表型的潜在分子机制。REACTOME PATHWAY分析显示,7个SNPs调控的基因涉及多个信号通路,其中rs72959395、rs2673431、rs1052240、rs112183859直接或间接参与Myogenesis信号通路的调节。Myogenesis信号通路与骨骼肌生长发育关系密切,也是Developmental Biology通路的一部分。Myogenesis信号通路涉及由生长因子信号传导介导的肌细胞增殖、细胞分化、细胞重组形成肌管和细胞融合等过程(Bentzinger et al.,2012;Chal et al.,2017)。与该信号通路相关的转录因子MYOD1、MYOG、MEF2A、MEF2B、MEF2C、MEF2D、TCF3、TCF4、TCF12、MYF5、MYF6控制骨骼肌发育过程中肌源性分化(Zammit,2017)。在细胞培养中,Mef2与MyoD家族成员在激活基因表达和将成纤维细胞转化为成肌细胞方面具有协同作用(Taylor et al.,2017)。人体实验发现,抗阻训练可增加肌肉横截面积以及MyoD和MyoG的表达水平(Angleri et al.,2022)。在过度抗阻训练的动物模型中,可观察到肌纤维面积、MyoD、MyoG表达水平的下降(Souza et al.,2014),提示MyoD、MyoG的表达对肌肉质量、力量表型有积极影响。MRF4-MEF2轴可控制骨骼肌质量,成人骨骼肌中MRF4基因的缺失会导致肌肉肥大并防止萎缩,这种效应是由促进肌肉生长的MEF2因子介导的,MRF4充当MEF2活性的抑制因子(Schiaffino et al.,2018)。研究发现,单次抗阻训练后,人类骨骼肌中的MYOD、MYOG和MRF4mRNA水平短暂升高,进而可能通过MRF4-MEF2轴参与骨骼肌肥大、纤维型转变的调节(Minnock et al.,2020;Psilander et al.,2003)。在肌肉细胞中,TCF3/E2A蛋白与肌肉调节因子(如MyoD)形成异二聚体,然后与DNA结合,并调节对肌肉分化至关重要的靶基因的转录(Sun et al.,2007)。TCF4在肌细胞中高度表达,并调节肌肉再生,有助于运动诱导肌纤维损伤的重建(Kanazawa et al.,2021)。在肌源性分化过程中,MyoD1/TCF12可形成异二聚体诱导肌细胞分化(Obikane et al.,2021),TCF12参与肌细胞增殖和分化的过程(Gao et al.,2021)。MYF5、MYOD和MYOG基因的表达水平可反映早期肌生成。研究发现,12周70%~85% 1RM的全身抗阻训练能够显著提高股外侧肌MYF5、MYOD和MYOGmRNA表达(Caldow et al.,2015)。16周的抗阻训练后,年轻和老年受试者股外侧肌MYOD和MYOGmRNA表达增加,Myf6蛋白表达增加,MYF5蛋白仅在年轻受试者股外侧肌中增加(Kosek et al.,2006)。以上研究提示,Myogenesis信号通路中的大多数基因同时也受抗阻训练的影响。
其他信号通路,例如FOXO-mediated transcription(MURF1、HDAC2、TRIM63、BCL6、SIN3A、FOXO4、FOXO3、FOXO1)、PPARA activates gene expression(RXRA、SP1、NR1H2、NR1H4、RORA、PPARG、PPARA、PPARGC1B)、MAP kinase activation(MEF2A、MEF2C、JUN、FOS、ELK1、NFKB1)可能也参与了骨骼肌生长发育的调节(Pietrosemoli et al.,2017;Santos et al.,2015;Yang et al.,2022),如FOXO1、MURF1可介导骨骼肌质量的调节(Vilchinskaya et al.,2022)。
3.5 研究局限性
较大样本量的GWAS分析有利于减少群体分层,消除遗传背景带来的假阳性,因此样本量较小是本研究的局限性之一,也是国内外研究同样面临的难题。但本研究通过将PCA纳入协变量,对群体分层进行了控制,并采用膨胀系数对GWAS结果进行了质控,研究结果准确可靠。首次筛选7个遗传标记解释卧推训练效果个体差异的39.6%,基于GWAS的训练效果预测模型为制定提升卧推1RM的精准化健身指导方案提供了依据。
4 结论
首次基于GWAS筛选出rs79726572、rs112183859、rs77187527等7个与卧推1RM训练效果相关的遗传标记。建立的基因组-表型综合预测模型对卧推1RM训练效果具有较好的预测能力。卧推1RM训练效果相关的SNPs与骨骼肌生长发育过程相关,其中Myogenesis信号通路是特别值得注意的一条通路。
