不同养殖密度虹鳟幼鱼生长和肌肉转录组比较分析
2023-09-27朱敏杰李爱平简少卿赵大显
朱敏杰,曾 起,程 超,肖 敏,李爱平,简少卿,赵大显
( 1.南昌大学 生命科学学院,江西省水产动物资源与利用重点实验室, 江西 南昌 330031;2.井冈山农业科技园,江西 吉安 343000;3.井冈山市竹佬总冷水鱼养殖专业合作社,江西 吉安 343000 )
虹鳟(Oncorhynchusmykiss)属硬骨鱼纲鲑科太平洋鲑属,是我国重要的冷水性经济鱼类,2020年国内鳟鱼产量达3.78×104t[1]。随着市场需求量增大和养殖产业发展,高密度集约化人工养殖迅速兴起,而这种养殖模式也导致养殖过程中疾病的发生,严重制约了该产业的健康发展。
在集约化水产养殖中,养殖密度被认为是影响鱼类生长性能和养殖产量的重要因素之一[2]。随着养殖密度增大,鱼体生长率、存活率和食物利用率都有所下降[3-5];养殖密度的增大,除降低养殖鱼体的生长性能外,还会引起鱼体氧化应激反应[4]、皮质醇水平变化[6]以及鱼体拥挤胁迫相关基因表达水平发生改变[7]等;而低放养密度由于空间利用效率低下,也可能导致更高的生产成本和利润率下降。因此,适宜的养殖密度对水产动物健康养殖至关重要。
由于鱼类在生态和经济方面的重要性,利用功能基因组学通过基因调控来研究鱼类和非生物环境之间的相互作用关系越来越受到广泛关注[8-12]。随着转录组测序技术在水产研究领域的广泛应用,其也被针对性地用于探讨鱼体受养殖密度诱导的应激反应的调节机制[13-14]。近年来,基于二代高通量测序平台的RNA-seq技术,可提供大量的序列和丰度信息,逐渐替代了微阵列技术,成为转录组分析更好的方法[15]。与传统的基因芯片技术相比,RNA-seq技术在注释编码和非编码基因上有较大优势,它不需要知道目标物种的基因信息,就可以对多数生物体的转录水平进行分析。母尹楠[16]利用RNA-seq技术研究了感染嗜水气单胞菌(Aeromonashydrophila)的大黄鱼(Larimichthyscrocea)转录组,分析得到了1996个差异表达基因(DEGs),包括1133个上调表达基因和863个下调表达基因,其中727个差异表达基因显著上调,489个差异表达基因显著下调,这些基因分布在Toll-like通路、JAK-STAT通路、MAPK通路以及参与T细胞受体信号通路中;Schunter等[17]采用RNA-seq技术研究德氏三鳍鳚(Tripterygiondelaisi)交配策略中雄性二态性的分子特征,结果发现,两种雄性表型间差异表达基因数目比雌雄表型间差异表达基因还多;RNA-seq技术还被用于识别大西洋鲑(Salmosalar)肌肉组织中的miRNA,作为毒理学应激的潜在生物标志物[18]。因此,RNA-seq技术在鱼类差异表达基因的筛选和候选基因的发掘上发挥重要功能。
笔者以3种养殖密度条件下的虹鳟幼鱼为试验对象,通过比较分析其生长指标变化,并采用RNA-seq高通量测序技术进行测序和分析,筛选获得养殖密度和生长应激响应之间的关联基因信息,为进一步开展虹鳟健康养殖和基因组学研究提供参考数据,也为进一步深入挖掘和开发利用虹鳟功能基因提供科学依据。
1 材料与方法
1.1 试验设计
养殖试验在井冈山市竹佬总冷水鱼养殖专业合作社进行,所有的虹鳟均由同一批受精卵孵化而来,采用室内流水养殖模式进行试验。试验用鱼均来自同一个养殖池(记为来源池),经人工捕捞、筛选出规格一致的虹鳟幼鱼,放养在规格一致、底面积为1 m2的9个养殖网箱中。试验水温17~21 ℃,pH 6.5~7.5,出水口水流速度为1 kg/s,池内水深0.4 m。
试验用虹鳟幼鱼初始平均体质量(4.66±0.26)g,初始平均体长(5.98±0.16)cm。试验设置3个养殖密度:6.99、4.66、2.33 kg/m3,比例为3∶2∶1。按养殖密度设为低密度组、中密度组、高密度组,每组设置3个平行。低密度组每池放养200尾,中密度组每池放养400尾,高密度组每池放养600尾。试验鱼体健康,规格基本一致。试验自2019年8月6日开始,每隔8 d从每个网箱中取30尾幼鱼进行体长和体质量的测量,养殖32 d后从每个网箱中取3尾虹鳟幼鱼肌肉组织混样作为1个样品,3个密度组每组3个平行,共9个样本进行转录组测序。
1.2 RNA提取
各组样品使用TRIzol©试剂(Invitrogen)并按照说明书操作提取总RNA,使用DNase I (TaKaRa)去除基因组DNA,使用Nanodrop ND-2000分光光度计(美国赛默飞)、Aglient 2100 分析仪器对总RNA的纯度、浓度和完整性进行检测。RIN值>7的RNA用于下游试验。
1.3 mRNA-seq文库构建和Illumina测序
使用NEBNext UltraTM RNA Library Prep Kit(NEB,美国)试剂盒生成测序文库。利用带有Oligo(dT)磁珠纯化mRNA,随机六聚体引物和M-MuLV逆转录酶合成第一链cDNA,DNA聚合酶I和RNase H合成第二链cDNA,AMPure XP系统(美国贝克曼库尔特有限公司)对文库片段进行纯化;然后使用USER酶(NEB,美国) 与大小合适的接头连接cDNA,37 ℃孵育15 min,95 ℃孵育5 min后进行PCR扩增并对产物进行纯化,利用Agilent Bioanalyzer 2100系统对文库质量进行评价,合格后使用Illumina-HiSeq 4000平台进行测序。
1.4 数据处理与分析
1.4.1 生长数据处理与分析
以每隔8 d取样称量获得的体长、体质量作为生长指标,按下式计算生长性能指标。
wWGR=(m2-m1)/m1×100%
RSG=(lnm2-lnm1)/t×100%
CF=100m/L3
式中,wWGR为质量增加率(%),RSG为特定生长率(%/d),CF为肥满度,m1为初始体质量(g),m2为终末体质量(g),t为养殖时间(d),m为体质量(g),L为体长(cm)。
试验数据均以平均值±标准误表示,采用Excel和SPSS 21.0软件进行单因素方差分析。
1.4.2 测序数据产出和质量控制
基于高通量测序平台IlluminaHiseq 4000对cDNA文库进行测序,通过Perl脚本对产出的原始数据进行处理,同时计算处理后的有效数据、Q20、Q30、GC含量和序列重复水平。
1.4.3 转录组数据和参考基因组序列比对
利用Hisat2软件 (http://ccb.jhu.edu/software/hisat2/index.shtml,参数:-p 24-dta-x-1-2-S) 对参考基因组进行比对,再利用StringTie软件(http://ccb.jhu.edu/software/stringtie/,参数:-p 24-G-o-l)进行组装和定量。
1.4.4 新基因的挖掘
基于参考基因组序列,将拼接好的序列与原有的基因组注释信息比对,寻找原来未被注释的转录区,发掘该物种性的转录本和新基因,补充和完善基因组注释信息。使用BLAST软件 (http://blast.ncbi.nlm.nih.gov/Blast.cgi,参数:-query-db-out-outfmt 6-max-target-seqs 1) 将新基因与数据库比对进行注释。
1.4.5 基因功能的注释
基因功能的注释基于Nr、Nt、Pfam、KOG/COG、Swiss Prot、KEGG、GO数据库。
1.4.6 差异表达分析
采用DESeq2软件(http://www.bioconductor.org/packages/release/bioc/html/DESeq.html)分析样品组间的差异表达。将差异倍数≥1.5且P<0.05作为筛选标准,对每组差异基因进行维恩图的绘制。
1.4.7 差异表达基因GO富集分析
GO数据库是一个结构化的标准生物学注释系统。GO注释系统是一个有向无环图,包括三个主要分支:生物学过程、分子功能和细胞组分。
1.4.8 差异表达基因KEGG通路富集分析
根据差异表达基因进行KEGG富集分析,并选取q值最小的前20个通路进行分析。
1.4.9 生长与应激相关基因的筛选
根据转录组测序结果,结合相关文献报道,筛选与生长和应激相关的基因。
2 结 果
2.1 养殖密度对虹鳟幼鱼生长的影响
养殖32 d后,低密度组终末体质量、质量增加率、特定生长率和肥满度均大于中密度组和高密度组,但3个组别之间上述4个生长指标数据间差异不显著(P>0.05)(表1)。
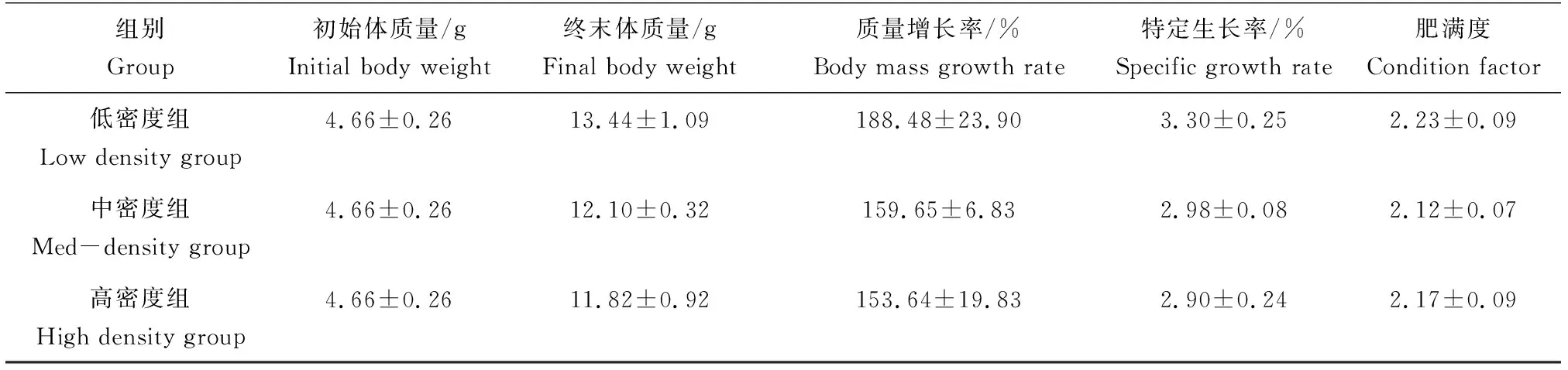
表1 不同养殖密度中虹鳟幼鱼生长参数统计
2.2 测序结果的产出及质控
本试验完成3个密度组(低密度组、中密度组、高密度组),每个密度组3个平行,共9个样本的转录组测序,共得到数据67.41 Gb,各样品的数据均达到5.99 Gb以上。Q20和Q30碱基百分比分别在98.07%和94.72%以上,各密度组GC碱基占总碱基平均百分比为:低密度组51.07%、中密度组51.33%、高密度组51.08%。有效数据总数(取各密度组3个平行组的有效数据平均值)为低密度组2.694×107、中密度组2.478×107、高密度组2.327×107(表2)。

表2 测序数据统计
2.3 转录组数据与参考基因组比对
将本试验转录组获得的有效数据与虹鳟参考基因组进行比对发现,各样品的序列与参考基因组的比对效率为91.67%~92.77%,比对效果大于70%。另外,单次匹配的比对率为79.26%~80.06%,多次匹配的比对率为11.82%~13.37%(表3)。

表3 样品测序数据与虹鳟参考基因组的序列比对结果统计
2.4 新基因的分析
基于虹鳟基因组序列作为参考基因组,使用StringTie软件对Mapped Reads进行拼接,并与原有的基因组注释信息进行比较,寻找原来未被注释的转录区,发掘虹鳟的新转录本和新基因,从而补充和完善原有的基因组注释信息。过滤掉编码的肽链过短(少于50个氨基酸残基)或只包含单个外显子的序列,共发掘5501个新基因,其中4121个基因得到注释,1380个基因未得到注释(表4)。

表4 新基因功能注释结果统计
2.5 差异表达分析
使用DESeq2软件对虹鳟养殖高、中、低密度组转录组两两比较进行差异表达分析(FDR<0.05)发现:低密度组与高密度组相比出现2760个差异表达基因,其中1279个差异表达基因上调,1481个差异表达基因下调;低密度组与中密度组相比有654个差异表达基因,其中307个上调,347个下调;中密度组与高密度组相比有2316个差异表达基因,其中1226个上调,1090个下调(表5)。
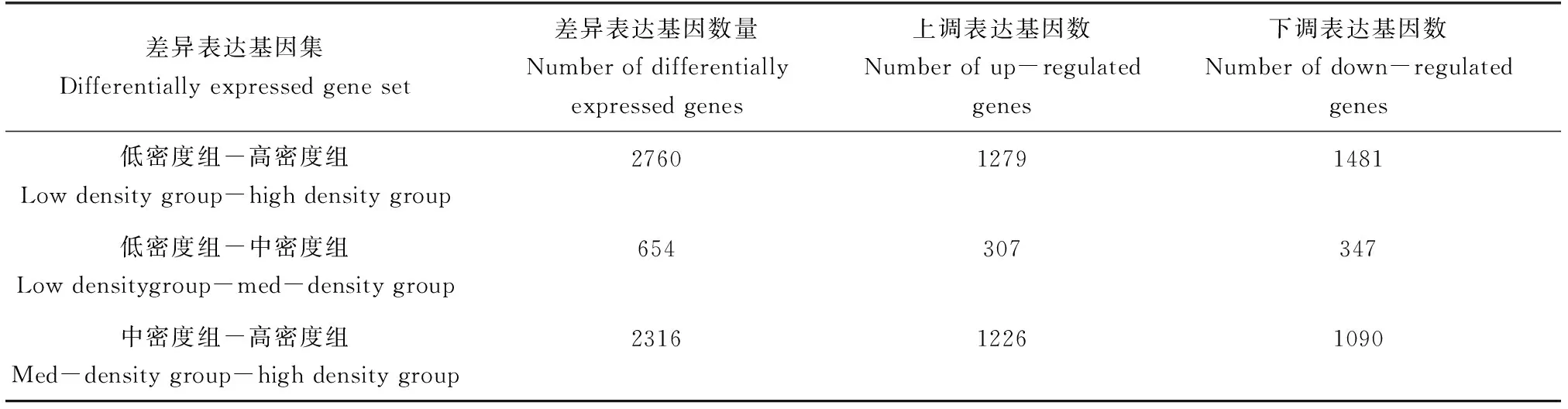
表5 差异表达基因数目统计
将每组差异基因绘制维恩图发现,共4030个非冗余差异表达基因中的38个是3个比较组之间共有的(图1)。
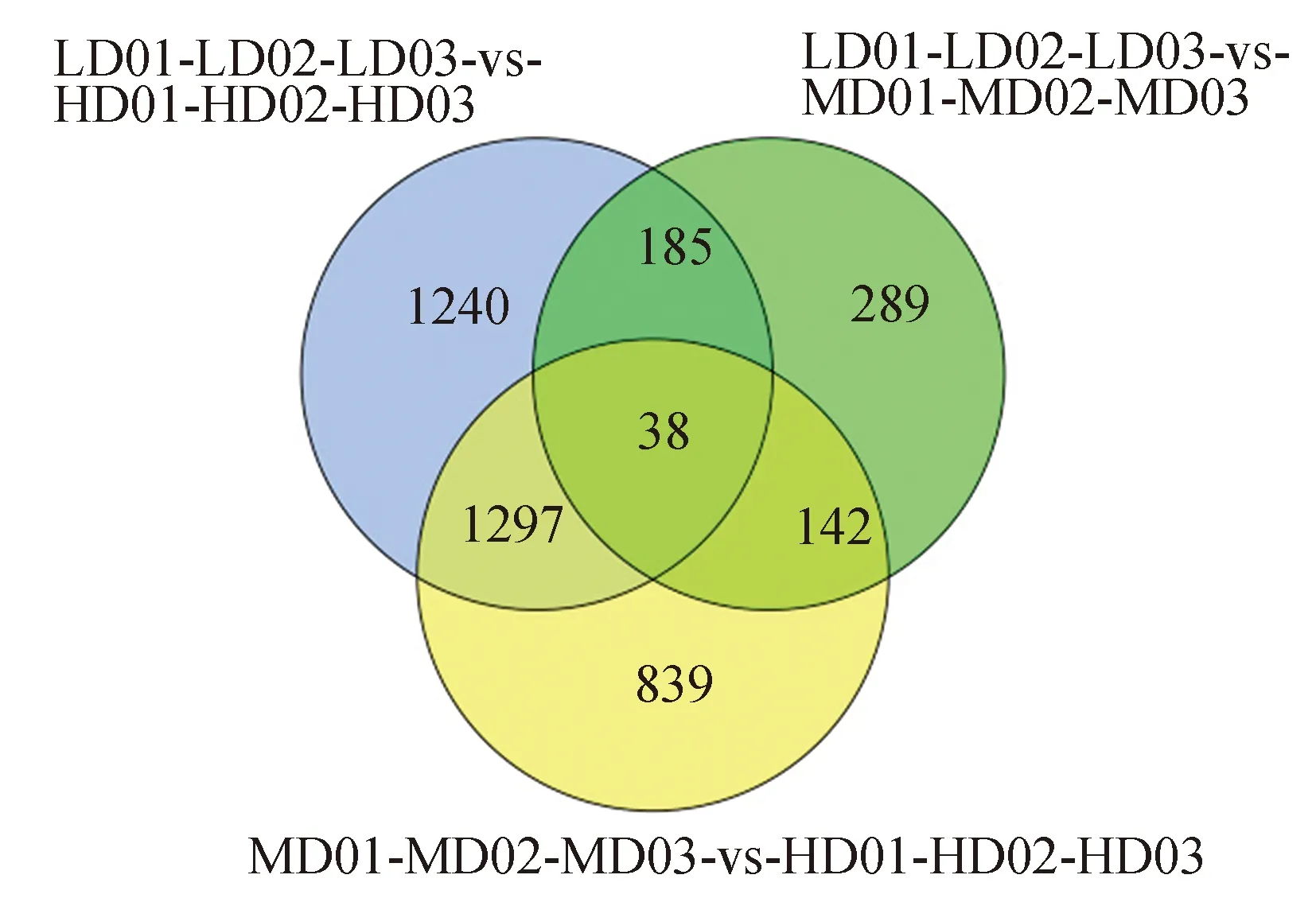
图1 差异表达基因维恩图Fig.1 Venn diagram of differentially expressed genes
2.6 差异基因GO富集分析
对上述差异表达基因进行GO分类发现,低密度组与高密度组比较共有1943个差异表达基因获得了GO注释,包括823个上调和1120个下调的差异表达基因。归类到GO 3大本体的59个子类别中,其中:生物学过程主要由代谢过程(741个)、细胞过程(950个)、单有机体过程(710个)和生物调控(559个)组成;细胞组分主要由细胞(920个)、膜(530个)、膜部件(463个)、细胞组分(909个)和细胞器(649个)组成;分子功能主要由催化活性(706个)和结合(1067个)组成(图2a)。
低密度组与中密度组比较共有438个具有显著性差异表达基因获得了GO注释信息,其中包括191个上调和247个下调的差异表达基因。代谢过程(131个)、细胞过程(172个)、单有机体过程(160个)和生物调控(157个)占据着生物学过程涉及的大部分差异表达基因;细胞组分主要由细胞(194个)、膜(151个)、膜部件(137个)、细胞组分(188个)和细胞器(137个)组成,分子功能主要由催化活性(135个)和结合(232个)这两个子类别组成(图2b)。
中密度组与高密度组进行比较共有1602个具有显著性差异表达基因获得了GO注释信息,其中包括811个上调和791个下调的差异表达基因。生物学过程中有611个差异表达基因参与代谢过程,755个差异表达基因参与细胞过程,586个差异表达基因参与单有机体过程,426个差异表达基因参与生物调控;细胞组分主要由细胞(680个)、膜(480个)、膜部件(439个)、细胞组分(667个)和细胞器(440个)组成,分子功能主要由催化活性(593个)和结合(844个)组成(图2c)。
通过进一步分析发现,低密度组与高密度组以及中密度组与高密度组的差异表达基因都主要富集在DNA整合、DNA介导的转位(生物学过程),褶皱(细胞组分)和细胞骨架的结构组成(分子功能),低密度组与中密度组的差异表达基因主要富集在DNA整合、DNA介导的转位(生物学过程),褶皱(细胞组分)和DNA结合、氧化还原酶活性作用于成对供体结合或重组(分子功能)过程(图2)。
2.7 差异基因KEGG富集通路分析
通过KEGG富集通路分析发现,低密度组与高密度组进行比较,920个差异表达基因定位到212个特定的KEGG代谢途径。低密度组与中密度组相比较,196个差异表达基因定位到112个特定的KEGG代谢途径;中密度组与高密度组进行对比,743个差异表达基因定位到189个特定代谢途径。
选取q值最小的20个通路进行分析发现:低密度组与高密度组相比,差异表达基因显著富集到RNA转运、剪切体、DNA复制和真核生物中核糖体合成4个通路(图3a);低密度组与中密度组相比,差异表达基因显著富集到紧密连接通路(图3b);中密度组与高密度组相比,差异表达基因显著富集到剪切体,甘氨酸、丝氨酸和苏氨酸代谢,氨基酸的生物合成和真核生物中核糖体合成4条信号途径(图3c)。
2.8 生长与应激相关基因的筛选
选择P值小且差异倍数大的差异表达基因,并根据相关文献报道进行筛选。从低密度组-高密度组中筛选获得丝氨酸/苏氨酸蛋白磷酸酶pp1-β催化亚基、胰岛素受体2、组蛋白结合蛋白4、无翅型MMTV整合位点家族成员2BA、肌肉骨骼-胚胎核蛋白1b、V型质子ATP酶亚基e1等与生长相关的差异表达基因及颗粒蛋白前体、整合素alpha-V、转换B细胞复合物亚基、组织蛋白酶、热休克蛋白等与生长相关的差异表达基因;从低密度组-中密度组分组中筛选获得肌肉生长抑制素、无翅型MMTV整合位点家族成员2BA等与生长相关的差异表达基因及与应激相关的视黄醛脱氢酶2等差异表达基因;从中密度组-高密度组组中筛选获得生长相关的差异表达基因,包括骨形态发生蛋白2、骨形态发生蛋白5、转化生长因子β-2前蛋白、肌肉生长抑制素B、组蛋白结合蛋白4(表6)。
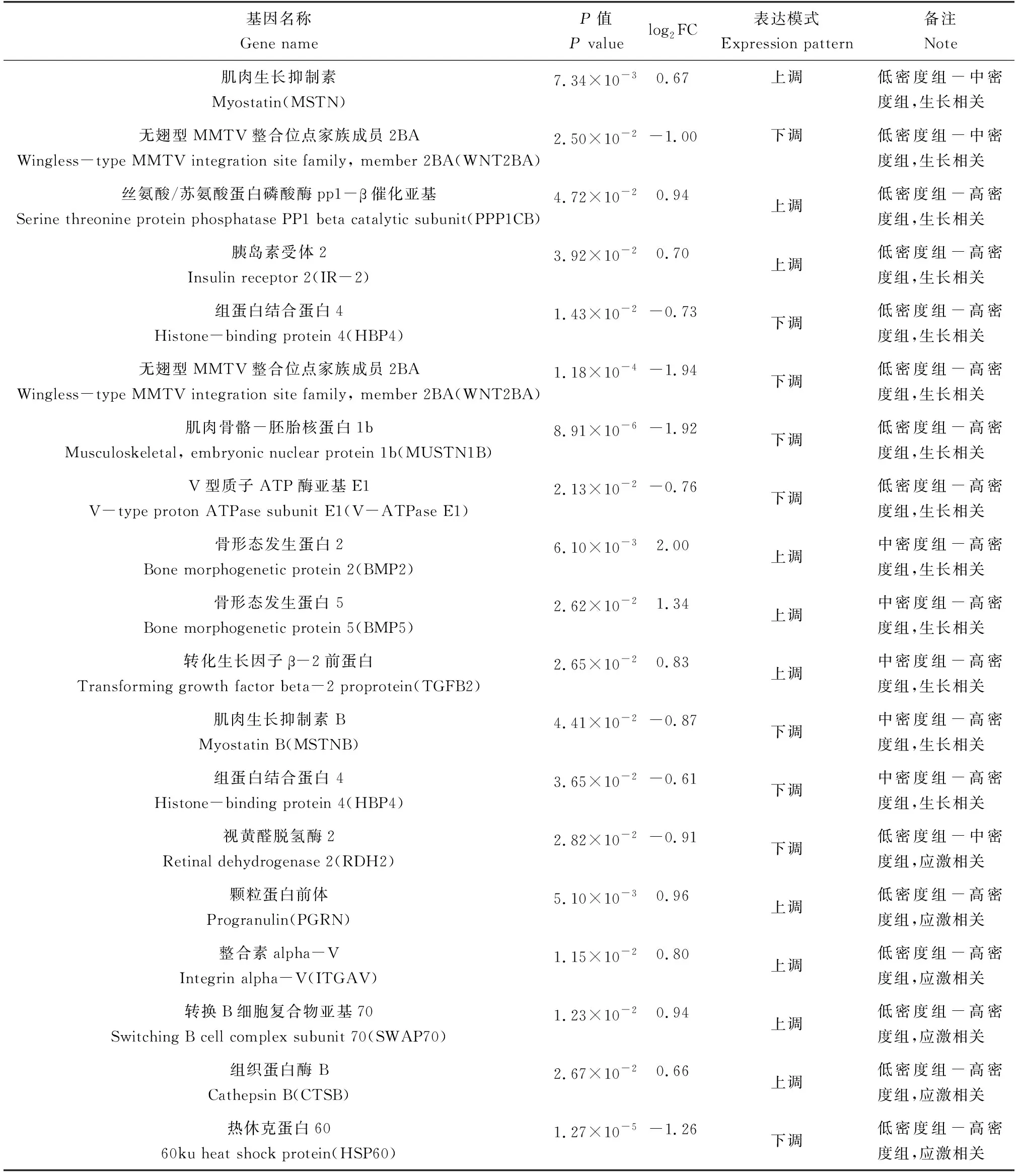
表6 筛选获得的与生长和应激相关的差异表达基因
3 讨 论
3.1 养殖密度对虹鳟幼鱼生长的影响
养殖密度作为水产健康养殖的一个重要因子,对鱼类生长性能产生重要影响。目前,关于养殖密度影响鱼类生长的机制还存在着争议。普遍认为在高密度环境生活的鱼类中,受到多种因素的影响,这些影响因素单独或者叠加地影响鱼类个体的生长,使得养殖密度对生长机制研究变得更为复杂。有些鱼类随着养殖密度的增加,其体质量增长(率)均不断降低,如地图鱼(Astronotusocellatus)[19]、大菱鲆(Scophthalmusmaximus)[20]、施氏鲟(Acipenserschrenckii)[21];有些鱼类高密度养殖条件下生长速度和饲料利用率更好,如大西洋白姑鱼(Argyrosomusregius)[22]等;有些鱼类养殖密度超过临界范围,其生长和存活不受密度的影响,如达氏鳇(Husodauricus)[23]、瓦氏海马(Hippocampuswhitei)[24]稚鱼和塞内加尔鳎(Soleasenegalensis)[25]等。笔者发现,随着养殖密度的增加,虹鳟幼鱼体质量增长率等生长指标均有不同程度的降低,虽然这种影响未呈现显著性差异(P>0.05),但从转录组分析结果可以发现,一些与生长和应激相关基因表达模式在不同养殖组之间发生了显著性变化,同时经过初步分析还发现,高密度养殖条件下虹鳟幼鱼抗氧化酶活性与低、中密度组之间存在显著差异(数据未列出),因此,综合分析可以推测,随着养殖时间的延长,高密度养殖条件下虹鳟幼鱼的生长受到负面影响。
3.2 不同养殖密度条件下虹鳟幼鱼转录组比较分析
为进一步阐明养殖密度对虹鳟幼鱼生长性能影响的分子机制,笔者通过RNA-seq技术对3个不同养殖密度虹鳟幼鱼肌肉组织进行了高通量转录组测序,对数据进行过滤和质控后,通过组装、拼接和转录本功能注释,得到67.41 Gb 的转录组有效数据信息,与其他淡水鱼类如江鳕(Lotalota)[26]、黄尾鲴(Xenocyprisdavidi)[27]、达氏鳇[28]、花斑裸鲤(Gymnocypriseckloni)[29]、乌鳢(Channaargus)和斑鳢(C.maculatus)[30]一样都处于较高水平。这些数据的积累为今后开展虹鳟基因组学、免疫学以及健康养殖提供了参考资料。
从转录组质量来看,大部分碱基质量打分能达到或超过Q30。通常Q30值在80%以上则可认为测序质量非常可靠,本试验9个样品中Q30值最低为94.72%,最高为95.62%,均大于90%,表明笔者利用RNA-seq技术构建的转录组数据库准确可靠,可用于后续虹鳟基因克隆及功能研究。
通过过滤掉编码的肽链过短(少于50个氨基酸残基)或只包含单个外显子的序列,笔者共发掘了5501个新基因,其中4121个基因得到注释,1380个基因未得到注释。这些基因的发现进一步补充和完善了虹鳟原有的基因组数据,对这些新基因的深入研究将有助于阐明养殖密度与虹鳟幼鱼响应的分子机制。
3.3 生长与应激相关基因在虹鳟幼鱼生长中的功能
对转录组进行GO和KEGG分析,并结合相关文献报道,人工筛选获得了19条与生长和应激相关的基因序列,其中:肌肉生长抑制素和肌肉生长抑制素B在鱼类早期发育、肌肉生长和免疫系统中发挥重要作用[31-33];无翅型MMTV整合位点家族成员2BA调控鱼类卵巢分化和发育[34];草鱼(Ctenopharyngodonidella)进食高淀粉饲料后,丝氨酸/苏氨酸蛋白磷酸酶pp1-β催化亚基可以使过量的碳水化合物转换为糖原[35];胰岛素受体2广泛分布于各个组织,在生长发育过程中有重要作用[36];肌肉骨骼-胚胎核蛋白1b是一种小型的核蛋白,参与了肌肉和骨骼的发育和再生[37];骨形态发生蛋白2和骨形态发生蛋白5参与了几个重要的胚胎过程和成脂前体细胞的诱导分化[38],骨形态发生蛋白5在1龄的团头鲂的肌间骨中广泛高表达[39];转化生长因子β-2前蛋白可作为间充质祖细胞和巨噬细胞(如破骨巨噬细胞[40]破骨细胞[41]、软骨细胞[42])的形态发生诱导因子[43];颗粒蛋白前体具有营养和抗炎活性[44];整合素alpha-V影响着身体和内脏的左右不对称[45];组织蛋白酶B参与了细菌感染和接种过程中的宿主免疫应答[46];热休克蛋白60可以在先天免疫系统细胞中引发强效的促炎反应,被认为是细胞受到压力或受损的危险信号[47];转换B细胞复合物亚基70作为外周肌动蛋白笼与吞噬体连接支架有着关键作用[48]。这些基因在不同养殖密度条件表达模式发现显著变化,说明它们在虹鳟幼鱼应对不同的养殖密度中发挥重要功能。这些基因的发现为进一步深入研究养殖密度对虹鳟生长性能影响的分子机制提供了基础数据,也为确定生产上适宜的养殖密度提供了参考指标。
4 结 论
本试验结果显示,随着养殖密度的增加,虹鳟幼鱼质量增加率等生长指标呈现降低的趋势。通过RNA-seq技术共获得了3个养殖密度条件下虹鳟幼鱼肌肉组织67.41 Gb转录组有效数据,通过序列比对共发掘5501个新基因,其中4121个基因得到注释,1380个基因未得到注释。差异表达基因分析发现:低密度组与高密度组以及中密度组与高密度组的差异表达基因都主要富集在DNA整合、DNA介导的转位(生物学过程)、褶皱(细胞组分)和细胞骨架的结构组成(分子功能),低密度组与高密度组进行比较,920个差异表达基因定位到212个特定的KEGG代谢途径,中密度组与高密度组进行对比,743个差异表达基因定位到189个特定代谢途径;低密度组与中密度组的差异表达基因主要富集在DNA整合、DNA介导的转位(生物学过程),褶皱(细胞组分)和DNA结合、氧化还原酶活性作用于成对供体结合或重组(分子功能)过程,196个差异表达基因定位到112个特定的KEGG代谢途径。筛选获得了19条与生长和应激相关的基因序列。此试验结果可为开展鲑科鱼类组学研究提供了基因资源库,也可为今后虹鳟功能基因的挖掘和健康养殖发提供理论依据。
