芋可溶性淀粉合成酶CeSS基因家族的克隆和表达分析
2023-09-15殷剑美韩晓勇郭文琦张培通
王 立, 殷剑美, 韩晓勇, 蒋 璐, 郭文琦, 金 林, 张培通
(江苏省农业科学院经济作物研究所,江苏 南京 210014)
淀粉是储存在叶片叶绿体或储存器官中的主要化合物,被用作种子休眠和植物生长的能量来源,也是人类和动物最重要的能量来源,并被广泛用作工业生产的原料。植物主要合成支链淀粉和直链淀粉,两者的结构和比例决定了淀粉的理化特性,如溶胀、溶解性、可塑性和黏度等[1-2]。
研究结果显示,可溶性淀粉合成酶(SSS)通过催化形成α-1,4糖苷键延长合成具有不同长度的支链淀粉,决定支链淀粉的链条长度和链长分布,从而影响支链淀粉的理化性质以及淀粉粒的结构和品质[2-3]。SSS主要存在于植物叶片和贮藏组织,主要分为4个亚家族,即SSI、SSII、SSIII和SSIV,大多存在于质体基质中。SSI主要参与支链淀粉的短链产生,SSII和SSIII参与支链淀粉的链条延伸[3]。研究结果显示,SSI在马铃薯块茎淀粉合成中的酶活性较弱,而在水稻和玉米胚乳中具有较强的活性;下调SSII的转录水平会影响马铃薯块茎中的链长分布,改变淀粉结构和其他淀粉合成酶活性[2,4-5]。此外,SSS每个亚家族又含有同工酶,但是这些同工酶如何影响植物中合成的支链淀粉的数量和组成尚待阐明。目前关于芋可溶性淀粉生物合成的研究相对较少,对于淀粉合成酶的功能尚不清楚。
本研究在靖江香沙芋转录组测序基础上,克隆获得3个芋可溶性淀粉合成酶CeSS家族基因,通过分析氨基酸序列、蛋白质结构和亲缘进化关系,检测3个基因的转录表达谱,探索其与淀粉丰度形成的关系,从而加快芋可溶性淀粉合成机理的深入研究。
1 材料与方法
1.1 试验材料
供试材料为靖江香沙芋, 种植于江苏省农业科学院六合试验基地。分别在种植60 d、90 d、120 d、150 d和180 d取样。地上部组织取样部位为叶片和叶柄,地下部组织取样部位为母芋、子芋和根。叶片打孔取样,叶柄切段取样,球茎去皮取样,根洗去杂质取样,统一贮存于-80 ℃冰箱。每个组织取样质量约1.5 g,设3份重复。
1.2 cDNA合成
采用 RNAprep Pure Plant Kit[天根生化科技(北京)有限公司产品]提取样品RNA,cDNA合成采用宝生物工程(大连)有限公司PrimeScript RT reagent Kit和RNA PCR Kit,试验方法参照试剂盒说明书。
1.3 基因克隆
根据已获得的靖江香沙芋转录组数据,通过BLASTx方法比对获得CeSS基因信息,使用Primer Premier 5.0设计基因克隆引物和基因表达引物(表1)。PCR扩增方法参考王立等[6]的方法。PCR扩增产物送北京鼎国科技公司完成序列测定。

表1 CeSS家族基因特异引物
1.4 氨基酸序列和进化分析
采用DNAman完成氨基酸序列同源性比较,在MEGA v4.0中构建系统进化树。采用SMART在线数据库(http://smart.embl-heidelberg.de/)进行蛋白质结构域预测,在ExPaSy网站(http://web.expasy.org/compute_pi/)计算等电点和相对分子质量,采用Cell-PLoc 2.0 package预测亚细胞定位。
1.5 转录表达分析
以Actin基因为内参基因。采用SYBR PremixExTaqKit进行qRT-PCR检测,按照试剂盒说明书操作。定量RT-PCR反应体系为20 μl,程序如下:95 ℃ 30 s;每循环95 ℃ 3 s,60 ℃ 30 s,40个循环;最后95 ℃ 15 s,60 ℃ 1 min,95 ℃ 15 s,每个样品3次重复。参考Livak等[7]的方法计算基因相对表达量。
1.6 支链淀粉含量测定
采用何洁等[8]双波长法测定芋球茎中的支链淀粉含量。取播种后不同时期球茎干燥样品,质量为0.1 g,用1 ml无水乙醇+9 ml 1 mol/L KOH研磨,沸水浴加热溶解10 min,冷却后定容至100 ml。检测535 nm、757 nm下的吸光值,计算支链淀粉含量。
1.7 数据处理
使用 Microsoft Excel 2010 软件和 SPSS 17.0 软件进行试验数据统计分析。
2 结果与分析
2.1 芋可溶性淀粉合成酶家族基因克隆及序列分析
经过BLAST比对,共鉴定获得3个芋可溶性淀粉合成酶SS基因,分别命名为CeSSI、CeSSII、CeSSIII。CeSSI基因碱基序列全长为1 932 bp,编码643个氨基酸(图1),蛋白质相对分子质量和理论等电点分别为70 795和6.02(表2)。CeSSII基因碱基序列全长为2 409 bp,编码802个氨基酸(图2),蛋白质相对分子质量和理论等电点分别为88 702和5.75(表2)。CeSSIII基因碱基序列全长为3 606 bp,编码1 202个氨基酸(图3),蛋白质相对分子质量和理论等电点分别为136 248和5.47(表2)。蛋白质亲水性预测和亚细胞定位预测结果(表2)显示,3个CeSS蛋白均为亲水性蛋白质,且均定位在叶绿体。

图2 CeSSII氨基酸序列分析Fig.2 Amino acid sequence analysis of CeSSII

图3 CeSSIII氨基酸序列分析Fig.3 Amino acid sequence analysis of CeSSIII

表2 CeSS基因家族及其编码蛋白质的生化特性
2.2 芋可溶性淀粉合成酶氨基酸序列和进化分析
图4显示,3个SS蛋白的氨基酸序列比较相似,均含有Glyco_transf结构域,其中CeSSI和CeSSII蛋白含有1个Glyco_transf_5结构域(CeSSI,Asn135~Ser393;CeSSII,Asn312~Glu555)和1个Glyco_trans_1_4结构域(CeSSI,Asp448~Ile597;CeSSII,Leu608~Thr762),而CeSSIII仅含有1个Glyco_trans_4结构域(Gly770~Gly944),以及3个CBM_25结构域(Gly316~Glu402、Glu492~Pro583、Gly654~Thr745)。

图4 CeSS蛋白Pfam结构分析Fig.4 Pfam structure of CeSS protein
系统进化分析结果(图5)表明,3个SS蛋白聚类分为3组,表现出进化差异:CeSSI与海枣(Phoenixdactylifera, XP_008783178.1)中SS蛋白最接近,序列相似度为62.9%,其次是菜豆(Phaseolusvulgaris,BAD18845.1)和拟南芥(Arabidopsisthaliana, NP_197818.1),序列相似度分别为59.7%和59.4%;CeSSII与芦笋(Asparagusofficinalis,XP_020257302.1)中相应蛋白质较相近,序列相似度为56.3%,其次是菠萝(Ananascomosus,XP_020104470.1)和繁穗苋(Amaranthuscruentus,ABA64552.1);CeSSIII与药用稻(Oryzaofficinalis,AIU99454.1)SS蛋白相似度最高,序列相似度为55.5%,其次是玉米(Zeamays, AFW59498.1)和拟南芥(Arabidopsisthaliana,NP_172637.2)。

Amaranthus cruentus:繁穗苋;Ananas comosus:菠萝;Arabidopsis lyrata subsp. lyrata:拟南芥琴亚科;Arabidopsis thaliana:拟南芥;Asparagus officinalis:芦笋;Brassica napus:甘蓝型油菜;Capsella rubella:芥菜;Colocasia esculenta:芋;Elaeis guineensis:油棕;Glycine max:大豆;Ipomoea batatas:甘薯;Malus domestica:苹果;Manihot esculenta:木薯;Morus notabilis:川桑;Nelumbo nucifera:莲;Oryza officinalis:药用稻;Oryza sativa Japonica group:粳稻;Phaseolus vulgaris:菜豆;Phoenix dactylifera:海枣;Populus euphratica:胡杨;Prunus mume:梅花;Ricinus communis:蓖麻;Solanum lycopersicum:番茄;Theobroma cacao:可可;Triticum aestivum:小麦;Vitis vinifera:葡萄;Zea mays:玉米。图5 不同植物SS家族基因系统进化分析Fig.5 Phylogenetic tree analysis of SS family genes in different plant species
2.3 芋可溶性淀粉合成酶家族基因的表达水平分析
图6显示,芋可溶性淀粉合成酶家族的3个基因CeSSI、CeSSII、CeSSIII在芋的不同组织中均有表达,但在母芋和子芋中高表达,在根、叶片和叶柄中低表达,说明同一基因的组织表达谱差异较大;而在同一组织中,尤其是母芋和子芋中,CeSSII基因表达量最高,其次是CeSSI和CeSSIII。图7显示,在芋发育过程中,叶片中CeSSI、CeSSII、CeSSIII基因表达趋势基本不变,而球茎中的表达水平均高于叶片。球茎中的CeSSI、CeSSII基因在种植60 d后表达量开始提高,在种植150 d表达水平达到峰值,随后略有下降;而CeSSIII基因在种植60 d后在球茎中表达量开始上调,在种植120 d时表达增速最快,随后表达水平趋缓,在种植180 d时表达水平最高。
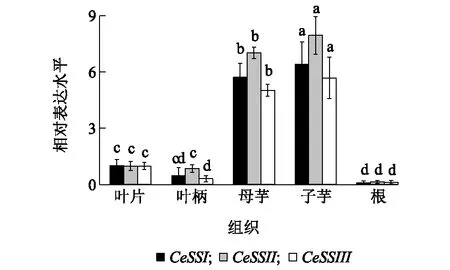
同一基因不同组织间不同小写字母表示差异显著(P<0.05)。图6 CeSSI、CeSSII和CeSSIII在不同芋组织中的表达Fig.6 Expression of CeSSI, CeSSII and CeSSIII in different tissues of taro
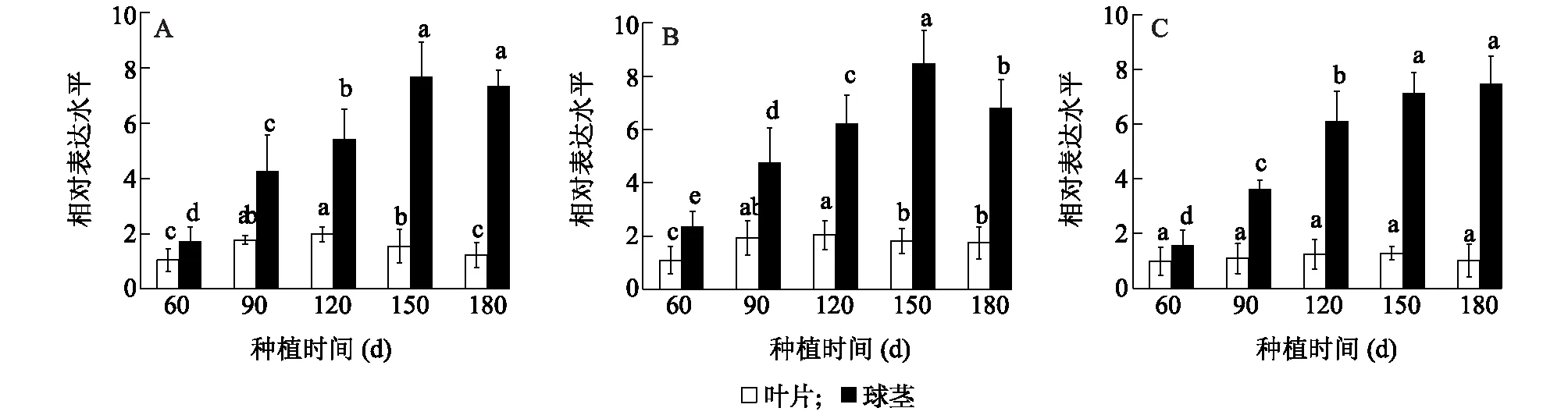
同一组织间不同小写字母表示差异显著(P<0.05)。图7 CeSSI (A)、CeSSII (B) 和 CeSSIII (C)在芋不同发育时期的表达Fig.7 Expression of CeSSI (A), CeSSII (B) and CeSSIII (C) at different developmental stages of taro
2.4 CeSSIII的基因表达与芋球茎支链淀粉含量相关性分析
图8显示,芋球茎支链淀粉含量呈持续增长趋势,在种植120~150 d增长速率最高,球茎支链淀粉含量在种植180 d达到较大值。相关性分析结果显示,芋球茎CeSSIII的表达水平与支链淀粉含量之间存在明显正相关性。
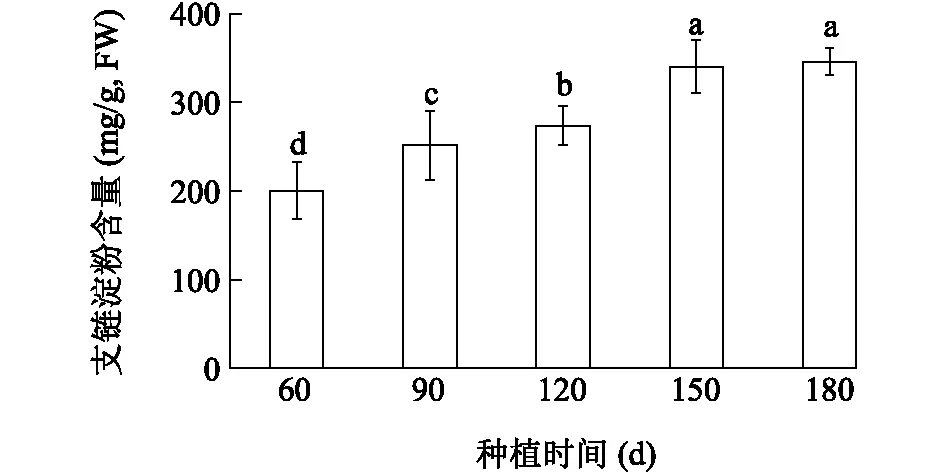
不同小写字母表示差异显著(P<0.05)。图8 不同发育时期芋球茎中支链淀粉含量分析Fig.8 Amylopectin contents in corms of taro at different development stages
3 讨 论
3.1 芋可溶性淀粉合成酶家族成员具有各自独特功能
在植物叶片和贮藏组织中已经发现了4种形式的淀粉合成酶:SSI、SSII、SSIII和SSIV[1,9]。在马铃薯上的研究结果显示,SSI和SSII是主要淀粉合酶,会影响淀粉含量和结构,SSIII是支链淀粉主要合成酶,其活性占块茎可溶性淀粉合成酶活性的80%[3]。本研究结果显示,靖江香沙芋中存在3个可溶性淀粉合成酶,均具有类似的Glyco_transf结构域,它们与腺苷二磷酸(ADP)葡萄糖结合,催化合成淀粉,而CeSSIII中除了同样的Glyco_transf结构域以外,还包含3个CBM_25结构域,具有淀粉结合特性,可能与淀粉链长有关[2]。前人研究发现,SSI氨基末端区域高度保守的KXGGL基序,是可溶性淀粉合成的起始酶,与糖原合成酶底物ADP-葡萄糖结合,可能对芋的短支链淀粉合成具有重要作用[10]。SSII可影响支链淀粉的分支合成,改变淀粉粒的晶体层结构和淀粉糊化温度[1]。SSIII中含有的六肽基序(RYGTVPVV)与14-3-3蛋白结合位点基序相关[1, 11],后者通过负调控拟南芥中可溶性淀粉合成酶活性,进而影响拟南芥叶片中淀粉积累,说明CeSSIII与芋淀粉储藏密切相关[2]。
3.2 3个芋可溶性淀粉合成酶基因与淀粉合成紧密相关
本研究结果表明,CeSSI、CeSSII和CeSSIII主要在芋球茎中表达,但在芋的不同发育阶段,3个基因表现出不同的表达模式。CeSSI和CeSSII在未成熟的芋球茎中表达水平较高,与马铃薯中的转录表达趋势相似,即在种植90 d左右明显上调表达,表明CeSSI和CeSSII更趋向于参与芋球茎中瞬时淀粉的合成[2, 5];而CeSSIII的转录上调时间相对较迟,在种植120 d时上调速率最高,在种植180 d的成熟球茎中表达水平最高,与在香蕉中的表达模式基本一致,说明CeSSIII更倾向于参与贮藏淀粉的合成[12]。
本研究发现,在芋发育过程中,芋球茎中支链淀粉含量持续增加,在种植180 d达到较大值,而且芋球茎中CeSSIII基因表达量与芋球茎中支链淀粉含量呈正相关,表明CeSSIII与芋球茎中淀粉贮藏有关。该结果与在马铃薯上的研究结果类似,马铃薯块茎中SSIII活性占总SS活性的80%,与贮藏淀粉的合成密切相关[3]。
本研究结果表明,芋可溶性淀粉合成酶基因家族的3个基因与球茎淀粉合成密切相关,其中CeSSI和CeSSII参与芋球茎中瞬时淀粉的合成,而CeSSIII更倾向于参与贮藏淀粉的合成。今后需要对芋可溶性淀粉合成酶基因进行深入研究,以明确和验证CeSSI、CeSSII和CeSSIII基因功能。
