聚乙烯降解菌白浅灰链霉菌LBX-2烷烃羟化酶基因SaalkB的克隆与表达分析
2023-09-10张榕麟高田蕊陈美菊何欣怡邵欢欢
张榕麟, 高田蕊, 陈美菊, 熊 爽, 何欣怡, 雍 彬, 邵欢欢
(四川师范大学 生命科学学院, 四川 成都 610101)
塑料因其具有强度大、重量轻、柔韧性好、易于生产和成本低廉等特点,被广泛应用于人类生活、工业生产等各个领域[1].据相关文献报道,全球塑料年产量约为3亿t[2],其中聚乙烯(polyethylene, PE)约占40%,是塑料生产的重要组成部分[3].然而废弃塑料造成的环境污染问题日益严重,随着时间推移,92.4%的塑料被风化分解为直径小于5 mm的微塑料,广泛分布于全球各地,对河流、湖泊、耕地等造成严重污染[4],且在海洋、淡水、土壤、陆地生物体内均发现了微塑料的存在,严重危害生物体健康[5].PE分子排列紧密、疏水性强、表面能低且化学性质稳定,因此在自然界中很难被降解,导致PE废弃物在环境中大量积累,如何彻底降解PE废弃物已经成为一个全球性难题[6].
生物降解PE是一种绿色环保、低能耗的降解方式.通过微生物降解PE将其无机矿化为CO2和H2O,该降解方式极有可能成为减少“白色污染”的有效途径之一[7].生物降解PE的第一步是PE氧化,由微生物产生胞外酶氧化PE长烃链断裂[8].研究发现烷烃羟化酶、漆酶以及过氧化物酶等多种酶类具有氧化降解PE的能力[9].Zhao等[10]发现过氧化物酶可以显著提高高密度聚乙烯薄膜的亲水性.Santo等[11]发现赤红球菌分泌的漆酶可降低低密度聚乙烯的分子量及增加酮羰基指数,认为可由漆酶催化打断聚乙烯长链.烷烃-1-单加氧酶(alkane-1-monooxygenase,AlkB)是烷烃羟化酶(alkane hydroxylase,AH)系统的催化组分,参与烃类代谢途径的第一步,通过末端或次末端氧化降解烃类低聚物.烷烃羟化酶系统包含3个组份,分别是1个非血红素铁的膜整合的单加氧酶(alkane-1-monooxygenase,AlkB)和2个电子转移蛋白红素氧还蛋白(rubredoxin,AlkG)、红素氧还蛋白还原酶(rubredoxin reductase,AlkT)[12].Jin等[13]通过实时荧光定量PCR检测烷烃羟化酶基因alkB表达量,以监测聚乙烯降解菌的丰度.Gyung等[14]将PE降解细菌铜绿假单胞菌E4的烷烃羟化酶基因alkB在大肠杆菌BL21中表达,经80 d堆肥生物降解将低分子量PE近20%的碳矿化为CO2,认为alkB基因在低分子量PE降解过程中发挥重要作用.Jeon等[15]克隆了PE降解细菌铜绿假单胞菌E7的烷烃羟化酶基因alkB并在大肠杆菌中表达,发现在含有低密度PE的培养基中转录水平增加了4倍,alkB重组菌株表现出对PE的高生物降解能力,而不含alkB的菌株对PE完全无降解作用,证实了alkB是降解聚乙烯不可或缺的基因.
本实验室前期从土壤中分离筛选得到一株能够降解聚乙烯的放线菌——白浅灰链霉菌LBX-2,该菌株对淀粉聚乙烯降解15 d的失重率可达26.54%[16].蛋白质组与代谢组数据表明SaalkB基因可能在白浅灰链霉菌LBX-2降解PE的过程中发挥重要作用.因此,本研究通过克隆白浅灰链霉菌LBX-2烷烃单加氧酶基因SaalkB,对SaalkB基因片段进行测序及序列同源性分析,并对其编码蛋白氨基酸序列、理化性质、信号肽等进行生物信息学分析.构建SaalkB基因的原核表达载体并进行重组蛋白的原核表达,通过AFM分析重组工程菌株对PE膜片的降解效果;通过荧光定量PCR分析SaalkB基因在聚乙烯降解过程中的表达情况;通过蛋白组数据分析AlkB蛋白在聚乙烯降解过程中的表达情况.本研究可为深入探索SaalkB基因在聚乙烯降解过程中的作用提供理论依据,也为研究白浅灰链霉菌LBX-2降解聚乙烯的过程及机制提供思路.
1 材料和方法
1.1 实验材料白浅灰链霉菌LBX-2由本实验室分离并保存.E.coliDH5α感受态、E.coliBL21(DE3)感受态与原核表达载体pDE1 Directional Expression Kit、DNA凝胶回收试剂盒均购自成都擎科梓熙生物技术有限公司.质粒提取试剂盒购自OMEGA公司.细菌基因组DNA提取试剂盒、DL2000 DNA Marker、DL5000 DNA Marker、250 bp DNA Ladder以及TaqPCR Mastermix购自天根生化科技(北京)有限公司.IPTG和卡那霉素购自AMERESCO公司.QPK-201、ReverTra Ace®qPCR RT Master Mix、TRIzolTMPlus RNA Purification Kit购自toyobo公司.RR047Q反转录试剂盒购自TaKaRa.PE粉末(MW=2 000)购自中国石油化工股份公司茂名分公司.PE膜片购自海门市扬子医疗器械有限公司.引物根据SaalkB基因序列与pDE1序列设计,由成都擎科梓熙生物技术有限公司合成(见表1).
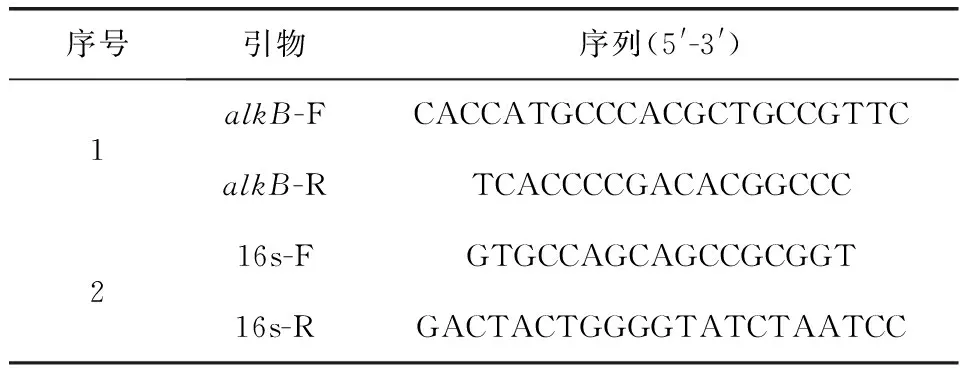
表 1 引物序列
1.2 培养基高氏一号培养基(g/L):可溶性淀粉20.0(用于蛋白质组学分析时为3.0),KNO31.0,NaCl 0.5,K2HPO4·3H2O 0.5,MgSO4·7H2O 0.5,FeSO4·7H2O 0.01,pH值7.2~7.4.
1.3 白浅灰链霉菌LBX-2总DNA和RNA提取采用细菌基因组DNA试剂盒制备白浅灰链霉菌LBX-2总DNA.由于白浅灰链霉菌LBX-2是革兰氏阳性菌,且在液体培养基中呈球状,较难破壁,故先将菌体混合石英砂在液氮中研磨,再按照试剂盒提供的操作步骤提取基因组DNA.白浅灰链霉菌LBX-2总RNA利用Trizol法提取,并利用凝胶电泳对其纯度和完整性进行检测;同时使用TaKaRa反转录试剂盒进行反转录得到该菌株的总cDNA.
1.4 白浅灰链霉菌LBX-2SaalkB基因扩增及测序以白浅灰链霉菌LBX-2总DNA为模板,alkB-F和alkB-R为扩增引物,扩增SaalkB基因.PCR反应体系为:基因组DNA 1 μL,2×PrimeSTAR GC Buffer(Mg2+plus) 25 μL,PCR Forward Primer(10 μmol/L) 2 μL,PCR Reverse Primer(10 μmol/L) 1 μL,dNTP Mixtuer(2.5 mmol/L) 4 μL,ddH2O补足至50 μL.扩增条件为:98 ℃预变性2 min,进入循环;94 ℃变性10 s;60 ℃退火5 s;72 ℃延伸30 s;循环32次;72 ℃延伸5 min.
使用DNA纯化回收试剂盒回收PCR目的DNA片段,取2 μL目的DNA,依次加入1 μL pDE1 Vector,1 μL 10×Topo Mix,6 μL ddH2O,混匀后20~30 ℃反应5 min,连接产物转化E.coliDH5α感受态细胞,取200 μL转化液涂布于含有100 μg/mL卡那霉素的LB固体培养基中,37 ℃过夜培养,挑取单菌落接种至含100 μg/mL卡那霉素的LB液体培养基中,37 ℃、180 r/min振荡培养10~12 h,提取质粒进行PCR验证.经PCR鉴定为阳性的菌株,送往成都擎科梓熙生物技术有限公司进行测序,对测序正确的菌株进行菌种保藏.
1.5 生物信息学分析将克隆得到的SaalkB基因序列进行BLAST(https://blast.ncbi.nlm.nih.gov/)相似性比对分析;AlkB蛋白的理化性质使用ProtParam(https://web.expasy.org/protparam/)在线分析;应用SignalP 3.0(http://www.cbs.dtu.dk/services/SignalP)进行AlkB蛋白信号肽预测;使用NPSA(http://npsa-pbil.ibcp.fr/npsa_automat.pl?page=/NPSA)在线预测AlkB蛋白二级结构;使用Swiss-Model(http://swissmodel.expasy.org)在线预测AlkB蛋白三级结构;运用TMPRED(http://www.ch.embnet.org/software/TMPRED/nn.html)预测AlkB蛋白的跨膜结构;应用NCBI中的BLASTP程序对AlkB蛋白进行序列同源性分析,利用软件MEGA 5.02比对序列,采用近邻相接法(neighbor-joining method)构建AlkB系统进化树,氨基酸的进化距离使用Poisson correction模型.
1.6 重组蛋白的诱导表达将测序验证正确的重组质粒转入E.coliBL21(DE3)中,使用含有卡那霉素(100 μg/mL)的LB平板进行筛选,37 ℃过夜培养,挑取单菌落至含100 μg/mL卡那霉素的液体LB培养基中,37 ℃、180 r/min振荡培养至OD600为0.5时,加入1 mmol/L IPTG,18 ℃低温诱导表达16 h.取1 mL菌液超声破碎3~4次至透明,离心取菌体沉淀,加入100 μL上样缓冲液后取样进行SDS-PAGE凝胶电泳.
1.7 重组菌株处理PE膜片使用以PE膜片为唯一碳源的无机盐培养基培养上述构建的重组工程菌株,以空载体E.coliBL21(DE3)作为对照组,每组设3个重复.培养30 d后,从对照组和实验组的摇瓶中分别回收PE膜片,用蒸馏水清洗膜片数次,再将膜片放入终质量分数为2%的SDS溶液中,超声清洗2 h后置于37 ℃环境中过夜,然后依次用无水乙醇、ddH2O清洗,干燥后用原子力显微镜(AFM, Dimension Icon, Veeco, Billerica, MA, USA)在1.0 Hz的扫描速度下观察PE膜片表面形貌.
1.8 荧光定量PCR以白浅灰链霉菌LBX-2总cDNA为模板,16s-F和16s-R为引物,采用SYBR染料法对SaalkB基因进行相对荧光定量检测.反应体系为:SYBR Green Realtime PCRMasterMix-plus(2×)10 μL,PCR Forward Primer(10 μmol/L)1 μL,PCR Reverse Primer(10 μmol/L)1 μL,cDNA 1 μL,Plus Solution 2 μL,ddH2O补足至20 μL.反应条件:95 ℃预变性3 min,进入循环;95 ℃变性15 s;58 ℃退火30 s;72 ℃延伸45 s;循环40次,72 ℃保温3 min,重复次数n=3.从55 ℃到95 ℃进行溶解曲线分析,去除引物二聚体和其他非特异性扩增.利用iQ 3.1-Cycler分析数据,定量方法为2-ΔΔCt法.
优美的水生态环境,是最公平的公共产品,是最普惠的民生福祉。必须树立以人民为中心的理念,坚持全民共建、全民共享,广泛凝聚保护河湖的强大合力。
1.9 AlkB蛋白的表达量分析分别使用以PE粉末为唯一碳源的无机盐培养基和高氏一号培养基培养白浅灰链霉菌LBX-2,每组设3个重复,180 r/min、37 ℃振荡培养.在LBX-2培养至48 h(指数生长期)和84 h(平台期)时进行取样,取菌液于5 000 r/min、4 ℃条件下离心10 min,收集全部菌体,以PE粉末为唯一碳源培养48 h和84 h的样品分别标记为PE48h、PE84h,用高氏一号培养基培养48 h和84 h的样品分别标记为G48h、G84h.对样品进行蛋白质测序并获得蛋白质组学数据,通过序列比对和信息注释找到AlkB蛋白,利用HEML软件制作热图分析其表达量,并比较不同碳源、不同时间组别的AlkB表达量(log2FC转化后数据)的差异,分别记为PE48h vs G48h、PE84h vs G84h、G84h vs G48h和PE84h vs PE48h.
2 结果
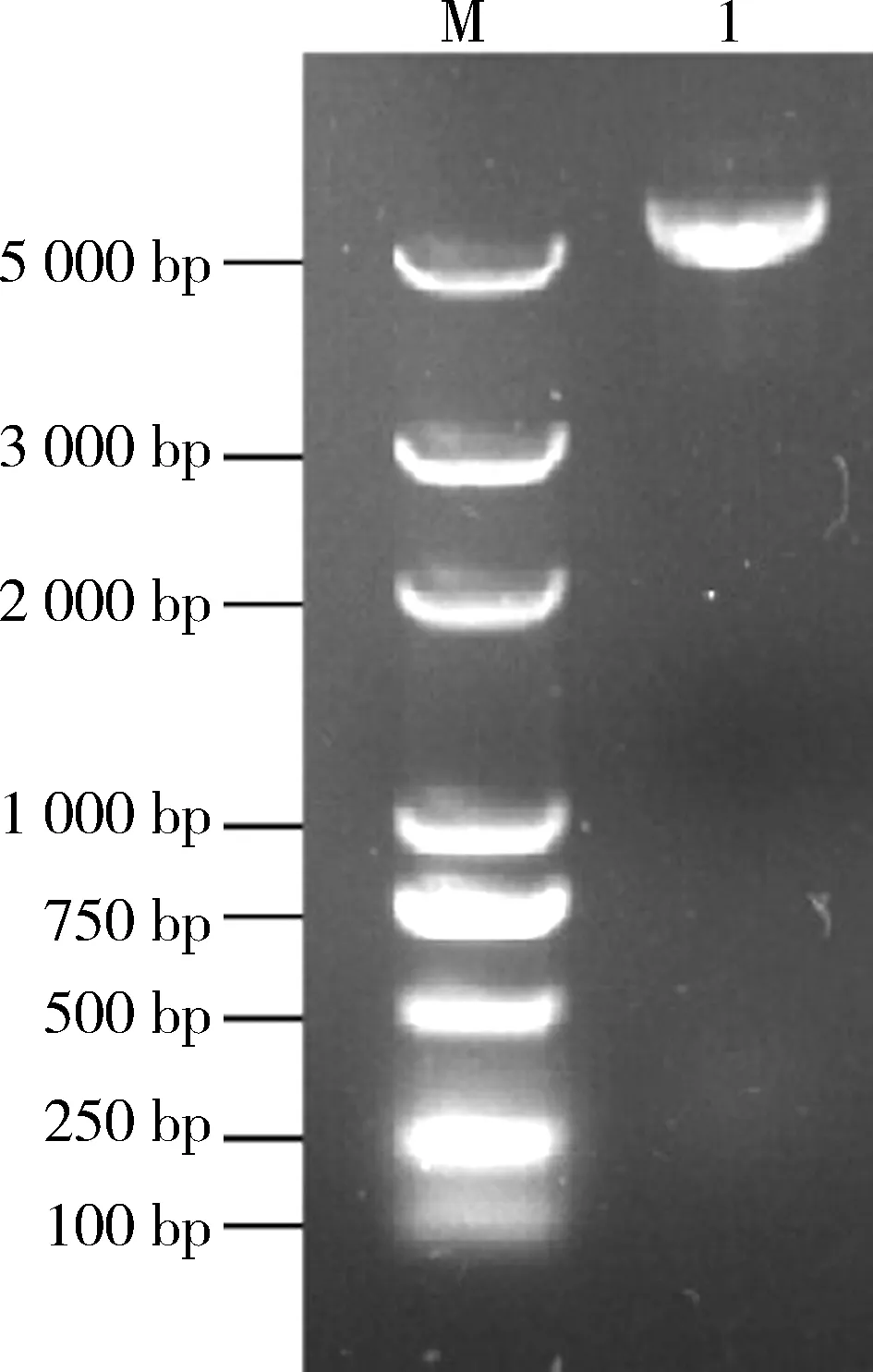
2.1 白浅灰链霉菌LBX-2SaalkB基因的克隆以白浅灰链霉菌LBX-2基因组DNA为模板,利用引物alkB-F和alkB-R进行PCR扩增,经纯化后获得长度约为1 000 bp的片段(见图1).

M: DNA分子量标准(DL2 000) ; 1: SaalkB基因的PCR扩增产物
2.2SaalkB基因的生物信息学分析
2.2.1SaalkB编码蛋白的核苷酸序列及氨基酸序列分析 白浅灰链霉菌LBX-2的alkB基因通过PCR扩增后,对其进行胶回收、克隆并测序,经测序后发现该基因长度为1 086 bp(NCBI GenBank索引号:OL804209),通过DNAMAN软件分析获得该基因编码蛋白质共具有361个氨基酸,且具有一个脂肪酸脱饱和功能域,该功能域长度为585 bp,起始位点为第316 bp,终止位点为第900 bp.
2.2.2SaalkB编码蛋白的理化性质分析 理化性质分析显示SaalkB基因编码蛋白共有361个氨基酸,分子式为C1851H2837N547O491S10,分子量为40.9 kDa,等电点(isoelectric point, pI)为10,不稳定系数为60.74,是一种不稳定的蛋白质,脂肪疏水系数为88.12,亲水性平均系数(GRAVY)为-0.209,属于亲水性蛋白.
2.2.3SaalkB编码蛋白的二级和三级结构预测 NPSA预测得到SaalkB编码蛋白的二级结构(见图2),其中α螺旋(alpha helix)、β折叠(beta folding)、无规则卷曲(random coil)所占比例分别为39.34%、9.42%、51.25%.因此,AlkB蛋白主要由无规则卷曲组成,其次为α螺旋,再次为β折叠.
利用Swiss Model同源构建模型预测AlkB蛋白的三维结构模型(见图3),该蛋白三级结构中仅有部分α螺旋和少量的β折叠,大部分由无规则卷曲组成,整体结构比较疏松,与蛋白质二级结构预测基本一致.
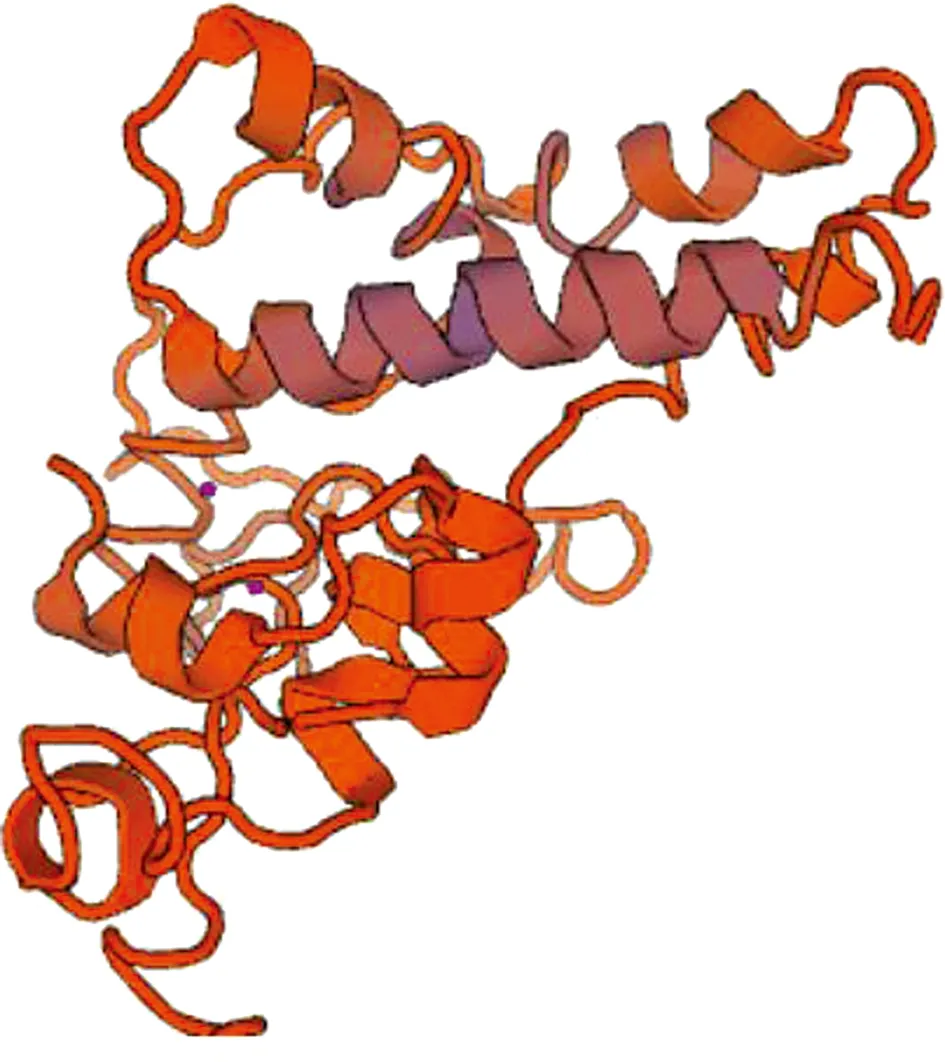
图3 SaalkB编码蛋白三级结构预测
2.2.4SaalkB编码蛋白信号肽及跨膜结构分析 Signal IP 3.0分析发现SaalkB编码蛋白不存在信号肽序列(见图4),属于非分泌蛋白.
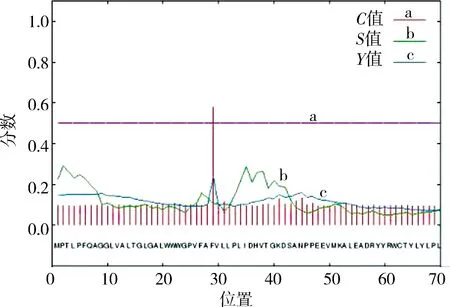
图4 SaalkB编码蛋白信号肽分析
TMpred软件预测SaalkB编码蛋白含有5个跨膜结构域(见图5),分别为氨基酸第10~35位(跨膜方向由内向外)、氨基酸第62~85位(跨膜方向由内向外)、氨基酸第87~110位(跨膜方向由外向内)、氨基酸第205~229位(跨膜方向由内向外)、氨基酸第305~325位(跨膜方向由外向内),表明SaalkB编码蛋白属于跨膜蛋白.
2.2.5SaalkB编码蛋白的系统进化分析 系统进化分析结果表明:经克隆获得的SaalkB编码氨基酸序列与Stretomycessp. XY006、Streptomycessp. B9173、Streptomycessp. CB01883以及Streptomycesaureus的alkB编码氨基酸序列亲缘关系最近(见图6),这一结果与alkB编码蛋白的物种分类系统的结果较为一致.

图6 SaalkB编码蛋白的系统进化树
2.3 原核载体构建及表达分析从转入重组质粒的E.coliDH5α中提取含有SaalkB基因的重组质粒pDE1(见图7)并进行PCR验证,片段大小与目的基因大小相同(见图8),证明含有SaalkB基因的原核表达载体pDE1-alkB构建成功.
M: DNA分子量标准(DL5 000); 1: 重组质粒pDE1-alkB.
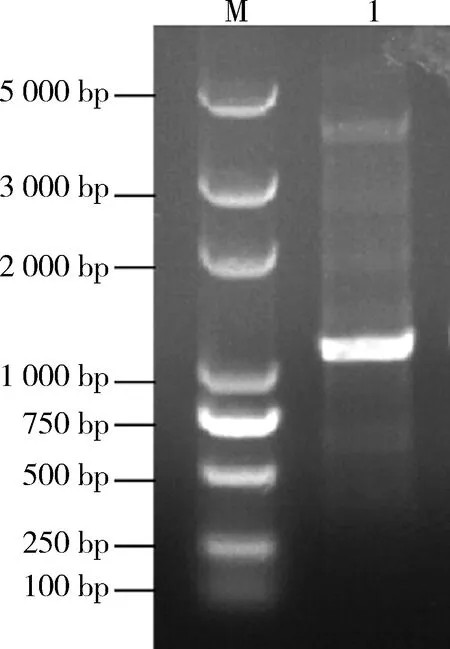
M: DNA分子量标准(DL5 000); 1: 重组质粒pDE1-alkB验证PCR.
经终浓度为1 mmol/L IPTG诱导的含有SaalkB基因的原核表达载体pDE1-alkB在E.coliBL21 (DE3)中成功表达(见图9),分子量约40 kDa,与预期的目标蛋白大小一致(40.9 kDa),表明在1 mmol/L IPTG诱导下,SaalkB基因在大肠杆菌中能够进行过表达.
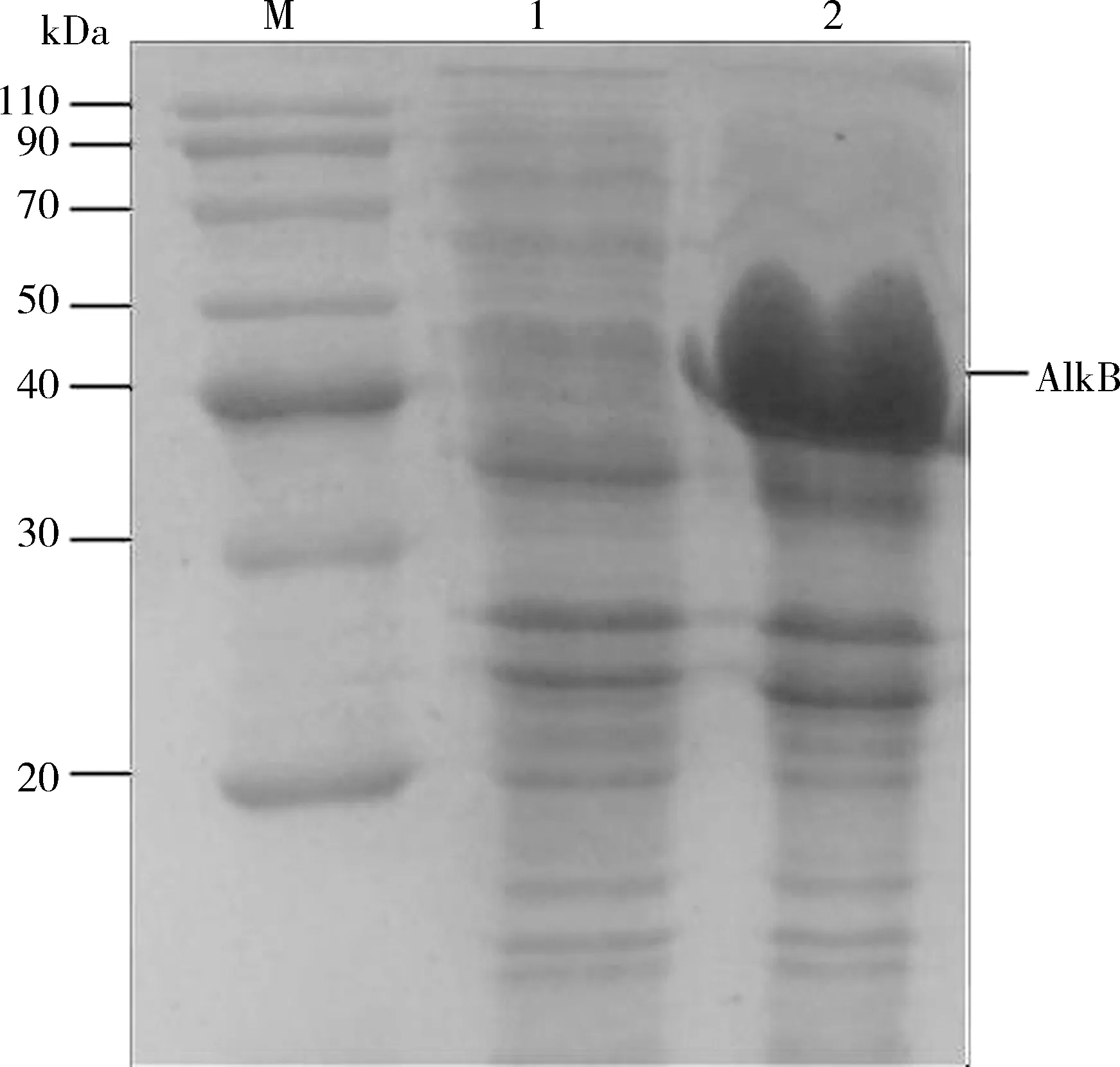
M: 蛋白Marker; 1: 空载体转化菌诱导表达产物; 2: 重组质粒转化菌诱导表达产物.
2.4 重组菌株对PE膜片的处理作用AFM图像显示重组工程菌株处理30 d后PE膜片局部降解,膜片上形成了明显的沟槽、凹陷和轻度侵蚀,粗糙程度高(见图10B),而对照组PE膜片相对更加平滑,虽有凹陷,但粗糙程度低(见图10A),表明重组工程菌株能够降解PE,通过与PE膜片表面的物理接触改变了聚合物表面的完整性,进一步表明SaalkB基因可能在聚乙烯降解过程中发挥重要作用.

图10 PE 膜片表面形貌的AFM测试
2.5 荧光定量PCR分析利用Trizol法提取白浅灰链霉菌LBX-2总RNA,反转录为cDNA进行荧光定量PCR,分析白浅灰链霉菌LBX-2在以PE粉末为唯一碳源的无机盐培养基中和在高氏一号培养基中SaalkB基因的表达水平,每组设3个重复.结果表明,在以PE粉末为唯一碳源的培养基中SaalkB基因的表达量约为在高氏一号培养基中的30倍(见图11),证明了SaalkB基因可能在白浅灰链霉菌LBX-2降解聚乙烯的过程中具有重要的作用.
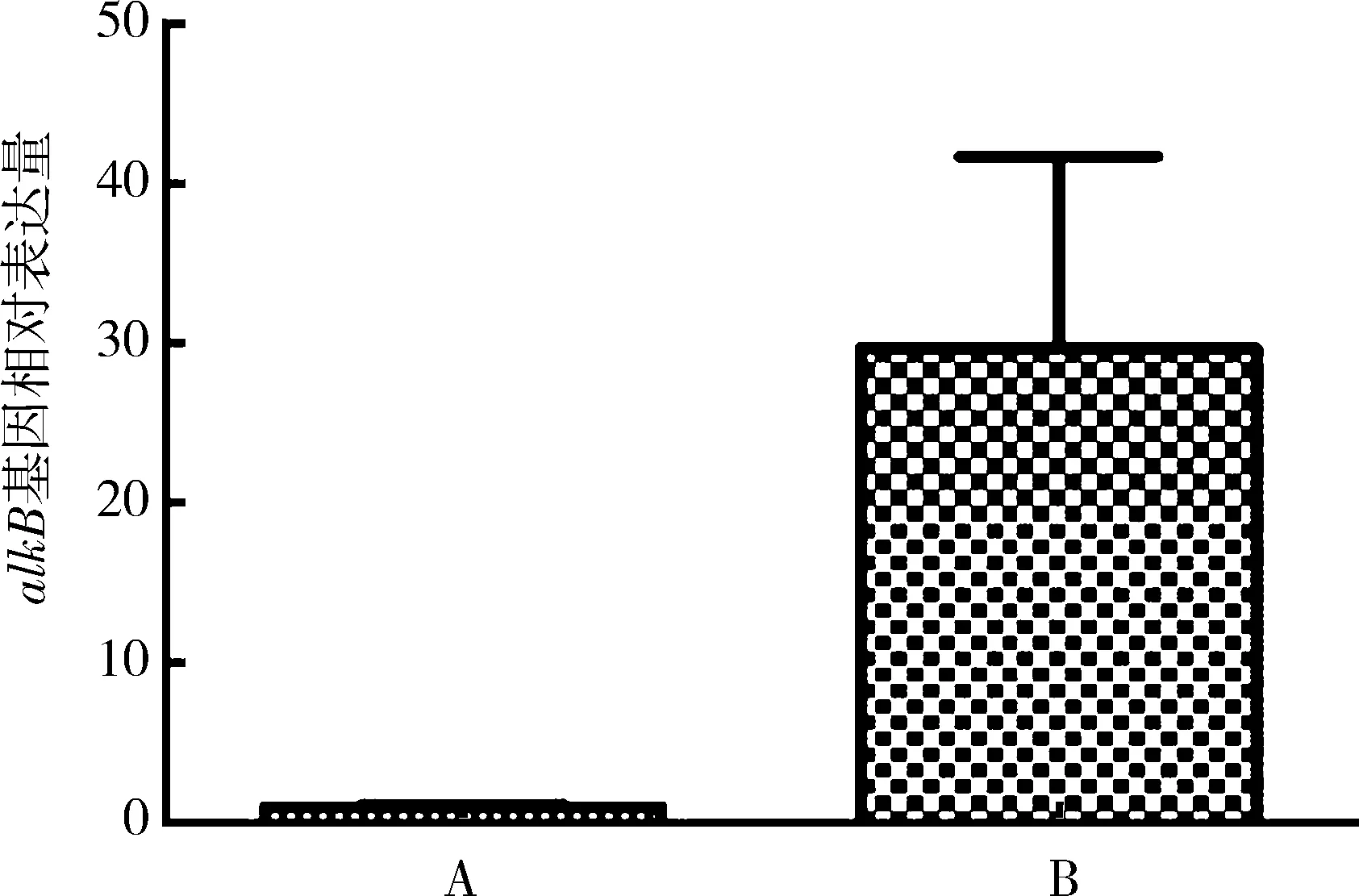
A: 高氏一号培养基中SaalkB基因表达量;
2.6 AlkB蛋白的表达量分析蛋白质表达量热图显示白浅灰链霉菌LBX-2的AlkB蛋白在PE48h和PE84h中的表达量显著高于G48h和G84h(见图12A),通过表达量log 2FC转化后的数据进行差异显著性分析,白浅灰链霉菌LBX-2在以PE为唯一碳源培养48 h和84 h时,AlkB蛋白的表达量比在高氏一号培养基中分别上调了2.35和1.57倍(图12B),表明AlkB蛋白可能参与了与PE降解相关的代谢途径,证明了SaalkB基因可能在白浅灰链霉菌LBX-2降解聚乙烯的过程中具有重要的作用.

A: 不同培养条件下AlkB蛋白表达量,横坐标表示3个重复S1-S3,纵坐标表示不同培养条件,深色表示上调,浅色表示下调;
3 讨论
自20世纪以来,随着塑料产品不断大规模生产,塑料废弃物由于处理不当,长期大量积累造成“白色污染”,严重危害生态环境的健康发展[17].目前,已有许多研究报道了微生物降解PE的潜力,分离出若干能利用PE为唯一碳源生长的菌株.然而,大多微生物存在降解率低以及降解需要预处理PE等问题,且降解速率受PE结晶度、氧化程度和分子量大小等多种因素的影响[18].白浅灰链霉菌LBX-2是一种好氧和高G+C含量(质量分数72.47%)的革兰氏阳性菌,能够利用PE作为唯一碳源,降解PE薄膜第15天薄膜表面出现皱褶和凹痕,第30天出现明显可见的破损,第45天出现孔洞,对分子量为5 000和10 000的PE粉末降解15 d的降解率分别为17.27%±0.48%、13.03%±0.74%[19].本研究成功地从白浅灰链霉菌LBX-2基因组克隆了SaalkB基因,其长度为1 086 bp,编码361个氨基酸,脂肪酸脱饱和功能域长度为585 bp,与PseudomonascitronellolisUAM-Ps1和Alcanivoraxsp. 2B5的alkB基因大小相似[20-21].对AlkB蛋白进行理化性质分析、信号肽、蛋白质二级和三级结构预测,结果表明,AlkB蛋白是一种分子量为40.9 kDa、具有5个跨膜结构域(见图5)且无信号肽(见图4)的不稳定型亲水蛋白;二级结构预测结果可知其主要由无规则卷曲和α螺旋组成(见图2).系统进化分析结果表明,AlkB蛋白氨基酸序列与Stretomycessp. XY006、Streptomycessp. B9173、Streptomycessp. CB01883以及Streptomycesaureus的alkB编码氨基酸序列亲缘关系最近,其中与Stretomycessp. XY006的alkB编码氨基酸序列相似性为100%(见图6).
大肠杆菌外源蛋白表达系统是生产外源蛋白的最优选系统[22-23].本研究成功构建了SaalkB基因的原核表达载体pDE1-alkB(见图7),通过SDS-PAGE凝胶电泳在大肠杆菌BL21中过量诱导表达得到分子量大小与预测一致的AlkB蛋白(40.9 kDa)(见图9),与Pseudomonascitronellolis的AlkB蛋白(42 kDa)分子量相近[20].但Al-Kanany等[24]将Pseudomonasaeruginosa的alkB基因在大肠杆菌BL21中表达,经IPTG诱导得到分子量为46 kDa.Luo等[25]发现从PseudomonasputidaGPo1中克隆得到的alkB基因在大肠杆菌中经不同浓度的柴油诱导表达,其分子量为51 kDa.可见不同菌株来源的AlkB蛋白分子量大小存在一定差异,可能与编码基因的大小或组成不同有关.来自Pseudomonassp. E4、PseudomonasaeruginosaE7的烷烃羟化酶基因alkB在大肠杆菌BL21中表达,80 d的堆肥降解结果表明重组工程菌株均对低分子量PE粉末具有降解作用,而空载体菌株无降解作用[14-15],证实了alkB对低分子量PE的降解能力.本研究中30 d的降解实验同样发现重组工程菌株能够降解PE,AFM观察PE膜片表面形成明显的沟槽、凹陷(见图10).通过荧光定量PCR分析发现,白浅灰链霉菌LBX-2在以PE粉末为唯一碳源的培养基中SaalkB基因的表达量约为在高氏一号培养基中的30倍(见图11),这与Kong等[26]发现AcinetobacterpittiiSW-1的alkB基因在以长链正构烷烃C20作为唯一碳源时转录水平提高78.28倍且通过RT-qPCR证实了C20显著诱导alkB表达,以及杨劼等[27]通过荧光定量PCR证明Acinetobactersp. Tust-DM21降解石油与alkB基因的上调表达相关具有一致性.通过蛋白质组学数据分析发现白浅灰链霉菌LBX-2在以PE为唯一碳源培养48 h和84 h时AlkB蛋白的表达量比在高氏一号培养基中分别上调了2.35和1.57倍(见图12),这与刘环[28]分析PseudomonasaeruginosaStrain SJTD-1蛋白质组数据发现烷烃羟化酶AlkB2在有烷烃诱导时表达量上调4倍的结果具有一致性.也有研究发现AlkB参与聚苯乙烯塑料降解的第一步,可以催化聚苯乙烯羟基化[29].由此可以推测alkB不仅是聚乙烯降解过程中的关键酶基因,也可能在其他塑料降解过程中起着重要作用.
本研究从白浅灰链霉菌LBX-2基因组克隆得到了SaalkB基因,并对其进行了生物信息学分析,为进一步探讨该基因的表达提供理论依据.此外,构建含有SaalkB基因的原核表达载体pDE1-alkB并在大肠杆菌内成功表达,且重组工程菌株具有降解PE的能力.荧光定量PCR分析SaalkB基因在PE降解过程中的表达情况,蛋白质组分析AlkB蛋白在PE降解过程中的表达情况,证明了SaalkB基因可能在白浅灰链霉菌LBX-2降解PE过程中起重要的作用.为进一步研究AlkB蛋白在聚乙烯降解过程中的功能以及深入探索白浅灰链霉菌LBX-2降解聚乙烯的机制提供理论支撑.
致谢四川师范大学“大学生创新性实验计划”项目(S201910636417)和四川师范大学开放实验项目(KFSY2018046)对本文给予了资助,谨致谢意.
