寡养单胞菌膜孔蛋白D0Y85_RS06780对细菌抗生素耐药性的作用研究
2023-08-25刘亚豫任钊锐谢欣茹王祯王高扬曹广祥付加芳
刘亚豫?任钊锐?谢欣茹?王祯?王高扬?曹广祥?付加芳


摘要:目的 探究寡养单胞菌Stenotrophomonas sp. G4菌株中膜孔蛋白D0Y85_RS06780对抗生素耐药性的作用。方法 通过氨基酸序列比对和同源建模预测D0Y85_RS06780蛋白的生物学功能;采用大肠埃希菌构建D0Y85_RS06780基因的异源表达菌株,通过检测抗生素MICs探讨D0Y85_RS06780对细菌抗生素抗性的作用,同时分析外排泵抑制剂对D0Y85_RS06780功能的影响;利用分子对接软件Discovery Studio的CDOCKER技术分析D0Y85_RS06780与抗生素和外排泵抑制剂的结合情况。结果 同源分析表明D0Y85_RS06780为膜孔蛋白,异源表达D0Y85_RS06780导致重组大肠埃希菌具有多黏菌素和四环素抗性,外排泵抑制剂利血平能够抑制其功能,分子对接显示D0Y85_RS06780能结合多黏菌素、四环素和利血平。结论 Stenotrophomonas sp. G4菌株中膜孔蛋白D0Y85_RS06780與其抗生素耐药性有关。本研究将为D0Y85_RS06780与抗生素耐药性的关联提供实验证据。
关键词:寡养单胞菌;膜孔蛋白;耐药性;异源表达;分子对接
中图分类号:R917文献标志码:A
Study on the effect of cell membrane porin D0Y85_RS06780
of Stenotrophomonas sp. on bacterial antibiotic resistance
Liu Ya-yu1, Ren Zhao-rui1, Xie Xin-ru1, Wang Zhen2, Wang Gao-yang1, Cao Guang-xiang1, and Fu Jia-fang1
(1 College of Biomedical Sciences, Shandong First Medical University & Shandong Academy of Medical Sciences, Jinan 250117;
2 School of Municipal and Environmental Engineering, Shandong Jianzhu University, Jinan 250101)
Abstract Objective To explore the effect of cell membrane porin D0Y85_RS06780 of Stenotrophomonas sp. G4 on bacterial antibiotic resistance. Methods Biological function of D0Y85_RS06780 was predicted based on amino acid sequence alignment and homologous modeling; D0Y85_RS06780 gene was heterologously expressed in Escherichia coli, and its effect on bacterial antibiotic resistance was determined through MICs; The effect of efflux pump inhibitors on D0Y85_RS06780 were also analyzed through antibiotic MICs; The molecular docking was carried out according to the CDOCKER protocol of Discovery Studio to determine whether D0Y85_RS06780 can bind antibiotics and efflux pump inhibitors. Results Homologous analysis showed that D0Y85_RS06780 is a membrane porin protein. Recombinant E. coli expressing D0Y85_RS06780 obtained polymyxin and tetracycline resistance, and the polymyxin resistance could be inhibited by the efflux pump inhibitor reserpine. Molecular docking revealed that D0Y85_RS06780 could bind polymyxin, tetracycline and the efflux pump inhibitor reserpine. Conclusion Cell membrane porin D0Y85_RS06780 of Stenotrophomonas sp. is associated with bacterial antibiotic resistance. This study provides experimental evidence for further research on antibiotic resistance mechanism of cell membrane porin D0Y85_RS06780.
Key words Stenotrophomonas sp.; Membrane porin; Antibiotic resistance; Heterologous express; Molecular docking
微生物通过多种机制产生耐药性[1],其中,细菌主动外排作用是其主要耐药机制之一[2]。根据外排泵蛋白的氨基酸序列和能量利用可分为5大类:耐药结节化细胞分化超家族(Resistance-nodulation-cell division,RND)、易化子超家族(Major facilitator superfamily,MFS)、多重耐药及毒性化合物蛋白家族(multi antimicrobial extrusion,MATE)、耐多药性家族(small multidrug resistance,SMR)和ATP结合盒超家族(ATP-binding cassette,ABC),这些外排泵广泛存在于革兰阳性菌和阴性菌中[3-9]。研究显示多数外排泵介导多重耐药性,而部分外排泵具有底物特异性[10]。除外排泵蛋白外,革兰阴性菌外膜还存在许多通道蛋白,亲水性的溶质和其他小分子物质可以通过膜孔蛋白进出细菌[11-12]。一方面,抗菌药物通过膜孔蛋白进入菌体将细菌杀死[13-14],另一方面膜孔蛋白也可能与多重耐药密切相关[15],但膜孔蛋白与抗生素耐药性的关联尚有待深入阐释。
嗜麦芽寡养单胞菌(Stenotrophomonas maltophilia)是重要的医源性感染菌,在重症患者和免疫缺陷患者中可引起致死性感染,其日益严重的多重耐药性加剧了临床治疗难度(Brooke JS, 2017)[16]。Stenotrophomonas sp. G4菌株为本课题组前期分离获得的一株多重耐药菌,对多种抗生素表现为抗性,其中多黏菌素MIC值>128 mg/L[17]。基因组注释显示D0Y85_RS06780为膜孔蛋白,转录组测序分析表明多黏菌素压力下D0Y85_RS06780转录水平显著上升,但其与抗生素抗性之间的关系仍然未知。本研究通过构建D0Y85_RS06780异源表达菌株,分析膜孔蛋白D0Y85_RS06780与细菌抗生素抗性之间的關联,可为阐释膜孔蛋白参与抗生素耐药机制提供依据。
1 材料和方法
1.1 材料
1.1.1 菌株
Stenotrophomonas sp. G4菌株为从污水中分离的多重耐药菌[17],保存于本实验室。Escherichia coli DH5α菌株,购自上海昂宇生物技术有限公司。D0Y85_RS06780的异源表达菌株R06780和P06780,为本研究构建。
1.1.2 试剂和仪器
限制酶Hind Ⅲ和 Xba Ⅰ(北京Sibenzyme公司),pMD18-T载体(北京Takara公司),2×clone express(南京诺唯赞生物科技股份有限公司),Pfu DNA Polymerase(北京艾德莱生物科技有限公司),胶回收试剂盒(美国Omega公司),质粒提取试剂盒(美国Omega公司)。多黏菌素、氟苯尼考、红霉素、环丙沙星、卡那霉素、美罗培南、头孢克肟、四环素、氨苄青霉素、羰基氰化物间氯苯腙、利血平、维拉帕米、N-甲基吡咯烷酮(麦克林公司)。MTH-100型恒温混匀仪(杭州米欧仪器有限公司),P-800型酶标仪ScanReady(杭州遂真生物技术有限公司)。
1.1.3 引物
根据Stenotrophomonas sp. G4菌株基因组(GenBank号:CP031741.1)序列,通过BioXM2.6软件设计引物序列。本研究所设计的引物序列及其扩增长度见表1。
1.2 实验方法
1.2.1 D0Y85_RS06780蛋白的同源分析
通过NCBI查找D0Y85_RS06780蛋白的同源序列,采用CLUSTALW软件比对同源蛋白的氨基酸序列,利用ESPript 3软件查看比对结果[18]。
1.2.2 D0Y85_RS06780自身启动子异源表达菌株的构建
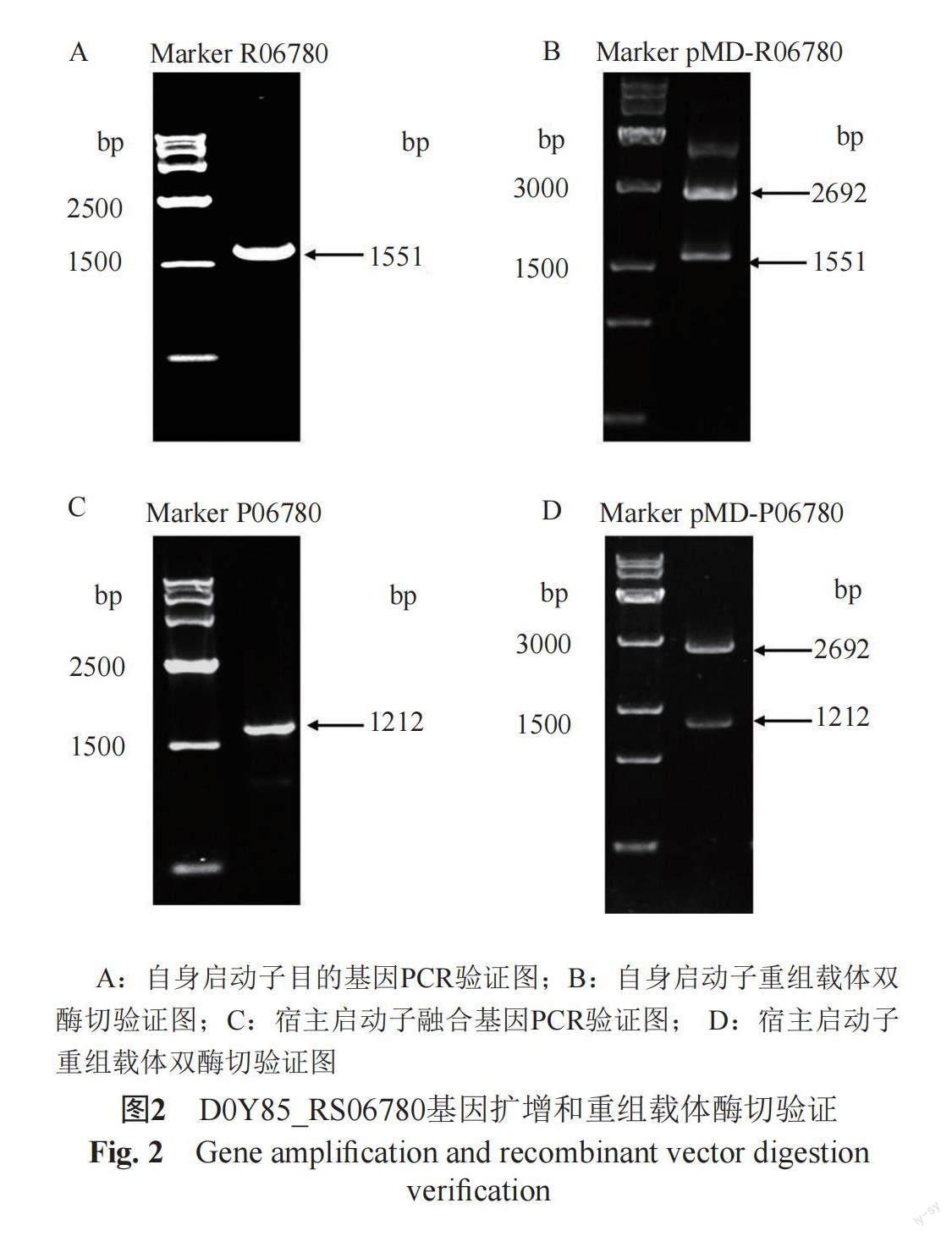
以G4菌株的基因组为模板,RS06780-F和RS06780-R为引物(表1),PCR扩增D0Y85_RS06780基因含启动子的DNA片段。采用2×clone express重组酶将PCR片段连接至 pMD18-T载体,转化到DH5α感受态细胞,用氨苄青霉素筛选转化子,提取重组载体进行Hind Ⅲ和Xba Ⅰ酶切验证以及DNA测序验证,获得异源表达D0Y85_RS06780基因(自身启动子)的R06780重组菌株。R06780重组菌株经过Hind Ⅲ和Xba Ⅰ酶切验证、PCR验证及DNA测序验证。
1.2.3 D0Y85_RS06780大肠埃希菌启动子异源表达菌株的构建
以G4的基因组为模板,以P06780-F和P06780-R为引物(表1),扩增D0Y85_RS06780的基因片段;以pMD18-T载体为模板,AP-F和AP-R为引物(表1),扩增β-内酰胺酶基因的启动子片段;切胶回收后混合上述两个PCR产物作为模板,以AP-F和P06780-R 为引物,采用Pfu DNA聚合酶扩增融合基因片段,最后加Taq DNA聚合酶在72℃继续反应30 min,使PCR产物的末端加3'-A尾巴。通过T4 DNA连接酶将PCR产物连接至 pMD18-T载体,转化到DH5α感受态细胞,用氨苄青霉素筛选转化子,提取重组载体进行HindⅢ和XbaⅠ酶切验证以及DNA测序验证,获得D0Y85_RS06780基因融合大肠埃希菌启动子的P06780重组菌株。P06780重组菌株经过HindⅢ和XbaⅠ酶切验证、PCR验证及DNA测序验证。
1.2.4 肉汤微量稀释法测定抗生素最低抑菌浓度(MICs)
R06780菌株和P06780菌株采用肉汤微量稀释法测定抗生素MICs,测定方法根据临床实验室标准研究所(CLSI 2018)指南所述[19]。抗生素包括多黏菌素、氟苯尼考、红霉素、环丙沙星、卡那霉素、美罗培南、头孢克肟和四环素。同时以携带pMD18-T载体的DH5α(18T)菌株作为对照。
1.2.5 抑制剂对抗生素MICs的影响
使用肉汤微量稀释法测定抑制剂对抗生素MICs的影响,如临床实验室标准研究所(CLSI 2018)指南所述[19]。抑制剂选择羰基氰化物间氯苯腙(CCCP)、利血平、维拉帕米和N-甲基吡咯烷酮,首先用肉汤微量稀释法测定P06780重组菌株对上述4种抑制剂的MICs,然后确定不影响P06780菌株生长的供试浓度,然后在供试浓度条件下测定抗生素MICs,同时以不加抑制剂的P06780菌株作为对照。
1.2.6 D0Y85_RS06780蛋白与抗生素和抑制剂的分子对接
从SWISS MODEL数据库进行蛋白同源建模[20],获取核心靶蛋白结构,从Chemspider数据库获取抗生素和抑制剂小分子配体结构式[21],用分子对接软件Discovery Studio的CDOCKER技术对接靶蛋白与小分子配体,分析D0Y85_RS06780蛋白与抗生素和抑制剂分子之间的结合情况[22]。
2 结果
2.1 D0Y85_RS06780功能注释与同源性分析
Stenotrphomonas sp. G4菌株的基因注释显示D0Y85_RS06780基因全长1212 bp,编码含402个氨基酸的蛋白。氨基酸序列分析显示,D0Y85_RS06780与S. maltophilia K279a菌株中OprO和OprP膜孔蛋白的相似性分别为96.5%和30.4%(图1),与Pseudoxanthomonas suwonensis 11-1菌株中OprO蛋白的相似性为69.1%,与Lysobacter sp. A03菌株中OprP蛋白的相似性为58%,说明D0Y85_RS06780可能为OprO类型的膜孔蛋白。
2.2 D0Y85_RS06780异源表达菌株的抗生素MICs分析
通过PCR扩增得到D0Y85_RS06780基因(含启动子区)的DNA片段(图2A),与pMD18-T载体连接获得重组载体pMD-R06780(图2B),最后转到DH5α菌株构建得到重组菌株R06780。同时通过重叠PCR获得融合大肠埃希菌启动子的D0Y85_RS06780片段(图2C),与pMD18-T载体连接获得重组载体pMD-P06780(图2D),构建得到重组菌株P06780。
肉汤微量稀释法检测R06780和P06780重组菌株的抗生素MICs,结果如表2所示。结果显示DH5α(18T)菌株的四环素MIC值为8 μg/mL,而P06780菌株(大肠埃希菌启动子)的四环素MIC值为
64 μg/mL,R06780菌株(自身启动子)的四环素MIC值为16 μg/mL,显著高于携带pMD18-T载体的DH5α(18T)菌株,说明异源表达D0Y85_RS06780会导致大肠埃希菌对四环素产生抗性。而P06780菌株(大肠埃希菌启动子)的多黏菌素和四环素MICs值分别为32 μg/mL和64 μg/mL,表明其对多黏菌素和四环素均具有抗性。此外,P06780菌株的多黏菌素和四环素MICs值显著高于R06780菌株,提示采用宿主启动子可能更有利于D0Y85_RS06780在大肠埃希菌中表达。
2.3 抑制剂对P06780菌株抗生素MICs的影响分析
羰基氰化物间氯苯腙(CCCP)、利血平、维拉帕米和N-甲基吡咯烷酮是常用的外排泵抑制剂。本研究首先通过肉汤微量稀释法测定了P06780菌株对这些外排泵抑制剂的MICs,确定0.1 μg/mL CCCP、
8 μg/mL利血平、8 μg/mL维拉帕米和8 μg/mL N-甲基吡咯烷酮不影响P06780菌株的生长,因此采用上述浓度作为供试浓度进行抑制剂试验。抑制剂对P06780菌株抗生素MICs的影响见表3,在供试浓度下CCCP、维拉帕米、N-甲基吡咯烷酮对抗生素MICs值无显著影响,而添加8 μg/mL 利血平时P06780菌株对多黏菌素的MIC下降为4 μg/mL,比不加抑制剂时的MIC值(32 μg/mL)明显降低,说明利血平能抑制D0Y85_RS06780蛋白对多黏菌素的抗性。文献显示利血平能够抑制ABC家族和MFS家族外排泵的功能[23],提示D0Y85_RS06780可能通过发挥外排泵作用使细菌产生对多黏菌素抗性。此外,添加8 μg/mL 利血平时P06780菌株对四环素的MIC仍为64 μg/mL,与不加抑制剂时的MIC值(64 μg/mL)一致,提示利血平能不能抑制D0Y85_RS06780蛋白对四环素的抗性。
2.4 膜孔蛋白D0Y85_RS06780与抗生素的分子对接
CDOCKER分子对接显示D0Y85_RS06780蛋白可以结合多黏菌素和四环素等抗生素分子(图3)。分子对接结果显示多黏菌素可结合D0Y85_RS06780蛋白的B环Ser-364、 Lys-104、Leu-129、Phe-120、Asn-102氨基酸残基,四环素结合B环Leu-99、Val-76、Asn-102、Ser-131、Asp-100氨基酸残基,表明膜孔蛋白D0Y85_RS0678可能具有运输多黏菌素和四环素的功能,与细菌的多黏菌素和四环素抗性有关。分子对接显示外排泵抑制剂利血平也可以结合D0Y85_RS06780蛋白的B环和C环 Leu-368、Phe-366、Val-103、Lys-104、Tyr-105、Ile-115氨基酸残基,考虑到利血平可以显著降低P06780菌株的多黏菌素MIC(表3),提示利血平可与多黏菌素竞争性结合D0Y85_RS06780蛋白并抑制其运输功能。此外,D0Y85_RS06780蛋白与氟苯尼考、环丙沙星、卡那霉素、红霉素、美罗培南和头孢克肟等抗生素均不能成功对接,分子对接数据与上述抗生素MICs的结果一致(表2)。
3 讨论
膜孔蛋白与抗生素耐药性的关联尚有待深入阐释[24]。生物信息学分析表明Stenotrphomonas sp. G4菌株中D0Y85_RS06780蛋白为OprO类型的膜孔蛋白,已有的研究显示假单胞菌中OprO膜孔蛋白与其多重耐药性相关 [25-26],提示D0Y85_RS06780可能与细菌的抗生素抗性有关。本文进一步探讨了Stenotrphomonas sp. G4菌株中D0Y85_RS06780膜孔蛋白与抗生素耐药性的关联。本研究表明异源表达D0Y85_RS06780时重组大肠埃希菌P06780的多黏菌素和四环素MICs值分别为32 μg/mL和64 μg/mL,表明D0Y85_RS06780介导了细菌的多黏菌素和四环素抗性。外排泵抑制试验分析显示添加8 μg/mL利血平时P06780菌株对多黏菌素的MIC比不加抑制剂时明显降低,提示D0Y85_RS06780可能发挥外排泵作用从而使细菌产生多黏菌素抗性。分子对接分析进一步表明多黏菌素和外排泵抑制剂利血平均能结合D0Y85_RS06780蛋白上的Lys-104氨基酸位点,上述结果表明Stenotrphomonas sp. G4中膜孔蛋白D0Y85_RS06780与细菌的多黏菌素抗性之间存在关联。但分子对接显示利血平与膜孔蛋白D0Y85_RS0678结合的氨基酸位点不同于四环素与D0Y85_RS0678结合的氨基酸位点,与抑制剂實验结果(利血平能不能降低D0Y85_RS06780蛋白对四环素的MIC值)一致,暗示D0Y85_RS0678虽具有四环素抗性但其抗性不能被利血平抑制。
P06780菌株(大腸埃希菌启动子)的多黏菌素和四环素MICs值均高于R06780菌株(自身启动子),提示膜孔蛋白D0Y85_RS06780的表达水平可能影响其对抗生素的抗性。此外,Stenotrphomonas sp. G4菌株的多黏菌素和四环素MICs值[17]与P06780菌株和R06780菌株均有较大差异,除膜孔蛋白D0Y85_RS06780在不同菌体内表达量不同的原因之外,也可能与细菌体内微环境有关,还可能与G4菌株存在其它抗生素耐药机制有关,提示细菌的抗生素抗性受到多因素的复杂影响。
细菌外排泵主动外排过程所消耗的能量来源于质子移动力(PMF)或ATP水解,研究认为SMR家族、MATE家族、MFS家族和RND家族都属于PMF型,ABC家族为ATP型[27]。研究显示外排泵抑制剂利血平会抑制ABC家族外排泵的功能[28]。尽管本文研究表明Stenotrphomonas sp. G4菌株中D0Y85_RS06780膜孔蛋白介导细菌的抗生素抗性,且其对多黏菌素的抗性可被利血平抑制,但D0Y85_RS06780能量驱动类型尚需进一步深入研究。
参 考 文 献
Rowe-Magnus D A, Guerout A M, Mazel D. Bacterial resistance evolution by recruitment of super-integron gene cassettes[J]. Mol Microbiol, 2002, 43(6): 1657-1669.
张刚, 冯婕. 细菌固有耐药的研究进展[J]. 遗传, 2016, 38(10): 872-880.
Coyne S, Courvalin P, Périchon B. Efflux-mediated antibiotic resistance in Acinetobacter spp[J]. Antimicrob Agents Chemother, 2011, 55(1): 947-953.
Magnet S, Courvalin P, Lambert T. Resistance-nodulation-cell division-type efflux pump involved in aminoglycoside resistance in Acinetobacter baumannii strain BM4454[J]. Antimicrob Agents Chemother, 2001, 45(12): 3375-3380.
Wong E W, Yusof M Y, Mansor M B, et al. Disruption of adeB gene has a greater effect on resistance to meropenems than adeA gene in Acinetobacter spp. isolated from University Malaya Medical Centre[J]. Singapore Med J, 2009, 50(8): 822-826.
Blanc V, Salah-Bey K, Folcher M, et al. Molecular characterization and transcriptional analysis of a multidrug resistance gene cloned from the pristinamycin-producing organism, Streptomyces pristinaespiralis[J]. Mol Microbiol, 1995, 17(5): 989-999.
Brown M H, Paulsen I T, Skurray R A. The multidrug efflux protein NorM is a prototype of a new family of transporters[J]. Mol Microbiol, 1999, 31(1): 394-395.
Bay D C, Rommens K L, Turner R J. Small multidrug resistance proteins: A multidrug transporter family that continues to grow[J]. Biochim Biophys Acta, 2008, 1778(9): 1814-1838.
Schuldiner S. EmrE, a model for studying evolution and mechanism of ion-coupled transporters[J]. Biochim Biophys Acta, 2009, 1794(5): 748-762.
Li X Z, Nikaido H. Efflux-mediated drug resistance in bacteria: An update[J]. Drugs, 2009, 69(12): 1555-1623.
Brunson D N, Maldosevic E, Velez A, et al. Porin loss in Klebsiella pneumoniae clinical isolates impacts production of virulence factors and survival within macrophages[J]. Int J Med Microbiol, 2019, 309(3-4): 213-224.
Srinivasan V B, Venkataramaiah M, Mondal A, et al. Functional characterization of a novel outer membrane porin KpnO, regulated by PhoBR two-component system in Klebsiella pneumoniae NTUH-K2044[J]. PLoS One, 2012, 7(7): e41505.
Li Y, Yang H, Zou X, et al. Analysis of the CYP2C19 genetic polymorphism in Han and Uyghur patients with cardiovascular and cerebrovascular diseases in the Kashi area of Xinjiang[J]. Med Sci Monit, 2014, 20: 2213-2218.
Chambers J R, Sauer K. The MerR-like regulator BrlR impairs Pseudomonas aeruginosa biofilm tolerance to colistin by repressing PhoPQ[J]. J Bacteriol, 2013, 195(20): 4678-4688.
Brunson D N, Maldosevic E, Velez A, et al. Porin loss in Klebsiella pneumoniae clinical isolates impacts production of virulence factors and survival within macrophages[J]. Int J Med Microbiol, 2019, 309(3-4): 213-224.
Brooke J S, Di Bonaventura G, Berg G, et al. Editorial: A multidisciplinary look at Stenotrophomonas maltophilia: An emerging multi-drug-resistant global opportunistic pathogen[J]. Front Microbiol, 2017, 8: 1511.
Li J, Liu S, Fu J, et al. Co-occurrence of colistin and meropenem resistance determinants in a Stenotrophomonas strain isolated from sewage water[J]. Microb Drug Resist, 2019, 25(1): 317-325.
Guo R, Huang D, Ji J, et al. A novel mutation GJA8 NM_005267.5: c.124G > A, p.(E42K) causing congenital nuclear cataract[J]. BMC Ophthalmol, 2022, 22(1): 172.
Tenover F C. Antimicrobial Susceptibility Testing Methods for Bacterial Pathogens[J]. Antimicrob Drug Resist, 2018, 46: W296-W303.
Waterhouse A, Bertoni M, Bienert S, et al. SWISS-MODEL: homology modelling of protein structures and complexes[J]. Nucleic Acids Res,2018, 46(W1): W296-W303.
Little J L, Williams A J, Pshenichnov A, et al. Identification of “known unknowns” utilizing accurate mass data and ChemSpider[J]. J Am Soc Mass Spectrom, 2012, 23(1): 179-185.
Rampogu S, Baek A, Son M, et al. Discovery of lonafarnib-like compounds: Pharmacophore modeling and molecular dynamics studies[J]. ACS Omega, 2020, 5(4): 1773-1781.
Pasca M R, Guglierame P, De Rossi E, et al. mmpL7 gene of Mycobacterium tuberculosis is responsible for isoniazid efflux in Mycobacterium smegmatis[J]. Antimicrob Agents Chemother, 2005, 49(11): 4775-4777.
Delcour A H. Outer membrane permeability and antibiotic resistance[J]. Biochim Biophys Acta, 2009, 1794(5): 808-8016.
Modi N, Ganguly S, Bárcena-Uribarri I, et al. Structure, dynamics, and substrate specificity of the OprO porin from Pseudomonas aeruginosa[J]. Biophys J, 2015, 109(5): 1429-1438.
Moraes T F, Bains M, Hancock R E, et al. An arginine ladder in OprP mediates phosphate-specific transfer across the outer membrane[J]. Nat Struct Mol Biol, 2007, 14(1): 85-87.
Rahman T, Yarnall B, Doyle D A. Efflux drug transporters at the forefront of antimicrobial resistance[J]. Eur Biophys J, 2017, 46(1): 647-653.
顧觉奋. 外排泵抑制剂研究进展[J]. 国外医药(抗生素分册),2020, 41(1): 1-10.
