姜科基因组复杂性的比较基因组学研究
2023-08-19胡福博王希胤
胡福博,王希胤
(1.华北理工大学,河北 唐山 063210; 2.华北理工大学基因组学与计算生物学研究中心,河北 唐山 063210)
0 引言
姜科(Zingiberaceae)是单子叶植物姜目(Zingiberales)的一个重要分支,在生产生活中占据重要地位。其下属植物具有重要的经济价值及药用价值,如姜(Zingiberofficinale)、草果(Amomumtsao-ko)及砂仁(Wurfbainiavillosa),作为中药应用了几千年[1]。多倍化是物种进化与分歧的重要推动力[2]。研究表明,单子叶植物在进化过程中均经历了全基因组加倍(WGD,whole genome duplication),包括姜科在内的大部分单子叶植物共同拥有一次全基因加倍事件,即τWGD[3-5]。全基因组加倍导致的基因丢失、易位等现象对基因组结构及复杂性造成了重大影响[5-7]。
研究表明,姜在进化过程中从单子叶植物祖先到现在共经历了3次全基因组加倍事件,草果基因组在近期没有经历过全基因组加倍事件,砂仁基因组的研究表明,砂仁和姜的共同祖先可能经历了1次全基因组加倍事件。但现有研究对姜科植物古多倍化的认识还很模糊。随着单子叶植物基部物种的测序完成,为姜科植物在进化过程中经历的更古老加倍事件的研究提供了条件。
对菖蒲(Acorustatarinowii)基因组的研究表明,它只经历过1次全基因组加倍事件[8]。椰子(Cocosnucifera)在进化过程中除与姜科祖先物种共享了古老的τWGD以外,只单独经历了1次全基因组加倍事件,两者基因组相对保守。[4]这有助于进一步了解姜科植物基因组结构的复杂性。
1 材料与方法
1.1 物种基因组数据
姜基因组数据下载自公共数据库Genebank (Index of /genomes/genbank/plant/Zingiber_officinale/latest_assembly_versions (nih.gov)/GCA_018446385.1_Zo_v1.1/)。草果基因组数据来自国家基因库生命大数据平台CNGBdb(https://db.cngb.org/search/project/CNP0003772/)。砂仁基因组数据来自公共数据库Refseq(https://ftp.ncbi.nlm.nih.gov/genomes/refseq/plant/Elaeis_guineensis/all_assembly_versions/GCF_000442705.1_EG5/。菖蒲基因组数据来自中国国家基因库(https://ftp.cngb.org/pub/CNSA/data4/CNP0001708/CNS0456199/CNA0036157/)。编写python 脚本,将下载得到的原始数据进行数据预处理得到所需的注释文件(gff)、染色体长度文件(lens)、蛋白序列文件(pep)及蛋白编码序列文件(cds)。
1.2 系统发育及共线性基因推断
使用蛋白序列比对工具BLAST+[9],对研究物种蛋白序列(pep)文件进行种内及种间同源基因搜索(E-value<1e-5,score >100)。使用orthofinder 提取待研究物种的单拷贝基因,构建系发育物种树(见图1)。根据blast得到的结果,运行生信分析流程软件WGDI的“-d”模块,绘制基因组内及基因组间的同源基因点阵图[10],运用共线性分析软件ColinearScan,提取所研究物种基因组内及基因组之间的共线性基因对(共线性片段的基因对>=5个)[11]。
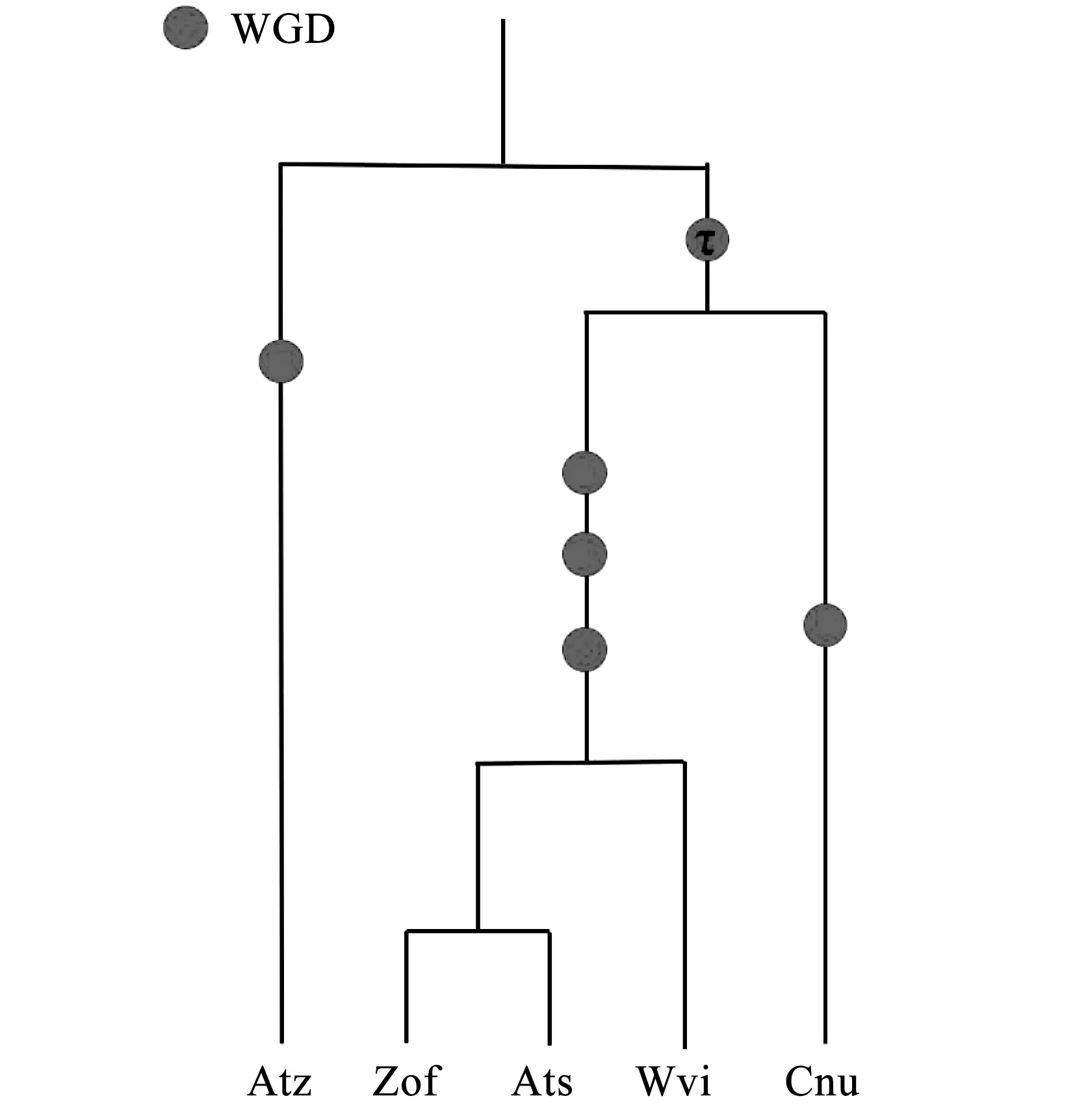
Ata代表菖蒲,Zof代表姜,Ats代表草果,Wvi代表砂仁,Cnu代表椰子图1 待研究植物系统发育树Fig.1 Plant phylogenetic tree to be studied
1.3 Ks计算
为了区分不同全基因组加倍事件产生的共线基因,计算了待研究物种间同源性基因对的核苷酸同义替代(Ks),以估计共线基因之间的分歧水平。利用clustalW将基因对的编码序列进行比对,调用PAML包的Nei-Gojobori的方法进行Ks分布计算[12]。利用WGDI对ks分布进行数学拟合,得到ks峰值。
1.4 染色体同源区域深度推断
编写Python脚本,将利用软件colinearscan提取的姜基因组与菖蒲同源的染色体片段投影到菖蒲染色体上,构建一个展示姜基因组染色体同源区域深度的列表,结合上述处理得到的染色体长度(lens)文件并运行软件WGDI的“-ci”模块将列表可视化。
2 结果分析
2.1 基因共线性分析
对共线性分析软件colinearscan获得的共线性片段进行分析,分别统计了菖蒲、姜、草果、砂仁的基因组内及基因组间的共线性基因对数量5个以上的共线性片段的共线性基因数(见表1)。姜基因组内共线性基因对有11 185对,砂仁有6815对,草果有668对。菖蒲与姜基因组间的共线性基因对有4809对,与砂仁有3323对,与草果有668对。同理,椰子与姜基因组间的共线性基因对有14 425对,与砂仁有10 189对,与草果有9935对。结果显示,姜基因组进化过程中的同源共线性基因对保留最多,砂仁次之,草果最少,故后续分析以姜作为主要研究对象,探究姜科基因组的复杂性。
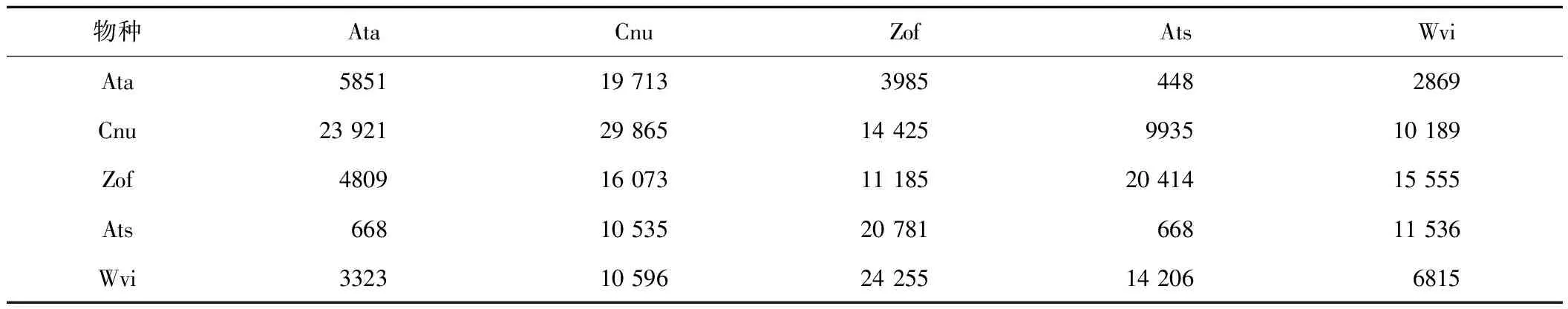
表1 同源基因统计
2.2 Ks分析
同义核苷酸替换(ks)是蛋白质编码序列的核苷酸变异不引起氨基酸改变[13]。有研究利用椰子基因组内共线性基因计算了ks分布并进行数字拟合,发现有两个明显的峰,认为ks峰值大的峰代表椰子经历的τWGD。提取了那部分共线性基因绘制了其ks柱状图,计算了姜、砂仁及菖蒲基因组内与组间共线性基因的ks分布并绘制ks 柱状图(见图2)。发现菖蒲和姜基因组间的共线性基因对的ks峰值为1.43,对应菖蒲和姜发生分歧的时间。姜和砂仁基因组间共线性基因对的ks峰值为0.36,对应姜和砂仁发生分歧的时间。椰子共线性基因对的ks峰值为0.99,对应椰子经历τWGD的时间。姜基因组内的同源共线性基因对的ks分布柱状图显示在0.36~1.43有很多小峰,推测在与菖蒲分歧之后,姜科各物种在发生分歧之前可能经历了包括τWGD在内至少4次全基因组加倍事件。

图2 菖蒲、椰子、姜、砂仁共线性基因同义核苷酸替换Fig.2 Collinear gene synonym nucleotide substitution of calamus, coconut,Zingiberaceae and amomum kernel
2.3 染色体同源区域深度推断
在一定程度上,染色体同源区域深度可以大致反映物种进化过程中经历的全基因组加倍次数。将姜与菖蒲基因组同源性区域映射到菖蒲染色体并绘制圈图(见图3)。结果显示,姜映射到菖蒲染色体上的同源区域深度最高达到13,由此推测,姜在进化过程中可能至少受到4次全基因组二倍乘事件的影响,越靠近外圈,姜的同源结构空白占比越大,代表同源基因丢失越多,说明姜经历的全基因组加倍事件次数较多,受古老的加倍事件影响产生的重复基因随物种进化而大量丢失。

外圈代表菖蒲映射的姜同源性基因图3 菖蒲映射姜的染色体同源片段深度Fig.3 Chromosomal homologous fragment depth of acorus mapping Zingiberaceae
2.4 基因组间同源基因点阵图
为探究姜科物种的基因组结构,绘制了菖蒲、椰子、姜等物种基因组间的同源基因点阵图。物种间的同源点阵图中的线性片段是由物种分化产生的同源基因片段。如姜与草果、砂仁的点图中(见图4),草果的2号染色体同源最好匹配姜的1号染色体,砂仁的2号染色体同源最好匹配姜的1号染色体,说明姜科祖先基因组在分化形成现存物种之后并没有再经历全基因组加倍事件。

点图中红点表示同源性最好,蓝点表示同源性次好,灰点表示同源性较差。图4 姜与草果、砂仁基因组间的同源点阵图Fig.4 Homologous dot map of the genome of Zingiberaceae, grass fruit and amomum kernel
菖蒲与姜的同源基因点图呈现的点非常散乱,不过部分菖蒲染色体对应的姜的同源片段数量显示大于8条(见图5)。椰子与姜的点图显示,椰子6号染色体对应姜的同源片段数量达到15~22条,再次说明姜基因组在进化过程中经历的多倍化事件至少有4次,甚至可能更多。至于椰子对应的姜的同源片段数量不同,可能是由于椰子受2次多倍化事件的影响,造成染色体基因的缺失。

图5 姜与菖蒲、椰子基因组间局部同源基因点阵图Fig.5 Local homologous gene dot map of Zingiberaceae, calamus and coconut genome
3 结论与讨论
以菖蒲为外类群,对姜科下属植物姜基因组进行比较研究,了解姜科经历的全基因组加倍事件可能多于3次,有可能是4次也可能更多,且姜科下属植物草果在进化过程中受多次加倍事件产生的重复基因大量丢失,导致其更为复杂的基因组结构,令人们对姜科基因组的深入挖掘难度增加。随着测序技术的不断更新换代,会有更多姜科乃至单子叶下属植物基因组完成更为精确的测序工作,有了更多精确的物种基因组数据做参考,会令姜科下属植物基因组复杂性结构的分析与挖掘得到长足进步,结果更为清晰、准确。生信分析软件及算法的开发,可为未来姜科基因组比较分析研究提供更强大的技术支撑。
