基于分子标记共祖的亚东鲑育种群体遗传背景分析
2023-07-31陈孟娟袁冬冬孙帅杰于光晴栾培贤王万良扎西拉姆周建设
陈孟娟,李 鑫,袁冬冬,孙帅杰,于光晴,户 国,栾培贤,王万良,扎西拉姆,周建设
(1.西藏自治区农牧科学院水产科学研究所,拉萨 850000;2.河南农业大学动物科技学院,郑州 450046;3.中国水产科学研究院黑龙江水产研究所,哈尔滨 150070)
亚东鲑(Salmotruttafario)又名褐鳟、棕鳟,属鲑形目鲑科鱼类,原产于欧洲、非洲北部和西亚一些地区,在19世纪初由英国引进,国内仅在西藏自治区亚东县内有分布,为西藏自治区特有的冷水经济鱼类[1,2]。该鱼周身有红色和灰色不规则圆点,鱼体肥满,肉质鲜美,其肌肉高度不饱和脂肪酸含量高并且具有丰富且平衡的氨基酸,营养价值较高[3],具有很大的商业价值。当地政府积极发展亚东鲑养殖业,现已成为亚东县重点发展的四大特色优势产业之一。但在亚东鲑产业化发展的过程也存在许多问题,如孵化率低、发病率高、体表色泽暗等,严重影响了养殖户的养殖成本和销售,亚东鲑由于引进时间较长[4],经过多代人工繁殖,缺乏人工选育,导致种质生长速度缓慢、个体小型化等现象时有发生。亚东鲑的良种化问题已成为制约其产业化发展的瓶颈。因此,无论亚东鲑作为商品化生产还是野外种质资源的养护需求,以其亲本遗传管理和建立选配策略解决亚东鲑良种化问题已成为当务之急。然而,因近亲繁殖导致的资源衰退,仅靠单纯数量意义上的增殖放流在经济、环境和增殖放流效果等方面不可持续,也不利于野生群体的种质资源多样性建立[5]。亚东鲑在无系谱背景的情况下,由于多代的人工繁殖缺乏实质性选育,如何避免近亲繁殖又成为一个难点。
分子标记共祖分析是通过高通量测序获得的SNP分子标记来替代传统系谱信息,结合多种统计学方法解析、分析群体遗传特征,计算如基因组共祖系数(genomic coancestry coefficient,CC)、血缘同源(identity by descent,IBD)、基因组近交系数(genomic inbreeding coefficient)、多维标度分析(multidimensional scaling,MDS)及群体遗传结构等估计值[6-8]。在没有系谱资料的育种群体中,通过分子标记共祖分析,获得个体间的亲缘关系信息,实现高效精准估算近交系数和群体近交率,进而从不同的角度揭示群体的近交关系,在水产动物育种中建立以维持物种多样性水平为保种目标的亲本选配策略。
本研究通过分子标记共祖分析,在无系谱信息的亚东鲑育种群体中,基于SNP分子标记替代系谱信息构建实现分子亲缘关系矩阵,精准估算个体间亲缘关系和群体近交率,以维持物种遗传多样性为目标,对亚东鲑育种群体遗传背景进行分析,为亚东鲑的家系选育及遗传多样性的管理提供新的技术方案。
1 材料和方法
1.1 样品采集
实验所用245尾亚东鲑均来自雅鲁藏布江渔业资源繁育基地,年龄为3~5龄,平均体长为(30.14±4.69)cm,平均体质量为(533.19±331.28)g。采样个体之间没有系谱记录。使用MS-222将采样个体进行麻醉后,采用PIT鱼类芯片标记的方法,将动物芯片打入背部肌肉进行标记,并剪取脂鳍放入90%的乙醇保存备用。
1.2 方法
1.2.1 样品DNA提取
利用无锡百泰克生物技术有限公司生产的海洋动物基因组DNA提取试剂盒提取亚东鲑脂鳍基因组DNA,通过琼脂糖检测和NanoDrop2000紫外分光光度计(Thermo fisher Scientific)对抽提的DNA质量和浓度进行检测。DNA产物置于-20 ℃保存备用。
1.2.2 文库构建和测序质量分析
按照 double digest restriction site-associated DNA sequencing(dd-RAD)的方式构建文库,参考基因组大小为2 371.88 Mb,选用的限制性内切酶为EcoRⅠ和BfaⅠ,插入片段在220~450 bp之间。利用第二代测序技术(Next-Generation Sequencing,NGS),基于Illumina NovaSeq测序平台,对文库进行双末端(Paired-end,PE)测序。得到的原始图像数据文件经碱基识别转化成原始测序序列,将拼接好的测序序列进行过滤以保证数据分析结果准确性。首先采用AdapterRemoval(version 2)[9]去除3′端的接头污染;随后进行质量过滤和长度过滤,过滤去除双末端长度≤50 bp的reads,由此获得可用于后续标记筛查、分型的高质量序列即SLAF标签。
1.2.3 全基因组序列比对和SNP标记检测
采用BWAmem[10]程序将过滤后得到的高质量数据比对到参考基因组上,均使用默认参数。采用GATK[11]软件进行SNP的检测,进一步对获得的SNP位点进行过滤,以获得高质量的SNPs用于遗传多样性分析。将获得的SNP结果进行统计,采用ANNOVAR[12]软件对SNP位点进行注释。
1.2.4 群体遗传结构分析
利用全基因组SNP标记对245个个体的遗传结构进行分析,设置K值为1-20个,迭代10 000次,通过ADMIXTURE[13]的5倍交叉验证来选择适宜的K值。使用PLINK软件进行多维标度(MDS)分析,以获得个体间的基因组关系。
1.2.5 群体遗传多样性分析
使用R软件包(DiveRsity)[14]估计群体内平均观测杂合度(Ho)和平均期望杂合度(He)。所有 SNPs的Ho和He值在群体内逐一计算,然后将平均值作为群体的Ho和He值。使用VCFtools[15]以10 Mb滑窗为单位,步长为2 Mb在整个基因组测量核苷酸多样性(Pi)。每个群体的平均Pi是由所有滑窗的Pi值的平均值计算出来的,使用R软件[16]对6个组别的均值进行多重比较,并进行Scheffe检验。
1.2.6 群体近交水平和亲缘关系
通过基因组共祖系数(CC)和血缘同源率(IBD)两个参数来衡量个体间的基因组亲缘关系。基因组共祖系数的估算是基于每个SNP自身效应的跨SNP标准化的混合模型获得的,并被计算为基因组成对加性关系的半值(基因组加性关系矩阵的非对角线元素),并由GVCBLUP软件[17]中的GCORRMX程序而得出,通过PLINK软件个体加性关系矩阵对角线元素计算个体近交系数(f)[18]。
1.2.7 群体遗传分化和基因流分析
通过VCFtools对全基因组以10 Mb滑窗为单位,长为2 Mb进行SNP扫描,估算群体间个体遗传分化指数(FST)[19],个体FST的显著性检验使用置换检验(10 000个置换)。通过高斯近似法从全基因组等位基因频率数据中发现遗传漂变,然后使用Treemix软件推断6个群体的历史分裂和混合的基因流。
2 结果
2.1 测序结果分析
对245个个体通过NGS测序,平均每个样本获得17.01 M clean reads,平均GC含量为42.28%,平均Q20百分比为97.84%,平均Q30碱基百分比率93.55%。平均测序深度为14.56,共获得1 092 509个SNP标记,经过筛选后获得17 058个SNP数据集,可用于后续的群体遗传分析。
2.2 群体遗传结构分析
群体结构是群体遗传分析的重要内容。本研究对245个个体进行Admixture检测分析,首先假设测试群体来源于1~20个祖先(K=1~20),交叉验证结果表明在K=11时错误率最低,其次为K=10。当K=10和11时,遗传聚类分析结果显示245个个体可明显地聚为6个群体(图1),其中StfA1、StfC2和StfC3群体中的遗传组成较为复杂,多数个体来源于不同祖先;StfB1、StfC1和StfC4群体遗传结构较为简单,多数个体具有单一的祖先来源(图1)。

图1 当K=10和11时,245个个体的遗传结构Fig.1 Genetic structure of 245 individuals when K=10 and 11
245个个体的多维标度(MDS)视图结果如图2a和图2b所示,遗传距离接近的群体会聚集在一起,从图中可以得到StfA1群体、StfB1群体各单独聚为一簇,并且与其它群体之间的遗传距离相距较远;StfC1、StfC2、StfC3、StfC4四个群体遗传距离较近,并且在二维图中4个群体间的个体有重叠部分(图2b)。

图2 245个个体的相互遗传距离多维标度分析(a,b)Fig.2 Multidimensional scaling analysis of mutual genetic distance of 245 individuals(a,b)
2.3 群体遗传多样性分析
分别统计了6个群体的Ho、He和Pi,结果显示(表1),6个群体的Ho范围为0.283~0.295,其中StfC3群体中遗传多样性最高,StfA1群体中遗传多样性最低。He范围为0.238~0.274,其中StfC2和StfC3群体遗传多样性相对更高,StfC1群体遗传多样性最低。Pi范围为1.658×10-6~1.884×10-6,He的趋势与Pi相似。
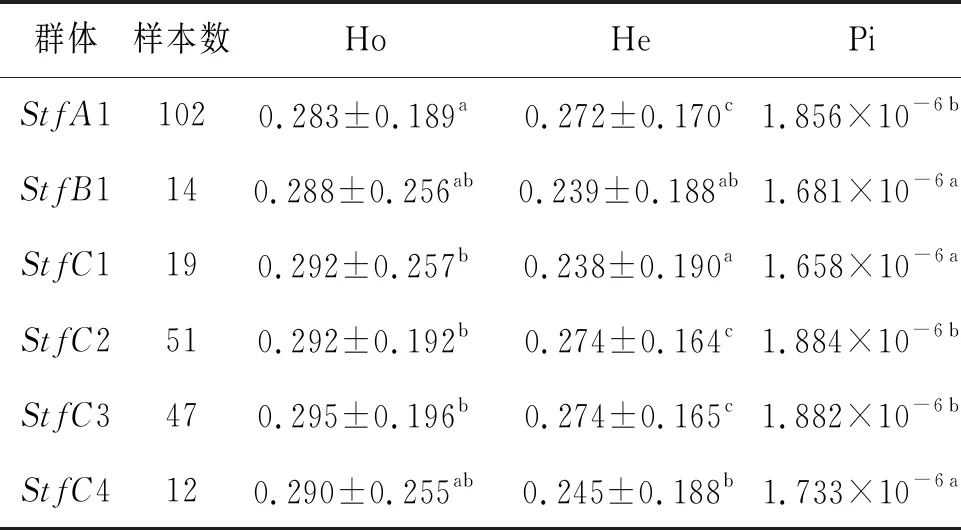
表1 6个群体的遗传多样性分析Tab.1 Genetic diversity among six sampling populations
2.4 群体近交水平估计
根据全基因组SNP数据估计的6个群体平均基因组近交系数如表2所示,StfA1群体基因组近交系数最高,平均基因组近交系数为0.046;StfC3群体基因组近交系数最低,平均基因组近交系数为0.007。StfC3群体的平均基因组近交系数显著小于StfA1群体,与其他4个群体比较差异不显著。
245个体中,基因组近交系数大于0.062 5的个体比例占所有检测个体的19.6%(48/245),其中StfA1群体为36.3%(37/102),StfC2群体为5.9%(3/51),StfC3群体为4.3%(2/47),StfC4群体为50%(6/12);基因组近交系数大于0.125的个体有14个,其中StfA1群体中12个,StfC3和StfC4群体中各有1个;基因组近交系数大于0.25的个体1个,在StfA1群体中(图3)。
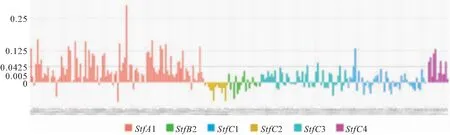
图3 245个个体的基因组近交系数Fig.3 Genomic inbreeding coefficients of the 245 individuals
2.5 群体内和群体间个体亲缘关系分析
群体内和群体间个体对亲缘关系可以通过CC和IBD来确定,并且可以通过两者的一致性来进行验证。六个群体内基因组平均成对亲缘关系见表2。StfC1群体中个体对的基因组亲缘关系最高(CC=0.169 5±0.043 7,IBD=0.346 4±0.086 7),StfC2和StfC3群体内个体对的基因组亲缘关系相对较远。群体内CC和IBD热图如图4所示,6个群体内共有115对个体为全同胞近交类型(CC≥0.25),近交程度属嫡亲,其中StfA1、StfB1、StfC1、StfC2、StfC3和StfC4群体内的个体对分别为55、1、12、22、12和13;958对个体为半同胞、祖孙或叔侄近交类型(0.125≤CC<0.25),近交程度属嫡亲,其中StfA1、StfB1、StfC1、StfC2、StfC3和StfC4群体内的个体对分别为487、36、142、176、85和32;3 653对个体为堂兄妹、半叔侄或曾祖孙近交类型(0.062 5≤CC<0.125),近交程度属近亲,其中StfA1、StfB1、StfC1、StfC2、StfC3和StfC4群体内的个体对分别为3 052、54、17、172、337和21。
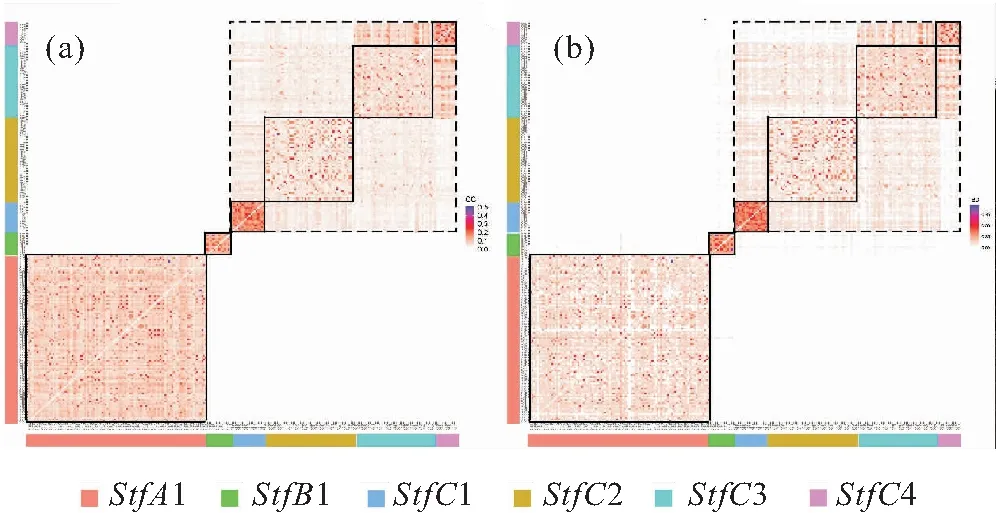
图4 245个个体间的基因组配对关系的概图Fig.4 The overview diagram of genomic pairwise relatedness between the 245 sequenced individualsa为基因组共祖系数(CC),b为血缘同源率(IBD)。
6个群体间个体亲缘关系如表3所示,无全同胞近交类型;半同胞、曾孙或叔侄近交类型的个体对为101对,其中StfC2和StfC3群体间28对,StfC3和StfC4群体间73对;堂兄妹、半叔侄或曾祖孙近交类型的个体对为584对,其中StfC1和StfC2群体间123对,StfC1和StfC3群体间45对,StfC2和StfC3群体间159对,StfC2和StfC4群体间3对,StfC3和StfC4群体间254对。不同群体间个体对基因组共祖系数有的无限接近零或为负数,血缘同源率分析结果除了无负数值出现外,与基因组共祖系数分析结果基本一致(图4b,表3)。
2.6 有效群体大小估算与基因流分析
6个群体间FST值在0.015~0.164之间(表4),其中StfA1群体与StfC1、StfC4群体之间的遗传分化指数较大,分别为0.164、0.160。BALLOUX等[20]以0.05、0.15和0.25为临界值将群体遗传分化划分为很小、中等、较大和很大4个等级。根据此划分依据,StfC3群体与StfC2、StfC4群体之间遗传分化程度很小(0≤FST≤0.05);StfA1群体与StfC1、StfC4群体之间表现出较大的分化水平(0.15≤FST≤0.25);其它种群之间表现为中等分化水平。
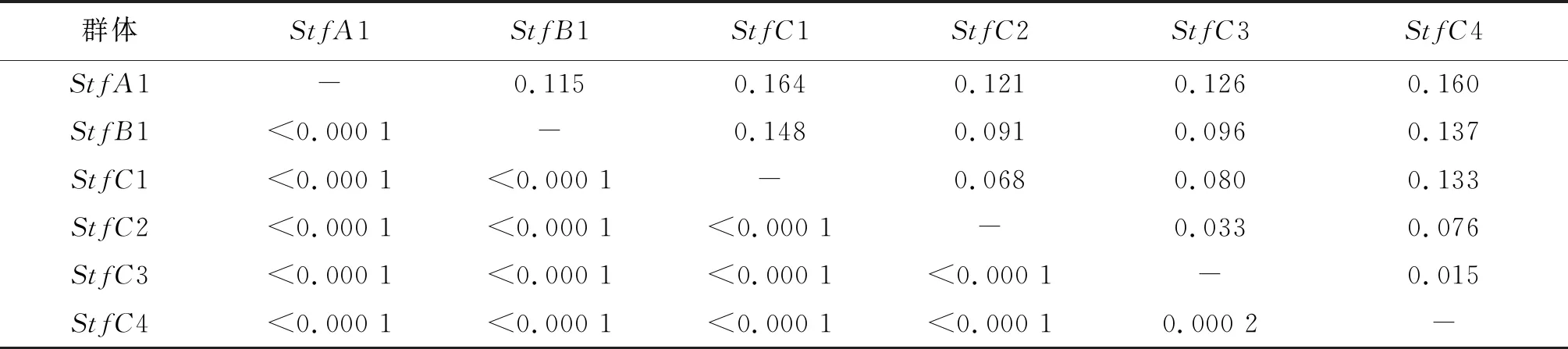
表4 群体间的成对FSTTab.4 Pairwise FST between populations
基因流结果显示,群体从StfC2群体分裂为两个分支,一个继续分裂为StfC1和StfC3群体,另一个继续分裂为两个子分支,一个分裂为StfC4群体,一个继续分裂为StfA1和StfB1群体。其中StfC3群体和StfC4群体基因交流最大,流向为StfC4群体到StfC3群体,其余各种群间也存在相当程度的基因交流(图5)。

图5 基因流分析和传播史推断Fig.5 Gene flow analysis and spread history inferring for the six populations黑线表示种群扩散的模式,带箭头的线表示推断的迁移方向。
3 讨论
3.1 亚东鲑繁殖群体遗传结构分析
群体遗传结构[21]指遗传变异在物种或群体中的一种非随机分布,按照地理分布或其他标准可将一个群体分为若干亚群,处于同一亚群内的不同个体亲缘关系较高,而亚群与亚群之间则亲缘关系稍远。亚东鲑最早是由英国引进养殖的[4],现已形成稳定的地方种群,并被农业农村部等八部门认定为第三批中国特色农产品。然而,由于引进时间长,未进行有效的人工选育,近亲繁殖导致的种质退化现象逐渐显现,户国等[22]对野生亚东鲑和褐鳟(Salmotrutta)日本品系群体进行个体亲缘关系聚类分析,结果显示野生型亚东鲑遗传结构较为简单,推测野生亚东鲑群体可能正经历遗传衰退的情况。亚东鲑群体系谱信息的缺失,使得人工选育工作不可避免地继续产生不同程度的近亲繁殖后代,使得选育周期延长。本研究基于分子标记共祖分析技术替代亚东鲑群体系谱信息,对雅鲁藏布江渔业资源繁育基地养殖的亚东鲑繁殖群体进行了遗传结构分析,结果显示,245尾亚东鲑明显地分为6个群体,从PCA主成分分析来看(图2a,图2b),6个群体中StfC1、StfC2、StfC3和StfC4群体间个体有掺杂的情况,说明这4个群体间遗传距离较近,在实际的育种应用中,避免这4个群体内个体间的选配,以防止近亲繁殖带来的种质退化。StfA1和StfB1两个群体在PCA主成分分析中分别单独聚为一簇,并与其他4个群体相聚较远,推断这2个群体个体间及与其他4个群体个体间遗传距离较远,在实际的亚东鲑育种应用中可以形成强杂种优势,为后代种质的提纯复壮提供育种基础。
3.2 亚东鲑繁殖群体遗传多样性分析
遗传多样性是优良性状选育的物质基础[23],表现在性状、控制性状位点的基因型在个体、群体或种的差异等方面,也是评价优良品种或品系的标准之一。本实验采用Ho、He和Pi遗传参数,从多个角度来估测亚东鲑繁殖群体的遗传多样性,Ho和He是衡量种群遗传变异程度的重要参数,与种群遗传多样性呈正相关[23]。实验结果显示6个群体的Ho范围为0.283±0.189~0.295±0.196,He范围为0.238±0.190~0.274±0.164,核苷酸多样性(Pi)范围为1.658×10-6~1.884×10-6,可以看出6个种群的平均观测杂合度均高于平均期望杂合度,群体的杂合度能够较准确反映群体遗传多样性,观测杂合度高一般情况下表明群体遗传多样性较丰富,王芳等[24]统计了日本品系褐鳟和亚东鲑He值分别为0.651 7和0.717 1,豪富华[25]发现亚东鲑野生群体的平均观测杂合度是0.708 3,而本研究中亚东鲑繁殖群体6个抽样群体的平均观测杂合度范围为0.283~0.295,均显著低于之前的研究结果,揭示了雅鲁藏布江鱼类资源繁育基地亚东鲑繁殖群体遗传多样性在下降,这与目前亚东鲑表现出来的生长速度慢、抗病能力低、体表色泽暗淡等均与种质退化相关,进一步说明目前该基地养殖的亚东鲑存在较严重的近亲繁殖现象。此外,6个群体的亚东鲑在绝大多数位点的Ho值都大于He值,提示在今后的人工选育过程中,从遗传多样性上适当增加群体的丰富性,避免遗传多样性的降低。
3.3 亚东鲑繁殖群体亲缘关系分析
近亲繁殖系数一定程度上反映了群体间亲缘关系远近和遗传变异程度。近亲繁殖系数越高,遗传距离越近,近交的程度越高。杂交亲本间遗传差异越大,亲缘关系越远,其后代杂种优势越明显[26]。基因组共祖系数(CC)和血缘同源率(IBD)是用来直接衡量个体间的亲缘关系的,用来反映近亲交配的程度。研究结果发现6个群体中,StfC1、StfC2、StfC3和StfC4四个群体(表3)个体间均达到了近亲繁殖的程度(0.031 25 遗传分化指数(FST)被认为是度量群体间基因分化相对大小的一个较好的指标,FST值高于0.15说明群体间存在显著性遗传分化[32]。本研究结果显示,StfA1群体与StfC1、StfC4群体间的遗传分化指数为0.164、0.160,遗传分化程度显著,说明StfA1群体与StfC1、StfC4群体间的遗传差异较大,其他群体间遗传分化程度都处于中等水平,说明群体间遗传差异处于中等及以上的程度。基因流是影响群体间遗传分化水平的重要因素,它能够减少群体间的遗传分化,使群体趋向于一致,当群体间基因流值大于1时,能够有效地抑制群体间遗传分化的产生[33]。在自然群体中,群体的分化差异程度与群体间的基因流大小紧密相关,即如果不同群体间的基因流强度越高,则该群体间的分化差异程度则相对越小[34]。本研究中6个群体的基因流均小于0.05,表明各群体间基因交流较少,产生了相应的遗传分化,这与FST的统计结果相一致。3.4 亚东鲑繁殖群体遗传分化分析
