关岭牛FABP1和FABP2基因克隆及其组织表达分析
2023-07-22石鹏飞许家利孙金魁许厚强
石鹏飞 许家利 孙金魁 许厚强

摘要:【目的】明確肝脏型脂肪酸结合蛋白(FABP1)和肠型脂肪酸结合蛋白(FABP2)基因的分子特征及其在不同年龄段和不同品种牛各组织中的表达情况,为后续研究FABP1和FABP2对牛脂肪酸的调控作用机制提供理论依据。【方法】采用RT-PCR扩增关岭牛FABP1和FABP2基因编码区(CDS)序列,通过ProtParam、ProtScale、SOPMA、SWISS-MODEL、TMHMM-2.0、SignalP-5.0和NetPhos-3.1等在线软件进行生物信息学分析,并以实时荧光定量PCR检测FABP1和FABP2基因在3日龄关岭牛(犊牛)、24月龄关岭牛(成年牛)及24月龄杂交牛(关岭牛×利木赞牛)各组织中的表达情况。【结果】FABP1、FABP2基因CDS序列全长分别为384和399 bp,对应编码127和132个氨基酸残基,其编码蛋白二、三级结构均以无规则卷曲和延伸链为主,无跨膜结构域和信号肽剪切位点,且属于亲水性稳定蛋白。FABP1、FABP2氨基酸序列分别包含19和18个磷酸化位点,其中苏氨酸在2个氨基酸序列中均广泛分布。FABP1、FABP2氨基酸序列同源比对及系统发育进化分析结果均显示,关岭牛与马、绵羊和山羊的亲缘关系较近,而与鸡和鸭的亲缘关系较远,与其他物种的相似性都在75.0%以上。FABP1、FABP2基因在不同年龄段和不同品种牛的各组织中均有表达,其中,FABP1基因以在肝脏和小肠中的相对表达量较高,且犊牛和杂交牛多个组织中的FABP1基因相对表达量极显著高于成年关岭牛(P<0.01,下同);FABP2基因则主要在小肠中高表达,且成年关岭牛小肠中的FABP2基因相对表达量极显著高于犊牛、显著高于杂交牛(P<0.05)。【结论】FABP1和FABP2基因在关岭牛中保守性较高,且在不同年龄阶段及不同组织中均有表达,尤其在肝脏和小肠中的表达水平较高,故推测FABP1和FABP2基因协同调控关岭牛脂肪酸的合成代谢。
关键词: 关岭牛;FABP1基因;FABP2基因;表达特征;脂肪酸
中图分类号:S823.81 文献标志码:A 文章编号:2095-1191(2023)02-0598-11
Abstract:【Objective】The molecular characteristics of liver-type fatty acid binding protein (FABP1) and intestinal-type fatty acid binding protein (FABP2) genes and their expression in various tissues of cattle of different ages and breeds were clarified to provide a theoretical basis for subsequent studies on the mechanisms of the regulatory effects of FABP1 and FABP2 on bovine fatty acids. 【Method】The sequences of the coding regions (CDS) of FABP1 and FABP2 genes in Guanling cattle were amplified by RT-PCR, and bioinformatics analysis was performed by online softwares such as ProtParam, ProtScale, SOPMA, SWISS-MODEL, TMHMM-2.0, SignalP-5.0 and NetPhos-3.1, and the expression of FABP1 and FABP2 genes in various tissues of 3-day-old Guanling cattle (calves), 24-month-old Guanling cattle (adults) and 24-month-old crossbred cattle (Guanling cattle × Limousin cattle) was examined by real-time fluorescence quantitative PCR. 【Result】The full-length CDS sequences of FABP1 and FABP2 genes were 384 and 399 bp, respectively, encoding 127 and 132 amino acid residues. The secondary and tertiary structures of their encoded proteins were dominated by random coil and extended chain without transmembrane structural domains and signal peptide shear sites, and they were hydrophilic stable proteins. The amino acid sequences of FABP1 and FABP2 contained 19 and 18 phosphorylation sites respectively, of which threonine was widely distributed in both amino acid sequences. Homology comparison and phylogenetic evolutionary analysis of the amino acid sequences of FABP1 and FABP2 showed that Guanling cattle were closely related to horses, sheep and goats, while they were more distantly related to chickens and ducks, and the similarities with other species were also above 75.0%. FABP1 and FABP2 genes were expressed in all tissues of cattle of different ages and breeds, among which, the relative expression of FABP1 gene was higher in liver and small intestine, and the relative expression of FABP1 gene in several tissues of calves and crossbred cattle was extremely significantly higher than that of adult Guanling cattle (P<0.01, the same below). The FABP2 gene was highly expressed mainly in the small intestine, and the relative expression of FABP2 gene in the small intestine of adult Guanling cattle was extremely significantly higher than that of calves and significantly higher than that of crossbred cattle (P<0.05). 【Conclusion】FABP1 and FABP2 genes are highly conserved in Guanling cattle and are expressed in different age stages and tissues, especially in the liver and small intestine, so it is hypothesized that FABP1 and FABP2 genes synergistically regulate fatty acid anabolism in Guanling cattle.
Key words: Guanling cattle; FABP1 gene; FABP2 gene; expression characteristics; fatty acid
Foundation items: National Natural Science Foundation of China (31571279); Ministry of Education Program to Promote Regional Research Cooperation and High-level Talent Training with U.S. University (〔2015〕2062)
0 引言
【研究意义】肝脏型脂肪酸结合蛋白(L-FABP或FABP1)和肠型脂肪酸结合蛋白(I-FABP或FABP2)同属于脂肪酸结合蛋白(Fatty acid binding protein,FABP)家族,该家族成员20%~70%的氨基酸序列表现出一致性,是一类与脂肪酸有高度亲和性的小分子蛋白(Chmurzyńska,2006;Storch and Thumser,2010)。FABPs能与长链脂肪酸结合,调节脂肪酸合成与分解的方式,进而调控细胞内的脂类代谢(Ono and Odani,2010)。因此,探究牛FABP1和FABP2基因的分子生物学特征及其组织表达特性,可为揭示FABP1和FABP2对脂肪酸的调控机制提供理论依据。【前人研究进展】长链脂肪酸作为代谢能源和生物膜的组成成分,在一些信号转导途径中发挥着重要作用(Schaap et al.,2002)。目前,已发现10多种类型的FABPs,其中FABP1是最早发现的FABPs(Chan et al.,1985)。FABP1调控细胞内脂质代谢和极低密度脂蛋白分泌,对脂肪酸的摄入及肝细胞中脂肪酸和胆固醇的稳态起重要作用(Newberry et al.,2003,2009)。与其他FABPs不同的是,FABP1不仅能与长链脂肪酸结合,还能与多种疏水配体结合,包括长链脂酰CoA、血红素、维生素K及胆固醇等(Ono and Odani,2010)。研究表明,敲除FABP1基因小鼠对脂肪酸的利用能力下降,但与长链脂酰CoA结合的相关蛋白表达量上升(Martin et al.,2003);而过表达FABP1基因能显著提高肝细胞对脂肪酸的摄取(Wu et al.,2016)、脂肪生成(Mukai et al.,2017)和极低密度脂蛋白分泌(Atshaves et al.,2010)。此外,在下调FABP1基因表达的小鼠中极低密度脂蛋白分泌减少,且能有效抑制小鼠脂肪肝的形成(Spann et al.,2006)。正常状态下,FABP1与脂肪酸的结合效率高于FABP2,但以饮食来源的脂肪酸为运输目标时FABP2与脂肪酸的结合效率高于FABP1,使其更有利于对食物中脂类物质的吸收和运输(Alpers et al.,2000),也说明FABP1和FABP2在脂肪酸的合成代谢过程中发挥着不同作用。FABP2能将细胞中的长链脂肪酸运送至内质网,合成三酰甘油,最后形成乳糜微粒并分泌到周圍的淋巴管中(Zimmerman et al.,2001)。Lackey等(2019)研究表明,敲除FABP2基因小鼠表现出肠道绒毛长度缩短、密度降低及肠道通透性增加,因此FABP2基因被认为是肠道完整性的重要标志基因。FABP1基因是调节家禽脂质代谢的重要候选基因,与过氧化物酶体增殖物激活受体α(Peroxisome proliferator activated receptor α,PPARα)协同调控脂肪酸的合成与分解,在肝脏和小肠的脂质代谢中发挥重要作用(He et al.,2013)。FABP1基因多态性与家禽肌内和腹部脂肪的沉积显著相关(He et al.,2012);FABP2基因多态性除了与鸡的腹脂重和腹脂率显著相关(王启贵,2004)外,还显著影响北京鸭的体尺和屠宰性状(廖秀冬等,2012)。综上所述,FABP1和FABP2基因可能是家禽脂肪性状的重要候选基因。【本研究切入点】目前,有关FABP1、FABP2基因的研究主要集中在人类和小鼠上,而针对牛FABP1和FABP2基因的生物功能及其表达特征尚未明确。【拟解决的关键问题】通过生物信息学分析关岭牛FABP1和FABP2基因的分子结构及其功能,运用实时荧光定量PCR检测这2个基因在不同年龄段和不同牛品种各组织中的表达差异,以期为后续研究FABP1和FABP2对牛脂肪酸的调控作用机制提供理论依据。
1 材料与方法
1. 1 试验材料
试验动物来自贵州省安顺市关岭县关岭牛产业园,随机选取相同饲养条件下的3日龄关岭牛(犊牛)、24月龄关岭牛(成年牛)及24月龄杂交牛(关岭牛×利木赞牛)各3头,采集心脏、肝脏、脾脏、肺脏、肾脏、背最长肌、脂肪及小肠等组织样品,-80 ℃保存备用。TRIzol购自美国Gibco公司;DL2000 DNA Marker、2×Taq PCR StarMix、逆转录试剂盒(Gen Star)购自西宝生物科技(上海)股份有限公司;荧光定量试剂Power UP SYBR GREEN Master Mix购自美国Thermo Fisher公司;胶回收试剂盒购自OMEGA公司;pMD19-T载体购自TaKaRa公司。主要仪器设备有电泳仪(DYY-2C,北京六一仪器厂)、超微量紫外分光光度计(NanoDrop 2000,美国Thermo Fisher公司)、普通PCR仪(C1000 Touch TM)、荧光PCR仪(Biosens SC710)及凝胶成像系统(Universal Hood Ⅱ,Bio-Rad)。
1. 2 引物设计与合成
根据NCBI已发布的牛FABP1基因序列(NM_175817.3)和FABP2基因序列(NM_001025332.1),使用Primer Premier 5.0设计编码区(CDS)扩增引物及荧光定量PCR特异性引物,综合选择得分最高的引物序列,委托北京擎科生物科技股份有限公司合成,引物序列信息见表1。
1. 3 组织总RNA提取及cDNA合成
采用TRIzol法提取各组织总RNA,以超微量分光光度计测定总RNA浓度及OD值;使用逆转录试剂盒(Gen Star)将符合条件的总RNA反转录合成cDNA第一链,-20 ℃保存备用。
1. 4 FABP1和FABP2基因克隆及测序分析
通过特异性引物扩增关岭牛FABP1和FABP2基因CDS区。PCR反应体系10.0 μL:cDNA模板0.4 μL,上、下游引物(10 μmol/L)各0.4 μL,2×Taq PCR StarMix 5.0 μL,ddH2O 3.8 μL。扩增程序:94 ℃预变性2 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 60 s/kb,进行35个循环;72 ℃延伸10 min。以1.0%琼脂糖凝胶电泳检测PCR扩增产物,使用胶回收试剂盒进行胶回收,纯化后的扩增产物与pMD19-T载体16 ℃连接30 min,然后转化DH5α感受态细胞,加入液体培养基培养1.5 h,再涂布至固体培养基上,37 ℃过夜培养,经氨苄青霉素筛选后挑选单个菌落进行扩大培养,PCR鉴定呈阳性的菌液送至北京擎科生物科技股份有限公司测序。
1. 5 FABP1和FABP2基因生物信息学分析
通过ProtParam和ProtScale分析FABP1、FABP2蛋白理化性质及亲/疏水性;采用SOPMA和SWISS-MODEL分别预测FABP1、FABP2蛋白二级结构及三级结构;利用TMHMM-2.0、SignalP-5.0和NetPhos-3.1预测FABP1、FABP2蛋白跨膜结构域、信号肽及磷酸化位点;使用MegAlign和MEGA 7.0分析FABP1、FABP2氨基酸序列相似性并构建系统发育进化树。
1. 6 实时荧光定量PCR检测FABP1和FABP2基因组织表达特征
以GAPDH为内参基因,通过实时荧光定量PCR检测犊牛、成年关岭牛及杂交牛各组织中的FABP1和FABP2基因表达情况。实时荧光定量PCR反应体系10.0 μL:Power UP SYBR GREEN Master Mix 5.0 μL,上、下游引物(10 μmol/L)各0.4 μL,cDNA模板0.5 μL,灭菌水3.7 μL。扩增程序:95 ℃预变性2 min;95 ℃ 15 s,60 ℃ 15 s,72 ℃ 60 s,进行39个循环。熔解曲线为仪器默认设置,每个样品设3个重复,获得完整光滑的熔解曲线,采用2-ΔΔCt法换算目的基因相对表达量。
2 结果与分析
2. 1 关岭牛FABP1和FABP2基因CDS序列扩增结果
以关岭牛cDNA为模板,扩增关岭牛FABP1和FABP2基因CDS序列,经1.0%琼脂糖凝胶电泳检测,结果显示扩增片段大小与预期结果相符(图1),初步判断成功扩增获得关岭牛FABP1和FABP2基因目的CDS序列。
2. 2 关岭牛FABP1和FABP2基因克隆及测序结果
菌液PCR鉴定结果显示成功获得关岭牛FABP1和FABP2基因的目的片段(图2),测序结果与NCBI已发布的牛FABP1和FABP2基因进行比对分析,结果(图3)发现,关岭牛FABP1基因CDS序列第273位核苷酸存在碱基突变(T→G),为同义突变,氨基酸序列未发生改变;关岭牛FABP2基因CDS序列与NCBI已发布的牛FABP2基因核苷酸序列完全一致,不存在碱基突变位点,表明pMD19-T-FABP1和pMD19-T-FABP2克隆载体构建成功。
2. 3 关岭牛FABP1和FABP2基因生物信息学分析结果
2. 3. 1 关岭牛FABP1和FABP2蛋白理化性质分析结果 ProtParam和ProtScale预测结果显示,关岭牛FABP1基因CDS序列长384 bp,共编码127个氨基酸残基,其蛋白分子式为C629H1009N165O195S7,原子数为2005,分子量为14227.34 Da,含量最高的氨基酸为赖氨酸(占12.6%),含量最低的为组氨酸(占0.8%)、半胱氨酸(占0.8%)和脯氨酸(占0.8%),理论等电点(pI)为7.78,蛋白不稳定指数为9.99,属于稳定蛋白。FABP1蛋白氨基酸序列的第55位甘氨酸疏水性最强(1.522)、第10位谷氨酰胺亲水性最强(-2.133),亲水性总体平均值为-0.402,即FABP1蛋白为亲水性蛋白(图4-A)。FABP2基因CDS序列长399 bp,共编码132个氨基酸残基,其蛋白分子式为C674H1065N179O202S4,原子数为2124,分子量为15036.19 Da,含量最高的氨基酸为谷氨酸(占10.6%)和赖氨酸(占10.6%),含量最低的为组氨酸(占0.8%),pI为6.62,蛋白不稳定指数20.27,属于稳定蛋白。FABP2蛋白氨基酸序列的第65位亮氨酸疏水性最强(2.000)、第14位天冬酰胺亲水性最强(-2.711),亲水性总体平均值为-0.414,也属于亲水性蛋白(图4-B)。
2. 3. 2 关岭牛FABP1和FABP2蛋白二、三级结构预测结果 通过SOMPA预测得知,FABP1蛋白二级结构由延伸链、无规则卷曲、α-螺旋和β-转角4种结构构成(图5-A),其中延伸链占比最高,为39.37%;FABP2蛋白同样由延伸链、无规则卷曲、α-螺旋和β-转角4种结构组成(图5-B),也是以延伸链占比最高(36.36%)。SWISS-MODEL预测得到的关岭牛FABP1、FABP2蛋白三級结构与对应的二级结构预测结果相符(图6)。
2. 3. 3 关岭牛FABP1和FABP2蛋白跨膜结构域、信号肽及磷酸化位点预测结果 通过TMHMM-2.0和SignalP-5.0分别预测关岭牛FABP1、FABP2蛋白的跨膜结构域及信号肽,结果显示二者均无跨膜结构域(图7)和信号肽剪切位点(图8)。NetPhos-3.1预测结果显示,关岭牛FABP1氨基酸序列共有19个磷酸化位点(图9-A),包括6个丝氨酸磷酸化位点、11个苏氨酸磷酸化位点和2个酪氨酸磷酸化位点;关岭牛FABP2氨基酸序列共有18个磷酸化位点(图9-B),包括5个丝氨酸磷酸化位点、9个苏氨酸磷酸化位点和4个酪氨酸磷酸化位点;其中苏氨酸磷酸化位点在2个氨基酸序列中均广泛分布。
2. 4 不同物种FABP1和FABP2氨基酸序列同源比对分析结果
对多个物种的FABP1和FABP2氨基酸序列进行同源比对分析,结果表明,关岭牛FABP1氨基酸序列与人类、小鼠、大鼠、马、猪、绵羊、山羊、鸡和鸭的FABP1氨基酸序列相似性分别为81.1%、76.4%、79.5%、96.1%、83.5%、97.6%、95.3%、67.7%和67.7%(图10-A);关岭牛FABP2氨基酸序列与人类、小鼠、大鼠、马、猪、绵羊、山羊、鸡和鸭的FABP2氨基酸序列相似性分别为84.1%、78.8%、82.6%、98.5%、87.9%、99.2%、99.2%、73.5%和73.5%(图10-B)。基于FABP1和FABP2氨基酸序列相似性,分别利用MEGA 7.0构建系统发育进化树(图11),结果均显示关岭牛与马、绵羊和山羊的亲缘关系较近,而与鸡和鸭的亲缘关系较远。
2. 5 FABP1和FABP2基因在犊牛、成年关岭牛和杂交牛不同组织中的表达情况
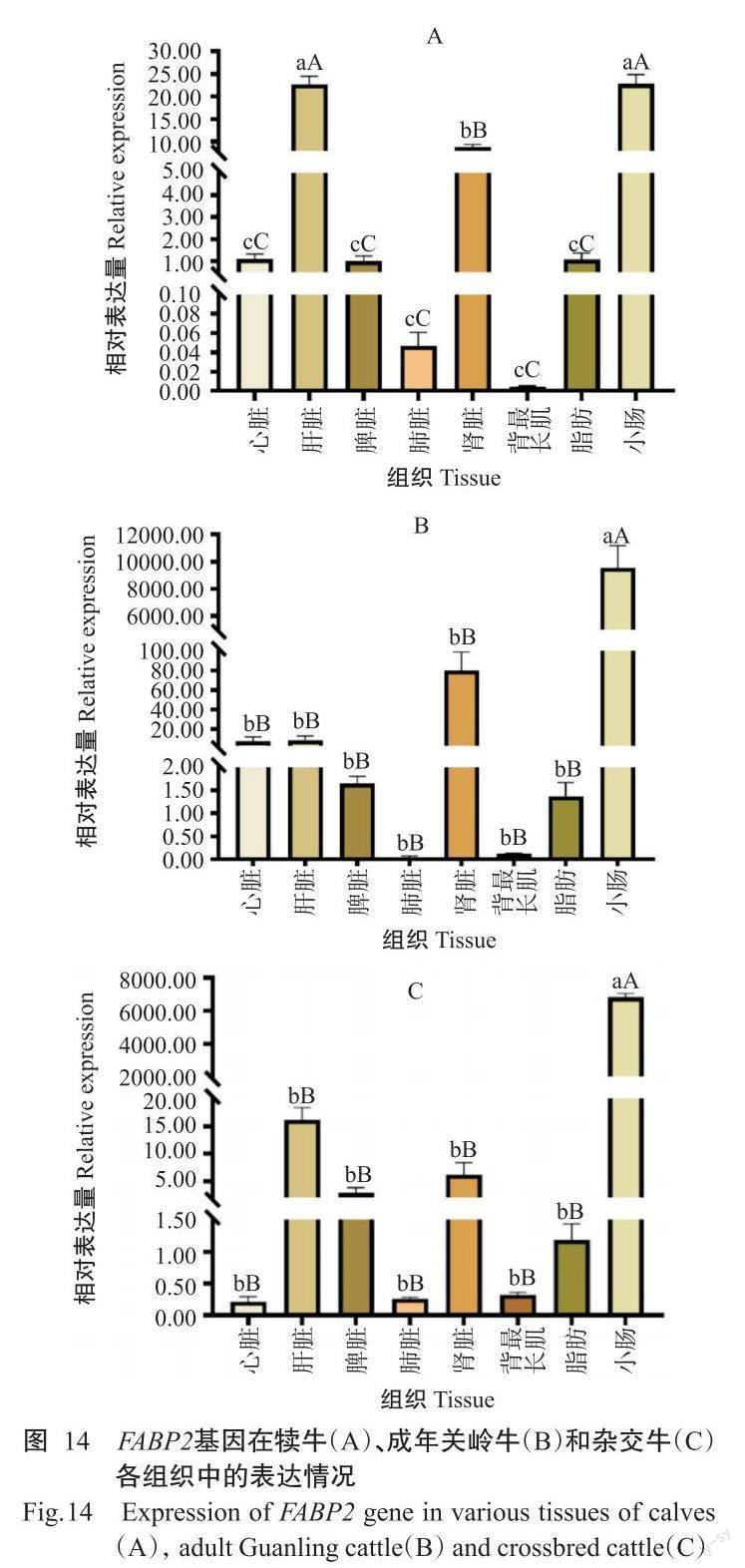
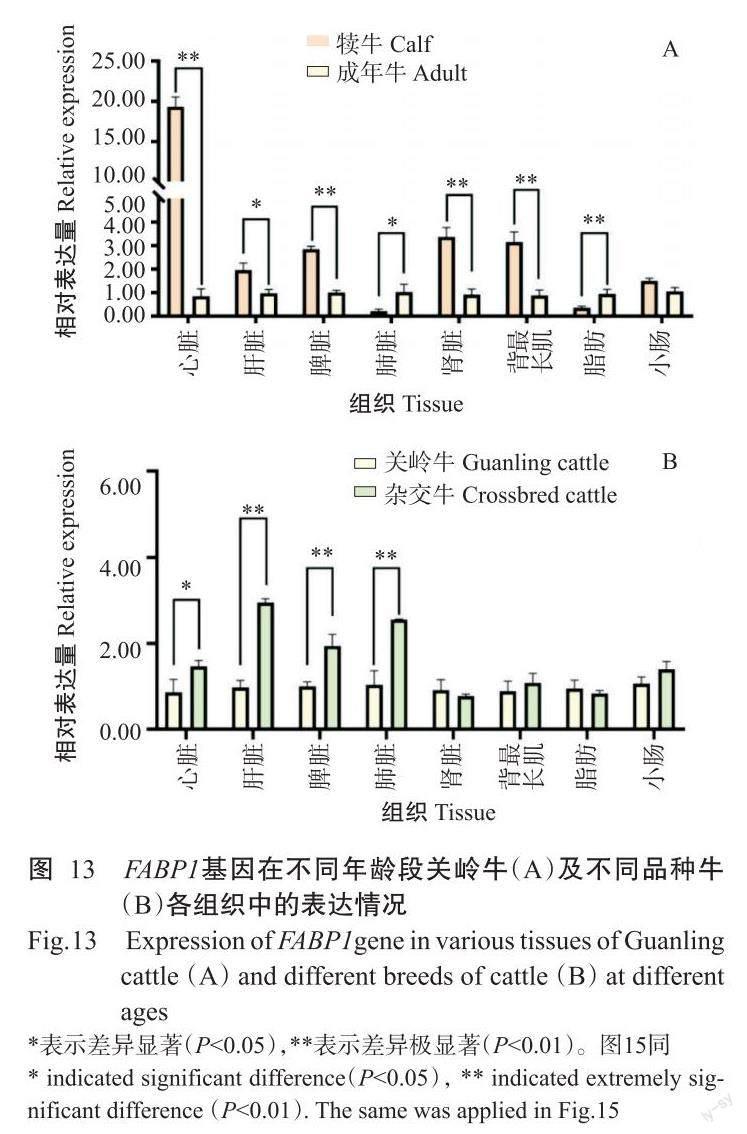
2. 5. 1 FABP1基因在犊牛、成年关岭牛和杂交牛不同组织中的表达情况 实时荧光定量PCR检测结果(图12)显示,FABP1基因在犊牛、成年关岭牛和杂交牛的不同组织中均有表达,且以肝脏中的相对表达量最高。FABP1基因在犊牛中的相对表达量排序为肝脏>小肠>肾脏>脂肪>脾脏>心脏>肺脏>背最长肌(图12-A),其中肝脏和小肠中的相对表达量极显著高于其他组织(P<0.01,下同);FABP1基因在成年关岭牛中的相对表达量排序為肝脏>小肠>脂肪>脾脏>肾脏>肺脏>心脏>背最长肌(图12-B),在肝脏和小肠中的相对表达量极显著高于其他组织;FABP1基因在杂交牛中的相对表达量排序为肝脏>小肠>脂肪>脾脏>肺脏>肾脏>心脏>背最长肌(图12-C),在肝脏和小肠中的相对表达量也极显著高于其他组织。此外,FABP1基因在犊牛心脏、脾脏、肾脏和背最长肌中的相对表达量极显著高于成年关岭牛,而在杂交牛肝脏、脾脏和肺脏中的相对表达量极显著高于关岭牛(图13)。
2. 5. 2 FABP2基因在犊牛、成年关岭牛和杂交牛不同组织中的表达情况 实时荧光定量PCR检测结果(图14)显示,FABP2基因在犊牛、成年关岭牛和杂交牛的不同组织中均有表达,且以小肠中的相对表达量最高。FABP2基因在犊牛中的相对表达量排序为小肠>肝脏>肾脏>心脏>脂肪>脾脏>肺脏>背最长肌(图14-A),在小肠和肝脏中的相对表达量极显著高于其他组织;FABP2基因在成年关岭牛中的相对表达量排序为小肠>肾脏>肝脏>心脏>脾脏>脂肪>背最长肌>肺脏(图14-B),在杂交牛中的相对表达量排序为小肠>肝脏>肾脏>脾脏>脂肪>背最长肌>肺脏>心脏(图14-C),FABP2基因在成年关岭牛和杂交牛小肠中的相对表达量均极显著高于其他组织。此外,FABP2基因在犊牛肝脏、肺脏和脂肪中的相对表达量显著高于成年关岭牛,但在小肠和背最长肌中表现为成年关岭牛极显著高于犊牛;FABP2基因在关岭牛心脏、脾脏、肾脏和脂肪中的相对表达量极显著高于杂交牛(图15)。
3 讨论
FABP1是FABP家族的重要成员之一,不仅可调控脂肪酸代谢,还影响细胞的生长。在细胞有丝分裂各时期,FABP1基因表达水平显著上调,且在切除部分肝脏组织后的再生过程中FABP1基因表达也呈显著上调趋势(Wang et al.,2004)。此外,FABP1对细胞具有保护作用,正常状态下可结合一些潜在的有毒脂肪酸、血红素及其他分子,解除结合即造成严重的细胞毒性(Wang et al.,2015)。在人类、猕猴及其他哺乳动物中,FABP2基因特异性表达于营养物质的主要吸收场所——小肠,与FABP2对食物中长链脂肪酸的吸收、转运及脂类物质代谢密切相关(Gajda and Storch,2015)。Baier等(1995)还研究证实,人类FABP2基因多态性与高血脂、糖尿病和肥胖症等疾病显著相关。目前,有关FABP1和FABP2基因的研究主要涉及人类的疾病及其免疫,在家畜上尤其是针对牛的研究鲜见报道。
本研究成功克隆出关岭牛FABP1和FABP2基因CDS序列,与NCBI已公布的相应序列对比分析发现,关岭牛FABP1基因核苷酸序列存在突变位点,但属于同义突变,不改变氨基酸序列,关岭牛FABP2基因CDS序列与NCBI已发布的牛FABP2基因核苷酸序列完全一致,表明FABP1和FABP2基因在关岭牛上遗传较稳定;FABP1和FABP2蛋白二、三级结构主要由延伸链和无规则卷曲构成,无规则卷曲是蛋白肽链中与配体和受体结合的重要部分(赵采芹等,2022),可能是FABP1和FABP2蛋白能有效结合并运输脂肪酸的原因之一。关岭牛FABP1、FABP2蛋白均无跨膜结构域和信号肽剪切位点,故推测二者不属于分泌蛋白。基于FABP1和FABP2氨基酸序列相似性构建的系统发育进化树均显示,关岭牛与马、绵羊和山羊的亲缘关系较近,而与鸡和鸭的亲缘关系较远,与其他物种的相似性都在75.0%以上。可见,关岭牛FABP1和FABP2基因保守性较高。
实时荧光定量PCR检测结果表明,FABP1、FABP2基因在关岭牛和杂交牛各组织中均有不同程度的表达,FABP1基因以在肝脏和小肠中的相对表达量较高,FABP2基因则主要在小肠中高表达,与姜延志和李学伟(2006)、Wang等(2019)的研究结果一致。肝脏和小肠是脂肪酸合成、运输的重要部位,调控机体的脂质代谢而影响肌内脂肪含量沉积,与肉品质密切相关(Jensen et al.,1990;Hunter,2001)。已有研究表明, FABP1基因启动子区含有过氧化物酶体增殖物受体元件,转录因子PPARα与之结合后调控FABP1基因表达(Venkatachalam et al.,2017),在肝脏和小肠中PPARα也呈丰富表达(Nakamura et al.,2014)。FABP1基因在犊牛多个组织(心脏、脾脏、肾脏和背最长肌)中的相对表达量极显著高于成年关岭牛,暗示着FABP1基因在关岭牛的早期生长发育过程中发挥重要作用。FABP1基因在杂交牛肝脏、脾脏和肺脏中的相对表达量极显著高于成年关岭牛,可能是FABP1基因对杂交牛的影响大于关岭牛。与成年关岭牛相比,FABP2基因在犊牛肝脏、肺脏和脂肪中的相对表达量极显著高于成年关岭牛,与FABP1基因在关岭牛中的表达情况相似;在成年关岭牛心脏、脾脏、肾脏和脂肪中的FABP2基因相对表达量极显著高于杂交牛,故推测FABP2基因对关岭牛的影响大于杂交牛。该结论为进一步揭示FABP1和FABP2基因对脂肪酸的调控机制提供了理论依据。
4 结论
FABP1和FABP2基因在关岭牛中保守性较高,且在不同年龄阶段及不同组织中均有表达,尤其在肝脏和小肠中的表达水平较高,故推测FABP1和FABP2基因协同调控关岭牛脂肪酸的合成代谢。
参考文献:
姜延志,李学伟. 2006. 猪I-FABP基因的分子克隆与组织特异性表达分析(英文)[J]. 遺传学报,33(2):125-132. [Jiang Y Z,Li X W. 2006. Molecular cloning and tissue-specific expression of intestinal-type fatty acid binding protein in porcine[J]. Acta Genetica Sinica,33(2):125-132.] doi:10.1016/S0379-4172(06)60031-0.
廖秀冬,任立明,王光瑛,李昂,黄银花. 2012. 北京鸭FABP2基因多态性与体尺和屠体性状的相关性研究[J]. 中国家禽,34(17):23-26. [Liao X D,Ren L M,Wang G Y,Li A,Huang Y H. 2012. Polymorphism of FABP2 gene and its association with body size and carcass traits in Peking duck[J].China Poultry,34(17):23-26.] doi:10. 16372/j.issn.1004-6364.2012.17.006.
王启贵. 2004. 鸡FABP基因克隆、表达特性及功能研究[D]. 哈尔滨:东北农业大学. [Wang Q G. 2004. Cloning,expression and function of chicken FABP genes[D]. Harbin:Northeast Agricultural University.] doi:10.7666/d.y631339.
赵采芹,王燕碧,张福平,唐宏,周磊,韩一帆,段志强. 2022. 鸡TRAF6和TIFA基因克隆及其组织表达特征分析[J]. 南方农业学报,53(2):546-556. [Zhao C Q,Wang Y B,Zhang F P,Tang H,Zhou L,Han Y F,Duan Z Q. 2022. Cloning and analysis tissue expression of chicken TRAF6 and TIFA genes[J]. Journal of Southern Agriculture,53(2):546-556.] doi:10.3969/j.issn.2095-1191.2022.02.028.
Alpers D H,Bass N M,Engle M J,DeSchryver-Kecskemeti K. 2000. Intestinal fatty acid binding protein may favor differential apical fatty acid binding in the intestine[J]. Biochimica et Biophysica Acta,1483(3):352-362. doi:10.1016/s1388-1981(99)00200-0.
Atshaves B P,Martin G G,Hostetler H A,McIntosh A L,Kier A B,Schroeder F. 2010. Liver fatty acid-binding protein and obesity[J]. The Journal of Nutritional Biochemistry,21(11):1015-1032. doi:10.1016/j.jnutbio.2010.01.005.
Baier L J,Sacchettini J C,Knowler W C,Eads J,Paolisso G,Tataranni P A,Mochizuki H,Bennett P H,Bogardus C,Prochazka M. 1995. An amino acid substitution in the human intestinal fatty acid binding protein is associated with increased fatty acid binding, increased fat oxidation,and insulin resistance[J]. The Journal of Clinical Investigation,95(3):1281-1287. doi:10.1172/JCI117778.
Chan L,Wei C F,Li W H,Yang C Y,Ratner P,Pownall H,Gotto Jr A M,Smith L C. 1985. Human liver fatty acid binding protein cDNA and amino acid sequence. Functional and evolutionary implications[J]. The Journal of Biological Chemistry,260(5):2629-2632.
Chmurzyńska A. 2006. The multigene family of fatty acid-binding proteins (FABPs):Function,structure and polymorphism[J]. Journal of Applied Genetics,47(1):39-48. doi:10.1007/BF03194597.
Gajda A M,Storch J. 2015. Enterocyte fatty acid-binding proteins (FABPs):Different functions of liver and intestinal FABPs in the intestine[J]. Prostaglandins Leukotrienes & Essential Fatty Acids,93:9-16. doi:10.1016/j.plefa. 2014.10.001.
He J,Chen J C,Lu L Z,Tian Y,Tao Z G,Wang D Q,Li J J,Li G Q,Shen J D,Fu Y,Niu D. 2012. A novel SNP of liver-type fatty acid-binding protein gene in duck and its associations with the intramuscular fat[J]. Molecular Bio-logy Reports,39(2):1073-1077. doi:10.1007/s11033-011-0833-z.
He J,Tian Y,Li J J,Shen J D,Tao Z G,Fu Y,Niu D,Lu L Z. 2013. Expression pattern of L-FABP gene in different tissues and its regulation of fat metabolism-related genes in duck[J]. Molecular Biology Reports,40(1):189-195. doi:10.1007/s11033-012-2048-3.
Hunter J E. 2001. Studies on effects of dietary fatty acids as related to their position on triglycerides[J]. Lipids,36(7):655-668. doi:10.1007/s11745-001-0770-0.
Jensen G L,Mascioli E A,Seidner D L,Istfan N W,Domnitch A M,Selleck K,Babayan V K,Blackburn G L,Bistrian B R. 1990. Parenteral infusion of long-and medium-chain triglycerides and reticuloendothelial system function in man[J]. Journal of Parenteral and Enteral Nutrition,14(5):467-471. doi:10.1177/0148607190014005467.
Lackey A I,Chen T N,Zhou Y X,Bottasso Arias N M,Gajda A M,Joseph L B,Storch J. 2019. Altered small intestinal morphology and whole-body energy homeostasis in the intestinal fatty acid binding protein (IFABP) knockout mouse[J]. The FASEB Journal,33(S1):583.5. doi:10. 1096/fasebj.2019.33.1_supplement.583.5.
Martin G G,Danneberg H,Kumar L S,Atshaves B P,Erol E,Bader M,Schroeder F,Binas B. 2003. Decreased liver fatty acid binding capacity and altered liver lipid distribution in mice lacking the liver fatty acid-binding protein gene[J]. Journal of Biological Chemistry,278(24):21429-21438. doi:10.1074/jbc.M300287200.
Mukai T,Egawa M,Takeuchi T,Yamashita H,Kusudo T. 2017. Silencing of FABP1 ameliorates hepatic steatosis,inflammation,and oxidative stress in mice with nonalcoholic fatty liver disease[J]. FEBS OpenBio,7(7):1009-1016. doi:10.1002/2211-5463.12240.
Nakamura M T,Yudell B E,Loor J J. 2014. Regulation of energy metabolism by long-chain fatty acids[J]. Progress in Lipid Research,53:124-144. doi:10.1016/j.plipres.2013. 12.001.
Newberry E P,Kennedy S M,Xie Y,Luo J Y,Davidson N O. 2009. Diet-induced alterations in intestinal and extrahepatic lipid metabolism in liver fatty acid binding protein knockout mice[J]. Molecular and Cellular Biochemi-stry,326:79-86. doi:10.1007/s11010-008-0002-4.
Newberry E P,Xie Y,Kennedy S M,Han X L,Buhman K K,Luo J Y,Gross R W,Davidson N O. 2003. Decreased hepatic triglyceride accumulation and altered fatty acid uptake in mice with deletion of the liver fatty acid-binding protein gene[J]. Journal of Biological Chemistry,278(51):51664-51672. doi:10.1074/jbc.M309377200.
Ono T,Odani S. 2010. Initial studies of the cytoplasmic FABP superfamily[J]. Proceedings of the Japan Academy. Series B:Physical and Biological Sciences,86(3):220-228. doi:10.2183/pjab.86.220.
Schaap F G,van der Vusse G J,Glatz J F C. 2002. Evolution of the family of intracellular lipid binding proteins in vertebrates[J]. Molecular & Cellular Biochemistry,239(1-2):69-77. doi:10.1023/a:1020519011939.
Spann N J,Kang S,Li A C,Chen A Z,Newberry E P,Davidson N O,Hui S T Y,Davis R A. 2006. Coordinate transcriptional repression of liver fatty acid-binding protein and microsomal triglyceride transfer protein blocks hepa-tic very low density lipoprotein secretion without hepatosteatosis[J]. Journal of Biological Chemistry,281(44):33066-33077. doi:10.1074/jbc.M607148200.
Storch J,Thumser A E. 2010. Tissue-specific functions in the fatty acid-binding protein family[J]. Journal of Biological Chemistry,285(43):32679-32683. doi:10.1074/jbc.R110.135210.
Venkatachalam A B,Parmar M B,Wright J M. 2017. Evolution of the duplicated intracellular lipid-binding protein genes of teleost fishes[J]. Molecular Genetics and Geno-mics,292(4):699-727. doi:10.1007/s00438-017-1313-5.
Wang G Q,Bonkovsky H L,de Lemos A,Burczynski F J. 2015. Recent insights into the biological functions of liver fatty acid binding protein 1[J]. Journal of Lipid Research,56(12):2238-2247. doi:10.1194/jlr.R056705.
Wang G Q,Chen Q M,Minuk G Y,Gong Y W,Burczynski F J. 2004. Enhanced expression of cytosolic fatty acid bin-ding protein and fatty acid uptake during liver regeneration in rats[J]. Molecular and Cellular Biochemistry,262:41-49. doi:10.1023/b:mcbi.0000038214.52184.82.
Wang Y J,Tang K Q,Zhang W,Guo W L,Wang Y N,Zan L S,Yang W C. 2019. Fatty acid-binding protein 1 increa-ses steer fat deposition by facilitating the synthesis and secretion of triacylglycerol in liver[J]. PLoS One,14(4):e0214144. doi:10.1371/journal.pone.0214144.
Wu Y L,Peng X E,Zhu Y B,Yan X L,Chen W N,Lin X. 2016. Hepatitis B virus X protein induces hepatic steatosis by enhancing the expression of liver fatty acid bin-ding protein[J].Journal of Virology,90(4):1729-1740. doi:10.1128/JVI.02604-15.
Zimmerman A W,van Moerkerk H T,Veerkamp J H. 2001. Ligand specificity and conformational stability of human fatty acid-bindingproteins[J]. The International Journal of Biochemistry and Cell Biology,33(9):865-876. doi:10.1016/s1357-2725(01)00070-x.
(責任编辑 陈德元)
